Биология внутренних вод, 2022, № 1, стр. 14-25
Филогеография куринского усача Barbus cyri De Filippi (Cyprinidae) по данным мтДНК
А. А. Гандлин 1, *, Б. Джапошвили 2, Г. Эпиташвили 2, Н. Дж. Мустафаев 3, А. Р. Рубенян 4, Б. А. Лёвин 1, 5
1 Институт биологии внутренних вод им. И.Д. Папанина Российской академии наук
пос. Борок, Некоузский р-н, Ярославская обл., Россия
2 Государственный университет Ильи, Институт зоологии
Тбилиси, Грузия
3 Азербайджанский государственный аграрный университет
Гянджа, Азербайджан
4 Институт гидроэкологии и ихтиологии Национальная академия наук Республики Армения
Ереван, Республика Армения
5 Череповецкий государственный университет
Череповец, Россия
* E-mail: gandlin.aleksander@yandex.ru
Поступила в редакцию 23.04.2021
После доработки 29.07.2021
Принята к публикации 06.08.2021
- EDN: JESIYX
- DOI: 10.31857/S0320965222010053
Аннотация
Представлено первое подробное исследование филогеографии куринского усача B. cyri (229 особей из 52 локальностей) с использованием двух молекулярных маркеров мтДНК – гена цитохрома b и контрольного региона. С помощью анализа сетей гаплотипов выявлена сложная генетическая структура популяций куринского усача, слабо коррелирующая с географией распространения этих популяций. Предполагается, что климатические флуктуации, колебания уровня Каспийского моря и тектоническая активность в регионе приводили к неоднократным вторичным контактам в сочетании с периодической изоляцией отдельных популяций усача (бассейны Ленкорани, Севана, Нижней Куры, южного побережья Каспия) в период плейстоцена–голоцена. Обнаруженная генетическая субизоляция популяций верхне-среднего течения р. Кура от популяций Нижней Куры, наряду с данными о высокой степени эндемизма ихтиофауны среднего течения Куры, позволяет высказать гипотезу о существовании в бассейне р. Кура локального рефугиума.
ВВЕДЕНИЕ
Усачи рода Barbus sensu stricto представляют собой линию эволюционных тетраплоидов в сем. Cyprinidae (2n=100) (Berrebi, Tsigenopoulos, 2003), обитающих в водоемах Европы, Кавказа и Ближнего Востока. Группа объединяет >30 видов и считается сложной в таксономическом и эволюционном отношениях. Исследования филогении и филогеографии европейских видов усачей обнаружили сложные паттерны их эволюционной истории, отображающие расселение из нескольких рефугиумов, существовавших в ледниковые периоды, наличие вторичных контактов и гибридизации (Kotlík, Berrebi, 2001; Lajbner et al., 2009; Meraner et al., 2013; Levin et al., 2019). Таким образом, группа Barbus представляет значительный интерес для эволюционной биологии и биогеографии. При этом кавказские усачи, представленные пятью видами – B. ciscaucasicus Kessler, 1877; B. cyri De Filippi, 1865; B. kubanicus Berg, 1912; B. rionicus Kamensky, 1899; B. tauricus Kessler, 1877, – являются слабо изученной группой (Levin, Rubenyan, 2006; Gandlin et al., 2017; Levin et al., 2019).
Куринский усач B. cyri – наиболее широко распространенный вид усачей на территории Кавказа – описан из р. Кура у г. Тбилиси. Его ареал охватывает пресные водоемы бассейна Каспийского моря как в Закавказье – реки Кура и Аракс (Грузия, Азербайджан, Армения, Турция), оз. Севан (Армения), так и водоемы Южного Прикаспия – бассейн оз. Урмия (Khaefi et al., 2018) и Закаспийского региона – р. Атрек в Иране (Bănărescu, Bogutskaya, 2003; Khaefi et al., 2017).
Систематическое положение куринского усача неоднократно менялось. Некоторые авторы синонимизировали B. cyri с B. lacerta Heckel, 1843 (Берг, 1949; Coad, 1995), описанного из р. Куэйк около г. Алеппо в Сирии, или считали его подвидом B. lacerta (Дадикян, 1986; Bănărescu, Bogutskaya, 2003). Другие авторы рассматривали его в качестве подвида B. cyclolepis Heckel, 1837 (Bianco, Bănărescu, 1982). В то же время, ряд описанных таксонов (B. toporovanicus Kamensky, 1899, B. sursunicus Kamensky, 1899, B. bortschalinicus Kamensky, 1899 и B. armenicus Kamensky, 1899) были синонимизированы с B. cyri (Берг, 1949), а B. goktschaicus Kessler, 1877 предложен к синонимизации (Levin et al., 2019). Дискуссия о таксономическом статусе куринского усача или его отдельных популяций, по-видимому, объясняется значительной морфологической изменчивостью широко распространенного вида (Абдурахманов, 1962; Лёвин, 2004). Ключевыми в понимании популяционной структуры вида и его таксономического статуса могут быть генетические данные, хотя сведений о генетическом полиморфизме куринского усача в литературе немного.
Цель настоящей работы – исследовать генетический полиморфизм куринского усача и реконструировать его филогеографию по результатам молекулярно-генетический анализа.
МАТЕРИАЛ И МЕТОДЫ ИССЛЕДОВАНИЯ
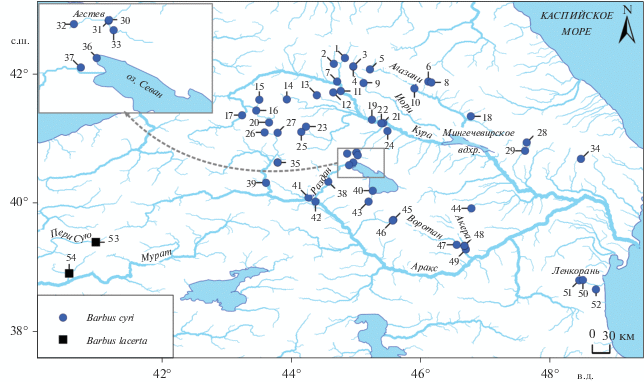
Для реконструкции эволюционной истории куринского усача и прояснения вопросов его филогеографии проведен молекулярно-генетический анализ 52 выборок, охватывающих бо́льшую часть ареала. Для сравнения в исследование также были взяты две выборки близкородственного куринскому усачу вида – Barbus lacerta. Материалом послужили коллекционные сборы Института биологии внутренних вод им. И.Д. Папанина РАН (ИБВВ РАН). Карта с местами сбора материала дана на рис. 1, географические координаты приведены в Дополнительном материале (табл. 1S).
Таблица 1.
Внутри- и межгрупповая генетическая изменчивость B. cyri и B. lacerta по последовательностям гена cyt b
| Группа | n/H | Hd ± SD | π ± SD | K | СВД ± SE | МВД | БГ | Дист. БГ ± SE |
|---|---|---|---|---|---|---|---|---|
| Barbus cyri | ||||||||
| Бассейн: р. Аракс | 27/10 | 0.843 ± 0.050 | 0.0024 ± 0.0002 | 2.4 | 0.0024 ± 0.0009 | 0.006 | оз. Севан | 0.003 ± 0.001 |
| р. Кура | 147/29 | 0.908 ± 0.012 | 0.0026 ± 0.0001 | 2.6 | 0.0026 ± 0.0007 | 0.008 | То же | 0.003 ± 0.001 |
| р. Ленкорань | 14/5 | 0.780 ± 0.085 | 0.0033 ± 0.0007 | 3.3 | 0.0033 ± 0.0011 | 0.007 | » | 0.003 ± 0.001 |
| оз. Севан | 17/9 | 0.882 ± 0.055 | 0.0024 ± 0.0003 | 2.4 | 0.0024 ± 0.0008 | 0.005 | р. Аракс | 0.003 ± 0.001 |
| Все бассейны | 205/44 | 0.938 ± 0.007 | 0.0028 ± 0.0001 | 2.7 | 0.0028 ± 0.0007 | 0.008 | – | – |
| Barbus lacerta | ||||||||
| Бассейн р. Евфрат | 11/7 | 0.909 ± 0.066 | 0.0039 ± 0.0004 | 3.9 | 0.0039 ± 0.0012 | 0.007 | р. Кура | 0.028 ± 0.005 |
Примечание. n/H – размер выборки/число гаплотипов; Hd ± SD – разнообразие гаплотипов ± стандартное отклонение; π – нуклеотидное разнообразие (на сайт); K – среднее количество нуклеотидных различий; СВД ± SE – средняя внутригрупповая p-дистанция ± стандартная ошибка; МВД – максимальная внутригрупповая p-дистанция; БГ – ближайшая группа; Дист. БГ – средняя p-дистанция до ближайшей группы (на основе 1000 реплик бутстрепа).
ДНК выделяли из плавников, фиксированных в 96%-ном этаноле, солевым методом (Aljanabi, Martinez, 1997). В дальнейшем работали с мтДНК, успешно используемой в изучении филогеографии рыб (Avise, 2000; Artaev et al., 2021). Исследовали последовательности фрагментов двух маркеров мтДНК – гена цитохрома b (cyt b) и контрольного региона (CR), которые хорошо себя зарекомендовали в филогеографических и филогенетических исследованиях усачей (Tsigenopoulos, Berrebi, 2000; Doadrio et al., 2002, Kotlík et al., 2004; Khaefi et al., 2018; Levin et al., 2019; Zaccara et al., 2019).
ПЦР проводили в реакционной смеси объемом 25 мкл [1× буфер, 1.5 мМ MgCl2, 10 мкМ каждого праймера, 0.2 мкМ каждого нуклеотидтрифосфата, 1 мкл ДНК-матрицы и 1 ед. Taq-полимеразы (Силекс, Москва)].
Амплификацию фрагмента гена цитохрома b (cyt b) проводили с праймерами GluDg 5′‑TGACTTGAARAACCAYCGTTG-3′ (Palumbi, 1996) и H16460 5′-CGAYCTTCGGATTACAAGACCG-3' (Perdices et al., 2002). В ряде случае использовали праймеры LA 5′-GCGACTTGAAAAACCACCGTT-3′ и CB2H 5′-CCCTCAGAATGATATTTGCCCTCA-3′ для восстановления нуклеотидной последовательности в начале 5′-конца гена цитохрома b (Espanhol et al., 2007). Для амплификации контрольного региона мтДНК (CR, согласно BLAST, амплифицированные фрагменты также включают 18 п.н. участка, кодирующего tRNA-Pro) были взяты праймеры DL623 5'-GGAATAGATATGTTATGCACTTG-3' (Levin et al., 2013) и LProf 5'‑AACTCTCACCCCTAGCTCCCAAAG-3' (Meyer et al., 1994), а также ESTFOR 5'-CATCGGTCTTGTAATCCGAAGAT-3' и NewCRev_AC 5'-GTTTAGGGGTTTGACAAGGATA-3', разработанные нами. ПЦР проводили согласно условиям, указанным в статье Лёвина и др. (Levin et al., 2012) для цитохрома b, и в статье Лёвина и др. (Levin et al., 2013) – для контрольного региона.
Полученные ПЦР-продукты визуализировали в 1.5%-ном агарозном геле, затем очищали ПЦР-продукт с использованием 96%-ного этанола и ацетата аммония в концентрации 3M. Секвенирование проводили на автоматическом секвенаторе ABI3500 в ИБВВ РАН в соответствии с инструкцией производителя. Гомологичные участки последовательностей проверены в программе FinchTV 1.4.0 (Rothgänger et al., 2005) и выравнены c использованием пакета программ MEGA7 (Tamura et al., 2007).
Сети гаплотипов строили с использованием программы PopART (Leigh, Bryant, 2015) при помощи алгоритма Median Joining (Bandelt et al., 1999). Генетическую дифференциацию между популяциями рассчитывали в Arlequin v. 3.5.2.2 (Excoffier, Lisсher, 2010) с использованием индекса фиксации FST (Weir, Cockerham, 1984). Нуклеотидное и гаплотипическое разнообразие, среднее количество нуклеотидных различий оценивали с помощью DnaSP v. 6 (Rozas et al., 2017). Расчет средней и максимальной внутригрупповой, а также средней межгрупповой p-дистанции проводили с помощью программы MEGA7 (Tamura et al., 2007).
Всего использовано 216 последовательностей гена cyt b длиною 993 п.н. B. cyri и B. lacerta. В это число входят 62 последовательности гена cyt b куринского усача, опубликованные в базе данных NCBI под номерами MK108197–MK108236, MK108254–MK108266 и MK108279–MK108287 из работы Лёвина и др. (Levin et al., 2019) и 154 последовательности (MZ547143–MZ547296), полученные в данном исследовании. Также впервые отсеквенированы 152 последовательности CR B. cyri длиною 652 п.н. (MZ547297–MZ547448). Кроме них, в качестве дополнительного материала использованы 68 последовательностей CR из базы данных NCBI под номерами MF599000–MF599067, опубликованные в работе Хаефи и др. (Khaefi et al., 2018). Таким образом, общее число анализируемых последовательностей CR достигало 220.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Филогеография куринского усача по данным последовательностей cyt b. Двести пять последовательностей гена cyt b куринского усача B. cyri из бассейнов рек Кура, Аракс, Ленкорань и бассейна оз. Севан относятся к 44 гаплотипам, в то время как 11 последовательностей сестринского вида B. lacerta из бассейна р. Евфрат представлены 7 гаплотипами (рис. 2, табл. 1). Сеть гаплотипов куринского усача характеризуется звездчатой структурой с гаплотипом 20 в центре и отходящими от него краевыми гаплотипами. Центральный гаплотип выявлен у особей трех бассейнов (рек Кура, Аракс и оз. Севан), остальные гаплотипы встречаются у представителей 1–2 бассейнов. В связи с этим разделение сети на отдельные гаплогруппы затруднительно. От центрального гаплотипа наиболее дистанцированными (на расстояние до пяти мутаций) оказались гаплотипы усача из р. Кура (гаплотипы 12, 37) и р. Ленкорань (гаплотипы 24 и 25) (рис. 2). Слабо дивергировавшие от центрального гаплотипа – гаплотипы усача из бассейнов р. Аракс и оз. Севан.
Рис. 2.
Сеть гаплотипов В. cyri и B. lacerta по 216 последовательностям гена cyt b мтДНК длиной 993 п.н. Число штрихов на отрезках, соединяющих гаплотипы, соответствует числу нуклеотидных замен; черные точки обозначают гипотетические гаплотипы.
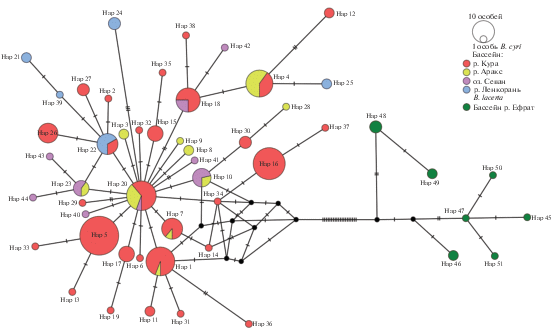
Наименьшее гаплотипическое разнообразие наблюдали в бассейне р. Ленкорань (Hd = 0.780), наибольшее – в бассейне р. Кура (Hd = 0.908) (табл. 1). Наименьшее нуклеотидное разнообразие обнаружено в бассейнах оз. Севан и р. Аракс (π = 0.0024), наибольшее – в бассейне р. Ленкорань (π = 0.0033). Среднее количество нуклеотидных различий и среднее генетическое расстояние между гаплотипами из бассейна р. Ленкорань также наибольшие (K = 3.3, СВД = 0.0033) по сравнению с другими бассейнами, что отражено и на гаплотипической сети (табл. 1, рис. 2).
Несмотря на небольшую выборку B. lacerta, его гаплотипическое разнообразие (Hd = 0.780) сравнимо или превышает таковое для B. cyri из бассейна р. Кура, имеющего самое высокое значение Hd среди всех бассейнов. Нуклеотидное разнообразие, средняя внутривидовая дистанция и среднее количество нуклеотидных различий B. lacerta существенно выше, чем у B. cyri (табл. 1). При рассмотрении более длинных последовательностей данного маркера (длиной 1068 п.н.), для B. lacerta почти каждая из них уникальна, хотя бо́льшая их часть происходит из одной локальности – р. Пери Сую (Peri Suyu). Таким образом, B. lacerta имеет высокое генетическое разнообразие. В целом усачи B. cyri и B. lacerta генетически хорошо различаются, что видно на сети гаплотипов. Минимальное генетическое расстояние между гаплотипами этих видов – 25 нуклеотидных замен.
Филогеография куринского усача по данным последовательностей контрольного региона (CR). Сеть гаплотипов контрольного региона куринского усача построена с использованием 152 последовательностей длиной 652 п.н. (рис. 3а). Выявлено 59 гаплотипов, 38 из них представлены уникальными последовательностями.
Рис. 3.
Филогеография усачей В. cyri по 152 последовательностям контрольного региона мтДНК длиной 652 п.н.: а – сеть гаплотипов (число штрихов на отрезках, соединяющих гаплотипы, соответствует числу нуклеотидных замен; черные точки обозначают гипотетические гаплотипы); б – географическое распределение гаплогрупп контрольного региона B. cyri (географически близкие локальности объединены в одну). 1–7 – гаплогруппы.
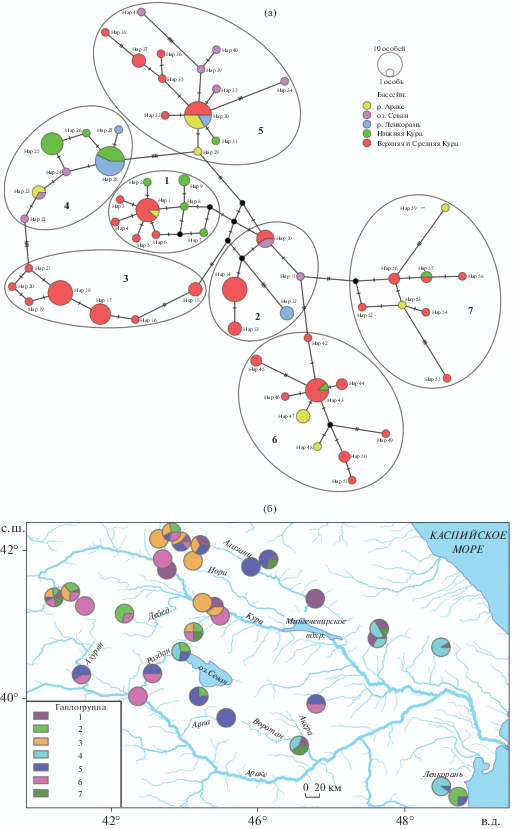
Сеть гаплотипов имеет сложную структуру. Учитывая топологию сети, можно выделить семь гаплогрупп (рис. 3а). Гаплогруппы не вполне соответствуют бассейнам или суб-бассейнам, хотя 3-я гаплогруппа включает гаплотипы исключительно из бассейна р. Кура. Гаплогруппы 1, 6 и 7 включают гаплотипы из бассейнов рек Кура и Аракс. Гаплогруппа 2 включает гаплотипы из всех бассейнов, кроме р. Аракс. Гаплогруппы 4 и 5, слабо дивергировавшие между собой (четыре мутации), включают гаплотипы из всех исследованных бассейнов (реки Кура, Аракс, Ленкорань и оз. Севан). Также обнаружено, что гаплотипы усача из оз. Севан и р. Ленкорань объединяются в одни и те же гаплогруппы: гаплотипы, выявленные в этих водоемах, входят в состав 2, 4 и 5-й гаплогрупп (рис. 3а). Гаплогрупп контрольного региона для лучшей визуализации их географического распределения представлены на карте (рис. 3б).
Все семь гаплогрупп обнаружены в бассейне р. Кура, пять гаплогрупп – в бассейне р. Аракс и по три гаплогруппы в бассейнах рек Ленкорань и оз. Севан. Гаплогруппы имеют разное распространение: например, 3-я гаплогруппа встречается исключительно в бассейне верхнего и среднего течения Куры. Также есть гаплогруппы, которые распространены преимущественно в отдельных бассейнах: 5-я гаплогруппа, распространенная в Верхней и Средней Куре, оз. Севан, р. Аракс и в р. Бадара (Нижняя Кура); 6-я гаплогруппа встречается в Верхней и Средней Куре, р. Аракс, а также в р. Бадара (Нижняя Кура); 4-я гаплогруппа обнаружена преимущественно в Нижней Куре, оз. Севан и р. Ленкорань; 1-я гаплогруппа встречается в одной локальности Араксинского бассейна, но широко распространена на всем протяжении бассейна р. Кура. Кроме того, есть гаплогруппы, которые отсутствуют в отдельных бассейнах (2-я гаплогруппа, не выявленная в бассейне р. Аракс). Как отмечали ранее, в бассейнах оз. Севан и р. Ленкорань распространены одни и те же гаплогруппы.
Весьма высокое гаплотипическое разнообразие куринского усача по сравнению с таковым для других бассейнов и для всех бассейнов вместе взятых отмечено в оз. Севан – Hd = 0.982 (табл. 2). Для этого бассейна почти каждая последовательность уникальна. Бассейн р. Ленкорань характеризуется низкими значениями почти всех показателей генетического разнообразия по сравнению с другими бассейнами (Hd = 0.648, K = 4.5, СВД = = 0.0069, π = 0.0069). Наибольшие значения нуклеотидного разнообразия, среднего количества нуклеотидных замен и средней внутригрупповой дистанции характерны для гаплотипов из бассейна р. Кура (π = 0.0097, K = 6.2, СВД = 0.0096), что совпадает с наличием последовательностей из этого бассейна в каждой гаплогруппе сети гаплотипов (рис. 3а).
Таблица 2.
Внутри- и межгрупповая генетическая изменчивость последовательностей контрольного региона B. cyri
| Группа | n/H | Hd± SD | π ± SD | K | СВД ± SE | МВД | БГ | Дист. БГ ± SE |
|---|---|---|---|---|---|---|---|---|
| Бассейн: р. Аракс | 14/8 | 0.890 ± 0.06 | 0.0086 ± 0.0012 | 5.6 | 0.0086 ± 0.0022 | 0.017 | р. Ленкорань | 0.009 ± 0.002 |
| р. Кура | 113/43 | 0.960 ± 0.006 | 0.0097 ± 0.0002 | 6.2 | 0.0096 ± 0.0022 | 0.02 | р. Аракс | 0.010 ± 0.002 |
| р. Ленкорань | 14/4 | 0.648 ± 0.116 | 0.0069 ± 0.0015 | 4.5 | 0.0069 ± 0.0021 | 0.017 | оз. Севан | 0.009 ± 0.002 |
| оз. Севан | 11/10 | 0.982 ± 0.046 | 0.0093 ± 0.001 | 6.1 | 0.0093 ± 0.0023 | 0.018 | р. Ленкорань | 0.009 ± 0.002 |
| Все бассейны | 152/59 | 0.965 ± 0.005 | 0.0096 ± 0.0002 | 6.3 | 0.0096 ± 0.0022 | 0.025 | – | – |
Примечание. Обозначения показателей как в табл. 1.
Для последовательностей контрольного региона мтДНК из разных бассейнов были рассчитаны попарные значения индекса фиксации FST (рис. 4). В связи с разницей в частотах гаплогрупп между бассейном р. Кура выше Мингечевирского водохранилища и ниже него (рис. 5), бассейн реки был разделен на два участка – верхне-среднее и нижнее течение реки.
Рис. 4.
График матрицы попарных значений FST между популяциями усача по данным последовательностей контрольного региона мтДНК. Звездочкой помечены значения FST. * p < 0.05.
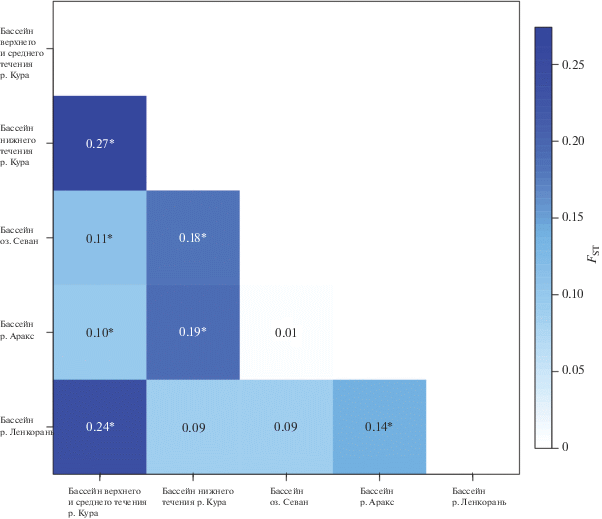
Рис. 5.
Диаграммы частот гаплогрупп в разных бассейнах. Для бассейна р. Кура частоты рассчитаны для всего бассейна и участков верхне-среднего и нижнего ее течений.
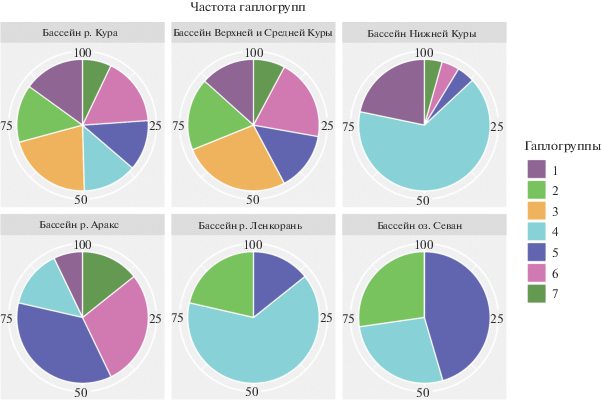
Наиболее высокое значение FST отмечено для популяций усача внутри бассейна р. Кура – между участками этой реки выше и ниже Мингечевирского водохранилища (FST = 0.27, p < 0.05). Наименьшее попарное значение FST выявлено для выборок из бассейна оз. Севан и р. Аракс (FST = 0.01, p > 0.05). Значения FST между выборками усача верхне-среднего течения р. Кура и выборками остальных бассейнов статистически значимо различаются. Популяции бассейна р. Ленкорань оказались более изолированы от популяций бассейна р. Аракс (FST = 0.14, p < 0.05), чем от популяций бассейна нижнего течения р. Куры (FST = 0.09, p > 0.05) или оз. Севан (FST = 0.09, p > 0.05). В то же время популяция бассейна Аракса генетически более изолирована от популяций Нижней Куры (FST = = 0.19, p < 0.05), чем от популяций верхнего и среднего течения этой реки (FST = 0.10, p < 0.05). Сходная ситуация в отношении Куринских суббасейнов наблюдается и для бассейна оз. Севан.
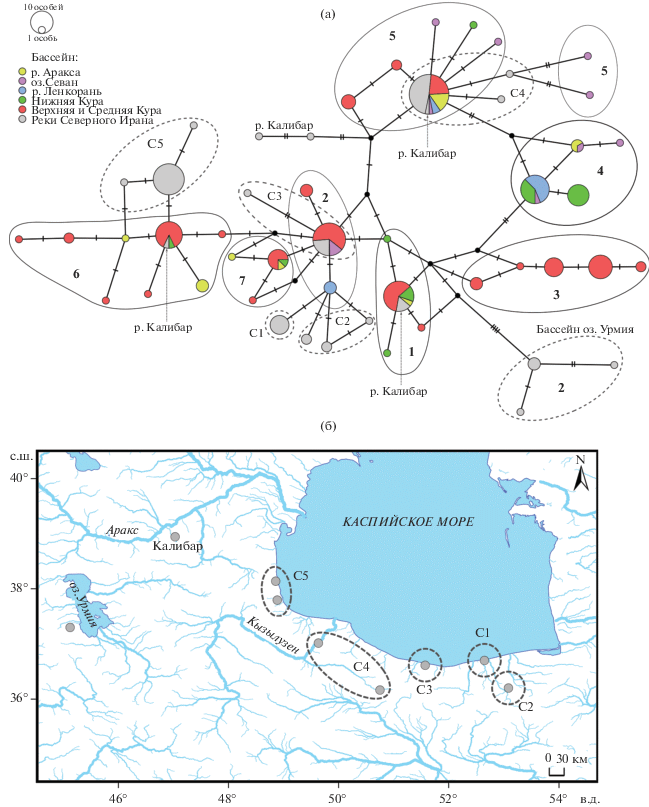
Для лучшего понимания филогеографии куринского усача нами сконструирована сеть гаплотипов (рис. 6) с добавлением более коротких последовательностей контрольного региона B. cyri из иранской части бассейна Каспийского моря (Khaefi et al., 2018), при этом семь ранее выделенных гаплогрупп (рис. 3а) сохраняются. Гаплотипы, описанные Хаефи и др. (Khaeti et al., 2018), входят в состав выделенных нами гаплогрупп или близки им (рис. 6). Некоторые из “иранских” последовательностей относятся к гаплотипам, общим с последовательностями усача из бассейнов рек Кура, Аракс, Ленкорань и оз. Севан. Вызывает интерес тот факт, что гаплотипы из р. Калибар (бассейн р. Аракс) расположены в разных частях сети на большом расстоянии друг от друга (5–9 замен, СВД = 0.011 ± 0.004, МВД = 0.019 ± 0.007) в составе разных гаплогрупп. Гаплогруппы C1–C3 близки географически и генетически, гаплогруппы С4 и C5, несмотря на географическую близость, генетически существенно дивергировали. Усачи из бассейна оз. Урмия группируются отдельно от остальных.
Рис 6.
Филогеография усачей В. cyri по 220 последовательностям контрольного региона мтДНК длиной 366 п.н. с учетом информации о выборках из Ирана (Khaefi et al., 2018): а – сеть гаплотипов (C1–C5, р. Калибар, оз. Урмия– гаплогруппы, описанные Хаефи с соавт., обведены пунктирной линией, 1–7 – гаплогруппы, выделенные нами ранее, обведены сплошной линией; б – географическое распространение гаплогрупп С1–С5, р. Калибар, оз. Урмия.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Два маркера мтДНК дают несколько различающуюся картину популяционно-генетической структуры куринского усача B. cyri. Так, сеть гаплотипов последовательностей гена cyt b представляет собой центральный гаплотип и отходящие от него отдельные гаплотипы и гаплотипические линии с недостаточно структурированными гаплогруппами (рис. 2). Такая структура может быть свидетельством того, что расселение куринского усача по водоемам и водотокам Кавказа было достаточно быстрым. В состав сети гаплотипов по гену cyt b также входят последовательности B. lacerta из двух водотоков бассейна р. Евфрат, представленные семью гаплотипами. В целом B. lacerta имеет высокое генетическое разнообразие.
В то же время анализ сети гаплотипов, построенной на основании некодирующих последовательностей контрольного региона (рис. 3а), показал существенную подразделенность выявленных гаплотипов на ряд гаплогрупп, не имеющих четкой приуроченности к тому или иному географическому региону или бассейну. На фоне общей слабой географической структурированности в р. Кура обнаружена уникальная группа гаплотипов B. cyri – 3-я гаплогруппа (рис. 3). Она представлена лишь в верхне-среднем течении реки, что может говорить о возможной частичной изоляции друг от друга популяций верхне-среднего и нижнего течений. Отсутствие в верхне-среднем течении представителей 4-ой гаплогруппы, которая встречается в нижнем течении р. Кура, также указывает на генетическую сегрегацию куринских популяций. О сниженном потоке генов между популяциями верхне-среднего и нижнего течения р. Кура свидетельствует и высокое значение FST между ними (рис. 4). Полученные результаты согласуются с информацией об эндемизме ихтиофауны области среднего течения Куры. В частности, в данном регионе обитают узколокальные эндемики Pseudophoxinus atropatenus (Derjavin 1937) (Артаев и др., 2018) и Cobitis derzhavini Vasil’eva, Solovyeva, Levin, Vasil’ev, 2020 (Vasil’eva et al., 2020). По-видимому, участок бассейна Средней Куры можно рассматривать как рефугиум водной фауны Закавказья.
Хотя бассейн р. Ленкорань изолирован от Кура-Араксинской речной системы, последовательности ленкоранского усача все же принадлежат к разным гаплогруппам, находясь на значительной дистанции друг от друга (табл. 2, рис. 2). Это может говорить о неоднократном заселении бассейна р. Ленкорань представителями разных гаплогрупп, возможно, несколькими путями. Так, за последний миллион лет уровень Каспийского моря существенно колебался, что сопровождалось изменениями солености в диапазоне 7–16‰ (Янина, 2012). Проникновение куринского усача в бассейн р. Ленкорань могло быть приурочено к крупным регрессиям или начальным этапам трансгрессий Каспийского моря, во время которых, как полагают, происходило снижение его солености с возможным возникновением локальных опресненных лиманов в местах сброса речных вод (Свиточ, 2016). Как предполагает Свиточ (2016), в трансгрессивные эпохи объем растворенных солей в водах Каспийского моря был несколько больше, чем в регрессивные. В ходе трансгрессий Каспия источником солей выступали соляные купола Северного Прикаспия и, вероятно, залив Кара-Богаз-Гол и засоленные грунты при их затоплении. Кроме того, во время регрессий при отступлении Каспия палеорусла рек могли соединяться ниже по течению, что также представляет собой возможность для расселения рыб. Случаи подобных слияний палеорусел известны для периодов регрессий уровня морей и океанов (Гулин, Коваленко, 2010; Swartz et al., 2014).
Не исключено, что бассейн р. Ленкорань был связан с Кура-Араксинским бассейном через систему перехватов верховий рек. О связи фауны р. Ленкорань с фауной Кура-Араксинского бассейна говорят также недавние генетические исследования быстрянок Alburnoides (Levin et al., 2018). Согласно попарным значениям FST, поток генов между бассейнами верхне-среднего течения р. Кура и р. Ленкорань ниже, чем между р. Ленкорань и другими бассейнами (рис. 4). В то же время, более высокие значения FST между выборками рек Ленкорань и Аракс по сравнению с таковыми между р. Ленкорань и Нижней Курой предположительно объясняются колонизацией р. Ленкорань из региона Нижней Куры или одновременно с ней.
Озеро Севан – высокогорное изолированное озеро, находящееся в бассейне р. Аракс. Изоляция оз. Севан произошла ~20 тыс. лет назад путем образования водопада высотой 15 м на вытекающей из озера р. Раздан (Давыдов, 1938; Милановский, 1968). Низкий показатель FST между популяциями бассейнов р. Аракс и оз. Севан указывает на их недавнюю генетическую изоляцию. Также, в оз. Севан, вероятно, сохранились остатки линий, которые когда-то заселяли р. Ленкорань, что объясняет встречаемость в двух бассейнах одних и тех же гаплогрупп (рис. 3).
По данным спорово-пыльцевого анализа, увлажненность климата и уровень воды в оз. Севан на протяжении голоцена существенно флуктуировали (Саядян, Алешинская, 1991). Изменения водного режима оз. Севан в этом случае могут служить индикатором для всего региона Южного Кавказа, отражая колебания в уровне водности речных систем. Климатические флуктуации могли способствовать как изоляции отдельных популяций при фрагментации речных сетей в период сухого климата, так и приводить к вторичным контактам в результате расширения гидрографической сети и перехватам рек в периоды влажного климата. В дополнение к этому высокая вулканическая активность Малого Кавказа (Милановский, 1968; Леонов, 2003) также оказывала влияние на перестройку гидрографической сети.
Примечательно, что для куринского усача Южного Каспия так же характерны изолированность отдельных популяций и следы их перемешивания (Khaefi et al., 2018). Популяции куринского усача иранского побережья Каспия имеют много общих гаплотипов с выборками Кавказа, и направление их расселения трудно установить. Нельзя исключать неоднократных повторных расселений куринского усача вдоль побережья Каспия в четвертичный период.
Выводы. Настоящее исследование – первая попытка подробно рассмотреть филогеографию усачей рода Barbus на примере куринского усача B. cyri на Кавказе. Анализ полиморфизма двух молекулярных маркеров мтДНК свидетельствует о сложной истории расселения и взаимодействия популяций куринского усача B. cyri. Кодирующий маркер cyt b указывает на относительно быстрое распространение исследуемого вида по водоемам и водотокам Кавказа. Согласно анализу полиморфизма некодирующей области мтДНК, контрольного региона (CR), популяции куринского усача генетически подразделены на ряд групп. Полученные результаты выявили генетическую сегрегацию популяций верхне-среднего и нижнего течения р. Кура, а также неоднократные заселения усачом бассейнов р. Ленкорань, оз. Севан и Нижней Куры. Вероятно, между рассматриваемыми бассейнами, в том числе и бассейнами иранского побережья Каспия, существовал обмен генетическими линиями. Сложная современная генетическая структура популяций куринского усача, по-видимому, складывалась в периоды интенсивных тектонических процессов и климатических колебаний плейстоцена–голоцена, существенно влиявших на гидрографию речных сетей и способствовавших как вторичным контактам, так и изоляции отдельных популяций.
Список литературы
Абдурахманов Ю.А. 1962. Рыбы пресных вод Азербайджана. Баку: Изд-во АН АзССР.
Артаев О.Н., Лёвин Б.А., Мустафаев Н.Д., Симонов Е.П. 2018. Ширванская плотва – закавказский реликт? // Природа. № 5. С. 57.
Берг Л.С. 1949. Рыбы пресных вод СССР и сопредельных стран. Ч. 2. Москва: Изд-во Акад. наук СССР. С. 693.
Гулин М.Б., Коваленко М.В. 2010. Палеорусла рек Черная и Бельбек на шельфе юго-западного Крыма – новый объект экологических исследований // Морской экол. журн. Т. 9. № 1. С. 23.
Давыдов В.К. 1938. Водный баланс озера Севан. Материалы по исследованию озера Севан и его бассейна. Ч. 6. Ленинград: Гидрометеорологическое изд-во.
Дадикян М.Г. 1986. Рыбы Армении. Ереван: Изд-во АН АрмССР.
Лёвин Б.А. 2004. Фенетические отношения усачей Кавказского региона и их положение в системе рода Barbus sensu stricto (Cyprinidae) // Вопр. ихтиологии. Т. 44. № 4. С. 496.
Леонов М.Г. 2003. Поэзия Кавказских гор // Природа. № 7. С. 25.
Милановский Е.Е. 1968. Новейшая тектоника Кавказа. Москва: Недра.
Саядян Ю.В., Алешинская З.В. 1991. Колебания увлажненности в бассейне оз. Севан // История озер Севан, Иссык-Куль, Балхаш, Зайсан и Арал. Ленинград: Наука. С. 38.
Свиточ А.А. 2016. Регрессивные эпохи большого Каспия // Водные ресурсы. Т. 43. № 2. С. 134. https://doi.org/10.7868/S0321059616020164
Янина Т.А. 2012. Неоплейстоцен Понто-Каспия: биостратиграфия, палеогеография, корреляция. Москва: МГУ.
Aljanabi S.M., Martinez I. 1997. Universal and rapid salt-extraction of high quality genomic DNA for PCR-based techniques // Nucleic Acids Res. V. 25. № 22. P. 4692. https://doi.org/10.1093/nar/25.22.4692
Artaev O.N., Ermakov O.A., Vekhov D.A. et al. 2021. Genetic screening of distribution pattern of roaches Rutilus rutilus and R. lacustris (Cyprinidae) in the broad range of secondary contact (Volga basin) // Inland Water Biol. V. 14. № 2. P. 205. https://doi.org/10.1134/S1995082921020024
Avise J.C. 2000. Phylogeography: the history and formation of species. Cambridge: Harvard University Press.
Bănărescu P.M., Bogustkaya N.G. 2003. The Freshwater Fishes of Europe. V. 5. Cyprinidae 2. Part II. Barbus. Wiesbaden: Aula.
Bandelt H.J., Forster P., Röhl A. 1999. Median-joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. V. 16. № 1. P. 37. https://doi.org/10.1093/oxfordjournals.molbev.a026036
Berrebi P., Tsigenopoulos C.S. 2003. Phylogenetic organization of the genus Barbus sensu stricto: A review based on data obtained using molecular markers // The Freshwater Fishes of Europe. V. 5. P. 11.
Bianco P.G., Bănărescu P. 1982. A contribution to the knowledge of the Cyprinidae of Iran (Pisces, Cypriniformes) // Cybium. V. 6. № 2. P. 75.
Coad B.W. 1995. Fresh water fishes of Iran // Acta Sc. Nat. Brno. V. 29. № 1. P. 1.
Doadrio I., Carmona J.A., Machordom A. 2002. Haplotype diversity and phylogenetic relationships among the Iberian barbels (Barbus, Cyprinidae) reveal two evolutionary lineages // J. Hered. V. 93. № 2. P. 140. https://doi.org/10.1093/jhered/93.2.140
Espanhol R., Almeida P.R., Alves M.J. 2007. Evolutionary history of lamprey paired species Lampetra fluviatilis (L.) and Lampetra planeri (Bloch) as inferred from mitochondrial DNA variation // Mol. Ecol. V. 16. № 9. P. 1909. https://doi.org/10.1111/j.1365-294X.2007.03279.x
Excoffier L., Lischer H.E.L. 2010. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Res. V. 10. № 3. P. 564. https://doi.org/10.1111/j.1755-0998.2010.02847.x
Gandlin A.A., Mustafaev N.D., Yakimov A.V., Levin B.A. 2017. Updating the geographical range of Terek barbel Barbus ciscaucasicus Kessler, 1877 (Cyprinidae) using the cytochrome b molecular marker // Inland Water Biol. V. 10. № 1. P. 115. https://doi.org/10.1134/S1995082917010072
Khaefi R., Esmaeili H.R., Geiger M.F., Eagderi S. 2017. Taxonomic review of the cryptic Barbus lacerta species group with description of a new species (Teleostei: Cyprinidae) // FishTaxa. V. 2. № 2. P. 90.
Khaefi R., Esmaeili H.R., Ansari M.H., Ebrahimi M. 2018. Genetic diversification and population structure of Barbus cyri De Filippi, 1865 (Teleostei: Cyprinidae) in northern Iran inferred from the mitochondrial D-loop gene sequence // Environ. Biol. Fishes. V. 101. № 1. P. 181. https://doi.org/10.1007/s10641-017-0690-2
Kotlík P., Berrebi P. 2001. Phylogeography of the barbel (Barbus barbus) assessed by mitochondrial DNA variation // Mol. Ecol. V. 10. № 9. P. 2177. https://doi.org/10.1046/j.0962-1083.2001.01344.x
Kotlík P., Bogutskaya N.G., Ekmekçi F.G. 2004. Circum Black Sea phylogeography of Barbus freshwater fishes: divergence in the Pontic glacial refugium // Mol. Ecol. V. 13. № 1. P. 87. https://doi.org/10.1046/j.1365-294X.2003.02021.x
Lajbner Z., Šlechtová V., Šlechta V. et al. 2009. Rare and asymmetrical hybridization of the endemic Barbus carpathicus with its widespread congener Barbus barbus // J. Fish Biol. V. 74. № 2. P. 418. https://doi.org/10.1111/j.1095-8649.2008.02098.x
Leigh J.W., Bryant D. 2015. POPART: full-feature software for haplotype network construction // Methods in Ecology and Evolution. V. 6. № 9. P. 1110. https://doi.org/10.1111/2041-210X.12410
Levin B.A., Rubenyan H.R. 2006. Threatened fishes of the world: Barbus goktschaicus Kessler, 1877 (Cyprinidae) // Environ. Biol. Fishes. V. 76. № 2–4. P. 409. https://doi.org/10.1007/s10641-006-9026-3
Levin B.A., Freyhof J., Lajbner Z. et al. 2012. Phylogenetic relationships of the algae scraping cyprinid genus Capoeta (Teleostei: Cyprinidae) // Mol. Phylogenet. Evol. V. 62. № 1. P. 542. https://doi.org/10.1016/j.ympev.2011.09.004
Levin B.A., Golubtsov A.S., Dgebuadze Y.Y., Mugue N.S. 2013. New evidence of homoplasy within the African genus Varicorhinus (Cyprinidae): an independent origin of specialized scraping forms in the adjacent drainage systems of Ethiopia inferred from mtDNA analysis // African Zoology. V. 48. № 2. P. 400. https://doi.org/10.3377/004.048.0208
Levin B.A., Simonov E., Matveyev M.P. et al. 2018. DNA barcoding of the fishes of the genus Alburnoides (Actinopterygii, Cyprinidae) from Caucasus // Mitochondrial DNA Part A. V. 29. № 1. P. 49. https://doi.org/10.1080/24701394.2016.1238900
Levin B.A., Gandlin A.A., Simonov E.S. et al. 2019. Phylogeny, phylogeography and hybridization of Caucasian barbels of the genus Barbus (Actinopterygii, Cyprinidae) // Mol. Phylogenet. Evol. V. 135. P. 31. https://doi.org/10.1016/j.ympev.2019.02.025
Meraner A., Venturi A., Ficetola G.F. et al. 2013. Massive invasion of exotic Barbus barbus and introgressive hybridization with endemic Barbus plebejus in Northern Italy: where, how and why? // Mol. Ecol. V. 22. № 21. P. 5295. https://doi.org/10.1111/mec.12470
Meyer A., Morrissey J.M., Schartl M. 1994. Recurrent origin of a sexually selected trait in Xiphophorus fishes inferred from a molecular phylogeny // Nature. V. 368. № 6471. P. 539. https://doi.org/10.1038/368539a0
Palumbi S.R. 1996. What can molecular genetics contribute to marine biogeography? An urchin’s tale // J. Exp. Mar. Biol. Ecol. V. 203. № 1. P. 75. https://doi.org/10.1016/0022-0981(96)02571-3
Perdices A., Bermingham E., Montilla A., Doadrio I. 2002. Evolutionary history of the genus Rhamdia (Teleostei: Pimelodidae) in central America // Mol. Phylogenet. Evol. V. 25. № 1. P. 172. https://doi.org/10.1016/S1055-7903(02)00224-5
Rothgänger J., Weniger M., Weniger T. et al. 2005. Ridom TraceEdit: a DNA trace editor and viewer // Bioinformatics. V. 22. № 4. P. 493. https://doi.org/10.1093/bioinformatics/btk002
Rozas J., Ferrer-Mata A., Sánchez-DelBarrio J.C. et al. 2017. DnaSP 6: DNA sequence polymorphism analysis of large data sets // Mol. Biol. Evol. V. 34. № 12. P. 3299. https://doi.org/10.1093/molbev/msx248
Swartz E.R., Chakona A., Skelton P.H., Bloomer P. 2014. The genetic legacy of lower sea levels: does the confluence of rivers during the last glacial maximum explain the contemporary distribution of a primary freshwater fish (Pseudobarbus burchelli, Cyprinidae) across isolated river systems? // Hydrobiologia. V. 726. № 1. P. 109. https://doi.org/10.1007/s10750-013-1755-7
Tamura K., Dudley J., Nei M., Kumar S. 2007. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0 // Mol. Biol. Evol. V. 24. № 8. P. 1596. https://doi.org/10.1093/molbev/msm092
Tsigenopoulos C.S., Berrebi P. 2000. Molecular phylogeny of North Mediterranean freshwater barbs (genus Barbus: Cyprinidae) inferred from cytochrome b sequences: biogeographic and systematic implications // Mol. Phylogenet. Evol. V. 14. №2. P. 165. https://doi.org/10.1006/mpev.1999.0702
Vasil’eva E.D., Solovyeva E.N., Levin B.A., Vasil’ev V.P. 2020. Cobitis derzhavini sp. nova – a new spined loach species (Teleostei: Cobitidae) discovered in the Transcaucasia // J. Ichthyol. V. 60. P. 135. https://doi.org/10.1134/S0032945220020198
Weir B.S., Cockerham C.C. 1984. Estimating F-statistics for the analysis of population structure // Evolution. V. 38. № 6. P. 1358. https://doi.org/10.1111/j.1558-5646.1984.tb05657.x
Zaccara S., Quadroni S., Vanetti I. et al. 2019. Morphologic and genetic variability in the Barbus fishes (Teleostei, Cyprinidae) of Central Italy // Zoologica Scripta. V. 48. № 3. P. 289. https://doi.org/10.1111/zsc.12341
Дополнительные материалы
- скачать ESM_1.docx
- Дополнительный материал (табл. 1S).
Инструменты
Биология внутренних вод