Биология внутренних вод, 2023, № 5, стр. 671-679
Уникальные гаплотипы Artemia salina (Crustacea, Branchiopoda, Anostraca) в гиперсоленом оз. Сасык-Сиваш (Крым)
А. О. Лантушенко a, Я. В. Мегер a, *, А. В. Гаджи a, Е. В. Ануфриева a, b, Н. В. Шадрин a, b
a Севастопольский государственный университет
Севастополь, Россия
b Федеральный исследовательский центр Институт биологии южных морей
им. А.О. Ковалевского Российской академии наук
Севастополь, Россия
* E-mail: meger_yakov@mail.ru
Поступила в редакцию 03.11.2022
После доработки 22.03.2023
Принята к публикации 14.04.2023
- EDN: REAHCK
- DOI: 10.31857/S032096522305008X
Аннотация
Исследована генетическая структура популяции вида Artemia salina (L., 1758), из гиперсоленого оз. Сасык-Сиваш на основе фрагмента гена первой субъединицы цитохром оксидазы с COI митохондриальной ДНК. Реконструкция филогении выполнена с использованием всех имеющихся в международной базе данных GenBank (NCBI) нуклеотидных последовательностей данного гена для вида salina. Установлены генеалогические связи между COI гаплотипами и выявлены филогеографические паттерны. В западно-средиземноморских популяциях отмечены общие гаплотипы, что может быть следствием их расположения в едином миграционном коридоре птиц, которые, как известно, способствуют пассивному распространению покоящихся стадий артемии. Географически изолированные группы популяций из Ливии, Туниса, Египта, Кипра и Крыма характеризуются уникальными гаплотипами, в настоящее время не обнаруженными в других средиземноморских популяциях. Высказано предположение, что уникальные гаплотипы могут быть эндемичными для географически удаленных регионов.
ВВЕДЕНИЕ
Фауна отдельных водных экосистем, например озер, состоит как из активно распространяющихся животных (амфибионтные летающие насекомые, амфибии), так и пассивно распространяемых беспозвоночных, переносимых ветром или подвижными животными (Fontaneto, 2019; Hessen et al., 2019). При этом птицами и летающими насекомыми переносятся чаще всего покоящиеся стадии этих животных (Green et al., 2005; Frisch et al., 2007; Naceur, 2020). Изучение структуры популяций и филогеографические образцы пассивно расселяемых водных беспозвоночных показало, что, несмотря на их высокую способность к колонизации новых местообитаний в покоящихся стадиях, эти организмы часто демонстрируют неожиданно высокую степень генетической дифференциации и локального эндемизма вследствие низкого потока генов между такими популяциями (Hebert, 1998; De Meester, 2002; Hebert et al., 2003b; Penton et al., 2004; De Gelas, De Meester, 2005; Paland et al., 2005; Ishida, Taylor, 2007). В настоящее время для таких популяций в основном рассматривается два крайних варианта филогеографической структуры. Во-первых, относительно старые популяции в плейстоценовых убежищах или вокруг них демонстрируют глубокую дивергенцию/диверсификацию генетической структуры и сильные межпопуляционные различия с высокой степенью локального эндемизма (Gómez et al., 2000, 2007; Zierold et al., 2007). Во втором случае обнаруживается незначительно дифференцированная географическая структура и пониженное генетическое разнообразие, что свидетельствует о недавнем и быстром расширении ареала в новую доступную область, либо после естественной колонизации, часто послеледниковой, или после интродукции человеком (Weider et al., 1999; Mergeay et al., 2005; Ishida, Taylor, 2007). Сочетание обоих вариантов может быть обнаружено у одного и того же вида в зависимости от возраста его локальных популяций по всему ареалу (Ishida, Taylor, 2007). Для понимания закономерностей формирования филогеографических образцов вида необходимы данные о молекулярно-генетической структуре его локальных популяций на различных пространственных масштабах (Gómez et al., 2002; Adamowicz et a., 2005, 2007).
Удобными модельными объектами для филогеграфических исследований являются виды рода Artemia (Crustacea, Branchiopoda, Anostraca), широко распространенные в мире, играющие важную роль в функционировании экосистем гиперсоленых водоемов и имеющие большую коммерческую ценность (Sanchez et al., 2016; Marden et al., 2020; Van Stappen et al., 2020). В водоемах Крыма при соленостях >100 г/л артемии часто доминируют и достигают весьма высокой численности в гиперсоленых озерах и лагуне Сиваш – >75 тыс. экз./м3 (Балушкина и др., 2009; Anufriieva et al., 2022). Они играют ключевую роль в пищевых сетях этих водоемов, питаясь фитопланктоном, и служат пищей для различных беспозвоночных и водных птиц (Sanchez et al., 2016; Marden et al., 2020). Как организмы-фильтраторы виды рода Artemia участвуют в образовании лечебных грязей (Балушкина и др., 2009) и очищении воды от ртути (Shadrin et al., 2022).
В настоящее время в мире наряду с партеногенетическими популяциями признано существование пяти валидных видов двуполых артемий: A. salina (L., 1758), A. urmiana Günther, 1890, A. monica Verrill, 1869 (=A. franciscana Kellogg, 1906) и A. sinica Cai, 1989, A. persimilis Piccinelliand Prosdocimi, 1968 (Sainz-Escudero et al., 2021). В Крыму, крупнейшем п-ве Черного моря, в многочисленных гиперсоленых водоемах отмечено существование четырех двуполых видов (A. salina, A. urmiana, A. monica и A. sinica) и множество разноплоидных партеногенетических популяций Artemia (Shadrin, Anufriieva, 2017; Lantushenko et al., 2022). Artemia salina – аборигенный вид Средиземноморско-Черноморского региона, характеризующийся высоким локальным генетическим разнообразием (Munoz et al., 2008). Вселение A. monica (=A. franciscana) в гиперсоленые водоемы этого региона ведет к исчезновению локальных популяций A. salina и, следовательно, к снижению генетического разнообразия вида (Munoz et al., 2008). Разнообразие гаплотипов в крымских популяциях A. salina ранее не изучали.
Цель работы – дать анализ гаплотипического разнообразия и оценить уровень его уникальности в популяции A. salina из оз. Сасык-Сиваш, крупнейшего гиперсоленого озера Крыма.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
В Крыму зарегистрировано >50 гиперсоленых озер, оз. Сасык-Сиваш (45°09′10″ с.ш., 33°31′04″ в.д.) – крупнейшее среди них (Shadrin et al., 2022). Площадь озера 75.3 км2. Озеро мелководное (глубина ≤1.5 м), полимиксное, характеризуется высокой сезонной и многолетней изменчивостью различных характеристик. В настоящее время озеро разделено на две части – с низкой и высокой соленостью. В период наблюдений (2016–2022 гг.) соленость в гиперсоленой части озера колебалась от 220 до 350 г/л (Shadrin et al., 2022), средняя соленость была ~280 г/л.
В июле 2021 г. в оз. Сасык-Сиваш собраны взрослые особи A. salina, плотность популяции рачков достигала 220 экз./м3 при солености 245 г/л и температуре ~35°С. Пробы отбирали путем фильтрации воды через малую планктонную сеть Джеди с размером ячеи газа 50 мкм. Живых рачков доставляли в лабораторию для дальнейшего анализа. Одновременно с взятием проб в озере измеряли соленость и температуру воды с помощью портативного рефрактометра WZ212 (Kelilong Electron Co. Ltd., Фуань, Китай) и электронного рН-метра PHH-830 (OMEGA Engineering Inc., Норуолк, США) соответственно.
Тотальную ДНК выделяли с помощью набора ДНК-Экстран 2 (Синтол, Россия) согласно инструкциям производителя. Качество выделенных продуктов оценивали на нанофотометре Implen N60 (Germany) и с помощью электрофореза на 1.5%-ном агарозном геле. Фрагмент маркерного митохондриального гена COI амплифицировали с помощью ПЦР набора Screen-Mix (Eurogen, Россия) в объеме 25 мкл с использованием двух праймеров 2COI_Fol-F (5′-ATTCTACGAATCACAAGGATATTGG-3′) и 2COI_Fol-R (5′-TACACTTCAGGATGGCCAAAA) (Munoz, 2008). Протокол амплификации включал следующие стадии: предварительная денатурация 5 мин – 95°C, 34 цикла амплификации 30 с – 95°С, 30 с – 48°С, 45 с – 72°С и финальная элонгация 5 мин – 72°С. Очистку от остаточных продуктов ПЦР-смеси осуществляли методом сорбционной экстракции ДНК набором Colgen (Синтол, Россия). Для проверки однородности размерного состава амплифицированных фрагментов проводили визуализацию с помощью электрофореза на 1.5%-ном агарозном геле, концентрацию полученного продукта контролировали спектрофотометрически на нанофотометре Implen N60. При секвенировании использовали набор реагентов Big Dye Terminator v.3.1 cycle sequencing kit (Thermo Fisher Scientific, США), визуализацию продуктов секвенирования осуществляли при помощи капиллярного электрофореза на генетическом анализаторе Нанофор 05 (Синтол, Россия) в Центре коллективного пользования “Молекулярная структура вещества” Севастопольского гос. университета. Всего получено и проанализировано восемь нуклеотидных последовательностей фрагмента гена COI мтДНК длиной 559 пн.
Редактирование полученных нуклеотидных последовательностей проводили вручную в программе Bioedit v.7.2.5 (Hall et al., 2011). В дополнение к полученным оригинальным данным в анализ включали все последовательности гена COI A. salina, доступные в международной базе данных GenBank (NCBI), общее число последовательностей длиной 559 пн ‒ 115 (табл. 1). Выравнивание проводили в программе Mega-X (Kumar, 2018) с помощью алгоритма MUSCLE, гаплотипический анализ – в DnaSP6 (Rozas, 2017). Гаплотипическую сеть реконструировали методом TCS (Templeton, Crandall and Sing, 1992) в программе PopART v.1.7 (Leigh, Bryant, 2015). Карту географического распространения гаплотипов составляли на основе координат, соответствующих исследованным популяциям. Филогенетический анализ для A. salina проводили с помощью байесовских методов в MrBaeys v.3.2 (Ronquist, 2012). Он включал 10 млн итераций, 25% первых исключали из дальнейшего анализа. Топологию результирующего дерева выбирали по лучшей достоверности клад, длину ветвей вычисляли по средним значениям параметров промежуточных деревьев.
Таблица 1.
Гаплотипы (H) Artemia salina, найденные в разных частях ареала и проанализированные в данной работе
| Гаплотип– число особей |
Сокращенное название популяции |
Географическое расположение | Номера в ГенБанке (NCBI) | Литературный источник |
|---|---|---|---|---|
| H1–5 | SS | оз. Сасык-Сиваш, Крым | OQ376365 ON872209 OP753708 ON872211 ON872210 |
Данные авторов |
| H2–1 | SS | То же | ON872208 | То же |
| H3–1 | SS | » | ON872206 | » |
| H4–1 | SS | » | ON872207 | » |
| H5–6 | BON, BRAS, MAT, PIN | Испания | DQ426827, DQ426836, DQ426851 | Munoz et al., 2008 |
| H6–1 | MAT | То же | DQ426828 | То же |
| H7–1 | MAT | » | DQ426829 | » |
| H8–1 | BON, BRAS, MAT | » | DQ426830 | » |
| H9–6 | DON, EBR, ROC, POR, MOL, SGI | Испания, Италия | DQ426831, DQ426832, DQ426853, DQ426857, EU543475, EU543477 | » |
| H10–1 | DON, POR, ROC | Испания | DQ426833 | » |
| H11–2 | DON, POR | То же | DQ426834, DQ426856 | » |
| H12–1 | BRAS | » | DQ426835 | » |
| H13–1 | BRAS | » | DQ426837 | » |
| H14–1 | BRAS | » | DQ426838 | » |
| H15–1 | BRAS | » | DQ426839 | » |
| H16–1 | BRAS | » | DQ426840 | » |
| H17–1 | PIN | » | DQ426841 | » |
| H18–1 | PIN | » | DQ426842 | » |
| H19–1 | PIN | » | DQ426843 | » |
| H20–1 | PIN | » | DQ426844 | » |
| H21–1 | PIN | » | DQ426845 | » |
| H22–1 | PIN | » | DQ426846 | » |
| H23–1 | CER | » | DQ426847 | » |
| H24–1 | CER | » | DQ426848 | » |
| H25–1 | CER | » | DQ426849 | » |
| H26–1 | BON, CER | » | DQ426850 | » |
| H27–1 | POR | » | DQ426854 | » |
| H28–1 | MOL, POR, SGI | Испания, Италия | DQ426855 | » |
| H29–1 | EBR | Испания | DQ426858 | » |
| H30–1 | ROS | То же | EU543444 | » |
| H31–7 | SCA, MAL, LAM, SAL | » | EU543445, EU543446, EU543449, OM486989, OM486990, OM486995, OM486996 | Sainz-Escuderoet al., 2022 |
| H32–1 | MAL | » | EU543447 | Munoz et al., 2008 |
| H33–1 | MAL | » | EU543448 | То же |
| H34–5 | CAM, SAL, SNT | » | EU543450, OM486997, OM486998, OM486999, OM487000 | Sainz-Escudero et al., 2022, Eimanifar et al., 2014 |
| H35–1 | CAM | » | EU543451 | Munoz et al., 2008 |
| H36–1 | JAD | Марокко | EU543452 | То же |
| H37–1 | JAD | То же | EU543453 | » |
| H38–6 | JAD, MEG | Марокко, Тунис | EU543454, KF691509, KF691510, KF691511, KF691512, KF691513 | Munoz et al., 2008, Eimanifar et al., 2014 |
| H39–1 | JAD | Марокко | EU543455 | Munoz et al., 2008 |
| H40–2 | ADH, SAH | Тунис | EU543456, EU543458 | То же |
| H41–2 | ADH, LIB | Тунис, Ливия | EU543457, EU543460 | » |
| H42–1 | ADH | Тунис | EU543459 | » |
| H43–1 | ADH | То же | EU543461 | » |
| H44–1 | ADH | » | EU543462 | » |
| H45–1 | LIB, SAH | » | EU543463 | » |
| H46–1 | LIB, SAH | » | EU543464 | » |
| H47–1 | SAH | » | EU543466 | » |
| H48–1 | GAR | Алжир | EU543467 | » |
| H49–2 | GAR, REL | То же | EU543468, KF691135 | Munoz et al., 2008, Sainz-Escudero et al., 2022 |
| H50–1 | GAR | » | EU543469 | Munoz et al., 2008 |
| H51–1 | WAD | Египет | EU543470 | То же |
| H52–1 | WAD | То же | EU543471 | » |
| H53–1 | LIB | Тунис | EU543472 | » |
| H54–2 | CYP, LAR | Кипр | EU543473, OM486981 | Munoz et al., 2008, Sainz-Escudero et al., 2022) |
| H55–1 | SGI | Италия | EU543474 | Munoz et al., 2008 |
| H56–1 | SGI | То же | EU543476 | То же |
| H57–1 | MOL | » | EU543478 | » |
| H58–1 | MOL | » | EU543479 | » |
| H59–1 | MES | » | EU543480 | » |
| H60–1 | MES | » | EU543481 | » |
| H61–1 | VEL | Южная Африка | EU543482 | » |
| H62–1 | VEL | То же | EU543483 | » |
| H63–1 | VEL | » | EU543484 | » |
| H64–1 | VEL | » | EU543485 | » |
| H65–1* | DAPH | – | HQ972028 | – |
| H66–2 | REL | Алжир | KF691133, KF691134 | Eimanifar et al., 2014 |
| H67–1 | REL | То же | KF691136 | То же |
| H68–1 | MEG | Тунис | KF691514 | » |
| H69–4 | SFA | То же | KF691515, KF691517, KF691518, KF691519 | » |
| H70–1 | SFA | » | KF691516 | » |
| H71–1 | LAR | Кипр | OM486980 | Sainz-Escudero et al., 2022 |
| H72–3 | SID | Марокко | OM486982, OM486984, OM486985 | То же |
| H73–1 | SID | То же | OM486983 | » |
| H74–1 | FOR | Испания | OM486988 | » |
| H75–2 | BRU | То же | OM486991, OM486992 | » |
| H76–2 | LAG | » | OM486993, OM486994 | » |
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
В исследованной популяции A salina обнаружено четыре гаплотипа Н1-Н4 (табл. 1, рис. 1). Один из выделенных гаплотипов был найден у пяти особей, три остальных – единожды. Важно отметить, что все четыре гаплотипа отмечены только в популяции из оз. Сасык-Сиваш, следовательно, их можно считать уникальными, и, возможно, эндемичными для п-ова Крым.
Рис. 1.
Карта ареала обитания A. salina и филогенетическое дерево вида. Дерево построено с помощью байесовских методов в MrBaeys v.3.2: цветом выделена клада, сформированная уникальными гаплотипами из различных популяций; на цветовой шкале отображена поддержка узлов. ⚫ – анализируемые популяции (всего 38).
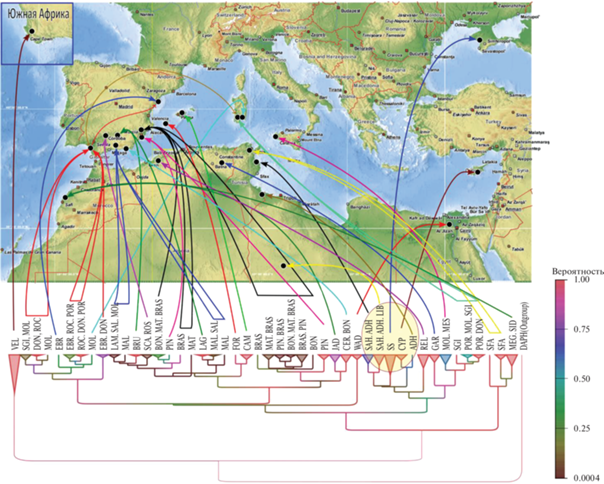
Всего для вида A. salina выявлено 76 гаплотипов (табл. 1), 16 из них – общие для разных популяций, 60 – уникальные, встречающиеся только в одной популяции. Популяционно-генетические параметры свидетельствуют о достаточно высоком разнообразии гаплотипов у A. salina (табл. 2). Для обитающих в Европе особей инвазивного вида A. franciscana характерны более низкие значения гаплотипического разнообразия Hd (0.54) (Eimanifar, 2014), чем для A. salina (0.98) (табл. 2). Для коренных обитателей Евразии также отмечены более высокие значения Hd: A. sinica – 0.86, A. urmiana – 0.90, A. tibetiana – 0.98 (Eimanifar, 2014). Можно предположить, что найденные уникальные гаплотипы A. salina будут обнаружены и в других популяциях вида из гиперсоленых водоемов п-ова Крым. Не вызывает сомнений, что при дальнейших более масштабных исследованиях разнообразие гаплотипов существенно увеличится.
Таблица 2.
Популяционно-генетические параметры исследованных A. salina
| N | S | h | Hd | π | k | M |
|---|---|---|---|---|---|---|
| 135 | 109 | 76 | 0.982 | 0.01868 | 9.6 | 115 |
На рис. 1 дана карта ареала обитания A. salina c указанием мест расположения 37 различных локальных популяций, данные по которым приведены в табл. 1. Исследованные в нашей работе особи из двуполых популяций оз. Сасык-Сиваш образовали общую кладу с A. salina из Туниса, Ливии и Кипра (рис. 1: выделенная область на филогенетическом дереве). Анализ данных позволил выделить в структуре гаплотипической сети группу, в которую входят уникальные гаплотипы A. salina из Ливии, Туниса, Египта, Кипра и Крыма, не обнаруженные в западной части средиземноморского региона (рис. 2: пунктирная область). Возможно, места обитания этих уникальных популяций можно отнести к плейстоценовым рефугиумам.
Рис. 2.
Генеалогические связи между COI гаплотипами из популяций A. salina (TCS сеть). Пунктирной линией выделены уникальные гаплотипы из Ливии, Туниса, Египта, Кипра и Крыма. Черные точки – число мутаций; в скобках ‒ число нуклеотидных замен. Жирным курсивом указана популяция озера Сасык-Сиваш (SS).
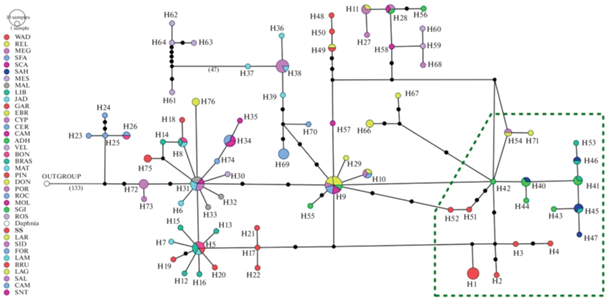
Выделенная группа популяций характеризуется уникальным генетическим разнообразием. Следует отметить, что западно-средиземноморские популяции с множеством общих гаплотипов находятся в одном миграционном коридоре птиц. Можно считать, что между популяциями существует регулярный обмен генами за счет транспортировки цист мигрирующими птицами (Green et al., 2005). Ранее было убедительно показано, что структура миграционных путей птиц – важный фактор, определяющий филогеграфию Artemia (Munoz et al., 2013). Подобное предположение было сделано и для другого вида ракообразных (копеподы Arctodiaptomus salinus (Daday, 1885)), также имеющего покоящиеся яйца и широко распространенного в Средиземноморско-Черноморском регионе (Anufriieva, Shadrin, 2015).
Появление инвазионных видов Artemia, в первую очередь A. monica (=A. franciscana), может повлечь за собой сокращение крымского ареала A. salina и, следовательно, быстрое и значительное снижение генетического разнообразия. Важно отметить, что высокой адаптируемости A. mo-nica (возможно, и других видов Artemia) в новых условиях обитания способствует наличие различных альтернативных паттернов экспрессии генов (De Vos et al., 2021; Lee et al., 2022). Таким образом, наличие альтернативных вариантов реализации генотипа обеспечивает возможность перехода в новых условиях от одних гомеостатических стратегий к другим, что может повышать инвазивность и адаптацию вида к новым местам обитаниям. Однако, исходя из большого разнообразия и временнóй изменчивости гиперсоленых водоемов в Крыму, высокой адаптивной способности видов Artemia и наличие у них цист, можно с высокой степенью вероятности предположить, что все четыре двуполых вида Artemia будут устойчиво сосуществовать с партеногенетическими популяциями в Крыму, сменяя друг друга. Будет ли при этом снижаться генетическое разнообразие аборигенных популяций A. salina, смогут показать лишь дальнейшие многолетние исследования.
Выводы. Изучение филогенетических связей и филогеографической структуры популяций A. salina, проведенное по митохондриальному маркерному гену COI, позволило выделить две разнородные группы популяций. Для западно-средиземноморских популяций характерно наличие множества общих гаплотипов, что может быть обусловлено их расположением в одном миграционном коридоре птиц. Географически изолированные группы популяций из Ливии, Туниса, Египта, Кипра и Крыма характеризуются уникальными гаплотипами, не встречающимися в западной части средиземноморского региона. В крымской популяции артемии зарегистрированы уникальные, предположительно эндемичные для Крыма, гаплотипы. Появление инвазионных видов Artemia, в первую очередь A. monica (=A. franciscana), может повлечь за собой быстрое сокращение крымского ареала A. salina и вызванное этим значительное снижение генетического разнообразия вида.
Список литературы
Балушкина Е.В., Голубков С.М., Голубков М.С. и др. 2009. Влияние абиотических и биотических факторов на структурно-функциональную организацию экосистем соленых озер Крыма // Журн. общ. биол. Т. 70. № 6. С. 504.
Adamowicz S.J., Purvis A. 2005. How many branchiopod crustacean species are there? Quantifying the components of underestimation // Global Ecol. Biogeogr. V. 14. P. 455. https://doi.org/10.1111/j.1466-822X.2005.00164.x
Adamowicz S.J., Menu-Marque S., Hebert P.D., Purvis A. 2007. Molecular systematics and patterns of morphological evolution in the Centropagidae (Copepoda: Calanoida) of Argentina // Biol. J. Linnean Soc. V. 90. P. 279. https://doi.org/10.1111/j.1095-8312.2007.00723.x
Anufriieva E.V., Shadrin N.V. 2015. Morphometric variability of Arctodiaptomussalinus (Copepoda) in the Mediterranean-Black Sea region // Zool. Res. V. 18. № 36(6). P. 328.
Anufriieva E., Kolesnikova E., Revkova T. et al. 2022. Human-Induced Sharp Salinity Changes in the World’s Largest Hypersaline Lagoon Bay Sivash (Crimea) and Their Effects on the Ecosystem // Water. V. 14 (3)403. https://doi.org/10.3390/w14030403
De Gelas K., De Meester L. 2005. Phylogeography of Daphnia magna in Europe // Mol. Ecol. V. 14. P. 753. https://doi.org/10.1111/j.1365-294X.2004.02434.x
De Meester L., Gómez A., Okamura B., Schwenk K. 2002. The Monopolization hypothesis and the dispersal-gene flow paradox in aquatic organisms // Acta Oecologica. V. 23. P. 121. https://doi.org/10.1016/S1146-609X(02)01145-1
De Vos S., Rombauts S., Coussement L. et al. 2021. The genome of the extremophile Artemia provides insight into strategies to cope with extreme environments // BMC Genom. V. 22 (1). P. 1. https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-021-07937-z
Eimanifar A., Van Stappen G., Marden B., Wink M. 2014. Artemia biodiversity in Asia with the focus on the phylogeography of the introduced American species Artemia franciscana Kellogg, 1906. // Mol. Phylogen. and Evol. V. 79. P. 392. https://doi.org/10.1016/j.ympev.2014.06.027
Frisch D., Green A.J., Figuerola J. 2007. High dispersal capacity of a broad spectrum of aquatic invertebrates via waterbirds // Aquat. Sci. V. 69(4). P. 568. https://doi.org/10.1007/s00027-007-0915-0
Fontaneto D. 2019. Long-distance passive dispersal in microscopic aquatic animals // Mol. Ecol. V. 7. P. 10. https://doi.org/10.1186/s40462-019-0155-7
Green A.J., Sánchez M.I., Amat F. et al. 2005. Dispersal of invasive and native brine shrimps Artemia (Anostraca) via waterbirds // Limnol., Oceanogr. V. 50. P. 737.
Gómez A., Carvalho G.R., Lunt D.H. 2000. Phylogeography and regional endemism of a passively dispersing zooplankter: mitochondrial DNA variation in rotifer resting egg banks // Proc. Royal Soc. Series B. V. 267. P. 2189. https://doi.org/10.4319/lo.2005.50.2.0737
Gómez A., Serra M., Carvalho G.R., Lunt D.H. 2002. Speciation in ancient cryptic species complexes: evidence from the molecular phylogeny of Brachionus plicatilis (Rotifera) // Evolution. V. 56. P. 1431. https://doi.org/10.1111/j.0014-3820.2002.tb01455.x
Gómez A., Montero-Pau J., Lunt D.H. et al. 2007. Persistent genetic signatures of colonization in Brachionus manjavacas rotifers in the Iberian Peninsula // Mol. Ecol. V. 16. P. 3228. https://doi.org/10.1111/j.1365-294X.2007.03372.x
Hebert P.D. 1998. Variable environments and evolutionary diversification in inland waters // Adv. Mol. Ecol. P. 267.
Hebert P.D., Witt J.D., Adamowicz S.J. 2003. Phylogeographical patterning in Daphnia ambigua: Regional divergence and intercontinental cohesion // Limnol., Oceanogr. V. 48. P. 261. https://doi.org/10.4319/lo.2003.48.1.0261
Hessen D.O., Jensen T.C., Walseng B. 2019. Zooplankton diversity and dispersal by birds; insights from different geographical scales // Frontiers in Ecol. and Evol. V. 20. P. 7. https://doi.org/10.3389/fevo.2019.00074
Hall T., Biosciences I., Carlsbad C. 2011. BioEdit: an important software for molecular biology // GERF Bull Biosci. V. 2(1) P. 60.
Ishida S., Taylor D.J. 2007. Mature habitats associated with genetic divergence despite strong dispersal ability in an arthropod // BMC Evol. Biol. V. 7. P. 52. https://doi.org/10.1186/1471-2148-7-52
Kumar S., Stecher G., Li M. et al. 2018. MEGA X: molecular evolutionary genetics analysis across computing platforms // Mol. Biol. Evol. V. 35(6). P. 1547. https://doi.org/10.1093/molbev/msy096
Lantushenko A., Meger Y., Gadzhi A. et al. 2022. Artemia spp. (Crustacea, Anostraca) in Crimea: New Molecular Genetic Results and New Questions without Answers // Water. V. 14(17). P. 2617. https://doi.org/10.3390/w14172617
Le J., Cho B.C., Park J.S. 2022. Transcriptomic analysis of brine shrimp Artemia franciscana across a wide range of salinities // Mar. Genom. V. 61: 100919. https://doi.org/10.1016/j.margen.2021.100919
Leigh J.W., Bryant D. 2015. POPART: full-feature software for haplotype network construction // Meth. Ecol. and Evol. V. 6. № 9. P. 1110. https://doi.org/10.1111/2041-210X.12410
Marden B., Brown P., Bosteels T. 2020. Great Salt Lake Artemia: ecosystem functions and services with a global reach // Great Salt Lake Biol. P. 175. https://doi.org/10.1080/10454438.2018.1484838
Mergeay J., Verschuren D., De Meester L. 2005. Cryptic invasion and dispersal of an American Daphnia in East Africa // Limnol., Oceanogr. V. 50. P. 1278. https://doi.org/10.4319/lo.2005.50.4.1278
Munoz J., Gomez A., Green A.J. et al. 2008. Phylogeography and local endemism of the native Mediterranean brine shrimp Artemia salina (Branchiopoda: Anostraca) // Mol. Ecol. V. 17(13). P. 3160. https://doi.org/10.1111/j.1365-294X.2008.03818.x
Muñoz J., Amat F., Green A.J. et al. 2013. Bird migratory flyways influence the phylogeography of the invasive brine shrimp Artemia franciscana in its native American range // Peer J. V. 1. P. 200. https://doi.org/10.7717/peerj.200
Naceur H.B., Romdhan M.S., Stappen G.V. 2020. Potential Use of fatty acid profile for Artemia spp. discrimination // Inland Water Biol. V. 13. № 3. P. 434. https://doi.org/10.1134/S199508292003013X
Paland S., Colbourne J.K., Lynch M. 2005. Evolutionary history of contagious asexuality in Daphnia pulex // Evolution. V. 59. P. 800. https://doi.org/10.1111/j.0014-3820.2005.tb01754.x
Penton E.H., Hebert P.D., Crease T.J. 2004. Mitochondrial DNA variation in North American populations of Daphnia obtusa: continentalism or cryptic endemism? // Mol. Ecol. V. 13. P. 97. https://doi.org/10.1046/j.1365-294X.2003.02024.x
Ronquist F., Teslenko M., Van Der Mark P. et al. 2012. MrBayes 3.2: Efficient Bayesian phylogenetic inference and model choice across a large model space // Syst. Biol. V. 61. P. 539. https://doi.org/10.1093/sysbio/sys029
Rozas J. 2017. DnaSP 6: DNA sequence polymorphism ana-lysis of large data sets // Mol. Biol. Evol. V. 34. P. 3299. https://doi.org/10.1093/molbev/msx248
Sainz-Escudero L., López-Estrada E.K., Rodríguez-Flores P.C., García-París M. 2021. Settling taxonomic and nomenclatural problems in brine shrimps, Artemia (Crustacea: Branchiopoda: Anostraca), by integrating mitogenomics, marker discordances and nomenclature rules // Peer J. V. 9. P. 10865. https://doi.org/10.7717/peerj.10865
Sainz-Escudero L., López-Estrada E.K., Rodríguez-Flores P.C., García-París M. 2022. Brine shrimps adrift: Historical species turnover in Western Mediterranean Artemia (Anostraca) // Biol. Invasions. V. 24. P. 2477. https://doi.org/10.1007/s10530-022-02779-6
Sanchez M.I., Paredes I., Lebouvier M., Green A.J. 2016. Functional role of native and invasive filter-feeders, and the effect of parasites: learning from hypersaline ecosystems // PLoS One. V. 11(8). e0161478. https://doi.org/10.1371/journal.pone.0161478
Shadrin N., Stetsiuk A., Anufriieva E. 2022. Differences in mercury concentrations in water and hydrobionts of the crimean saline lakes: does only salinity matter? // Water. V. 14(17): e2613. https://doi.org/10.3390/w14172613
Templeton A., Crandall K., Sing C. 1992. A cladistic analysis of phenotypic associations with haplotypes inferredfrom restriction endonuclease mapping and DNA sequencedata. III. Cladogram estimation // Genetics. № 132. P. 619. https://doi.org/10.1093/genetics/132.2.619
Van Stappen G., Sui L., Hoa V.N. et al. 2020. Review on integrated production of the brine shrimp Artemia in solar salt ponds // Rev. Aquac. V. 12. P. 1054. https://doi.org/10.1111/raq.12371
Weider L.J., Hobaek A., Hebert P.D., Crease T.J. 1999. Holarctic phylogeography of an asexual species complex-II. Allozymic variation and clonal structure in Arctic Daphnia // Mol. Ecol. V. 8. P. 1. https://doi.org/10.1046/j.1365-294X.1999.00522.x
Zierold T., Hanfling B., Gómez A. 2007. Recent evolution of alternative reproductive modes in the “living fossil” Triops cancriformis // BMC Evol. Biol. V. 7. P. 161. https://doi.org/10.1186/1471-2148-7-161
Дополнительные материалы отсутствуют.
Инструменты
Биология внутренних вод


