Доклады Российской академии наук. Науки о жизни, 2020, T. 493, № 1, стр. 417-422
ОПЫТ ПОСТРОЕНИЯ ФИЛОГЕНИИ ЛИСТОНОСОВ РОДА HIPPOSIDEROS ПО ЯДЕРНЫМ ГЕННЫМ МАРКЕРАМ
А. П. Юзефович 1, И. В. Артюшин 1, А. А. Распопова 1, А. А. Банникова 1, С. В. Крускоп 2, *
1 Биологический факультет Московского государственного университета
имени М.В. Ломоносова
Москва, Россия
2 Зоологический музей Московского государственного университета имени М.В. Ломоносова
Москва, Россия
* E-mail: selysius@mail.ru
Поступила в редакцию 23.03.2020
После доработки 14.04.2020
Принята к публикации 15.04.2020
Аннотация
Род Hipposideros – один из крупнейших родов рукокрылых. Новейшие исследования применительно к его азиатским представителям были сделаны только на митохондриальных маркерах либо на единичных ядерных. В настоящей работе, используя выборку, включающую все основные группы видов, мы сравнили результаты филогенетических построений, основанные на митохондриальных, на единичных ядерных генах и на множественных ядерных маркерах, проанализированных с использованием алгоритма MRP (Matrix representation with parsimony). Последние дают наиболее поддержанную и хорошо интерпретируемую филогенетическую реконструкцию. Была показана монофилия крупных азиатских листоносов, претендующих на самостоятельный таксон ранга подрода, а также монофилия ряда видовых комплексов, парафилетических по данным анализа мтДНК.
ВВЕДЕНИЕ
Hipposideros – центральный род в семействе Hipposideridae и один из крупнейших родов млекопитающих, включающий не менее 65 современных видов [1]. Большинство авторов используют систему, предложенную Хиллом [2], в рамках которой род был поделен на несколько групп видов, с очень приблизительной оценкой иерархии взаимоотношений между ними. Недавние молекулярно-генетические исследования [3, 4] подтвердили монофилию рода Hipposideros (в каковой были сомнения [5, 6]), при условии возведения групп видов “commersoni” и “cyclops” в ранг самостоятельных родов – Macronycteris и Doryrhina соответственно. Единственная работа, основанная на множественном использовании ядерных генов [4] сфокусирована на специфических африканских линиях и практически не уделяет внимания структуре собственно рода Hipposideros, особенно его азиатских представителей. Таким образом, филогенетическая структура рода остается малоизученной. В то же время результаты генного баркодинга [7] и частные исследования отдельных групп видов (напр. [8, 9]) показывают, что и видовое разнообразие рода недооценено.
Располагая материалом по большинству групп видов рода Hipposideros (в первую очередь – из Юго-Восточной Азии), мы построили схему филогенетических отношений по нескольким генетическим маркерам.
МАТЕРИАЛЫ И МЕТОДЫ
Геномную ДНК выделяли из фиксированных в этаноле проб тканей стандартным методом фенол-хлороформной депротеинизации [10]. Были получены последовательности митохондриального гена цитохрома b (cytb; 1129 п.о.) и фрагменты семи ядерных генов (ABHD11, 460 bp; ACOX2, 569 bp; COPS, 722 bp; RAG2, 1035 bp; ROGDI2, 501 bp; SORBS2, 594 bp; и THY, 546 bp). Выбор этих ядерных маркеров определен их вариабильностью и информативностью при изучении альфа-таксономии млекопитающих [11]. Для cytb и SORBS2 были использованы оригинальные праймеры (H_cyt_Rh: TAGAATTTCAGCTTTGGGTGCT, L_cyt_Rh: AAACCACGACCAATGAC-ATGAAA, SORBS2_L730a: CCATCGAGTCTYTGCTGGAGGAAG и SORBS2_R1300a: CCTTCTCTTTTCAGC-CTTAATYTGGTCAC). Для остальных ядерных маркеров последовательности праймеров были взяты из опубликованных работ [11–13]. Реакция амплификации включала 35 циклов и осуществлялась при следующих условиях: денатурация – 94°C в течение 30 с, отжиг – 59°C (для cytb) и 60°C (для ядерных генов) в течение 1 мин, синтез – 72°C в течение 1 мин. Конечный синтез осуществлялся при 72°C в течение 6 мин. Опыты выполнены на приборе My Cycler BioRAD. Контроль амплификации проводили в 1% агарозном геле. Очистку продуктов ПЦР проводили с помощью набора Diatom DNA Clean-Up. Автоматическое секвенирование проведено на секвенаторе ABI PRISM 3500xl. Всего получено 34 последовательности cytb и 213 последовательностей ядерных генов для 19 видов; виды родственного рода Aselliscus взяты в качестве внешней группы.
Последовательности выровнены с помощью программ BioEdit v7.1.3. и SeqMan Pro v.7.1. и скорректированы вручную. Реконструкция филогенетических деревьев выполнена методами максимального правдоподобия (ML) и максимальной экономии (MP) в программах PAUP* 4.0b10 и MEGA 5 и методом байесова анализа (BA) с помощью программы MrBayes 3.1.2. Далее выборка частных деревьев, полученных ML методом, была объединена в супердерево с использованием алгоритма MRP [14] при помощи программы RAxML (v8.2.9) с моделью GTRGAMMA. Для определения достоверности полученной топологии использовали процедуру “stratified bootstrap” со 100 репликами.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Наши данные по гену cytb в целом согласуются с ранее опубликованными (напр. [3]). Топологии деревьев, построенных методами Байеса и ML, не имели существенных различий. Монофилия рода (относительно Aselliscus) поддержана на дереве, построенном методом Байеса (рис. 1).
Рис. 1.
Дерево филогенетических отношений внутри рода Hipposideros по последовательностям митохондриального гена cyt b, построенное методом Байеса (апостериорные вероятности получены для цепи в 6000000 генераций).
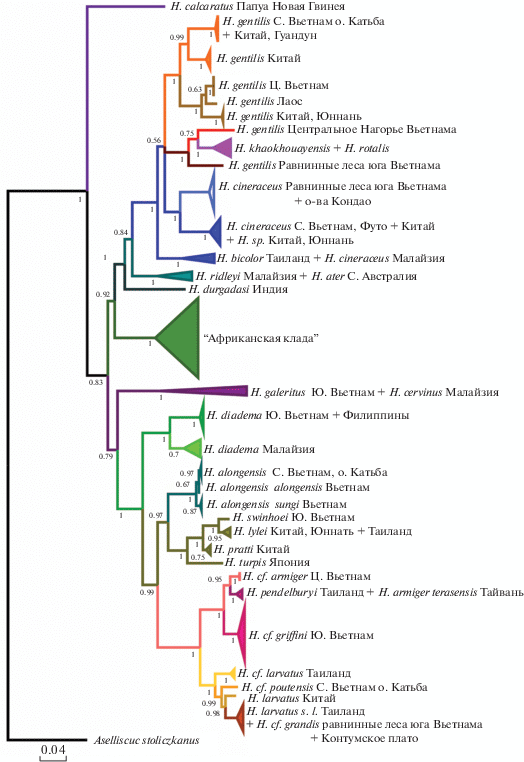
Монофилия всех крупных форм рода имеет поддержки на нижней границе значимых. Среди крупных листоносов базальное положение занимает H. diadema. Вид H. alongensis занял сестринское положение к группе “swinhoei” (а не к “armiger”, как традиционно считали). H. armiger из Вьетнама оказался сестринским к H. pendleburyi (Таиланд) и H. armiger terasensis (Тайвань). В комплексе “larvatus” базальное положение заняла мелкая форма из Таиланда. Также отличия уровня вида от прочих включенных в анализ форм имеет H. cf. poutensis (из Северного Вьетнама). Популяции из Южного и Центрального Вьетнама, относимые к H. grandis, объединены в компактный кластер с другой формой из Таиланда.
Среди мелких листоносов многие клады оказались не поддержанными. Базальное положение ко всему роду занял H. calcaratus. Африканские H. ruber, H. cafer и H. fuliginosus в составе “Африканской клады” и азиатские H. galeritus, H. cervinus образуют хорошо поддержанные монофилетические ветви. Группа “bicolor” в узком понимании имеет хорошие поддержки только методом Байеса. Базальное положение в ней занял индийский H. durgadasi. Два распространенных вида, H. gentilis и H. cineraceus, парафилетичны: H. gentilis из равнинных лесов юга Вьетнама и с Центрального Нагорья занимают сестринское положение к H. khaokhouayensis, а H. cineraceus из Малайзии – к H. bicolor s. str. из Таиланда.
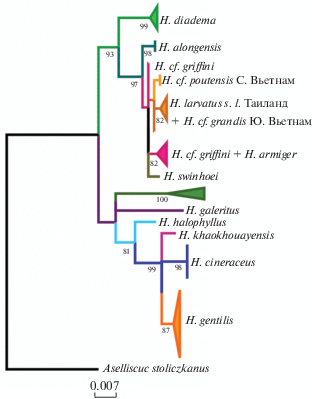
Большинство ядерных генов, проанализированных по-отдельности, не дали деревьев с хорошо поддержанной топологией. Узлы внутривидовых дивергенций по отдельным ядерным генам не имеют значимых поддержек, как не имеет поддержек и монофилия рода в целом (относительно Aselliscus). Наиболее информативные деревья (большинство узлов которых, однако, также не имеет значимых поддержек) дают интрон THY (рис. 2) и экзон RAG2.
Рис. 2.
Дерево филогенетических отношений внутри рода Hipposideros по данным ядерного интрона THY, построенное методом Максимального подобия (ML). Под узлами ветвей указаны значения бутстреп-поддержек (получены из 1000 реплик).
Дерево, полученное для семи ядерных генов методом MRP, разрешило все узлы дивергенции надвидового уровня с максимальными поддержками (рис. 3), в том числе и базальное положение африканского H. jonesi. Все крупные азиатские листоносы формируют монофилетичную кладу. Среди них самое базальное положение занимает H. diadema. Ветка H. alongensis отходит базальнее, чем H. swinhoei. Последний занимает сестринское положение к кладе “armiger/larvatus”. В отличие от данных по митохондриальной ДНК, разделение H. armiger и H. griffini внутри данной клады не поддержано. Клады, соответствовавшие видовым комплексам “armiger” и “larvatus” имеют максимальные поддержки. Из них вторая имеет хорошо поддержанную внутреннюю дивергенцию, возможно, видового уровня. В частности, обособлена мелкая форма из северного Вьетнама (H. cf. poutensis) (рис. 4а).
Рис. 3.
Обобщенное дерево филогенетических отношений внутри рода Hipposideros, полученное по 7 ядерным генам методом Matrix Representation Parsimony (MRP). Виды рода Aselliscus использованы в качестве внешней группы. Под узлами ветвей указаны значения бутстреп-поддержек (получены из 1000 реплик).
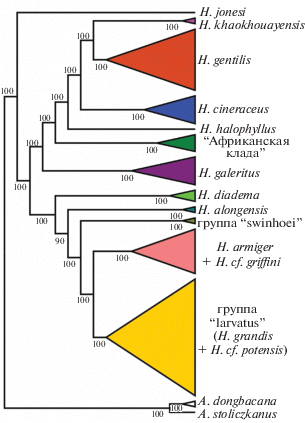
Рис. 4.
Фрагменты филогенетического дерева, полученного по 7 ядерным генам методом MRP: а) филогенетические отношения форм комплекса “larvatus”; б) филогенетические отношения вьетнамских форм H. gentilis. Под узлами ветвей указаны значения бутстреп-поддержек (получены из 1000 реплик).
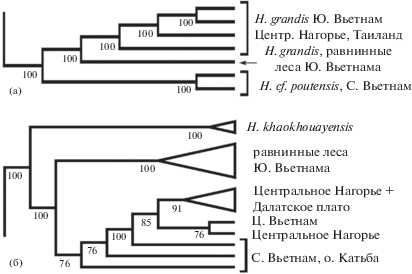
Предполагавшееся родство африканских мелких листоносов и группы “galeritus” [2] на MRP-дереве не поддержано, однако родство обеих линий с группой “bicolor” несомненно. При этом азиатский H. galeritus образует более базальную кладу, чем африканские виды. Оба широко распространенных в Индокитае вида, парафилетичные по данным митохондриальной ДНК, формируют монофилетические клады. При этом H. gentilis из равнинных лесов Южного Вьетнама формируют в своей кладе наиболее базальную ветвь (рис. 4б).
ЗАКЛЮЧЕНИЕ
К сожалению, на настоящий момент не от всех видов, проанализированных по митохондриальным генам, удалось получить достаточный материал для включения их в итоговое MRP-дерево. Однако даже предварительные результаты показывают эффективность использованного подхода. Показано, что результаты анализа отдельных ядерных генов могут быть использованы лишь как аргументы “за” или “против” в решении вопросов филогении рукокрылых, однако объединение топологий нескольких маркеров дает имеющую высокие поддержки и хорошо интерпретируемую филогенетическую гипотезу.
В более частных аспектах подтверждено, что крупные азиатские листоносы представляют собой монофилетичную группу, заслуживающую ранг подрода, для которого в качестве действительного названия может быть использовано Phyllorhina Bonaparte 1837. Окончательное решение о названии требует изучения H. speoris, типового для Hipposideros, который пока не был вовлечен в молекулярные исследования, но особенности его морфологии [15] позволяют предполагать родство с мелкими, а не с крупными листоносами. Положение на дереве H. galeritus позволяет предположить, что мелкие африканские листоносы происходят от азиатских предков. Вид H. alongensis, вопреки ожиданиям, не является частью комплекса “larvatus/armiger”. Для комплексов форм, считавшихся парафилетическими (H. larvatus s. lato, H. gentilis и H. cineraceus), показано существование монофилетических клад. В качестве причины противоречий можно подозревать древние интрогрессии митохондриальной ДНК (известной и в других семействах рукокрылых; напр. [13]). Детальное рассмотрение этих ситуаций – предмет отдельных публикаций с применением большего массива генетических данных, включая данные по Y хромосоме, зарекомендовавшие себя в решении сложных таксономических ситуаций альфа-уровня.
Список литературы
Monadjem A., Soisook P., Thong V.D., et al. Family Hipposideridae (Old World Leaf-nosed Bats) / Eds. Wilson D.E., Mittermeier R.A. Handbook of the Mammals of the World, Volume 9: Bats. Barcelona: Lynx Edicions, 2019. 1008 p.
Hill J.E. A revision of the genus Hipposideros // Bulletin of British Museum (Natural History). 1963. V. 11. № 1. P. 1–129.
Murray S.W., Campbell P., Kingston T., et al. Molecular phylogeny of hipposiderid bats from Southeast Asia and evidence of cryptic diversity // Molecular Phylogeny and Evolution. 2012. V. 62. P. 597–611.
Foley N.M., Goodman S.M., Whelan C.V., et al. Towards navigating the Minotaur’s labyrinth: cryptic diversity and taxonomic revision within the speciose genus Hipposideros (Hipposideridae) // Acta Chiropterologica. 2017. V. 19. № 1. P. 1–18.
Bogdanowicz W., Owen R.D. In the Minotaur’s labyrinth: The phylogeny of the bat family Hipposideridae / Eds. Kunz T.H., Racey P.A. Bat biology and conservation. Washington: Smithsonian Institution Press, 1998.
Wang H., Liang B., Feng J., et al. Molecular phylogenetic of Hipposiderids (Chiroptera: Hipposideridae) and Rhinolophids (Chiroptera: Rhinolophidae) in China based on mitochondrial cytochrome b sequences // Folia Zoologica. 2003. V. 52. № 3. P. 259–268.
Francis C.M., Borisenko A.V., Ivanova N.V., et al. The Role of DNA Barcodes in Understanding and Conservation of Mammal Diversity in Southeast Asia // PLoS ONE, 2010. V. 5. № 9. e12575.
Guillen-Servent A., Francis C.M. A new species of bat of the Hipposideros bicolor group (Chiroptera: Hipposideridae) from Central Laos, with evidence of convergent evolution with Sundaic taxa // Acta Chiropterologica. 2006. V. 8. № 1. P. 39–61.
Kruskop S.V. Dull and bright: cryptic diversity within the Hipposideros larvatus group in Indochina (Chiroptera: Hipposideridae) // Lynx, nová série (Praha). 2015. V. 46. P. 29–42.
Sambrook J., Fritsch E.F., Maniatis T. Molecular cloning – a laboratory manual. Sec. ed. N.Y.: Cold Spring Harbour Lab. Press, 1989. 385 p.
Igea J., Juste J., Castresana J. Novel intron markers to study the phylogeny of closely related mammalian species // BMC Evolutionary Biology. 2010. V. 10. № 369. P. 1–13.
Matthee C.A., Burzlaff J.D., Taylor J.F., et al. Mining the mammalian genome for artiodactyl systematics // Systematic Biology. 2001. V. 50. P. 1–24.
Artyushin I.V., Bannikova A.A., Lebedev V.S., et al. Mitochondrial DNA relationships among North Palaearctic Eptesicus (Vespertilionidae, Chiroptera) and past hybridization between Common Serotine and Northern Bat // Zootaxa. 2009. V. 2262. P. 40–52.
Ragan M.A. Matrix representation in reconstructing phylogenetic relationships among the eukaryotes // Biosystems. 1992. V. 28. № 1. P. 47–55.
Dar T.H., Kamalakannan M., Venkatraman C., et al. New record of Hipposideros speoris (Chiroptera: Hipposideridae) from Myanmar hidden in the National Zoological Collections of the Zoological Survey of India // Mammalia. 2018. V. 83. № 5. P. 513–515.
Дополнительные материалы отсутствуют.
Инструменты
Доклады Российской академии наук. Науки о жизни


