Доклады Российской академии наук. Науки о жизни, 2022, T. 503, № 1, стр. 166-171
Прогностическая значимость белков-регуляторов сплайсинга РНК для пациентов с глиобластомой
Т. Д. Ларионова 1, Т. Ф. Коваленко 1, М. И. Шахпаронов 1, М. С. Павлюков 1, *
1 Государственный Научный Центр ФГБУ ИБХ РАН
им. академиков М.М. Шемякина и Ю.А. Овчинникова
Москва, Россия
* E-mail: marat.pav@mail.ru
Поступила в редакцию 25.11.2021
После доработки 13.12.2021
Принята к публикации 13.12.2021
- EDN: KSQAGM
- DOI: 10.31857/S2686738922020135
Аннотация
Глиобластома (GBM) считается одной из наиболее агрессивных опухолей человека. Ранее нашей группой была продемонстрирована важная роль альтернативного сплайсинга РНК в регуляции фенотипа GBM. Чтобы дополнить это исследование, мы проанализировали тип сплайсинга РНК и уровни экспрессии сплайсосомных генов в большом количестве образцов опухолевой ткани и первичных культур нейросфер GBM. Мы показали, что, основываясь на экспрессии факторов сплайсинга, можно разделить пациентов на группы, характеризующиеся различной выживаемостью, а также предположить фенотип опухоли. Кроме того, мы выявили события альтернативного сплайсинга, которые могут играть важную роль в регуляции фенотипа клеток GBM. Наконец, мы впервые исследовали экспрессию сплайсосомных генов в различных частях одной и той же опухоли, а также выявили регуляторы сплайсинга, количество которых наиболее тесно связано с выживаемостью пациентов. Перечисленные выше данные подчеркивают важную роль сплайсинга пре-РНК в прогрессии GBM.
Глиобластома является наиболее распространенной первичной опухолью головного мозга у взрослых пациентов. Несмотря на громадные усилия, направленные на борьбу с этим заболеванием, существующие методы лечения глиобластом в основном носят лишь паллиативный характер и позволяют увеличить среднюю выживаемость пациентов с трех месяцев (без лечения) до двух лет (при условии максимального хирургического удаления опухоли с последующей химио- и радиотерапией) [1, 2]. Причина столь негативного исхода кроется в чрезвычайной внутри- и меж- опухолевой гетерогенности. Так, на основании генетических мутаций, клетки глиобластомы делят на IDH-мутантные и IDH дикого типа [3]; на основании метилирования ДНК опухоли разделяют на CIMP+ и CIMP- [4]; наконец, по уровню экспрессии генов глиобластомы могут относиться к классическому, мезенхимальному или пронейрональному фенотипам [5]. Для каждой из этих многочисленных групп характерно свое течение болезни – различная скорость роста опухоли, чувствительность к терапии и, как следствие, различная выживаемость пациентов. Таким образом, для того чтобы достичь наилучших результатов в лечении, крайне важно правильно и своевременно определить молекулярный фенотип опухоли и, исходя из этого, выбрать лекарственный препарат, который будет оптимален для лечения каждого конкретного пациента.
Ранее нашей и другими группами были получены результаты, говорящие о том, что важную роль в формировании гетерогенности глиобластом играет то, каким именно образом осуществляется сплайсинг молекул пре-мРНК в опухолевых клетках [6]. Например, тип сплайсинга Ras и CyclinD1 определяет скорость пролиферации опухолевых клеток [7], альтернативный сплайсинг PKM изменяет тип метаболизма [8], а изоформы белков MDM4 и MDM2 регулируют чувствительность опухоли к терапии [9, 10]. Однако факторы, обусловливающие различия в сплайсинге пре-мРНК между опухолевыми клетками, роль изменений сплайсинга в прогрессии заболевания, а также механизмы влияния сплайсинга на фенотип клеток глиобластомы, до сих пор не ясны.
Для того, чтобы определить белки-регуляторы сплайсинга, наиболее сильно связанные с основными свойствами глиом низкой степени злокачественности (степени II–III по классификации ВОЗ) и глиобластом (степень IV), мы решили провести биоинформатический анализ общедоступных данных секвенирования РНК из базы данных TCGA (The Cancer Genome Atlas), а также осуществить секвенирование РНК из образцов опухолевой ткани и первичных культур клеток глиобластомы, собранных нашей лабораторией за последние 5 лет.
Мы начали наш анализ с определения белков, вовлеченных в сплайсинг пре-мРНК. С помощью баз данных KEGG и NCBI мы создали список из 197 белков, предположительно способных влиять на сплайсинг. Необходимо отметить, что этот список, вероятно, является несколько избыточным, так как, исходя из анализа структуры белков и белок-белковых взаимодействий, лишь 50–150 белков в клетке могут регулировать сплайсинг напрямую [11, 12].
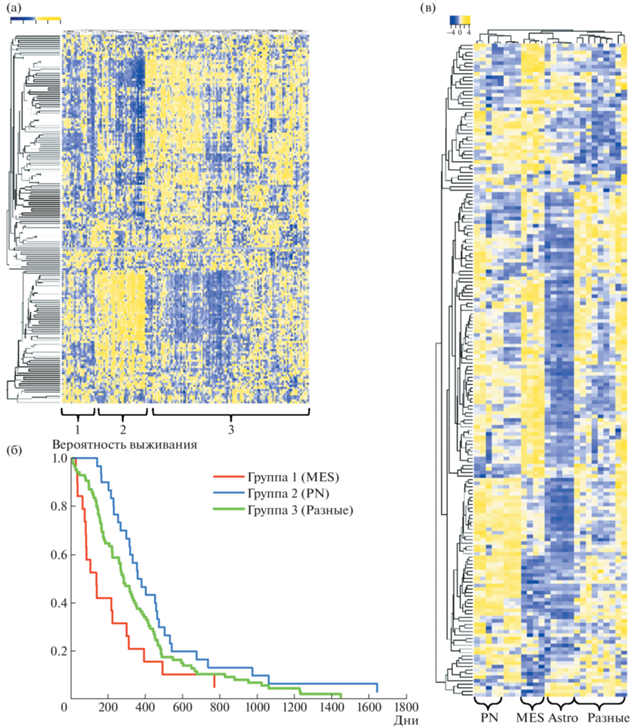
Проанализировав уровни экспрессии генов сплайсосомных белков у более чем 150 пациентов с глиобластомами, мы показали, что все глиобластомы можно разделить на несколько групп по экспрессии сплайсинговых факторов (рис. 1а). В первую группу, в основном, входят мезенхимальные опухоли (более 75%), и она характеризуется наихудшим прогнозом в отношении выживаемости пациентов (рис. 1б). Вторая группа состоит преимущественно из опухолей пронейронального или нейронального фенотипов (более 70%) и, наоборот, характеризуется наибольшим сроком жизни больных. Для оставшихся образцов на данном этапе работы нам не удалось выявить ярких отличительных особенностей. Далее, мы провели секвенирование транскриптома первичных культур клеток глиобластомы (18 линий), человеческих астроцитов (5 линий) и предшественников нервных клеток (3 линии). Рисунок 1в показывает, что все эти группы клеток хорошо отличимы друг от друга по уровню экспрессии генов сплайсосомных белков. В данном эксперименте легко выделялись отдельные кластеры пронейрональных и мезенхимальных клеток, а также группа, содержащая плохо характеризуемые линии. Эти данные хорошо согласуются с результатами нашего анализа базы данных TCGA.
Рис. 1.
Связь экспрессии сплайсосомных генов с фенотипом глиобластомы. (а) Тепловая карта, отображающая экспрессию сплайсосомных генов у пациентов с глиобластомой. (б) Кривые Каплана–Майера, показывающие выживаемость пациентов c GBM, разделенных на группы исходя из экспрессии сплайсосомных генов. (В) Тепловая карта, отображающая экспрессию сплайсосомных генов в первичных культурах нейросфер глиобластомы с различным фенотипом (PN, MES, Разные) и человеческих астроцитов (Astro).
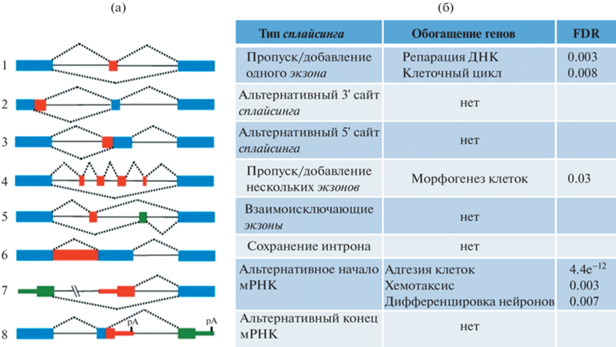
Чтобы понять, как наблюдаемые нами изменения экспрессии сплайсосомных генов влияют на процесс сплайсинга пре-мРНК, мы сравнили события альтернативного сплайсинга, обнаруженные с помощью секвенирования транскриптомов пронейрональных и более агрессивных мезенхимальных первичных культур клеток глиобластомы. В общей сложности мы обнаружили 3370 событий альтернативного сплайсинга, статистически значимо отличающихся между двумя группами образцов. Эти события затрагивали такие важные опухоль-ассоциированные гены, как MDM2, MDM4, FoxM1, hRAS, Notch1, ATM, TGM2, Drosha и другие. Далее мы разделили все детектированные изменения сплайсинга на 8 типов: (1) измененный 3' сайт сплайсинга, (2) измененный 5' сайт сплайсинга, (3) альтернативный конец мРНК, (4) альтернативное начало мРНК (строго говоря, это изменение не относится к сплайсингу, а является следствием использования альтернативного промотора), (5) пропуск/добавление одного экзона, (6) пропуск/добавление нескольких экзонов, (7) взаимоисключающие экзоны, (8) сохранение интрона (рис. 2а). Анализ обогащения генов в каждом из восьми типов событий сплайсинга показал, что для групп 1, 2, 3, 7 и 8 значимого обогащения какими-либо генами не наблюдается, однако для группы 4 мы обнаружили существенное обогащение генами, ответственными за адгезию клеток, хемотаксис и нейрональную дифференцировку. Группа 5 была обогащена транскриптами, участвующими в репарации ДНК и регуляции клеточного цикла, а группа 6 генами, кодирующими белки клеточного морфогенеза (рис. 2б).
Рис. 2.
Различия в сплайсинге пре-мРНК между пронейрональными и мезенхимальными клетками глиобластомы. (а) Схематичное изображение различных типов событий альтернативного сплайсинга. (б) Анализ обогащения генов, мРНК которых по-разному сплайсирована в пронейрональных и мезенхимальных клетках глиобластомы.
Этот результат хорошо согласуется с фенотипическими отличиями пронейрональных и мезенхимальных клеток глиобластомы. Так, ранее нами было показано, что мезенхимальные клетки обладают повышенной скоростью миграции и инвазивностью, большей устойчивостью к повреждающим ДНК химиотерапевтическим препаратам, ускоренной пролиферацией, а также измененной клеточной морфологией [13]. Таким образом, можно утверждать, что сплайсинг пре-мРНК, действительно, определяет некоторые фенотипические особенности клеток глиобластомы.
Получив данные об экспрессии сплайсосомных генов в опухолях от разных пациентов, мы перешли к анализу уровней регуляторов сплайсинга РНК внутри одной опухоли. Ранее нами были продемонстрированы большие фенотипические различия между клетками из центра и периферии глиобластомы [14]. По этой причине мы сравнили уровни экспрессии генов-регуляторов сплайсинга в парных образцах опухолевой ткани, полученной из края и центра опухолей от 3 различных пациентов. Проанализировав данные РНК-секвенирования, мы показали, что центральная и периферическая части опухоли сильно отличаются по профилю экспрессии сплайсинговых факторов. В соответствии с этим исследованные образцы кластеризуются друг с другом не по тому, от какого пациента они были получены, а по тому, к какой зоне глиобластомы они относятся (рис. 3а). Также, интересно отметить, что, согласно нашим данным, в центральной зоне опухоли присутствует значительно большее разнообразие сплайсосомных белков, чем в клетках, инфильтрирующих нормальный мозг.
Рис. 3.
Влияние белков-регуляторов сплайсинга РНК на выживаемость пациентов с глиобластомой. (а) Тепловая карта, отображающая экспрессию сплайсосомных генов в опухолевой ткани, полученной из центра (красный) или периферии (зеленый) глиобластомы от трех различных пациентов (указан номер пациента). (б) Диаграмма, иллюстрирующая факторы сплайсинга, экспрессия которых статистически значимо коррелирует с выживаемостью пациентов с глиобластомой и глиомой II–III стадии. Красными точками обозначены сплайсосомные гены, для которых низкий уровень экспрессии коррелирует с плохим прогнозом для выживаемости пациентов с GBM. Синими точками обозначены сплайсосомные гены, для которых высокий уровень экспрессии коррелирует с плохим прогнозом для выживаемости пациентов.

В заключение мы проанализировали базу данных TCGA с целью установить сплайсинговые факторы, экспрессия которых статистически достоверно связана с выживаемостью пациентов с глиомой II–III стадии и глиобластомой. Из рис. 3б видно, что для глиобластомы было обнаружено 27 сплайсосомных генов, экспрессия которых может служить прогностическим фактором для пациентов. Интересно, что для глиомы II–III стадии таких генов оказалось в 4 раза больше (109). Также стоит отметить, что для большинства сплайсинговых факторов (21 из 27) именно пониженная экспрессия соответствующего гена была связана с плохой выживаемостью пациентов. Этот результат хорошо согласуется с гипотезой о том, что для опухолевых и стволовых клеток характерен пониженный уровень контроля сплайсинга пре-мРНК, что позволяет увеличить разнообразие белковых изоформ в клетке, и таким образом поддерживать плюрипотентные свойства. Также эти данные являются важным подтверждением наших ранее опубликованных результатов о том, что для более эффективного противодействия терапии опухолевые клетки используют разнообразные механизмы для понижения уровня сплайсосомных белков [15].
Суммируя результаты, приведенные выше, можно утверждать, что белки-регуляторы сплайсинга играют чрезвычайно важную роль в определении фенотипа клеток глиобластомы, и, теоретически, могут быть использованы для предсказания течения заболевания пациентов с глиобластомой, однако, очевидно, необходимы дальнейшие исследования для выявления функций конкретных сплайсосомных белков.
Список литературы
Stupp R., Mason W.P., van den Bent M.J., et al. // N. Engl. J. Med. 2005. V. 352. № 10. P. 987–996.
Walid M.S. // Perm. J. 2008. V. 12. № 4. P. 45–48.
Brat D.J., Verhaak R.G.W., Aldape K.D., et al. // N. Engl. J. Med. 2015. V. 372. P. 2481–2498.
Malta T.M., de Souza C.F., Sabedot T.S., et al. // Neuro-oncol. 2018. V. 20. № 5. P. 608–620.
Phillips H.S., Kharbanda S., Chen R., et al. // Cancer cell. 2006. V. 9. № 3. P. 157–173.
Pavlyukov M.S., Yu H., Bastola S., et al. // Cancer cell. 2018. V. 34. № 1. P. 119–135.e10.
Li Z., Jiao X., Wang C., et al. // Cancer res. 2010. V. 70. № 21. P. 8802–8811.
David C.J., Manley J.L. // Genes dev. 2010. V. 24. № 21. P. 2343–2364.
Mancini F., Di Conza G., Moretti F. // Curr. genomics. 2009. V. 10. № 1. P. 42–50.
Cheng T.H., Cohen S.N. // Mol. Cell. Biol. 2007. V. 27. № 1. P. 111–119.
Chen M., Manley J.L. // Nat. Rev. Mol. Cell Biol. 2009. V. 10. № 11. P. 741–754.
Schmidt C., Grønborg M., Deckert J., et al. // RNA. 2014. V. 20. № 3. P. 406–420.
Mao P., Joshi K., Li J., et al. // Proc. № atl. Acad. Sci. U S A. 2013. V. 110. № 21. P. 8644–8649.
Bastola S., Pavlyukov M.S., Yamashita D., et al. // Nat. Commun. 2020. V. 11. № 1. P. 4660.
Anufrieva K.S., Shender V.O., Arapidi G.P., et al. // Genome med. 2018. V. 10. № 1. P. 49.
Дополнительные материалы отсутствуют.
Инструменты
Доклады Российской академии наук. Науки о жизни


