Доклады Российской академии наук. Науки о жизни, 2023, T. 509, № 1, стр. 147-154
Мультилокусный анализ филогенетических отношений в видовом комплексе Crocidura suaveolens sensu lato: сравнение с митохондриальными данными
В. А. Грицышин 1, *, А. А. Лисенкова 1, А. С. Сперанская 2, И. В. Артюшин 1, Б. И. Шефтель 3, В. С. Лебедев 4, А. А. Банникова 1, **
1 Московский государственный университет
имени М.В. Ломоносова
Москва, Россия
2 НИИ системной биологии и медицины Роспотребнадзора
117246 Москва, Россия
3 Институт проблем экологии и эволюции
им. А.Н. Северцова Российской академии наук
Москва, Россия
4 Зоологический музей МГУ им. М.В. Ломоносова
Москва, Россия
* E-mail: vladimir.sokol.gritsyshin@gmail.com
** E-mail: hylomys@mail.ru
Поступила в редакцию 10.10.2022
После доработки 07.11.2022
Принята к публикации 08.11.2022
- EDN: LZGICT
- DOI: 10.31857/S2686738922600820
Аннотация
Предпринята первая попытка изучения филогении видового комплекса малая белозубка с помощью мультилокусного анализа. Данные секвенирования 16 ядерных генов указывают на существование в рамках С. suaveolens s. l. нескольких обособленных форм, при этом структура видового комплекса в основных чертах не противоречит митохондриальной филогении. Сибирская белозубка характеризуется определенным своеобразием ядерного генома, но уровень ее генетического отличия не соответствует видовому. Прояснены связи Crocidura aff. suaveolens из южного Ганьсу и Сычуани с другими формами видового комплекса. Белозубки из Бурятии и Хэнтэя также относятся к этой форме, но их мтДНК, по-видимому, заимствована от C. shantungensis. В недавнее время происходила также гибридизация C. suaveolens s.str. с C. aff. suaveolens и C. güeldenstaedtii. Ввиду неоднократных событий интрогрессии в истории C. suaveolens s.l., анализ филогении комплекса в целом требует значительного расширения выборки генетических локусов.
ВВЕДЕНИЕ
В настоящее время в центре филогенетических исследований находится эволюционная история многовидовых групп близкородственных видов относительно недавнего происхождения, так как их изучение позволяет приблизиться к пониманию исходных процессов видообразования. Однако молекулярно-филогенетическая реконструкция кладогенеза близкородственных видов может быть осложнена анцестральным полиморфизмом, событиями гибридизации в прошлом и сохранением потока генов в настоящем.
Видовой комплекс малая белозубка – Crocidura suaveolens sensu lato, занимает огромный ареал, простирающийся через всю Евразию от Пиренейского полуострова до о. Цусима через несколько природных зон. Все формы малой белозубки, кроме С. sibirica, имеют идентичный кариотип [1], вследствие чего существовало мнение, что все популяции с 2n = 40 должны быть объединены в одном виде C. suaveolens [2, 3]. Но имеются также свидетельства морфологического и высокого молекулярного разнообразия этой группы [4–10]. В настоящее время в составе видовой группы “малая белозубка” по морфологическим данным и/или митохондриальной ДНК (мтДНК) различают не менее шести форм, однако, точка зрения на их видовой статус варьирует у разных авторов. Обычно распознают собственно малую белозубку C. suaveolens Pallas, 1811, сибирскую C. sibirica Dukelsky, 1930, кавказскую длиннохвостую C. güeldenstaedtii Pallas, 1811, каспийскую C. caspica Thomas, 1907, манчжурскую С. shantungensis Miller, 1901 и белозубку Зарудного C. zarudnyi Ognev, 1928 [4, 8, 11]. Одни авторы придают также видовой статус и C. mimula Miller, 1901 из Западной Европы [8], другие – C. aleksandrisi Vesmanis, 1977 из Киренаики средиземноморского побережья Северной Африки [11]. Некоторые авторы включают mimula в состав C. güeldenstaedtii в качестве подвида наряду с C. g. iculisma Mottaz, 1908 и C. g. cypria Bate, 1903 [11]. По митохондриальным данным все эти формы (за исключением сибирской белозубки [12]), а также популяции Эгейского побережья и центрально-восточного Ирана формируют самостоятельные филогруппы [10]. Однако данные по изменчивости маркеров ядерной ДНК до сих пор практически отсутствовали, вследствие чего остается не ясным, в какой степени мтДНК отражает общий уровень обособленности их геномов.
МАТЕРИАЛЫ И МЕТОДЫ
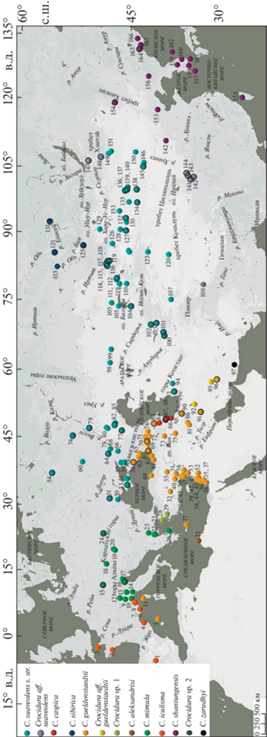
В работе использовано 420 образцов, географическое происхождение которых отражено на карте (рис. 1). Из них для 11 образцов получены последовательности митохондриального гена cytb (1140 п.о.), для 91-го образца отсеквенированы 16 ядерных локусов: ABHD11, ApoB, BDNF, BRCA1, GHR10, ITPK1, MCGF, NHSL1, PALLD, PTGER4, RAG1, RAI14, ROGDI2, SORBS2, SPECC1L, vWF (номера ГенБанка OP599577–OP599587, OP599602–OP599612, OQ374926–OQ376279). Для филогенетического анализа cytb из базы ГенБанк взято 398 образцов (номера сиквенсов AB077075–AB077090, AB0770278–AB077280, AY843448–AY843461, AY843487–AY843500, AY843502, AY843511, AY994368–AY994370, AY994372, AY994373, AY994375–AY994377, AY994386–AY994389, DQ059023, DQ242541, DQ630057–DQ630061, DQ630064–DQ630106, DQ630108, DQ630110–DQ630112, DQ630114–DQ630118, DQ630120, DQ630121, DQ630395, DQ641270, EU742583–EU742594, EU742605–EU742614, HM586991–HM586996, KX354172–KX354178, KX354180, KX354181, LR536317–LR536326, LR536367, LR536372, MF136304–MF136385, MF152782, MN690925–MN690944, MN691017, MW297680–MW297698, MW297700–MW297721, MW297723–MW297727, MW297729–MW297733, MW297735–MW297757, MW297759–MW297791. В качестве внешней группы в митохондриальном и ядерном анализах использованы C. dsinezumi (AB077274, AB077277), C. lasiura (AB077072, MW381915, MW381936, MW381956, MW381979, MW382002, MW382020, MW382042, MW410130), C. leucodon (MW381916, MW381937, MW381980, MW382003, MW382021, MW382043, MW410131) и C. zaitsevi (OL451379, OL451380, OL451373, OL451417, OL451434). В митохондриальный анализ включен дополнительный материал, содержащий внутривидовые выборки большего размера и последовательности отсутствующих у нас форм C. suaveolens s.l. (zarudnyi, aleksandrisii, iculisma) для демонстрации разнообразия митохондриальных линий в этой группе.
Рис. 1.
Локалитеты образцов, использованных в работе: 1 – Испания, Канделарио; 2 – Франция, о. Оэдик; 3 – Великобритания, о. Сарк; 4 – Испания, Фигерассес; 5 – Испания, Менорка; 6 – Франция, о. Поркероль; 7 – Италия, Вентимилья; 8 – Италия, Бадалукко, Фраитуза; 9 – Италия, Верчелли; 10 – Италия, Варацце; 11 – Франция, о. Корсика; 12 – Швейцария, Гордевио; 13 – Италия, Пьяченца, Сан-Николо; 14 – Италия, Фивиццано; 15 – Германия; 16 – Италия, Венеция; 17 – Италия, Латизана; 18 – Австрия, Вена; 19 – Венгрия, Фулопхаза; 20 – Венгрия; 21 – Ливия, Вади эль-Хуф; 22 – Греция, Эпаноми; 23 – Греция, Салоники; 24 – Украина, Воловец; 25 – Болгария, Сандански; 26 – Греция, Афины; 27 – Греция, Крит; 28 – Греция, Лесбос; 29 – Турция, Измир, Юкарыкызылджа; 30 – Украина, Одесса; 31 – Украина, Николаевская обл.; 32 – Турция, Конья; 33 – Крым; 34 – Израиль, Цеэлим; 35 – Израиль, ха-Шфела; 36 – Израиль; 37 – Израиль, Мата; 38 – Израиль, Ахзив; 39 - Израиль, Эйн Яаков; 40 – Турция, Адана; 41 – Крым, Феодосия; 42 – Израиль, Цивон; 43 – Израиль, Цфат; 44 – Израиль, Киннерет Мошава; 45 – Израиль, Тивериадское оз.; 46 – Сирия, Рас аль-Басит; 47 – Сирия, Джебла; 48 – Сирия, Рабия; 49 – Турция, Самсун, Чакаллы; 50 – Сирия, Силинфа; 51 – Турция, Ашагыкарафакылы; 52 – Сирия, Эс-Сувайда, Канават; 53 – Сирия, Каттина; 54 – Россия, Московская обл.; 55 – Турция, Кахраманмараш, Таныр; 56 – Сирия, Хама; 57 – Россия, Азовское море; 58 – Россия, Краснодарский край, Туапсе; 59 – Россия, Краснодарский край; 60 – Россия, Воронеж; 61 – Турция, Алтындере; 62 – Абхазия, Гагра; 63 – Турция, Ризе; 64 – Россия, Ростовская обл.; 65 – Абхазия, Сухум; 66 – Россия, Сальск; 67 – Грузия, Аджария, Батуми; 68 – Россия, Цимлянск; 69 – Грузия, Ахалцихе; 70 – Грузия, Кахетия; 71 – Грузия, Душети; 72 – Грузия, Шулавери; 73 – Иран, Западный Азербайджан, Бастам; 74 – Россия, Пензенская обл.; 75 – Иран, Мохаммедиар; 76 – Грузия, Алазанская долина; 77 – Россия, Калмыкия, зап. “Черные земли”; 78 – Россия, Астраханская обл., пески Шкили; 79 – Россия, Саратовская обл., с. Дьяковка; 80 – Россия, оз. Баскунчак; 81 – Иран, Бехистун; 82 – Россия, Астраханская обл.; 83 – Азербайджан, Кызылагадж; 84 – Азербайджан; 85 – Россия, Досанг; 86 – Иран, Хамадан, Аланьи; 87 – Азербайджан, Астара, Сиов; 88 – Азербайджан, Ленкорань, Бюрждали; 89 – Иран, Гилян, Эсалем; 90 – Иран, Казвин, Фейзабад; 91 – Иран, Бахтиария, Бахшабад; 92 – Иран, Исфахан, Эсфидан; 93 – Иран, Голестан, д. Имер; 94 – Иран, С Хорасан, п. Даркаш; 95 – Иран, Керман; 96 – Иран, Керман, хр. Джебаль-Барез; 97 – Иран, Пир Сохраб; 98 – Казахстан, Аральск; 99 – Казахстан, Косколь; 100 – Узбекистан, Гиссарский зап.; 101 – Таджикистан, р. Сарытаг; 102 – Узбекистан, Чаткальский зап.; 103 – Таджикистан, Похтапор; 104 – Казахстан, Чу-Илийские горы; 105 – Казахстан, СЗ Балхаш; 106 – Казахстан, С Балхаш; 107 – Китай, Синцзянь, Ташкурган; 108 – Китай, Дзанда; 109 – Казахстан, Бельсексеул; 110 – Казахстан, Капал; 111 – Казахстан, Алма-Атинская обл.; 112 – Казахстан, Шыбынды; 113 – Россия, Новосибирск; 114 – Казахстан, Тассай; 115 – Казахстан, река Иртыш; 116 – Казахстан, Айгыркум; 117 – Казахстан, Аксуат; 118 – Казахстан, Шекельмес; 119 – Казахстан, Тугыл; 120 – Китай, Синцзянь, Черчен; 121 – Россия, Кемерово; 122 – Китай, Синцзянь; 123 – Китай, Синцзянь, Корла; 124 – Китай, Синцзянь, Мосован; 125 – Россия, Алтай, Телецкое оз.; 126 – Китай, Синцзянь, уезд Алтай; 127 – Монголия, Байтаг-Богд-Уул; 128 – Монголия, р. Булган-гол; 129 – Монголия, оз. Шаазгай-нуур; 130 – Монголия, хр. Тахин-Шара-нуруу; 131 – Россия, зап. Красноярские столбы; 132 – Монголия, окр. Гун-Тамга-булаг; 133 – Монголия, Говь-Алтай ай-к., Шарга; 134 – Монголия, Барун-Шаргын-Гол; 135 – Монголия, Их-Дзармаг; 136 – Монголия, Баянхонгор айм., Ба-Цаган; 137 – Монголия, Баянхонгор аймак, ю. скл. Баги-Богд-уул; 138 – Монголия, оазис Дзулганай; 139 – Монголия,оз. Орог-нуур; 140 – Монголия, Их-Богд-Уул; 141 – Китай, Ю Ганьсу, монастырь Гойн Ба; 142 – Китай, Сычуань, Дзёгё; 143 – Китай, Ю Ганьсу, Ляньхуашань; 144 – Китай, Ю Ганьсу, зап. Ляньхуашань; 145 – Монголия, Гоби, Гашуун-булаг; 146 – Монголия, Таван-Алдын Худун; 147 – Россия, Бурятия; 148 – Монголия, аймак Сэлэнгэ; 149 – Монголия, окр. Баян-Овоо, Хар-Ямат; 150 – Монголия, окр. Хатан-булаг; 151 – Монголия, В. Хантэй, Баян Уул; 152 – Китай, Шаньси, Нинъу; 153 – Китай, Хэбэй, Цзуньхуа; 154 – Монголия, аймак Дорнод, хр. Хинган; 155 – Тайвань, Тайчжун; 156 – Китай, Ляонин, Синьбинь; 157 – Южная Корея, о. Чеджудо; 158 – Южная Корея, прибрежные о-ва; 159 – Южная Корея; 160 – Южная Корея, Кенджу; 161 – Япония, о. Цусима; 162 – Южная Корея, о. Уллындо; 163 – Россия, Южное Приморье; 164 – Россия, Приморский край, о. Попова; 165 – Россия, Приморский край, о. Путятина; 166 – Россия, Приморский край, Лазовский зап. Локалитеты образцов, использованных в анализе ядерных генов, обведены черным.
Выделение ДНК, амплификацию и секвенирование по Сэнгеру проводили, как указано ранее [12, 13]. Для cytb и BRCA1 использовали праймеры из публикаций [13], для других локусов – из публикаций [14, 15]. Значительная часть данных получена секвенированием ПЦР-продуктов методом Сэнгера. Для подавляющего числа образцов последовательности локусов ABHD11, ITPK1, NHSL1, PALLD, RAI14, ROGDI2, SORBS2, SPECC1L, а также локусов ApoB, BDNF, BRCA1, GHR10, MCGF, PTGER4, RAG1 и vWF для меньшей части образцов получены путем секвенирования на платформе Illumina MiSeq. Библиотеки приготовлены с использованием набора от Illumina Inc. согласно рекомендации производителя из продуктов ПЦР всех локусов, пулированных в равных пропорциях для каждого образца отдельно. Из полученных прочтений удалялись последовательности праймеров, адаптеров и низкокачественные основания при помощи программы Trimmomatic v. 0.33 [16], картирование осуществляли программами bowtie v. 1.1.2 [17] и samtools [18] на референсные последовательности С. suaveolens s.l. или, для отдельных локусов, секвенированных нами впервые, ближайших родственных видов из базы GeneBank (C. indochinensis и Sorex araneus), с последующей заменой на консенсусные последовательности локусов. Коллинг замен осуществлен GATK 4.1.2.0 [19], создание консенсусных последовательностей проводили с помощью bcftools [20], позиции, в которых встречены делеции/инсерции, полностью заменены в выравниваниях на N. Основания с покрытием менее 40 считали непрочитанными. Консенсусные последовательности верифицировали по IGV 2.11.2 [21]. Последовательности выровнены с помощью программ BioEdit 7.2.5. и MEGA 11.0.8.
Для определения аллельного состава видов ядерные последовательности фазировали с помощью модуля Phase в пакете DnaSP 5.10.1 [22] и уточняли по IGV. Выделения групп популяций проводили в программе Structure 2.3.4 [23] (модель “Admixture” при 2,5 млн. MCMC повторов и 250 тыс. повторностей в качестве burn-in). Cтруктуру разнообразия тестировали при заданном числе кластеров от 4 до 10 для последующей оценки оптимального числа кластеров в программе Structure Harvester [24].
Филогенетические деревья строили методом максимального правдоподобия (maximum likelihood, ML) в программе IQTREE 1.6.9 [25] и Байесовым методом (Bayesian inference, BI) в программе Mr.Bayes 3.2.6 [26]. Индивидуальные генные деревья и 5000 бутстрэп-реплик по всем 16 ядерным локусам использовали для построения видового дерева в ASTRAL 5.15.5 [27].
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Митохондриальная филогения С. suaveolens s.l.
В составе С. suaveolens s.l. на дереве по cytb (рис. 2а) обнаруживаются 12 митохондриальных линий: (1) C. suaveolens s.str+C. sibirica, (2) Crocidura aff. suaveolens – гаплотипы из южного Ганьсу и Сычуани, (3) C. caspica, (4) C. iculisma, (5) C. aleksandrisi, (6) güeldenstaedtii, (7) C. aff. güeldenstaedtii – гаплотипы из центрально-восточного Ирана, (8) Crocidura sp.1 – гаплотипы с о-ов Эгейского моря и близлежащей территории Анатолии, (9) C. mimula, (10) C. shantungensis, включая гаплотипы из северо-центральной Монголии (Хэнтэй) и Бурятии, (11) C. zarudnyi и (12) сестринская к ней ветвь из Дзанда в Западном Тибете – Crocidura sp.2. Наиболее близкое положение к корню занимают С. shantungensis и C. zarudnyi/Crocidura sp.2, однако их отношения не разрешены. Остальные виды довольно четко делятся на две клады определенной географической приуроченности: западная и восточная.
Рис. 2.
(а) Филогенетическое дерево видовой группы C. suaveolens s.l. по результатам Байесового (BI) анализа митохондриального гена cytb. Вблизи узлов дерева приведены апостериорные вероятности (BPP) в Байесовом анализе и бутстрэп-поддержки в анализе методом максимального правдоподобия (ML); (б) Видовое дерево группы C. suaveolens s.l., построенное в программе ASTRAL 5.15.5 по данным 16 ядерных генов. В узлах указаны локальные апостериорные вероятности (local PP), рассчитанные программой, бутстрэп-поддержки в анализе методом максимального правдоподобия (ML) и апостериорные вероятности (BPP) в Байесовом анализе конкатенированных последовательностей ядерных локусов (PP/ML/BPP). Внешняя группа для каждого дерева удалена.
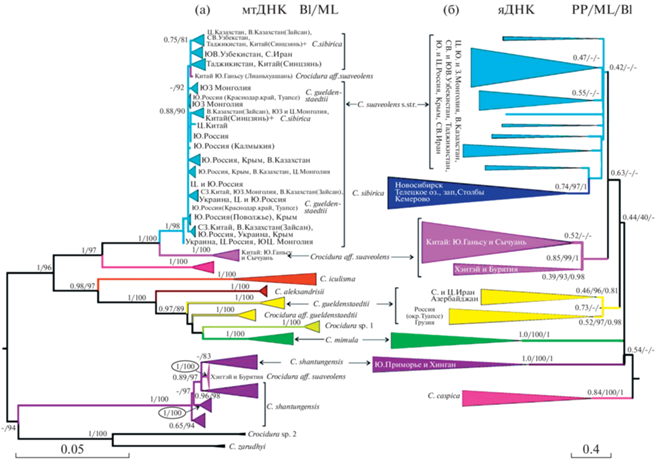
Восточная клада состоит из восточно-европейских и азиатских представителей C. suaveolens s.str., C. aff. suaveolens, C. sibirica и C. caspica. Каспийская белозубка уверенно образует сестринскую ветвь группировки C. aff. suaveolens/(C. suaveolens s.str. + C. sibirica). Гаплотипы из ареала C. sibirica располагаются внутри гаплогруппы C. suaveolens s.str., не образуя монофилетической группы, также как гаплотипы C. güeldenstaedtii с Черноморского побережья Кавказа (Туапсе).
Западная клада включает белозубок из Западной и Центральной Европы, Ближнего Востока, Эгейского моря и близлежащей территории Анатолии, которые образуют несколько гаплогрупп, родственных C. güeldenstaedtii и С. mimula. Базальное положение в западной кладе занимает C. iculisma (Пиренейский п-ов, юг и запад Франции).
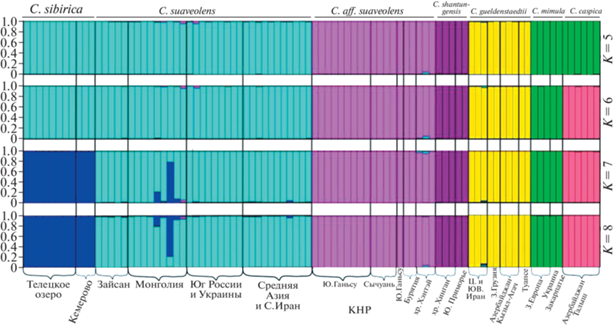
Анализ ядерных данных в программе Structure
Результаты, полученные в программе Structure, оказались нестабильны, т.к. характер разбиения на группы различался при повторении анализов при одном и том же числе кластеров (К). Оптимальное значение К, предложенное программой Harvester, соответствует разбиению на пять групп (рис. 3). В лучшем из решений при данном К (с максимальным Ln Prob) в самостоятельные кластеры выделяются С. shantungensis, C. aff. suaveolens и C. güeldenstaedtii; в единые кластеры объединяются C. suaveolens s.str. + C. sibirica и C. mimula + + C. caspica. В лучшем решении при К = 6 C. mimula и C. caspica формируют самостоятельные кластеры; при К = 7 C. sibirica отделяется от C. suaveolens s.str.; при К = 8 характер разбиения не отличается от К = 7. Заметный сигнал обмена генами отмечается только между C. sibirica и C. suaveolens s.str.
Филогенетический анализ ядерных данных
Видовое дерево. Для того чтобы получить видовое дерево в условиях, когда генные деревья могут достоверно различаться, был выполнен анализ в программе Astral. На полученном видовом дереве (рис. 2б) обнаружены 6 группировок, взаимоотношения между которыми не разрешены: (1) C. shantungensis (Южное Приморье и Хинган); (2) C. mimula (Закарпатье на Украине и Западная Европа); (3) C. güeldenstaedtii, включающая две сестринские линии из северного и центрального Ирана и Азербайджана, с одной стороны, и Грузии и Черноморского побережья Краснодарского края России, с другой; (4) C. caspica; (5) C. aff. suaveolens (КНР, Бурятия и Хэнтэй); (6) группировка C. suaveolens s.str. + C. sibirica, монофилия которой слабо поддержана; C. sibirica внутри нее формирует поддержанный кластер.
ML и Байесов анализ объединенной последовательности ядерных генов. На филогенетическом дереве по результатам ML и Байесова анализа объединенной последовательности ядерных генов (дерево не приведено) в рамках С. suaveolens s.l. состав группировок не значительно отличается от видового дерева (рис. 3б). Две ветви C. güeldenstaedtii не объединяются в единую кладу; C. caspica и C. sibirica оказываются среди множества ветвей C. suaveolens s.str., не связанных географической принадлежностью, где образуют поддержанные монофилетические группировки. Объединение C. suaveolens s.str. + C. caspica + C. sibirica в Байесовом анализе имеет только низкую поддержку, в ML поддержки вообще отсутствуют (0.74/27).
ОБСУЖДЕНИЕ
Мультилокусное филогенетическое дерево демонстрирует существование в рамках видового комплекса С. suaveolens s.l. нескольких обособленных форм. Хотя порядок их дивергенции установить не удается, своеобразие большинства из них вполне очевидно.
C. mimula – монофилетическая самостоятельная форма, представленная в нашем исследовании небольшим числом особей, происходящих из разных частей ареала. По нашим данным именно этот вид населяет Закарпатскую Украину. Следов гибридизации между C. mimula и C. suaveolens s.str. не обнаружено.
В составе C. güeldenstaedtii по ядерным данным обнаружены две сестринские линии, населяющие северо-западную и восточную часть ареала, соответственно. Представители западной линии с Черноморского побережья Кавказа (окрестности Туапсе) несут заимствованную мтДНК C. suaveolens s.str. Однако в ядерных генах следов заимствования не обнаружено. Возможно, это объясняется недавней гибридизацией, сопровождающейся интрогрессией мтДНК, о чем говорит полная идентичность митохондриальных гаплотипов C. güeldenstaedtii с Черноморского побережья Кавказа гаплотипам малой белозубки.
Собственно малая белозубка, C. suaveolens s.str., парафилетична относительно сибирской и имеет крайне слабую структуру, что соответствует и митохондриальным данным [7, 12]. C. caspica монофилетична как по митохондриальным, так и по ядерным данным, однако в последнем случае восстановить ее филогенетические связи с другими видами комплекса C. suaveolens s.l. пока не удается.
Существенным отличием от митохондриального дерева является поддержанная монофилия C. sibirica. Ранее по митохондриальным данным мы предположили, что сибирская белозубка образовалась в период изоляции от C. suaveolens s.str. около 20 тыс. лет назад [12]. Основываясь на анализе ядерных генов, можно предположить, что гипотеза недавнего происхождения сибирской белозубки в целом подтверждается и одна из митохондриальных линий C. sibirica является аутентичной. Отсутствие монофилии сибирской белозубки по мтДНК может объясняться заимствованием части митотипов в результате вторичного контакта с малой белозубкой. Таким образом, хотя сибирская белозубка и характеризуется определенным своеобразием ядерного генома, однако она все же крайне близка к C. suaveolens s.str. Уровень ее генетического отличия не соответствует видовому. Приходится признать, что генетические данные не дают основания для придания сибирской белозубке статуса полного вида.
Наиболее сложный вопрос – это взаимоотношения формы C. aff. suaveolens с другими группировками видового комплекса. На митохондриальном дереве эта форма складывается только из образцов южного Ганьсу и Сычуани (КНР) и занимает сестринское положение по отношению к C. suaveolens s.str./C. sibirica. На ядерном дереве наблюдается похожая тенденция, однако теперь в эту группу входят также образцы из Бурятии и Хэнтея, которые на митохондриальном дереве образуют одну из гаплогрупп в составе C. shantungensis. Образцы из Южного Приморья и Хингана, имеющие митотип настоящей C. shantungensis, и на ядерном дереве составляют самостоятельную кладу, соответствующую маньчжурской белозубке. Таким образом, белозубки из Бурятии и Хэнтэя относятся к форме C. aff. suaveolens, однако их мтДНК, по-видимому, заимствована в прошлом от C. shantungensis. Интересно, что в южном Ганьсу экземпляр из нижней части гор Лиaньхуашани, наоборот, имел обычный митотип малой белозубки, но на ядерном дереве группировался с C. aff. suaveolens из субальпийского пояса гор севера Сычуани. Это показывает, что в разное время и на разных участках ареала происходила (и происходит до сих пор?) гибридизация C. aff. suaveolens не только с маньчжурской, но и с малой белозубкой. Удивительным фактом является огромный разрыв ареала C. aff. suaveolens, поскольку на юге Монголии встречается только типичная C. suaveolens s.str. Направление этого заимствования и происхождение самой формы, как и ее статус нуждаются в дальнейшем пристальном изучении.
Итак, с учетом вероятной интрогрессии мтДНК C. suaveolens s.str. и C. shantungensis в геном C. aff. suaveolens, а также недавней гибридизации между C. suaveolens s.str. и C. güeldenstaedtii на восточно-черноморском побережье, полученная ядерная филогения в основных чертах не противоречит митохондриальной. Однако не вполне уверенные поддержки самых внутренних узлов митохондриального дерева и их отсутствие на ядерном дереве не дают возможности уверенно выстроить порядок кладогенетических событий в эволюционной истории C. suaveolens s.l.
Таким образом, видовой комплекс “малая белозубка” – это наглядный пример недавней серии событий видообразования, в которых большую роль играли не только дивергентные, но и ретикулярные процессы. По митохондриальным оценкам [8] максимальный возраст дивергенции безусловных видов этой группы составляет не более 1.3 млн лет (C. shantungensis), а минимальный – 680 тыс. лет (C. caspica/(C. suaveolens s.str. + C. sibirica)), а о чрезвычайно недавней дивергенции линий C. suaveolens s.str. + C. sibirica мы упоминали выше. Для разрешения ветвей филогенетического дерева такой группы, пережившей быструю и недавнюю диверсификацию, сопровождавшуюся ретикулярными событиями, требуется серьезное умножение числа информативных генетических локусов. Разработка таких маркеров составляет предмет наших дальнейших исследований.
Список литературы
Графодатский А.С., Раджабли С.И., Шаршов А.В., и др. Кариотипы пяти видов землероек-белозубок фауны СССР // Цитология. 1988. Т. 30. № 10. С. 1247–1251.
Catzeflis F., Maddalena T., Hellwing S., et al. Unexpected findings on the taxonomic status of East Mediterranean Crocidura russula auct. (Mammalia, Insectivora) // Zeitschrift für Säugetierkd. 1985. V. 50. № 4. P. 185–201.
Vogel P., Maddalena T., Catzeflis F. A contribution to the taxonomy and ecology of shrews (Crocidura zimmermanni and C. suaveolens) from Crete and Turkey // Acta Theriol. 1986. V. 31. № 39. P. 537–545.
Зайцев М.В. Видовой состав и вопросы систематики землероек-белозубок (Mammalia, Insectivora) фауны СССР // Труды Зоологического института Академии наук СССР. 1991. Т. 243. С. 3–46.
Jiang X.L., Hoffmann R.S. A revision of the white-toothed shrews (Crocidura) of Southern China // Journal of Mammalogy. 2001. V. 82. №4. P. 1059–1079.
Vogel P., Cosson J.-F., Jurado L.F.L. Taxonomic status and origin of the shrews (Soricidae) from the Canary islands inferred from a mtDNA comparison with the European Crocidura species // Molecular Phylogenetics and Evolution. 2003. V. 27. № 2. P. 271–282.
Ohdachi S.D., Iwasa M.A., Nesterenko V.A., et al. Molecular Phylogenetics of Crocidura Shrews (Insectivora) in East and Central Asia // Journal of Mammalogy. 2004. V. 85. № 3. P. 396–403.
Bannikova A.A., Lebedev V.S., Kramerov D.A., et al. Phylogeny and systematics of the Crocidura suaveolens species group: corroboration and controversy between nuclear and mitochondrial DNA markers / Phylogénie et systématique du groupe d’espèces Crocidura suaveolens: coordination et contradiction des marqu // Mammalia. 2006. V. 70. № 1–2. P. 106–119.
Dubey S., Zaitsev M., Cosson J.-F., et al. Pliocene and Pleistocene diversification and multiple refugia in a Eurasian shrew (Crocidura suaveolens group) // Molecular Phylogenetics and Evolution. 2006. V. 38. № 3. P. 635–647.
Dubey S., Cosson J.F., Magnanou E., et al. Mediterranean populations of the lesser white-toothed shrew (Crocidura suaveolens group): an unexpected puzzle of Pleistocene survivors and prehistoric introductions // Molecular Ecology. 2007. V. 16. № 16. P. 3438–3452.
Burgin C.J., He K., Haslauer R., et al. Family Soricidae (shrews). In: Wilson D.E., Mittermeier R.A. (Eds.), Vol. 8. Insectivores, Sloths and Colugos. Handbook of the Mammals of the World. Barcelona, Lynx Edicions; 2018. P. 332–551.
Грицышин В.А., Артюшин И.В., Бурская В.О., и др. Филогеография малой Crocidura suaveolens и сибирской Crocidura sibirica белозубок: В поисках географической родины // Известия Российской академии наук. Серия Биологическая. 2022. № 2. С. 1–14.
Bannikova A.A., Zemlemerova E.D., Lebedev V.S., et al. The phylogenetic relationships within the Eastern Afromontane clade of Crocidura based on mitochondrial and nuclear data // Mammalian Biology. 2021. V. 101. № 6. P. 1005–1018.
Esselstyn J.A., Achmadi A.S., Siler C.D., et al. Carving out turf in a biodiversity hotspot:multiple, previously unrecognized shrew species co-occur on Java Island, Indonesia //Molecular ecology. 2013. V. 22. № 19. P. 4972–4987.
Igea J., Juste J., Castresana J. Novel intron markers to study the phylogeny of closely related mammalian species // BMC evolutionary biology. 2010. V. 10. № 369.
Bolger A.M., Lohse M., Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data // Bioinformatics. 2014. V. 30. № 15. P. 2114–2120.
Langmead B., Salzberg S.L. Fast gapped-read alignment with Bowtie 2 // Nature Methods. 2012. V. 9. P. 357.
Li H., Handsaker B., Wysoker A., et al. The Sequence Alignment/Map format and SAMtools // Bioinformatics. 2009. V. 25. № 16. P. 2078–2079.
Van der Auwera G.A., O’Connor B.D. Genomics in the Cloud: Using Docker, GATK, and WDL in Terra (1st Edition). O’Reilly Media, 2020.
Narasimhan V., Danecek P., Scally A., et al. BCFtools/RoH: a hidden Markov model approach for detecting autozygosity from next-generation sequencing data // Bioinformatics. 2016. V. 32. № 11. P. 1749–1751.
Robinson J.T., Thorvaldsdóttir H., Winckler W., et al. Integrative Genomics Viewer // Nature Biotechnology. 2011. V. 29. № 1. P. 24–26.
Librado P., Rozas J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data // Bioinformatics. 2009. V. 25. № 11. P. 1451–1452.
Pritchard J.K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data // Genetics. 2000. V. 155. № 2. P. 945–959.
Earl D.A., von Holdt B.M. STRUCTURE HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method // Conservation Genetics Resources. 2012. V. 4. № 2. P. 359–361.
Nguyen L.-T., Schmidt H.A., von Haeseler A., et al. IQ-TREE: A fast and effective stochastic algorithm for estimating maximum likelihood phylogenies // Molecular Biology and Evolution. 2015. V. 32. № 1. P. 268–274.
Ronquist F., Teslenko M., van der Mark P., et al. MrBayes 3.2: Efficient Bayesian Phylogenetic Inference and model choice across a large model space // Systematic Biology. 2012. V. 61. № 3. P. 539–542.
Chao Z., Rabiee M., Sayyari E., et al. ASTRAL-III: Polynomial Time Species Tree Reconstruction from Partially Resolved Gene Trees // BMC Bioinformatics. 2018. V. 19. № 6. P. 15–30.
Дополнительные материалы отсутствуют.
Инструменты
Доклады Российской академии наук. Науки о жизни