Доклады Российской академии наук. Науки о жизни, 2023, T. 509, № 1, стр. 196-201
Активность экзонуклеазы nsp14 вируса SARS-CoV-2 по отношению к РНК с модифицированными 3'-концевыми нуклеотидами
С. К. Ююкина 1, 2, *, А. Е. Барматов 2, С. Н. Бизяев 1, 3, 4, Д. А. Стеценко 1, 3, О. В. Сергеева 5, Т. С. Зацепин 5, 6, член-корреспондент РАН Д. О. Жарков 1, 2, **
1 Новосибирский государственный университет
Новосибирск, Россия
2 Институт химической биологии и фундаментальной медицины Сибирского отделения Российской академии наук
Новосибирск, Россия
3 Институт цитологии и генетики Сибирского отделения Российской академии наук
Новосибирск, Россия
4 Новосибирский институт органической химии
им. Н.Н. Ворожцова Сибирского отделения
Российской академии наук
Новосибирск, Россия
5 Сколковский институт науки и технологий
Москва, Россия
6 Московский государственный университет
имени М.В. Ломоносова, Химический факультет
Москва, Россия
* E-mail: sonyayuyukina@gmail.com
** E-mail: dzharkov@niboch.nsc.ru
Поступила в редакцию 25.11.2022
После доработки 05.12.2022
Принята к публикации 09.12.2022
- EDN: NBTFEQ
- DOI: 10.31857/S268673892370018X
Аннотация
Пандемия COVID-19 показала необходимость создания новых средств терапии коронавирусных инфекций. Для некоторых вирусов успешно применяются аналоги нуклеозидов, ингибирующие репликацию за счет включения в растущую цепь ДНК или РНК. Однако репликативный аппарат коронавирусов содержит неструктурный белок nsp14 – 3' → 5'-экзонуклеазу, которая удаляет неправильно включенные и модифицированные нуклеотиды с 3'-конца растущей цепи РНК. В работе исследована эффективность гидролиза РНК, содержащей разные модификации в 3'-концевой области, экзонуклеазой nsp14 SARS-CoV-2 и ее комплексом со вспомогательным белком nsp10. Показано, что одноцепочечная РНК представляет собой предпочтительный субстрат по сравнению с двуцепочечной, что подтверждает предложенную на основе структурного анализа модель переноса субстратной цепи в активный центр фермента. Наибольшее влияние на активность nsp14 оказывала фосфотиоатная и в несколько меньшей степени –N-мезилфосфорамидная модификация фосфодиэфирной связи между предпоследним и последним нуклеотидами, а также введение 2'-фтор заместителя в предпоследний нуклеотид субстрата.
Коронавирусы (семейство Coronaviridae, порядок Nidovirales) представляют собой оболочечные вирусы с одноцепочечным РНК-геномов размером около 26–32 т. п. н. [1]. Пандемия COVID-19 остро поставила вопрос о создании низкомолекулярных терапевтических средств для лечения коронавирусных инфекций. Для некоторых групп вирусов (вирус иммунодефицита человека, герпесвирусы, вирусы гепатита B и гепатита C) успешно применяются модифицированные аналоги нуклеозидов, ингибирующие репликацию вирусного генома за счет включения в растущую цепь ДНК или РНК, после чего синтез либо обрывается, либо продолжается с отложенными летальными последствиями для вируса. На сегодня в разных странах одобрены либо применяются в экспериментальных условиях несколько препаратов для лечения SARS-CoV-2, действующих по описанному механизму – молнупиравир [2], ремдесивир [3], фавипиравир [4] и рибавирин [5]. Сложность использования нуклеозидных ингибиторов в случае SARS-CoV-2 заключается в присутствии в репликативном аппарате коронавирусов неструктурного белка nsp14, обладающего 3' → 5'-экзонуклеазной активностью, которая способна удалять неправильно включенные нуклеотиды с 3'-конца растущей цепи РНК [6].
Белок nsp14 представляет собой бифункциональный фермент, состоящий из N-концевого экзонуклеазного домена (ExoN), участвующего в процессе репликации, и C-концевого метилтрансферазного домена, участвующего в кэпировании мРНК [7]. Домен ExoN относится к суперсемейству DEDD-экзонуклеаз, куда входят многие корректирующие репликативные экзонуклеазы бактерий и эукариот [8]. Экзонуклеазная активность зависит от двухвалентных катионов и стимулируется в несколько раз в присутствии еще одного неструктурного белка коронавирусов – nsp10 [6]. Структурные исследования nsp14 и nsp10 коронавирусов показывают, что связывание с nsp10 приводит к конформационным изменениям в белке nsp14, которые формируют оптимальную структуру его активного центра [7, 9].
Нуклеотидные последовательности, кодирующие белки nsp14 и nsp10 SARS-CoV-2, были оптимизированы для экспрессии в E. coli и получены методом полного синтеза генов. На N-конец обоих белков был введен гексагистидиновый пептид для последующей очистки методом металлхелатной аффинной хроматографии. Для продукции белка nsp10 использовали штамм E. coli Arctic Express(DE3) (Agilent Technologies, США). Успешная наработка белка наблюдалась при индукции 1 мМ IPTG в течение 16 ч при 16°С. Выделение производилось в одну стадию на сорбенте SepFast-Ni-IDA (BioToolomics, Великобритания). Белок nsp14 удалось получить с использованием штамма E. coli BL21(DE3) Star (Thermo Fisher Scientific, США) при индукции 50 мкМ IPTG в течение 6 ч при 37°С. Для выделения белка сначала была проведена металлхелатная аффинная хроматография на сорбенте SepFast-Ni-IDA, а затем гель-фильтрация на колонке Superdex 75 (Cytiva, США). Гомогенность полученных препаратов белка составляла около 90%. Природу обоих рекомбинантных белков подтверждали иммуноблоттингом с антителами на гексагистидиновый пептид, а в случае белка nsp14 дополнительно доказывали методом масс-спектрометрического пептидного картирования и секвенирования продуктов трипсинолиза.
Олигонуклеотиды были получены с использованием амидофосфитного метода синтеза. Для введения межнуклеозидной N-мезилфосфорамидной группы использовали метод, предложенный ранее [10, 11]. Удаление защитных групп проводили смесью насыщенного водного аммиака и водного метиламина, 3 ч при комнатной температуре с последующим упариванием и удалением диметилтретбутилсилильных защитных групп тригидрофторидом триэтиламина в ДМСО в течение 1.5 ч при 65°C. Очистку олигонуклеотидов проводили методом ионообменной хроматографии, их структуру подтверждали методом электрораспылительной масс-спектрометрии. Структуры модифицированных нуклеотидов в составе РНК-субстратов приведены на рис. 1.
Рис. 1.
а – схема РНК-субстратов для исследования экзонуклеазной активности белка nsp14. Подчеркнуты нуклеотиды с тиофосфатными связями. X = G, A или C, Y = C или A. б – общая структура двух 3'-концевых нуклеотидов в субстратах. В дальнейшем для обозначения субстрата используются сокращения вида “серия”-“3'-концевой нуклеотид” (напр., II-G) для одноцепочечных субстратов и “серия”-“3'-концевой нуклеотид”:“нуклеотид напротив 3'-концевого” (напр., II-G:A) для двуцепочечных субстратов. В работе использовано по 5 субстратов из каждой серии.
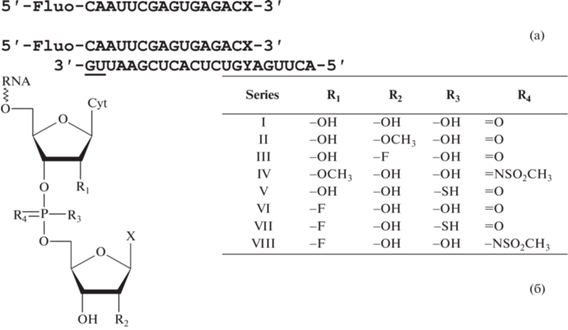
Для детекции продуктов после электрофореза на 5'-конец модифицированной олигонуклеотидной цепи был при синтезе введен остаток флуоресцеина. Для предотвращения неспецифичного расщепления второй цепи в составе РНК-дуплексов два ее 3'-концевых нуклеотида несли защитную модификацию в виде межнуклеозидной тиофосфатной группы.
По литературным данным, фермент nsp14 из других коронавирусов – SARS-CoV-1 и вируса мышиного гепатита – гидролизует как одно-, так и двуцепочечную РНК [5, 12]. С использованием полученных препаратов белков была проведена оценка экзонуклеазной активности выделенного nsp14 и комплекса nsp14/nsp10 по отношению к флуоресцентно меченным одноцепочечным РНК-субстратам I-G–VIII-G (рис. 1). Активность белка исследовали как в присутствии nsp10, так и без его добавления. Дополнительно были проведены реакции только с nsp10 без добавления nsp14. Как и ожидалось, белок nsp14 эффективно гидролизовал немодифицированную РНК, однако эта реакция практически не стимулировалась белком nsp10 (рис. 2а, б, рис. 3а). На геле наблюдались две выраженные полосы, соответствующие расщеплению по связям G7–A8 и G9–U10, что, скорее всего, объясняется паузой 3'→5'-экзонуклеазной активности в этих позициях. Аналогичная картина двух преимущественных точек расщепления в 5–7 нуклеотидах от 3'-конца наблюдается для фермента nsp14 из близкородственных коронавирусов SARS-CoV-1 и MERS-CoV [6, 13]. Можно предположить, что паузы вызваны особенностями взаимодействия фермента с выступающей частью двуцепочечного РНК-субстрата; например, они могут возникать, когда выступающая часть становится достаточно длинной, чтобы образовывать контакты с метилтрансферазным доменом фермента. Введение модификаций в остаток рибозы 3'-концевого нуклеотида (2'-O-метил, 2'-фтор; субстраты II-G и III-G) не оказывало заметного влияния на эффективность расщепления. Напротив, модификации, затрагивающие фосфодиэфирную связь между последним и предпоследним рибонуклеозидами (тиофосфат, мезилфосфорамид; субстраты IV-G, V-G, VII-G, VIII-G), снижали скорость реакции в 2–4 раза (рис. 2в, г, рис. 3а). Введение 2'-F-заместителя в предпоследнее положение РНК (субстрат VI-G) также оказывало ингибирующее действие, хотя и не столь заметное, как модификация межнуклеотидного фосфата.
Рис. 2.
Расщепление одноцепочечных олигорибонуклеотидных субстратов I-G (а, б) и V-G (в, г) рибонуклеазой nsp14. а, в – фотографии гелей после электрофореза. S – субстрат, P – продукты реакции. б, г – графики зависимости уровня расщепления субстрата от времени. ($ \bullet $) – реакции в присутствии nsp14 и nsp10, ($\bigcirc $) – только nsp14, ($\blacksquare $) – только nsp10. Приведены среднее и стандартная ошибка по 3 независимым экспериментам. В состав реакционной смеси входили 40 мМ Трис–HCl (pH 7,5), 50 мМ NaCl, 5 мМ MgCl2, 1 мМ дитиотреит, 100 нМ РНК-субстрат, 500 нМ белки nsp14 и nsp10. Смесь инкубировали при 37°C указанное время, после чего инактивировали добавлением равного объема формамида, содержащего 10 мМ этилендиаминтетраацетат натрия, и прогреванием в течение 3 мин при 95°C. Продукты реакции разделяли электрофорезом в 12%-ном полиакриламидном геле в присутствии 7.2 М мочевины. Гель сканировали с помощью системы Typhoon FLA 9500 (Danaher, США).
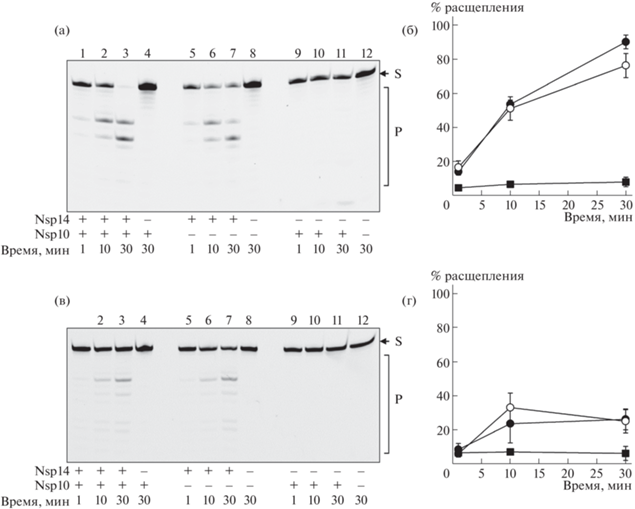
Рис. 3.
Эффективность расщепления олигорибонуклеотидных субстратов разной структуры рибонуклеазой nsp14. Общая структура субстратов: одноцепочечная РНК с 3'-концевым остатком G (а), двуцепочечная РНК с 3'-концевым остатком G на конце меченой цепи, образующим комплементарную пару G:C (б), двуцепочечная РНК с 3'-концевым остатком G на конце меченой цепи, образующим некомплементарную пару G:A (в), одноцепочечная РНК с 3'-концевым остатком А (г), одноцепочечная РНК с 3'-концевым остатком C (д). Реакцию вели 30 мин в таких же условиях, как на рис. 2. Приведены среднее и стандартная ошибка по 3 независимым экспериментам. (е) – сводная таблица влияния модификации на эффективность расщепления. Субстраты с достоверно сниженным расщеплением в сравнении с аналогичным немодифицированным субстратом отмечены звездочками на панелях (а–д) и заливкой на панели (е) (* или светло-серый цвет заливки – p < 0.05, ** или темно-серый цвет – p < 0.01; *** или черный цвет – p < 0.005 по критерию Стьюдента). Субстраты с достоверно повышенным расщеплением в присутствии nsp10 в сравнении с отсутствием nsp10 отмечены крестиком на панелях (а–д) и кружком на панели Е (p < 0.05 по критерию Стьюдента).
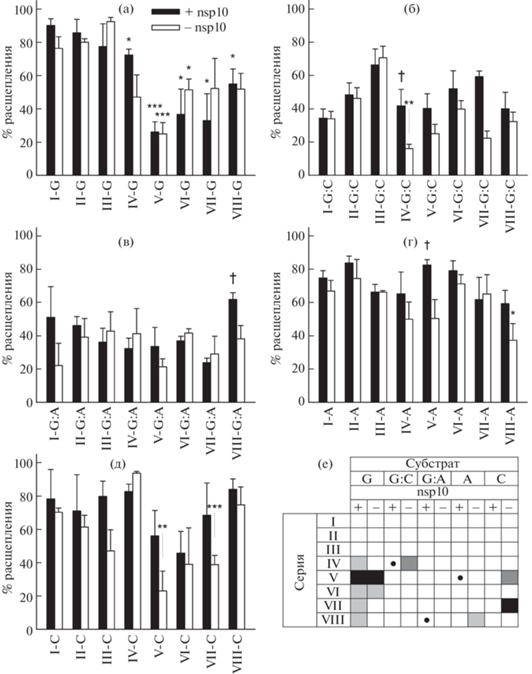
Для изучения активности фермента nsp14 по отношению к двуцепочечным РНК-субстратам использовали наборы дуплексов I-G:C–VIII-G:C и I-G:A–VIII-G:A. В целом экзонуклеазная активность была несколько ниже, чем при использовании одноцепочечных субстратов, и не наблюдалось достоверных различий между полностью комплементарными и частично некомплементарными субстратами (рис. 3б, в). Такая специфичность согласуется с моделью, недавно предложенной на основе структуры репликативного комплекса SARS-CoV-2 [15], в которой гидролиз 3'-концевых нуклеотидов при включении неправильного NMP происходит именно в одноцепочечном состоянии – после обратного движения РНК-полимеразы, разделения цепей РНК на протяжении последних ~6 пар нуклеотидов и переноса 3'-конца синтезируемой цепи в активный центр субъединицы nsp14. Стимуляция гидролиза РНК-дуплексов белком nsp10 достоверно наблюдалась лишь при наличии мезилфосфорамидной модификации фосфодиэфирной связи (субстраты IV-G:C и VIII-G:A, рис. 3Б,В). Вполне вероятно, что требования к аллостерической активации nsp14 для олигонуклеотидных РНК-субстратов не столь критичны.
Для сравнения специфичности nsp14 по отношению к 3'-концевым рибонуклеозидам в составе одноцепочечных субстратов была определена активность фермента в реакции гидролиза олигорибонуклеотидов, содержащих на 3'-конце AMP, CMP либо их модификации (рис. 3г, д). Как и в случае GMP, фермент nsp14 эффективно гидролизовал такие субстраты как в присутствии, так и в отсутствие nsp10. Однако достоверное снижение активности при модификациях наблюдалось только в отсутствие nsp10 для субстратов VIII-A (3'-концевой A, предпоследний 2'-фторрибонуклеозид, мезилфосфорамидная связь между последним и предпоследним нуклеозидами), V-C (3'-концевой C, тиофосфатная связь между последним и предпоследним нуклеозидами) и VII-C (3'-концевой C, предпоследний 2'-фторрибонуклеозид, тиофосфатная связь между последним и предпоследним нуклеозидами). Можно предположить, что наибольшее влияние модификаций проявляется именно в GMP за счет наибольшего объема основания Gua, затрудняющего конформационную подгонку активного центра фермента при наличии дополнительных заместителей.
В настоящее время аналоги нуклеотидов используются в качестве терапевтических средств не так часто, как аналоги нуклеозидов, из-за менее эффективного проникновения в клетки. Однако такие средства, как, например, цидофовир, используемый для терапии цитомегаловирусной инфекции, а также тенофовир и адефовир, применяемые при гепатите B, относятся именно к аналогам нуклеотидов с модифицированным фосфатом. При этом для всех них существуют соединения-предшественники с заместителями, нейтрализующими заряд и облегчающими проникновение в клетки. Таким образом, аналоги нуклеотидов, способные включаться в РНК и модифицированные по 5'-фосфатной группе, представляют собой группу соединений, перспективную для разработки устойчивых к корректирующей экзонуклеазе ингибиторов репликации коронавирусов.
Список литературы
Su S., Wong G., Shi W., et al. // Trends Microbiol. 2016. V. 24. № 6. P. 490–502.
Jayk Bernal A., Gomes da Silva M.M., Musungaie D.B., et al. // N. Engl. J. Med. 2022. V. 386. № 6. P. 509–520.
Bravo J.P.K., Dangerfield T.L., Taylor D.W., John-son K.A. // Mol. Cell. 2021. V. 81. № 7. P. 1548–1552.
Shannon A., Selisko B., Le N.-T.-T., et al. // Nat. Commun. 2020. V. 11. 4682.
Ferron F., Subissi L., Silveira De Morais A.T., et al. // Proc. Natl Acad. Sci. U.S.A. 2018. V. 115. № 2. P. E162–E171.
Bouvet M., Imbert I., Subissi L., et al. // Proc. Natl Acad. Sci. U.S.A. 2012. V. 109. № 24. P. 9372–9377.
Ma Y., Wu L., Shaw N., et al. // Proc. Natl Acad. Sci. U.S.A. 2015. V. 112. № 30. P. 9436–9441.
Zuo Y., Deutscher M.P. // Nucleic Acids Res. 2001. V. 29. № 5. P. 1017–1026.
Liu C., Shi W., Becker S.T., et al. // Science. 2021. V. 373. № 6559. P. 1142–1146.
Челобанов Б.П., Буракова Е.А., Прохорова Д.В., и др. // Биоорган. химия. 2017. Т. 43. № 6. С. 644–649.
Miroshnichenko S.K., Patutina O.A., Burakova E.A., et al. // Proc. Natl Acad. Sci. U.S.A. 2019. V. 116. № 4. P. 1229–1234.
Minskaia E., Hertzig T., Gorbalenya A.E., et al. // Proc. Natl Acad. Sci. U.S.A. 2006. V. 103. № 13. P. 5108–5113.
Ogando N.S., Zevenhoven-Dobbe J.C., van der Meer Y., et al. // J. Virol. 2020. V. 94. № 23. e01246-20.
Yan L., Yang Y., Li M., et al. // Cell. 2021. V. 184. № 13. P. 3474–3485.e11.
Дополнительные материалы отсутствуют.
Инструменты
Доклады Российской академии наук. Науки о жизни


