Экология, 2022, № 5, стр. 355-361
Разнообразие и экологические особенности филогенетических линий настоящего трутовика на Урале
Е. В. Жуйкова a, *, В. А. Мухин a, b
a Институт экологии растений и животных УрО РАН
620144 Екатеринбург, ул. 8 Марта, 202, Россия
b Институт естественных наук и математики, Уральский федеральный университет им. первого Президента России Б.Н. Ельцина
620002 Екатеринбург, ул. Мира, 19, Россия
* E-mail: e.zhuykova@list.ru
Поступила в редакцию 10.03.2022
После доработки 05.05.2022
Принята к публикации 07.05.2022
- EDN: UXIWNY
- DOI: 10.31857/S0367059722050110
Аннотация
Филогенетический анализ внутреннего транскрибируемого спейсера (ITS) рибосомальной ДНК, выделенной из 51 базидиокарпа настоящего трутовика, показывает, что на Урале и в Северном Казахстане данный вид ксилотрофных грибов – комплексный, состоящий из двух криптических филогенетических линий (А и В), представленных сублиниями А2 и В2. Их ареалы перекрываются и охватывают Южный, Средний и Северный Урал, а также Северный Казахстан, но А2 чаще встречается на Северном и Среднем Урале, а В2 – на Южном Урале и в Северном Казахстане. Их симпатрия основывается на трофической специализации: представители сублинии А2 встречаются на Alnus, Betula, Prunus, Salix, Sorbus, а В2 – на Acer, Populus, Salix, Tilia. Для первых основным субстратом является Betula, а для вторых – Populus. По ITS региону рДНК сублиния А2 соответствует Fomes fomentarius sensu stricto, а В2 – Fomes inzengae (Ces. & De Not.) Cooke, двум симпатрическим криптическим видам, описанным в Европе. По уровню нуклеотидной дивергенции F. fomentarius s. s. и F. inzengae в равной степени удалены от F. fasciatus (Sw.) Cooke (6.26 и 7.05% соответственно), но уровень дивергенции между ними (1.48%) не превышает среднего уровня внутривидовой ITS изменчивости базидиальных грибов. Скорее всего, их следует рассматривать как криптические симпатрические инфравидовые филогенетические линии Fomes fomentarius sensu lato подвидового ранга.
Настоящий трутовик – Fomes fomentarius sensu lato (Polyporaceae, Agaricomycetes) – один из широко распространенных в Евразии, в том числе и на Урале, видов ксилотрофных грибов [1–6]. Данная группа организмов определяет многие аспекты процессов разложения древесных остатков, в том числе и такой их параметр, как углеродный газообмен, уступающий по объему эмиссии СО2 только почвенному дыханию [7]. Это объясняет наблюдаемый в настоящее время интерес к изучению экологии древесного дебриса и ксилотрофных грибов [8]. Большой интерес F. fomentarius s. l. представляет и для биотехнологии [9–15], но его использование затрудняет неопределенность популяционной структуры и границ как биологического вида [14, 16].
Как и большинство других видов трутовых грибов, F. fomentarius s. l. описан по морфологическим и анатомическим признакам базидиокарпов и рассматривается как морфологически гомогенный, или мономорфный вид [1–3, 5, 17, 18]. Однако на генетическом уровне настоящий трутовик не является однородным, и, например, в Европе на основе вариабельности последовательностей внутреннего транскрибируемого спейсера (ITS) рибосомальной ДНК обнаружены два генотипа (А и В), которые рассматривают как два симпатрических криптических вида [19, 20]. J. Gaper et al. [14] рассматривают их как филогенетические линии (lineages), из которых одна (А) имеет две сублинии (А1 и А2), встречающиеся в Северной Америке и Европе соответственно. Линия В, как нами было показано ранее [15], также имеет две сублинии: одна в Азии (В1), а вторая – в Европе (В2). U. Peintner et al. [16] идентифицировали две филогенетические линии в Европе как два криптических симпатрических вида: F. fomentarius sensu stricto (s. s.) и F. inzengae (Ces. & De Not.) Cooke.
Для более полного изучения разнообразия филогенетических линий F. fomentarius s. l. и их экологических особенностей необходимо расширение географии исследований, и прежде всего за счет азиатской части России (Урал, Сибирь, Дальний Восток), где находится один из трех (азиатский, европейский и североамериканский) крупных участков его обширного ареала. Цель настоящей работы – анализ разнообразия, распространения и экологических особенностей филогенетических линий настоящего трутовика на Урале – границе европейского и азиатского субконтинентов.
МАТЕРИАЛ И МЕТОДЫ
Районы исследования
Биоматериалом для молекулярно-генетического анализа послужили базидиокарпы настоящего трутовика, коллекция которых была собрана в ходе полевых работ с 2001 г. по 2020 г. на Южном, Среднем и Северном Урале, а также в Костанайской области Казахстана. Протяженность эколого-генетического профиля с севера на юг составила около 1000 км. Базидиокарпы собирали случайным образом в ходе маршрутных учетов по одному с каждого субстрата в разных локалитетах, расстояние между субстратами составляло не менее 100 м. В общей сложности для филогенетического анализа был отобран 51 базидиокарп, их видовая диагностика выполнена с использованием традиционных микологических методов [3]. Подробная информация об образцах представлена в базе данных GBIF [21], а их коллекция находится в гербарии Института экологии растений и животных УрО РАН (г. Екатеринбург).
Выделение ДНК и ПЦР амплификация
Работы по выделению ДНК и ПЦР с визуализацией выполнены в лаборатории молекулярных исследований растений и грибов Института естественных наук и математики Уральского федерального университета им. первого Президента России Б.Н. Ельцина (г. Екатеринбург, Россия). Источником ДНК были как непосредственно собранные в полевых условиях базидиокарпы настоящего трутовика (32 шт.), так и полученные из них чистые дикариотические культуры (19 шт.) на сусло-агаре.
Выделение ДНК осуществляли с помощью набора для выделения ДНК из растительной ткани (diaGene, Россия) с разведением ДНК в 100 μL деионизированной воды. Два праймера: ITS1F и ITS4b [22], были использованы для амплификации внутреннего транскрибируемого спейсера (ITS) рибосомальной ДНК в полимеразной цепной реакции. ПЦР проводили с помощью C1000 Touch Amplifier (Bio-Rad Laboratories, США) в 25 μL реакционной смеси, содержащей буферный раствор с 2.5 мM MgCl2, 0.2 мM dNTP, 0.1 мкM каждого праймера, 0.08 ед./мкл Taq ДНК-полимеразы (ЗАО “Евроген”, Россия) и 5 мкл раствора ДНК.
Программа ПЦР включала предденатурацию 95°C – 5 мин, 35 циклов денатурации при 95°C – 30 с, отжига праймеров при 55°C – 45 с, элонгации при 72°C – 45 с, и финальную элонгацию при 72°C – 10 мин. Результаты ПЦР детектировали визуально в 1.2%-ном агарозном ТВЕ геле с окраской бромистым этидием. Длину фрагментов сверяли относительно маркера длин ДНК 100+ bp DNA Ladder (ЗАО “Евроген”, Россия) с помощью системы гель-документирования Gel Doc XR+ Gel (Bio-Rad Laboratories, США).
Ферментативная очистка или очистка электрофорезом в агарозном геле ПЦР-продуктов, а также секвенирование как в прямом, так и обратном направлениях выполнены ООО “Синтол” (Москва, Россия). Обработка первичных данных секвенирования проведена с использованием программ Sequencing Analysis Software v. 5.3.1 (Applied Biosystems, США), Finch TV v. 1.4.0 (Geopriza, Inc.) и MEGA v. 7.0.18 [23]. Идентификация полученных последовательностей проведена по результатам поиска схожих в базе данных GenBank с помощью алгоритма Blast (blast.ncbi.nlm. nih.gov). Полученные в ходе исследования оригинальные последовательности депонированы в базу данных GenBank (ncbi.nlm.nih.gov/genbank).
Филогенетический анализ
Для филогенетического анализа были подготовлены два набора референсных последовательностей. Первый был сформирован на основе данных U. Peintner et al. [16]. Он включал последовательности F. fomentarius s. s. (EF155495, GU062198, HQ189534, KM360127, MK295658), F. fomentarius II (HM584810, JX126886, JX183719, JX183720, KC505546), Fomes sp. Asia (DQ513402, EU273503, JX290073, KJ668550, MH114657), Fomes sp. Iran (MK050587) и F. inzengae (AY849306, AM981233, FN539045, GQ184604, MG719676, MK184456, MK184458). Второй набор включал последовательности линии А2 (GQ184603, JF927720, KM360128, KM396269) и В (FN539043, GQ184600, HQ189535, JX910366), описанные J. Gaper et al. [14]. В качестве референсных были использованы и последовательности, описанные нами ранее: сиквенсы сублиний А2 (MF563971, MF563973, MF563977, MF563978) и В2 (MF563981, MF563984, MF563985) [15]. Все последовательности (38) были взяты из базы данных GenBank (ncbi.nlm. nih.gov/genbank) в соответствии с их номерами. Оригинальные последовательности были добавлены к референсным и выравнены с помощью алгоритма Muscle с ручной проверкой результатов. В качестве внешней группы для всех построений использована одна последовательность Fomes fasciatus (Sw.) Cooke JX126900.
Филогенетические деревья были построены методом максимального правдоподобия, основанного на модели Hasegawa-Kishino-Yano [24]. Все позиции с охватом сайта менее 90% были исключены, в окончательном наборе было 520 нуклеотидов. Бутстрэп анализ (500 повторений) проводился с помощью алгоритма Subtree-Pruning-Regrafting уровня 5, исходное дерево было получено автоматически методом максимальной экономии. Деревья нарисованы в масштабе с длинами ветвей, измеряющимися количеством нуклеотидных замен на сайт; значения бутстрэп больше 60 указаны рядом с узлами. Описание каждой последовательности содержит GenBank accession number, страну или физико-географической район происхождения образца, а также субстрат. Эволюционный анализ проводился в MEGA7 [23].
База данных для расчета полиморфизма ДНК включала 51 последовательность, полученную в настоящем исследовании, и 5 последовательностей F. fasciatus (JX126900, JX126901, JX126906, JX126907, JX126908) [16]. Выравнивание было проведено алгоритмом Muscle с ручной проверкой результатов. Полиморфные позиции в невариабельных во всей выборке сайтах трактовались в пользу сохранения гомогенности. При наличии в выборке однозначных замен в рассматриваемой позиции хотя бы у одной последовательности полиморфизм не учитывался. Расчет количества однонуклеотидных замен в попарных сравнениях последовательностей отдельных линий и нуклеотидной дивергенции (Dxy) проводился при помощи программы DnaSP v. 6.12 [25].
Статистический анализ
Оценка географического распространения филогенетических линий была проведена с помощью однофакторного дисперсионного анализа с использованием широты в качестве фактора. Статистический анализ субстратной приуроченности проведен с использованием критерия χ2 с поправкой Йейтса. Все расчеты выполнены в статистическом пакете Statistica 8.0 (StatSoft, Inc.).
РЕЗУЛЬТАТЫ
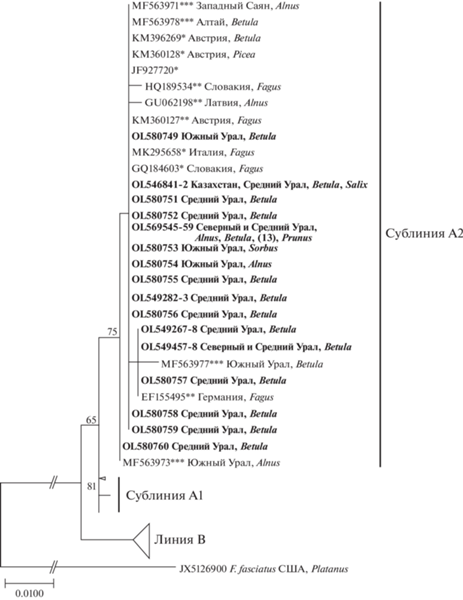
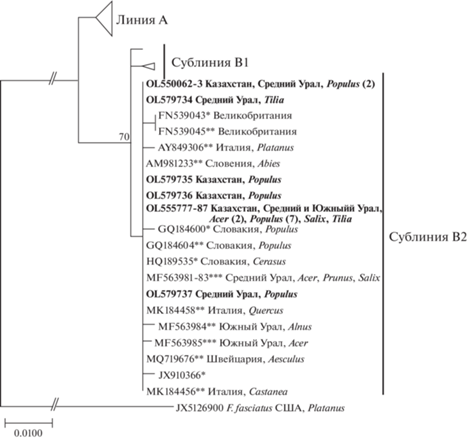
Как показывает филогенетический анализ последовательностей внутреннего транскрибируемого спейсера рибосомальной ДНК, базидиокарпы F. fomentarius s. l. с Северного, Среднего и Южного Урала, а также из Северного Казахстана относятся к филогенетическим сублиниям А2 и В2. Большая часть изолятов (34) образует гомогенный кластер с референсными изолятами сублинии А2, меньшая (17) – сублинии В2 (рис. 1, 2). Данные кластеры включают не только референсные для сублиний А2 и В2, но и для F. fomentarius s. s. и F. inzengae последовательности соответственно. Поэтому сублинию А2 можно соотнести с F. fomentarius s. s., а В2 – с F. inzengae.
Однозначные нуклеотидные замены между последовательностями в пределах каждой из филогенетических сублиний отсутствуют, но между собой сублинии отличаются нуклеотидными заменами в 8 позициях, и соответственно их нуклеотидная дивергенция составляет 8 пар оснований (Dxy = 0.01486, или 1.49%). Между сублиниями и F. fasciatus нуклеотидная дивергенция в 4–5 раз выше: F. fomentarius s. s. – в среднем 33 п. о. (Dxy = = 0.06255, или 6.26%), а F. inzengae – в среднем 38 п. о. (Dxy = 0.07046, или 7.05%).
Грибы обоих филогенетических сублиний встречаются одновременно на Среднем, Южном Урале и в Северном Казахстане (см. рис. 1, 2). Большая часть образцов, идентифицируемых как F. fomenarius s. s., были собраны на Северном и Среднем Урале (30 находок из 34), а идентифицируемых как F. inzengae – на Южном Урале и в Северном Казахстане (12 находок из 17). Значимость указанных различий географического распределения филогенетических линий подтверждают результаты однофакторного дисперсионного анализа: F(2, 48) = 13.163, p = 0.00003.
Грибы сублинии F. fomentarius s. s. обнаружены на древесных остатках Alnus, Betula, Prunus, Salix, Sorbus, а F. inzengae – Acer, Populus, Salix, Tilia. Общим для них субстратом является лишь Salix. Основная часть образцов базидиокарпов сублинии F. fomentarius s. s. (29 из 34) были собраны на древесных остатках Betula, тогда как сублинии F. inzengae ─ на древесных остатках Populus (12 из 17). Более того, грибы сублинии F. fomentarius s. s. не были найдены на Populus, а F. inzengae – на Betula. Указанные особенности субстратного распределения грибов филогенетических линий статистически значимы: χ2 = 36.31, p = 0.0000.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
На Урале F. fomentarius s. l. – комплексный вид, состоящий из двух описанных для Европы филогенетических линий А и В, представленных в регионе сублиниями А2 и В2. Первая из них как широко распространенный вариант линии А встречается в Европе, Азии, Северной Америке [14, 19, 20]. Сублиния B2, ассоциируемая с европейским субконтинентом, по имеющимся на настоящий момент времени данным прослеживается до Урала [15].
На уровне ITS последовательностей рибосомальной ДНК сублиния А2 соответствует F. fomentarius s. s., а В2 – F. inzengae (Ces. & De Not.) Cooke, являющимися, по мнению U. Peintner et al. [16], двумя криптическими симпатрическими видами. По морфологическим признакам базидиокарпы F. inzengae отличаются лишь более мелкими, чем у F. fomentarius s. s., порами гименофора и соответственно большим их количеством на единицу площади [16]. При почти полном отсутствии морфологических различий они заметно отличаются по географическому распространению: U. Peintner et al. [16] характеризуют F. inzengae как средиземноморский, а F. fomentarius s. s. как северный вид. Грибы филогенетической линии F. fomentarius s. s. встречаются на древесных остатках Fagus и Betula, реже Alnus, Quercus, Populus, Acer, Picea, а F. inzengae − на Quercus, Castanea, Carpinus, Platanus, Populus, в виде исключения – на Abies и Cerasium [16, 26].
На Урале F. fomentarius s. s. и F. inzengae также существенно различаются по субстратным спектрам и трофическим преферендумам. Субстратный спектр первого включает Alnus, Betula, Prunus, Salix, Sorbus, а F. inzengae − Acer, Populus, Tilia, Salix. Как и в Европе, общим для них субстратом служит лишь Salix. Для F. fomentarius s. s. основным и специфическим субстратом является Betula, а для F. inzengae – Populus. По мнению K. Náplavová et al. [27], южная граница распространения филогенетической линии А2 в Европе, диагностируемая нами как F. fomentarius s. s., определяется южной границей ареала Betula pendula. Следует также отметить, что F. inzengae был впервые описан по базидиокарпам с Populus nigra [16].
Дифференцированное отношение F. fomentarius s. s. и F. inzengae по отношению к различным видам древесных остатков лежит в основе их симпатрии. Обе филогенетические линии встречаются на Среднем и Южном Урале, в Северном Казахстане, но, если судить по количеству секвенированных базидиокарпов, F. fomenarius s. s. чаще встречается на Северном и Среднем Урале, а F. inzengae – на Южном Урале и в Северном Казахстане. Подобное распределение соответствует их распространению в Европе, где F. inzengae также более южный по отношению к F. fomenarius s. s. вид.
Как было отмечено выше, филогенетические линии F. fomentarius s. s. и F. inzengae рассматриваются как криптические, симпатрические виды [16]. Однако наши данные позволяют в этом усомниться. Во-первых, различия между F. fomentarius s. s. и F. inzengae (8 п. о.; 1.48%) не превышают средний для ITS региона уровень внутривидовой изменчивости у базидиальных грибов – 3.33% [28]. Во-вторых, различия между ними ниже видового уровня, показателем которого, на наш взгляд, может служить их нуклеотидная дивергенция с F. fasciatus: в среднем 33 п. о., 6.23% с F. fomentarius s. s. и 38 п. о., 7.04% с F. inzengae. Аналогичные данные приводят P. Dresch et al. [29] и U. Peintner et al. [16] – нуклеотидная дивергенция между F. fomentarius s. s. и F. inzengae составляет 9–18 п. о., а между ними и F. fasciatus – 41–62 п. о.
Мы считаем, что F. fomentarius s. s. и F. inzengae необходимо рассматривать не как виды, а как филогенетические линии инфравидового уровня. Этот подход соответствует традиционным взглядам на F. inzengae, как на морфологическую форму F. fomentarius s. l. [1, 17, 18, 30, 31]. Однако, учитывая генетическую, географическую и экологическую обособленности F. fomentarius s. s. и F. inzengae, их таксономический ранг, скорее всего, соответствует подвиду – таксону, объединяющему географически и экологически обособленные группы популяций одного вида [32]: в нашем случае Fomes fomentarius sensu lato.
ЗАКЛЮЧЕНИЕ
На Урале настоящий трутовик не является генетически гомогенным и состоит из двух симпатрических криптических филогенетических линий, идентифицируемых по ITS рДНК как F. fomentarius s. s. и F. inzengae. По своим генетическим, географическим и экологическим характеристикам они не отличаются принципиально от аналогичных филогенетических линий в Европе. Представители линии F. fomentarius s. s. преимущественно встречаются на Северном и Среднем Урале, а F. inzengae – на Южном Урале и в Северном Казахстане. Грибы линии F. fomentarius s. s. в основном встречаются на Betula, а F. inzengae – на Populus, и это лежит в основе их симпатрии в районах совместного обитания. Уровень филогенетической дивергенции F. fomentarius s. s. и F. inzengae соответствует внутривидовому уровню, и, скорее всего, их надо рассматривать как криптические и симпатрические подвиды настоящего трутовика.
Исследование выполнено при поддержке РФФИ (проект № 20-34-90054), РФФИ и Правительства Свердловской области (проект № 20-44-660012), РНФ (проект № 22-24-00970) и Гос. соглашения ИЭРиЖ УрО РАН (122021000092-9). Авторы выражают благодарность В.Л. Семерикову и С.А. Семериковой за помощь при обработке данных.
Авторы подтверждают отсутствие конфликта интересов.
Настоящая статья не содержит исследований с участием людей или животных в качестве объектов исследования.
Список литературы
Бондарцев А.С. Трутовые грибы европейской части СССР и Кавказа. М.; Л.: Изд-во АН СССР, 1953. 1106 с.
Jülich W. Deutschlands Kryptogamenflora. Bd. IIb/1. Basidiomyceten. 1. Teil. Die Nichtblätterpilze, Gallertpilze und Bauchpilze/. Jena: VEB Gustav Fisher Verlag, 1984. 626 s.
Ryvarden L., Gilbertson R.L. European Polypores. Part 1 (Abortiporus-Lindtneria). Oslo, Norway: Fungiflora, 1993. 387 p.
Мухин В.А. Биота ксилотрофных базидиомицетов Западно-Сибирской равнины. Екатеринбург: УИФ “Наука”, 1993. 231 с.
Бондарцева М.А. Определитель грибов России. Порядок афиллофоровые. Вып. 2: Семейства альбатрелловые, апорпиевые, болетопсиевые, бондарцевиевые, ганодермовые, кортициевые (виды с порообразным гименофором), лахнокладиевые (виды с трубчатым гименофором), полипоровые (роды с трубчатым гименофором), пориевые, ригидопоровые, феоловые, фистулиновые. СПб.: Наука, 1998. 391 с.
Aphyllophoroid fungi of Sverdlovsk region, Russia: biodiverstiy, distribution and the IUCN threat categories / A.G. Shiryaev, H. Kotiranta, V.A. Mukhin et al. Ekaterinburg: Goshchitskiy Publisher, 2009. 304 p.
Кудеяров В.Н., Заварзин Г.А., Благодатский С.А. и др. Пулы и потоки углерода в наземных экосистемах России. М.: Наука, 2007. 315 с.
Mukhin V.A., Diyarova D.K., Gitarskiy M.L., Zamolodchikov D.G. Carbon and Oxygen Gas Exchange in Woody Debris: the Process and Climate-Related Drivers // Forests. 2021. V. 12. № 9. P. 1156–1171. https://doi.org/10.3390/f12091156
Peintner U., Pöder R., Pümpel T. The Iceman’s fungi // Mycological Research. 1998. V. 102. P. 1153–1162. https://doi.org/10.1017/s0953756298006546
Pöder R., Peintner U. Laxatives and the ice man // The Lancet. 1999. V. 353. P. 926–926. https://doi.org/10.1016/S0140-6736(05)75032-7
Peintner U., Pöder R. Ethnomycological remarks on the Iceman’s fungi // The Iceman and His Natural Environment / Eds. Bortenschlager S., Oeggl K. Vienna: Springer, 2000. P. 143–150.
Roussel B., Rapior S., Masson C., Boutié P. Fomes fomentarius (L.: Fr.) Fr.: un champignon aux multiples usages // Cryptogamie Mycologie. 2002. V. 23. P. 349–366.
Grienke U., Zöll M., Peintner U., Rollinger J.M. European medicinal polypores – a modern view on traditional uses // Journal of Ethnopharmacology. 2014. V. 154. P. 564–583. https://doi.org/10.1016/j.jep.2014.04.030
Gáper J., Gáperova S., Pristas P., Naplavova K. Medicinal value and taxonomy of the tinder polypore, Fomes fomentarius (Agaricomycetes): a review // Int. J. Med. Mushrooms. 2016. V. 18. № 10. P. 851–859. https://doi.org/10.1615/intjmedmushrooms.v18.i10.10
Mukhin V.A., Zhuykova E.V., Badalyan S.M. Genetic variability of the medicinal tinder bracket polypore, Fomes fomentarius (Agaricomycetes), from the Asian Part of Russia // Int. J. Med. Mushrooms. 2018. V. 20. № 6. P. 561–568. https://doi.org/10.1615/IntJMedMushrooms.2018026278
Peintner U., Kuhnert-Finkernagel R., Wille V. et al. How to resolve cryptic species of polypores: an example in Fomes // IMA Fungus. 2019. V. 10. № 17. P. 1–21. https://doi.org/10.1186/s43008-019-0016-4
Gilbertson R.L., Ryvarden L. North American Polypores. V. 1: Abortiporus-Lindtneria. Oslo, Norway: Fungiflora, 1986. 433 p.
Lécuru C., Courtecuisse R., Moreau P.-A. Nomenclatural novelties // Index Fungorum. 2019. V. 384. P. 1–2.
Judova J., Dubikova K., Gáperova S. et al. The occurrence and rapid discrimination of Fomes fomentarius genotypes by ITS-RFLP analysis // Fungal Biol. 2012. V. 116. P. 155–160. https://doi.org/10.1016/j.funbio.2011.10.010
Pristas P., Gáperova S., Gáper J., Judova J. Genetic variability in Fomes fomentarius reconfirmed by translation elongation factor 1-α DNA sequences and 25S LSU rRNA sequences // Biologia. 2013. V. 68. № 5. P. 816–820. https://doi.org/10.2478/s11756-013-0228-9
Zhuykova E., Mukhin V. Fomes fomentarius in the Urals and adjacent territories from the Institute of Plant and Animal Ecology herbaria. Version 1.2. Institute of Plant and Animal Ecology (IPAE). Occurrence dataset. 2021. https://doi.org/ accessed via GBIF.org on 2022-02-22https://doi.org/10.15468/xnqzar
Gardes M., Bruns T.D. ITS primers with enhanced specificity for Basidiomycetes – application to the identification of mycorrhizae and rusts // Mol. Ecol. 1993. V. 2. P. 113–118. https://doi.org/10.1111/j.1365-294x.1993.tb00005.x
Kumar S., Stecher G., Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets // Mol. Biol. Evol. 2016. V. 33. P. 1870–1874. https://doi.org/10.1093/molbev/msw054
Hasegawa M., Kishino H., Yano T. Dating the human-ape split by a molecular clock of mitochondrial DNA // J. of Mol. Evol. 1985. V. 22. P. 160–174. https://doi.org/10.1007/BF02101694
Rozas J., Ferrer-Mata A., Sánchez-DelBarrio J.C. et al. DnaSP 6: DNA Sequence Polymorphism Analysis of Large Datasets // Mol. Biol. Evol. 2017. V. 34. P. 3299–3302. https://doi.org/10.1093/molbev/msx248
Garrido-Benavent I., Velasco-Santos J.M., Pérez-De-Gregorio M.Ȧ. et al. Fomes inzengae (Ces. & De Not.) Cooke in the Iberian Peninsula // Butll. Soc. Micol. Valenciana. 2020. V. 24. P. 151–170.
Náplavová K., Gáper J., Gáperová S. et al. Genetic and plant host differences of Fomes fomentarius in selected parts of Southern Europe // Plant Biosystems. 2020. V. 154. № 1. P. 125–127. https://doi.org/10.1080/11263504.2019.1701129
Nilsson R.H., Kristiansson E., Ryberg M. et al. Intraspecific ITS Variability in the Kingdom Fungi as Expressed in the International Sequence Databases and Its Implications for Molecular Species Identification // Evolutionary Bioinformatics. 2008. V. 4. P. 193–201. https://doi.org/10.4137/EBO.S653
Dresch P., D’Aguanno M., Rosam K. et al. Fungal Strain Matters: Colony Growth and Bioactivity of the European Medicinal Polypores Fomes fomentarius, Fomitopsis pinicola and Piptoporus betulinus // AMB Express. 2015. V. 5. № 4. P. 1–14. https://doi.org/10.1186/s13568-014-0093-0
Donk M.A. Revision der Nederländischen Homobasidiomyceteae-Aphyllophoraceae II // Mededeelingen van het botanisch Museum en Herbarium van de Rijks Universiteit te Utrecht. 1933. V. 9. 279 p.
Pilát A. Atlas des Champignons de l’Europe. T. III. Polyporaceae. Praha,1941. P. 344–348.
Биологический энциклопедический словарь / Ред. Гиляров М.С., Баев А.А., Винберг Г.Г. и др. М.: Сов. энциклопедия, 1986. 831 с.
Дополнительные материалы отсутствуют.