Физиология растений, 2020, T. 67, № 3, стр. 259-269
Регуляторные контексты в 5'-области мРНК растений Arabidopsis thaliana и их роль в эффективности трансляции
К. В. Кабардаева a, А. А. Тюрин a, Ф. Кучоро a, О. Н. Мустафаев b, И. В. Дейнеко a, В. С. Фадеев a, И. В. Голденкова-Павлова a, *
a Федеральное государственное бюджетное учреждение науки Институт физиологии растений
им. К.А. Тимирязева Российской академии наук
Москва, Россия
b Институт генетических ресурсов Национальной академии наук Азербайджана
Баку, Азербайджан
* E-mail: irengold58@gmail.com
Поступила в редакцию 16.08.2019
После доработки 18.10.2019
Принята к публикации 30.10.2019
Аннотация
В этом исследовании применили метод профилирования полисом для разделения мРНК в зависимости от их загрузки рибосомами, а именно на полисомные и моносомные фракции. Разделение пулов таких мРНК и анализ транскриптов (мРНК), характеризующихся постоянным уровнем транскрипции в широком диапазоне абсолютных значений на всех этапах онтогенеза растений и ассоциированных с каждым пулом мРНК, за счет секвенирования РНК, позволили получить представление о трансляционной эффективности индивидуальных мРНК, а последующий in silico анализ – провести поиск регуляторных контекстов в 5'-области мРНК растений A. thaliana, которые могут быть потенциально важными для эффективной трансляции мРНК. Результаты проведенного исследования позволили установить, что пиримидиновые ди-нуклеотиды и мотивы характерны для 5'-нетранслируемой области мРНК с высокой трансляционной эффективностью, тогда как пуриновые ди-нуклеотиды и мотивы ассоциированы с транскриптами, имеющими низкую трансляционную эффективность.
ВВЕДЕНИЕ
Трансляция – многоэтапный процесс с неочевидным для исследователей механизмом контроля, включает три ключевых этапа – инициацию, элонгацию и терминацию, а также возобновление трансляции с данной мРНК. На каждом этапе трансляции могут быть задействованы один или совокупность нескольких регуляторных контекстов мРНК, и сложная сеть механизмов их взаимодействия [1–4]. Согласно текущему мнению, инициация трансляции является круциальным этапом. На этапе инициации трансляции происходит иммобилизация рибосом на матрице мРНК, которая обеспечивается факторами инициации трансляции и сайтами связывания малой субъединицы рибосомы. Контексты в мРНК, которые важны на передовом фланге инициации трансляции – это кэп-структуры и 5'-нетранслируемая область (5'-НТО), последняя может действовать и как самостоятельный регуляторный контекст, и как контейнер разнообразных регуляторных мотивов меньшего размера [1–4]. Для инициации трансляции критическим также является правильное и эффективное распознавание стартового AUG кодона, решающую роль в котором играет его нуклеотидное окружение, так называемая консенсусная последовательность Козак [5].
Применение новых экспериментальных инструментов геномного мониторинга экспрессии генов (таких, например, как RNA-Seq и ДНК-микрочипы), а также системных трансляционных измерений in vivo (таких, например, как профилирование рибосом, Ribosome Profiling (или RiboSeq)) [6–8] выявило широкую и удивительную сложность декодирования генома растений. В ряде исследований корреляционный анализ между накоплением транскрипта и белка в масштабе всего генома позволил предположить, что накопление белков у растений A.thaliana, в первую очередь, регулируется уровнем индивидуального транскрипта [9]. В других исследованиях представлены убедительные доказательства диссонанса между обилием индивидуальных мРНК (транскриптом) и обилием соответствующего белка (протеом) [2]. Более удивительным стал тот факт, что наблюдаемые флуктуации в уровне транскриптов не обязательно приводят к ожидаемому изменению уровня их трансляции, как во время воздействия различных стрессовых факторов [6, 10], так и во время роста и развития растений [7]. Например, продемонстрировано, что трансляция мРНК у растений снижается при тепловом стрессе, так как отмечается избирательное обогащение рибосомами определенных мРНК (в основном тех, которые кодируют белки, участвующие в ответах на стресс), что обеспечивает их избирательную трансляцию [10]. Несоответствие между уровнями мРНК и белка относят, главным образом, к вариабельности трансляции и/или оборота белка [2, 9, 10].
Учитывая ключевую роль трансляции в общем механизме реализации генетической информации, а также тот факт, что правила регуляции и декодирования мРНК у растений, как и других эукариот, еще до конца не ясны, можно заключить, что новые знания о регуляторных контекстах в мРНК и их роли в эффективности трансляции, крайне необходимы исследователям. Важным представляется применение не только новых экспериментальных методов, но и вычислительных алгоритмов, которые позволили бы выяснить, какие регуляторные контексты в мРНК растений могут быть потенциально критичными для их эффективной трансляции при различных условиях жизнедеятельности или на разных этапах роста и развития растений.
В этом исследовании применили метод профилирования полисом, основанный на том, что в клетке присутствуют активно транслируемые мРНК и трансляционно покоящиеся мРНК, которые связаны со множеством рибосом (полисомы) или одиночными рибосомами (моносомы). Разделение двух пулов таких мРНК в градиенте сахарозы и анализ транскриптов (мРНК), ассоциированных с каждым пулом мРНК, за счет секвенирования РНК, позволяет оценить трансляционную эффективность индивидуальных мРНК. Поиск регуляторных контекстов в 5'-области мРНК растений A. thaliana, которые ассоциированы с транскриптами, имеющими разный трансляционный статус, за счет in silico анализа позволяет выявить регуляторные контексты, которые потенциально могут быть важными для эффективной трансляции мРНК.
МАТЕРИАЛЫ И МЕТОДЫ
Растительный материал. В работе использовали растения Arabidopsis thaliana экотипа Columbia-0 в стадии развития третьего розеточного листа (стадия развития 3.20; ~28 дней). Растения выращивали при 22 ± 1°C, 12-часовом фотопериоде, освещенности 100 мкмоль квантов/(м2 с).
Получение моносомных и полисомных фракций, выделение РНК из фракций и тотальной РНК. Разделение моносомных и полисомных фракций проводили дифференциальным центрифугированием в градиенте сахарозы согласно методу, описанному раннее [6]. Кратко, растительный материал (листья) гомогенизировали в буфере, содержащем 0.2 M Tris pH 9.0, 0.2 M KCl, 0.025 M ЭГТА, 0.035 M MgCl2, 1% DOC, 1% Тритон, 5 mM DTT, 50 мг/мл циклогексимида, 50 мг/мл хлорамфеникола. Клеточные экстракты наносили поверх 5 мл 15–60% (W/v) градиента сахарозы и центрифугировали при 237 000 g в течение 1.5 часов при 4°С. Фракции, объемом 400 мкл, отбирали вручную. Из каждой фракции экстрагировали суммарную РНК с использованием набора ExtractRNA (ЗАО “Евроген”, Россия). В каждой фракции оценивали содержания РНК с помощью прибора Nanodrop ND-1000 (“LabTech International”, Великобритания). Суммарную РНК выделяли из неочищенного клеточного экстракта растений в буфере до загрузки на градиент сахарозы и экстрагировали с использованием набора ExtractRNA (ЗАО “Евроген”, Россия). Качество и количество препаратов суммарной РНК (далее обозначаемая как ТФ) и РНК из полисомных (далее обозначаемая как ПФ) и моносомных (далее обозначаемая как МФ) фракций растений оценивали на Agilent Bioanalyzer 2100.
Подготовка образцов РНК, секвенирование, сборка транскриптома и картирование. Образцы РНК (суммарной, а также из моносомной и полисомной фракций) обрабатывали ДНКазой (Qiagen, США), и очищали с использованием RNeasy kit (Qiagen, США). Образцы РНК для секвенирования получали с использованием набора TruSeq Stranded mRNA Sample Prep Kit (Illumina). Качество полученных библиотек проверяли с помощью Agilent Bioanalyzer 2100 и ПЦР в реальном времени (qPCR). Секвенирование библиотек проводили на 1 дорожке Illumina HiSeq 4000 (101 цикл с двух сторон фрагментов) с использованием реактивов HiSeq 4000 sequencing kit version 1 (ЗАО “Евроген”, Россия). Файлы FASTQ получены с помощью программного обеспечения bcl2fastq v2.17.1.14 Conversion Software (Illumina). Формат записи строки данных о качестве – Phred 33. Качество прочтений оценено в программе FastQC. Прочтения, полученные в результате секвенирования, отфильтрованы для удаления последовательностей адаптера; прочтений, содержащих более 10% неизвестных оснований; и последовательностей низкого качества.
Относительный уровень транскрипции (lgCPM) рассчитывали, как логарифмическое отношение количества прочтений на последовательности аннотированного гена, которые нормализованы на миллион (counts per million – CPM) согласно протоколу [11]. Показатель трансляционной эффективности (lgFC ПФ/МФ) рассчитывали, как логарифмическое отношение количества прочтений для отдельных транскрипционных единиц, представленных в полисомной (ПФ) и моносомной (МФ) фракциях [12]. Для сравнения логарифмических соотношений использован t-критерий Стьюдента (двусторонняя неравная дисперсия) [12].
Метод количественной ПЦР в реальном времени для валидации результатов РНК-секвенирования. Данные RNA-Seq подтверждали при помощи qPCR с праймерами и зондами к генам At2g05170, At3g57890, At5g06600, At4g09000, At2g47170, At5g46210, At4g27960, At3g20290 и At5g65940 (Supplementary Table 1 ). qPCR проводили, используя StepOnePlus Real-Time PCR System (“Applied Biosystems”, США), SYBR Green как флуоресцентный краситель. Программа амплификации: 95°С – 3 минуты, затем 40 циклов с детекцией на стадии отжига: 95°С – 10 с, 61°С – 20 с. Три повторности qPCR и значения Ct были использованы для количественной оценки уровня транскрипции.
In silico анализ. Первоначально сформировали списки последовательностей 5'-НТО, согласно принадлежности их мРНК к кластерам с различной эффективностью трансляции (высокая, средняя и низкая) (принцип разделения последовательностей 5'-НТО на кластеры описан в разделе “Результаты и обсуждение”). Для получения данных о нуклеотидных мотивах (контекстах) в 5'-НТО мРНК A. thaliana применен следующий новый алгоритм их поиска. Первоначально заданы базовый список вида [A, T, G, C], списки анализируемых последовательностей 5'-НТО и порог встречаемости мотивов в каждом кластере 5'-НТО, выраженный в % (для анализа использовали нижний порог встречаемости мотива – 50%). Для каждого нуклеотида рассчитывали его встречаемость в каждом кластере последовательностей 5'-НТО. Далее на основе каждого нуклеотида генерировали 4 ди-нуклеотида за счет присоединения к их 3'-концу одного из четырех нуклеотидов, в тестовом списке проверяемый нуклеотид заменялся на 4 его потомка – ди-нуклеотида (обозначали их как мотивы) и рассчитывали их встречаемость в каждом кластере последовательностей 5'-НТО. В том случае, если встречаемость мотива превышала заданный порог, данный мотив и частота его встречаемости записывались в результирующий список. Далее на основе мотива с допустимым порогом встречаемости генерировали 4 новых мотива за счет присоединения к его 3'-концу одного из четырех нуклеотидов, и в тестовом списке проверяемый мотив вновь заменялся на 4 его потомка. В случае если встречаемость мотива была меньше заданного порога встречаемости (50%), данный мотив удалялся из тестового списка. Анализ повторяли рекурсивно, пока тестовый список мотивов не становился пустым. Заключительный этап анализа – результирующий список мотивов и их частот встречаемости в итоговом списке 5'-НТО для каждого кластера последовательностей, а именно: количество последовательностей 5'-НТО с мотивом; максимальное число мотивов в каждой последовательности 5'-НТО и среднее число мотивов в последовательностях 5'‑НТО из анализируемого кластера. Консенсусные последовательности окружения стартового кодона оценены с использованием программного обеспечения WebLogo [13].
Статистический анализ. Статистическую обработку результатов проводили с использованием программы Statistica for Windows v. 9.0 (применяли t-критерий Стьюдента для независимых выборок, Р = 0.05) и графопостроителя Microsoft Office Excel 2007. Для сравнения логарифмических соотношений использован t-критерий Стьюдента (двусторонняя неравная дисперсия) [12]. Выделение суммарной РНК и РНК из полисомных и моносомных фракций растений A. thaliana, а также РНК секвенирование проведено в трех независимых биологических повторностях.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Согласно текущему мнению, ключевую роль в инициации трансляции играет 5'-область транскрипта, которая не только задействована в иммобилизации малой субъединицы рибосомы, но и обеспечивает дальнейшее продвижение трансляционного комплекса вдоль мРНК до стартового AUG кодона, с которого и начнется синтез соответствующего белка [3, 4, 7]. Подобный сценарий инициации трансляции характерен для подавляющего большинства мРНК эукариот, и растения – не исключение.
В этом исследовании мы попытались выяснить, какие регуляторные контексты в 5'-области мРНК растений A. thaliana могут быть потенциально важными для эффективной трансляции мРНК.
Для прояснения этого вопроса первоначально сформировали выборку генов для проведения экспериментальных работ и последующего in silico анализа. Формирование выборки генов проведено, исходя из следующего критерия: выбранные гены характеризуются постоянным уровнем транскрипции в широком диапазоне абсолютных значений на всех этапах онтогенеза растений (Supplementary Table 2 ) [14]. Этот выбор базируется на следующем соображении – у таких генов уровень мРНК каждого индивидуального гена сохраняется на постоянном уровне во время онтогенеза, что позволит избежать модуляции в эффективности трансляции, потенциально возможной за счет изменения количества индивидуального транскрипта. В выборку включено 130 последовательностей с низким коэффициентом вариации транскрипции (показатель SD/MV, Supplementary Table 2 ), который характеризует стабильность транскрипции на всех изученных ранее стадиях развития A. thaliana [14].
Анализ транскрипционной и трансляционной эффективности мРНК
Далее мы попытались выяснить, есть ли корреляция между уровнем транскрипции у сформированной выборки генов и трансляционной эффективностью их мРНК. Для этого проведено выделение и секвенирование суммарной мРНК, а также разделение мРНК с дифференциальной загрузкой рибосомами, а именно на фракции полисомных и моносомных РНК методом дифференциального центрифугирования, с последующим формированием суммарной полисомной (ПФ) и суммарной моносомной (МФ) фракций (рис. 1), мРНК из которых также выделена и секвенирована.
Рис. 1.
Полисомный профиль, оцененный по содержанию РНК во фракциях. I – фракции моносом, II – фракции полисом.
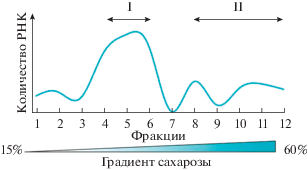
В результате RNA-Seq получены высококачественные прочтения (Q30 > 84%) в общей сложности: для МФ – 1.57 × 108; для ПФ – 1.43 × 108; и для образцов тотальной мРНК (ТФ) – 1.68 × 108. Полученные прочтения с удаленными адаптерами картированы на геном A. thaliana (TAIR10), оценены профиль охвата прочтений вдоль хромосом и профиль покрытия прочтений по длине отдельных транскрипционных единиц для трех наборов прочтений – ТФ, МФ и ПФ. Сравнение этих показателей выявило, что все наборы данных имеют сходные профили охвата хромосом и сопоставимые профили покрытия аннотированных кДНК TAIR10 (данные в статье не приводятся). Полученные результаты свидетельствуют о возможности использования трех наборов данных RNA-seq для проведения объективного сравнительного анализа транскрипционной и трансляционной эффективности генов [15].
Для каждого гена из сформированной выборки оценивали: (1) относительный уровень транскрипции – показатель lgCPM (см. Материалы и методы), [11]; и (2) трансляционную эффективность – показатель lgFC ПФ/МФ (см. Материалы и методы) (Supplementary Table 2 ).
Используя показатели относительного уровня транскрипции (lgCPM) и трансляционной эффективности (lgFC ПФ/МФ), построена эмпирическая функция распределения мРНК для каждого транскрипта (рис. 2). Далее все транскрипты разделяли на кластеры, в зависимости от (1) уровня транскрипции, использовали следующие критерии для классификации: lgCPM > 6 – высокий, 3 < lgCPM < 6 – средний и lgCPM < 3 – низкий; и (2) эффективности трансляции, использовали следующие критерии для классификации: lgFC ПФ/МФ > 0.5 – высокая, –0.5 > lgFC ПФ/МФ < 0.5 – средняя и lgFC ПФ/МФ < –0.5 – низкая (табл. 1).
Рис. 2.
Распределения мРНК в зависимости от уровня транскрипции и трансляции. Заливка фона отражает разделение мРНК на кластеры в зависимости от уровня транскрипции и эффективности трансляции; транскрипция, логарифмическая шкала – значения LgCPM; трансляция, логарифмическая шкала – значения LgFC ПФ/МФ.
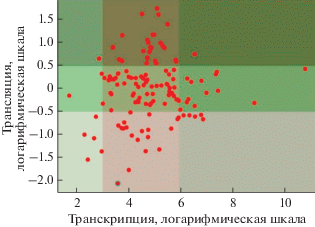
Таблица 1.
Классификация выборки генов в зависимости от уровня транскрипции и трансляционной эффективности
| Эффектность трансляции | Уровень транскрипции | ||
|---|---|---|---|
| высокий | средний | низкий | |
| Высокая (кластер 1) | 2 | 26 | 1 |
| Средняя (кластер 2) | 12 | 58 | 2 |
| Низкая (кластер 3) | 5 | 19 | 5 |
Как видно из представленных данных, каждый кластер, характеризующийся различной эффективностью трансляции (высокая, средняя и низкая) включает гены с разным уровнем транскрипции (табл. 1). Мы рассчитали коэффициенты корреляции между эффективностью трансляции (значения lgFC ПФ/МФ) и уровнями транскрипта (значения lgCPM) для общей выборки, а также для каждого кластера транскриптов. Для всей выборки транскриптов коэффициент корреляции составил 0.127, для транскриптов кластера 1 (высокая эффективность трансляции) – 0.349, для кластера 2 (средняя эффективность трансляции) – 0.134, и кластера 3 (низкая эффективность трансляции) – 0.098. Эти результаты свидетельствуют о том, что корреляция между уровнем транскрипции генов и трансляционной эффективностью их мРНК у сформированной выборки слабая.
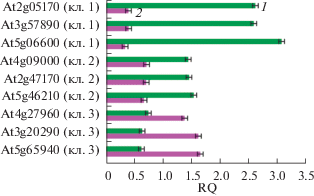
Классификация генов по эффективности трансляции подтверждена методом количественной ПЦР в реальном времени. Для этого из каждого кластера генов выбраны последовательности (см. Материалы и методы), и оценены относительные количества (RQ) транскриптов этих генов в суммарной полисомной (ПФ) и суммарной моносомной (МФ) фракциях (рис. 3). Как видно, распределение представленности транскриптов из каждого кластера между полисомной и моносомной фракциями соответствует ожидаемому распределению.
Рис. 3.
Относительные количества (RQ) транскриптов генов из кластеров с разной трансляционной эффективностью. Зеленые (1) и сиреневые (2) бары – полисомная и моносомная фракция соответственно; At2g05170, At3g57890, At5g06600, At4g09000, At2g47170, At5g46210, At4g27960, At3g20290 и At5g65940 – AGI (Arabidopsis Gene Identification) генов из разных кластеров транскриптов; кл.1, кл.2 и кл.3 – обозначение кластеров 1, 2 и 3 соответственно, где кластер 1, 2 и 3 – транскрипты с высокой, средней и низкой эффективностью трансляции соответственно.
Поиск регуляторных контекстов в кластерах генов с разной трансляционной эффективностью
В настоящее время длина, присутствие стартовых кодонов, локализованных выше основного (upstream start codons – uAUGs), нуклеотидный состав в 5'-НТО мРНК, а также нуклеотидный контекст вокруг стартового AUG кодона пристально исследуются с точки зрения их вклада в эффективность трансляции [3, 4, 7].
Для того чтобы выяснить, какие характеристики 5'-области мРНК, в том числе и их регуляторные контексты характерны для генов с разной трансляционной эффективностью, проведен сравнительный in silico анализ последовательностей 5'-областей из разных кластеров генов (Supplementary Table 2 ).
Длина 5'-НТО и эффективность трансляции
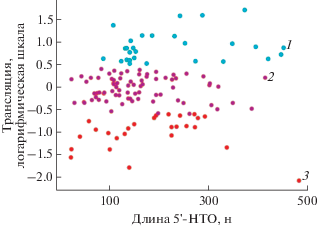
Ранние результаты исследований по системному анализу взаимосвязи между длиной 5'-НТО и нагрузкой транскриптов рибосомами у A. thaliana показали, что (i) 5'-НТО длиной от 50 до 75 нуклеотидов обеспечивают оптимальную нагрузку рибосомами, а (ii) короткие 5'-НТО (менее 25 нуклеотидов) и длинные 5'-НТО (от 175 нуклеотидов и выше) могут ингибировать нагрузку транскрипта рибосомами [1, 16]. Результаты нашего сравнительного исследования длин 5'-НТО из разных кластеров генов демонстрируют отсутствие столь явной зависимости длины этой области и эффективности трансляции (рис. 4). Так, длина 5'-НТО у транскриптов в каждом кластере варьирует примерно в сходных границах: от 85 до 451 н., от 25 до 414 н. и от 24 до 482 н., для кластеров с высокой, средней и низкой трансляционной эффективностью соответственно (Supplementary Table 2 ). Ряд современных экспериментальных данных согласуется с полученными нами результатами о том, что взаимосвязь между длиной 5'-НТО и нагрузкой транскриптов рибосомами не всегда имеет место [17–19]. Предполагается, что длина 5'-НТО, вероятно, не является самостоятельным фактором, определяющим эффективность трансляции, но увеличивает вероятность вхождения в ее последовательность элементов, которые могут модулировать эффективность трансляции. Ниже оценили присутствие других регуляторных контекстов в 5'-НТО и их потенциальный вклад в трансляционный статус мРНК.
Рис. 4.
Распределение транскриптов из разных кластеров в зависимости от длины 5’-НТО и эффективности трансляции. Голубые (1), сиреневые (2) и красные (3) кружки – обозначение распределения транскриптов из кластеров 1, 2 и 3 соответственно, где кластер 1, 2 и 3 – транскрипты с высокой, средней и низкой эффективностью трансляции соответственно.
Наличие стартовых кодонов, локализованных выше основного (uAUGs) в 5'-НТО и эффективность трансляции
Ряд 5'-НТО клеточных мРНК содержит так называемые стартовые кодоны, локализованные выше основного (обозначаемые как uAUGs). С одной стороны, такие стартовые кодоны, в том числе и многочисленные, могут, в конечном счете, привести к синтезу множества функциональных белков. С другой стороны, их функциональная роль может заключаться в тонкой регуляции трансляции, а именно, сделать в целом трансляцию с подобной мРНК неэффективной и, таким образом, уменьшить количество синтезируемого белка [1, 2].
Мы провели in silico анализ 5'-НТО мРНК из разных кластеров на наличие в них uAUGs. При поиске учитывали нуклеотидное окружение таких стартовых кодонов, согласно классической последовательности Козак – присутствие пурина (A/G) в положении –3 и гуанина в +4 [20]. Сравнительный анализ показал, что у генов из кластера 3 (низкая трансляционная эффективность, оцененная по загрузке мРНК рибосомами) количество uAUGs в 5'-НТО мРНК достоверно выше. У 5'-НТО мРНК из этого кластера 20% последовательностей имеют uAUGs с оптимальным нуклеотидным окружением согласно последовательности Козак, по сравнению с кластером 1 (высокая трансляционная эффективность) – 3% и кластером 2 (средняя трансляционная эффективность) – 6% (табл. 2 и Supplementary Table 2 ).
Таблица 2.
Процент встречаемости uAUGs, моно- и динуклеотидов в 5'-НТО транскриптов из кластеров с разной трансляционной эффективностью
| Параметр | Кластеры мРНК | ||
|---|---|---|---|
| 1 | 2 | 3 | |
| Наличие uAUGs, % | 3 | 6 | 20* |
| Мононуклеотиды, % | |||
| A | 27.6 | 30.4* | 28.3 |
| C | 22.8* | 21 | 21.8 |
| G | 17.7 | 18 | 19.3* |
| U | 31.9* | 30.6 | 30.6 |
| Динуклеотиды, % | |||
| AA | 10.7 | 12.5* | 10.7 |
| AC | 4.1 | 4.3 | 5.2* |
| AG | 5.1 | 6.3 | 6.2 |
| AU | 7.7* | 7.2 | 6.3 |
| CA | 5.4 | 5.8 | 5.4 |
| CC | 4.4 | 4.1 | 4.8 |
| CG | 4.9 | 4.1 | 4.7 |
| CU | 8.2* | 7 | 6.9 |
| GA | 7.1 | 7.2 | 7.4 |
| GC | 3 | 3 | 3.1 |
| GG | 3.1 | 3.5 | 4* |
| GU | 4.4 | 4.4 | 4.6 |
| UA | 4.4 | 4.9 | 4.9 |
| UC | 11.3* | 9.7 | 8.7 |
| UG | 4.6 | 4.1 | 4.4 |
| UU | 11.6 | 11.9 | 12.7 |
Таким образом, нами продемонстрировано, что uAUGs с оптимальным для трансляции нуклеотидным окружением потенциально могут быть важны для эффективной трансляции. Это предположение согласуется с современными экспериментальными данными, в которых приведены убедительные доказательства, а именно, доказано, что удаление uAUGs из мРНК приводит к значительному (на порядок) увеличению уровня синтеза целевого белка [1, 2].
Нуклеотидный контекст в 5'-НТО мРНК и его роль в эффективности трансляции
За счет полногеномного секвенирования многих видов растений выявлено, что нуклеотидные композиции 5'-НТО мРНК проявляют большое разнообразие. При этом крайне важно понять, как такое разнообразие отражается на эффективности трансляции, т.е. есть ли особенности в нуклеотидной композиции 5'-НТО мРНК растений, которые различаются по своему трансляционному статусу.
Ранние результаты системного анализа взаимосвязи между композицией моно- и ди-нуклеотидов в 5'-НТО и нагрузкой транскриптов рибосомами у A. thaliana показали, что: (i) мРНК с высокой рибосомной нагрузкой обычно имеют в 5'-НТО высокое содержание аденина (A), и ди-нуклеотидов AU и AC; (ii) 5'-НТО мРНК слабо нагруженных рибосомами, как правило, имеют повышенное содержание гуанина (G), и ди-нуклеотида GU [1].
Результаты нашего сравнительного in silico исследования композиции моно- и динуклеотидов 5'-НТО из разных кластеров транскриптов позволили установить следующие закономерности: (1) отмечена достоверно высокая представленность нуклеотидов С и U и ди-нуклеотидов CU и UC в дополнении к известному AU ди-нуклеотиду в 5'-НТО мРНК с высокой трансляционной эффективностью (кластер 1); (2) установлена достоверно высокая представленность нуклеотида G и ди-нуклеотида GG в 5'-НТО мРНК с низкой трансляционной эффективностью (кластер 3); (3) выявлена достоверно высокая представленность нуклеотида А и ди-нуклеотида АА в 5'-НТО мРНК со средней трансляционной эффективностью (кластер 2) (табл. 2).
В результате дальнейшего in silico анализа получены данные о нуклеотидных мотивах в 5'-НТО мРНК A. thaliana, которые встречаются не менее, чем в 50% последовательностей для каждого кластера транскриптов. Результирующий список включал 64, 102 и 37 мотивов, содержащих 3, 4 и 5 нуклеотидов соответственно. Следует отметить, что мотивов большего размера, чем 5 нуклеотидов при заданном пороге поиска (не менее, чем в 50% 5'-НТО) не выявлено. Дальнейший анализ результирующего списка мотивов акцентирован на том, чтобы выбрать из него общие мотивы для всех кластеров транскриптов, а также уникальные нуклеотидные мотивы в 5'-НТО для каждого кластера транскриптов.
В результате этого анализа из большого числа мотивов первичного списка было экстрагировано 26, 1 и 5 уникальных нуклеотидных мотива длиной 5 нуклеотидов, характерных для 5'-НТО транскриптов из кластеров с высокой, средней и низкой трансляционной эффективностью соответственно (рис. 5), один мотив, который является общим для транскриптов всех кластеров – CAAAA, а также четыре мотива, которые выявлены в 5'-НТО у транскриптов кластера 1 и 2 (рис. 5).
Рис. 5.
Уникальные нуклеотидные мотивы длиной 5 нуклеотидов, характерные для 5'-НТО транскриптов из кластеров с высокой (голубые бары, 1), средней (фиолетовые бары, 2) и низкой (красные бары, 3) трансляционной эффективностью. На оси абсцисс указано среднее количество таких мотивов в 5'-НТО проанализированных мРНК. На оси ординат указаны пуриновые (I), пиримидиновые (II) и смешанные (III) мотивы длиной 5 нуклеотидов, характерные для 5'-НТО транскриптов разных кластеров.
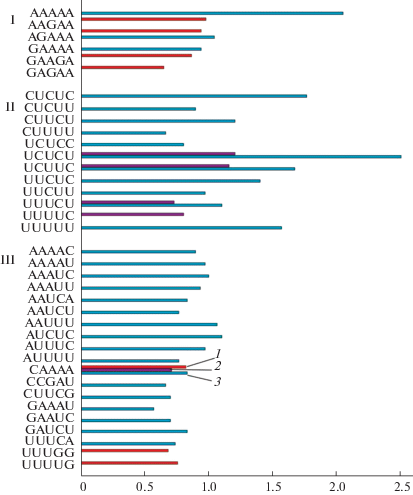
Как видно из представленных данных, выявленные мотивы длиной 5 нуклеотидов можно разделить на следующие группы: пуриновые, пиримидиновые и смешанные (содержащие пуриновые и пиримидиновые основания). При этом смешанные мотивы представлены, как мотивами преимущественно содержащими пурины (A и/или G), так и преимущественно содержащими пиримидины (C и/или U). Согласно ассоциации таких мотивов и их представленности в индивидуальных 5'-НТО, эти области у транскриптов кластера 1 и 2 несут, в основном, мотивы, имеющие преимущественно пиримидиновые мотивы или смешанные мотивы с преимущественным содержанием пиримидинов (рис. 5, голубые (1) и фиолетовые (2) бары соответственно), тогда как в 5'-НТО транскриптов кластера 3 такие мотивы представлены, в основном, пуринами, за исключением мотивов UUUGG и UUUUG (рис. 5, красные бары (3)).
Основываясь на данных о количестве последовательностей 5'-НТО с индивидуальным мотивом можно выделить наиболее значимые для каждого кластера последовательностей мотивы: для кластера 1 (транскрипты с высокой трансляционной эффективностью) – это UCUCU (этот мотив имеет двадцать одна 5'-НТО), и CUCUC (шестнадцать 5'-НТО); AAGAA (шестнадцать 5'-НТО), UUUUC (сорок две 5'-НТО) для кластера 2 (транскрипты со средней трансляционной эффективностью) и GAAGA (пятнадцать 5'-НТО) для кластера 3 (транскрипты с низкой трансляционной эффективностью).
Таким образом, результаты проведенного анализа позволяют предположить, что пиримидины, в том числе пиримидиновые ди-нуклеотиды и мотивы длиной 5 нуклеотидов характерны для 5'-НТО мРНК с высокой трансляционной эффективностью (кластер 1), тогда как пурины, пуриновые ди-нуклеотиды, а также пуриновые мотивы длиной 5 нуклеотидов являются характерным параметром для 5'-НТО мРНК со средней (кластер 2) и низкой (кластер 3) трансляционной эффективностью. Высокая представленность этих мотивов у транскриптов из разных кластеров, может указывать на то, что такие мотивы могут быть потенциально задействованы в контроле эффективности трансляции. В пользу этого предположения свидетельствуют современные данные, полученные другими исследователями. Так, в 5'-НТО мРНК выявлены некоторые регуляторные контексты, которые вовлечены в программирование инициации трансляции. Один из них – это пиримидиновый трек, локализованный в непосредственной близости от кэп-структуры 5'-НТО, и который функционирует как трансляционный энхансер. Этот регуляторный элемент обнаружен у многих мРНК растений [21, 22], прежде всего, у мРНК, белковые продукты которых вовлечены в клеточное деление и сопутствуют многим этапам развития организмов. Согласно текущему мнению, этот механизм регуляции трансляции может быть более распространенным, чем сейчас полагают [22]. Мы оценили локализацию мотивов на последовательностях 5'-НТО из разных кластеров. Выявлено, что пиримидиновые мотивы в 5'-НТО транскриптов 1 кластера локализованы преимущественно в 3'‑области 5'-НТО (данные в статье не приводятся), а пуриновые мотивы в 5'-НТО транскриптов 3 кластера – преимущественно в 5'-области 5'-НТО (данные в статье не приводятся).
В другом исследовании показано наличие светового запуска трансляционного контроля для транскриптов, имеющих TAGGGTTT или AAAACCCT мотивы в их 5'-НТО мРНК, и такие мотивы выявлены в 5'-НТО многих генов у A. thaliana [23]. Функциональная роль этих мотивов в регуляции экспрессии генов именно на трансляционном уровне подтверждена экспериментально, хотя механизм, посредством которого происходит контроль, пока не установлен. Высказано лишь предположение, что они могут контролировать трансляцию за счет формирования шпилеподобной структуры на мРНК [17].
Последние исследования по глубокому секвенированию фрагментов мРНК, предоставили доказательства того, что вторичная структура вокруг стартового кодона снижает эффективность трансляции [17]. Полагают, что высокое содержание AU в 5'-НТО, которое уменьшает формирование вторичной структуры, позволяет рибосоме лучше сканировать стартовый AUG кодон и, следовательно, в положительной манере сказывается на эффективности трансляции [22, 24]. Следует подчеркнуть, что последовательности 5'-НТО транскриптов 1 кластера также характеризуются достоверно большим содержанием ди-нуклеотида AU, чем таковые 2 и 3 кластеров (табл. 2).
Окружение стартового кодона
Согласно текущему мнению, решающую роль в инициации элонгации играет правильное и эффективное распознавание стартового AUG кодона, которое зависит от его нуклеотидного окружения, присутствия так называемой консенсусной последовательности Козак [5]. Нуклеотиды, которые окружают стартовый кодон, и которые определяют его точное распознавание, локализованы в двух областях – 5'-НТО и 5'-лидере кодирующей области.
Консенсусная последовательность вокруг стартового AUG кодона была экспериментально определена при анализе генов A. thaliana [1]. Эти результаты показали, что последовательности благоприятные для эффективной трансляции у растений не полностью соответствуют последовательности Козак, характерной для позвоночных. Так, анализ показал, что нуклеотиды A/G в позиции –3, A/C в позиции –2 и C в позиции +5 были общими для всех исследованных видов. Следует отметить, что поскольку нуклеотид в позиции +5 проявляется во втором кодоне (кодон, следующий за стартовым кодоном), это может определять химические свойства второй аминокислоты, и, возможно, этот нуклеотид находится в стадии функциональных ограничений аминокислотных последовательностей [25].
Нуклеотидная композиция вокруг стартового кодона у транскриптов из разных кластеров оценена за счет in silico анализа нуклеотидов в позициях –3 и +4, а консенсусные последовательности окружения стартового кодона выведены с использованием программы Logo (рис. 6) [13]. Весьма примечательно, что для транскриптов всех кластеров оптимальное окружение стартового кодона, согласно последовательности Козак присуще только 30% транскриптов (Supplementary Table 2 ). При этом сигналы консенсусных последовательностей окружения стартового кодона для каждого кластера транскриптов были следующими: AANAUGNNG, AAСAUGNNN и AANAUGNCN для кластера 1, 2 и 3 соответственно. Таким образом, в нашем исследовании отмечено отсутствие строгой корреляции между оптимальным окружением стартового кодона и загрузкой транскриптов рибосомами. Следует отметить, что недавние исследования с использованием баз данных по всем последовательностям генома из различных эукариотических видов показали, что предпочтительные нуклеотиды вокруг AUG кодона весьма разнообразны у разных видов организмов, включая и растения [25, 26]. Совокупность имеющихся литературных данных и собственные результаты этого исследования могут свидетельствовать о том, что роль контекста вокруг стартового кодона в эффективности трансляции у растений, еще до конца не прояснена. Так, все еще не полностью ясно насколько критическим является данный регуляторный контекст для эффективности трансляции в целом [25, 26].
Рис. 6.
Консенсусные последовательности вокруг стартового AUG кодона для транскриптов из разных кластеров: с высокой (а), средней (b) и низкой (c) трансляционной эффективностью. На оси ординат (bits) – высота каждой буквы в столбце обозначает относительную частоту, с которой нуклеотид встречается в этой позиции в анализируемой выборке 5'-НТО (относительные значения, которые рассчитаны программным обеспечением WebLogo [13]).
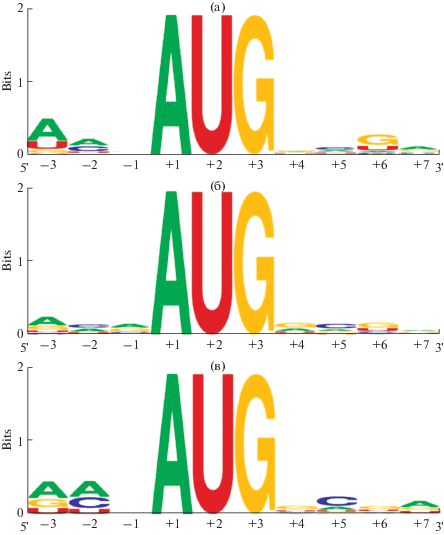
Таким образом, в ходе исследования нами выявлены новые нуклеотидные контексты в 5'-области мРНК растений A. thaliana, которые потенциально могут быть важными для эффективности трансляции мРНК растений на исследуемом этапе онтогенеза. Безусловно, функциональная значимость выявленных мотивов и нуклеотидного смещения, наблюдаемого вокруг стартового кодона у транскриптов с различным трансляционным статусом, требует экспериментального подтверждения.
Работа выполнена при финансовой поддержке Российского фонда фундаментальных исследований (Проект № 17-04-00783).
Авторы заявляют, что у них нет конфликта интересов. Статья не содержит каких-либо исследований с участием людей и животных в качестве объектов исследований.
Список литературы
Kawaguchi R. mRNA sequence features that contribute to translational regulation in Arabidopsis // Nucleic A-cids Res. 2005. V. 33. P. 955. https://doi.org/10.1093/nar/gki240
Sablok G., Powell J.J., Kazan K. Emerging roles and landscape of translating mRNAs in plants // Front. Plant Sci. 2017. V. 8. https://doi.org/10.3389/fpls.2017.01443
Goldenkova-Pavlova I., Pavlenko O., Mustafaev O., Deyneko I., Kabardaeva K., Tyurin A. Computational and experimental tools to monitor the changes in translation efficiency of plant mRNA on a genome-wide scale: Advantages, limitations, and solutions // Int. J. Mol. Sci. 2018. V. 20. P. 33. https://doi.org/10.3390/ijms20010033
Kabardaeva K.V., Tyurin A.A., Pavlenko O.S., Gra O.A., Deyneko I. V., Kouchoro F., Mustafaev O.N., Goldenkova-Pavlova I.V. Fine tuning of translation: A complex Web of mechanisms and its relevance to plant functional genomics and biotechnology // Russ. J. Plant Physiol. 2019. V. 66. P. 835.
Kozak M. How do eucaryotic ribosomes select initiation regions in messenger RNA? // Cell. 1978. V. 15. P. 1109. https://doi.org/10.1016/0092-8674(78)90039-9
Mustroph A., Zanetti M.E., Jang C.J.H., Holtan H.E., Repetti P.P., Galbraith D.W., Bailey-Serres J. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis // Proc. Natl. Acad. Sci. 2009. V. 106. P. 18843. https://doi.org/10.1073/pnas.0906131106
Bai B., Peviani A., van der Horst S., Gamm M., Snel B., Bentsink L., Hanson J. Extensive translational regulation during seed germination revealed by polysomal profiling // New Phytol. 2017. V. 214. P. 233. https://doi.org/10.1111/nph.14355
Ingolia N.T., Ghaemmaghami S., Newman J.R.S., Weissman J.S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling // Science. 2009. V. 324. P. 218. https://doi.org/10.1126/science.1168978
Baerenfaller K., Grossmann J., Grobei M.A., Hull R., Hirsch-Hoffmann M., Yalovsky S., Zimmermann P., Grossniklaus U., Gruissem W., Baginsky S. Genome-scale proteomics reveals Arabidopsis thaliana gene models and proteome dynamics // Science. 2008. V. 320. P. 938. https://doi.org/10.1126/science.1157956
Baerenfaller K., Massonnet C., Walsh S., Baginsky S., Bühlmann P., Hennig L., Hirsch-Hoffmann M., Howell K.A., Kahlau S., Radziejwoski A., Russenberger D., Rutishauser D., Small I., Stekhoven D., Sulpice R. et al. Systems-based analysis of Arabidopsis leaf growth reveals adaptation to water deficit // Mol Syst Biol. 2012. V. 8. P. 606. https://doi.org/10.1038/msb.2012.39
Qi W., Schlapbach R., Rehrauer H. RNA-Seq data analysis: From raw data quality control to differential expression analysis // Meth. Mol. Biol. 2017. P. 295. https://doi.org/10.1007/978-1-4939-7286-9_23
Larsson O., Sonenberg N., Nadon R. Identification of differential translation in genome wide studies // Proc. Natl. Acad. Sci. 2010. V. 107. P. 21487. https://doi.org/10.1073/pnas.1006821107
Crooks G.E. WebLogo: A sequence logo generator // Genome Res. 2004. V. 14. P. 1188. https://doi.org/10.1101/gr.849004
Czechowski T., Stitt M., Altmann T., Udvardi M.K., Scheible W.-R. Genome-wide Identification and testing of superior reference genes for transcript normalization in Arabidopsis // Plant Physiol. 2005. V. 139. P. 5. https://doi.org/10.1104/pp.105.063743
Zhang X., Rosen B.D., Tang H., Krishnakumar V., Town C.D. Polyribosomal RNA-Seq reveals the decreased complexity and diversity of the Arabidopsis translatome // PLoS One. 2015. V. 10: e0117699. https://doi.org/10.1371/journal.pone.0117699
Hsu P.Y., CalvielloL., Wu H.L., Li F.W., Rothfels C.J., Ohler U., Benfey P.N. Super-resolution ribosome profiling reveals unannotated translation events in Arabidopsis // Proc. Natl. Acad. Sci. 2016. V. 113: E7126. https://doi.org/10.1073/pnas.1614788113
Kim Y., Lee G., Jeon E., Sohn E.J., Lee Y., Kang H., Lee D.W., Kim D.H., Hwang I. The immediate upstream region of the 5'-UTR from the AUG start codon has a pronounced effect on the translational efficiency in Arabidopsis thaliana // Nucleic Acids Res. 2014. V. 42. P. 485. https://doi.org/10.1093/nar/gkt864
Kanoria S., Burma P.K. A 28 nt long synthetic 5′UTR (synJ) as an enhancer of transgene expression in dicotyledonous plants // BMC Biotechnol. 2012. V. 12. P. 85. https://doi.org/10.1186/1472-6750-12-85
Тюрин А.А., Кабардаева К.В., Гра О.А., Мустафаев О.Н., Садовская Н.С., Павленко О.С., Голденкова-Павлова И.В. Эффективность экспрессии гетерологичного гена в растениях зависит от нуклеотидного состава 5'-области мРНК // Физиология растений. 2016. Т. 63. С. 546.
Kozak M. Point mutations define a sequence flanking the AUG initiator codon that modulates translation by eukaryotic ribosomes // Cell. 1986. V. 44. P. 283. https://doi.org/10.1016/0092-8674(86)90762-2
Tzeng T.-Y., Kong L.-R., Chen C.-H., Shaw C.-C., Yang C.-H. Overexpression of the lily p70s6k gene in Arabidopsis affects elongation of flower organs and indicates TOR-dependent regulation of AP3, PI and SUP translation // Plant Cell Physiol. 2009. V. 50. P. 1695. https://doi.org/10.1093/pcp/pcp114
Ortega J.L., Wilson O.L., Sengupta-Gopalan C. The 5′ untranslated region of the soybean cytosolic glutamine synthetase β1 gene contains prokaryotic translation initiation signals and acts as a translational enhancer in plants // Mol. Genet. Genomics. 2012. V. 287. P. 881. https://doi.org/10.1007/s00438-012-0724-6
Liu M.-J., Wu S., Chen H.-M., Wu S.-H. Widespread translational control contributes to the regulation of Arabidopsis photomorphogenesis // Mol. Syst. Biol. 2012. V. 8. P. 566. https://doi.org/10.1038/msb.2011.97
Agarwal P., Garg V., Gautam T., Pillai B., Kanoria S., Burma P.K. A study on the influence of different promoter and 5′UTR (URM) cassettes from Arabidopsis thaliana on the expression level of the reporter gene β glucuronidase in tobacco and cotton // Transgenic Res. 2014. V. 23. P. 351.
Nakagawa S., Niimura Y., Gojobori T., Tanaka H., Miura K.-I. Diversity of preferred nucleotide sequences around the translation initiation codon in eukaryote genomes // Nucleic Acids Res. 2007. V. 36. P. 861. https://doi.org/10.1093/nar/gkm1102
Gupta P., Rangan L., Ramesh T.V., Gupta M. Comparative analysis of contextual bias around the translation initiation sites in plant genomes // J. Theor. Biol. 2016. V. 404. P. 303. https://doi.org/10.1016/j.jtbi.2016.06.015
Дополнительные материалы отсутствуют.
Инструменты
Физиология растений


