Физиология растений, 2020, T. 67, № 4, стр. 357-368
Предсказание характеристик потенциальных сайтов связывания миРНК в мРНК генов транскрипционных факторов TCP растений
А. К. Рахметуллина a, А. Ю. Пыркова a, А. В. Гончарова a, А. Т. Иващенко a, *
a Казахский Национальный университет им. аль-Фараби,
факультет биологии и биотехнологии
Алматы, Казахстан
* E-mail: a_ivashchenko@mail.ru
Поступила в редакцию 07.08.2019
После доработки 20.01.2020
Принята к публикации 20.01.2020
Аннотация
Экспрессия генов семейства транскрипционных факторов ТСР зависит от миРНК, действие которых на трансляцию их мРНК слабо изучено. Анализ взаимодействия миРНК с мРНК проведен с помощью программы MirTarget, определяющей количественные характеристики связывания всей нуклеотидной последовательности миРНК с мРНК. При анализе связывания 125 миРНК c мРНК 28 генов семейства ТСР Triticum aestivum обнаружены восемь генов-мишеней для miR319-3p, miR444a-3p, miR5086-5p, miR9666a-3p, miR9780-3p. Сайты связывания миРНК в мРНК генов семейства ТСР T. aestivum располагались только в CDS. Из 738 миРНК с мРНК 22 генов семейства ТСР Oryza sativa только 12 генов связывались с miR1437b-5p, miR1846a-5p, miR1848-5p, miR1858a-5p, miR1858b-5p, miR1861d-5p, miR1861h-5p, miR2102-3p, miR2102-5p, miR2919, miR2925-5p, miR319a-3 miR408-3p, miR5075-3p, miR5819-5p. Сайты связывания миРНК в мРНК генов семейства ТСР O. sativa располагались в 5'UTR и CDS. Из 46 генов семейства TCP Zea mays только десять генов были мишенями для miR164g-3p, miR164h-5p, miR166a-5p, miR168a-5p, miR171d-5p, miR399d-3p и miR408a-3p из 325 миРНК Z. mays. С мРНК генов GRMZM2G031905_P01, GRMZM2G055024_P01, GRMZM2G062711_P01 и GRMZM2G170232_P01 связывались zma-miR166a-5p. С мРНК генов AC213524.3_FGP003, AC233950.1_FGP002, GRMZM2G034638_P01 связывались по две миРНК. мРНК генов GRMZM2G035944_P01, AC190734.2_FGP003 и AC205574.3_FGP006 связывали по одной миРНК. Сайты связывания миРНК располагались 5'UTR и CDS мРНК генов семейства ТСР Z. mays. Из 27 генов семейства TCP Arabidopsis thaliana десять были мишенями для miR319-3р, miR4228-5p, miR4228-3p, miR5021-5p, miR5658-5p и miR8181-5p из 429 миРНК A. thaliana. мРНК генов AT1G53230 и AT2G31070 имели сайты связывания для miR319c-3p, miR5021-5p и miR5658-5p, а мРНК гена AT3G15030 имела сайты связывания для miR319a-3p и miR4228-3p. По две миРНК связывались с мРНК генов AT1G69690, AT3G02150 и AT3G47620. По одной миРНК связывалось с мРНК генов AT1G30210, AT4G18390 и AT5G08330. Сайты связывания миРНК располагались в 5'UTR, CDS и 3'UTR мРНК генов семейства ТСР A. thaliana. В мРНК шести групп генов семейства TCP установлены сайты связывания с miR319-3p, miR444a-3p, mir5021-5p, miR5658-5p, miR2102-5p, которые кодируют олигопептиды QRGPLQS, STSETS, SSSSSS, HHHHHH, GGGGGG, AAAAAA, консервативные в группах белков семейства ТСР различных видов растений. Предсказаны количественные характеристики связывания миРНК с мРНК транскрипционных факторов растений семейства ТСР, участвующих в регуляции роста и развития растений.
ВВЕДЕНИЕ
Транскрипционные факторы (ТФ) семейства TCP участвуют во многих процессах развития и роста растений [1]. Согласно базе данных факторов транскрипции растений, семейство TCP включает 4187 генов различных видов растений. Гены семейства TCP участвуют в апикальном доминировании [2], контроле двусторонней цветовой симметрии [2, 3], регуляции клеточного цикла [4], способствуют развитию листьев путем клеточного деления, роста и дифференциации [2]. Эти процессы регулируются многими миРНК, подавляющими экспрессию генов-мишеней на этапе трансляции. После открытия миРНК у растений [5] были проведены исследования биогенеза и участия миРНК во многих физиологических процессах [6–10]. Было показано участие miR159 в регуляции транскрипционных факторов семейства MYВ, а miR319 на TCP [11, 12]. Показано участие miR156, miR159, miR160, miR164, miR172 и miR319 в развитии цветков путем регуляции транскрипционных факторов SPL, MYB, ARF, NAC, AP2 и TCP, соответственно [13]. Обнаружено участие miR156, miR164, miR167, miR319, miR399, miR444, miR1507, miR2673 в регуляции метаболизма ананаса, кукурузы и люпина [14–16]. Показано, что miR160, miR166, miR156, miR164 и miR394 участвуют в индукции эмбриогенеза каллуса, дифференцировки растений, ауксиновых ответов и тотипотентности [17]. Функции генов-мишеней miR159, miR167, miR396, miR166, miR168, miR156, miR165, miR162 и miR394 включают передачу сигнала гормонов женьшеня, регуляцию транскрипции, макромолекулярный метаболизм и передачу сигналов ауксина [18]. miR156, miR160, miR166, miR167, miR168, miR172, miR319, miR390, miR399, miR408 и miR2275 участвуют в регуляции деления клеток и развития путем действия на различные транскрипционные факторы кукурузы [19]. С помощью биоинформатического анализа были обнаружены, pbr-miR156, pbr-miR164, pbr-miR399 и pbr-miR482, участвующие в защите груши от вирусов, а pbr-miR160, pbr-miR168, pbr-miR171 и pbr-miR319 посредством регуляции сигнальных путей с участием ауксина [20]. Установлена значительная разнонаправленная регуляция экспрессии miR164, miR171, miR396, miR399, miR395, miR408, miR528, miR529, miR820, miR1432, miR1846, miR1861, miR2102 и miR2907 в ответ на действие мышьяка на растения риса [21]. В большинстве цитируемых выше работ, изучалось действие миРНК на экспрессию генов и физиологические процессы без установления конкретных связей миРНК с их генами-мишенями. Влияние миРНК на экспрессию генов ТФ TCP, играющих важную роль в росте и развитии растений, мало исследовано. В настоящей работе, используя биоинформатические подходы, изучены количественные характеристики взаимодействия миРНК с мРНК генов ТФ TCP, для установления ассоциации миРНК и генов-мишеней способных регулировать физиологические процессы в растениях.
МАТЕРИАЛЫ И МЕТОДЫ
Нуклеотидные последовательности генов семейства TCP Triticum aestivum, Oryza sativa, Zea mays, Arabidopsis thaliana и других видов растений были взяты из базы ТФ растений v4.0 (http:// planttfdb.cbi.pku.edu.cn; http://www.ncbi.nlm.nih.gov). Нуклеотидные последовательности миРНК были взяты из miRBase v.22 (http://www.mirbase.org/). Вариабельность аминокислотных последовательностей оценивали программой WebLogo (https:// weblogo.berkeley.edu/logo.cgi). Сайты связывания миРНК в мРНК генов были предсказаны с помощью программы MirTarget [22]. Эта программа определяет следующие особенности связывания миРНК с мРНК: а) начало сайтов связывания миРНК с мРНК; б) расположение сайтов в 5'-нетранслируемом участке (5'UTR), в белок-кодирующей части (CDS) и в 3'-нетранслируемом участке (3'UTR) мРНК; в) свободную энергию взаимодействия миРНК и мРНК (∆G, кДж/моль); г) схемы взаимодействия нуклеотидов между миРНК и мРНК. По всей длине сайта связывания в мРНК допускается образование только одного однонуклеотидного пузыря “bulge”. Расчетные характеристики взаимодействия миРНК с мРНК приведены для отношения их концентраций 1 : 1. При изменении отношения концентраций доля комплексов миРНК с мРНК будет существенно изменяться согласно кинетическим уравнениям ингибитор-мишень. Для каждого сайта рассчитывали отношение ΔG/ΔGm (%), где ΔGm равно свободной энергии связывания миРНК с полностью комплементарной нуклеотидной последовательностью. Внутримолекулярные взаимодействия в мРНК с участием сайта связывания миРНК всегда меньше, чем взаимодействия между миРНК и мРНК в сайтах связывания. Это нами многократно проверялось с помощью программ мРНК-фолдинг. Мы делали вставку нуклеотидной последовательности миРНК в разные участки мРНК, и миРНК всегда взаимодействовала с предсказанным сайтом связывания. То есть, итоговая свободная энергия взаимодействия миРНК с мРНК способствовала связыванию миРНК.
Позиция сайтов связывания указана от первого нуклеотида мРНК. Учитываются связи не только между аденином (А) и урацилом (U), гуанином (G) и цитозином (C), но и взаимодействия между A и C, G и U посредством одной водородной связи. Расстояния между A и C (1.04 нанометра), G и U (1.02 нм) были аналогичны и между G и C, A и U и равны 1.03 нм [23]. Число водородных связей во взаимодействиях G–C, A–U, G–U и A–C было равным 3, 2, 1 и 1 соответственно. MirTarget по сравнению с программами для нахождения сайтов связывания миРНК с мРНК генов растений [24] отличается тем, что: 1) учитывает взаимодействие миРНК с мРНК по всей последовательности миРНК; 2) учитывает неканонические пары G–U и A–C; 3) рассчитывает свободную энергию взаимодействия миРНК с мРНК.
Список сокращенных названий видов растений, используемых в данной работе: Aegilops tauschii – Ata, Aethionema arabicum – Aar, Amborella trichopoda – Atr, Ananas comosus – Aco, Arabidopsis halleri – Aha, Arabidopsis lyrata – Aly, Arabidopsis thaliana – Ath, Arabis alpina – Aal, Azadirachta indica – Ain, Boechera stricta – Bst, Brachypodium distachyon – Bdi, Brachypodium stacei – Bsa, Brassica napus – Bna, Brassica oleracea – Bol, Brassica rapa – Bra, Camelina sativa – Csa, Capsella grandiflora – Cgr, Capsella rubella – Cru, Carica papaya – Cpa, Citrus clementina – Ccl, Citrus sinensis – Csi, Dichanthelium oligosanthes – Dol, Elaeis guineensis – Egu, Eucalyptus grandis – Egr, Eutrema salsugineum – Esa, Glycine max – Gma, Gossypium arboreum – Gar, Gossypium hirsutum – Ghi, Gossypium raimondii – Gra, Hordeum vulgare – Hvu, Leersia perrieri – Lpe, Musa acuminata – Mac, Oropetium thomaeum – Oth, Oryza barthii – Oba, Oryza brachyantha – Obr, Oryza glaberrima – Ogl, Oryza glumaepatula – Ogu, Oryza longistaminata – Olo, Oryza meridionalis – Ome, Oryza nivara – Oni, Oryza punctata – Opu, Oryza rufipogon – Oru, Oryza sativa subsp. indica – Osai, Oryza sativa subsp. japonica – Osa j, Panicum hallii – Pha, Panicum virgatum – Pvi, Phalaenopsis equestris – Peq, Phoenix dactylifera – Pda, Phyllostachys heterocycla – Phe, Populus trichocarpa – Ptr, Raphanus raphanistrum – Rra, Raphanus sativus – Rsa, Setaria italica – Sit, Setaria viridis – Svi, Sisymbrium irio – Sir, Solanum lycopersicum – Sly, Sorghum bicolor – Sbi, Spirodela polyrhiza – Spo, Tarenaya hassleriana – Tha, Thellungiella parvula – Tpa, Theobroma cacao – Tca, Triticum aestivum – Tae, Zea mays – Zma, Zostera marina – Zmr, Zoysia matrella – Zmt, Zoysia japonica – Zja, Zoysia pacifica – Zpa.
РЕЗУЛЬТАТЫ
Характеристики потенциальных сайтов связывания миРНК с мРНК генов ТСР Triticum aestivum
Для выявления миРНК, мишенями которых являются гены семейства ТФ TCP, был проведен поиск сайтов связывания 125 миРНК генома пшеницы с мРНК 28 генов семейства ТСР T. aestivum. Обнаружено восемь генов-мишеней для miR319-3p, miR444a-3p, miR5086-5p, miR9666a-3p, miR9780-3p (табл. 1). Два сайта связывания в мРНК гена Traes_2AL_EA60A06AC.1 имела miR444a-3p, расстояние между которыми составляет 223 н. Такое же расстояние между двумя сайтами связывания имела miR444a-3p в мРНК гена Traes_2BL_36A3AB3A2.1 и один сайт связывания выявлен для miR9780-3p. Две или более миРНК, которые связываются с мРНК одного гена, мы называли альтернативными миРНК. Гены Traes_2AL_EA60A06AC.1 и Traes_2BL_36A3AB3A2.1 были альтернативными генами-мишенями для miR444a-3p. Для miR5086-5p альтернативными генами-мишенями являются Traes_4DS_59A46B69A.1 и Traes_4AL_75D069945.1 (табл. 1). Сайты связывания миРНК в мРНК генов семейства ТСР T. aestivum располагались только в CDS. Эффективность действия миРНК определяется свободной энергией взаимодействия с мРНК и соотношением концентрации миРНК и мРНК. Две и более миРНК или одна миРНК, имеющая несколько сайтов связывания, могут оказывать повышенное влияние на экспрессию генов-мишеней.
Таблица 1.
Характеристики потенциальных сайтов связывания миРНК в CDS мРНК генов ТСР Triticum aestivum
| Ген | миРНК | Начало сайта, н. | ΔG, кДж/моль | ΔG/ΔGm, % | Длина, н. |
|---|---|---|---|---|---|
| TRAES3BF014900010CFD_t1 | miR319-3p | 1106 | –98 | 85 | 21 |
| Traes_2AL_EA60A06AC.1 | miR444a-3p | 839 | –98 | 85 | 21 |
| Traes_2AL_EA60A06AC.1 | miR444a-3p | 1062 | –102 | 89 | 21 |
| Traes_2BL_36A3AB3A2.1 | miR444a-3p | 866 | –98 | 85 | 21 |
| Traes_2BL_36A3AB3A2.1 | miR444a-3p | 1089 | –102 | 89 | 21 |
| Traes_4DS_59A46B69A.1 | miR5086-5p | 132 | –98 | 87 | 21 |
| Traes_4AL_75D069945.1 | miR5086-5p | 132 | –98 | 87 | 21 |
| Traes_2AL_B05A4E316.1 | miR9666a-3p | 17 | –104 | 86 | 22 |
| Traes_2BL_4B6057E06.1 | miR9666a-3p | 161 | –104 | 86 | 22 |
| Traes_2BL_36A3AB3A2.1 | miR9780-3p | 991 | –113 | 88 | 21 |
Мишенями для миРНК являются не только гены семейства TCP T. aestivum, но и гены семейства TCP других организмов. tae-miR319-3p, имеющая сходные нуклеотидные последовательности в A. thaliana, O. sativa и Z. mays, связывалась в мРНК генов TRAES3BF014900010CFD_t1 T. aestivum, гена LOC_Os01g11550.1 O. sativa, генов AC205574.3_FGP006 и GRMZM2G015037_P01 Z. mays, генов AT3G15030 и AT1G53230 A. thaliana с близкими характеристиками. Учет образования неканонических пар A–C и G–U увеличивает свободную энергию взаимодействия миРНК с мРНК. Нуклеотидные последовательности сайтов связывания tae-miR319-3p в этих мРНК гомологичны и кодируют олигопептид QRGPLQS (рис. 1а). Сайты связывания tae-miR319-3p кодируют консервативный олигопептид QRGPLQS в белках факторов транскрипции TCP у 54 видов растений (Aar, Aco, Ain, Aly, Ata, Ath, Atr, Bdi, Bna, Bol, Bra, Bsa, Bst, Ccl, Cgr, Csa, Dol, Egr, Egu, Esa, Gar, Ghi, Gma, Gra, Hvu, Lpe, Mac, Oba, Ogl, Ogu, Ome, Oni, Opu, Oru, Osai, Osaj, Peq, Pha, Ptr, Pvi, Rra, Sbi, Sir, Sit, Sly, Spo, Svi, Tae, Tca, Tha, Tpa, Zja, Zma, Zmr). Нуклеотидные последовательности сайтов связывания кодирующих олигопептид QRGPLQS были идентичны (рис. 1а′). Вариабельность аминокислот, примыкающих к олигопептиду QRGPLQS, и вариабельность нуклеотидов, фланкирующих сайт связывания, свидетельствует, что только связь tae-miR319-3p с генами-мишенями сохранялась в течение многих миллионов лет дивергенции изученных видов растений.
Рис. 1.
Вариабельность аминокислот олигопептидов, кодируемых сайтами связывания миРНК c мРНК, и вариабельность нуклеотидов сайтов связывания, кодирующих белки TCP разных растений. (а) – Олигопептид QRGPLQS, кодируемый сайтами связывания tae-miR319-3p в мРНК 56 генов у 54 видов растений; (а′) – олигонуклеотид CAGAGGGGACCCCUUCAGUCC сайтов связывания, кодирующий олигопептид QRGPLQS. (б) – Олигопептид STSETS, кодируемый сайтами связывания tae-miR444a-3p в мРНК 29 генов у 28 видов растений; (б′) – олигонуклеотид AGCACGUCGGAGACCAGC сайтов связывания, кодирующий олигопептид STSETS. (в) – Олигопептид SSSSSS, кодируемый сайтами связывания ath-mir5021-5p в мРНК 30 генов у 17 видов растений; (в′) – Weblogo для набора нуклеотидов, кодирующих олигопептид SSSSSS. (г) – Олигопептид HHHHHH, кодируемый сайтами связывания ath-miR5658-5p в мРНК 23 генов у 19 видов растений; (г′) – Weblogo для набора нуклеотидов, кодирующих олигопептид HHHHHH. (д) – Олигопептид GGGGGG, кодируемый сайтами связывания osa-miR2102-5p в мРНК 8 генов у 6 видов растений; (д′) – олигонуклеотид GGCGGCGGCGGCGGCGGC сайтов связывания, кодирующий олигопептид GGGGGG. (е) – Олигопептид AAAAAA, кодируемый сайтами связывания osa-miR2102-5p в мРНК 10 генов у 7 видов растений; (е′) – Weblogo для набора нуклеотидов, кодирующих олигопептид AAAAAA. Вариабельность аминокислотных последовательностей оценивали программой WebLogo (https://weblogo.berkeley.edu/logo.cgi).
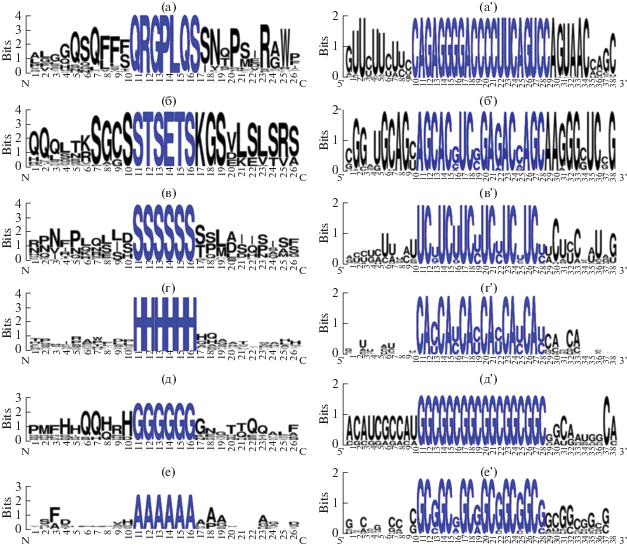
Нуклеотидная последовательность miR444a-3p известна только для T. aestivum и O. sativa. Эта миРНК имела сайты связывания в CDS мРНК двух генов T. aestivum и одного гена Z. mays. Характеристики сайтов связывания tae-miR444a-3p с мРНК генов Traes_2AL_EA60A06AC.1, Traes_ 2BL_36A3AB3A2.1, GRMZM2G020805_P01 семейства TCP сходны и сайты связывания кодируют гексапептид STSETS. Потенциальные сайты связывания tae-miR444a-3p выявлены в мРНК 29 генов у 28 видов растений (Bdi, Bsa, Dol, Hvu, Lpe, Mac, Oba, Ogl, Ogu, Ome, Oni, Opu, Oru, Osai, Osaj, Oth, Pda, Peq, Pha, Phe, Sbi, Sit, Spo, Svi, Tae, Zja, Zma, Zmt). Гексапептид STSETS, кодируемый сайтами связывания tae-miR444a-3p, входит в состав консервативного олигопептида SSTSETSKGS белков ТСР (рис. 1б). Примыкающие к этому олигопептиду аминокислоты вариабельны. Нуклеотидные последовательности сайтов связывания, кодирующих гексапептид STSETS, состоят из синонимических кодонов, в которых изменяется только третий нуклеотид (рис. 1б′). Следовательно, взаимодействие между tae-miR444a-3p и мРНК генов ТФ изученных видах растений семейства TCP сохраняется в течение многих миллионов лет.
Характеристики потенциальных сайтов связывания миРНК в мРНК генов ТСР Oryza sativa
Проведен поиск возможных сайтов связывания 738 миРНК с мРНК 22 генов семейства TCP O. sativa. Только 12 генов были мишенями для miR1437b-5p, miR1846a-5p, miR1848-5p, miR1858a-5p, miR1858b-5p, miR1861d-5p, miR1861h-5p, miR2102-3p, miR2102-5p, miR2919, miR2925-5p, miR319a-3 miR408-3p, miR5075-3p, miR5819-5p (табл. 2). У гена LOC_Os07g05720.1 обнаружены сайты связывания для miR2102-5p, miR2925-5p, miR319a-3p.2-3p, miR408-3p. Три миРНК имели сайты связывания с мРНК генов LOC_Os03g57190.1, LOC_Os05g43760.1. По две миРНК связывались с мРНК генов LOC_Os01g11550.1, LOC_Os01g55750.1, LOC_Os02g51280.1, LOC_Os03g49880.1, LOC_ Os06g12230.1, LOC_Os12g42190.1. мРНК других генов могли связываться только с одной миРНК. miR2102-5p имеет восемь альтернативных генов-мишеней, что свидетельствует о важной роли этой миРНК в регуляции нескольких физиологических функций растений. Сайты связывания миРНК в миРНК генов семейства TCP O. sativa расположены в 5'UTR и CDS. Сайты связывания osa-miR2102-5p в мРНК двух генов O. sativa и генов GRMZM2G003944_P01, ONIVA07G01330.1, ORGLA07G0028600.1, Zpz_sc01752.1.g00080.1.sm.mk кодировали консервативный гексапептид АААААА.
Таблица 2.
Характеристики потенциальных сайтов связывания миРНК в мРНК генов ТСР Oryza sativa
| Ген | миРНК | Начало сайта, н. | Участок мРНК | ΔG, кДж/моль | ΔG/ΔGm, % | Длина, н. |
|---|---|---|---|---|---|---|
| LOC_Os03g49880.1 | miR1437b-5p | 557 | 5'UTR | –102 | 89 | 21 |
| LOC_Os02g51280.1 | miR1846a-5p | 8 | 5'UTR | –110 | 90 | 21 |
| LOC_Os03g57190.1 | miR1848-5p | 246 | 5'UTR | –113 | 88 | 21 |
| LOC_Os02g51280.1 | miR1858a-5p | 1358 | CDS | –106 | 88 | 21 |
| LOC_Os05g43760.1 | miR1861d-5p | 706 | CDS | –108 | 91 | 22 |
| LOC_Os05g43760.1 | miR1861h-5p | 706 | CDS | –106 | 88 | 20 |
| LOC_Os08g43160.1 | miR2102-3p | 872 | CDS | –113 | 88 | 22 |
| LOC_Os01g11550.1 | miR2102-5p | 1670 | CDS | –110 | 91 | 20 |
| LOC_Os01g55750.1 | miR2102-5p | 923 | CDS | –104 | 86 | 20 |
| LOC_Os01g69980.1 | miR2102-5p | 1007 | CDS | –110 | 91 | 20 |
| LOC_Os05g43760.1 | miR2102-5p | 428 | CDS | –108 | 89 | 20 |
| LOC_Os06g12230.1 | miR2102-5p | 644 | 5'UTR | –108 | 89 | 20 |
| LOC_Os07g05720.1 | miR2102-5p | 1357 | CDS | –110 | 91 | 20 |
| LOC107277536 | miR2102-5p | 997 | CDS | –110 | 91 | 20 |
| LOC_Os03g49880.1 | miR2919 | 827 | CDS | –96 | 88 | 19 |
| LOC_Os07g05720.1 | miR2925-5p | 912 | CDS | –100 | 89 | 19 |
| LOC_Os03g57190.1 | miR319a-3p.2-3p | 1214 | CDS | –98 | 88 | 20 |
| LOC_Os07g05720.1 | miR319a-3p.2-3p | 1441 | CDS | –98 | 88 | 20 |
| LOC_Os07g05720.1 | miR408-3p | 904 | CDS | –106 | 89 | 21 |
| LOC_Os01g55750.1 | miR5075-3p | 965 | CDS | –108 | 88 | 21 |
| LOC_Os06g12230.1 | miR5075-3p | 642 | 5'UTR | –108 | 88 | 21 |
| LOC_Os12g42190.1 | miR5075-3p | 120 | CDS | –110 | 90 | 21 |
| LOC_Os01g11550.1 | miR5819-5p | 1163 | CDS | –110 | 90 | 21 |
| LOC_Os03g57190.1 | miR5819-5p | 721 | CDS | –110 | 90 | 21 |
Характеристики потенциальных сайтов связывания миРНК в мРНК генов ТСР Zea mays
Анализ связывания 325 миРНК с мРНК 46 генов семейства ТСР Z. mays предсказал только десять генов-мишеней для miR164g-3p, miR164h-5p, miR166a,с,k-5p, miR168a-5p, miR171d-5p, miR399d-3p, miR408a-3p-5p (табл. 3). С мРНК генов GRMZM2G031905_P01, GRMZM2G055024_P01, GRMZM2G062711_P01 и GRMZM2G170232_P01 связывались zma-miR166a-5p и zma-miR166с-5. С мРНК генов AC213524.3_FGP003, AC233950.1_ FGP002, GRMZM2G034638_P01 связывались по две миРНК. мРНК генов GRMZM2G035944_P01, AC190734.2_FGP003 и AC205574.3_FGP006 связывали по одной миРНК. Сайты связывания миРНК располагались 5'UTR и CDS мРНК генов семейства ТСР Z. mays (табл. 3).
Таблица 3.
Характеристики потенциальных сайтов связывания миРНК с мРНК генов мишеней ТФ семейства TCP Zea mays
| Ген | миРНК | Начало сайта, н. | Участок мРНК | ΔG, кДж/моль | ΔG/ΔGm, % | Длина, н. |
|---|---|---|---|---|---|---|
| AC205574.3_FGP006 | miR164g-3p | 157 | CDS | –102 | 86 | 21 |
| AC233950.1_FGP002 | miR164h-5p | 87 | 5'UTR | –102 | 87 | 21 |
| GRMZM2G170232_P01 | miR166a-5p | 158 | 5'UTR | –106 | 91 | 21 |
| GRMZM2G031905_P01 | miR166a-5p | 123 | CDS | –104 | 89 | 21 |
| GRMZM2G062711_P01 | miR166a-5p | 112 | CDS | –106 | 91 | 21 |
| GRMZM2G055024_P01 | miR166a-5p | 270 | CDS | –104 | 89 | 21 |
| GRMZM2G170232_P01 | miR166c-5p | 158 | 5'UTR | –102 | 89 | 21 |
| GRMZM2G055024_P01 | miR166c-5p | 270 | CDS | –100 | 87 | 21 |
| GRMZM2G031905_P01 | miR166c-5p | 123 | CDS | –100 | 87 | 21 |
| GRMZM2G062711_P01 | miR166c-5p | 112 | CDS | –102 | 89 | 21 |
| GRMZM2G170232_P01 | miR166k-5p | 158 | 5'UTR | –104 | 89 | 21 |
| GRMZM2G062711_P01 | miR166k-5p | 112 | CDS | –104 | 89 | 21 |
| GRMZM2G034638_P01 | miR168a-5p | 460 | CDS | –100 | 85 | 21 |
| AC213524.3_FGP003 | miR168a-5p | 477 | CDS | –100 | 85 | 21 |
| GRMZM2G035944_P01 | miR171d-5p | 328 | 5'UTR | –104 | 91 | 21 |
| AC190734.2_FGP003 | miR399d-3p | 355 | CDS | –100 | 85 | 21 |
| AC233950.1_FGP002 | miR399d-3p | 764 | CDS | –100 | 85 | 21 |
| GRMZM2G034638_P01 | miR408a-3p | 364 | CDS | –102 | 86 | 21 |
| AC213524.3_FGP003 | miR408a-3p | 381 | CDS | –102 | 86 | 21 |
Характеристики потенциальных сайтов связывания миРНК в мРНК генов ТСР Arabidopsis thaliana
Анализ взаимодействия 429 миРНК с мРНК 27 генов семейства ТСР A. thaliana показал, что только десять генов были мишенями для miR319-3р, miR4228-5p, miR4228-3p, miR5021-5p, miR5658-5p и miR8181-5p (табл. 4). мРНК генов AT1G53230 и AT2G31070 имели сайты связывания для miR319c-3p, miR5021-5p и miR5658-5p, а мРНК гена AT3G15030 имела сайты связывания для miR319a-3p и miR4228-3p. По две миРНК связывались с мРНК генов AT1G69690, AT3G02150 и AT3G47620. По одной миРНК связывалось с мРНК генов AT1G30210, AT4G18390 и AT5G08330. Сайты связывания миРНК располагались в 5'UTR, CDS и 3'UTR мРНК генов семейства ТСР A. thaliana.
Таблица 4.
Характеристики потенциальных сайтов связывания миРНК с мРНК генов мишеней ТФ семейства TCP Arabidopsis thaliana
| Ген | миРНК | Начало сайта, н. | Участок мРНК | ΔG, кДж/моль | ΔG/ΔGm, % | Длина, н. |
|---|---|---|---|---|---|---|
| AT3G15030.1 | miR319a-3p | 1619 | CDS | –100 | 87 | 21 |
| AT1G53230.1 | miR319c-3p | 1184 | CDS | –98 | 87 | 21 |
| AT2G31070.1 | miR319c-3p | 1379 | CDS | –98 | 87 | 21 |
| AT3G15030.1 | miR319c-3p | 1620 | CDS | –100 | 89 | 21 |
| AT3G15030.1 | miR4228-3p | 1370 | CDS | –100 | 87 | 21 |
| AT3G47620.1 | miR4228-5p | 1732 | 3'UTR | –98 | 87 | 21 |
| AT2G31070.1 | miR5021-5p | 52 | 5'UTR | –89 | 91 | 20 |
| AT1G53230.1 | miR5021-5p | 91 | 5'UTR | –91 | 93 | 20 |
| AT5G08330.1 | miR5021-5p | 359 | 5'UTR | –89 | 91 | 20 |
| AT3G02150.1 | miR5021-5p | 697 | CDS | –89 | 91 | 20 |
| AT1G69690.1 | miR5021-5p | 945 | CDS | –91 | 93 | 20 |
| AT1G30210.2 | miR5021-5p | 1114 | CDS | –93 | 96 | 20 |
| AT1G30210.1 | miR5021-5p | 1241 | CDS | –93 | 96 | 20 |
| AT1G69690.1 | miR5658-5p | 415 | 5'UTR | –100 | 98 | 21 |
| AT3G02150.1 | miR5658-5p | 972 | CDS | –91 | 90 | 21 |
| AT4G18390.1 | miR5658-5p | 1214 | CDS | –98 | 96 | 21 |
| AT1G53230.1 | miR5658-5p | 1236 | CDS | –96 | 94 | 21 |
| AT2G31070.1 | miR5658-5p | 1287 | CDS | –91 | 90 | 21 |
| AT3G47620.1 | miR8181-5p | 295 | CDS | –100 | 85 | 20 |
Гексасерин и гексагистидин обнаружены в белках TCP, кодируемых соответственно сайтами связывания ath-miR-5021-5p, ath-miR-5658-5p. В таблице 5 приведены характеристики взаимодействия ath-miR-5021-5p с генами мРНК семейства TCP, которые могут связываться со значением ΔG/ΔGm 91–96%. Схемы взаимодействия ath-miR5658-5p с генами мРНК ТФ TCP показаны на рисунке 2. Приведенные схемы взаимодействия ath-miR5658-5p с мРНК нескольких генов отчетливо показывают эффективность нашей программы MirTarget. Все нуклеотиды ath-miR5658-5p образуют водородные связи с мРНК генов AT1G53230.1 и GSBRNA2T00009950001. В последнем случае образуется семь связей А–С, которые сохраняют двухспиральную структуру комплекса миРНК и мРНК с сохранением стекинг взаимодействий всех их нукдеотидов. мРНК других генов связывают ath-miR5658-5p образуя подряд 19–20 водородных связей, что тоже сохраняет стекинг взаимодействия в этих участках миРНК и мРНК.
Таблица 5.
Характеристики потенциальных сайтов связывания ath-miR5021-5p в CDS мРНК генов ТСР растений
| Ген | Начало сайта, н. | ΔG, кДж/моль | ΔG/ΔGm, % |
|---|---|---|---|
| 473241 | 808 | –93 | 96 |
| 476101 | 511 | –91 | 93 |
| 476101 | 67 | –85 | 87 |
| 676708942 | 877 | –89 | 91 |
| 676720890 | 67 | –85 | 87 |
| AA54G00421 | 961 | –85 | 87 |
| Araha.15691s0008.1.p | 508 | –91 | 93 |
| Araha.15691s0008.1.p | 67 | –85 | 87 |
| Araha.6346s0001.1.p | 829 | –93 | 96 |
| AT1G30210.1 | 1241 | –93 | 96 |
| AT1G69690.1 | 945 | –91 | 93 |
| AT3G02150.2 | 1416 | –85 | 87 |
| AT3G02150.2 | 697 | –89 | 91 |
| Bostr.10273s0396.1.p | 505 | –87 | 89 |
| Bostr.10273s0396.1.p | 64 | –85 | 87 |
| Cagra.13056s0010.1.p | 508 | –91 | 93 |
| Cagra.13056s0010.1.p | 64 | –85 | 87 |
| Carubv10011113m | 832 | –93 | 96 |
| Carubv10020637m | 508 | –91 | 93 |
| Csa03g033390.1 | 742 | –93 | 96 |
| Csa05g087530.1 | 307 | –91 | 93 |
| GSBRNA2T00025745001 | 769 | –87 | 89 |
| GSBRNA2T00025745001 | 775 | –91 | 93 |
| GSBRNA2T00112877001 | 58 | –85 | 87 |
| RrC2920_p3 | 55 | –85 | 87 |
| Rsa1.0_00361.1_g00006.1 | 787 | –87 | 89 |
| Rsa1.0_00361.1_g00006.1 | 793 | –91 | 93 |
| Rsa1.0_01246.1_g00003.1 | 505 | –91 | 93 |
| Rsa1.0_01246.1_g00003.1 | 61 | –85 | 87 |
| Thhalv10008175m | 811 | –87 | 89 |
| Thhalv10008175m | 817 | –91 | 93 |
| Thhalv10018834m | 55 | –85 | 87 |
| Tp1g26030 | 832 | –93 | 96 |
| Tp5g24820 | 67 | –85 | 87 |
| XP_009104987.1 | 61 | –85 | 87 |
| XP_009109609.1 | 751 | –87 | 89 |
| XP_009109609.1 | 757 | –91 | 93 |
| XP_010551531.1 | 508 | –91 | 93 |
| XP_013616237.1 | 67 | –85 | 87 |
| XP_013622999.1 | 751 | 87 | 89 |
| XP_013622999.1 | 757 | –91 | 93 |
Рис. 2.
Cхемы взаимодействия ath-miR5658-5p с генами мРНК ТФ TCP. Ген; начало сайта связывания (н.); участок мРНК; свободная энергия, ΔG, (кДж/моль); ΔG/ΔGm, %; длина миРНК (н.); верхняя и нижняя нуклеотидные последовательности мРНК и ath-miR5658-5p соответственно. Жирным шрифтом обозначены нуклеотиды неканонических пар U–G и A–C.

Cайты связывания миРНК в мРНК генов ТСР многих видов растений кодируют консервативные олигопептиды, что свидетельствует о раннем возникновении функциональной связи между миРНК и генами-мишенями, которая сохраняется на протяжении многих миллионов лет. Обнаружены сайты связывания ath-miR5021-5p в мРНК трех генов A. thaliana и в мРНК 27 генов 17 видов растений, кодирующих олигопептид SSSSSS (Aar, Aha, Aly, Ath, Bna, Bol, Bra, Bst, Cgr, Cru, Csa, Esa, Rra, Rsa, Sir, Tha, Tpa). В этих белках олигопептид SSSSSS является высоко консервативным, что отражено на рисунке 1в. Белок AA54G00421 A. thaliana содержит декасерин, двенадцать белков содержат октасерин и пять белков содержат гептасерин. Сайты связывания, кодирующие гексасерин, состоят из синонимических кодонов серина, в которых изменяется третий нуклеотид (рис. 1в′). Это не изменяет состав олигопептида, но влияет на свободную энергию взаимодействия ath-miR5021-5p с мРНК генов ТСР. В данном случае преобладающей причиной консервативности аминокислот является функциональное значение гексасерина по сравнению с величиной свободной энергии взаимодействия ath-miR5021-5p с мРНК генов ТФ ТСР.
Характеристики сайтов связывания ath-miR-5658-5p с мРНК генов TCP показаны в таблице 6. Гены-мишени для ath-miR-5658-5p содержали эффективные сайты связывания для этой миРНК, поскольку ΔG/ΔGm варьировала от 90 до 98%. Сайты связывания ath-miR5658-5p кодируют гексапептид HHHHHH в 18 белках. Из приведенных данных следует, что гексапептид HHHHHH является консервативным в TCP-белках, кодируемых сайтами связывания ath-miR5658-5p в мРНК 23 генов у 19 видов растений: Aly, Ath, Bdi, Bna, Bol, Bst, Cgr, Cru, Csa, Gar, Ghi, Gma, Gra, Osaj, Rra, Rsa, Tca, Tha, Tpa (рис. 1г). Сайты связывания ath-miR-5658-5p в мРНК генов GSBRNA2T00009950001, Bostr.0124s0115.1.p и Tp1g39800 кодируют октагистидин, а мРНК гена Thecc1EG031706T1 декагистидин.
Таблица 6.
Характеристики потенциальных сайтов связывания ath-miR-5658-5p в мРНК CDS генов ТСР растений
| Ген | Начало сайта, н. | ΔG, кДж/моль | ΔG/ΔGm, % |
|---|---|---|---|
| 474447 | 960 | –96 | 94 |
| AT1G53230.1 | 1236 | –96 | 94 |
| AT3G02150.2 | 11 183 | –91 | 90 |
| Bostr.0124s0115.1.p | 978 | –91 | 90 |
| Cagra.1472s0007.1.p | 970 | –96 | 94 |
| Carubv10009348m | 966 | –96 | 94 |
| Cotton_A_20110_BGI-A2_v1.0 | 738 | –89 | 87 |
| Csa14g063740.1 | 957 | –96 | 94 |
| Gh_D01G0419 | 738 | –89 | 87 |
| Glyma.08G097900.1.p | 1078 | –100 | 98 |
| Gorai.002G064500.1 | 738 | –89 | 87 |
| GSBRNA2T00009950001 | 610 | –87 | 85 |
| LOC_Os05g43760.1 | 930 | –87 | 85 |
| LOC_Os07g05720.1 | 1476 | –89 | 87 |
| RrC11789_p1 | 817 | –96 | 94 |
| Rsa1.0_02089.1_g00003.1 | 895 | –93 | 92 |
| Thecc1EG031706t1 | 1584 | –89 | 87 |
| Tp1g39800 | 475 | –96 | 94 |
| XP_010519240.1 | 1071 | –87 | 85 |
| XP_013630036.1 | 729 | –87 | 85 |
Нуклеотидные последовательности сайтов связывания ath-miR-5658-5p, кодирующих гексагистидин, состоят из синонимических кодонов с вариацией третьего нуклеотида (рис. 1г′). Причиной разной консервативности нуклеотидного состава сайтов связывания ath-miR-5658-5p и соответствующих аминокислот, вероятно, является большая значимость роли гексагистидина в белках ТСР и по сравнению со свободной энергией связывания этой миРНК.
Характеристики потенциальных сайтов связывания osa-miR2102-5p в мРНК CDS генов семейства TCP растений
Сайты связывания некоторых миРНК имеют гомологичные нуклеотидные последовательности, которые могут быть прочитаны в разных открытых рамках считывания. Например, нуклеотидная последовательность сайтов связывания osa-miR2102-5p GCGGCGGCGGCGGCGGCGGCG в первой рамке считывания будет кодировать олигопептид AAAAAAA, во второй рамке считывания RRRRRR, а в третьей рамке считывания сайтов связывания osa-miR2102-5p будет кодироваться олигопептид GGGGGG. На рисунке 1д показаны содержащие полиглицин области белков шести растений: Oba, Ogl, Ome, Oru, Osaj, Tae. Нуклеотидные последовательности сайтов связывания osa-miR2102-5p консервативны (рис. 1д′).
Для группы 10 генов семейства TCP видов Ogl, Oni, Osaj, Spo, Tae, Zma и Zpa, сайты связывания osa-miR2102-5p, кодировали гексааланин. Из рисунка 1е видно, что области белков, смежные с гексапептидом AAAAAA, являются вариабельными. Это указывает на консерватизм сайтов связывания osa-miR2102-5p в мРНК десяти генов ТСР. Сайты связывания osa-miR2102-5p состоят из синонимических кодонов, что обусловливает консервативность только первых двух нуклеотидов (рис. 1е′). По-видимому, функциональная роль гексапептида AAAAAA преобладает над зависимостью экспрессии гена от osa-miR2102-5p.
В некоторых белках TCP T. aestivum, O. sativa, Z. mays, A. thaliana были обнаружены полиглутамин, политреонин, полиаспарагин, полипролин, полиаспаргиновая кислота (табл. 7). Нуклеотидные последовательности, кодирующие эти олигопептиды не являются сайтами известных миРНК T. aestivum, O. sativa, Z. mays, A. thaliana. Эти олигопептиды могут кодироваться сайтами связывания пока не обнаруженных миРНК. В генах животных и человека эти олигопептиды кодируются сайтами связывания миРНК [25–27]. Основные свойства взаимодействий миРНК с генами мРНК у растений и животных похожи, что указывает на сходство регуляции экспрессии генов посредством миРНК у представителей разных царств эукариотических организмов.
Таблица 7.
Олигопептиды, которые могут кодироваться сайтами связывания миРНК Triticum aestivum, Oryza sativa, Zea mays, Arabidopsis thaliana
| Ген | Сокращенное название вида | Олигопептиды |
|---|---|---|
| AT2G31070.1 | Ath | QQQQQQ, TTTTTT |
| AT3G15030.1 | Ath | QQQQQQQQ |
| AT3G47620.1 | Ath | QQQQQQQ |
| AT5G23280.1 | Ath | NNNNNNNNNNN, QQQQQQ |
| LOC_Os01g69980.1 | Osaj | QQQQQQ |
| LOC_Os02g51280.1 | Osaj | QQQQQQQQQQQQQQQQQ |
| LOC_Os03g57190.1 | Osaj | QQQQQQ |
| LOC_Os07g05720.1 | Osaj | QQQQQQ, QQQQQQQQ |
| LOC_Os08g43160.1 | Osaj | QQQQQQQ |
| LOC_Os09g24480.1 | Osaj | QQQQQQQ |
| LOC_Os12g07480.1 | Osaj | DDDDDD, NNNNNN |
| TRAES3BF146000020CFD_t1 | Tae | QQQQQQ, QQQQQQQ |
| Traes_5BL_2DDBAA0C2.1 | Tae | QQQQQQ, QQQQQQQ |
| Traes_5BL_B94C45A8F.1 | Tae | QQQQQQQ |
| Traes_6AL_DA27ABCA61.2 | Tae | QQQQQQQQ |
| Traes_6DL_136DE13FB.1 | Tae | QQQQQQQQ |
| GRMZM2G003944_P01 | Zma | QQQQQQ |
| GRMZM2G034638_P01 | Zma | QQQQQQ, QQQQQQ |
| GRMZM2G060319_P01 | Zma | TTTTTT |
| GRMZM2G077755_P01 | Zma | QQQQQQQ |
| GRMZM2G078077_P01 | Zma | QQQQQQQQ |
| GRMZM2G089361_P01 | Zma | PPPPPPP |
| GRMZM2G089638_P01 | Zma | QQQQQQ |
| GRMZM2G107031_P01 | Zma | DDDDDD |
| GRMZM2G142751_P01 | Zma | QQQQQQ |
| GRMZM2G148022_P02 | Zma | QQQQQQQ, QQQQQQQ |
ОБСУЖДЕНИЕ
Количество генов и миРНК, способных взаимодействовать друг с другом, отличаются для T. aestivum, O. sativa, Z. mays, A. thaliana. Полученные результаты показывают, что экспрессия генов семейства TCP может регулироваться путем связывания миРНК с их мРНК. Значительная часть ТФ может быть мишенью миРНК, о чем свидетельствуют предыдущие исследования [28, 29]. Сайты связывания миРНК в генах мРНК растений расположены в основном в CDS (табл. 1, 2, 5, 6). Часть генов семейства TCP имеет сайты связывания в 5'UTR и 3'UTR мРНК (табл. 3, 4).
Сайты белков, содержащих олигопептиды, кодируемые сайтами связывания миРНК, имеют следующие особенности. В некоторых белках TCP фланкирующие аминокислоты варьируют по сравнению с олигопептидом, кодируемым сайтами связывания миРНК. Для некоторых миРНК олигопептид, кодируемый сайтом связывания миРНК, является консервативным, вместе с фланкирующими аминокислотами (рис. 1). В транскрипционных факторах SPL арабидопсиса выявлен олигопептид который консервативен в нескольких белках и нуклеотидные последовательности сайтов связывания miR156j в мРНК соответствующих генов полностью комплементарны и тоже консервативны [29]. В ортологичных генах восьми видов растений олигопептид ALSLLS, кодируемый сайтами связывания miR156a, и нуклеотидные последовательности этих сайтов были высоко консервативны [29]. Нуклеотидные последовательности сайтов связывания miR396 и кодируемые ими олигопептиды в транскрипционных факторах GRF были консервативны по сравнению с фланкирующими их последовательностями нуклеотидов и аминокислот [28]. Особый интерес представляют сайты связывания миРНК, которые кодируют аминокислотные повторы (рис. 1). Это свойство растительных миРНК напоминает миРНК животных, сайты связывания которых тоже кодируют аминокислотные повторы [25, 26]. Основные свойства взаимодействий миРНК с генами мРНК у растений и животных очень похожи, что указывает на сходство регуляции экспрессии генов посредством миРНК у представителей разных царств эукариотических организмов [27]. miR319-3p, miR444a-3p, mir5021-5p, miR5658-5p имели сайты связывания в мРНК генов семейства ТФ TCP. Эти сайты связывания кодируют олигопептиды QRGPLQS, STSETS, SSSSSS, HHHHHH, соответственно. miR2102-5p имела сайты связывания, кодирующие олигопептиды GGGGGG и AAAAAA. Участки мРНК, длиной 18 н. и более нуклеотидов, являющиеся сайтами связывания миРНК, кодируют полисерин, полигистидин, полиглицин и полиаланин, которые могут служить фенотипическим признаком белка, свидетельствующим о зависимости его синтеза от соответствующих миРНК. Консервативность олигопептидов, кодируемых сайтами связывания соответствующих миРНК в генах различных видов растений, указывает на эволюционно раннее появление регуляции экспрессии генов с использованием миРНК (рис. 1). Благодаря программе MirTarget, предсказаны количественные характеристики взаимодействия миРНК с мРНК генов транскрипционных факторов семейства ТСР, которые существенно увеличивают возможность установления взаимодействия конкретных миРНК с мРНК генов соответствующих транскрипционных факторов.
В результате проведенных исследований можно сделать следующие выводы: 1) для миРНК предсказаны гены-мишени из семейства TCP T. aestivum, O. sativa, Z. mays и A. thaliana; 2) сайты связывания некоторых миРНК консервативны в мРНК генов-мишеней различных растений и кодируют консервативные олигопептиды; 3) некоторые миРНК могут иметь более одного сайта связывания в одной мРНК и более одного гена-мишени; 4) мРНК некоторых генов-мишеней могут иметь сайты связывания для двух или более разных миРНК; 5) tae-miR319-3p имеет консервативные сайты связывания в мРНК генов семейства TCP T. aestivum, O. sativa, Z. mays и A. thaliana; 6) сайты связывания миРНК растений могут располагаться в 5'UTR, CDS и 3'UTR; 7) выявлены миРНК, которые могут связываться с мРНК генов из разных семейств ТФ растений; 8) перечисленные свойства миРНК растений подобны свойствам миРНК животных.
Авторы заявляют об отсутствии конфликта интересов. Настоящая статья не содержит каких-либо исследований с участием людей и животных в качестве объектов исследований.
Список литературы
Li S. The Arabidopsis thaliana TCP transcription factors: a broadening horizon beyond development // Plant Signal. Behav. 2015. V. 10: e1044192. https://doi.org/10.1080/15592324.2015.1044192
Manassero N., Viola I., Welchen E., Gonzalez D. TCP transcription factors: architectures of plant form // Biomol. Concepts. 2013. V. 4. P. 111. https://doi.org/10.1515/bmc-2012-0051
Sengupta A., Hileman L. Novel traits, flower symmetry, and transcriptional autoregulation: new hypotheses from bioinformatic and experimental data // Front. Plant Sci. 2018. V. 9. P. 1. https://doi.org/10.3389/fpls.2018.01561
Mart M., Cubas P. TCP genes: a family snapshot ten years later // Trends Plant Sci. 2010. V. 15. P. 31. https://doi.org/10.1016/j.tplants.2009.11.003
Reinhart B., Weinstein E., Rhoades M., Bartel B., Bartel D. MicroRNAs in plants // Genes Dev. 2002. V. 16. P. 1616. https://doi.org/10.1101/gad.1004402
Rogers K., Chen X. Biogenesis, turnover, and mode of action of plant microRNAs // Plant Cell. 2013. V. 25. P. 2383. https://doi.org/10.1105/tpc.113.113159
Navarro L., Dunoyer P., Jay F., Arnold B., Dharmasiri N. A plant miRNA contributes to antibacterial resistance by repressing auxin signaling // Science. 2006. V. 312. P. 436. https://doi.org/10.1126/science.aae0382
Zhang L., Zheng Y., Jagadeeswaran G., Li Y., Gowdu K., Sunkar R. Identification and temporal expression analysis of conserved and novel microRNAs in Sorghum // Genomics. 2011. V. 98. P. 460. https://doi.org/10.1016/j.ygeno.2011.08.005
Phillips J.R., Dalmay T., Bartels D. The role of small RNAs in abiotic stress // FEBS Lett. 2007. V. 581. P. 3592. https://doi.org/10.1016/j.febslet.2007.04.007
Yan Y., Wang H., Hamera S., Chen X., Fang R. miR444a has multiple functions in the rice nitrate-signaling pathway // Plant J. 2014. V. 78. № 1. P. 44. https://doi.org/10.1111/tpj.12446
Palatnik J.F., Wollmann H., Schommer C., Schwab R., Boisbouvier J., Rodriguez R., Warthmann N., Allen E., Dezulian T., Huson D., Carrington J.C., Weigel D. Sequence and expression differences underlie functional specialization of Arabidopsis microRNAs miR159 and miR319 // Dev. Cell. 2007. V. 13. № 1. P. 115. https://doi.org/10.1016/j.devcel.2007.04.012
Tomotsugu K., Fumihiko S., Masaru O. Roles of miR319 and TCP transcription factors in leaf development // Plant Physiology. 2017. V. 175. P. 874. https://doi.org/10.1104/pp.17.00732
Li Z., An X., Zhu T., Yan T., Wu S., Tian Y., Li J., Wan X. Discovering and constructing ceRNA-miRNA-target gene regulatory networks during anther development in maize // Int. J. Mol. Sci. 2019. V. 20. № 14. P. 1. https://doi.org/10.3390/ijms20143480
Yusuf N.H., Ong W.D., Redwan R.M., Latip M.A., Kumar S.V. Discovery of precursor and mature microRNAs and their putative gene targets using high-throughput sequencing in pineapple (Ananas comosus var. comosus) // Gene. 2016. V. 571. № 1. P. 71. https://doi.org/10.1016/j.gene.2015.06.050
Wu F.Y., Tang C.Y., Guo Y.M., Yang M.K., Yang R.W., Lu G.H., Yang Y.H. Comparison of miRNAs and their targets in seed development between two maize inbred lines by high-throughput sequencing and degradome analysis // PLoS One. 2016. V. 17. № 1. P. 1. https://doi.org/10.1371/journal.pone.0159810
De Boer K., Melser S., Sperschneider J., Kamphuis L.G., Garg G., Gao L.L., Frick K., Singh K.B. Identification and profiling of narrow-leafed lupin (Lupinus angustifolius) microRNAs during seed development // BMC Genomics. 2019. V. 20. № 1. P. 1. https://doi.org/10.1186/s12864-019-5521-8
López-Ruiz B.A., Juárez-González V.T., Sandoval-Zapotitla E., Dinkova T.D. Development-related miRNA expression and target regulation during staggered in vitro plant regeneration of tuxpeño VS-535 maize cultivar // Int. J. Mol. Sci. V. 20. № 9. https://doi.org/10.3390/ijms20092079
Wang Y., Peng M., Wang W., Chen Y., He Z., Cao J., Lin Z., Yang Z., Gong M., Yin Y. Verification of miRNAs in ginseng decoction by high-throughput sequencing and quantitative real-time PCR // Heliyon. 2019. V. 5. № 4. P. 1. https://doi.org/10.1016/j.heliyon.2019.e01418
Aydinoglu F., Lucas S.J. Identification and expression profiles of putative leaf growth related microRNAs in maize (Zea mays L.) hybrid ADA313 // Gene. 2019. V. 690. P. 57. https://doi.org/10.1016/j.gene.2018.12.042
Zhang Q., Zhang Y., Wang S., Hao L., Wang S., Xu C., Jiang F., Li T. Characterization of genome-wide microRNAs and their roles in development and biotic stress in pear // Planta. 2019. V. 249. № 3. P. 693. https://doi.org/10.1007/s00425-018-3027-2
Sharma D., Tiwari M., Lakhwani D., Tripathi R.D., Trivedi P.K. Differential expression of microRNAs by arsenate and arsenite stress in natural accessions of rice // Metallomics. 2015. V. 7. № 1. P. 174. https://doi.org/10.1039/c4mt00264d
Ivashchenko A., Pyrkova A., Niyazova R., Alybayeva A., Baskakov K. Prediction of miRNA binding sites in mR-NA // Bioinformation. 2016. V. 12. P. 237. https://doi.org/10.6026/97320630012237
Garg A., Heinemann U. A novel form of RNA double helix based on G·U and C·A+ wobble base pairing // RNA. 2018. V. 24. № 2. P. 209. https://doi.org/10.1261/rna.064048.117
Dai X., Zhuang Z., Zhao P. Computational analysis of miRNA targets in plants: current status and challenges // Briefings in Bioinformatics. 2011. V. 12. P. 115. https://doi.org/10.1093/bib/bbq065
Niyazova R., Berillo O., Atambayeva Sh., Pyrkova A., A-lybayeva A., Ivashchenko A. miR-1322 binding sites in paralogous and orthologous genes // BioMed Research Int. 2015. V. 1. P. 1. https://doi.org/10.1155/2015/962637
Kondybayeva A.M., Akimniyazova A.N., Kamenova S.U., Ivashchenko A.T. The characteristics of miRNA binding sites in mRNA of ZFHX3 gene and its orthologs // Vavilov journal of Genetics and Breeding. 2018. V. 22. P. 438. https://doi.org/10.18699/VJ18.380
Yurikova O.Yu., Aisina D.E., Niyazova R.E., Atambayeva Sh.A., Labeit S., A. Ivashchenko A.T. The interaction of miRNA-5p and miRNA-3p with the mRNAs of orthologous genes // Mol. Biol. 2019. V. 53. №. 4. P. 612. https://doi.org/10.1134/S0026893319040174
Bari A., Sagaidak I., Pinskii I., Orazova S., Ivashchenko A. Binding of miR396 to mRNA of genes encoding growth-regulating transcription factors of plants // Russian Journal of Plant Physiology. 2014. V. 61. P. 807. https://doi.org/10.1134/S1021443714050033
Bari A., Orazova A., Ivashchenko A. miR156- and miR171-binding sites in the protein-coding sequences of several plant genes // BioMed Res. Int. 2013. V. 10. P. 1. https://doi.org/10.1155/2013/307145
Дополнительные материалы отсутствуют.
Инструменты
Физиология растений


