Генетика, 2019, T. 55, № 11, стр. 1312-1326
Идентификация генетических локусов, ассоциированных с устойчивостью яровой мягкой пшеницы (T. aestivum L.) к мучнистой росе
И. Н. Леонова *
Федеральный исследовательский центр Институт цитологии и генетики Сибирского отделения
Российской академии наук
630090 Новосибирск, Россия
* E-mail: leonova@bionet.nsc.ru
Поступила в редакцию 05.04.2019
После доработки 13.05.2019
Принята к публикации 29.05.2019
Аннотация
Мучнистая роса, вызываемая грибным патогеном Blumeria graminis f. sp. tritici (Bgt), является одним из вредоносных заболеваний мягкой пшеницы T. aestivum L. Выращивание сортов, характеризующихся генетической устойчивостью, – один из эффективных и экологически важных способов защиты от патогена. В работе было изучено генетическое разнообразие российских сортов яровой мягкой пшеницы по локусам устойчивости к мучнистой росе. Результаты фитопатологического скрининга 97 сортов показали, что не более 10% изученных образцов характеризуются низкой восприимчивостью к популяции Bgt, специфичной для региона Западной Сибири. С помощью ассоциативного картирования, выполненного на основании данных генотипирования маркерами SNP и фитопатологической оценки в течение трех полевых сезонов, были идентифицированы восемь локусов в хромосомах 1AL, 1DS, 2BL, 5AS, 5DS, 6AL, 6DL и 7AL. Высокий вклад в фенотипическое проявление признака установлен для генетических факторов, локализованных в хромосомах 5AS, 6AL и 6DL. В длинном плече хромосомы 6D картирован ген Pm6Ai=2, интродуцированный от пырея Thinopyrum intermedium, который обеспечивает эффективную защиту сортов от возбудителя мучнистой росы. На основании сравнительного анализа хромосомной локализации известных генов устойчивости и локусов, картированных в настоящей работе, было сделано предположение, что QTLs в хромосомах 1DS, 5AS, 6AL являются новыми, ранее неописанными локусами резистентности. Полученные результаты могут быть использованы для разработки молекулярных маркеров и отбора источников генетических локусов, определяющих устойчивость мягкой пшеницы к Bgt.
Мягкая пшеница (T. aestivum L.) является наиболее важной продовольственной культурой во всем мире и одним из основных источников белка и углеводов. Урожайность мягкой пшеницы в значительной степени зависит от устойчивости сортов к болезням. Мучнистая роса, вызываемая грибным патогеном Blumeria graminis (DC.) Speer f. sp. tritici Marchal (Bgt), входит в группу наиболее вредоносных болезней и встречается во всех регионах выращивания, включая Россию, Европу, Северную и Южную Америку, Австралию и Китай [1, 2]. По сравнению с другими грибными заболеваниями мучнистая роса появляется значительно раньше, влияет на фотосинтетическую активность листовой пластины, уменьшает размер зерна, что приводит к существенному снижению продуктивности.
До недавнего времени созданию сортов с генетической устойчивостью к мучнистой росе уделялось меньше внимания по сравнению с устойчивостью к бурой и стеблевой ржавчине. Однако в последние годы наблюдается значительное повышение вредоносности патогена, симптомы заболевания которым регистрируются в регионах с менее увлажненным климатом [3, 4]. Поэтому проблема поиска источников генетических факторов, определяющих устойчивость к Bgt, становится все более актуальной.
В настоящее время в геном мягкой пшеницы перенесено более 80 генов ювенильной и возрастной устойчивости к мучнистой росе (символ Pm), более половины из которых имеют чужеродное происхождение [5]. Для генов Pm1, Pm2, Pm3, Pm4, Pm5, Pm24, локализованных в хромосомах 7AL, 5DS, 1AS, 2AL, 7BL и 1DS соответственно, выявлено от двух до шести аллелей, имеющих различное происхождение и различный защитный эффект по отношению к Bgt [6]. Для некоторых генов, таких как Pm8, Pm38, Pm39, было показано, что они локализованы в группах сцепления, включающих локусы устойчивости к бурой, стеблевой и желтой ржавчинам, что обеспечивает комплексную устойчивость сортов к грибным патогенам [7–9]. Кроме генов с постоянными и временными символами были идентифицированы локусы количественных признаков (QTLs), для которых определена вероятность ассоциации с признаком и интервалы маркеров, включающие максимальное проявление признака [10].
Классические методы выявления генов Pm с использованием расоспецифических изолятов Bgt трудоемки и не всегда информативны, особенно при наличии в геноме нескольких генов или аллелей одного и того же гена. Использование диагностических молекулярных маркеров в сочетании с фитопатологическим тестированием – один из эффективных способов выявления локусов, определяющих устойчивость к патогену. Генетическое картирование проведено для большинства известных генов Pm, информация о хромосомной локализации генов и подобранных для их выявления маркерах представлена в ряде обзорных публикаций и на интернет-сайте, посвященном маркер-ориентированной селекции [5, 6, 11, 12, https://maswheat.ucdavis.edu/].
В настоящее время для изучения генетического разнообразия злаков по генам устойчивости к патогенам, изучения генетической архитектуры хозяйственно важных признаков, определения хромосомной локализации генов и QTLs применяется новый подход, основанный на полногеномном поиске ассоциаций (GWAS, genome-wide association study). Это стало возможным в результате создания платформ для высокопроизводительного генотипирования, включающих маркеры SNP [13, 14]. Для проведения GWAS могут быть использованы коллекции стародавних и современных сортов, наборы селекционных, интрогрессивных и рекомбинантных инбредных линий, которые отличаются гораздо большим генетическим разнообразием по сравнению с картирующими популяциями, полученными на основе двуродительских скрещиваний.
Для пшеницы большинство работ, в которых использован GWAS, сфокусировано на поиске новых локусов устойчивости к грибным заболеваниям. На данный момент при изучении образцов пшеницы различного географического происхождения выявлено большое число ассоциаций маркер–признак для устойчивости к бурой, стеблевой, желтой ржавчине, фузариозу колоса и септориозу [15–17]. Однако только ограниченное число работ проведено по ассоциативному картированию локусов устойчивости к Bgt [18, 19]. Информация по генам устойчивости к мучнистой росе в сортах пшеницы, рекомендованных для выращивания в Российской Федерации, практически отсутствует. Целью данной работы была оценка разнообразия сортов яровой мягкой пшеницы российской селекции по генетическим локусам, ассоциированным с устойчивостью к Bgt.
МАТЕРИАЛЫ И МЕТОДЫ
В работе использовали 97 сортов яровой мягкой пшеницы, созданных в различных селекционных центрах Российской Федерации и рекомендованных для выращивания в Поволжье, Уральском и Сибирском регионах России. Семенной материал получен из Всероссийского института генетических ресурсов растений им. Н.И. Вавилова (ВИР, г. Санкт-Петербург), поддерживался и размножался в Федеральном исследовательском центре ИЦиГ СО РАН. Список использованных образцов представлен в табл. 1, более детальную информацию можно найти в Интернет-ресурсах GRIS (Genetic Resources Information System for Wheat and Triticale, http://wheatpedigree.net) и в разделе биоресурсных коллекций ИЦиГ СО РАН (http://ckp.icgen.ru/plants/fond).
Таблица 1.
Устойчивость сортов и селекционных линий яровой мягкой пшеницы к мучнистой росе (2016–2018 гг., Новосибирская область)
| № п/п | Наименование сорта | Оригинатор | Год допуска | Устойчивость к Bgt (баллы по шкале иммунности) | Постулированные гены/QTL | ||
|---|---|---|---|---|---|---|---|
| 2016 г. | 2017 г. | 2018 г. | |||||
| 1 | Куйбышевская 2 | НИИСХ, г. Самара | 1988 | 9-8 | 7-6 | 7-6 | |
| 2 | Лютесценс 101 | 1994 | 2-1 | 2-1 | 2-1 | Pm6Ai=2, QPm.icg-5A, QPm.icg-6A | |
| 3 | Лютесценс 840 | 1987 | 7-6 | 7-6 | 7-6 | ||
| 4 | Тулайковская белозерная | 1995 | 5 | 5 | 7-6 | QPm.icg-2B, QPm.icg-7A | |
| 5 | Тулайковская степная | 1998 | 5 | 5 | 5 | QPm.icg-2B, QPm.icg-7A | |
| 6 | Тулайковская золотистая | 2006 | 2-1 | 2-1 | 2-1 | Pm6Ai=2, QPm.icg-5A, QPm.icg-6A | |
| 7 | Тулайковская 1 | 1994 | 4-3 | 4-3 | 2-1 | QPm.icg-1D, QPm.icg-5D | |
| 8 | Тулайковская 10 | 2003 | 2-1 | 2-1 | 2-1 | Pm6Ai=2, QPm.icg-5A, QPm.icg-6A | |
| 9 | Кинельская 40 | 1975 | 7-6 | 7-6 | 7-6 | ||
| 10 | Кинельская 60 | 1998 | 2-1 | 4-3 | 4-3 | QPm.icg-5A, QPm.icg-1D, QPm.icg-6A | |
| 11 | Волгоуральская | 2001 | 5 | 5 | 5 | QPm.icg-1D, QPm.icg-7A | |
| 12 | Лютесценс 80 | Алтайский НИИ земледелия и селекции, г. Барнаул | – | 7-6 | 9-8 | 9-8 | |
| 13 | Лютесценс 85 | – | 4-3 | 5 | 5 | QPm.icg-1A, QPm.icg-7A | |
| 14 | Лютесценс 148 | – | 7-6 | 7-6 | 7-6 | ||
| 15 | Алтайский простор | 1997 | 7-6 | 7-6 | 9-8 | ||
| 16 | Алтайская 92 | 1997 | 7-6 | 7-6 | 7-6 | ||
| 17 | Алтайская 99 | 2005 | 7-6 | 7-6 | 7-6 | ||
| 18 | Алтайская 100 | 2005 | 7-6 | 9-8 | 9-8 | ||
| 19 | Алтайская 325 | 2004 | 7-6 | 7-6 | 9-8 | ||
| 20 | Алтайская 530 | 2007 | 7-6 | 7-6 | 9-8 | ||
| 21 | Эритроспермум 72 | – | 4-3 | 5 | 5 | QPm.icg-1D, QPm.icg-6A | |
| 22 | Сибирская 12 | СибНИИРС, г. Новосибирск | 2006 | 5 | 7-6 | 7-6 | |
| 23 | Новосибирская 15 | 2003 | 7-6 | 7-6 | 7-6 | ||
| 24 | Новосибирская 22 | 1991 | 5 | 7-6 | 7-6 | ||
| 25 | Новосибирская 29 | 2003 | 7-6 | 7-6 | 7-6 | ||
| 26 | Новосибирская 67 | 1974 | 5 | 5 | 5 | QPm.icg-6A | |
| 27 | Новосибирская 81 | 1986 | 7-6 | 7-6 | 7-6 | ||
| 28 | Новосибирская 89 | 1993 | 5 | 7-6 | 7-6 | ||
| 29 | Новосибирская 91 | 2002 | 7-6 | 7-6 | 5 | ||
| 30 | Лютесценс 25 | 1990 | 7-6 | 9-8 | 7-6 | ||
| 31 | Обская 2 | 2014 | 5 | 5 | 5 | QPm.icg-1D | |
| 32 | Обская 14 | 1990 | 5 | 7-6 | 7-6 | QPm.icg-7A | |
| 33 | Кантегирская 89 | 1993 | 7-6 | 9-8 | 7-6 | ||
| 34 | Александрина | 2007 | 7-6 | 9-8 | 7-6 | ||
| 35 | Удача | 2006 | 7-6 | 7-6 | 9-8 | ||
| 36 | Полюшко | 2008 | 7-6 | 9-8 | 9-8 | ||
| 37 | Баганская 93 | 1999 | 5 | 5 | 5 | QPm.icg-1A, QPm.icg-7A | |
| 38 | Сирена | НИИСХ, г. Красноярск | 1992 | 7-6 | 7-6 | 9-8 | |
| 39 | Краса 2 | 2002 | 9-8 | 9-8 | 9-8 | ||
| 40 | Красноярская 90 | 1991 | 5 | 7-6 | 7-6 | ||
| 41 | Веснянка 8 | 1990 | 7-6 | 9-8 | 7-6 | ||
| 42 | Альбидум 73 | 1963 | 7-6 | 9-8 | 7-6 | ||
| 43 | Рыбинская 127 | 1990 | 5 | 5 | 5 | QPm.icg-1D, QPm.icg-7A | |
| 44 | Казачка | 2001 | 7-6 | 7-6 | 7-6 | ||
| 45 | Ангарида | 2002 | 7-6 | 9-8 | 7-6 | ||
| 46 | Мана 2 | 2005 | 7-6 | 9-8 | 9-8 | ||
| 47 | Тулеевская | НИИСХ, г. Кемерово | 2002 | 4-3 | 4-3 | 4-3 | QPm.icg-5A, QPm.icg-7A |
| 48 | Изида | 2002 | 5 | 7-6 | 7-6 | ||
| 49 | Мария | 1999 | 7-6 | 7-6 | 7-6 | ||
| 50 | АН-34 | 2002 | 5 | 5 | 5 | QPm.icg-1A, QPm.icg-7A | |
| 51 | Мариинка | 2006 | 7-6 | 7-6 | 7-6 | ||
| 52 | Салимовка | 2001 | 9-8 | 9-8 | 7-6 | ||
| 53 | Кийская | 2001 | 5 | 7-6 | 7-6 | ||
| 54 | Ностальгия | 2001 | 5 | 7-6 | 7-6 | ||
| 55 | Алешина | 2003 | 7-6 | 7-6 | 7-6 | ||
| 56 | Дарница | 2008 | 7-6 | 9-8 | 9-8 | ||
| 57 | Серебрина | НИИСХ Северного Зауралья, г. Тюмень | 2000 | 7-6 | 7-6 | 7-6 | |
| 58 | Речка | 2000 | 7-6 | 7-6 | 9-8 | ||
| 59 | Латона | 2000 | 7-6 | 7-6 | 9-8 | ||
| 60 | Провинция | 2002 | 9-8 | 9-8 | 9-8 | ||
| 61 | Бэль | 2008 | 7-6 | 7-6 | 9-8 | ||
| 62 | Устя | 1999 | 7-6 | 5 | 9-8 | ||
| 63 | Чернява 13 | 2000 | 5 | 7-6 | 9-8 | ||
| 64 | Златозара | 1999 | 7-6 | 7-6 | 9-8 | ||
| 65 | Тюменская 99 | 2000 | 7-6 | 9-8 | 9-8 | ||
| 66 | Икар | 2001 | 7-6 | 7-6 | 9-8 | ||
| 67 | Скэнт 1 | 1998 | 4-3 | 5 | 5 | QPm.icg-1D, QPm.icg-7A | |
| 68 | Ильинская | 1997 | 7-6 | 7-6 | 9-8 | ||
| 69 | Туринская | 1995 | 7-6 | 7-6 | 9-8 | ||
| 70 | Сурента 1 | 2000 | 7-6 | 7-6 | 7-6 | ||
| 71 | Сурента 4 | 2002 | 5 | 7-6 | 9-8 | ||
| 72 | Сурента 5 | 2002 | 7-6 | 9-8 | 9-8 | ||
| 73 | Сурента 6 | 2003 | 7-6 | 9-8 | 9-8 | ||
| 74 | Сурента 7 | 2003 | 7-6 | 7-6 | 9-8 | ||
| 75 | Диас 2 | Сибирский НИИСХ, г. Омск | 1992 | 7-6 | 7-6 | 9-8 | |
| 76 | Катюша | 2008 | 5 | 5 | 5 | QPm.icg-6A | |
| 77 | Тарская 6 | 2002 | 7-6 | 5 | 7-6 | ||
| 78 | Соната | 2002 | 7-6 | 7-6 | 9-8 | ||
| 79 | Страда Сибири | 2002 | 7-6 | 9-8 | 9-8 | ||
| 80 | Отрада Сибири | 1998 | 7-6 | 7-6 | 9-8 | ||
| 81 | Терция | 1995 | 7-6 | 9-8 | 9-8 | ||
| 82 | Прииртышская 86 | 1986 | 9-8 | 9-8 | 9-8 | ||
| 83 | Росинка 2 | 1999 | 7-6 | 9-8 | 9-8 | ||
| 84 | Омская 20 | 1993 | 5 | 7-6 | 9-8 | ||
| 85 | Омская 23 | 1997 | 7-6 | 9-8 | 9-8 | ||
| 86 | Омская 24 | 1996 | 9-8 | 9-8 | 9-8 | ||
| 87 | Омская 26 | 1998 | 9-8 | 9-8 | 9-8 | ||
| 88 | Омская 28 | 1997 | 7-6 | 7-6 | 9-8 | ||
| 89 | Омская 29 | 1999 | 7-6 | 7-6 | 7-6 | ||
| 90 | Омская 32 | 2001 | 7-6 | 7-6 | 9-8 | ||
| 91 | Омская 33 | 2002 | 7-6 | 7-6 | 9-8 | ||
| 92 | Омская 34 | 2001 | 7-6 | 7-6 | 9-8 | ||
| 93 | Омская 36 | 2007 | 7-6 | 7-6 | 7-6 | ||
| 94 | Саратовская 29 | НИИСХ Юго-Востока, г. Саратов | 1957 | 7-6 | 7-6 | 7-6 | |
| 95 | Саратовская 42 | 1973 | 7-6 | 7-6 | 9-8 | ||
| 96 | Лютесценс 62 | 1924 | 7-6 | 7-6 | 9-8 | ||
| 97 | Тулун 15 | Тулунская государственная селекционная станция, г. Тулун | 1998 | 9-8 | 9-8 | 9-8 | |
Сорта выращивали на селекционном поле СибНИИРС-филиал ИЦиГ СО РАН (Новосибирская область) в течение трех полевых сезонов 2016–2018 гг. Образцы высевали рандомизированным методом в двух повторностях на делянках шириной 1 м, по 60 зерен в ряду. Восприимчивость к мучнистой росе оценивали на естественном инфекционном фоне в период от всходов до цветения, начиная с появления первых симптомов заболевания до полного развития болезни. Степень инфицирования определяли в баллах по шкале Саари и Прескотта [20]: 2-1 – устойчивый тип (слабая инфекция на нижних листьях); 4-3 – среднеустойчивый тип (растение поражено на одну треть, сильная инфекция на нижних листьях, умеренная – на вышерасположенных); 5 – средневосприимчивый тип (сильно поражены нижние листья; от умеренной до слабой инфекции до середины растения); 7-6 – восприимчивый тип (сильное поражение нижних и вышерасположенных листьев, инфекция доходит до флагового листа или частично его затрагивает); 9-8 – сильно восприимчивый тип (инфицирование флагового листа, иногда инфекция распространяется на колос).
Геномную ДНК выделяли из 5–7-дневных проростков как описано ранее [21]. Панель сортов была генотипирована с помощью Illumina Infinium 15K чип в компании TraitGenetics GmbH (Германия, www.traitgenetics.de). Перед проведением GWAS выполняли фильтрацию: маркеры с частотой аллелей меньше 5% и маркеры, которые не амплифицировали фрагменты у 20% и более образцов, в анализ не включали. После фильтрации число использованных маркеров составило 9285. Ассоциации маркер–признак (MTAs, marker-trait association) определяли с помощью программы TASSEL v. 5.2.24 [22], структура популяции (Q-матрикс) и генетическое родство (K-матрикс) были использованы в качестве ковариаты. Q-матрикс рассчитывали исходя из результатов генотипирования по 5950 SNP с помощью Байесовского алгоритма, реализованного в программе STRUCTURE 2.3.4 [23]. Вероятное число субкластеров подсчитано на основании Delta K (ΔK) статистики [24] с помощью web-программы Structure Harvester [25]. К-матрикс рассчитан в программе TASSEL v. 5.2.24. Для кластерного анализа использован метод ближайших соседей, реализованный в программе PAST v. 3.15 [26]. Для выявления достоверных ассоциаций использовали два критерия: критерий множественных сравнений Бонферрони c α = 0.1, что соответствовало величине p < 1.06 × 10–5, и FDR-контроль при p < 0.001. Предполагаемую генетическую локализацию локусов, ассоциированных с устойчивостью к мучнистой росе, определяли с помощью консенсусных карт хромосом гексаплоидной пшеницы, представленных в работе Ванга с соавт. [13].
РЕЗУЛЬТАТЫ
Оценка устойчивости образцов мягкой пшеницы к мучнистой росе
Погодные условия в регионе в 2016–2018 гг. благоприятствовали развитию инфицирования растений патогеном мучнистой росы, при этом инфекционный фон в 2016 г. был ниже, чем в остальные годы. Количество выпавших осадков в третьей декаде мая и июне 2016 и 2017 гг. было сравнимо со среднемноголетними показателями, условия 2018 г. отличались существенным переувлажнением. Гидротермический коэффициент по Селянинову в 2018 г. составил 3.1, что в 3 раза превышало среднемноголетние показатели для региона.
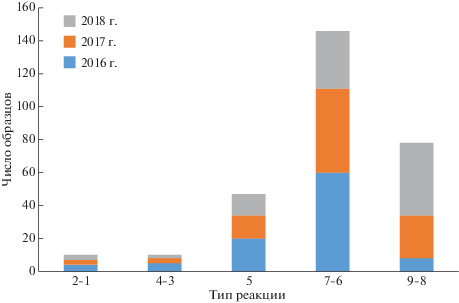
Скрининг сортов по степени инфицирования возбудителем мучнистой росы, проведенный в течение трех полевых сезонов, показал, что для большинства из них характерен восприимчивый тип реакции (балл 7-6 по шкале иммунности) (табл. 1). Из рис. 1 видно, что распределение сортов имеет отклонение в сторону восприимчивости. Фенотипическая оценка также указывает на то, что для сортов Тулайковская золотистая, Тулайковская 1, Тулайковская 10, Кинельская 60, Лютесценс 101 и Тулеевская был характерен высоко- и среднеустойчивый тип реакции во все сезоны вегетации. 11 сортов (Тулайковская степная, Волгоуральская, Лютесценс 85, Эритроспермум 72, Новосибирская 67, Обская 2, Баганская 93, Рыбинская 127, АН-34, Скэнт 1, Катюша) показали среднюю восприимчивость (балл 5). Поражение остальных сортов варьировало в зависимости от сезона вегетации, при этом следует отметить, что в 2018 г. увеличилось число образцов, у которых наблюдался высокочувствительный тип реакции (рис. 1).
Полногеномный поиск ассоциаций
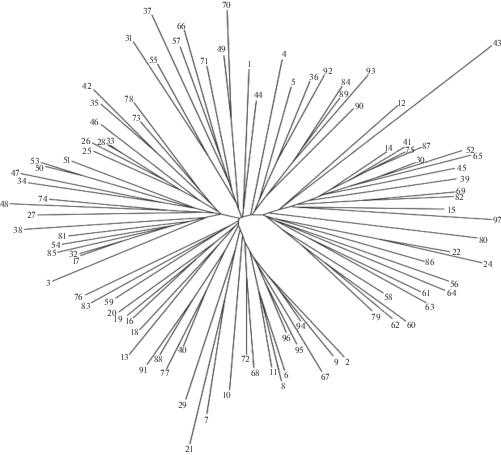
Для анализа структуры популяции было использовано 5950 маркеров SNP, представляющих все хромосомы мягкой пшеницы. На основании ΔK статистики число постулируемых субкластеров составило пять (рис. 2). Для уточнения генетических взаимосвязей был проведен кластерный анализ, для которого использовано 9285 маркеров SNP (рис. 3). Состав субкластеров и группировка сортов на дендрограмме генетического сходства свидетельствуют об отсутствии четкого разделения сортов по группам согласно их происхождению из различных селекционных институтов.
Рис. 2.
Популяционная структура сортов яровой мягкой пшеницы, рассчитанная на основании генотипирования с помощью 5950 маркеров SNP. На вертикальной оси обозначены коэффициенты принадлежности образца к субкластеру. Столбцы и цифры под ними соответствуют нумерации сортов пшеницы, представленной в табл. 1.
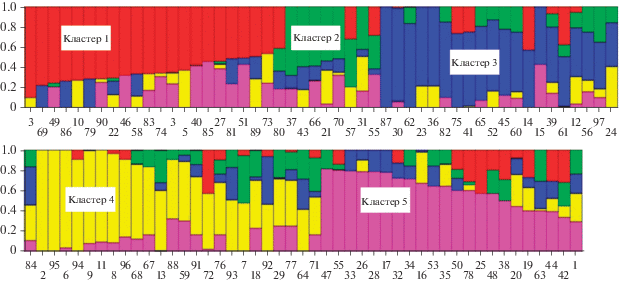
Рис. 3.
Дендрограмма генетического сходства сортов яровой мягкой пшеницы, построенная методом ближайших соседей на основании данных генотипирования маркерами SNP. Нумерация сортов соответствует нумерации, представленной в табл. 1.
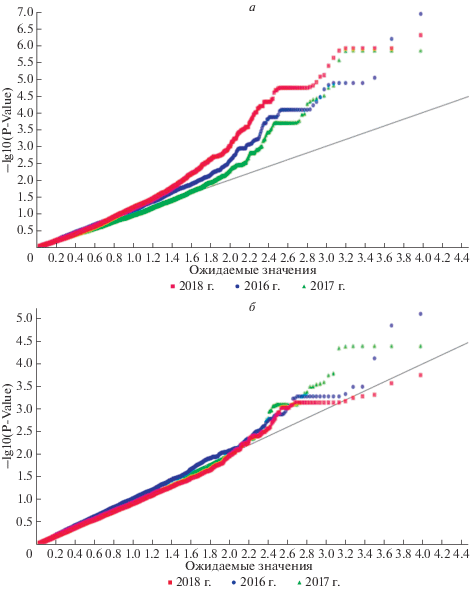
Оптимальная модель для проведения GWAS была выбрана на основании квантиль-квантиль (QQ) графика, построенного с использованием фенотипических оценок признака и данных генотипирования, который демонстрирует соответствие между наблюдаемыми и ожидаемыми величинами (рис. 4). Были проверены две модели: обобщенная линейная модель (GLM), где в качестве ковариаты использовались данные по структуре популяции (Q-матрикс), и смешанная линейная модель (MLM). Исходя из QQ-графика, модель MLM, учитывающая кроме Q-матрикса генетическое родство (К-матрикс), оказалась более оптимальной, чем GLM, и была использована для проведения GWAS.
Полногеномный поиск выявил 33 маркера SNP, ассоциированных с устойчивостью к Bgt, при этом для 25 маркеров установлены значимые ассоциации во все три года проведения фитопатологических оценок (табл. 2). Согласно консенсусным генетическим картам мягкой пшеницы [13] маркеры локализованы в хромосомах 1A, 1D, 2B, 5A, 5D, 6A, 6D и 7A. Наиболее высокие MTAs были обнаружены для десяти SNP короткого плеча хромосомы 5A. Маркеры находятся в районе 35.38–36.95 сМ, что предполагает их принадлежность к одному локусу (рис. 5). В хромосоме 6АS выявлено шесть информативных SNPs, картированных в диапазоне 0.5 сМ (табл. 2). Три значимых MTAs идентифицированы в хромосоме 6DL. Для BS00063175_51 показано, что маркер находится в районе 84.54 сМ. Для маркеров RAC875_c7178_404 и GENE-4008_395 данные по локализации на хромосомах консенсусных карт отсутствуют.
Таблица 2.
Маркеры SNP, ассоциированные с устойчивостью сортов мягкой пшеницы к Bgt, их хромосомная локализация и вклад в фенотипическое проявление признака
| Наименование маркера | Индекс SNP | Хромосома | Позиция на хромосоме* | Аллель** | p | R2 |
|---|---|---|---|---|---|---|
| tplb0041a22_935 | IWB74701 | 1AL | 71.09 | A/G | 1.10E-05 | 0.19 |
| Excalibur_c15098_59116, 18*** | IWB22559 | 1AL | 71.47 | T/C | 4.28E-05 | 0.05 |
| JD_c3737_285 | IWB37391 | 1DS | 67.72 | A/G | 1.28E-05 | 0.16 |
| BS00095225_51 | IWB11921 | 1DS | 67.72 | T/G | 1.90E-05 | 0.18 |
| IACX589 | IWB36051 | 1DS | 67.72 | G/A | 2.20E-05 | 0.18 |
| BS00012936_51 | IWB6579 | 1DS | 67.72 | T/C | 4.89E-05 | 0.16 |
| Kukri_c5113_108216, 18 | IWB46138 | 1DS | 71.47 | C/T | 4.85E-04 | 0.05 |
| RAC875_c3752_890 | IWB57293 | 2BL | 97.69 | A/G | 6.47E-05 | 0.16 |
| wsnp_Ex_c41558_48356814 | IWA3785 | 2BL | 103.34 | G/A | 3.62E-05 | 0.03 |
| BS00100185_51 | IWB12123 | 5AS | ̶ | T/C | 1.21E-07 | 0.16 |
| Excalibur_rep_c115510_314 | IWB30698 | 5AS | 35.38 | C/T | 2.42E-06 | 0.22 |
| BS00099700_51 | IWB12084 | 5AS | 36.58 | A/C | 5.15E-07 | 0.22 |
| Kukri_c8835_112 | IWB48151 | 5AS | 36.58 | T/C | 1.27E-06 | 0.22 |
| wsnp_CAP7_c2282_1107112 | IWA1062 | 5AS | 35.95 | T/C | 1.27E-06 | 0.22 |
| TA002772-0920 | IWB65602 | 5AS | 35.95 | G/A | 1.60E-05 | 0.16 |
| wsnp_Ex_rep_c69526_68472787 | IWA5615 | 5AS | 35.95 | T/G | 1.27E-06 | 0.18 |
| GENE-3321_201 | IWB33331 | 5AS | 36.73 | A/C | 1.27E-06 | 0.22 |
| BS00094095_51 | IWB11853 | 5AS | 36.87 | T/C | 1.27E-06 | 0.18 |
| BobWhite_rep_c50145_387 | IWB4848 | 5AS | 36.87 | G/T | 1.48E-06 | 0.22 |
| IAAV211517,18 | IWB34503 | 5DL | 67.49 | G/T | 4.21E-05 | 0.17 |
| Ra_c70331_77917,18 | IWB52508 | 5DL | 67.49 | C/T | 4.21E-05 | 0.17 |
| RAC875_rep_c70595_321 | IWB62708 | 5DL | 67.49 | A/G | 4.21E-05 | 0.17 |
| Kukri_rep_c68344_627 | IWB49782 | 6AS | 71.24 | C/T | 6.63E-07 | 0.20 |
| wsnp_CAP7_c1839_908011 | IWA1049 | 6AS | 71.24 | A/G | 4.13E-06 | 0.18 |
| Kukri_c19338_413 | IWB42115 | 6AS | 71.24 | G/A | 9.42E-06 | 0.17 |
| BS00021747_51 | IWB6752 | 6AS | 71.24 | T/G | 1.87E-05 | 0.18 |
| BS00023847_51 | IWB7511 | 6AS | 71.24 | G/A | 1.87E-05 | 0.18 |
| BS00021999_51 | IWB6851 | 6AS | 71.73 | G/A | 6.15E-05 | 0.13 |
| BS00063175_51 | IWB9015 | 6DL | 84.54 | G/A | 1.83E-06 | 0.26 |
| RAC875_c7178_40416, 18 | IWB60307 | 6DL | ̶̶ | T/C | 6.30E-06 | 0.24 |
| GENE-4008_39517, 18 | IWB33728 | 6DL | ̶ | C/A | 1.94E-05 | 0.18 |
| RAC875_c37085_31716, 18 | IWB57246 | 7AL | 152.76 | A/G | 2.41E-04 | 0.13 |
| RAC875_c11283_72216, 18 | IWB53301 | 7AL | – | A/G | 7.43E-04 | 0.13 |
* Позиция на хромосоме указана в сМ согласно консенсусным картам мягкой пшеницы [13]. ** Полужирным шрифтом отмечен благоприятный аллель. *** Отмечены годы, для которых выявлены значимые ассоциации.
Рис. 5.
Схематическая иллюстрация локализации локусов, определяющих устойчивость мягкой пшеницы к Bgt, в хромосомах 1A, 1D, 2B, 5A, 5D, 6A, 6D и 7A. Полужирным шрифтом обозначены маркеры и QTLs, идентифицированные в настоящей работе. Порядок маркеров соответствует консенсусным картам хромосом для маркеров SNP [18] и SSR [29].
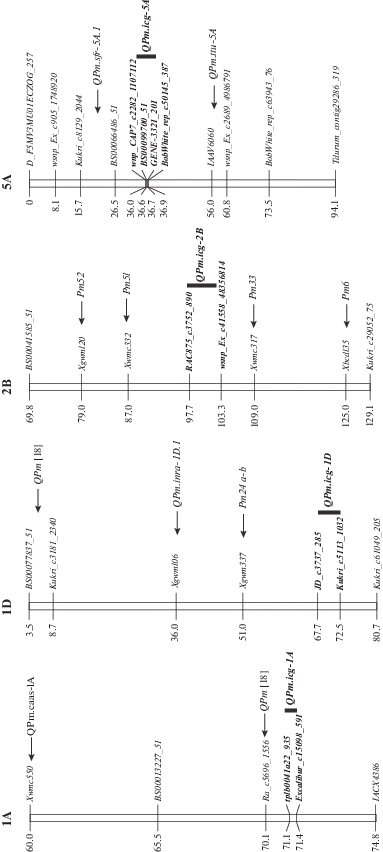
Рис. 5.
Окончание.

Пять минорных QTLs с вероятностью p < < 0.001–0.00001 обнаружены в хромосомах 1DS, 1AL, 2BL, 5DL и 7AL, при этом следует отметить, что все маркеры были определены по результатам не менее двух полевых сезонов. Наибольшее число ассоциаций установлено в хромосоме 1DS, из которых четыре наиболее значимых SNP локализуются в районе 67.72 сМ (табл. 2).
В хромосоме 5DL в районе 67.49 cM по данным 2017 и 2018 гг. идентифицированы маркеры IAAV2115, Ra_c70331_779 и RAC875_rep_c70595_321, проявляющие ассоциации с вероятностью 0.000042. Для ассоциаций, относящихся к хромосомам 1AL, 2BL и 7AL, было детектировано по два маркера, при этом SNPs в хромосоме 1AL расположены в пределах одного локуса в диапазоне 0.4 сМ.
Полученные результаты также свидетельствуют, что благоприятные аллели пяти маркеров, локализованных в хромосомах 5AS (BS00100185_51, Excalibur_rep_c115510_314, Kukri_c8835_112, BS00094095_51) и 6DL (BS00063175_51), выявляются только у средне- и высокоустойчивых сортов, что означает наличие в данных районах хромосом эффективных локусов резистентности (табл. 1). Данные маркеры могут быть использованы для диагностики локусов устойчивости к Bgt.
ОБСУЖДЕНИЕ
Результаты оценки восприимчивости сортов мягкой яровой пшеницы к мучнистой росе, представленные в настоящей работе, свидетельствуют о том, что только шесть сортов из 97 имеют высоко- и среднеустойчивый статус по отношению к популяции Bgt Западносибирского региона (табл. 1). Восприимчивость остальных варьирует по годам в зависимости от погодных условий и инфекционной нагрузки. Особенно это характерно для высоковосприимчивых сортов (балл 9-8 по шкале иммунности), число которых увеличилось от 8 в 2016 г. до 44 в 2018 г., когда наблюдался более высокий инфекционный фон. Эти результаты позволяют предположить, что большинство изученных сортов не содержат эффективных генов устойчивости к Bgt. Аналогичные результаты были получены при изучении образцов мягкой пшеницы из коллекции ВИР им. Н.И. Вавилова [27, 28]. Проверка восприимчивости более чем 800 образцов яровой и озимой мягкой пшеницы различного географического происхождения в условиях Ленинградской области свидетельствует, что только 10% образцов не поражались Bgt, при этом основная часть устойчивых сортообразцов происходила из США, Австралии, Аргентины и стран Европы. Наиболее впечатляющие результаты получены М.И. Киселевой с соавт. [2], которые при изучении 158 коллекционных образцов озимой пшеницы в полевых условиях Московской области не выявили ни одного образца с устойчивостью.
Для анализа генетических локусов устойчивости к Bgt в настоящей работе был использован GWAS, с помощью которого идентифицированы восемь локусов в хромосомах 1A, 1D, 2B, 5A, 5D, 6A, 6D и 7A. Наиболее значимые ассоциации были установлены для маркеров, локализованных в хромосомах 5А, 6A и 6D. Для постуляции генов устойчивости результаты GWAS были сопоставлены с литературными данными по хромосомной локализации известных генов/QTL (рис. 5). Позицию локусов устанавливали согласно консенсусным картам хромосом гексаплоидной пшеницы для SNP и SSR маркеров [13, 29].
В длинном плече хромосомы 6D значимые ассоциации показаны для трех маркеров, один из которых (BS00063175_51) локализован в районе 84.54 сМ. Ранее нами было установлено, что сорта Тулайковская 5 и Тулайковская 10 содержат замещение хромосомы 6D на хромосому пырея 6Ai = 2 с геном устойчивости к бурой ржавчине от Thinopyrum intermedium [30, 31]. Поскольку многолетние испытания свидетельствуют, что эти сорта характеризуются высоким уровнем устойчивости к грибным болезням, можно предположить, что хромосома пырея 6Ai = 2 несет комплекс генов устойчивости, в том числе ген Pm6Ai = 2, определяющий резистентность к Bgt. На данный момент в хромосоме 6DL известна только одна транслокация T6DL.6VS, полученная с участием Haynaldia villosa, содержащая предположительно ген Pm21 [32].
Шесть значимых SNPs детектированы в коротком плече хромосомы 6A. В хромосому 6AS интродуцированы гены Pm21 от H. villosa (транслокация T6AL.6VS) и Pm56 от Secale cereale (транслокация T6RS.6AL) [33, 34]. С использованием панели сортов озимой мягкой пшеницы в 6AS локализован QTL в районе 43.1 сМ [18]. Однако локус QPm.icg-6A, картированный в настоящей работе, по всей видимости, является новым, так как его локализация не совпадает с этими генами/QTLs.
Десять достоверных MTAs показаны для SNP, локализованных в хромосоме 5AS в районе 35.38–36.87 сМ (табл. 2, рис. 5). Локус QPm.sfr-5A.1, унаследованный от мягкой пшеницы, и QPm.ttu-5A от Triticum militinae были картированы ранее [35, 36] в диапазоне 16–26 и 55–60 сМ соответственно, что отличается от локализации локуса QPm.icg-5A, установленной в нашей работе. Полученные результаты указывают на то, что QPm.icg-5A является ранее неописанным локусом устойчивости к Bgt.
В длинное плечо хромосомы 5DL мягкой пшеницы интродуцированы гены Pm34 и Pm35, происходящие из генома Ae. tauschii, и Pm55 от Dasypyrum villosum [37, 38]. Pm34 находится в теломерной области длинного плеча вблизи маркера Xgwm272, тогда как Pm35 картирован на расстоянии 11.9 сМ дистальнее Xcfd26, что соответствует дистанции 67 сМ на генетической карте хромосом мягкой пшеницы для маркеров SSR. Хромосомная локализация локуса QPm.icg-5D, выявленного в нашей работе, совпадает с локализацией гена Pm35, поэтому нельзя исключить, что QPm.icg-5D является его аллелем.
В хромосому 1DS перенесены два аллеля гена Pm24, происходящие от китайских сортов пшеницы Chiyacao и Baihulu [5]. Pm24a и Pm24b картированы в интервале SSR-маркеров Xgwm1291 и Xgwm337, что соответствует области 51 cM на консенсусной карте [6, 39]. Три высокоинформативных SNPs были выявлены в районе локализации гена Pm24 у коллекции сортов озимой мягкой пшеницы при проведении GWAS [18]. Дополнительно к этому в районе 36 сМ (маркер Xgwm106) идентифицирован локус QPm.inra-1D.1 [10]. QPm.icg-1D, картированный нами, расположен на расстоянии не менее 16 сМ от упомянутых выше генов/QTLs, что позволяет считать его новым не описанным ранее локусом.
Три минорных локуса QPm.icg-1A, QPm.icg-2B и QPm.icg-7A были идентифицированы в хромосомах 1AL, 2BL и 7AL на основании результатов двух полевых сезонов. На данный момент имеется информация всего о двух QTLs, картированных в хромосоме 1А. Один из них QPm.caas-1A [40], тесно сцепленный с Xwmc550, находится на расстоянии 10 сМ от QPm.icg-1A (рис. 5). Второй локус был выявлен у озимых сортов мягкой пшеницы методом GWAS [18] и его положение на генетической карте совпадает с локализацией QPm.icg-1A.
В хромосому 2BL перенесено несколько чужеродных генов: Pm6 от Triticum timopheevii [5], Pm33 от Triticum carthlicum [41], Pm51 от Thinopyrum ponticum [42], Pm57 от Ae. searsii [43] и ген Pm52 от мягкой пшеницы [44]. Исходя из хромосомной локализации генов/QTLs, а также их происхождения локус QPm.icg-2B нельзя отнести ни к одному из известных на данный момент генов (рис. 5). В хромосоме 7АL картирован ген Pm1, для которого на данный момент известно пять аллелей, происходящих от T. aestivum, T. monococcum и T. spelta [5]. Pm1 находится в дистальном районе длинного плеча в комплексе с генами устойчивости к бурой и стеблевой ржавчине – Lr20, Sr15 вблизи SSR и SNP маркеров Xsfr325-7A и wsnp_JD_c1219_1766041 соответственно [45, 46]. Поскольку локализация ранее опубликованных локусов по литературным данным варьирует в диапазоне 150–200 сМ на референсных картах хромосом мягкой пшеницы, можно предположить, что локус QPm.icg-7A является аллелем гена Pm1.
Несмотря на большое число известных генов Pm, только ограниченная их часть используется в селекции. В основном это гены Pm2, Pm3, Pm4a, Pm4b, Pm6, Pm8, Pm13, Pm21 и Pm24 [12]. Согласно литературным данным в популяции Bgt, специфичной для европейской части РФ и Республики Беларусь, присутствуют клоны, вирулентные к генам Pm1a, Pm2, Pm3a,b,c, Pm4a, Pm5, Pm6, Pm7, Pm8, Pm9, Pm16, Pm17, Pm19 [27, 28, 47]. Показано, что защитный эффект генов Pm1a, Pm2, Pm3a-c, Pm4b, Pm5, Pm6, Pm8 преодолен во многих европейских странах, в США и Китае, начиная с конца 20 в. [48].
Оценка степени восприимчивости сортов и изогенных линий мягкой пшеницы к полевой популяции Bgt Западносибирского региона показывает, что гены Pm1, Pm2, Pm3b, Pm4b слабо защищают сорта от патогена. Эффективность сохраняют гены Pm5, Pm3d и ген Pm, интродуцированный от пырея Agropyron intermedium [49]. Также следует отметить значительный сдерживающий эффект пирамид, состоящих из нескольких генов устойчивости – Pm6 + Pm3d + Pm1, Pm6 + Pm4b + Pm3d + + Pm2, Pm4b + Pm5 + Pm1, Pm4b + Pm5, Pm4b + + Pm3d [50]. Это подтверждается результатами GWAS, при использовании которого нами не было выявлено значимых ассоциаций в районах локализации Pm5 (7BS) и Pm3d (1AS). Высокую степень устойчивости к Bgt имеют сорта Тулайковская золотистая, Тулайковская 10 и Лютесценс 101, защищенные генами Pm6Ai = 2 от пырея среднего (табл. 1). Наличие нескольких минорных QTLs в геномах сортов Лютесценс 85, Тулайковская белозерная, Тулайковская степная, Волгоуральская, Эритроспермум 72, Баганская 93, Рыбинская 127, АН-34 и Скэнт 1 снижает восприимчивость к патогену.
Таким образом, несмотря на то, что большая часть изученных сортов была восприимчивой к мучнистой росе, тем не менее полногеномный поиск ассоциаций позволил выявить ряд генетических факторов с различным вкладом в фенотипическое проявление признака. Результаты, полученные в настоящей работе, могут быть использованы для отбора источников целевых локусов при создании новых линий пшеницы и для разработки молекулярных маркеров к локусам Pm.
Работа выполнена при финансовой поддержке гранта РНФ 16-16-00011-П. Размножение и культивирование коллекции сортов яровой мягкой пшеницы проводились в рамках проекта Мин-обрнауки РФ № 0324-2019-0039.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Cunfer B.M. Powdery mildew // Bread Wheat: Improvement and Production. Italy: FAO, 2002. P. 317–330.
Киселева М.И., Коломиец Т.М., Пахолкова Е.В. и др. Дифференциация сортов озимой мягкой пшеницы (Triticum aestivum L.) по устойчивости к наиболее вредоносным возбудителям грибных болезней // С.-х. биология. 2016. Т. 51. № 3. С. 299–309. https://doi.org/10.15389/agrobiology.2016.3.299rus
Санин С.С., Назарова А.Н. Фитосанитарная обстановка на посевах пшеницы в Российской Федерации (1991–2008 гг.) // Защита и карантин растений. 2010. № 2. С. 70–78.
Теплякова О.И., Тепляков В.И. Локальный мониторинг болезней листьев яровой пшеницы в Сибири // Защита и карантин растений. 2011. № 6. С. 39–41.
McIntosh R.A., Yamazaki Y., Dubcovsky J. et al. Catalogue of Gene Symbols for Wheat, 2013. Suppl. 2014–2017. Available at: www.shigen.nig.ac.jp/wheat/komugi/genes/.
Huang X.O., Röder M.S. Molecular mapping of powdery mildew resistance genes in wheat: A review // Euphytica. 2004. V. 137. P. 203–223. https://doi.org/10.1023/B:EUPH.0000041576.74566.d7
Mohler V., Hsam S.L.K., Zeller F.J., Wenzel G. An STS marker distinguishing the rye-derived powdery mildew resistance alleles at the Pm8/Pm17 locus of common wheat // Plant Breeding. 2001. V. 120. P. 448–450. https://doi.org/10.1046/j.1439-0523.2001.00622.x
Spielmeyer W., Mcintosh R.A., Kolmer J., Lagudah E. Powdery mildew resistance and Lr34/Yr18 genes for durable resistance to leaf and stripe rust co-segregate at a locus on the short arm of chromosome 7D of wheat // Theor. Appl. Genet. 2005. V. 111. P. 731–735. https://doi.org/10.1007/s00122-005-2058-9
Lillemo M., Asalf B., Singh R.P. et al. The adult plant rust resistance loci Lr34/Yr18 and Lr46/Yr29 are important determinants of partial resistance to powdery mildew in bread wheat line Saar // Theor. Appl. Genet. 2005. V. 116. P. 1155–1166. https://doi.org/10.1007/s00122-008-0743-1
Li Z., Lan C., He Z. et al. Overview and application of QTL for adult plant resistance to leaf rust and powdery mildew in wheat // Crop Sci. 2014. V. 54. P. 1907–1925. https://doi.org/10.2135/cropsci2014.02.0162
Alam Md., Xue F., Wang C., Ji W. Powdery mildew resistance genes in wheat: identification and genetic analysis // J. Mol. Biol. Res. 2011. V. 1. P. 20–39. https://doi.org/10.5538/jmbr.v1n1p20
Shah L., Rehman S., Ali A. et al. Genes responsible for powdery mildew resistance and improvement in wheat using molecular marker-assisted selection // J. Plant Disease Protection. 2018. V. 125. P. 145–158. https://doi.org/10.1007/s41348-017-0132-6
Wang S., Wong D., Forrest K. et al. Characterization of polyploid wheat genomic diversity using a high-density 90 000 single nucleotide polymorphism array // Plant Biotechnology J. 2014. V. 12. P. 787–796. https://doi.org/10.1111/pbi.12183
Maccaferri M., Ricci A., Salvi S. et al. A high-density, SNP-based consensus map of tetraploid wheat as a bridge to integrate durum and bread wheat genomics and breeding // Plant Biotechnology J. 2015. V. 13. P. 648–663. https://doi.org/10.1111/pbi.12288
Gerard G.S., Börner A., Lohwasser U. et al. Genome-wide association mapping of genetic factors controlling Septoria tritici blotch resistance and their association with plant height and heading date in wheat // Euphytica. 2017. V. 213. P. 27–41. https://doi.org/10.1007/s10681-016-1920-1
Kankwatsa P., Singh D., Thomson P.C. et al. Characterization and genome-wide association mapping of resistance to leaf rust, stem rust and stripe rust in a geographically diverse collection of spring wheat landraces // Mol. Breeding. 2017. V. 37. P. 113–136. https://doi.org/10.1007/s11032-017-0707-8
Tessmann E.W., Van Sanford D.A. GWAS for Fusarium Head Blight related traits in winter wheat (Triticum aestivum L.) in an artificially warmed treatment // Agronomy. 2018. V. 8. Article № 68. https://doi.org/10.3390/agronomy8050068
Liu N., Bai G., Lin M. et al. Genome-wide association analysis of powdery mildew resistance in U.S. winter wheat // Sci. Reports. 2017. V. 7. Article No. 11743. https://doi.org/10.1038/s41598-017-11230-z
Ren Y., Hou W., Lan C. et al. QTL analysis and nested association mapping for adult plant resistance to powdery mildew in two bread wheat populations // Front. Plant Sci. 2017. V. 8. Article No. 1212. https://doi.org/10.3389/fpls.2017.01212
Saari E.E., Prescott J.M. A scale for appraising the foliar intensity of wheat diseases // Plant Disease Rep. 1975. V. 59. P. 377–380.
Леонова И.Н., Добровольская О.Б., Каминская Л.Н. и др. Молекулярный анализ линий тритикале, содержащих различные системы Vrn-генов, с помощью микросателлитных маркеров и гибридизации in situ // Генетика. 2005. Т. 41. № 9. С. 1236–1243.
Bradbury P.J., Zhang Z., Kroon D.E. et al. TASSEL: Software for association mapping of complex traits in diverse samples // Bioinformatics. 2007. V. 23. P. 2633–2635. https://doi.org/10.1093/bioinformatics/btm308
Pritchard J., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data // Genetics. 2000. V. 155. P. 945–959.
Evanno G., Regnaut S., Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study // Mol. Ecol. 2005. V. 14. P. 2611–2620. https://doi.org/10.111/j.1365-294X.2005.02553.x
Earl D.A., vonHoldt B.M. STRUCTURE HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method // Conserv. Genet. Resour. 2012. V. 4. P. 359–361. https://doi.org/10.1007/s12686-011-9548-7
Hammer Ø., Harper D.A.T., Ryan P.D. PAST: Paleontological statistics software package for education and data analysis // Palaeontologia Electronica. 2001. V. 4. P. 1–9.
Лебедева Т.В. Генетическое разнообразие мягкой пшеницы Triticum aestivum L. по устойчивости к Blumeria graminis DC. f. sp. tritici Golovin // Информ. вестник ВОГиС. 2008. Т. 12. С. 685–690.
Лебедева Т.В., Зуев Е.В., Брыкова А.Н. Проявление устойчивости к мучнистой росе у образцов яровой мягкой пшеницы из коллекции генетических ресурсов растений ВИР // Тр. по прикладной ботанике и селекции. 2018. Т. 179. № 3. С. 272–277. https://doi.org/10.30901/2227-8834-2018-3-272-277
Somers D.J., Isaac P., Edwards K. A high-density microsatellite consensus map for bread wheat (Triticum aestivum L.) // Theor. Appl. Genet. 2004. V. 109. P. 1105–1114. https://doi.org/10.1007/s00122-004-1740-7
Salina E.A., Adonina I.G., Badaeva E.D. et al. A Thinopyrum intermedium chromosome in bread wheat cultivars as a source of genes conferring resistance to fungal diseases // Euphytica. 2015. V. 204. P. 91–101. https://doi.org/10.1007/s10681-014-1344-5
Leonova I.N., Stasyuk A.I., Skolotneva E.S., Salina E.A. Enhancement of leaf rust resistance of Siberian winter wheat varieties by marker-assisted selection // Cereal Res. Commun. 2017. V. 45. P. 621–632. https://doi.org/10.1556/0806.45.2017.048
Li H., Chen X., Xin Z.Y. et al. Development and identification of wheat-Haynaldia villosa 6DL.6VS chromosome translocation lines conferring resistance to powdery mildew // Plant Breeding. 2005. V. 124. P. 203–205. https://doi.org/10.1111/j1439-0523.2004.01062.x
Qi L.L., Chen P.D., Liu D.J. et al. The gene Pm21 – a new source of resistance to wheat powdery mildew // Acta Agriculture Sinica. 1995. V. 21. P. 257–261.
Hao M., Liu M., Luo J. et al. Introgression of powdery mildew resistance gene Pm56 on rye chromosome arm 6RS into wheat // Front. Plant Sci. 2018. V. 9. Article No. 1040. https://doi.org/10.3389/fpls.2018.01040
Keller M., Keller B., Schachermayr G. et al. Quantitative trait loci for resistance against powdery mildew in a segregating wheat x spelt population // Theor. Appl. Genet. 1999. V. 98. P. 903–912. https://doi.org/10.1007s001220051149
Jakobson I., Peusha H., Timofejeva L., Jarve K. Adult plant and seedling resistance to powdery mildew in a Triticum aestivum × Triticum militinae hybrid line // Theor. Appl. Genet. 2006. V. 112. P. 760–769. https://doi.org/10.1007/s00122-005-0181-2
Miranda L.M., Murphy J.P., Marshall D. et al. Pm34: a new powdery mildew resistance gene transferred from Aegilops tauschii Coss. to common wheat (Triticum aestivum L.) // Theor. Appl. Genet. 2006. V. 113. P. 1497–1504. https://doi.org/10.1007/s00122-006-0397-9
Zhang R., Sun B., Chen J. et al. Pm55, a developmental-stage and tissue-specific powdery mildew resistance gene introgressed from Dasypyrum villosum into common wheat // Theor. Appl. Genet. 2016. V. 129. P. 1975–1985. https://doi.org/10.1007/s00122-016-2753-8
Xue F., Wang C., Li C. et al. Molecular mapping of a powdery mildew resistance gene in common wheat landrace Baihulu and its allelism with Pm24 // Theor. Appl. Genet. 2012. V. 125. P. 1425–1432. https://doi.org/10.1007/s00122-012-1923-6
Lan C., Liang S., Wang Z. et al. Quantitative trait loci mapping for adult-plant resistance to powdery mildew in Chinese wheat cultivar Bainong 64 // Phytopathology. 2009. V. 99. P. 1121–1126. https://doi.org/10.1094/PHYTO-99-10-1121
Zhu Z., Zhou R., Kong X. et al. Microsatellite markers linked to 2 powdery mildew resistance genes introgressed from Triticum carthlicum accession PS5 into common wheat // Genome. 2005. V. 48. P. 585–590. https://doi.org/10.1139/g05-016
Zhan H., Li G., Zhang X. et al. Chromosomal location and comparative genomics analysis of powdery mildew resistance gene Pm51 in a putative wheat-Thinopyrum ponticum introgression line // PLoS One. 2014. V. 9. Article No. e113455. https://doi.org/10.1371/journal.pone.0113455
Liu W., Koo D.H., Xia Q. et al. Homoeologous recombination-based transfer and molecular cytogenetic mapping of powdery mildew-resistant gene Pm57 from Aegilops searsii into wheat // Theor. Appl. Genet. 2017. V. 130. P. 841–848. https://doi.org/10.1007/s00122-017-2855-y
Zhao Z., Sun Y., Somg W. et al. Genetic analysis and detection of the gene MlLX99 on chromosome 2BL conferring resistance to powdery mildew in the wheat cultivar Liangxing 99 // Theor. Appl. Genet. 2013. V. 126. P. 3081–3089. https://doi.org/10.1007/s00122-013-2194-6
Neu C., Stein N., Keller B. Genetic mapping of the Lr20-Pm1 resistance locus reveals suppressed recombination on chromosome arm 7AL in hexaploid wheat // Genome. 2002. V. 45. P. 737–744. https://doi.org/10.1139/g02-040
Aoun M., Breiland B., Turner M.K. et al. Genome-wide association mapping of leaf rust response in a durum wheat worldwide germplasm collection // Plant Genome. 2016. V. 9. № 3. P. 1–24. https://doi.org/10.3835/plantgenome2016.01.0008
Булойчик А.А., Борзяк В.С. Встречаемость генов вирулентности в белорусской популяции Blumeria graminis f. sp. tritici // Микология и фитопатология. 2013. Т. 47. С. 405–409.
Cowger C., Mehra L., Arellano C. et al. Virulence differences in Blumeria graminis f. sp. tritici from the Central and Eastern United States // Phytopathology. 2018. V. 108. P. 402–411. https://doi.org/10.1094/PHYTO-06-17-0211-R
Сочалова Л.П., Лихенко И.Е. Изучение устойчивости пшеницы к листовым патогенам в условиях Западной Сибири // Сиб. вестник с.-х. науки. 2011. № 1. С. 18–25.
Сочалова Л.П., Пискарев В.В. Устойчивость сортов яровой мягкой пшеницы к возбудителям инфекционных заболеваний в условиях изменяющегося климата Западной Сибири // Достижения науки и техники АПК. 2017. Т. 31. № 2. С. 21–25.
Дополнительные материалы отсутствуют.