Генетика, 2019, T. 55, № 4, стр. 390-397
Молекулярно-генетический полиморфизм Puccinia triticina в Южном Дагестане – центре совместной эволюции возбудителя бурой ржавчины и пшеницы
Е. И. Гультяева 1, *, И. А. Казарцев 1, Е. Л. Шайдаюк 1
1 сероссийский научно-исследовательский институт защиты растений
196608 Санкт- Петербург-Пушкин, Россия
* E-mail: eigultyaeva@gmail.com
Поступила в редакцию 23.04.2018
После доработки 06.06.2018
Принята к публикации 23.07.2018
Аннотация
Проведена оценка генетического полиморфизма по вирулентности, микросателлитным локусам (SSR) и однонуклеотидным заменам (SNP) у изолятов возбудителя бурой ржавчины Puccinia triticina Erikss., собранных в Южном Дагестане на видах Triticum L. sp. и Aegilops L. sp. Наличие дифференциации популяций патогена по результатам микросателлитного анализа и изучения вирулентности указывает на существование в дербентской популяции нескольких генетически различающихся групп изолятов. С использованием SNP маркеров определено высокое филогенетическое родство между дербентскими изолятами P. triticina и референсными с твердой и мягкой пшеницы из разных стран, информация по которым получена из Генбанка.
Южный Дагестан относится к Переднеазиатскому генцентру происхождения пшениц и их совместной эволюции с паразитами, в том числе и с возбудителем бурой ржавчины (Puccinia triticina Erikss.). В растительных сообществах низинной и предгорной зон широко представлены виды родов Aegilops L. (Ае. tauschii (Coss.) Schmalh., Ae. triuncialis L., Ae. biuncialis Viz., Ae. triaristata Willd. и Ae. cylindrica), Agropyron L., Cynondon Rich., Thalictrum Tourn ex L., Anchusa L. и Triticum L. (T. persicum Vav. ex Zhuk., T. dicoccum (Schrank) Schubl.) [1–3]. Благодаря длительному периоду с оптимальными температурами и влажностью воздуха, высокому разнообразию растений-хозяев в регионе поддерживается высокая численность паразита. Н.И. Вавилов считал это место уникальным для проведения генетико-популяционных исследований хозяина и паразита [1].
На Дагестанской опытной станции ВИР (ДОС ВИР), расположенной в Южном Дагестане (Дербентский район), ежегодно изучается генетически разнообразная коллекция пшениц и эгилопсов, которая в целом представляет генофонд устойчивости к бурой ржавчине. Вокруг станции произрастают дикие злаки, в том числе виды пырея и эгилопсов, восприимчивые к бурой ржавчине. Осенний посев пшеницы и теплая зима создают благоприятные условия для перезимовки и воспроизведения популяции патогена [4]. Существует мнение [1], что основной (материнской) популяцией P. triticina в этом регионе является совокупность клонов патогена, обитающих в течение года на пырее и других многолетних злаках и лишь сезонно распространяющихся на эфемерные злаки и пшеницу. В результате изучения генетического разнообразия образцов популяций P. triticina, собранных с дикорастущих злаков в Дагестане в 1970–1980 гг., показано их высокое разнообразие. Определены клоны, авирулентные к девяти линиям-дифференциаторам Thatcher (ТсLr1, ТсLr2a, ТсLr2c, ТсLr3a, ТсLr3ka, ТсLr14, ТсLr16, ТсLr17, ТсLr18), и клоны с определенными сочетаниями аллелей вирулентности/авирулентности, которые характеризовались разной специфичностью к видам пшеницы и эгилопса [1, 5].
Существенное селективное влияние на отбор патогена по вирулентности отмечено и на видах Triticum, и на Aegilops, изучаемых на ДОС ВИР [1, 5, 6]. В 2014 г. нами [5–7] был изучен состав дербентской популяции P. triticina, собранной с диплоидного вида Ae. tauschii, тетраплоидных T. aethiopiсum Jakubz., T. turaniсum Jakubz., T. dicoccoides (Körn. ex Asch. & Graebner) Schweinf., Ae. crassa Boiss., и гексаплоидных видов Ae. juvenalis (Thell.) Eig., Ae. trivialis (Zhuk.) Migusch. et Chak., T. compactum Host., T. macha Dekapr. et Menabde, T. petropavlovskyi Udacz. et Migusch., T. spelta L., T. sphaerococcum Perc., T. vavilovii и T. aestivum L. Отмечено, что изоляты P. triticina на тетраплоидных видах характеризовались меньшим числом аллелей вирулентности по сравнению с изолятами на гексаплоидах и диплоидном виде Ae. tauschii. Изоляты, выделенные из гексаплоидных видов Triticum sp. и Ae. crassa, характеризовались высоким генетическим родством по микросателлитным локусам, а изоляты с тетраплоидных видов T. aethiopiсum, T. turaniсum и T. dicоccum отличались от них. В данных исследованиях была хорошо представлена группа изолятов P. triticina, выделенных из гексаплоидных пшениц (с девяти видов) и умеренно – из тетраплоидных (с четырех видов) и диплоидных (с одного вида). Для уточнения влияния растений-хозяев разной плоидности на генетическую дифференциацию патогена представляло интерес дополнить ранее проведенный анализ коллекциями возбудителя с диплоидных и тетраплоидных видов Triticum и Aegilops.
Молекулярные подходы, применяемые для изучения популяций P. triticina, постоянно совершенствуются. До недавнего времени для этого использовали RAPD [8–10], AFLP [11] и SSR маркеры [12, 13]. В середине 2010 г. были подобраны SNP маркеры, которые позволяют оценить филогенетическое родство между изолятами с разных видов-хозяев и их дивергенцию. С использованием SNP маркеров изучена микроэволюция гриба P. triticina на мягкой и твердой пшеницах и Ae. speltoides Tausch. [14]. Показано, что сопряженная эволюция шла по вектору Ae. speltoides (донор генома B и цитоплазмы аллополиплоидных рядов пшеницы) – T. durum Desf. (популяции из Эфиопии) – T. aestivum. Представляло интерес изучить полиморфизм по олигонуклеотидным заменам (SNP) у дербентских изолятов P. triticina, поражающих виды Aegilops и Triticum, и сравнить его с задепонированным в Генбанке Лью с соавт. [14] для изолятов с твердой и мягкой пшеницы из других стран.
Цель работы – характеристика молекулярно-генетического полиморфизма дербентских изолятов P. triticina, поражающих ди-, тетра- и гексаплоидные виды Triticum и Aegilops в южном Дагестане.
МАТЕРИАЛЫ И МЕТОДЫ
Инфекционный материал, представленный листьями с урединиопустулами, был собран с четырех образцов диплоидных, шести тетраплоидных и двух гексаплоидных видов пшеницы и эгилопса, изучаемых на ДОС ВИР в 2017 г. (табл. 1). Для получения монопустульных изолятов и их размножения использовали метод лабораторного культивирования гриба P. triticina. Анализ вирулентности проводили на живых растениях. Для этого 10–14-дневные проростки пшеницы (фаза первого листа) инокулировали суспензией возбудителя, помещали во влажную камеру на 12–18 ч и далее инкубировали при температуре 20–24°С [15]. Учет проводили на 10-й день после заражения по балловой шкале Mains и Jackson [16], где 0, 1, 2 балла – устойчивый тип реакции, 3, 4 и Х – восприимчивый.
Таблица 1.
Характеристика инфекционного материала Puccinia triticina, собранного на Дагестанской опытной станции ВИР в 2017 г.
| Вид | Источник инфекции | Изучено изолятов (анализ вирулентности) |
Фенотипы вирулентности | Среднее число аллелей вирулентности | Изучено изолятов (SSR анализ) |
SSR генотип |
|---|---|---|---|---|---|---|
| T. aestivum BAuD |
к-65372 Донской маяк, к-65223 Гром, к-65223 Васса, Россия | 9 | PHPTH THPTR | 15 | 3 | II, IV |
| T. compactum BAuD |
к-41428 Монголия к-13800 Армения к-28564 Казахстан к-56573 Узбекистан к-21045 Турция к-21047 Турция |
25 | PHPTH PHPTG PHTTG PCPTG THPTQ | 13.6 | 4 | II, IV, VII |
| T. durum ВAu | Kofa Италия Kundyru 1149 Турция Audin 93 Турция Firat 93 Турция |
10 | MHMKG MHMKH | 10 | 17 | V, VI |
| T. dicоccoides ВAu |
к-61816, к-15903,
к-61817 Израиль к-17157 Сирия |
12 | MHMKG MHMKH LHCFG | 9.3 | 6 | IV |
| T. dicоccum ВAu | к-7497 к-7492 Россия |
7 | MHMKG MHRKG | 10.5 | 2 | IV |
| T. aethiopicum ВAu | Не известен | 14 | MHMKG MHMKH | 10.5 | – | – |
| T. polonicum BAu | Не известен | 6 | MHMKG | 10 | 2 | III |
| T. persicum AuB | Не известен | 3 | MHMKG | 10 | 2 | V |
| T. monococcum Аb | к-46140 Балканы к-39414 Албания |
5 | TCPTR | 15 | 4 | II, VIII |
| Ae. tauschii D | Не известен | 10 | TCPTR | 15 | 3 | I, II |
| Ae. sharonensis Sl | Не известен | 3 | MCPKG | 10 | 2 | III, IV |
| Ae. caudata C | Не известен | 5 | MCPKG | 10 | 2 | I |
В анализе вирулентности использовали 20 почти изогенных линий Thatcher с генами Lr (ТсLr). Для обозначения фенотипов вирулентности линии TcLr были разделены на пять наборов по четыре линии в каждом: 1-й набор – Lr1, Lr2a, Lr2c, Lr3а; 2-й – Lr9, Lr16, Lr24, Lr26; 3-й – Lr3ka, Lr11, Lr17, Lr30, 4-й – Lr2b, Lr3bg, Lr14a, Lr14b; 5-й – Lr15, Lr18, Lr19, Lr20. Первые три группы линий соответствовали традиционному североамериканскому набору [17], остальные включали линии, использовавшиеся нами в предыдущих исследованиях популяций P. triticinа [6]. Буквенный код фенотипов (рас), частоты вирулентности и фенотипов определяли с использованием пакета программ VirulenceAnalysisTool (VAT) [18]. Для оценки генетической дифференциации коллекций патогена с Triticum sp. и Aegilops sp. использовали индексы генетических расстояний Нея (Nei_D, Nei Distance) и FST, которые были рассчитаны с помощью алгоритма AMOVA в пакете программ GenAlEx 6.5 (Genetic analysis in Excel, 6.5 http://biology.anu.edu.au/GenAlEx).
Выделение ДНК гриба проводили по методике Джастисен с соавт. [19]. Для SSR анализа использовали 16 микросателлитных маркеров (PtSSR13, PtSSR50, PtSSR55, PtSSR61, PtSSR68, PtSSR91, PtSSR92, PtSSR151, PtSSR152, PtSSR158, PtSSR164, PtSSR173, PtSSR186, RB8, RB26, RB35) [12, 13]. SSR анализ проводили по ранее описанной методике [7]. Статистическую обработку результатов SSR анализа осуществляли в пакете программ GenAlEx 6.5 (Genetic analysis in Excel, 6.5 http://biology.anu.edu.au/GenAlEx). Для характеристики внутрипопуляционной изменчивости по микросателлитным локусам использовали следующие показатели: среднее число аллелей на локус (Na), число эффективных аллелей (Ne), % полиморфных локусов, ожидаемая (HE) и наблюдаемая (HO) гетерозиготность, индекс фиксации (F) и индекс Шеннона (I). Генетическую дифференциацию популяций определяли по индексам FST и Нея (Nei_D, Nei Genetic Distance), которые были рассчитаны с использованием алгоритма AMOVA (GenAlЕx) (для 999 пермутаций).
Многомерные дендрограммы родства между коллекциями изолятов P. triticina с разных видов-хозяев по вирулентности и микросателлитным локусам построены по индексу FST в пакете программ GenAlEx 6.5 (опция PCoA).
Для SNP анализа использовано 24 изолята P. triticina. Среди них 12 дагестанских изолятов, изученных в представленной работе (один с Ae. tauschii, два c Ae. sharonensis Eig., один c T. monococcum, один c T. dicoccum, один с T. dicoccoides, три c T. durum, один с T. polonicum, один с T. aephiopicum, один с T. aestivum), и 12, охарактеризованных в предыдущих исследованиях [6, 7] (один с Ae. tauschii, один c T. boeticum Boiss., один с T. aephiopicum, один с T. spelta, один c T. vavilovii, один c T. petropavlovskyi Udacz., один с T. sphaerococcum (Дагестан), один с T. durum (Казахстан), один с T. dicoccoides, один с T. aestivum (Новосибирск), два с T. aestivum (Алтай)).
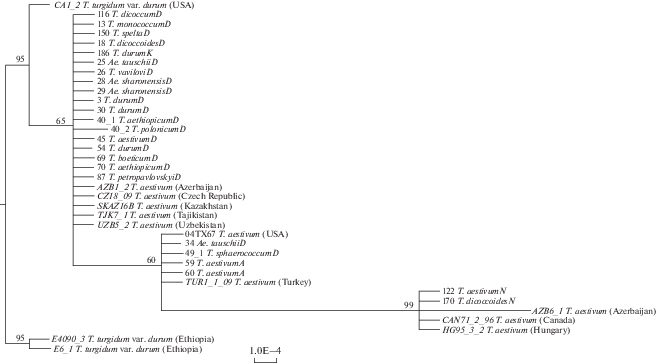
Для осуществления SNP анализа было выбрано три стабильно амплифицирующихся локуса (ctg1-3, ctg5-1, ctg84-1), при работе с которыми не наблюдалась аномальная гетерозиготность, обусловленная повышенной неспецифичностью ПЦР, которую проводили согласно протоколам разработчиков маркеров, представленным в работе Лью с соавт. [14]. Секвенирующую ПЦР проводили по классическому методу обрыва цепи [20] с использованием набора BigDye Terminator v. 3.1 Cycle Sequencing Kit (ABI, США). Определение нуклеотидных последовательностей осуществляли на генетическом анализаторе ABI PRIZM 3500 (ABI-Hitachi, Япония). Полученные последовательности редактировали в программе Vector NTI Advance 11.5.1. Выравнивание последовательностей выполняли в программе MEGA 6.0 [21]. Полученные для российских изолятов данные сравнили с представленными Лью с соавт. [14]. Для этого из базы данных GenBank были получены последовательности локусов ctg1-3, ctg5-1, ctg84-1 для репрезентативного числа изолятов с твердой и мягкой пшеницы из разных стран [14]. В итоге в группу для сравнения вошли следующие изоляты с T. durum: E4090-3, E6-1 (Эфиопия), CA1_2 (Северная Америка), и с T. aestivum: 04ТХ67 (Северная Америка), CAN71_2_96 (Канада), HG95_3_2 (Венгрия), CZ18_09 (Чехия), TUR1_1_09 (Турция), AZB1_2, AZB6_1 (Азербайджан), SKAZ16B (Южный Казахстан), TJK7_1 (Таджикистан), UZB5_2 (Узбекистан) [14]. Нуклеотидные последовательности каждого локуса были конкатенированы в программе Sequence Matrix v. 1.7.8 [22]. Филогенетическое дерево для мультилокусных последовательностей построено с помощью программы MrBayes v. 3.2 с использованием модели нуклеотидных замен GTR+G+I и генерации 1 × × 106 марковских цепей методом Монте-Карло [23]. Все последовательности были помещены в Международную базу данных нуклеотидных последовательностей (INSD) через GenBank (CTG 84-1 MH215368–MH215391, CTG 1-3 MH215416–MH215439, CTG 5-1 MH215392–MH215415).
РЕЗУЛЬТАТЫ
По признаку вирулентности охарактеризовано 109 изолятов гриба (табл. 1). Высоким уровнем устойчивости характеризовались линии с генами Lr9, Lr19 и Lr24. Абсолютной неэффективностью характеризовались гены Lr1, Lr14a, Lr14b, Lr18 и Lr30 (частоты вирулентности 100%). На других линиях наблюдали вариабельность по типу инфекции (табл. 2). Изоляты, выделенные из тетраплоидных видов, характеризовались меньшим числом аллелей вирулентности (10.1) по сравнению с изолятами из гексаплоидов (14). Полученные результаты согласуются с ранее проведенными исследованиями для изолятов с других тетра- и гексаплоидных видов (9.4 и 12.7 соответственно [6]). Все дербентские изоляты с тетраплоидных видов были авирулентны к TcLr2a, TcLr2b, TcLr2c, TcLr15 и TcLr17. Изоляты с диплоидных видов T. tauschii и T. monococcum характеризовались большей вирулентностью (15 аллелей) по сравнению с Ae. sharonensis и Ae. caudata L. (10). Для T. tauschii результаты согласуются с полученными нами в 2014 г. (14.6) [6].
Таблица 2.
Частота изолятов Puccinia triticina, вирулентных к линиям Thatcher, на видах пшеницы и эгилопсов (%)
| Линия Thatcher с геном Lr |
2n = 42 | 2n = 28 | 2n = 14 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| T. aestivum | T. compactum | T. durum | T. dicоccoides | T. dicоccum | T. aethiopicum | T. polonicum | T. persicum | T. monococcum | Ae. tauschii | Ae. sharonensis | Ae. caudata | |
| 9, 19, 24 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| 2a | 22 | 8 | 0 | 0 | 0 | 0 | 0 | 0 | 100 | 100 | 0 | 0 |
| 2b | 100 | 100 | 0 | 0 | 0 | 0 | 0 | 0 | 100 | 100 | 0 | 0 |
| 2c | 100 | 100 | 0 | 0 | 0 | 0 | 0 | 0 | 100 | 100 | 0 | 0 |
| 3a | 100 | 100 | 100 | 100 | 100 | 50 | 100 | 100 | 100 | 100 | 100 | 100 |
| 3bg | 100 | 100 | 100 | 100 | 100 | 50 | 100 | 100 | 100 | 100 | 100 | 100 |
| 3ka | 100 | 100 | 100 | 100 | 100 | 50 | 100 | 100 | 100 | 100 | 100 | 100 |
| 11 | 0 | 12 | 0 | 0 | 14 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| 15 | 22 | 8 | 0 | 0 | 0 | 0 | 0 | 0 | 100 | 100 | 0 | 0 |
| 16 | 100 | 16 | 100 | 100 | 100 | 100 | 100 | 100 | 0 | 0 | 0 | 0 |
| 17 | 100 | 100 | 0 | 0 | 0 | 0 | 0 | 0 | 100 | 100 | 100 | 100 |
| 20 | 100 | 16 | 80 | 17 | 0 | 64 | 0 | 0 | 100 | 100 | 0 | 0 |
| 26 | 100 | 100 | 80 | 100 | 100 | 100 | 100 | 100 | 100 | 100 | 100 | 100 |
| 1, 14a, 14b, 18, 30 | 100 | 100 | 100 | 100 | 100 | 100 | 100 | 100 | 100 | 100 | 100 | 100 |
| Среднее число аллелей вирулентности | 14 | 13.6 | 10.6 | 9.3 | 10.1 | 10 | 10 | 10 | 15 | 15 | 10 | 10 |
Всего выявлено 13 фенотипов патогена, которые представлены в табл. 1. Общие фенотипы определены на гексаплоидных видах T. aestivum и T. compactum (PHPTH); на тетраплоидных видах T. durum, T. dicоccoides, T. dicоccum, T. aethiopicum, T. polonicum и T. persicum (MHMKG); диплоидных T. monococcum и Ae. tauschii (TCPTR), Ae. sharonensis и Ae. caudata (MCPKG). Согласно индексам генетических расстояний Нея (Nei_D) и FST определена дифференциация по вирулентности между изолятами P. triticina на разных видах пшеницы и эгилопса. Генетическое расстояние между изолятами P. triticina, полученными с гексаплоидных видов, по индексу Нея составляло 0.07; в группе c тетраплоидных видов варьировало от 0 до 0.1, в группе c диплоидных видов от 0 до 0.29. Аналогичные результаты дифференциации получены по индексу FST (рис. 1,а).
Рис. 1.
Многомерная дендрограмма генетического родства между коллекциями изолятов с видов Triticum и Aegilops по вирулентности (а) и по микросателлитным локусам (б) (по индексу FST).
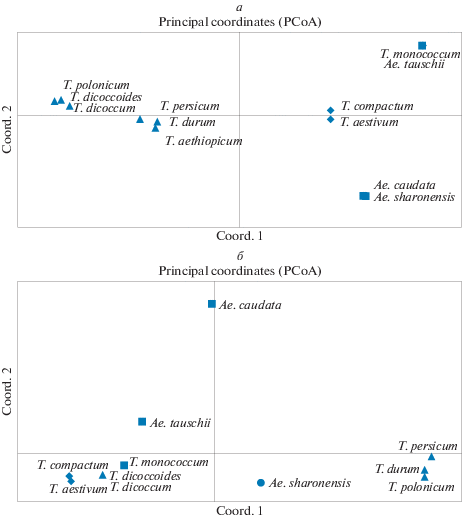
Полиморфизм микросателлитных локусов изучили у 47 изолятов (табл. 1). Идентифицировано 25 полиморфных аллелей и восемь SSR генотипов. В табл. 1 они обозначены как I–VIII. Среднее число аллелей на локус (Na) составило 1.25 ± 0.05, число эффективных аллелей (Ne) 1.22 ± ± 0.03, процент полиморфных локусов 25 ± 2.5 и индекс Шеннона (I) 0.16 ± 0.02. Уровень наблюдаемой гетерозиготности (НО) во всех коллекциях изолятов был выше уровня ожидаемой (НЕ), что подтверждается отрицательными значениями индекса фиксации (F). Внутрипопуляционное разнообразие дербентской популяции P. triticina по микросателлитным локусам было ниже, чем по вирулентности (восемь SSR генотипов и 13 фенотипов вирулентности). Согласно индексу Нея (Nei_D), высоким родством по микросателлитным локусам характеризовались изоляты патогена на T. aestivum и T. compactum (Nei_D = 0.004). Изоляты на T. dicoccum, T. dicoccoides (0.004–0.008), T. monococcum (0.003–0.02), Ae. tauschii (0.01–0.02) и Ae. sharonensis (0.02) были близки по сходству с этой группой. В отдельную близкородственную группу выделились изоляты с T. durum, T. polonicum и T. persicum (0.004–0.02), и эта группа умеренно дифференцировалась от всех других изученных (0.08–0.1). Изоляты с Ae. caudatа отличались от всех изученных (0.1–0.2). Аналогичные результаты получены по индексу FST (рис. 1,б). Изоляты с твердой пшеницы достоверно отличались (Р > 0.05) от изолятов с T. compactum, T. aestivum, Ae. tauschii, Ae. caudatа, T. monococcum; изоляты с T. persicum – от изолятов с T. aestivum, T. monococcum, T. dicoccum; изоляты с T. polonicum – от изолятов с T. compactum, T. dicoccum, T. dicoccoides, T. monococcum, Ae. tauschii, Ae. caudatа; изоляты с Ae. caudatа – от изолятов с T. compactum, T. aestivum, T. monococcum.
SNP анализ выявил 14 полиморфных сайтов при использовании маркера ctg1-3, 15 сайтов для ctg5-1 и 3 сайта для ctg84-1. В исследованиях Лью с соавт. [14] число сайтов составляло 26, 10 и 8 соответственно. Филогения изученных нами изолятов P. triticina и исследованных Лью с соавт. [14] представлена на рис. 2. Изоляты P. triticina разделились на две клады (достоверность 95%). В исследованиях Лью с соавт. [14] эфиопские изоляты с твердой пшеницы существенно дифференцировались от всех изученных. Показано [14], что в историко-эволюционном контексте они рассматриваются в качестве более ранних по отношению к изолятам P. triticina на твердой и мягкой пшенице. На дендрограмме, построенной нами для российских изолятов и отобранных из Генбанка [14], изоляты из Эфиопии с твердой пшеницы также кластеризовались в отдельную группу (рис. 2). Большинство дербентских изолятов с мягкой пшеницы формировали две слабо поддерживаемых клады, что свидетельствует об их низком уровне дивергенции и согласуется с результатами Лью с соавт. [14]. При этом в отдельную кладу выделились изоляты с мягкой пшеницы из Новосибирска и три референсных из Канады, Венгрии, Азербайджана. Такой уровень сходства для референсных сиквенсов ранее не был отмечен их авторами (данные были получены для большего числа локусов), вероятно, несмотря на высокий уровень поддержки, эту группу можно расценивать как артефакт, и она, возможно, элиминируется при увеличении числа локусов и изолятов.
ОБСУЖДЕНИЕ
Считается, что наибольшим генетическим разнообразием характеризуются центры происхождения видов. В данном исследовании подтверждено высокое генотипическое разнообразие дербентской популяции возбудителя бурой ржавчины на видах Triticum sp. и Aegilops sp. по признаку вирулентности и по микросателлитным локусам, что согласуется с проведенными нами ранее исследованиями для других гексаплоидных и тетраплоидных видов-хозяев патогена [6, 7]. Изоляты с гексаплоидных и диплоидных видов Triticum и Aegilops характеризовались более высоким полиморфизмом по вирулентности, чем изоляты с тетраплоидных видов. По полиморфизму микросателлитных локусов разнообразие патогена было равнозначным на всех изученных видах-хозяевах.
Показано, что диплоидные пшеницы в целом характеризуются высокой устойчивостью к бурой ржавчине, гексаплоидные – сильной восприимчивостью, а тетраплоидные занимают промежуточное положение (наблюдаются существенные различия по устойчивости между разными видами) [1]. В данном исследовании изоляты с тетраплоидных видов характеризовались меньшим числом аллелей вирулентности и отличались от изолятов с гексаплоидных и диплоидных видов. Аналогичные результаты были получены нами ранее [6] для изолятов с тетраплоидных видов T. turanicum, T. aethiopicum, T. dicoccum, T. dicoccoides и Ae. crassa. Дифференциация изолятов, выделенных из близкородственных видов T. durum, T. polonicum и T. persicum, от других, изученных по полиморфизму микросателлитных локусов, согласуется с ранее полученными результатами для генетически близких к ним видов T. turanicum и T. aethiopicum [7].
Существенная вариабельность по вирулентности и микросателлитным локусам отмечена у изолятов на диплоидных видах Ae. caudatа, Ae. tauschii, Ae. sharonensis и T. monococcum. Эти виды имеют разные геномы, характеризуются как устойчивые к бурой ржавчине и широко используются в селекции для расширения генетического разнообразия мягкой пшеницы [24].
Наличие дифференциации популяций патогена, выявленной по результатам микросателлитного анализа и вирулентности, указывает на существование нескольких генетически различающихся групп изолятов в пределах дербентской популяции. Основной источник спор бурой ржавчины в окрестностях ДОС ВИР – дикие злаки. Многочисленные споры, летящие с них, оседают на изучаемых образцах пшеницы и эгилопсов. Вначале отбираются споры, которые имеют вирулентность к соответствующим растениям-хозяевам. Дальнейший их отбор обусловливается процессом конкуренции на генотипах хозяев [1, 5, 25]. Эти изменения затрагивают не только генетические механизмы вирулентности патогена, но и полиморфизм микросателлитных локусов. В мировой литературе такие сведения ранее были представлены только для изолятов с T. durum, T. aestivum и Ae. speltoides [26, 27].
Адаптация и специфичность к растению-хозяину являются ключевым движущим фактором эволюции и дивергенции клонально размножающихся фитопатогенов, к которым относится бурая ржавчина [14]. Несмотря на дифференциацию между дербентскими изолятами по вирулентности и микросателлитным локусам, определено высокое филогенетическое родство между ними по SNP полиморфизму. Дербентские изоляты характеризовались высоким сходством по SNP полиморфизму с изолятами, полученными с твердой и мягкой пшениц в разных странах, информация о которых представлена в Генбанке [14], за исключением группы изолятов из Эфиопии. Считается, что Эфиопия является центром разнообразия тетраплоидных пшениц и адаптация возбудителя бурой ржавчины к ним произошла свыше тысячи лет назад. В наших исследованиях SNP маркеры были менее полиморфны по сравнению с вирулентностью и микросателлитными маркерами, что, вероятно, обусловлено малым количеством анализируемых локусов. Однако в целом сведения о филогенетическом родстве между изученными изолятами патогена согласуются с полученными Лью с соавт. [14].
Комплексный анализ с использованием признака вирулентности и молекулярных маркеров разного типа позволяет наиболее полно охарактеризовать генетическую изменчивость и микроэволюционные процессы в популяциях патогена. В современный период данный методический подход является приоритетным в генетико-популяционных исследованиях.
Работа выполнена при поддержке гранта Российского научного фонда (проект № 14-26-00067).
Список литературы
Берлянд-Кожевников В.М., Дмитриев А.П., Будашкина Е.Б. и др. Устойчивость пшеницы к бурой ржавчине (Генетическое разнообразие популяций гриба и растения-хозяина). Новосибирск: Наука, 1978. 442 с.
Богуславский Р.Л., Голик О.В. Род Aegilops L. как генетический ресурс селекции. Харьков, 2004. 236 с.
Дорофеев В.Ф., Удачин Р.А., Семенова Л.В. и др. Пшеницы мира. Л.: Колос, 1987. 487 с.
Mихайлова Л.А., Абдуллаев К.М., Шеломова Л.Ф. Изменение структуры популяции Рuccinia recondita Rob. ex Desm. f. sp. tritici в окрестностях Дербента (Дагестан) в 1970–1995 гг. // Микол. и фитопатол. 1997. Т. 31. № 2. С. 71–77.
Михайлова Л.А. Генетика взаимоотношений возбудителя бурой ржавчины и пшеницы / Под ред. Левитина М.М. СПб.: ВИЗР, 2006. 80 с.
Gultyaeva E.I., Shaydayuk E.L., Goncharov N.P. et al. Virulence of Puccinia triticina on Tritcum and Aegilops species // Austral. Plant Pathol. 2016. V. 45(2). P. 155–163. doi 10.1007/s13313-016-0395-6
Gultyaeva E.I., Shaydayuk E.L., Kazartsev I.A. et al. Microsatellite analysis of Puccinia triticina from Triticum and Aegilops hosts // Austral. Plant Pathol. 2018. V. 47(2). P. 163–170. doi 10.1007/s13313-018-0542-3
Kolmer J.A., Liu J.Q., Sies M. Virulence and molecular polymorphism in Puccinia recondita f. sp. tritici in Canada // Phytopathology. 1995. V. 85. P. 276–285. doi 10.1094/Phyto-85-276
Kolmer J.A., Liu J.Q. Virulence and molecular polymorphism in international collections of the wheat leaf rust fungus Puccinia triticina // Phytopathology. 2000. V. 90. № 4. P. 427–436. doi 10.1094/PHYTO.2000.90.4.427
Gultyaeva E.I., Dmitriev A.P., Kosman E. Regional diversity of Russian populations of Puccinia triticina in 2007 // Canad. J. Plant Pathol. 2012. V. 34(2). P. 213–224. doi 10.1080/07060661.2011.633562
Kolmer J.A. Molecular polymorphism and virulence phenotypes of the wheat leaf rust fungus Puccinia triticina in Canada // Canad. J. Bot. 2001. V. 79. P. 917–926. doi 10.1139/b01-075
Duan X., Enjalbert J., Vautrin D. et al. Isolation of 12 microsatellite loci, using an enrichment protocol, in the phytopathogenic fungus Puccinia triticina // Mol. Ecol. Notes. 2003. V. 3. P. 65–67. doi 10.1046/j.1471-8286.2003.00350.x
Szabo L.S., Kolmer J.A. Development of simple sequence repeat markers for the plant pathogenic rust fungus Puccinia triticina // Mol. Ecol. Notes. 2007. V. 7. P. 708–710. doi 10.1111/j.1471-8286.2007.01686.x
Liu M., Rodrigue N., Kolmer J. Population divergence in the wheat leaf rust fungus Puccinia triticina is correlated with wheat evolution // Heredity. 2014. V. 112. P. 443–453. doi 10.1038/hdy.2013.123
Гультяева Е.И., Солодухина О.В. Ржавчинные болезни зерновых культур. Изучение генетических ресурсов зерновых культур по устойчивости к вредным организмам: Методич. пособие. М., 2008. С. 5–11.
McIntosh R.A., Wellings C.R., Park R.F. Wheat rusts. An atlas of resistance genes. Plant Breeding Institute, University of Sydney, CSIRO, Australia, 1995.
Long D.L., Kolmer J.A. North American system of nomenclature for Puccinia recondita f. sp. tritici // Phytopathology. 1989. V. 79. P. 525–529. doi 10.1094/Phyto-79-525
Kosman E., Dinoor A., Herrmann A., Schachtel G.A. Virulence Analysis Tool (VAT). User Manual. 2008. http://www.tau.ac.il/lifesci/departments/plants/members/kosman/VAT.html.
Justesen A.F., Ridout C.J., Hovmoller M.S. The recent history of Puccinia striiformis f. sp. tritici in Denmark as revealed by disease incidence and AFLP markers // Plant Pathol. 2002. V. 51. P. 13–23. doi 10.1046/ j.0032-0862.2001.00651.x
Sanger F., Niclein S., Coulson A.R. DNA sequencing with chain-terminating inhibitors // Proc. Natl Acad. Sci. USA. 1977. V. 74. P. 5463–5467.
Tamura K., Stecher G., Peterson D. et al. MEGA6: Molecular Evolutionary Genetics Analysis version 6.0 // Mol. Biol. Evol. 2013. V. 30. P. 2725–2729.
Vaidya G., Lohman D.J., Meier R. SequenceMatrix: concatenation software for the fast assembly of multi-gene datasets with character set and codon information // Cladistics. 2011. V. 27. № 2. P. 171–180. doi 10.1111/ j.1096-0031.2010.00329.x
Huelsenbeck J.P., Ronquist F. MRBAYES: Bayesian inference of phylogeny // Bioinformatics. 2001. V. 17. P. 754–755. doi 10.1093/bioinformatics/17.8.754
Чикида Н.Н., Максимов И.В., Давоян Р.О. Перспективы использования разногеномных видов эгилопсов (диких родичей пшеницы) для расширения генетического потенциала продовольственной пшеницы // Здоровье – основа человеческого потенциала: проблемы и пути их решения. 2011. Т. 6. № 1. С. 622–628.
Дмитриев А.П., Михайлова Л.А., Шеломова Л.Ф., Деревянкин А.И. Исследование расового и генотипического состава дербентской популяции Рuccinia recondita Rob. ex Desm. в 1972–1973 гг. // Микология и фитопатология. 1976. Т. 10. № 4. С. 61–64.
Ordoñez M.E., Kolmer J.A. Virulence phenotypes of a worldwide collection of Puccinia triticina from durum wheat // Phytopathology. 2007. V. 97. P. 344–351. doi 10.1094/PHYTO-97-3-0344
Mantovani P., Maccaferri M., Tuberosa R., Kolmer J. Virulence phenotypes and molecular genotypes in collections of Puccinia triticina from Italy // PlantDis. 2010. V. 94. P. 420–424.
Дополнительные материалы отсутствуют.