Генетика, 2020, T. 56, № 3, стр. 313-320
Дифференциация гибридного потомства белого (Hypophthalmichthys molitrix Val.) и пестрого (H. nobilis Rich.) толстолобиков на основании полиморфизма микросателлитных локусов
А. Ю. Носова 1, В. Н. Кипень 1, *, А. И. Царь 1, В. А. Лемеш 1, **
1 Институт генетики и цитологии Национальной академии наук Беларуси
220072 Минск, Республика Беларусь
* E-mail: v.kipen@igc.by
** E-mail: v.lemesh@igc.by
Поступила в редакцию 22.03.2019
После доработки 15.05.2019
Принята к публикации 16.07.2019
Аннотация
В работе дана оценка дифференцирующего потенциала 11 микросателлитных локусов – Hmo11, Hmo13, Hmo15, Hmo25, Hmo26, Hmo31, Hmo33, Hmo34, Hmo36, Hmo37, Hmo40 – для различения белого (Hypophthalmichthys molitrix Val.) и пестрого (H. nobilis Rich.) толстолобиков с использованием молекулярно-генетического анализа. Наибольшие рассчитанные значения FST показаны для STR-локусов Hmo15 (0.5235), Hmo33 (0.4957), Hmo40 (0.3500), Hmo25 (0.3365) и Hmo31 (0.2458). Точность классификации пестрых толстолобиков с использованием данных микросателлитных локусов составила 99.5 ± 0.3%, белых толстолобиков – 99.4 ± 0.8%. С использованием пяти STR-локусов предложена схема для выявления гибридных особей между пестрым и белым толстолобиками, выращиваемых в аквакультуре на территории Республики Беларусь. Данный подход может быть использован в качестве экспресс-теста в селекционных и воспроизводительных программах для данных видов растительноядных рыб.
В условиях коммерческой аквакультуры с довольно высокой частотой происходит скрещивание между белым (Hypophthalmichthys molitrix Val.) и пeстрым (H. nobilis Rich.) толстолобиками. Для повышения эффективности формирования ремонтных групп данных видов и в дальнейшем для отбора из них элитных низкоинбредных экземпляров в маточное стадо должны учитываться генетическая и фенотипическая изменчивость, а также физиологическое состояние. При формировании маточных стад растительноядных рыб необходимо использовать как минимум двухлинейное разведение – воспроизводство двух неродственных групп рыб с подбором самок и самцов различного происхождения. Это позволит избежать близкородственного скрещивания и рассчитывать на получение высокопродуктивного, быстрорастущего рыбопосадочного материала.
При сравнении диких и искусственных (заводских) популяций карпа из разных частей ареала было показано, что максимальные оценки генетической изменчивости характерны для природных аутбредных популяций. При искусственном воспроизводстве может наблюдаться снижение уровня полиморфизма за счет эффекта основателя и выраженной инбредной депрессии [1, 2]. Ранее показано, что одними из наиболее доступных и информативных показателей полиморфизма для природных популяций рыб являются оценки аллельного разнообразия и гетерозиготности [3]. Оценка генетического разнообразия для особей белого и пестрого толстолобиков с использованием STR-локусов была проведена в работах Gheyas et al., Feng et al., Wang et al. и др. [4–10].
В предыдущем нашем исследовании по данным генотипирования 11 STR-локусов (Hmo11, Hmo13, Hmo15, Hmo25, Hmo26, Hmo31, Hmo33, Hmo34, Hmo36, Hmo37, Hmo40) дана оценка генетического разнообразия производителей белого (БТ) и пестрого (ПТ) толстолобиков, разводимых в отделении “Белоозерское” ОАО “Опытный рыбхоз “Селец”” (агрогородок Селец, Брестская обл.) [11], который является единственным держателем маточных стад белого и пестрого толстолобиков на территории Республики Беларусь. Полученные результаты позволили сделать заключение о достаточно высоком генетическом разнообразии изученных выборок белого и пестрого толстолобиков, вплоть до возможности выделить несколько групп для последующей работы по получению линейного материала и дальнейшего воспроизводства. Однако открытым оставался вопрос о возможности идентификации среди изученных выборок производителей гибридных особей белого и пестрого толстолобиков с использованием молекулярно-генетического анализа.
Межвидовая гибридизация и последующая интрогрессия между белым и пестрым толстолобиками потенциально могут иметь негативные последствия для маточных и ремонтных стад, а также для эффективности их применения в аквакультуре. В то же время эффект гетерозиса для потомства белого и пестрого толстолобика изучен в недостаточной степени для использования его в коммерческой аквакультуре [12].
Ранее в ряде исследований была продемонстрирована возможность с использованием STR-локусов дифференцировать особи близкородственных видов [13–15] или даже проводить классификацию особей с высокой точностью в пределах подвидов [16]. По данным Mia et al. [17] аллельные вариации по ряду STR-локусов позволяют также различать белого и пестрого толстолобиков, сводя к минимуму ошибки идентификации по фенотипическим признакам. Однако полученные исследователями результаты в одном географическом регионе не могут быть прямо перенесены на другие выборки за пределами исследуемой территории по причине различия популяционных частот. В этой связи любая предлагаемая исследователями схема дифференциации близкородственных видов должна быть адаптирована под новую территориальную единицу.
Таким образом, цель настоящего исследования – оценить дифференцирующий потенциал 11 STR-локусов (Hmo11, Hmo13, Hmo15, Hmo25, Hmo26, Hmo31, Hmo33, Hmo34, Hmo36, Hmo37, Hmo40) для идентификации гибридных особей белого и пестрого толстолобиков.
МАТЕРИАЛЫ И МЕТОДЫ
Материалом для исследований служили образцы тканей от 129 производителей растительноядных рыб (ПТ – 63 особи, БТ – 66 особей), отобранных в отделении “Белоозерское” ОАО “Опытный рыбхоз “Селец””.
В качестве биологических проб для молекулярно-генетических исследований использовался биопсийный материал (плавники, мышечная ткань, кровь и др.). Выделение тотальной ДНК проводили с использованием коммерческих наборов DNA Purification Kit (ThermoFisherScientific®, США), ДНК-сорб-С (AmpliSens®, РФ), а также методик фенол-хлороформной и солевой экстракции [18] с модификациями.
Проведен анализ разнообразия аллелей по 11 STR-локусам – Hmo11, Hmo13, Hmo15, Hmo25, Hmo26, Hmo31, Hmo33, Hmo34, Hmo36, Hmo37, Hmo40 [11].
Статистический анализ данных проводили с использованием программ GenAIEx v.6.5 [19], STRUCTURE v.2.3.4 [20], PAST v.3.17 [21] и POPHELPER v.1.0.10 [22]. С использованием GenAlEx v.6.5 был проведен расчет генетических дистанций по методу AMOVA (Analysis of molecular variance) [23]. В программе STRUCTURE v.2.3.4 был рассчитан критерий Q, который характеризует принадлежность каждой отдельной особи к соответствующему кластеру (субгруппе в пределах группы). С использованием веб-приложения POPHELPER v.1.0.10 произведена графическая интерпретация результатов, полученных в STRUCTURE v.2.3.4. В программе PAST v.3.17 выполнено построение графика главных компонент на основе расчета генетических дистанций по методу AMOVA.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Анализ субпопуляционной структуры исследуемых групп толстолобиков с использованием программы STRUCTURE v.2.3.4 [20] по данным генотипирования 11 STR-локусов, а также наглядная демонстрация полученного результата, выражающаяся в отнесении особей ко всей выборке или конкретной группе и графически представленная в веб-приложении POPHELPER v.1.0.10 [22], приведены на рис. 1.
Рис. 1.
Результаты анализа генетической структуры исследуемых групп толстолобиков для наиболее вероятного числа кластеров (K) от 3 до 5. Ось Х (подпись вверху) – изучаемый вид; ось Y – параметр Q (значение K).

В результате проведенного моделирования (длительность Burn-in периода – 5000, количество MCMC [Markov chain Monte Carlo] повторов после Burn-in периода – 50 000) показано, что имеются два четко выраженных кластера (K = 2). В программе STRUCTURE v.2.3.4 по методу J.K. Pritchard для двух групп (“ПТ” и “БТ”) также был проведен расчет критерия Q, который характеризует принадлежность каждой отдельной особи к соответствующей группе (виду). Значение Q, равное 75% или выше, подтверждает членство особи в своем кластере. В табл. 1 представлены результаты анализа, проведенного в STRUCTURE v.2.3.4.
Таблица 1.
Точность отнесения особи к одной из двух групп – “ПТ” (пестрый толстолобик) или “БТ” (белый толстолобик)
| ID | Кластер | ID | Кластер | ID | Кластер | |||
|---|---|---|---|---|---|---|---|---|
| № 1 | № 2 | № 1 | № 2 | № 1 | № 2 | |||
| ПТ-01 | 99.56 | 0.44 | ПТ-44 | 99.80 | 0.20 | БТ-24 | 0.20 | 99.80 |
| ПТ-02 | 99.80 | 0.20 | ПТ-45 | 99.80 | 0.20 | БТ-25 | 0.20 | 99.80 |
| ПТ-03 | 99.80 | 0.20 | ПТ-46 | 43.02 | 56.98 | БТ-26 | 0.20 | 99.80 |
| ПТ-04 | 99.81 | 0.19 | ПТ-47 | 99.80 | 0.20 | БТ-27 | 0.20 | 99.80 |
| ПТ-05 | 99.80 | 0.20 | ПТ-48 | 46.87 | 53.13 | БТ-28 | 0.20 | 99.80 |
| ПТ-06 | 99.80 | 0.20 | ПТ-49 | 99.84 | 0.16 | БТ-29 | 0.20 | 99.80 |
| ПТ-07 | 99.80 | 0.20 | ПТ-50 | 99.80 | 0.20 | БТ-30 | 0.21 | 99.79 |
| ПТ-08 | 99.61 | 0.39 | ПТ-51 | 99.74 | 0.26 | БТ-31 | 0.20 | 99.80 |
| ПТ-09 | 99.62 | 0.38 | ПТ-52 | 99.81 | 0.19 | БТ-32 | 0.30 | 99.70 |
| ПТ-10 | 99.70 | 0.30 | ПТ-53 | 99.80 | 0.20 | БТ-33 | 0.20 | 99.80 |
| ПТ-11 | 99.78 | 0.22 | ПТ-54 | 99.71 | 0.29 | БТ-34 | 0.20 | 99.80 |
| ПТ-12 | 99.70 | 0.30 | ПТ-55 | 99.80 | 0.20 | БТ-35 | 0.20 | 99.80 |
| ПТ-13 | 99.79 | 0.21 | ПТ-56 | 99.80 | 0.20 | БТ-36 | 0.21 | 99.79 |
| ПТ-14 | 99.80 | 0.20 | ПТ-57 | 99.80 | 0.20 | БТ-37 | 0.20 | 99.80 |
| ПТ-15 | 99.70 | 0.30 | ПТ-58 | 99.80 | 0.20 | БТ-38 | 0.20 | 99.80 |
| ПТ-16 | 99.75 | 0.25 | ПТ-59 | 99.45 | 0.55 | БТ-39 | 0.20 | 99.80 |
| ПТ-17 | 99.80 | 0.20 | ПТ-60 | 0.20 | 99.80 | БТ-40 | 0.28 | 99.72 |
| ПТ-18 | 99.79 | 0.21 | ПТ-61 | 0.20 | 99.80 | БТ-41 | 0.20 | 99.80 |
| ПТ-19 | 99.80 | 0.20 | ПТ-62 | 0.20 | 99.80 | БТ-42 | 0.20 | 99.80 |
| ПТ-20 | 44.18 | 55.82 | ПТ-63 | 0.20 | 99.80 | БТ-43 | 0.20 | 99.80 |
| ПТ-21 | 99.86 | 0.14 | БТ-01 | 0.20 | 99.80 | БТ-44 | 0.20 | 99.80 |
| ПТ-22 | 99.82 | 0.18 | БТ-02 | 0.30 | 99.70 | БТ-45 | 0.20 | 99.80 |
| ПТ-23 | 99.80 | 0.20 | БТ-03 | 0.20 | 99.80 | БТ-46 | 0.29 | 99.71 |
| ПТ-24 | 99.80 | 0.20 | БТ-04 | 0.20 | 99.80 | БТ-47 | 0.20 | 99.80 |
| ПТ-25 | 99.80 | 0.20 | БТ-05 | 0.31 | 99.69 | БТ-48 | 0.81 | 99.19 |
| ПТ-26 | 99.85 | 0.15 | БТ-06 | 0.21 | 99.79 | БТ-49 | 1.90 | 98.10 |
| ПТ-27 | 99.79 | 0.21 | БТ-07 | 0.21 | 99.79 | БТ-50 | 0.20 | 99.80 |
| ПТ-28 | 99.83 | 0.17 | БТ-08 | 0.20 | 99.80 | БТ-51 | 0.30 | 99.70 |
| ПТ-29 | 99.76 | 0.24 | БТ-09 | 0.002 | 0.998 | БТ-52 | 0.20 | 99.80 |
| ПТ-30 | 99.83 | 0.17 | БТ-10 | 0.002 | 0.998 | БТ-53 | 0.20 | 99.80 |
| ПТ-31 | 99.81 | 0.19 | БТ-11 | 0.002 | 0.998 | БТ-54 | 0.20 | 99.80 |
| ПТ-32 | 99.81 | 0.19 | БТ-12 | 63.72 | 36.28 | БТ-55 | 0.20 | 99.80 |
| ПТ-33 | 99.80 | 0.20 | БТ-13 | 0.20 | 99.80 | БТ-56 | 0.21 | 99.79 |
| ПТ-34 | 99.80 | 0.20 | БТ-14 | 0.21 | 99.79 | БТ-57 | 0.20 | 99.80 |
| ПТ-35 | 99.80 | 0.20 | БТ-15 | 0.20 | 99.80 | БТ-58 | 0.20 | 99.80 |
| ПТ-36 | 99.80 | 0.20 | БТ-16 | 0.20 | 99.80 | БТ-59 | 0.20 | 99.80 |
| ПТ-37 | 99.80 | 0.20 | БТ-17 | 0.20 | 99.80 | БТ-60 | 0.20 | 99.80 |
| ПТ-38 | 99.80 | 0.20 | БТ-18 | 0.21 | 99.79 | БТ-61 | 0.20 | 99.80 |
| ПТ-39 | 99.85 | 0.15 | БТ-19 | 0.20 | 99.80 | БТ-62 | 0.20 | 99.80 |
| ПТ-40 | 99.70 | 0.30 | БТ-20 | 0.30 | 99.70 | БТ-63 | 0.20 | 99.80 |
| ПТ-41 | 99.79 | 0.21 | БТ-21 | 0.20 | 99.80 | БТ-64 | 0.30 | 99.70 |
| ПТ-42 | 99.80 | 0.20 | БТ-22 | 0.20 | 99.80 | БТ-65 | 0.20 | 99.80 |
| ПТ-43 | 99.70 | 0.30 | БТ-23 | 0.20 | 99.80 | БТ-66 | 0.20 | 99.80 |
Точность классификации подавляющего большинства особей в пределах группы “Пестрый толстолобик (ПТ)” (60 особей из 63, включенных в данный анализ) варьировала в пределах 99.8 ± ± 0.1%, аналогичный показатель для особей в пределах группы “Белый толстолобик (БТ)” (65/66) – 99.7 ± 0.3%. В первом кластере “ПТ” были выявлены три особи, вероятность отнесения которых к группе находилась в пределах 43.0–46.9%; во втором кластере “БТ” была выявлена одна особь, вероятность отнесения которой к группе составила 36.3%. Данные особи были дифференцированы как гибриды между ПТ и БТ. При значении K = 3 в группе “БТ” представляется возможным выделить уже два субкластера, для группы “ПТ” данное подразделение наблюдается только при K = 5. Данный факт еще раз подчеркивает полученные нами ранее данные [11] о более выраженном генетическом разнообразии производителей БТ в сравнении с ПТ.
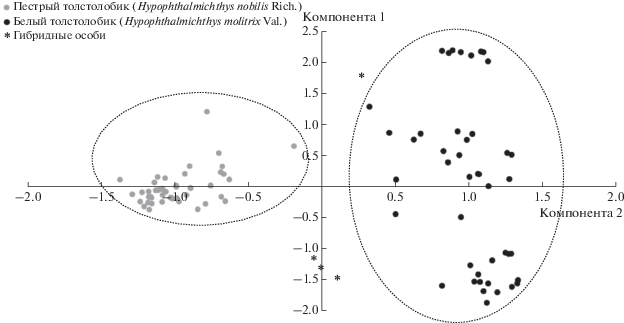
На основании анализа генетических дистанций, рассчитанных по алгоритму AMOVA, был построен график главных компонент (principal component analysis, PCA) – рис. 2. Особи в группе “ПТ” расположены очень компактно, в отличие от особей в группе “БТ”. Данные группы на графике разнесены друг относительно друга и образуют два неперекрывающихся массива, гибридные особи (звездочки) располагаютя между группами “ПТ” и “БТ”.
Из 11 STR-локусов, анализируемых в рамках данного исследования, наибольшим потенциалом для решения задачи по дифференциации белого и пестрого толстолобиков обладают те, для которых рассчитанные значения FST являются максимальными – табл. 2. При расчетах FST не учитывали те особи, которые были отнесены к гибридам между ПТ и БТ.
Таблица 2.
Дифференцирующий потенциал 11 STR-локусов (результаты анализа locus-by-locus AMOVA)
| STR | FST | p-уровень |
|---|---|---|
| Hmo11 | 0.2214 | <0.001 |
| Hmo13 | 0.1492 | <0.001 |
| Hmo15# | 0.5235 | <0.001 |
| Hmo25 | 0.3356 | <0.001 |
| Hmo26 | 0.1272 | 0.001 |
| Hmo31 | 0.2458 | <0.001 |
| Hmo33 | 0.4957 | <0.001 |
| Hmo34 | 0.2147 | 0.001 |
| Hmo36 | 0.2134 | 0.001 |
| Hmo37 | 0.1592 | 0.001 |
| Hmo40 | 0.3500 | <0.001 |
Набольшие рассчитанные значения FST показаны для STR-локусов Hmo15, Hmo33, Hmo40, Hmo25 и Hmo31. Информация об аллельном разнообразии и частоте распространенности конкретных аллелей для перечисленных STR-локусов представлена в табл. 3.
Таблица 3.
Частота распространенности аллелей среди производителей ПТ и БТ (исключая гибридных особей) для пяти STR-локусов с наибольшим дифференцирующим потенциалом (по результатам FST)
| Аллель | Hmo15 | Аллель | Hmo33 | Аллель | Hmo40 | Аллель | Hmo25 | Аллель | Hmo31 | |||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ПТ | БТ | ПТ | БТ | ПТ | БТ | ПТ | БТ | ПТ | БТ | |||||
| 139 | 0.537 | 0.012 | 76 | 0.034 | – | 207 | – | 0.023 | 142 | – | 0.012 | 131 | – | 0.107 |
| 141 | 0.463 | – | 82 | 0.932 | – | 210 | – | 0.581 | 146 | 0.011 | 0.105 | 133 | – | 0.036 |
| 146 | – | 0.826 | 86 | – | 0.012 | 212 | – | 0.012 | 148 | 0.011 | 0.419 | 135 | – | 0.060 |
| 148 | – | 0.070 | 88 | – | 0.085 | 215 | – | 0.140 | 150 | 0.057 | 0.326 | 139 | – | 0.381 |
| 153 | – | 0.023 | 90 | – | 0.073 | 217 | – | 0.233 | 152 | 0.034 | 0.023 | 141 | – | 0.071 |
| 158 | – | 0.047 | 92 | – | 0.268 | 220 | – | 0.012 | 154 | 0.659 | – | 143 | – | 0.119 |
| 160 | – | 0.023 | 95 | – | 0.171 | 237 | 0.024 | – | 156 | 0.057 | – | 145 | – | 0.119 |
| 97 | – | 0.012 | 246 | 0.415 | – | 159 | 0.125 | 0.023 | 147 | – | 0.083 | |||
| 100 | – | 0.183 | 248 | 0.463 | – | 192 | – | 0.023 | 152 | 0.012 | – | |||
| 103 | – | 0.049 | 250 | 0.098 | – | 195 | – | 0.012 | 154 | 0.037 | – | |||
| 107 | 0.011 | 0.037 | 208 | 0.023 | 0.023 | 156 | 0.024 | – | ||||||
| 109 | – | 0.012 | 210 | – | 0.012 | 159 | 0.085 | – | ||||||
| 115 | 0.011 | 0.085 | 214 | – | 0.023 | 161 | 0.573 | – | ||||||
| 118 | – | 0.012 | 246 | 0.023 | – | 163 | 0.220 | – | ||||||
| 132 | 0.011 | – | 165 | 0.012 | – | |||||||||
| 167 | 0.037 | – | ||||||||||||
| 207 | – | 0.012 | ||||||||||||
| 221 | – | 0.012 | ||||||||||||
В результате аллельные диапазоны для групп “ПТ” и “БТ” либо не пересекаются (например, для Hmo40), либо представленность мажорных аллелей сильно различается. В частности, для Hmo15 в группе “ПТ” мажорными аллелями были 139 и 141 (суммарная частота распространенности – 100%), для группы “БТ” мажорный аллель – 146 (встречаемость 82.6%). Для Hmo33 в группе “ПТ” мажорный аллель 82 распространен с частотой 93.2% и не представлен в группе “БТ”. Для STR-локуса Hmo40 в группе “ПТ” наиболее часто встречающимися являлись аллели 246 (41.5%) и 248 (46.3%), в то время как в группе “БТ” мажорный аллель – 210 (58.1%). Для локусов Hmo25 и Hmo31 наблюдается схожая тенденция. В локусе Hmo25 для группы “ПТ” наиболее распространенным являлся аллель 154 (65.9%), для “БТ” – 148 (41.9%) и 150 (32.6%). В локусе Hmo31 для группы “ПТ” наиболее распространен аллель 161 (57.3%), для “БТ” – 139 (38.1%).
На основании полученных данных был проведен повторный анализ субпопуляционной структуры исследуемых выборок толстолобиков с использованием программы STRUCTURE v.2.3.4 на основании результатов генотипирования только пяти из 11 STR-локусов. В результате точность классификации (отнесения к одной из двух групп) оказалась сопоставима с результатами, полученными на 11 STR-локусах. В частности, точность дифференциации ПТ с использованием STR-локусов Hmo15, Hmo33, Hmo40, Hmo25 и Hmo31 составила 99.5 ± 0.3%, БТ – 99.4 ± 0.8%. Также были выявлены и четыре гибридные особи: для ПТ-20 вероятность принадлежности к кластеру “ПТ” составила 48.8%, к кластеру “БТ” – 51.2%; для ПТ-46 – 50.8 и 49.2% соответственно; для ПТ-48 – 54.4 и 45.6%; БТ-12 – 64.6 и 35.4%.
Отдельно необходимо отметить, что в научной литературе представленность информации о частоте встречаемости гибридов между белым и пестрым толстолобиками в аквакультуре или естественных условиях весьма скудна. В исследовании Mia et al. [17] было показано, что среди особей белого толстолобика, выращиваемых в аквакультуре (коммерческие инкубаторы в различных регионах Бангладеш), процент гибридных особей варьировал в широких пределах – от 2.6% в Мименсингхе (Mymensingh) до 18.3% в Борге (Bogra) при среднем значении по стране 8.3% (35/422). Среди особей пестрого толстолобика частота распространенности гибридов была выше – от их полного отсутствия в Комиллe (Comilla) до 43.8% в Богре (Bogra) при среднем значении по стране 23.3% (55/236). В рамках проведенного нами настоящего исследования частота гибридов среди особей пестрого толстолобика в маточном стаде составила 4.8% (3/63), среди особей белого толстолобика в маточном стаде – 1.5% (1/66). В целом частота распространенности гибридов в маточных стадах белого и пестрого толстолобиков в Республике Беларусь незначительна. Возможные причины, лежащие в основе различий, заключаются, на наш взгляд, в технологиях выращивания и разведения особей в аквакультурах двух стран. Использование же предложенной схемы по дифференциации данных близкородственных видов в аквакультуре в Республике Беларусь должно свести данный показатель к минимуму или сделать его в перспективе гетерозисных исследований полностью контролируемым.
Для Беларуси растительноядные рыбы – белый (Hypophthalmichthys molitrix Val.) и пестрый (H. nobilis Rich.) толстолобики – интродуцированные виды. Их используют для зарыбления и мелиорации водоемов, но самоподдерживающиеся популяции не формируются из-за низких температур для начала нереста. Данные виды, будучи растительноядными, представляют исключительно большой интерес для повышения рыбопродуктивности как прудов, так и иных водоемов. Особенностью доместикации этих видов, отличающей их от традиционного объекта рыбоводства – карпа, является способ их воспроизводства. В прудовых условиях эти рыбы не размножаются, и их разведение основывается лишь на физиологическом методе стимуляции созревания с применением гормональных препаратов.
Известно, что в условиях коммерческой аквакультуры с довольно высокой частотой происходит скрещивание между белым и пестрым толстолобиками, а дальнейшая межвидовая гибридизация и последующая интрогрессия между этими видами могут иметь негативные последствия для маточных и ремонтных стад.
В данном исследовании была дана оценка дифференцирующего потенциала 11 STR-локусов для решения задачи по идентификации особей белого и пестрого толстолобиков, а также для выявления гибридных особей между данными видами.
Анализ субпопуляционной структуры исследуемых двух групп толстолобиков позволил сделать заключение, что на основании результатов генотипирования по 11 STR-локусам точность классификации особей пестрого толстолобика составляет 99.8 ± 0.1%, особей белого толстолобика – 99.7 ± 0.3%. Точность классификации сохраняется и при генотипирования только пяти STR-локусов – Hmo15, Hmo33, Hmo40, Hmo25 и Hmo31, дифференцирующий потенциал которых по данным FST был наибольшим и варьировал в пределах 0.2458–0.5235. Предложенная нами схема по дифференциации особей белого и пестрого толстолобиков с использованием пяти STR-локусов позволит также выявлять гибридные особи между данными видами растительноядных видов рыб, что может быть использовано в качестве экспресс-теста в селекционных и воспроизводительных программах.
На основании полученных результатов в лаборатории генетической и клеточной инженерии Института генетики и цитологии НАН Беларуси (Минск, Республика Беларусь) совместно с сотрудниками Института рыбного хозяйства (Минск, Республика Беларусь) разработана и используется технология генетической идентификации растительноядных рыб (белого толстолобика, пестрого толстолобика и их гибридов), которая позволит повысить продуктивность за счет реализации эффективных селекционных приемов, а также за счет контроля формирования межвидовых гибридов.
Работа выполнена в рамках мероприятия 21ИБ “Разработать и внедрить технологию генетической идентификации растительноядных и лососевых видов рыб” подпрограммы 1 “Инновационные биотехнологии – 2020” ГП “Наукоемкие технологии и техника” на 2016–2020 гг., Республика Беларусь.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Агеец В.Ю. Перспективы развития рыбоводства в Беларуси // Весцi Нацыянальнай акадэмii навук Беларусi. Сер. аграрных навук. 2013. № 2. С. 102–109.
Агеец В.Ю. Экологические проблемы рыбоводства в Республике Беларусь и пути их решения // Весцi Нацыянальнай акадэмii навук Беларусi. Сер. аграрных навук. 2015. № 2. С. 95–101.
Chistiakov D.A., Hellemans B., Volckaert F.A.M. Microsatellites and their genomic distribution, evolution, function and applications: A review with special reference to fish genetics // Aquaculture. 2006. V. 255. № 1–4. P. 1–29. https://doi.org/10.1016/j.aquaculture.2005.11.031
Gheyas A.A., Cairney M., Gilmour A.E. et al. Characterization of microsatellite loci in silver carp (Hypophthalmichthys molitrix), and cross-amplification in other cyprinid species // Mol. Ecol. Notes. 2006. V. 6. № 3. P. 656–659. https://doi.org/10.1111/j.1471-8286.2006.01288.x
Feng X., Yu X., Fu B. et al. Development of 159 transcript-associated microsatellite markers in silver carp (Hypophthalmichthys molitrix) // Conserv. Genet. Res. 2014. V. 6. № 1. P. 111–113. https://doi.org/10.1007/s12686-013-0017-3
Wang C.Z., Liang H.W., Zou G.W. et al. Genetic variation analysis of two silver carp populations in the middle and upper Yangtze River by microsatellite // Yi Chuan. 2008. V. 30. № 10. P. 1341–1348. https://doi.org/10.3724/SP.J.1005.2008.01341
Liao M., Yang G., Wang X. et al. Development of microsatellite DNA markers of silver carp (Hypophthalmichthys molitrix) and their cross-species application in bighead carp (Aristichthys nobilis) // Mol. Ecol. Notes. 2007. V. 7. № 1. P. 95–99. https://doi.org/10.1111/j.1471-8286.2006.01542.x
Guo W., Xiaomu Yu, Tong J. et al. Development of 134 novel polynucleotide-repeat microsatellite markers in silver carp (Hypophthalmichthys molitrix) // Conserv. Genet. Res. 2012. V. 5. № 2. P. 525–528. https://doi.org/10.1007/s12686-012-9843-y
Cheng L., Liu L., Yu X. et al. Sixteen polymorphic microsatellites in bighead carp (Aristichthys nobilis) and cross-amplification in silver carp (Hypophthalmichthys molitrix) // Mol. Ecol. Res. 2008. V. 8. № 3. P. 656–658. https://doi.org/10.1111/j.1471-8286.2007.02037.x
Nazish N., Abbas K., Abdullah S. et al. Microsatellite diversity and population structure of Hypophthalmicthys molitrix in hatchery populations of Punjab // Turkish J. Fisheries and Aquatic Sci. 2018. V. 18. P. 1113–1122. https://doi.org/10.4194/1303-2712-v18_9_10
Носова А.Ю., Кипень В.Н., Царь А.И. и др. Полиморфизм микросателлитных локусов у белого (Hypophthalmichthys molitrix Val.) и пестрого (Hypophthalmichthys nobilis Rich.) толстолобиков, выращиваемых в аквакультуре в Республике Беларусь // Докл. НАН Беларуси. 2019. Т. 63. № 1. С. 79–86.
Wang J., Yang G., Zhou G. Quantitative trait loci for morphometric body measurements of the hybrids of silver carp (Hypophthalmichthys molitrix) and bighead carp (H. nobilis) // Acta Biol. Hungarica. 2013. V. 64. № 2. P. 169–183. https://doi.org/10.1556/ABiol.64.2013.2.4
Li Y., Ludwig A., Pengcorresponding Z. Geographical differentiation of the Euchiloglanis fish complex (Teleostei: Siluriformes) in the Hengduan Mountain Region, China: Phylogeographic evidence of altered drainage patterns // Ecol. Evol. 2017. V. 7. № 3. P. 928–940. https://doi.org/10.1002/ece3.2715
Christopher G. Genetic variation and differentiation in African catfish, Clarias gariepinus, assessed by heterologous microsatellite DNA // Ind. J. Biotechnology. 2015. V. 14. № 3. P. 388–393. https://doi.org/10.1002/ece3.2715
Deniskova T.E., Sermyagin A.A., Bagirov V.A. et al. Comparative analysis of the effectiveness of STR and SNP markers for intraspecific and interspecific differentiation of the genus ovis // Genetika. 2016. V. 52. № 1. P. 90–96. https://doi.org/10.1134/S1022795416010026
Conyers C.M., Allnutt T.R., Hird H.J. et al. Development of a microsatellite-based method for the differentiation of European wild boar (Sus scrofa scrofa) from domestic pig breeds (Sus scrofa domestica) in food // J. Agricultural and Food Chemistry. 2012. V. 60. № 13. P. 3341–3347.https://doi.org/10.1021/jf205109b
Mia M.Y., Taggart J.B., Gilmour A.E. et al. Detection of hybridization between Chinese carp species (Hypophthalmichthys molitrix and Aristichthys nobilis) in hatchery broodstock in Bangladesh, using DNA microsatellite loci // Aquaculture. 2005. V. 247. № 1–4. P. 267–273. https://doi.org/10.1016/j.aquaculture.2005.02.018
Sambrook J., Russell D.W. Molecular Cloning: A Laboratory Manual, 3rd ed. N.Y.: Cold Spring Harbor Lab. Press, 2001.
Peakall R., Smouse P.E. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research – an update // Bioinformatics. 2012. V. 28. № 19. P. 2537–2539. https://doi.org/10.1093/bioinformatics/bts460
Pritchard J.K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data // Genetics. 2000. V. 155. № 2. P. 945–959.
Hammer Ø., Harper D.A.T., Ryan P.D. Past: Paleontological statistics software package for education and data analysis // Palaeontologia Electronica. 2001. V. 4. № 1. P. 1–9.
Francis R.M. Pophelper: an R package and web app to analyse and visualize population structure // Mol. Ecol. Resour. 2017. V. 17. № 1. P. 27–32. https://doi.org/10.1111/1755-0998.12509
Excoffier L., Smouse P.E., Quattro J.M. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data // Genetics. 1992. V. 131. № 2. P. 479–491.
Дополнительные материалы отсутствуют.