Генетика, 2020, T. 56, № 4, стр. 421-432
Молекулярно-филогенетическая характеристика эндемичных дальневосточных близкородственных видов секции Orobia рода Oxytropis (Fabaceae)
М. М. Козыренко 1, А. Б. Холина 1, *, Е. В. Артюкова 1, М. Н. Колдаева 2, В. В. Якубов 1, С. В. Прокопенко 1
1 Федеральный научный центр биоразнообразия наземной биоты Восточной Азии Дальневосточного отделения Российской академии наук
690022 Владивосток, Россия
2 Ботанический сад-институт Дальневосточного отделения Российской академии наук
690024 Владивосток, Россия
* E-mail: kholina@biosoil.ru
Поступила в редакцию 22.04.2019
После доработки 30.05.2019
Принята к публикации 24.06.2019
Аннотация
До сих пор остаются нерешенными вопросы о таксономическом статусе и филогенетических связях дальневосточных близкородственных видов Oxytropis ochotensis, O. litoralis, O. erecta, O. ruthenica и O. kunashiriensis секции Orobia рода Oxytropis. Исследование полиморфизма нуклеотидных последовательностей межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG хпДНК показало, что популяции O. ochotensis и O. erecta характеризуются низким (0.378–0.495) гаплотипическим (h) и низким (0.0006–0.0009) нуклеотидным (π) разнообразием, а в популяциях O. ruthenica h варьирует от 0.154 до 0.872, π – от 0.0002 до 0.0016. Одна популяция O. ochotensis из Магаданской области и одна популяция O. ruthenica с о. Русский (Приморский край) оказались мономорфными. Низкая нуклеотидная дивергенция хпДНК между видами O. ochotensis, O. erecta и O. litoralis, а также статистически незначимая генетическая дифференциация между ними, формирование одной гаплогруппы в генеалогической сети и отсутствие видовых молекулярных маркеров указывают на единство генофонда. Исследование полиморфизма ITS рДНК выявило индивидуальные риботипы у O. ruthenica и O. kunashiriensis, наличие общего риботипа у O. ochotensis, O. erecta и O. litoralis и внутривидовой полиморфизм у O. ochotensis и O. erecta. Установленные различия в хлоропластном и ядерном геномах подтверждают самостоятельность видов O. ruthenica и O. kunashiriensis и позволяют предположить, что O. erecta и O. litoralis представляют собой локальные фенотипы широко распространенного полиплоидного вида O. ochotensis. Анализ генеалогических связей гаплотипов хпДНК показал четкое разделение популяций O. ruthenica на две эволюционные линии, но с одним ITS риботипом.
Ключевым этапом при изучении биоразнообразия является определение границ видов в эволюционно молодых группах растений и установление их родства. Отсутствие подобной информации может привести к ошибочной выборке таксонов, некорректной оценке биоразнообразия и другим негативным последствиям, поэтому разграничение видов внутри близкородственных групп весьма актуально для систематики и эволюционной биологии [1, 2]. До сих пор остаются нерешенными вопросы о таксономическом статусе и филогенетических связях дальневосточных близкородственных эндемичных видов Oxytropis ochotensis Bunge, O. litoralis Kom., O. erecta Kom., O. ruthenica Vass. и O. kunashiriensis Kitam. секции Orobia Bunge подрода Oxytropis крупного полиморфного рода Oxytropis DC. (Fabaceae). Названия видов, секций и подродов приведены по Н.С. Павловой [3]. O. ochotensis – восточносибирский метаарктический горный вид, эндемик северо-востока Азии, распространен в дальневосточном секторе Арктики (низовья Колымы, Анюйское и Чукотское нагорья) и за ее пределами (хр. Сунтар-Хаята, Нерское нагорье, хр. Черского (восток), п-ов Камчатка и Охотское побережье) [3–6]. Виды O. litoralis и O. erecta являются эндемами п-ова Камчатка [3, 5]. Таким образом, ареалы трех видов – O. ochotensis, O. litoralis и O. erecta перекрываются в восточной части п-ова Камчатка. O. ruthenica – эндемик Уссурийского флористического района Дальнего Востока России [3, 7]. Существуют противоречивые мнения о таксономическом статусе O. erecta, O. litoralis и O. ruthenica. Так, В.Н. Ворошилов [8] считал O. litoralis синонимом O. erecta; В.В. Якубов и О.А. Чернягина [5] предложили рассматривать O. erecta и O. litoralis как подвиды O. ochotensis. По мнению Н.С. Павловой [3], O. erecta, O. litoralis и O. ochotensis это самостоятельные виды. Позднее Л.И. Малышев [6, 9, 10] обнаружил фенетическую близость O. litoralis и O. ruthenica и понизил ранг последнего до подвида – O. litoralis subsp. ruthenica (Vass.) Malyshev. O. kunashiriensis – эндемик о. Кунашир Курильского архипелага [3, 10, 11]. Н.С. Павлова отмечала [3], что по морфологии O. kunashiriensis близок к O. erecta и O. litoralis, а Л.И. Малышев [9] – что самостоятельность вида O. kunashiriensis сомнительна.
Причиной такой таксономической неопределенности является отсутствие надежных морфологических диагностических признаков, что связано с недавним видообразованием, высоким уровнем межвидовой гибридизации и полиплоидии среди представителей Oxytropis. Так, у O. ochotensis в зависимости от места обитания обнаружены две вариации цитотипов: 2n = 64 в популяциях из низовьев Колымы, Анюйского нагорья и Северо-Восточной Якутии [12]; 2n = 48 в резко обособленной популяции в нижнем течении р. Алярмагтын на Чукотском нагорье [4], число хромосом O. erecta 2n = 48 [12], О. ruthenica 2n = 16 [13]. Число хромосом O. litoralis и O. kunashiriensis на данный момент неизвестно.
В современных генетических исследованиях растений успешно используют маркеры ядерного и хлоропластного геномов. К таковым относятся полные или частичные последовательности ITS региона (ITS1–5.8S рРНК–ITS2) рибосомного оперона ядерной ДНК (рДНК) и некодирующие регионы (быстро эволюционирующие интроны и межгенные спейсеры) хлоропластной ДНК (хпДНК), комбинированное использование которых особенно эффективно на уровне близкородственных видов. Так, по данным секвенирования маркеров двух геномов были уточнены эволюционные отношения близкородственных видов Paspalum (Poaceae) с различными цитотипами [14], 30 видов древесных покрытосеменных растений из Южной Бразилии [15], проведена оценка состояния природных популяций ряда редких и эндемичных видов рода Oxytropis DC., уточнена секционная принадлежность и таксономический статус некоторых видов, а также реконструированы филогенетические взаимоотношения в некоторых группах рода [16–25].
Настоящая работа посвящена изучению генетического разнообразия, популяционной структуры и оценке степени дивергенции хлоропластного генома дальневосточных близкородственных видов O. ochotensis, O. erecta, O. litoralis, О. ruthenica и O. kunashiriensis с целью уточнения таксономического статуса четырех последних по данным полиморфизма нуклеотидных последовательностей межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG хпДНК и ITS рДНК.
МАТЕРИАЛЫ И МЕТОДЫ
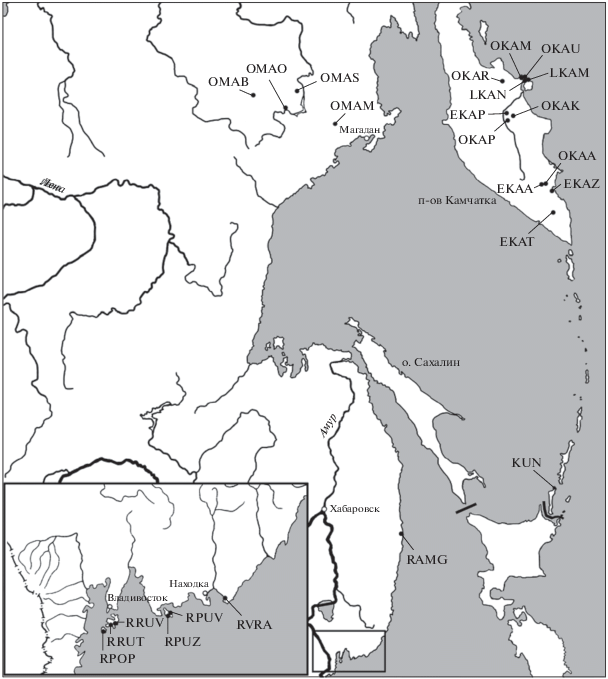
Материалом служили 177 растений из природных популяций O. ochotensis (55 образцов), O. erecta (19), O. litoralis (2), O. ruthenica (99), O. kunashiriensis (2) (табл. 1, рис. 1).
Таблица 1.
Исследуемые популяции видов Oxytropis и параметры генетического разнообразия по данным хпДНК
| Местонахождение (число образцов) |
Код | Разнообразие (стандартное отклонение) |
Гаплотип (число образцов) |
|
|---|---|---|---|---|
| гаплотипическое | нуклеотидное | |||
| O. ochotensis | ||||
| Камчатка, вулк. Ключевская сопка, склон (10) | OKAК | 0.378 (0.181) | 0.0009 (0.0006) | H1 (8) H2 (1) H3 (1) |
| Камчатка, вулк. сопка Плоская, юго-вост. макросклон (13) | OKAP | 0.385 (0.132) | 0.0006 (0.0005) | H1 (10) H2 (3) |
| Камчатка, вулк. Авачинская сопка, южный макросклон (14) | OKAA | 0.495 (0.151) | 0.0008 (0.0006) | H4 (10) H5 (2) H6 (1) H7 (1) |
| Камчатка, окр. г. Усть-Камчатск, склон горы Увальная (1) | OKAU | – | – | H8 (1) |
| Камчатка, среднее течение р. Радуга (1) | OKAR | – | – | H9 (1) |
| Камчатка, горы Камчатского мыса, истоки р. Угловая (1) | OKAM | – | – | H10 (1) |
| Магаданская обл., окр. пос. Оротук (10) | OMAO | 0.000 (0.000) | 0.0000 (0.0000) | H11 (10) |
| Магаданская обл., окр. пос. Буркандья (3) | OMAB | – | – | H12 (2) H13 (1) |
| Магаданская обл., окр. пос. Штурмовой (1) | OMAS | – | – | H14 (1) |
| Магаданская обл., окр. пос. Мадаун, гора Лебединая, склон (1) | OMAM | – | – | H15 (1) |
| O. erecta | ||||
| Камчатка, Авачинский залив, береговые песчаные валы (16) | EKAZ | 0.425 (0.133) | 0.0009 (0.0006) | H16 (3) H17 (12) H18 (1) |
| Камчатка, вулк. сопка Плоская, склон (1) | EKAP | – | – | H19 (1) |
| Камчатка, вулк. Авачинская сопка, окр. г. Петропавловск-Камчатский (1) | EKAA | – | – | H17 (1) |
| Камчатка, оз. Толмачева, береговая терраса (1) | EKAT | – | – | H20 (1) |
| O. litoralis | ||||
| Камчатка, юго-западная часть, морская терраса (1) | LKAM | – | – | H10 (1) |
| Камчатка, окр. Крутоберегово, оз. Нерпичье, береговая терраса (1) | LKAN | – | – | H10 (1) |
| O. ruthenica | ||||
| Приморский край, о. Русский, мыс Тобизина, прибрежно-морские скалы (17) | RRUT | 0.000 (0.000) | 0.0000 (0.0000) | H21 (17) |
| Приморский край, о. Русский, мыс Вятлина, морская терраса (17) | RRUV | 0.228 (0.129) | 0.0002 (0.0002) | H21 (15) H22 (1) H23 (1) |
| Приморский край, о. Попова, бух. Пограничная, морская терраса (14) | RPOP | 0.363 (0.130) | 0.0002 (0.0002) | H23 (11) H24 (3) |
| Приморский край, о. Путятина, западный берег, прибрежно-морские скалы (19) | RPUZ | 0.637 (0.104) | 0.0011 (0.0007) | H25 (1) H26 (2) H27 (11) H28 (1) H29 (4) |
| Приморский край, о. Путятина, восточный берег, прибрежно-морские скалы (13) | RPUV | 0.154 (0.126) | 0.0004 (0.0003) | H27 (12) H30 (1) |
| Приморский край, окр. пос. Амгу, прибрежно-морские скалы (6) | RAMG | 0.600 (0.129) | 0.0002 (0.0002) | H31 (3) H32 (3) |
| Приморский край, окр. пос. Врангель, мыс Каменского, склон у моря (13) | RVRA | 0.872 (0.067) | 0.0016 (0.0010) | H32 (1) H33 (1) H34 (2) H35 (4) H36 (3) H37 (1) H38 (1) |
| O. kunashiriensis | ||||
| Курильские о-ва, о. Кунашир, мыс Ловцова (2) | KUN | – | – | H39 (1) |
Рис. 1.
Карта-схема с указанием мест сбора растений Oxytropis ochotensis, O. erecta, O. litoralis, O. ruthenica и O. kunashiriensis. Код популяции см. табл. 1.
Методы выделения ДНК, амплификации и секвенирования межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG приведены в наших предыдущих работах [22, 24–26]. Нуклеотидные последовательности прямых и обратных цепей определяли на генетическом анализаторе ABI 3500 (Applied Biosystems, США), затем редактировали и собирали с помощью пакета программ Staden Package v. 1.5 [27]. Для каждого образца последовательности регионов выравнивали вручную в программе SeaView v. 4.7 [28]. Матрицу объединенных последовательностей использовали для расчета числа гаплотипов и частот встречаемости их в популяциях, гаплотипического (h) и нуклеотидного (π) разнообразия (для популяций с числом образцов пять и более), уровней дифференциации и распределения генетической изменчивости внутри и между популяциями и/или группами популяций (анализ молекулярной дисперсии, AMOVA) в пакете программ Arlequin v. 3.5 [29]. Статистическую значимость (P) индексов фиксации (ΦST) оценивали на основе 1023 пермутаций. Генный поток (Nm) и степень дивергенции между популяциями/видами (Dxy) на основе нуклеотидных замен определяли в программе DnaSP v. 5.0 [30]. Генеалогические связи гаплотипов хпДНК анализировали методом медианного связывания (Median-Joining, MJ) в программе Network v. 5.0.1.1 [31], кодируя каждую делецию или вставку, независимо от их размера, как единичное мутационное событие. В качестве внешней группы использовали полученные нами ранее [24] нуклеотидные последовательности межгенных спейсеров psbA–trnH, trnL–trnF, trnS–trnG хпДНК (номера доступа в GenBank LT856572, LT856585, LT856598 соответственно) O. glabra секции Mesogaea Bunge подрода Phacoxytropis Bunge.
Регион ITS рДНК амплифицирован у 62 образцов: O. ochotensis (20 образцов), O. erecta (13), O. litoralis (2), O. ruthenica (25) и O. kunashiriensis (2), представляющих все выявленные в данной работе гаплотипы хпДНК, с праймерами ITS1 и ITS4, в реакционных условиях и температурном режиме, приведенных в работе [32]. Редактирование, сборку, выравнивание и анализ последовательностей ITS проводили с помощью программ, приведенных выше. Филогенетический анализ последовательностей проводили методом максимальной экономии (Maximum Parsimony, MP), используя эвристический поиск оптимальной топологии, в пакете программ PAUP v. 4.0b10 [33]. Статистическую достоверность порядка ветвления оценивали с помощью бутстреп-анализа 1000 альтернативных деревьев (Bootstrap Percentage, BP, %).
Работа проводилась с использованием оборудования ЦКП “Биотехнология и генетическая инженерия” ФНЦ Биоразнообразия ДВО РАН.
РЕЗУЛЬТАТЫ
Нуклеотидные последовательности каждого из регионов psbA–trnH, trnL–trnF и trnS–trnG хпДНК у 177 образцов O. ochotensis, O. erecta, O. litoralis, O. ruthenica и O. kunashiriensis характеризуются относительно низкой нуклеотидной изменчивостью и разной длиной вследствие присутствия инсерций/делеций (инделей), моно- и динуклеотидных повторов. Длина объединенной матрицы трех регионов после выравнивания составила 2476 сайтов (1–484, 485–1270 и 1271–2476 соответственно), из них 2359 мономорфных и 14 вариабельных. 12 нуклеотидных замен в позициях 240, 281, 414, 1269, 1270, 1313, 1557, 1631, 1804, 1848, 2244 и 2300 информативны согласно методу максимальной экономии. Анализ полученной матрицы выявил 39 гаплотипов (H1–H39), из них 18 (46.1%) уникальные (табл. 1). Последовательности трех регионов хпДНК депонированы в GenBank под номерами доступа MK806162–MK806278. O. ochotensis принадлежит 15 гаплотипов (H1–H15), O. erecta – 5 (H16–H20), O. litoralis – 1 (H10), O. ruthenica – 18 (H21–H38), O. kunashiriensis – 1 (H39). Общий гаплотип (H10) обнаружен у O. ochotensis и O. litoralis (табл. 1).
Популяции O. ochotensis и O. erecta характеризуются низким (0.378–0.495) гаплотипическим и низким (0.0006–0.0009) нуклеотидным разнообразием, а в популяциях O. ruthenica h варьирует от 0.154 до 0.872, π – от 0.0002 до 0.0016 (табл. 1). Популяции OMAO O. ochotensis и RRUT O. ruthenica оказались мономорфными (табл. 1). Одним из показателей степени генетической разобщенности между популяциями является дивергенция нуклеотидных последовательностей (Dxy). У O. ochotensis наибольшие значения Dxy определены между камчатскими популяциями OKAK, OKAP и магаданской популяцией OMAB, а также между магаданскими популяциями OMAB и OMAM (табл. 2). Нуклеотидная дивергенция между популяциями O. erecta и O. litoralis, как и между популяциями каждого из видов, отсутствует или очень низкая (табл. 2). Необходимо отметить, что значения Dxy между большинством популяций O. ochotensis, O. erecta и O. litoralis низкие и соответствуют внутривидовому уровню O. ochotensis. У O. ruthenica популяции RRUT, RRUV, RPOP с о-вов Русский и Попова значительно дивергированы от всех других (табл. 3). На высокую степень дифференциации популяций видов указывают и результаты анализа молекулярной дисперсии. Согласно AMOVA (табл. 4), у O. ochotensis, O. ruthenica и O. erecta основная доля всей генетической изменчивости приходится на межпопуляционные различия (ΦST = 0.8703, P < 0.0001; ΦST = 0.8898, P < 0.0001; ΦST = 0.8215, P > 0.05 соответственно). Значение индекса фиксации у O. erecta статистически незначимое, что связано с малым размером трех из четырех исследуемых выборок. Генный поток (Nm) между популяциями у O. ochotensis и у O. ruthenica составил 0.11 и 0.09 мигрантов на поколение соответственно. Иерархический AMOVA показал, что статистически значимых генетических различий между O. erecta и O. litoralis, а также между O. ochotensis, O. erecta и O. litoralis нет (табл. 4). Дивергенция нуклеотидных последовательностей между парами O. ochotensis–O. erecta, O. ochotensis–O. litoralis и O. erecta–O. litoralis крайне низка (табл. 5) и соответствует межпопуляционному уровню O. ochotensis и O. ruthenica (табл. 2, 3). Наибольшие значения Dxy определены между O. kunashiriensis и всеми другими видами (табл. 5). По результатам AMOVA 35.2% изменчивости обусловлено различиями между видами, а на меж- и внутрипопуляционную составляющие приходится 56.9 и 7.9% соответственно (табл. 4).
Таблица 2.
Нуклеотидная дивергенция между популяциями Oxytropis ochotensis, O. erecta и O. litoralis по данным хпДНК
| Код | OKAK | OKAP | OKAA | OKAU | OKAR | OKAM | OMAO | OMAB | OMAS | OMAM | EKAZ | EKAP | EKAA | EKAT | LKAM | LKAN |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| OKAK | – | 0.00 (0) | 1.43 (0) | 3.00 (3) | 3.00 (3) | 1.00 (1) | 3.00 (3) | 4.00 (4) | 1.00 (1) | 0.00 (0) | 0.75 (0) | 1.00 (1) | 1.00 (1) | 1.00 (1) | 1.00 (1) | 1.00 (1) |
| OKAP | 0.000 | – | 1.43 (0) | 3.00 (3) | 3.00 (3) | 1.00 (1) | 3.00 (0) | 4.00 (4) | 1.00 (1) | 0.00 (0) | 0.75 (0) | 1.00 (1) | 1.00 (1) | 1.00 (1) | 1.00 (1) | 1.00 (1) |
| OKAA | 0.060 | 0.060 | – | 1.57 (1) | 1.57 (1) | 2.43 (1) | 1.57 (1) | 2.57 (2) | 2.43 (1) | 1.43 (0) | 2.18 (0) | 2.43 (1) | 2.43 (1) | 2.43 (1) | 2.43 (1) | 2.43 (1) |
| OKAU | 0.125 | 0.125 | 0.066 | – | 0.00 (0) | 2.00 (2) | 0.00 (0) | 1.00 (1) | 2.00 (2) | 3.00 (3) | 2.25 (2) | 2.00 (2) | 2.00 (2) | 2.00 (2) | 2.00 (2) | 2.00 (2) |
| OKAR | 0.125 | 0.125 | 0.066 | 0.000 | – | 2.00 (2) | 0.00 (0) | 1.00 (1) | 2.00 (2) | 3.00 (3) | 2.25 (2) | 2.00 (2) | 2.00 (2) | 2.00 (2) | 2.00 (2) | 2.00 (2) |
| OKAM | 0.042 | 0.042 | 0.101 | 0.083 | 0.083 | – | 2.00 (2) | 3.00 (3) | 0.00 (0) | 1.00 (1) | 0.25 (0) | 0.00 (0) | 0.00 (0) | 0.00 (0) | 0.00 (0) | 0.00 (0) |
| OMAO | 0.125 | 0.125 | 0.066 | 0.000 | 0.000 | 0.083 | – | 1.00 (0) | 2.00 (2) | 3.00 (3) | 2.25 (2) | 2.00 (2) | 2.00 (2) | 2.00 (2) | 2.00 (2) | 2.00 (2) |
| OMAB | 0.167 | 0.167 | 0.107 | 0.042 | 0.042 | 0.125 | 0.042 | – | 3.00 (3) | 4.00 (4) | 3.25 (3) | 3.00 (3) | 3.00 (3) | 3.00 (3) | 3.00 (3) | 3.00 (3) |
| OMAS | 0.042 | 0.042 | 0.101 | 0.083 | 0.083 | 0.000 | 0.083 | 0.125 | – | 1.00 (1) | 0.25 (0) | 0.00 (0) | 0.00 (0) | 0.00 (0) | 0.00 (0) | 0.00 (0) |
| OMAM | 0.000 | 0.000 | 0.060 | 0.125 | 0.125 | 0.042 | 0.124 | 0.167 | 0.041 | – | 1.00 (1) | 1.00 (1) | 1.00 (1) | 1.00 (1) | 1.00 (1) | |
| EKAZ | 0.031 | 0.031 | 0.091 | 0.094 | 0.094 | 0.010 | 0.094 | 0.136 | 0.010 | 0.031 | – | 0.25 (0) | 0.25 (0) | 0.25 (0) | 0.25 (0) | 0.25 (0) |
| EKAP | 0.042 | 0.042 | 0.101 | 0.083 | 0.083 | 0.000 | 0.083 | 0.125 | 0.000 | 0.042 | 0.010 | – | 0.00 (0) | 0.00 (0) | 0.00 (0) | 0.00 (0) |
| EKAA | 0.042 | 0.042 | 0.101 | 0.083 | 0.083 | 0.000 | 0.083 | 0.125 | 0.000 | 0.042 | 0.010 | 0.000 | – | 0.00 (0) | 0.00 (0) | 0.00 (0) |
| EKAT | 0.042 | 0.042 | 0.101 | 0.083 | 0.083 | 0.000 | 0.083 | 0.125 | 0.000 | 0.042 | 0.010 | 0.000 | 0.000 | – | 0.00 (0) | 0.00 (0) |
| LKAM | 0.042 | 0.042 | 0.101 | 0.083 | 0.083 | 0.000 | 0.083 | 0.125 | 0.000 | 0.042 | 0.010 | 0.000 | 0.000 | 0.000 | – | 0.00 (0) |
| LKAN | 0.042 | 0.042 | 0.101 | 0.083 | 0.083 | 0.000 | 0.083 | 0.125 | 0.000 | 0.042 | 0.010 | 0.000 | 0.000 | 0.000 | 0.000 | – |
Примечание. Выше диагонали – среднее число нуклеотидных различий (число фиксированных различий), ниже диагонали – среднее число нуклеотидных замен на один сайт ×10–2. Код популяции см. табл. 1.
Таблица 3.
Нуклеотидная дивергенция между популяциями Oxytropis ruthenica по данным хпДНК
| Код популяции |
RRUT | RRUV | RPOP | RPUZ | RPUV | RAMG | RVRA |
|---|---|---|---|---|---|---|---|
| RRUT | – | 0.294 (0) | 2.000 (2) | 2.895 (2) | 2.923 (2) | 2.500 (2) | 2.000 (2) |
| RRUV | 0.012 | – | 1.824 (0) | 3.189 (2) | 3.217 (2) | 2.794 (2) | 2.294 (2) |
| RPOP | 0.083 | 0.076 | – | 4.895 (4) | 4.923 (4) | 4.500 (4) | 4.000 (4) |
| RPUZ | 0.121 | 0.133 | 0.205 | – | 0.263 (0) | 1.395 (0) | 0.895 (0) |
| RPUV | 0.122 | 0.135 | 0.206 | 0.011 | – | 1.423 (0) | 0.923 (0) |
| RAMG | 0.104 | 0.117 | 0.188 | 0.058 | 0.059 | – | 0.500 (0) |
| RVRA | 0.084 | 0.096 | 0.167 | 0.037 | 0.039 | 0.021 | – |
Примечание. Выше диагонали – среднее число нуклеотидных различий (число фиксированных различий), ниже диагонали – среднее число нуклеотидных замен на один сайт ×10–2. Код популяции см. табл. 1.
Таблица 4.
Распределение генетической изменчивости (AMOVA) между группами Oxytropis по данным хпДНК
| Источник дисперсии | Генетические различия (%) между | ||
|---|---|---|---|
| группами | популяциями внутри групп | особями в популяции | |
| Популяции видов Oxytropis | |||
| Одна группа: (все популяции O. ochotensis) | – | 87.03* | 12.97 |
| Одна группа: (все популяции O. erecta) | – | 82.15 ns | 17.85 |
| Одна группа: (все популяции O. ruthenica) | – | 88.98* | 11.02 |
| Две группы: (все популяции O. erecta) и (популяция O. litoralis) | –69.53 ns | 133.63 ns | 35.90** |
| Две группы: (популяция O. litoralis) и (все популяции O. ruthenica) | 42.54 ns | 51.06* | 6.41* |
| Три группы: (все популяции O. ochotensis), (все популяции O. erecta) и (популяция O. litoralis) | 8.73 ns | 78.11* | 13.16* |
| Пять групп: (все популяции O. ochotensis), (все популяции O. erecta), (популяция O. litoralis), (все популяции O. ruthenica) и (O. kunashiriensis) | 35.20** | 56.90* | 7.90* |
| Гаплогруппы, выявленные в Network-анализе | |||
| Одна группа: (гаплогруппа I) | – | 86.22* | 13.78 |
| Две группы: (гаплогруппа II) и (гаплогруппа III) | 84.64** | 8.16* | 7.20* |
| Четыре группы: (гаплогруппа I), (гаплогруппа II), (гаплогруппа III) и (гаплогруппа IV) | 62.86** | 29.69* | 7.46* |
Таблица 5.
Нуклеотидная дивергенция между видами Oxytropis ochotensis, O. erecta, O. litoralis, O. ruthenica и O. kunashiriensis по данным хпДНК
| Вид | O. ochotensis | O. erecta | O. litoralis | O. ruthenica | O. kunashiriensis |
|---|---|---|---|---|---|
| O. ochotensis | – | 1.576 (0) | 1.655 (0) | 4.017 (1) | 5.655 (4) |
| O. erecta | 0.066 | – | 0.211 (0) | 2.867 (1) | 4.211 (4) |
| O. litoralis | 0.069 | 0.009 | – | 2.657 (1) | 4.000 (4) |
| O. ruthenica | 0.169 | 0.120 | 0.111 | – | 5.626 (4) |
| O. kunashiriensis | 0.237 | 0.176 | 0.167 | 0.236 | – |
Для уточнения филогенетических отношений между исследуемыми видами построена медианная сеть генеалогических связей гаплотипов хпДНК (рис. 2,а). Выявленные гаплотипы формируют четыре гаплогруппы. Гаплогруппу I составили все гаплотипы O. ochotensis (H1–H15), O. erecta (H16–H20) и O. litoralis (H10), гаплогруппы II и III – гаплотипы O. ruthenica: H21–H24 (о-ва Русский и Попова) и H25–H38 (материковая часть и о. Путятина) соответственно, а гаплогруппа IV представлена только гаплотипом O. kunashiriensis (H39). Гаплогруппы I–III расходятся от гипотетического гаплотипа (необнаруженный в нашем исследовании или вымерший предковый) и разделены 5–7 мутационными шагами (рис. 2,а). Гаплотип H39 O. kunashiriensis через 22 мутационных шага и гипотетический гаплотип связан с гаплотипами H32 и H38 O. ruthenica гаплогруппы III. Такое генетическое удаление O. kunashiriensis от всех других видов обусловлено высокой нуклеотидной дивергенцией (табл. 5). Распределение гаплотипов O. ochotensis, O. erecta и O. litoralis в гаплогруппе I не соответствует ни популяционной, ни таксономической принадлежности, что в совокупности с низкой нуклеотидной дивергенцией между большинством популяций этих видов (табл. 2) указывает на генетическую однородность данной группы. Согласно AMOVA (табл. 4), основная доля генетической изменчивости в гаплогруппе I приходится на изменчивость между популяциями (ΦST = 0.8621, P < < 0.0001). Высокая степень дивергенции (около 85%) между гаплогруппами II и III O. ruthenica (табл. 4) свидетельствует о значительной генетической обособленности популяций с о-вов Русский и Попова от всех других. Наличие альтернативных связей (петлевые структуры в сети) между гаплотипами в гаплогруппах I и III (рис. 2,а) не позволяет однозначно установить взаимоотношения между камчатскими популяциями O. ochotensis и O. erecta, а также между популяциями O. ruthenica с о. Путятина и популяцией из окрестностей пос. Врангель.
Рис. 2.
Филогенетические связи близкородственных видов Oxytropis ochotensis, O. erecta, O. litoralis, O. ruthenica и O. kunashiriensis: а – генеалогическая сеть гаплотипов хпДНК (H1–H39), построенная с помощью MJ-метода. Размер окружностей отражает частоту встречаемости гаплотипов, маленькие черные кружки – гипотетические гаплотипы, поперечные тонкие штрихи на ветвях – мутационные события, черные толстые штрихи – вставка нуклеотидов, белые толстые штрихи – делеция нуклеотидов, пунктирной линией обозначены гаплогруппы I–IV. * Мутации для O. glabra, используемого в качестве внешней группы, не указаны и не рассматриваются; б – филогенетическое некорневое MP-дерево (длина – 12 шагов, индекс соответствия CI – 1.0, индекс гомоплазии HI – 0.0, индекс удерживания RI – 1.0), построенное на основе анализа последовательностей ITS рДНК 62 образцов. Числами обозначены значения индекса бутстрепа (выше 50%). Внизу рисунка указана единица измерения длин ветвей – 10 шагов. Сплошной линией обозначены кластеры, объединяющие образцы с одинаковым риботипом (R1–R6). Код популяции см. табл. 1, в скобках дано число образцов данной популяции.
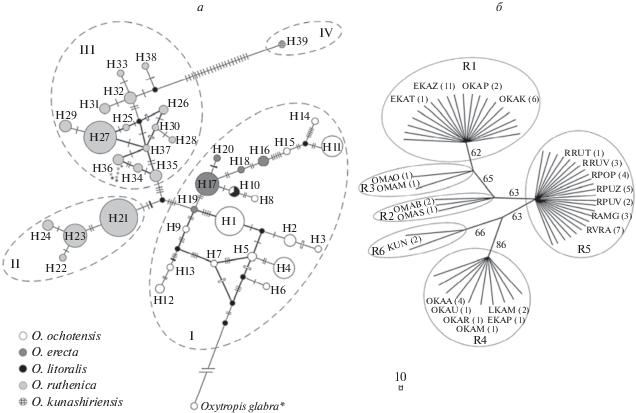
В гаплогруппах I, II и IV выявлены маркерные нуклеотидные замены и индели: в I – T в позиции 2300; во II – T в позиции 1631 и вставка 10 нуклеотидов (TTTTTATATC, позиции 1381–1390); в IV – делеция четырех нуклеотидов (GAAA, позиции 302–305) и C в позициях 1557, 1804 и 1848. Гаплогруппы II и III, объединяющие гаплотипы O. ruthenica, имеют общую маркерную замену (A в позиции 2300), гаплогруппы III и IV – С в позиции 2244. Нуклеотидная дивергенция последовательностей между гаплогруппами I–IV (табл. 6) сравнима с таковой между видами O. ochotensis–O. ruthenica и O. ochotensis–O. kunashiriensis (табл. 5). Иерархический AMOVA показал, что около 63% изменчивости обусловлено различиями между гаплогруппами (табл. 4). Таким образом, генеалогический анализ выявил четкое разделение гаплотипов хпДНК пяти близкородственных видов Oxytropis на четыре эволюционные ветви: 1) O. ochotensis–O. erecta–O. litoralis; 2) O. ruthenica с о-вов Русский и Попова; 3) O. ruthenica с о. Путятина и материковой части Приморского края; 4) O. kunashiriensis.
Таблица 6.
Нуклеотидная дивергенция между гаплогруппами, выявленными в Network-анализе гаплотипов хпДНК Oxytropis ochotensis, O. erecta, O. litoralis, O. ruthenica и O. kunashiriensis
| Гаплогруппа | I | II | III | IV |
|---|---|---|---|---|
| I | – | 4.050 (2) | 3.557 (2) | 5.200 (4) |
| II | 0.170 | – | 4.107 (2) | 7.750 (6) |
| III | 0.150 | 0.172 | – | 4.357 (4) |
| IV | 0.218 | 0.324 | 0.183 | – |
Примечание. Выше диагонали – среднее число нуклеотидных различий (число фиксированных различий), ниже диагонали – среднее число нуклеотидных замен на один сайт ×10–2. Гаплогруппы I–IV – см. рис. 2,а и раздел Результаты.
Нуклеотидные последовательности ITS рДНК 62 образцов исследуемых видов Oxytropis, представляющих все выявленные в данном исследовании гаплотипы хпДНК, характеризуются одинаковой длиной (603 пн) и низкой нуклеотидной изменчивостью. Границы трех участков региона определены путем сравнения с последовательностью ITS O. viscida Nutt. из базы данных GenBank под номером доступа AF121758 [34]. Размеры последовательностей ITS1, гена 5.8S рДНК и ITS2 составили 227, 164 и 212 пн соответственно. Из 603 сайтов семь были вариабельными и информативными согласно методу максимальной экономии: четыре замены (позиции 68, 119, 122, 227) в ITS1 и три (позиции 466, 523, 564) в ITS2. В 62 последовательностях выявлено шесть риботипов (R1–R6), последовательности которых депонированы в GenBank под номерами доступа MK795939–MK795946. O. ochotensis принадлежат четыре риботипа (R1–R4), R1 выявлен также у 12 образцов O. erecta, а R4 – у одного образца O. erecta и у O. litoralis. В 25 образцах O. ruthenica, представляющих 18 гаплотипов хпДНК, обнаружен только один риботип (R5), у O. kunashiriensis – R6. Филогенетический анализ ITS последовательностей методом MP дал одно дерево (длина – 12 шагов, индекс соответствия CI – 1.0, индекс гомоплазии HI – 0.0, индекс удерживания RI – 1.0). Образцы разных популяций и видов сгруппированы в соответствии с определенным риботипом со средней (BP 62–86%) статистической поддержкой (рис. 2,б). Растения из камчатских популяций видов O. ochotensis и O. erecta распределены в двух кластерах с риботипами R1 и R4, а образцы из магаданских популяций O. ochotensis – в двух кластерах с риботипами R2 и R3. Образцы O. litoralis объединились вместе с камчатскими образцами O. ochotensis и O. erecta, имеющими риботип R4. Отдельные кластеры образуют образцы O. ruthenica и O. kunashiriensis с риботипами R5 и R6 соответственно.
ОБСУЖДЕНИЕ
Анализ изменчивости нуклеотидных последовательностей межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG хлоропластного генома у представителей 12 популяций дальневосточных эндемичных видов O. ochotensis, O. erecta, O. litoralis и O. ruthenica выявил, что семь из них характеризуются низким гаплотипическим (h) разнообразием и три – высоким, и низким нуклеотидным (π) разнообразием, две популяции мономорфны (табл. 1). Низкие значения показателей генетического разнообразия обычно связывают с прохождением популяции через так называемое “бутылочное горлышко” – резкое сокращение численности с последующим ее восстановлением; высокие значения h и низкие π характерны для популяций с быстрым ростом численности от небольшого числа основателей, когда прошло достаточное время для восстановления гаплотипической изменчивости за счет мутационного процесса, но не достаточное для накопления значительных нуклеотидных различий в последовательностях ДНК [35]. Отсутствие генетического разнообразия в магаданской популяции OMAO O. ochotensis и в популяции RRUT с о. Русский O. ruthenica может указывать на происхождение их из небольшой группы близкородственных растений. Так, для островных эндемиков Eriogonum arborescens Greene и E. giganteum S. Watson [36] и Astragalus edulis Bunge [37] характерно наличие только одного гаплотипа на каждом из островов, что объясняется однократной их колонизацией, изоляцией и сокращением или полным прекращением обмена генами между островной и родительской популяциями [36, 37]. В целом генетическое разнообразие исследуемых популяций ниже, чем в популяциях эндемичных видов O. chankaensis Jurtz. [16], O. bargusinensis Peschk., O. interposita Sipl. и O. triphylla (Pall.) Pers. [24] и в баргузинских популяциях O. glandulosa Turcz. [25].
Низкая нуклеотидная дивергенция и статистически незначимая генетическая дифференциация между O. ochotensis, O. erecta и O. litoralis (табл. 4, 5), отсутствие видовых молекулярных маркеров, присутствие у всех одной маркерной нуклеотидной замены (T в позиции 2300) и формирование единой гаплогруппы в медианной сети (рис. 2,а) позволяет предположить, что все они относятся к одному виду – O. ochotensis. Это подтверждает ранее высказанные предположения о синонимичности O. erecta и O. litoralis [8], о необходимости рассматривать их как подвиды или разновидности O. ochotensis [5]. Значительная нуклеотидная дивергенция видов O. litoralis и O. ruthenica (табл. 5), принадлежность гаплотипов к разным гаплогруппам (рис. 2,а) и отсутствие общих молекулярных маркеров ставят под сомнение вывод Л.И. Малышева об их таксономической близости [9]. На дендрограмме различий, построенной по данным анализа 47 качественных морфологических признаков, O. litoralis и O. ruthenica оказались в одном кластере, различие составило 22%, на основании этого автор понизил ранг последнего до подвида O. litoralis. В то же время, рассматривая умеренно различающиеся виды O. alpestris Schischkin и O. helenae N.S. Pavlova (различие 24%), Л.И. Малышев предположил: “они могут быть отдельными видами, поскольку имеют далеко разобщенный ареал: один обитает на западе Горного Алтая, другой на Сахалине, и их морфологическое сходство может быть проявлением параллельной (гомологической) эволюции” [9]. Возвращаясь к O. litoralis и O. ruthenica, необходимо отметить, что ареалы этих видов также географически значительно разобщены (рис. 1), это Северо-Восточная Камчатка и Приморье соответственно [3], и промежуточных форм не обнаружено. Таким образом, на основании вышесказанного и полученных нами молекулярно-генетических данных об изменчивости хлоропластного генома можно утверждать, что O. litoralis и O. ruthenica это разные виды, как было принято считать со времени первого описания O. ruthenica [3, 7, 8].
Высокая (около 89%) статистически значимая популяционная дифференциация у O. ruthenica указывает на активно идущие процессы видообразования. Формирование двух четко обособленных эволюционных линий у O. ruthenica подтверждается отсутствием общих гаплотипов, выраженной дифференциацией (около 85%) двух гаплогрупп (рис. 2,а) и выявленными молекулярными маркерами в каждой из них. Наличие двух линий: одна включает гаплотипы популяций о-вов Русский и Попова, другая – гаплотипы популяций материковой части Приморского края и о. Путятина, может быть результатом отсутствия генетического обмена между ними на протяжении многих лет. Так, время изоляции о-вов Русский и Попова составляет около 8500 лет, а о. Путятина – 7000 лет [38]. Ранее у O. glandulosa [28] были выявлены три эволюционные линии, но, в отличие от O. ruthenica, в популяциях с различной плоидностью и сделано предположение о существовании криптических видов, заменяющих или сосуществующих с O. glandulosa, появление которых у растений связывают в первую очередь с полиплоидией. Проведенный анализ полиморфизма маркерных регионов хпДНК выявил высокую нуклеотидную дивергенцию хлоропластного генома O. kunashiriensis от других таксонов (табл. 5, 6; рис. 2,а), что подтверждает его видовой статус.
Ранее нами [17, 39] и другими исследователями [18, 20, 23] было показано, что у видов Oxytropis, как близких, так и из разных секций, несмотря на морфологическую дифференциацию, последовательности ITS рДНК одинаковы или имеют незначительные различия. У O. ruthenica и O. kunashiriensis выявлены индивидуальные риботипы R5 и R6 соответственно, что указывает на их самостоятельность, а наличие общего риботипа R4 у O. ochotensis, O. erecta и O. litoralis является подтверждением их генетического сходства. Обнаруженный внутривидовой полиморфизм ITS у O. ochotensis и O. erecta – четыре (R1–R4) и два (R1 и R4) риботипа соответственно, отражает, по-видимому, аллополиплоидное происхождение гексаплоидов O. ochotensis и O. erecta, и октоплоида O. ochotensis в результате гибридизации полиморфных диплоидных предков.
Таким образом, анализ полиморфизма межгенных спейсеров хпДНК и ITS рДНК близкородственных видов секции Orobia подтверждает видовой статус O. ruthenica и O. kunashiriensis; O. erecta и O. litoralis представляют собой, вероятнее всего, локальные фенотипы широко распространенного полиплоидного вида O. ochotensis. Для окончательного установления статуса O. erecta, O. litoralis и двух эволюционных линий хлоропластного генома O. ruthenica необходимы дополнительные генетические, морфологические и цитологические исследования расширенной выборки растений, охватывающей весь ареал этих таксонов.
Список литературы
Шанцер И.А. Филогения и систематика недавно дивергировавших групп на примере рода Rosa // Тр. Зоол. ин-та РАН. 2013. Приложение № 2. С. 202–216.
Pinheiro F., Dantas-Queiroz M.V., Palma-Silva C. Plant species complexes as models to understand speciation and evolution: a review of South American studies // Crit. Rev. Plant Sci. 2018. V. 37. № 1. P. 54–80. https://doi.org/10.1080/07352689.2018.1471565
Павлова Н.С. Бобовые – Fabaceae // Сосудистые растения советского Дальнего Востока. Л.: Наука, 1989. Т. 4. С. 191–339.
Юрцев Б.А. Oxytropis DC. // Арктическая флора СССР. Л.: Наука, 1986. Вып. 9. Ч. 2. С. 61–146.
Якубов В.В., Чернягина О.А. Каталог флоры Камчатки (сосудистые растения). Петропавловск-Камчатский: Камчатпресс, 2004. 165 с.
Малышев Л.И. Разнообразие рода Остролодка (Oxytropis) в Азиатской России // Turczaninowia. 2008. Т. 11. № 4. С. 5–141.
Васильченко И.Т., Федченко Б.А., Шишкин Б.К. Род Oxytropis // Флора СССР. М.; Л.: Изд-во АН СССР, 1948. Т. 13. С. 1–229.
Ворошилов В.Н. Определитель растений советского Дальнего Востока. М.: Наука, 1982. 672 с.
Малышев Л.И. Фенетика и хорология остролодок секции Orobia Bunge (род Oxytropis DC., Fabaceae) в Азиатской России // Растительный мир Азиатской России. 2008. № 1. С. 3–9.
Малышев Л.И. Род Oxytropis DC. // Конспект флоры Азиатской России: Сосудистые растения. Новосибирск: Изд-во СО РАН, 2012. С. 237–248.
Баркалов В.Ю. Флора Курильских островов. Владивосток: Дальнаука, 2009. 468 с.
Жукова П.Г. Числа хромосом у некоторых видов семейства Fabaceae с северо-востока Азии // Бот. журн. 1983. Т. 68. № 7. С. 925–932.
Пробатова Н.С. Хромосомные числа сосудистых растений Приморского края. Владивосток: Дальнаука, 2014. С. 98.
Bonasora M.G., Lopez A., Vaio M. et al. Origins of polyploidy in Paspalum stellatum and related species (Poaceae, Panicoideae, Paspaleae) inferred from phylogenetic and cytogenetic analyses // Bot. J. Linn. Soc. 2018. V. 188. P. 21–33. https://doi.org/10.1093/botlinnean/boy046
Bolson M., De Camargo Smidt E., Brotto M.L., Silva-Pereira V. ITS and trnH-psbA as efficient DNA barcodes to identify threatened commercial woody angiosperms from Southern Brazilian Atlantic Rainforests // PLoS One. 2015. V. 10. № 12: e0143049. https://doi.org/10.1371/journal.pone.0143049
Artyukova E.V., Kozyrenko M.M., Kholina A.B., Zhuravlev Yu.N. High chloroplast haplotype diversity in the endemic legume Oxytropis chankaensis may result from independent polyploidization events // Genetica. 2011. V. 139. № 2. P. 221–232.
Артюкова Е.В., Козыренко М.М. Филогенетические отношения Oxytropis chankaensis Jurtz. и Oxytropis oxyphylla (Pall.) DC. (Fabaceae) по данным секвенирования ITS рибосомного оперона ядерной ДНК и межгенных спейсеров хлоропластного генома // Генетика. 2012. Т. 48. № 2. С. 186–193.
Archambault A., Strömvik M.V. Evolutionary relationships in Oxytropis species, as estimated from the nuclear ribosomal internal transcribed spacer (ITS) sequences point to multiple expansions into the Arctic // Botany. 2012. V. 90. № 8. P. 770–779. https://doi.org/10.1139/B2012-023
Gao L., Lu P., Jin F. et al. TrnL-F sequences analysis and molecular phylogeny of 10 species of Oxytropis // Acta Botanica Boreali-Occidentalia Sinica. 2013. № 2. P. 266–271.
Tekpinar A., Karaman Erkul S., Aytac Z., Kaya Z. Phylogenetic relationships between Oxytropis DC. and Astragalus L. species native to an Old World diversity center inferred from nuclear ribosomal ITS and plastid matK gene sequences // Turk. J. Biol. 2016. V. 40. P. 250–263. https://doi.org/10.3906/biy-1502-5
Dizkirici Tekpinar A., Karaman Erkul S., Aytac Z., Kaya Z. Phylogenetic relationships among native Oxytropis species in Turkey using trnL intron, trnL–F IGS, and trnV intron cpDNA regions // Turk. J. Bot. 2016. V. 40. P. 472–479. https://doi.org/10.3906/bot-1506-45
Холина А.Б., Козыренко М.М., Артюкова Е.В. и др. Филогенетические взаимоотношения видов Oxytropis DC. subg. Oxytropis и Phacoxytropis (Fabaceae) Азиатской России на основе анализа нуклеотидных последовательностей межгенных спейсеров хлоропластного генома // Генетика. 2016. Т. 52. № 8. С. 895–909.
Shavvon R.S., Kazempour-Osaloo S., Maassoumi A.A. et al. Increasing phylogenetic support for explosively radiating taxa: The promise of high-throughput sequencing for Oxytropis (Fabaceae) // J. Syst. Evol. 2017. V. 55. № 4. P. 385–404. https://doi.org/10.1111/jse.12269
Холина А.Б., Козыренко М.М., Артюкова Е.В., Санданов Д.В. Современное состояние популяций эндемичных видов Oxytropis Байкальской Сибири и их филогенетические связи по данным секвенирования маркеров хлоропластной ДНК // Генетика. 2018. Т. 54. № 7. С. 795–806.
Kholina A., Kozyrenko M., Artyukova E. et al. Plastid DNA variation of the endemic species Oxytropis glandulosa Turcz. (Fabaceae) // Turk. J. Bot. 2018. V. 42. P. 38–50. https://doi.org/10.3906/bot-1706-11
Артюкова Е.В., Холина А.Б., Козыренко М.М., Журавлев Ю.Н. Анализ генетической изменчивости редкого эндемичного вида Oxytropis chankaensis Jurtz. (Fabaceae) на основе RAPD маркеров // Генетика. 2004. Т. 40. № 7. С. 877–884.
Bonfeld J.K., Smith K.F., Staden R. A new DNA sequence assembly program // Nucl. Acids Res. 1995. V. 23. P. 4992–4999.
Gouy M., Guindon S., Gascuel O. SeaView version 4: A multiplatform graphical user interface for sequence alignment and phylogenetic tree building // Mol. Biol. Evol. 2010. V. 27. P. 221–224. https://doi.org/10.1093/molbev/msp259
Excoffier L., Lischer H.E.L. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Resour. 2010. V. 10. P. 564–567.
Librado P., Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data // Bioinformatics. 2009. V. 25. № 11. P. 1451–1452.
Bandelt H.-J., Forster P., Röhl A. Median-joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16. № 1. P. 37–48.
Mir B.A., Koul S., Kumar A. et al. Intraspecific variation in the internal transcribed spacer (ITS) regions of rDNA in Withania somnifera (Linn.) Dunal // Indian J. Biotechnol. 2010. V. 9. P. 325–328.
Swofford D.L. PAUP*: Phylogenetic analysis using parsimony (* and other methods): version 4.04. Sunderland: Sinauer Associate, 2003.
Wojciechowski M.F., Sanderson M.J., Hu J.-M. Evidence on the monophyly of Astragalus (Fabaceae) and its major subgroups based on nuclear ribosomal DNA ITS and chloroplast DNA trnL intron data // Syst. Bot. 1999. V. 24. № 3. P. 409–437.
Абрамсон Н.И. Филогеография: итоги, проблемы, перспективы // Информ. вестник ВОГиС. 2007. Т. 11. № 2. С. 307–331.
Riley L., McGlaughlin M.E., Helenurm K. Narrow water barriers prevent multiple colonizations and limit gene flow among California Channel Island wild buckwheats (Eriogonum: Polygonaceae) // Bot. J. Linn. Soc. 2016. V. 181. P. 246–268.
Bobo-Pinilla J., Penas de Giles J., Lopez-Gonzalez N. et al. Phylogeography of an endangered disjunct herb: long-distance dispersal, refugia and colonization routes // AoB PLANTS. 2018. V. 10. № 5. ply047. https://doi.org/10.1093/aobpla/ply047
Велижанин А.Г. Время изоляции материковых островов северной части Тихого океана // ДАН СССР. 1976. Т. 231. № 1. С. 205–207.
Холина А.Б., Козыренко М.М., Артюкова Е.В. ДНК-типирование видов рода Oxytropis DC. // Росс. конф. с междунар. участием “Регионы нового освоения: современное состояние природных комплексов и вопросы их охраны”: сб. материалов. Хабаровск: ИВЭП ДВО РАН, 2015. С. 106–109.
Дополнительные материалы отсутствуют.


