Генетика, 2020, T. 56, № 6, стр. 648-656
Специфические lux-биосенсоры Escherichia coli, содержащие плазмиды pRecA::lux, pColD::lux и pDinI::lux, для детекции генотоксичных агентов
С. К. Абилев 1, В. Ю. Котова 2, С. В. Смирнова 1, *, Т. Н. Шапиро 1, Г. Б. Завильгельский 2
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
2 Национальный исследовательский центр “Курчатовский институт” (ГосНИИгенетика)
117545 Москва, Россия
* E-mail: s.v.smirnova.genet@gmail.com
Поступила в редакцию 14.11.2019
После доработки 09.12.2019
Принята к публикации 13.12.2019
Аннотация
Сконструирован lux-биосенсор E. coli MG1655 (pDinI::lux) и проведено сравнительное изучение SOS-ответа трех биосенсоров: E. coli MG1655 (pRecA::lux), E. coli MG1655 (pColD::lux) и E. coli MG1655 (pDinI::lux) при воздействии генотоксичных агентов. Перечисленные биосенсоры обозначены соответственно: PRecA, PColD и PDinI. В качестве показателя уровня SOS-ответа lux-биосенсоров учитывали амплитуду ответа (АО). Показано, что АО биосенсора PDinI более выражена (чем АО биосенсора PRecA) при действии перекиси водорода, алкилирующих агентов НММ, ММС и стрептозотоцина, а также антибактериального средства диоксидина и цитостатиков митомицина С и цис-платины. Антиметаболит 5-фторурацил показал активность только на PDinI. Фурацилин и 4‑НХО, метаболиты которых образуют аддукты с ДНК, были более активны на PColD, чем на PRecA и PDinI. Ингибиторы ДНК-гиразы: налидиксовая кислота и ципрофлоксацин, были менее активны на PDinI, чем на PColD и PRecA. В целом из 13 тестированных веществ восемь более активно индуцировали SOS-ответ у биосенсора PDinI, чем у PColD и PRecA. При этом 5-фторурацил индуцировал SOS-ответ только у биосенсора PDinI. Сделано заключение, что биосенсор PDinI может быть с успехом использован для первичного выявления потенциальных генотоксикантов по их способности индуцировать SOS-ответ в клетках E. coli.
В настоящее время lux-биосенсоры нашли применение как для экологического мониторинга, так и для изучения специфической биологической активности химических соединений. Такой биосенсор представляет собой бактериальную клетку, содержащую гибридную плазмиду, несущую два основных элемента: регуляторный участок (промотор и оператор) и гены-репортеры, кодирующие белки, образующие светящиеся комплексы. Lux-биосенсоры характеризуются высокой чувствительностью и специфичностью, что определяется особенностью взаимодействия белка-рецептора с тестируемым химическим веществом.
Для изучения генотоксичности химических и физических факторов широко применяют бактериальные lux-биосенсоры, содержащие гибридные плазмиды, в состав которых встроены регуляторные участки (промотор и оператор) генов recA и cda (сolD), а также lux-оперон люминесцирующей бактерии в качестве репортера [1–8]. В этом случае генотоксичность обнаруживается как результат активации SOS-системы у E. coli. При изучении с помощью lux-биосенсоров 47 химических соединений (главным образом фармакологических средств) 16 из них индуцировали SOS-ответ, а шесть – окислительный стресс [9]. Сравнение полученных результатов с ранее опубликованными данными о мутагенной активности изученных веществ в тесте Эймса показало полное совпадение результатов для 42 веществ. Это указывает на то, что lux-биосенсоры могут быть успешно использованы для выявления генотоксичных агентов при скрининге больших массивов химических соединений, а также для мониторинга образцов воды, аэрозолей воздуха и других компонентов окружающей среды. В этой связи становится актуальным изучение, помимо промоторов precA и pcda, характеристик других промоторов генов, входящих в SOS-систему E. coli, для использования их в lux-биосенсорах.
В настоящей работе проведено сравнительное изучение ответа биосенсоров E. coli MG1655 (pRecA::lux), E. coli MG1655 (pColD::lux) и E. coli MG1655 (pDinI::lux) на генотоксичные химические соединения с различным механизмом ДНК-повреждающего действия.
МАТЕРИАЛЫ И МЕТОДЫ
Конструирование lux-биосенсора E. coli MG1655 (pDinI::lux)
Промоторно-регуляторная часть гена dinI была амплифицирована из генома E. coli MG1655 c помощью праймеров:
DIR 5'-GATGACACGCTGGTGCCATTCC-3'
REV 5'-GTTTTCGCTATGGTGACTTCAATTCG-3'.
Размер ампликона составил 740 пн. Полученный ПЦР-фрагмент встраивали в вектор pTZ57R. Трансформацию клеток штамма E. coli TG-1 проводили с помощью электропорации. Ориентацию встроенного фрагмента определяли, разрезая гибридную плазмиду по сайтам BamH1-EcoR1. Затем фрагмент ДНК, содержащий регуляторную область, вырезали из вектора pTZ57R и встраивали в беспромоторный вектор pDW201 [10]. В результате была сконструирована плазмидаp DinI::lux размером 10 940 пн.
Для конструирования lux-биосенсора был выбран штамм E. coli MG1655. Данный штамм хорошо культивируется в стандартных средах при температурах 30 и 37°С. Биосенсорные штаммы на основе клеток E. coli MG1655 долго сохраняют свои свойства при лиофилизации. В результате трансформации был получен lux-биосенсор E. coli MG1655 (pDinI::lux) (далее обозначен как PDinI).
Химические соединения
Все химические соединения были аналитической чистоты. Митомицин С, цис-платина (цис-диаминодихлорплатина(II)), 4-НХО (4-нитрохинолин-1-оксид), N-нитрозо-N-метилмочевина, метилметансульфонат, стрептозотоцин, 2-аминопурин, 5-фторурацил и 5-бромурацил от “SigmaChemical Со” (США). Ципрофлоксацин от “Промед Экспортс Пвт. Лтд” (Индия), налидиксовая кислота от “Хиноин” (Венгрия). Лекарственные препараты из российских фармацевтических компаний: диоксидин (“Мосхимфармпрепараты”), фурацилин (“Татхимфармпрепараты”), перекись водорода (“Ferraine”). Все растворы тестируемых соединений готовили непосредственно перед их использованием.
Измерение интенсивности биолюминесценции
Культуры клеток lux-биосенсоров выращивали на полноценной среде Луриа-Бертани (LB) с добавлением ампициллина в конечной концентрации 100 мкг/мл в течение ночи при температуре 37°С. Ночную культуру разбавляли свежей средой в 100 раз. Измерения плотности суспензии (OD) проводили на денситометре DEN-1B (“Biosan”). Затем суспензию подращивали в течение двух часов при 37°С до логарифмической фазы.
Для определения активных концентраций генотоксикантов аликвоты культуры (по 180 мкл) переносились в стерильные ячейки планшета, в них же добавлялись индукторы в разных концентрациях в объеме 20 мкл, в качестве контроля использовали дистиллированную воду или соответствующий растворитель. Через 60 и 90 мин инкубации планшетов при 37°С проводили измерения интенсивности биолюминесценции на микропланшетном ридере StatFax 4400, AwarenessTechnologyInc (США).
В качестве главного параметра индукции люминесценции исследуемыми генотоксикантами определяли амплитуду ответа (АО) биосенсоров по следующей формуле: R = (It – I0)/(Ik – I0), где I0 – интенсивность люминесценции в момент добавления генотоксиканта (t = 0), Ik – интенсивность люминесценции в контроле (отсутствие генотоксиканта) в момент t, It – интенсивность люминесценции под действием генотоксиканта в момент t.
РЕЗУЛЬТАТЫ
Определение основных параметров lux-биосенсора PDinI (E. coli MG1655 (pDinI::lux))
Для калибровки lux-биосенсора PDinI использовался митомицин С, который формирует в ДНК как аддукты, так и межнитевые сшивки, для репарации которых необходимы и система рекомбинационной пост-репликативной репарации и система эксцизионной репарации [11].
Интенсивность люминесценции клеток штамма E. coli MG1655 (pDinI::lux) зависела от времени инкубации с различными концентрациями митомицина С (рис. 1).
Рис. 1.
Зависимость интенсивности люминесценции клеток штамма E. coli MG1655 (pDinI::lux) от времени инкубации без митомицина С (контроль) и с митомицином С в концентрациях: С1 – 10–5, С2 – 10–6, С3 – 10–7, С4 – 10–8, С5 – 10–9 моль/л.
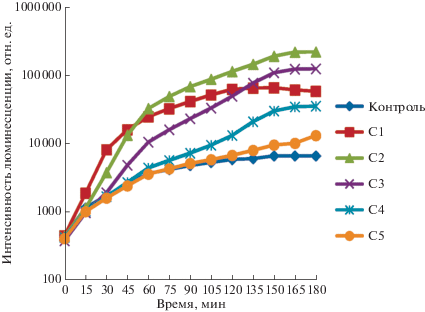
Была определена амплитудa ответа lux-биосенсора E. coli MG1655 (pDinI::lux) от времени инкубации с митомицином С для оптимальной и пороговой концентраций (табл. 1). Минимальное время ответа биосенсора E. coli MG1655 (pDinI::lux) для концентрации митомицина С 10–6 М составило 20 мин, а пороговая концентрация митомицина С, при которой АО удваивается, – 10–9 М.
Ответ биосенсоров E. coli MG1655 (pRecA::lux) (PRecA), E. coli MG1655 (pColD::lux) (PColD) E. coli MG1655 (pDinI::lux) (PDinI) на действие генотоксичных химических соединений
В работе были использованы химические соединения, относящиеся к различным группам по своим биологическим активностям. В первую группу вошли применяемые в медицинской практике бактерицидные средства: диоксидин, фурацилин, ципрофлоксацин, налидиксовая кислота и перекись водорода, а также противоопухолевые цитостатики – митомицин С, цис-платина, стрептозотоцин и антиметаболит 5-фторурацил. Во вторую группу вошли известные мутагены, которые используются в работах по мутагенезу: 4-нитрохинолин-1-оксид (4-НХО), метилметансульфонат (ММС), N-нитрозо-N-метилмочевина (НММ) и аналоги оснований 2-аминопурин и 5-бромурацил.
Тестирование перечисленных соединений на всех трех lux-биосенсорах проводили в восьми повторностях в трех независимых экспериментах. На рис. 2 приведены результаты изучения способности диоксидина индуцировать люминесценцию у биосенсоров PRecA, PColD и PDinI. Регистрировали зависимость интенсивности люминесценции биосенсоров в относительных единицах (RLU) от концентраций диоксидина в моль/л.
Рис. 2.
Показатели интенсивности люминесценции биосенсоров PColD (а), PRecA (б) и PDinI (в) при действии диоксидина; экспозиция 60 (синяя линия) и 90 (красная линия) мин.
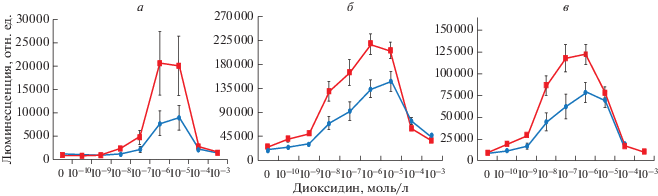
Из представленных графиков следует, что диоксидин у биосенсора PColD индуцирует люминесценцию в интервале концентраций 10–6–10–4 моль/л, тогда как на биосенсорах PRecA и PDinI диапазон активных концентраций диоксидина составляет от 10–8 до 10–4 моль/л. Диоксидин в концентрациях более 10–4 подавляет свечение бактерий вследствие бактерицидного действия.
Пересчет данных по интенсивностям люминесценции биосенсоров в амплитуды ответа по тестированным концентрациям диоксидина при 60 и 90 мин экспозиции приведен на рис. 3 АО отражает отношение интенсивности люминесценции в присутствии индуктора к таковой в контроле на момент измерения и выражается в относительных единицах.
Рис. 3.
Показатели АО биосенсоров PColD, PRecA и PDinI при действии диоксидина; экспозиция 60 (синяя штриховка) и 90 (красные столбики) мин.
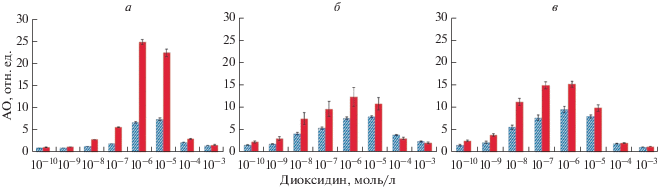
По показателям АО биосенсоров, представленных на рис. 3, можно судить о максимально эффективных концентрациях тестируемого агента. В случае диоксидина такими концентрациями являются: 10–6–10–5 на биосенсоре PColD (АО-25.0), 10–6 на биосенсоре PRecA (АО-12.0) и 10–7–10–6 на биосенсоре PDinI (АО-15). Наибольший АО наблюдается при использовании PColD. Сравнение показателей для PRecA и PDinI указывает на большую эффективность второго. Об этом свидетельствует не только величина АО, но и снижение эффективных концентраций диоксидина.
Количественные показатели амплитуды ответа lux-биосенсоров при действии каждого из исследуемых соединений приведены в табл. 2. АО биосенсора PColD при действии митомицина С, диоксидина, 4-НХО, ципрофлоксацина, налидиксовой кислоты, метилметансульфоната и перекиси водорода превышает таковую у PRecA и PDinI. Однако повышенное значение АО у PColD в случае тестирования налидиксовой кислоты, ципрофлоксацина, НММ и ММС наблюдалось при более высоких концентрацияхэтих соединений, чем на PRecA и PDinI.
Таблица 2.
АО биосенсоров PColD, PRecA и PDinI при действии генотоксичных агентов в течение 60 и 90 мин в условных единицах
| Химическое соединение | Диапазон концентраций, моль/л | PColD | PRecA | PDinI | |||
|---|---|---|---|---|---|---|---|
| 60 | 90 | 60 | 90 | 60 | 90 | ||
| Митомицин С | 10–8–5 × 10–5 | 22.8 (5 × 10–5) |
47.1 (5 × 10–6) |
5.4 (5 × 10–5) |
7.0 (10–5) |
15.0 (5 × 10–6) |
26.0 (5 × 10–6) |
| Диоксидин | 10–12–4.5 × 10–3 | 7.4 (10–5) |
24.9 (10–6) |
7.6 (10–5) |
12.0 (10–6) |
9.4 (10–6) |
15.0 (10–6) |
| Фурацилин | 10–3–10–1 | 1.5 (2.5 × 10–2) |
4.4 (5 × 10–2) |
1.9 (2.5 × 10–2) |
4.0 (2.5 × 10–2) |
2.2 (10–2) |
4.8 (2.5 × 10–2) |
| 4-НХО | 2 × 10–5–3.2 × 10–4 | 18.5 (3.2 × 10–4) |
40.1 (3.2 × 10–4) |
5.7 (1.6 × 10–4) |
9.6 (1.6 × 10–4) |
3.5 (8 × 10–5) |
8.3 (1.6 × 10–4) |
| Налидиксовая кислота | 10–6 –10–2 | 5.3 (10–2) |
12.9 (10–2) |
8.6 (5 × 10–3) |
12.4 (5 × 10–3) |
3.7 (5 × 10–3) |
4.7 (5 × 10–4) |
| Ципрофлоксацин | 5 × 10–8–5 × 10–4 | 7.5 (10–5) |
11.5 (10–5) |
5.5 (5 × 10–5) |
6.5 (10–6) |
5.2 (10–6) |
6.7 (10–6) |
| НММ | 5 × 10–4–10–1 | 15.7 (10–2) |
18.6 (10–2) |
6.8 (10–2) |
7.9 (10–2) |
20.0 (10–2) |
19.6 (10–2) |
| ММС | 10–4–5 × 10–2 | 26.8 (10–2) |
30.1 (10–2) |
6.8 (5 × 10–3) |
8.6 (5 × 10–3) |
11.3 (5 × 10–3) |
15.0 (5 × 10–3) |
| Перекись водорода | 10–3–5 × 10–2 | 23.1 (2.5 × 10–3) |
41.3 (2.5 × 10–3) |
6.3 (7.5 × 10–3) |
7.5 (7.5 × 10–3) |
17.6 (2.5 × 10–3) |
29.2 (5 × 10–3) |
| цис-Платина | 5 × 10–7–10–4 | 2.6 (10–4) |
7.4 (10–4) |
3.0 (10–4) |
5.1 (10–4) |
3.0 (10–4) |
8.6 (10–4) |
| Стрептозотоцин | 10–7–5 × 10–4 | 3.1 (5 × 10–5) |
3.1 (5 × 10–5) |
2.4 (5 × 10–5) |
2.9 (5 × 10–5) |
4.6 (10–5) |
7.3 (5 × 10–6) |
| 2-Аминопурин | 10–4–10–2 | – | 2.4 (2.5 × 10–2) |
– | 2.9 (10–2) |
– | 2.0 (10–2) |
| 5-Бромурацил | 10–4 –10–2 | – | – | 2.6 (10–2) |
4.6 (10–2) |
2.5 (10–2) |
4.6 (10–2) |
| 5-Фторурацил | 10–4 –10–2 | – | – | – | – | 1.8 (5 × 10–3) |
3.6 (7.5 × 10–3) |
Особый интерес представляет сравнение данных по АО lux-биосенсоров PRecA и PDinI, поскольку промоторы генов recA и dinI задействованы, в отличие от промотора гена cda (colD), на начальных этапах SOS-ответа.
Выявили, что АО PDinI более выражена, чем АО PRecA, при действии алкилирующих агентов НММ и ММС, антибактериального препарата диоксидина и цитостатиков митомицина С и цис-платины. При действии нитрофуранового соединения фурацилина, нитрохинолинового соединения 4-НХО, а также ингибиторов ДНК-гиразы, налидиксовой кислоты и ципрофлоксацина, различия в АО этих биосенсоров минимальны (табл. 2).
Биосенсор PDinI при действии цитостатика митомицина С показал более высокую АО (27), чем PRecA (6.9) в случае 90 мин экспозиции. В случае цис-платины, которая так же как и митомицин С, вызывает внутринитевые аддукты и межнитевые сшивки в ДНК, АО у обоих биосенсоров была ниже: PDinI – 8.6, PRecA – 5.1. Алкилирующий цитостатик стрептозотоцин был более активен на PDinI (АО-7.3), чем на PRecA – (АО-5.1).
Аналоги оснований тестировались в концентрациях 10–2–10–4 моль/л. Активность 2-аминопурина для всех трех lux-биосенсоров определяли лишь через 90 мин инкубации. При этом Фактор Индукции (нарастание АО) составлял от 2.0 до 2.9, что свидетельствует об очень низком уровне индукции SOS-ответа. 5-Бромурацил проявил активность на PRecA и PDinI. 5-Фторурацил индуцировал SOS-ответ только у биосенсора PDinI (табл. 3).
Таблица 3.
Минимальные концентрации генотоксичных агентов, достоверно повышающих уровень SOS-ответа биосенсоров. Воздействие 60 и 90 мин
| Химическое соединение | PColD | PRecA | PDinI | |||
|---|---|---|---|---|---|---|
| 60 | 90 | 60 | 90 | 60 | 90 | |
| Митомицин С | 5 × 10–8 (7.3 × 10–4) |
5 × 10–8 (6.7 × 10–4) |
5 × 10–7 (4.5 × 10–7) |
5 × 10–8 (3.4 × 10–3) |
5 × 10–8 (6.6 × 10–9) |
5 × 10–8 (1.9 × 10–11) |
| Диоксидин | 10–8 (2.7 × 10–2) |
10–8 (5.2 × 10–5) |
10–10 (1.3 × 10–2) |
10–10 (1.6 × 10–4) |
10–9 (1.7 × 10–4) |
10–11 (1.5 × 10–2) |
| Фурацилин | 10–2 (1.2 × 10–2) |
5 × 10–3 (2.5 × 10–2) |
2.5 × 10–3 (8.3 × 10–8) |
10–3 (1.1 × 10–5) |
10–3 (1.0 × 10–3) |
10–3 (2.0 × 10–8) |
| 4-НХО | 2 × 10–5 (2.6 × 10–6) |
2 × 10–5 (3.5 × 10–10) |
2 × 10–5 (4.5 × 10–6) |
2 × 10–5 (2.2 × 10–6) |
2 × 10–5 (1.1 × 10–13) |
2 × 10–5 (1.1 × 10–15) |
| Налидиксовая кислота | 5 × 10–5 (6.0 × 10–4) |
5 × 10–5 (1.3 × 10–4) |
5 × 10–5 (4.2 × 10–5) |
10–6 (1.1 × 10–2) |
5 × 10–5 (4.3 × 10–4) |
5 × 10–5 (1.5 × 10–3) |
| Ципрофлоксацин | 5 × 10–8 (2.8 × 10–4) |
5 × 10–8 (7.0 × 10–6) |
5 × 10–8 (1.2 × 10–7) |
5 × 10–8 (8.3 × 10–10) |
5 × 10–8 (3.1 × 10–10) |
5 × 10–8 (1.6 × 10–12) |
| НММ | 5 × 10–4 (4.0 × 10–8) |
5 × 10–4 (2.5 × 10–6) |
5 × 10–4 (1.8 × 10–9) |
5 × 10–4 (1.1 × 10–8) |
5 × 10–4 (6.9 × 10–13) |
5 × 10–4 (8.9 × 10–14) |
| ММС | 10–4 (3.3 × 10–3) |
10–4 (1.5 × 10–3) |
10–4 (3.8 × 10–8) |
10–4 (3.0 × 10–10) |
10–4 (9.3 × 10–10) |
10–4 (3.4 × 10–11) |
| Перекись водорода | 10–3 (1.2 × 10–10) |
10–3 (3.3 × 10–8) |
10–3 (3.1 × 10–18) |
10–3 (1.7 × 10–14) |
10–3 (9.7 × 10–14) |
10–3 (1.8 × 10–16) |
| цис-Платина | 5 × 10–5 (8.5 × 10–5) |
10–5 (1.7 × 10–3) |
5 × 10–6 (3.7 × 10–3) |
5 × 10–6 (4.1 × 10–7) |
5 × 10–6 (5.6 × 10–4) |
10–6 (1.4 × 10–5) |
| Стрептозотоцин | 5 × 10–5 (4.7 × 10–6) |
5 × 10–5 (1.2 × 10–5) |
10–5 (6.5 × 10–9) |
10–5 (9.7 × 10–8) |
10–7 (3.8 × 10–7) |
10–7 (1.8 × 10–9) |
| 2-Аминопурин | 10–4 (10–3) |
10–4 (3.5 × 10–2) |
2.5 × 10–4 (3.0 × 10–4) |
10–4 (1.7 × 10–3) |
– | 10–4 (1.3 × 10–2) |
| 5-Бромурацил | 7.5 × 10–3 (1.9 × 10–2) |
2.5 × 10–3 (1.7 × 10–2) |
5 × 10–3 (3.8 × 10–2) |
10–3 (2.2 × 10–2) |
10–3 (10–2) |
5 × 10–4 (1.8 × 10–3) |
| 5-Фторурацил | – | 7.5 × 10–4 (8.4 × 10–3) |
– | 2.5 × 10–4 (2.8 × 10–3) |
2.5 × 10–4 (4.2 × 10–8) |
10–4 (3.1 × 10–10) |
Минимальные концентрации изучаемых соединений, вызывающие статистически достоверное повышение индуцированной люминесценции над контролем, приведены в табл. 3. В скобках приведены количественные значения уровней значимости (p < 0.05). Наиболее эффективными индукторами SOS-ответа по данному критерию оказались: митомицин С, цис-платина, диоксидин и ципрофлоксацин. Если анализировать представленные минимальные концентрации тестированных соединений в связке со значениями уровней значимости, то можно утверждать, что для НММ и ММС концентрация 10–3 при p ≤ 10–7 не является реально минимальной и нуждается в уточнении.
ОБСУЖДЕНИЕ
В генетической токсикологии для выявления и оценки мутагенной активности химических факторов окружающей среды принят поэтапный подход. На первом этапе тестирования (этап скрининга) используются бактериальные тест-системы и клеточные культуры. На втором этапе вещества, показавшие активность на этапе скрининга, тестируются in vivo по способности индуцировать микроядра или хромосомные аберрации в клетках костного мозга мышей или крыс [12]. Для первичной оценки мутагенной активности химических соединений применяют тест Эймса Salmonella/микросомы [13]. Для регистрации мутагенной активности в данном тесте используется набор из пяти штаммов S. typhimurium, ауксотрофных по гистидину и ревертирующих к дикому типу в результате индукции обратных мутаций. Они позволяют устанавливать вероятные механизмы мутагенного действия. Большое число штаммов является одним из ограничивающих факторов применения теста Эймса для скрининга массива химических соединений. В этой связи возникает вопрос, можно ли обойтись минимальным набором тест-штаммов S. typhimurium, например ТА 98 и ТА 100, и включить в схему тестирования второй скрининговый метод, дополняющий тест Эймса и отличающийся простотой выполнения. В качестве такового был предложен метод, получивший название “SOS-хромотест”, с использование мштамма E. coli PQ37, у которого ген lacZ слит с промотором гена sfiA, входящего в систему SOS-системы [14]. Ответ штамма E. coli PQ37 регистрируется по результатам биохимической реакции галактозидазы с субстратом ОНФГ (о-нитрофенил-β-D-галактопиранозил). При этом из бесцветного субстрата ОНФГ высвобождается нитрофенил желтого цвета, и по интенсивной желтой окраски можно судить об активности β-галактозидазы, т.е. об уровне SOS-ответа. Ретроспективный анализ результатов тестирования 452 химических соединений в SOS-хромотесте и в тесте Эймса показал 82% совпадение результатов, то есть 378 соединений проявили одинаковую активность в обоих тестах [15].
Метод измерения SOS-ответа с помощью lux-биосенсоров E. сoli по индукции интенсивности биолюминесценции технически более экономичен и удобен, чем колориметрическое детектирование ферментативной активности в SOS-хромотесте. При люминесцентном анализе регистрация ответа происходит по свечению бактерий и не требует дополнительных манипуляций, таких как лизис бактерий после инкубации и определение ферментативной активности. Непосредственный анализ люминесценции бактерий позволяет одновременно регистрировать зависимость SOS-ответа как от концентрации тестируемого соединения, так и от продолжительности инкубирования. Lux-тест легко поддается автоматизации.
В настоящее время для изучения генотоксичности химических агентов среды используют lux-биосенсоры PColD и PRecA. У каждого из них свои преимущества и недостатки. Например, у PColD для генетического мониторинга загрязнений окружающей среды недостает чувствительности к низким концентрациям генотоксиканта. Биосенсор PRecA характеризуется высоким уровнем спонтанной люминесценции, связанной с частичной открытостью промотора pRecA. Это снижает амплитуду ответа, необходимого для достоверной регистрации индуцированного SOS-ответа. В этой связи нами было проведено сравнение таких характеристик биосенсоров PRecA, PColD и PDinI, как амплитуда ответа и минимальная концентрация генотоксикантов, статистически значимо повышающих SOS-ответ. Биосенсор PDinI был сконструирован в настоящей работе. Белок DinI играет значимую роль в регуляции индукции как SOS-ответа, так и SOS-мутагенеза [16–18]. На начальном этапе SOS-ответа начинается синтез DinI, который, формируя комплекс с RecA на более позднем этапе SOS-ответа, ингибирует образование филаментов RecA/ssDNA, что приводит к снижению SOS-ответа [19]. Однако наличие в среде генотоксичного агента приводит вновь к усилению SOS-ответа и т.д. [16, 19].
Белок DinI участвует и в регуляции SOS-индуцированного мутагенеза. На раннем этапе SOS-ответа DinI блокирует протеолиз белка UmuD до состояния UmuD', компонента ДНК-полимеразы V, что предотвращает запуск мутагенеза в начале SOS-репарации [19, 20].
Полученные нами результаты показали, что биосенсор PDinI выгодно отличается от PRecA. Например АО биосенсора PDinI более выражен, чем АО биосенсора PRecA при действии перекиси водорода, алкилирующих агентов НММ, ММС и стрептозотоцина, а также антибактериального средства диоксидина и цитостатиков митомицина С и цис-платины. Антиметаболит 5-фторурацил показал активность только на PDinI. Фурацилин и 4-НХО, метаболиты которых образуют аддукты с ДНК, были более активны на PColD, чем на PRecA и PDinI. Ингибиторы ДНК-гиразы налидиксовая кислота и ципрофлоксацин были мене активны на PDinI, чем на PColD и PRecA. В целом из 13 тестированных веществ восемь более активно индуцировали SOS-ответ у биосенсора PDinI, чем у PColD и PRecA. При этом 5-фторурацил индуцировал SOS-ответ только у биосенсора PDinI. Надо отметить, 5-фторурацил, являющийся структурным аналогом пиримидина, используется в медицинской практике в качестве противоопухолевого агента. Препарат метаболизируется в опухолевой ткани с образованием 5-фторуридинтрифосфата и 5-фтордезоксиуридина. Последний ингибирует тимидилатсинтетазу, что приводит к дефициту тимидина и ингибированию синтеза ДНК [21]. По-видимому он и в бактериальной клетке ингибирует синтез ДНК, приводящий к индукции SOS-ответа у биосенсора PDinI. Такая специфичность требует дальнейшего исследования. В результате проведенных исследований можно сделать заключение, что биосенсор PDinI может быть с успехом использован для первичного выявления потенциальных генотоксикантов по их способности индуцировать SOS-ответ в клетках E. coli.
Работа выполнена при финансовой поддержке гранта РФФИ 190400200.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Vollmer A.C., Belkin S., Smulski D.R. et al. Detection of DNA damage by use of Escherichia coli carrying recA':::lux, uvrA'::lux, or alkA'::lux reporter plasmids // Appl. Environ. Microbiol. 1997. V. 63. P. 2566–2571.
Davidov Y., Rozen R., Smulski D.R. et al. Improved bacterial SOS promoter&Colon: Lux fusions for genotoxicity detection // Mut. Res. 2000. V. 466. P. 97–107.
Norman A.A., Hansen L.H., Sorensen S.J. Construction of a ColD cda promoter – based SOS-Green fluorescent protein whole-cell biosensor with higher sensitivity toward genotoxic compounds than constructs based on recA, umuDC, or sulA promoters // Appl. Environ. Microbiol. 2005. V. 71. P. 2338–2346. https://doi.org/10.1128/AEM.71.5.2338-2346.2005
Manukhov I.V., Kotova V.Yu., Zavilgelsky G.B. Action of 1,1-dimethylhydrazine on bacterial cells is determined by hydrogen peroxide // Mut. Res. 2007. V. 634. P. 176–179.
Song Y., Li G., Thornton S.F., Thompson I.P. Optimization of bacterial whole cell bioreporters for toxicity assay of environmental samples // Environ. Sci. Technol. 2009. V. 43. № 20. P. 7931–7938.
Котова В.Ю., Манухов И.В., Завильгельский Г.Б. Lux-биосенсоры для детекции SOS-ответа, теплового шока и окислительного стресса // Биотехнология. 2009. № 6. С. 16–25. https://doi.org/10.1134/S0003683810080089
Завильгельский Г.Б., Котова В.Ю., Манухов И.В. Сенсорные биолюминесцентные системы на основе lux-оперонов для детекции токсичных веществ // Хим. физика. 2012. Т. 31. № 10. С. 15–20.
Ahn J.-M., Hwang E.T., Youn C.-H. et al. Prediction and classification of the modes of genotoxic actions using bacterial biosensors specific for DNA damages // Biosensors and Bioelectronics. 2009. V. 25. P. 767–772. https://doi.org/10.1016/j.bios.2009.08.025
Игонина Е.В., Марсова М.В., Абилев С.К. Lux-биосенсоры: скрининг биологически активных соединений на генотоксичность // Экол. генет. 2016. Т. 14. С. 52–62. https://doi.org/10.17816/ecogen14452-62
Van Dyk T.K., Rosson R.A. Photorhabdus luminescens lux CDABE promoter probe vectors // Meth. Mol. Biol. 1998. V. 102. P. 85–95.
Weng M.-W., Zheng Y., Jasti V.P. et al. Repair of mitomycin C mono- and interstrand cross-linked DNA adducts by UvrABC: a new model // Nucl. Ac. Res. 2010. V. 38. № 20. P. 6976–6984.
Абилев С.К., Глазер В.М. Генетическая токсикология: итоги и проблемы // Генетика. 2013. Т. 49. № 1. С. 81–93. https://doi.org/10.7868/S0016675813010025
McCann J., Choi E., Yamasaki E., Ames B.N. Detection of carcinogens as mutagens in the Salmonella/microsome test: Assay of 300 chemicals // Proc. Natl Acad. Sci. USA. 1975. V. 72. № 12. P. 5135–5139.
Quillardet P., Huisman O., D’Ari R. et al. SOS chromotest, a direct assay of induction of an SOS function in Escherichia coli K12 to measure genotoxity // Proc. Natl Acad. Sci. USA. 1982. V. 79. № 19. P. 5971–5975. https://doi.org/10.1073/pnas.79.19.5971
Quillardet P., Hofnung M. SOS chromotest: A review // Mut. Res. 1993. V. 297. P. 235–279. https://doi.org/10.1016/0165-1110(93)90019-j
Cox M.M. Regulation of bacterial RecA protein function // Crit. Rev. Biochem. Mol. Biol. 2007. V. 42. № 1. P. 41–63. https://doi.org/10.1080/10409230701260258
Galkin V.E., Britt R.L., Bane L.B. et al. Two modes of binding of DinI to RecA filament provide a new insight into the regulation of SOS response by DinI protein // J. Mol. Biol. 2011. V. 408. P. 815–824. https://doi.org/10.1016/j.jmb.2011.03.046
Voloshin O.N., Ramirez B.E., Bax A., Camerini-Otero R.D. A model for the abrogation of the SOS response by an SOS protein: A negatively charged helix in DinI mimics DNA in its interaction with RecA // Genes. Dev. 2001. V. 15. P. 415–427. https://doi.org/10.1101/gad.86290
Yasuda T., Morimatsu K., Kato R. et al. Physical interactions between DinI and RecA nucleoprotein filament for the regulation of SOS mutagenesis // EMBO J. 2001. V. 20. P. 1192–1202. https://doi.org/10.1093/emboj/20.5.1192
Yasuda T., Morimatsu K., Horii K. et al. Inhibition of Escherichia coli RecA coprotease by DinI // EMBO J. 1998. V. 17. № 11. P. 3207–3216. https://doi.org/10.1093/emboj/17.11.3207
Longley D.S., Harkin D.P., Johnston P.G. 5-Fluorouracil: Mechanisms of action and clinical strategies // Nature Rev. Cancer. 2003. V. 3. № 5. P. 330–337. https://doi.org/10.1038/nrc1074
Дополнительные материалы отсутствуют.


