Генетика, 2020, T. 56, № 6, стр. 657-666
Феномен высокой частоты мутаций в генах 35S рРНК С-субгенома у полиплоидных видов Avena L.
А. В. Родионов 1, 2, *, А. В. Амосова 3, Л. М. Крайнова 2, Э. М. Мачс 1, Ю. В. Михайлова 1, А. А. Гнутиков 4, О. В. Муравенко 3, И. Г. Лоскутов 2, 4
1 Ботанический институт им. В.Л. Комарова Российской академии наук
197376 Санкт-Петербург, Россия
2 Санкт-Петербургский государственный университет
199034 Санкт-Петербург, Россия
3 Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
4 Федеральный исследовательский центр Всероссийский институт генетических ресурсов растений
им. Н.И. Вавилова
190000 Санкт-Петербург, Россия
* E-mail: avrodionov@mail.ru
Поступила в редакцию 09.06.2019
После доработки 30.07.2019
Принята к публикации 11.09.2019
Аннотация
Используя локус-специфичное ДНК-секвенирование на платформе Roche 454, мы изучили внутригеномный полиморфизм одного из районов гена 35S рРНК (фрагмент 18S рДНК–ITS1–фрагмент 5.8S рДНК) у трех гексаплоидных видов Avena c кариотипом AACCDD и у тетраплоида A. insularis (AAСС или CCDD). В геномах этих полиплоидов найдены рДНК, полученные от предков-диплоидов как с А-, так и с С-геномами. У A. insularis вместо ожидаемых 50% ITS1 С-типа было только 3.3%, а у гексаплоидов рДНК, полученной от предков с С-геномами, только 1.4–2.4%. Последовательности 18S рДНК, ITS1 и 5.8S рДНК С-субгеномов были в 10 раз более вариабельны, чем те же последовательности A-субгеномов. Некоторые из последовательностей С-субгеномов содержали делеции, в том числе делеции в районе, кодирующем 18S рРНК. Результаты FISH с зондами pTa71 и pTa794 подтверждают факт утраты полиплоидами значительной части 35S рДНК и 5S рДНК субгенома, полученного от диплоида с кариотипом СС. Последовательности ITS1 С-субгеномов полиплоидных видов разнообразны, но среди них можно выделить основной (core) вариант, примерно равноудаленный от ITS диплоидов, несущих Сv- и Cp-геномы. Наши результаты показывают, что утрата 35S рДНК С-субгеномов происходит на фоне накопления в этих последовательностях множества однонуклеотидных замен (SNPs) и делеций. В “репрессируемых” локусах 35S рРНК С-субгеномов множественное мутирование, по-видимому, не сопровождается гомогенизацией рДНК – есть основания думать, что процессы изогенизации рДНК и процессы транскрипции/сайленсинга – связанные явления.
Все современные виды сосудистых растений прошли через несколько раундов полиплоидизации генома (полногеномных дупликаций) – это важные этапы в процессах прогрессивной эволюции растений [1, 2]. Геномы неополиплоидов и первых поколений гибридов, как правило, нестабильны. В них наблюдаются экспансия транспозонов, потеря части генов одного из родителей, изменение паттерна транскрипции и сплайсинга некоторых из дуплицированных генов. У гибрида в сравнении с родительскими видами происходят неаддитивные изменения генома, транскриптома, протеома и метаболома – изменения, направление которых в общем коррелирует с уклонениями в морфологии гибрида в ту или иную сторону [2–5]. Постепенно геном неополиплоида перестраивается за счет транспозиций, делеций и транслокаций. Постепенная утрата части генов и хромосом одного из “родителей” стабилизирует геном гибрида, он переходит в состояние эуполиплоида [4–6]. На этой стадии кариотип аллополиплоида относительно стабилен и выглядит как кариотип типичного полиплоида, у которого можно более или менее надежно идентифицировать гомологичные и гомеологичные хромосомы. Существенно, что геном каждого из межвидовых гибридов и видов, прошедших через стадию полиплоидизации, уже через одно или несколько поколений представляет собой уникальную генетическую систему, причудливую мозаику в той или иной степени измененных родительских геномов, репитомов, транскриптомов, протеомов, метаболомов, всегда разную в каждом конкретном случае [2, 7, 8].
Классическое проявление взаимодействия отцовского и материнского геномов у полиплоидов гибридного происхождения (аллополиплоидов) – ядрышковое доминирование [9]. Суть явления состоит в том, что у гибрида в этом случае работают гены 35S рРНК одного из родительских геномов. Ядрышковые организаторы (ЯОР), полученные от второго родителя, транскрипционно инертны и/или в той или иной степени утрачены [10–12].
Когда в геноме полиплоида сохраняются рДНК обоих родителей, их можно использовать для определения происхождения предполагаемого гибрида [11, 13–15]. Сделать это удается не всегда. Так, секвенирование ITS-последовательностей гена 35S рРНК овсов по Сэнгеру выявляло у всех полиплоидных видов рода Avena только ITS-последовательности, характерные для A-субгеномов; последовательности С-субгеномов, непременного элемента кариотипов всех полиплоидных видов овсов, при этом не обнаруживаются [16–18]. В какой-то мере это согласуется с данными цитогенетики. FISH-гибридизация показала, что в кариотипах полиплоидных видов A. insularis, A. sterilis, A. fatua, A. ludoviciana основные сайты 35S рРНК (ЯОР) лежат на хромосомах А-субгенома, а на С‑субгеномных хромосомах регистрируются только минорные сайты [19–22]. Причем нельзя исключить, что в этих минорных сайтах уже лежит “отредактированная” путем конверсии или иного механизма рДНК, неотличимая от рДНК A-субгеномов. Однако какие-то гены рРНК, происходящие от диплоидного предка с С-геномом, у одного или нескольких полиплоидных видов овсов, пусть в минорных количествах, но остаться могли. Косвенно об этом свидетельствовали данные Пенга и соавт. [18], секвенировавших 113 клонов рДНК 12 полиплоидных видов Avena и нашедших среди них только у одного вида A. fatua и только одну последовательноcть района ITS C-типа, близкую к ITS диплоидных видов A. eriantha и A. clauda.
Для того чтобы выявить в геномах полиплоидных Avena остаточные копии ITS-последовательностей C-типа, Николоудакис и Кациотис [17] и Тюпа с соавт. [23] разработали два набора праймеров, позволяющих специфично амплифицировать ITS С-субгеномов Avena, и таким образом амплифицировали и секвенировали минорные варианты ITS-последовательностей С-типа из генома нескольких полиплоидных видов. Они оказались ближе к ITS A. ventricosa, чем к A. clauda и A. eriantha [15, 17, 23].
Цель данной работы – исследовать внутригеномное разнообразие ITS-последовательностей у четырех полиплоидных видов Avena, A. insularis (геномная формула AACC или CCDD – [21]), A. fatua, A. ludoviciana и A. sterilis (AACCDD), используя метод локус-специфичного секвенирования на платформе 454. Геномы А и С различаются по положениям центромеры и ЯОР на хромосомах, паттерну микросателлитов и по дифференциальному С-окрашиванию [19–22, 24, 25]. Диплоидные виды с кариотипами АА подразделяются на группы с кариотипами AsAs, AlAl; AdAd, AcAc, ApAp, различающиеся по числу акроцентрических хромосом [25]. Диплоидные виды с кариотипами СС также подразделяются на группы. Различают кариотипы CvCv (A. ventriсosa, A. bruhnsiana) и СрСр (A. pilosa, A. clauda). Признак, который отличает Ср-геном от Cv-генома – это число спутничных хромосом: в геномах Ср-типа две спутничные хромосомы, с большим и малым спутником, в Cv – одна пара хромосом с малым спутником [25]. У тетраплоидного вида A. macrostachya кариотип CmCmCmCm [16, 22]. Геном D у диплоидных видов не найден, он есть только в составе полиплоидных геномов. Считается, что это дериват A-генома [19, 21].
Изучая в ходе настоящей работы внутригеномный полиморфизм рДНК полиплоидных видов Avena, мы обнаружили, что потеря рДНК С-субгеномов полиплоидных овсов происходит на фоне высокого разнообразия их рДНК. Каждая из выявленных нами последовательностей рДНК С‑субгеномов несла множественные уникальные однонуклеотидные замены (SNPs), некоторые из этих последовательностей имели протяженные делеции, в том числе такие, которые захватывали и районы, кодирующие 18S рРНК. Это означает, что в “исчезающих” ЯОР С-субгеномов не идут процессы гомогенизации рДНК. Возможно, гомогенизация рДНК происходит только в транскрибируемых копиях рибосомных генов.
МАТЕРИАЛЫ И МЕТОДЫ
В работе использовались семена видов овса из коллекции Всероссийского института генетических ресурсов растений им. Н.И. Вавилова: 1) Avena insularis Ladiz (VIR 2067), происхождение образца – Сицилия; 2) Avena fatua L. (VIR30) – Тува (два растения); 3) Avena ludoviciana Durieu (VIR389) – Венесуэла (два растения); 4) Avena sterilis L. (VIR511) – Израиль (два растения).
ДНК выделяли CTAB-методом [26]. Для исследования внутригеномного полиморфизма района 18S рДНК (фрагмент)–ITS1–5.8S рДНК (фрагмент) мы использовали подход, предложенный Матиашек и соавт. [11]. Исследуемый район амплифицировался с помощью праймеров ITS1P [27] или ITS5А [28] и ITS2 [29] с адаптерами для 454-секвенирования. Секвенирование по технологии 454 Life Sciences выполняли на секвенаторе Junior GC (Roche) на базе ЦКП ВНИИ сельскохозяйственной микробиологии. На первом этапе анализа полученных данных проводилась техническая фильтрация данных стандартными методами с использованием программ FastQC (http://www.bioinformatics.babraham.ac.uk/projects/ fastqc/), Trimmomatic [30] и Fastq-join [31]. Дальнейшая фильтрация данных более сложна, поскольку анализ SNPs в данном случае проводится на основе безреференсного выравнивания. Алгоритм действий был следующим: 1) консенсусная фильтрация (исключение из анализа заведомо не выравниваемых вариантов – очевидных контаминантов); 2) с использованием программы COUNT рассчитывалась частота каждого варианта последовательности в выборке – одинаковые гаплотипы представлены в дальнейшем анализе одной последовательностью с индексом – частота повторяемости. Для подсчета частот и сортировки гаплотипов использовали алгоритмы попарного сравнения и пузырьковой сортировки; 3) далее использовали программное обеспечение UGENE [32] и MEGA7 [33]. Сравнивали секвенированные варианты ITS с секвенированными по Сэнгеру ITS-последовательностями, характерными для С- и А-геномов диплоидных видов овсов [16–18, 23], и выявляли среди них последовательности ITS C-субгеномов, несущих ранее идентифицированные нами уникальные делеции, по которым последовательности ITS1 С- и А-геномов овсов различаются. В геноме полиплоидов обнаруживаются в большем или меньшем количестве гаплотипы близкие, но не идентичные рДНК ныне существующих диплоидных видов, последовательностей ITS A-типа много, ITS C-типа – единичные. Дальнейшую обработку полученных данных выполняли с использованием алгоритма TCS [34]. Алгоритм TCS позволяет определять вероятность связи между всеми гаплотипами с индикацией числа мутаций, по которым различаются исследуемые гаплотипы. Результаты TCS-расчетов далее обрабатывались в программе tcsBU [35].
Препараты для FISH готовили в соответствии со стандартным протоколом [36, 37]. Районы 35S рДНК выявляли с помощью плазмиды pTa71, содержащей последовательность 18S рДНК (фрагмент)–5.8S рДНК–26S рДНК (фрагмент) [38], гены 5S рРНК – с помощью плазмиды pTa794, содержащей ДНК-последовательность генома пшеницы длиной 420 пн [39]. ДНК pTa71 и рTa794 метили методом ник-трансляции biotin-16-dUTP (Roche Diagnostics, Mannheim, Germany) и digoxigenin-11-dUTP (Roche Diagnostics, Mannheim, Germany). Процедура FISH описана ранее [40].
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
По результатам 454-пиросеквенирования была определена последовательность нуклеотидов 5637 “прочтений” района 18S рДНК (фрагмент)–ITS1–5.8S рДНК (фрагмент) гена 35S рРНК ядерного генома семи растений, относящихся к четырем полиплоидным видам рода Avena. У части из них были выявлены характерные для С-геномов индели [16–18, 23], при этом абсолютное большинство ITS1 в геномах исследованных полиплоидов относилось к семейству ITS1, характерному для A-геномов. Нами было рассчитано, какая часть рДНК полиплоидов несет в ITS1 делеции, характерные для С-геномов. Напомним, что у A. fatua, A. ludoviciana и A. sterilis геномная формула кариотипа AACCDD [25, 41]. Геномная формула кариотипа тетраплоида A. insularis – предмет дискуссий: полагают, что это AACC или CCDD [23, 24, 41]. Можно думать, что у аллотетраплоида A. insularis, в момент его возникновения, было примерно равное количество рДНК, пришедших от диплоидного предка с C-геномом и предка с А‑геномом. В кариотипах гексаплоидов AACCDD только около трети рДНК могло относиться к С‑типу. На деле доля ITS1 C-типа оказалась 3.2% у A. insularis и 1.4–2.4% у гексаплоидов (табл. 1).
Таблица 1.
Абсолютное и относительное содержание С- и А-геномспецифичных последовательностей в образцах
| Вид и геномный состав кариотипа | Общее число полученных последовательностей ITS1 | C-геномспецифичные | А-геномспецифичные |
|---|---|---|---|
| Avena insularis, AACC или CCDD | 828 100% |
27 3.26% |
801 96.74% |
| A. ludoviciana, растение 1, AACCDD | 791 100% |
11 1.39% |
780 98.61% |
| A. ludoviciana, растение 2, AACCDD | 758 100% |
12 1.58% |
746 98.42% |
| A. fatua, растение 1, AACCDD | 873 100% |
21 2.41% |
852 97.59% |
| A. fatua, растение 2, AACCDD | 776 100% |
17 2.19% |
759 97.81% |
| A. sterilis, растение 1, AACCDD | 785 100% |
12 1.53% |
773 98.47% |
| A. sterilis, растение 2, AACCDD | 828 100% |
13 1.57% |
815 98.42% |
В то время как среди последовательностей ITS А-типа были такие, которые повторены сотни и десятки раз, все последовательности С-субгеномов у полиплоидов, выявляемые при секвенировании 454, были уникальны, все содержали однонуклеотидные мутации, делеции и вставки, отличающие их друг от друга и от референс-последовательности ITS1 Avena. Обращает на себя внимание, что часть последовательностей С-субгеномов содержала делеции, имеющие длину 30 и 34 пн, лежащие на 5'‑границе ITS1 и захватывающие 18S рРНК (рис. 1). Их доля составила 6–8% от числа ITS1 C-типа. Оба варианта делеций найдены у трех гексаплоидов, у тетраплоида A. insularis выявлен только вариант делеции 34 пн.
Рис. 1.
Два варианта делеций на границе гена 18S рРНК и ITS1, обнаруженные у полиплоидных видов Avena insularis, A. ludoviciana и A. fatua. Стрелка – граница последовательности гена 18S рРНК (слева) и ITS1 (строчные буквы).

Несмотря на то что образцы видов, взятые в исследование, получены из разных мест (A. insularis – Сицилия, A. ludoviciana – Венесуэла, A. fatua – Тува, A. sterilis – Израиль), в недавнем прошлом, с точки зрения агростологов, эти виды имеют общее происхождение. Так, дикорастущий сицилийский тетраплоидный овес A. insularis является наиболее вероятным, в сравнении с другими тетраплоидами, предком гексаплоидных овсов [22, 42, 43]. Из гексаплоидных овсов древнейшей формой является A. sterilis, возникший в азиатском центре происхождения овсов [44, 45]. В процессе распространения на запад A. sterilis, по-видимому, приобрел мутацию типа fatua, что привело к появлению A. fatua. Затем А. sterilis стал дифференцироваться и приобрел более адаптивные мелкосеменные формы, характерные для A. ludoviciana, который часто рассматривается как подвид A. sterilis [45].
Найденные нами у всех четырех изученных видов минорные варианты генов 35S рРНК с делецией, захватывающей ДНК, кодирующую 3'-конец молекулы 18S рРНК (рис. 1), косвенно подтверждают гипотезу об общем происхождении этих видов, причем вариант делеции с длиной 30 пн может быть возник уже после того, как появился гексаплоид A. sterilis.
Мы рассчитали средние р-расстояния (p-distance) отдельно для трех выборок: последовательности 18S (фрагмент), 5.8S рРНК (фрагмент) и последовательности ITS1 для C-субгеномов и А-субгеномов полиплоидных видов Avena. Результаты расчетов приведены в табл. 2. Во всех случаях среднее р-расстояние, измеряемое как доля различий в нуклеотидах между попарно сравниваемыми последовательностями, для последовательностей 18S рДНК (короткий фрагмент длиной 34–51 пн), ITS1 (226–250 пн) и 5.8S рДНК (короткий фрагмент длиной 10–34 пн) в С-субгеномах было на порядок выше, чем в А-субгеномах. При этом внутригеномный полиморфизм по SNPs в ITS1 во всех случаях был выше, чем в изученных фрагментах 18S и 5.8S рДНК, но разница в уровне полиморфизма между “смысловыми” участками гена 35S рРНК и спейсером для С-субгеномов во всех случаях была ниже, чем в А-субгеномах (табл. 2). Вероятно, это есть следствие стабилизирующего отбора в работающих генах 35S рРНК A‑субгеномов и ослабления (возможно, до полного отсутствия) влияния отбора в “репрессируемых” генах 35S рРНК, происходящих от диплоидных предков с С-геномами. Гипотеза эта требует проверки путем сравнения полных последовательностей рДНК A- и C-субгеномов.
Таблица 2.
Изменчивость генов 35S рРНК у аллополиплоидов в роде Avena
| Вид, субгеном | 18S рДНК 34–51 пн |
ITS1 226–250 пн |
Фрагмент 5.8S рДНК 10–34 пн |
|---|---|---|---|
| A. insularis, C-субгеном | 0.038 27 |
0.090 27 |
0.052 22 |
| A. insularis, A-субгеном | 0.004 800 |
0.012 801 |
0.008 578 |
| A. sterilis, растение 1, C-субгеном | 0.053 12 |
0.067 12 |
0.064 11 |
| A. sterilis, растение 1, A-субгеном | 0.003 773 |
0.013 773 |
0.004 507 |
| A. sterilis, растение 2, C-субгеном | 0.062 13 |
0.077 13 |
0.052 10 |
| A. sterilis, растение 2, A-субгеном | 0.004 815 |
0.011 815 |
0.004 515 |
| A. ludoviciana, растение 1, C-субгеном | 0.036 11 |
0.119 11 |
0.084 7 |
| A. ludoviciana, растение 1, А-субгеном | 0.005 778 |
0.010 780 |
0.005 551 |
| A. ludoviciana, растение 2, C-субгеном | 0.036 12 |
0.089 12 |
0.051 12 |
| A. ludoviciana, растение 2, A-субгеном | 0.004 746 |
0.013 746 |
0.008 484 |
| A. fatua, растение 1, C-субгеном | 0.049 21 |
0.101 21 |
0.057 11 |
| A. fatua, растение 1, A-субгеном | 0.004 852 |
0.013 852 |
0.006 553 |
| A. fatua, растение 2, C-субгеном | 0.050 17 |
0.102 17 |
0.026 9 |
| A. fatua, растение 2, A-субгеном | 0.004 759 |
0.016 759 |
0.007 525 |
Для того чтобы определить, к какому из современных диплоидных видов с рДНК С-типа ближе те рДНК С-типа полиплоидов, которые выявлены с помощью локус-специфичного секвенирования нового поколения, мы обработали результаты targeted-секвенирования “популяции” ITS-последовательностей полиплоидных геномов Avena с помощью программы TCS [33]. Алгоритм TCS основан на вероятностном методе статистической парсимонии и позволяет определять вероятность связи между всеми гаплотипами с индикацией числа мутаций, по которым они различаются. Результаты TCS-расчетов были обработаны в программе tcsBU [34, 35].
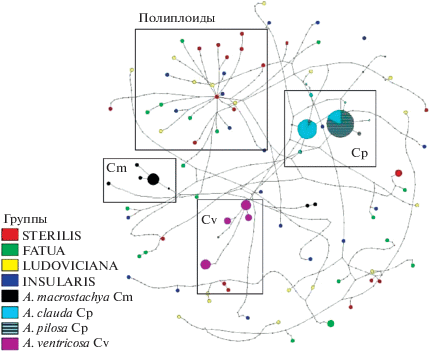
Из данных, представленных на рис. 2, однозначно следует, что A. pilosa (синоним A. eriantha) и A. clauda действительно имеют общий С-геном (Сp), отличный от С-генома типа Сv, характерного для A. ventricosa, и от Cm-генома A. macrostachya. С-субгеномы исследованных полиплоидных видов высоко изменчивы и разнообразны, но среди них можно выделить основной (core) вариант, примерно равноудаленный от ITS диплоидов, несущих С-геном, и от A. macrostachya.
Рис. 2.
Система риботипов С-субгеномов полиплоидных видов Avena и риботипов диплоидных видов Avena с геномами С-типа.
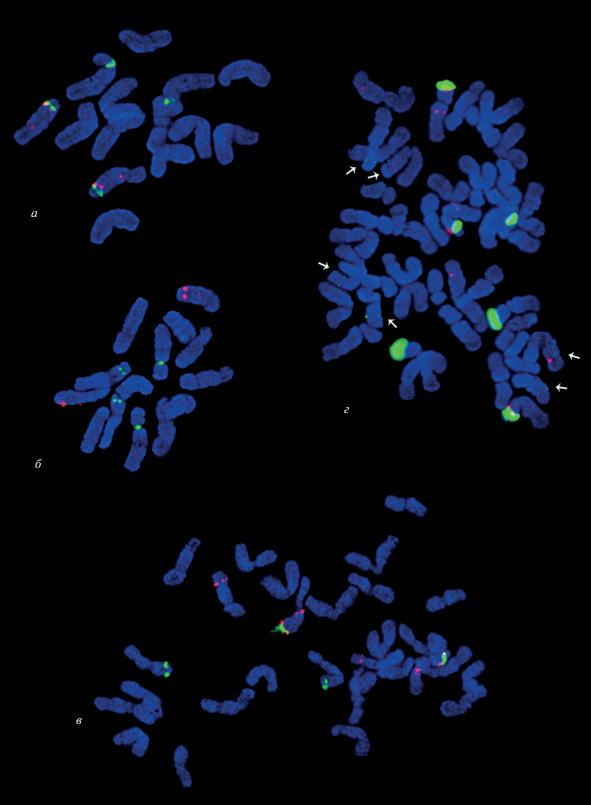
Результаты FISH-гибридизации с зондом, позволяющим выявлять гены 35S рРНК на хромосомах диплоидных видов с С-геномами и гексаплоидных видов с С-субгеномами, подтверждают тот факт, что у гексаплоида на хромосомах, пришедших от диплоида с кариотипом СС, произошла утрата значительной части генов 35S рРНК и 5S рРНК, так как на хромосомах, происходящих от С-геномных предков, они лежат в минорных сайтах, интенсивность сигнала на которых заметно меньше, чем на ЯОР и в локусах 5S рРНК диплоидов (рис. 3). Эти наблюдения полностью согласуются с ранее опубликованными данными [22, 46].
Рис. 3.
Флуоресцентная гибридизация in situ c пробами pTa71 (зеленый) и pTa794 (красный) на хромосомах видов овсов: а – А. longiglumis k-1811 (Al-геном), б – A. clauda K-2095 (С-геном), в – A. murphyi k-1986 (AC-геном), г – A. ludoviciana k-379 (ADC-геном). Стрелками указаны минорные сайты 35S рДНК.
Размеры минорных сайтов 35S рДНК на хромосомах, относящихся к С-субгеному, заметно меньше, чем размер локусов 35S рДНК на хромосомах A- и D-субгенома, однако их размер определенно больше, чем 4% от общей площади сигнала после FISH-гибридизации с зондом pTa71. Факт этот может означать, что часть генов 35S рРНК в минорных сайтах на хромосомах С-субгенома уже содержит рДНК А-типа благодаря конверсии или иным гипотетическим механизмам “concerted evolution”. Второе возможное объяснение: явление множественного мутирования в рДНК “репрессируемого” генома привело к тому, что какая-то часть рДНК С-типа уже не амплифицируется из-за некомплементарности стандартному набору праймеров, используемых для амплификации ITS1.
Сравнение паттернов рестрикции хлоропластной [47] и митохондриальной [48] ДНК показало, что пластиды и митохондрии гексаплоидных и тетраплоидных овсов получены от А-геномных, а не от С-геномных предков. Поскольку пластидная и митохондриальная ДНК у злаков наследуется по материнской линии [49], это означает, что С-субгеном произошел от предков со стороны отца. Филогенетический анализ, основанный на сравнении хлоропластных межгенных спейсеров trnL-trnF и psbA-trnH [17, 50], так же как мультиплексное секвенирование хлоропластного и митохондриального геномов [51], подтверждает умозаключение, что “репрессируемые” ЯОР, гены которых остались у полиплоидных Avena в минорных количествах, были получены их предками со стороны отца.
Наши результаты показывают, что потеря генов 35S рРНК “репрессируемого” ЯОР отцовского происхождения происходит на фоне накопления в этих последовательностях множества однонуклеотидных замен (SNPs) и делеций в рДНК С-субгеномов. То, что все без исключения последовательности ITS1 С-субгеномов были в наших выборках представлены единичными (уникальными) копиями, вероятно свидетельствуют о том, что в локусах 35S рРНК С-субгеномов не идут процессы гомогенизации рДНК – одного из механизмов явления “concerted evolution”, характерного для рДНК и других повторяющихся последовательностей [52–54]. Подчеркнем, что рДНК работающих генов, как правило, гомогенизированы в пределах каждого генома, хотя быстро изменяются в ходе дивергенции видов, различаясь по последовательностям даже близких видов [54, 55].
Есть все основания думать, что процессы изогенизации рДНК и процессы транскрипции/сайленсинга – связанные явления [10, 55–57]. Мыслимы, по крайней мере, два пути, обеспечивающих такую связь. Первый путь связан с тем, что в районах транскрибируемых генов повышена частота двунитевых разрывов и тем самым увеличена вероятность рекомбинационных событий [58, 59]. Гомогенизация рДНК при этом может происходить или в результате конверсии, или за счет мультипликации одной из копий 35S рДНК путем rolling-репликации и интеграции новообразованных копий рДНК в ЯОР, как это происходит при формировании HSR-районов [60].
Возможен и принципиально иной механизм гомогенизации повторов – уменьшение разнообразия 35S рРНК вследствие обратной связи между транскрибированной рРНК и ДНК-матрицей за счет механизмов, подобных механизмам редактирования генома созревающего макронуклеуса у инфузорий [61].
Исследование выполнено при финансовой поддержке РФФИ в рамках научного проекта № 17-00-00340 (17-00-00336, 17-00-00337, 17-00-00338).
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Van de Peer Y., Mizrachi E., Marchal K. The evolutionary significance of polyploidy // Nat. Rev. Genet. 2017. V. 18. P. 411–424. https://doi.org/10.1038/nrg.2017.26
Soltis D.E., Visger C.J., Marchant D.B., Soltis P.S. Polyploidy: pitfalls and paths to a paradigm // Am. J. Bot. 2016. V. 103. P. 1146–1166. https://doi.org/10.3732/ajb.1500501
Hu G., Wendel J.F. Cis–trans controls and regulatory novelty accompanying allopolyploidization // New Phytol. 2019. V. 221. P. 1691–1700. https://doi.org/10.1111/nph.15515
Родионов А.В., Амосова А.В., Беляков Е.А. и др. Генетические последствия межвидовой гибридизации, ее роль в видообразовании и фенотипическом разнообразии растений // Генетика. 2019. Т. 55. № 3. С. 255–272.
Родионов А.В. Межвидовая гибридизация и полиплоидия в эволюции растений // Вавилов. журн. генет. и селек. 2013. Т. 17. № 4(2). С. 916–929.
Родионов А.В., Носов Н.Н., Ким Е.С. и др. Происхождение полиплоидных геномов мятликов (Poa L.) и феномен потока генов между Северной Пацификой и субантарктическими островами // Генетика. 2010. Т. 46. № 12. С. 1598–1608.
Xiong Z., Gaeta R.T., Pires J. C. Homoeologous shuffling and chromosome compensation maintain genome balance in resynthesized allopolyploid Brassica napus // Proc. Natl Acad. Sci. USA. 2011. V. 108. P. 7908–7913. https://doi.org/10.1073/pnas.1014138108
Lipman M.J., Chester M., Soltis P.S., Soltis D.E. Natural hybrids between Tragopogon mirus and T. miscellus (Asteraceae): a new perspective on karyotypic changes following hybridization at the polyploid level // Am. J. Bot. 2013. V. 100. P. 2016–2022. https://doi.org/10.3732/ajb.1300036
Navashin M. Chromosomal alterations caused by hybridization and their bearing upon certain general genetic problems // Cytologia. 1934. V. 5. P. 169–203.
Kovarik A., Dadejova M., Lim Y.K. et al. Evolution of rDNA in Nicotiana allopolyploids: a potential link between rDNA homogenization and epigenetics // Ann. Bot. 2008. V. 101. P. 815–823. https://doi.org/10.1093/aob/mcn019
Matyášek R., Renny-Byfield S., Fulneček J. et al. Next generation sequencing analysis reveals a relationship between rDNA unit diversity and locus number in Nicotiana diploids // BMC Genomics. 2012. V. 13. P. 722. https://doi.org/10.1186/1471-2164-13-722
Long H., Chen C., Wang B., Feng Y. rDNA genetic imbalance and nucleolar chromatin restructuring is induced by distant hybridization between Raphanus sativus and Brassica alboglabra // PLoS One. 2015. V. 10(2). e0117198. https://doi.org/10.1371/journal.pone.0117198
Пунина Е.О., Мачс Э.М., Крапивская Е.Е. и др. Межвидовая гибридизация в роде Paeonia (Paeoniaceae): полиморфные сайты в транскрибируемых спейсерах генов 45S рРНК как индикаторы происхождения природных и искусственных гибридов пионов // Генетика. 2012. Т. 48. № 7. С. 812–826.
Родионов А.В., Добрякова К.С., Пунина Е.О. Полиморфные сайты в районе ITS1–5.8S рДНК–ITS2 у гибридогенного рода × Elyhordeum и предполагаемых межвидовых гибридов Elymus (Poaceae: Triticeae) // Генетика. 2018. Т. 54. № 9. С. 999–1014. https://doi.org/10.1134/S0016675818090126
Rodionov A.V., Gnutikov A.A., Kotsinyan A.R. et al. ITS1–5.8S rDNA–ITS2 sequence in 35S rRNA genes as a marker for reconstruction of phylogeny of grasses (Poaceae Family) // Biol. Bull. Reviews. 2017. V. 7. № 2. P. 85–102.
Родионов А.В., Тюпа Н.Б., Ким Е.С. и др. Геномная конституция автотетраплоидного овса Avena macrostachya, выявленная путем сравнительного анализа последовательностей ITS1 и ITS2: к вопросу об эволюции кариотипов овсов и овсюгов на ранних этапах дивергенции видов рода Avena // Генетика. 2005. Т. 41. № 5. С. 646–656.
Nikoloudakis N., Katsiotis A. The origin of the C-genome and cytoplasm of Avena polyploids // Theor. Appl. Genet. 2008. V. 117. P. 273–281. https://doi.org/10.1007/s00122-008-0772-9
Peng Y.Y., Baum B.R., Ren C.Z. et al. The evolution pattern of rDNA ITS in Avena and phylogenetic relationship of the Avena species (Poaceae: Aveneae) // Hereditas. 2010. V. 147. P. 183–204. https://doi.org/10.1111/j.1601-5223.2010.02172.x
Jellen E.N., Gill B.S., Cox T.S. Genomic in situ hybridization differentiates between A/D-and C-genome chromatin and detects intergenomic translocations in polyploid oat species (genus Avena) // Genome. 1994. V. 37. P. 613–618.
Irigoyen M.L., Loarce Y., Linares C. et al. Discrimination of the closely related A and B genomes in AABB tetraploid species of Avena // Theor. Appl. Genet. 2001. V. 103. P. 1160–1166.
Shelukhina O., Badaeva E., Loskutov I., Pukhal’sky V.A. A comparative cytogenetic study of the tetraploid oat species with the A and C genomes: Avena insularis, A. magna, and A. murphyi // Rus. J. Genet. 2007. V. 43. P. 613–626.
Badaeva E.D., Shelukhina O.Y., Diederichsen A. et al. Comparative cytogenetic analysis of Avena macrostachya and diploid C-genome Avena species // Genome. 2010. V. 53. P. 125–137. https://doi.org/10.1139/g09-089
Тюпа Н.Б., Ким Е.С., Лоскутов И.Г., Родионов А.В. К происхождению полиплоидов в роде Avena L.: молекулярно-филогенетическое исследование // Тр. по прикл. бот., ген. и селекции. 2009. Т. 165. С. 13–20.
Fominaya A., Loarce Y., Montes A., Ferrer E. Chromosomal distribution patterns of the (AC) 10 microsatellite and other repetitive sequences, and their use in chromosome rearrangement analysis of species of the genus Avena // Genome. 2016. V. 60. P. 216–227. https://doi.org/10.1139/gen-2016-0146
Rajhathy T., Thomas H.T. Chromosomal differentiation and speciation in diploid Avena // Can. J. Genet. Cytol. 1967. V. 9. P. 52–68.
Doyle J.J., Doyle J.L. A rapid DNA isolation procedure for small quantities of fresh leaf tissue // Phytochem. Bull. 1987. V. 19. P. 11–15.
Ridgway K.P., Duck J.M., Young J.P.W. Identification of roots from grass swards using PCR-RFLP and FFLP of the plastid trnL (UAA) intron // BMC Ecol. 2003. V. 3: 8. https://doi.org/10.1186/1472-6785-3-8
Stanford A.M., Harden R., Parks C.R. Phylogeny and biogeography of Juglans (Juglandaceae) based on matK and ITS sequence data // Am. J. Bot. 2000. V. 87. P. 872–882.
White T.J., Bruns T., Lee S., Taylor J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics // PCR Protocols: A Guide to Methods and Applications. 1990. V. 18. P. 315–322.
Bolger A.M., Lohse M., Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data // Bioinformatics. 2014. V. 30. P. 2114–2120. https://doi.org/10.1093/bioinformatics/btu170
Aronesty E. Comparison of sequencing utility programs // The Open Bioinformatics J. 2013. V. 7. № 1. https://doi.org/10.2174/18750362013070100011
Okonechnikov K., Golosova O., Fursov M. et al. Unipro UGENE: a unified bioinformatics toolkit // Bioinformatics. 2012. V. 28. P. 1166–1167. https://doi.org/10.1093/bioinformatics/bts0915
Kumar S., Stecher G., Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets // Mol. Biol. Evol. 2016. V. 33. P. 1870–1874. https://doi.org/10.1093/molbev/msw054
Clement M., Posada D.C.K.A., Crandall K.A. TCS: a computer program to estimate gene genealogies // Mol. Ecol. 2000. V. 9. P. 1657–1659.
Múrias dos Santos A., Cabezas M.P., Tavares A.I. et al. tcsBU: a tool to extend TCS network layout and visualization // Bioinformatics. 2015. V. 32. P. 627–628. https://doi.org/10.1093/bioinformatics/btv636
Badaeva E.D., Badaev N.S., Gill B.S., Filatenko A.A. Intraspecific karyotype divergence in Triticum araraticum (Poaceae) // Plant Syst. Evol. 1994. V. 192. P. 117–145. https://doi.org/10.1007/BF00985912
Amosova A.V., Bolsheva N.L., Samatadze T.E. et al. Molecular cytogenetic analysis of Deschampsia antarctica Desv. (Poaceae), Maritime Antarctic // PLoS One. 2015. V. 10(9): e0138878. https://doi.org/10.1371/journal.pone.0138878
Gerlach W.L., Bedbrook J.R. Cloning and characterization of ribosomal RNA genes from wheat and barley // Nucl. Acids Res. 1979. V. 7. P. 1869–1885. PMID:537913.https://doi.org/10.1093/nar/7.7.1869
Gerlach W.L., Dyer T.A. Sequence organization of the repeating units in the nucleus of wheat which contain 5S rRNA genes // Nucl. Acids Res. 1980. V. 8. P. 4851–4855.
Muravenko O.V., Yurkevich O.Y., Bolsheva N.L. et al. Comparison of genomes of eight species of sections Linum and Adenolinum from the genus Linum based on chromosome banding, molecular markers and RAPD analysis // Genetica. 2009. V. 135. P. 245–255. https://doi.org/10.1007/s10709-008-9273-7
Лоскутов И.Г. Межвидовые скрещивания в роде Avena L. // Генетика. 2001. Т. 37. № 5. С. 581–590.
Ladizinsky G. A new species of Avena from Sicily, possibly the tetraploid progenitor of hexaploid oats // Genet. Res. Crop Evol. 1998. V. 45. P. 263–269.
Jellen E.N., Ladizinsky G. Giemsa C-banding in Avena insularis Ladizinsky // Genet. Res. Crop Evol. 2000. V. 47. P. 227–230.
Zhou X., Jellen E.N., Murphy J.P. Progenitor germplasm of domisticated hexaploid oat // Crop Sci. 1999. V. 39. P. 1208–1214.
Loskutov I.G. On evolutionary pathways of Avena species // Genet. Res. Crop Evol. 2008. V. 55. P. 211–220.
Irigoyen M.L., Linares C., Ferrer E., Fominaya A. Fluorescence in situ hybridization mapping of Avena sativa L. cv. SunII and its monosomic lines using cloned repetitive DNA sequences // Genome. 2002. V. 45. P. 1230–1237.
Murai K., Tsunewaki K. Chloroplast genome evolution in the genus Avena // Genetics. 1987. V. 116. P. 613–621.
Rines H.W., Gengenbach B.G., Boylan K.L., Storey K.K. Mitochondrial DNA diversity in oat cultivars and species // Crop Sci. 1988. V. 28. P. 171–176.
Mogensen H.L. The hows and whys of cytoplasmic inheritance in seed plants // Am. J. Bot. 1996. V. 83. P. 383–404.
Peng Y.Y., Wei Y.M., Baum B.R. et al. Phylogenetic investigation of Avena diploid species and the maternal genome donor of Avena polyploids // Taxon. 2010. V. 59. P. 1472–1482.
Fu Y.B. Oat evolution revealed in the maternal lineages of 25 Avena species // Sci. Reports. 2018. V. 8(1). P. 4252. https://doi.org/10.1038/s41598-018-22478-4
Kovarik A., Matyasek R., Lim K.Y. et al. Concerted evolution of 18–5.8–26S rDNA repeats in Nicotiana allotetraploids // Biol. J. Linnean Soc. 2004. V. 82. P. 615–625.
Ganley A.R., Kobayashi T. Highly efficient concerted evolution in the ribosomal DNA repeats: total rDNA repeat variation revealed by whole-genome shotgun sequence data // Genome Res. 2007. V. 17. P. 184–191.
Eickbush T.H., Eickbush D.G. Finely orchestrated movements: evolution of the ribosomal RNA genes // Genetics. 2007. V. 175. P. 477–485.
Dadejová M., Lim K.Y., Soucková-Skalická K. et al. Transcription activity of rRNA genes correlates with a tendency towards intergenomic homogenization in Nicotiana allotetraploids // New Phytol. 2007. V. 174. P. 658–668.
Sochorová J., Coriton O., Kuderová A. et al. Gene conversion events and variable degree of homogenization of rDNA loci in cultivars of Brassica napus // Ann. Bot. 2016. V. 119. P. 13–26. https://doi.org/10.1093/aob/mcw187
Lunerová J., Renny-Byfield S., Matyášek R. et al. Concerted evolution rapidly eliminates sequence variation in rDNA coding regions but not in intergenic spacers in Nicotiana tabacum allotetraploid // Plant Syst. Evol. 2017. V. 303. P. 1043–1060.
Peng J.C., Karpen G.H. H3K9 methylation and RNA interference regulate nucleolar organization and repeated DNA stability // Nat. Cell Biol. 2007. V. 9. P. 25–35.
Gaillard H., Aguilera A. Transcription as a threat to genome integrity // Ann. Rev. Biochem. 2016. V. 85. P. 291–317. https://doi.org/10.1146/annurev-biochem-060815-014908
Okamoto H., Watanabe T.A., Horiuchi T. Double rolling circle replication (DRCR) is recombinogenic // Genes to Cells. 2011. V. 16. P. 503–513.
Noto T., Kataoka K., Suhren J.H. et al. Small-RNA-mediated genome-wide trans-recognition network in Tetrahymena DNA elimination // Mol. Cell. 2015. V. 59. P. 229–242. https://doi.org/10.1016/j.molcel.2015.05.024
Дополнительные материалы отсутствуют.


