Генетика, 2020, T. 56, № 6, стр. 667-676
Внутриродовая динамика размера генома у диплоидных семенных растений
А. М. Бутанаев 1, *, Л. Г. Немчинов 2, **
1 Пущинский научный центр биологических исследований, Институт фундаментальных проблем биологии
Российской академии наук
142290 Московская область, Пущино, Россия
2 Аграрный исследовательский центр Министерство агрокультуры Соединенных Штатов,
Служба исследования агрокультуры
МД, Белтсвиль, США
* E-mail: boutanaev@mail.ru
** E-mail: lev.nemchinov@ars.usda.gov
Поступила в редакцию 20.06.2019
После доработки 24.07.2019
Принята к публикации 03.10.2019
Аннотация
Размер генома растений зависит от многих факторов, действующих как внутри клетки, так и во внешней среде, что предполагает его значительную роль в адаптации и эволюции видов. Изучение размера генома и его изменений имеет длительную историю, тем не менее проблема все еще остается актуальной в связи с тем, что далеко не все факторы, влияющие на этот эволюционный процесс, известны. Несмотря на значительные усилия в исследовании изменения размера генома в основных эволюционных линиях растений, общая картина в настоящее время представляется статичной, поскольку не всегда принимается во внимание эволюционный возраст видов. В настоящей работе мы попытались на систематической основе изучить внутриродовую динамику размера генома у диплоидных семенных растений. В результате исследования была обнаружена неожиданно сильная линейная зависимость между размером генома и эволюционным возрастом видов, принадлежащих к одному и тому же роду. Такая линейность внутриродовой динамики размера генома прослеживается на протяжении миллионов лет, что скорее предполагает постепенные изменения в количестве ДНК и существенно меньшее влияние естественного отбора. Незначительное число видов выпало из общей линейной тенденции и, по-видимому, подверглось резким изменениям размера генома в течение короткого периода эволюционного времени. Таким образом, изменение размера генома внутри родов многих диплоидных семенных растений не случайно и следует линейной зависимости от эволюционного возраста видов.
Размер генома (РГ) эукариотических организмов является фундаментальной характеристикой, зависящей от многих внутриклеточных и внешних факторов и способной существенно влиять на адаптацию и эволюцию видов. По традиции размер гаплоидного генома в пикограммах ДНК называют значением “С” (С-value) или 1С. Происхождение этого термина рассматривается в [1, 2]. Так называемый парадокс значения “С” берет начало от того факта, что содержание ДНК у двух разных геномов с приблизительно одинаковым количеством генов может значительно отличаться. Однако уже давно стало очевидным, что в увеличение РГ растений и животных огромный вклад вносит некодирующая ДНК. Существует немало примеров адаптивного значения негенной ДНК, хотя ее пассивную роль также нельзя исключать (см. обзоры [3–9]). Позднее эту проблему стали рассматривать с точки зрения молекулярных механизмов, отвечающих за изменение количества ДНК, других внутриклеточных факторов, фенотипических признаков и факторов эволюции, влияющих на приобретение или потерю ДНК. В процессе эволюции у видов растений и животных происходило как увеличение, так и уменьшение количества ДНК.
Известна ассоциация РГ с размером клетки, длительностью клеточного цикла [10, 11] (см. обзор [12]), организацией хроматина в клетках животных [13] и метилированием ДНК у растений [14]. Из других характеристик, влияющих на РГ, можно назвать фенотипические признаки листовой стратегии (leaf strategy) [15], жизненный цикл растений [16], уровень сложности индивидуального развития [17] и увеличение сложности организации в процессе эволюции [18], а также интенсивность метаболизма у животных [19]. Имеются данные о связи РГ с ответом на стрессы у растений [20], образом жизни и продолжительностью генерации травянистых покрытосеменных [21], а также об ограничении РГ для больших геномов [22, 23]. Возможно влияние РГ на взаимодействие растений и растительноядных видов [24]. Функциональная эффективность генома в зависимости от его размера рассматривается в [25]. В работах [26, 27] обсуждали влияние РГ на видовую адаптацию, а в работе [28] – возможную роль небольших вставок и делеций в изменении РГ.
Полагают, что полиплоидия и размножение подвижных генетических элементов осуществляют основной вклад в увеличение содержания ДНК в клетке [29–31], в то время как потеря генетического материала связана с делециями посредством известных механизмов рекомбинации и репарации [32–35] (см. обзор [36]).
Исследование динамики РГ в царстве растений насчитывает длительную историю и в процессе исследований значительные усилия были предприняты для понимания возможных причин и последствий этого эволюционного процесса. В более поздних работах динамику РГ изучали в связи с филогенией отдельных таксонов. Многие исследования посвящены глобальным изменениям в содержании ДНК в основных таксономических линиях растений [37–40], а также изменениям, происходящим в отдельных семействах и родах [41–48]. Тем не менее общая картина изменений РГ у растений достаточно статична, поскольку временная составляющая, т.е. время дивергенции видов, по большому счету не рассматривалась. Основываясь на значительном объеме информации относительно РГ видов растений, принадлежащих к одному и тому же роду, и на их детальном филогенетическом датировании, мы провели анализ внутриродовой динамики РГ в значительном числе родов, принимая во внимание временную переменную величину.
МАТЕРИАЛЫ И МЕТОДЫ
База данных и компьютерное программирование
Величины размера генома растений получены из базы данных “C-value database” (Royal Botanic Gardens, Kew, UK. 1997; http://data.kew.org/cvalues) [49]. База имеет несколько полей, из которых в работе использовали следующие: “Family”, “Genus”, “Species”, “Ploidy level”, “C-value” и “Estimation method”. Компьютерная программа для обработки текстовой информации, специально созданная в рамках исследования для анализа базы данных, извлекала необходимую информацию и приписывала РГ каждого вида соответствующему роду и семейству.
Времена дивергенции видов и статистика
Для времен дивергенции видов использовали опубликованные калиброванные филогенетические деревья [50–72] (см. также дополнительную табл.: Supplementary Table S1 ). Каждое такое дерево имеет временную шкалу, которая позволяет найти время дивергенции отдельного вида. Так как филогенетические деревья публикуются, как правило, в виде графических образов без указания времени дивергенции, единственный способ его нахождения – прямое измерение длины ветвей дерева. Для этой цели использовали простой метод “линейки”. Прежде всего находили единицу шкалы (один миллион лет), измеренную в миллиметрах. Затем длину каждой ветви дендрограммы в миллиметрах делили на единицу шкалы. В результате получали время дивергенции конкретного вида, выраженное в миллионах лет. Надежность такого вынужденного подхода доказана, как будет видно из результатов, сильной ассоциацией между размером генома видов и их временем дивергенции, что было бы невозможно в том случае, если бы исходные данные, полученные прямым измерением длины ветвей дендрограммы, не воспроизводились (см. раздел “Результаты”).
Корреляционный анализ, регрессионный анализ и статистические критерии были выполнены с использованием программы SPSS Statistics 17.0.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
База данных значений “С” (С-values), т.е. размеров генома, на момент начала исследования насчитывала 190 семейств, 894 рода и 2988 видов диплоидных покрытосеменных растений [49]. Голосеменные были представлены в количестве 13 семейств, 81 рода и 312 видов. Поле базы данных “Ploidy level” использовали для того, чтобы исключить из анализа полиплоидные виды, поскольку существует возможность влияния полиплоидности на внутриродовую динамику РГ.
Далее был выполнен корреляционный анализ между значениями размера генома (пг ДНК) и временами дивергенции (млн лет) видов, принадлежащих к одному и тому же роду. Для того чтобы избежать двусмысленности в терминологии, в настоящем исследовании термины “время дивергенции видов” и “эволюционный возраст” использовались исключительно для значений времени дивергенции, которые или были опубликованы, или вычислены как указано выше, исходя из длин ветвей филогенетических деревьев. Необходимо отметить, что лишь рода с шестью или более видами были включены в анализ для большей репрезентативности выборки. В целом коэффициенты корреляции (r) были получены для 12 родов покрытосеменных и двух родов голосеменных растений, представляющих 10 семейств и 143 вида покрытосеменных, а также два семейства и 29 видов голосеменных растений (табл. 1). Примеры линейной зависимости между размером генома и временем дивергенции представлены на рис. 1. Количество видов в роду варьировало от 6 до 19. Несколько видов, явно выпадающих из общей линейной тенденции (8 из 172), были исключены из анализа, так как их значения радикально изменяли корреляцию (табл. 1 и рис. 1). Для абсолютного числа родов количество таких видов было несущественно (по одному в родах Acacia, Anthurium, Cistus и Vitis). Исключение составил род Araucaria, в котором исключенных видов было четыре. Таким образом, 9 родов из 14 вообще не содержали виды, не следующие общей линейной зависимости между их РГ и эволюционным временем (табл. 1).
Таблица 1.
Корреляция между временем дивергенции и размером генома видов внутри родов
| Семейство | Род | r | p | Виды | |
|---|---|---|---|---|---|
| в анализе | исключенные | ||||
| Покрытосеменные | |||||
| Двудольные | |||||
| Asteraceae | Artemisia | 0.64 | 0.003 | 16 | 0 |
| Cistaceae | Cistus | 0.50 | 0.03 | 15 | 1 |
| Fabaceae | Acacia | 0.85 | 0.002 | 9 | 1 |
| Rosaceae | Rosa1 | 0.26 | 0.47 | 10 | 0 |
| Vitaceae | Vitis | 0.64 | 0.02 | 11 | 1 |
| Rosaceae | Crataegus | –0.56 | 0.02 | 13 | 0 |
| Plumbaginaceae | Limonium | –0.92 | 0.005 | 6 | 0 |
| Rosaceae | Malus1 | –0.40 | 0.05 | 17 | 0 |
| Caprifoliaceae | Valerianella | –0.65 | 0.02 | 10 | 0 |
| Однодольные | |||||
| Araceae | Anthurium | 0.61 | 0.04 | 8 | 1 |
| Poaceae | Hordeum | 0.65 | 0.001 | 19 | 0 |
| Liliaceae | Lilium | 0.81 | 0.01 | 9 | 0 |
| Голосеменные | |||||
| Araucariaceae | Araucaria | 0.60 | 0.01 | 14 | 4 |
| Pinaceae | Pinus | 0.65 | 0.004 | 15 | 0 |
Примечание. Показано число видов, включенных в корреляционный анализ и исключенных из анализа по причине выпадения из линейной тенденции. r – коэффициент корреляции, p – уровень значимости r. 1 Корреляция статистически не достоверна (p ≥ 0.05; см. также рис. 1).
Рис. 1.
Примеры динамики размера генома в родах Hordeum, Pinus, Crataegus, Limonium, Rosa и Malus. Точки графика относятся к отдельным видам. Виды родов Hordeum и Pinus потеряли соответственно 37.6% ДНК за 5.0 млн лет и 38.9% ДНК за 18.2 млн лет; напротив, виды родов Crataegus и Limonium увеличили количество ДНК на 20.2% за 18.2 млн лет и на 72.9% за 3.3 млн лет. В родах Rosa и Malus корреляция отсутствует вследствие относительной стабильности размера генома в процессе эволюции этих родов (см. также табл. 1).
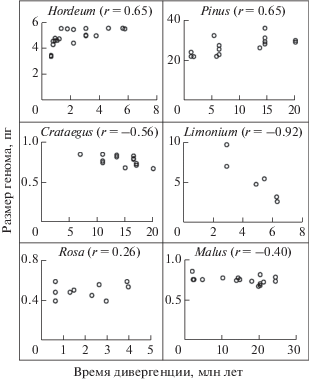
Значения коэффициентов корреляции были как положительными, так и отрицательными, т.е. имела место как прямая, так и обратная корреляция между изучаемыми величинами, что указывает на приобретение ДНК и на ее потерю в процессе эволюции видов внутри рода. Прямая корреляция указывает на то, что РГ уменьшался с уменьшением эволюционного возраста от прошлого к настоящему. Напротив, обратная корреляция указывает на то, что РГ увеличивался с уменьшением эволюционного возраста. Корреляция оказалась неожиданно сильной в большинстве родов как покрытосеменных, так и голосеменных растений. У покрытосеменных 8 родов из 12 имели абсолютные значения r ≥ 0.6. То же было справедливо и для двух родов голосеменных (табл. 1). Для родов Rosa и Malus значения r были статистически незначимы, по-видимому вследствие относительно стабильных РГ видов в пределах этих родов (табл. 1 и рис. 1).
Для аппроксимации эмпирических распределений к линейной модели использовали регрессионный анализ. В результате линейность обсуждаемой зависимости была подтверждена для 10 родов с уровнем значимости <0.05. Исключение составили Valerianella (p = 0.16) и Anthurium (p = 0.11). Два рода, Rosa и Malus, не рассматривали вследствие отсутствия корреляции Пирсона. Виды, не следующие общей внутриродовой тенденции изменения РГ, в терминах статистики представляют собой выбросы на графике зависимости РГ от времени дивергенции (рис. 2, слева). Поэтому для более формального основания исключения таких видов из анализа использовали стьюдентизированные остатки значений РГ родов Acacia, Cistus, Araucaria, Vitis и Anthurium (рис. 2, справа). Остатки для исключенных видов в родах Acacia, Cistus, Araucaria и Vitis попадают в интервал 2 < σ < 3, в то время как стьюдентизированные остатки значений РГ для остальных видов всех четырех родов находятся в интервале σ < 2 (рис. 2).
Рис. 2.
Динамика размера генома в родах с исключенными видами (обозначены символом “×”). Представлены графики зависимости размера генома от времени дивергенции видов (слева) и соответствующие графики стьюдентизированных остатков регрессии (справа). Виды, для которых значения остатков были больше двух сигм, принимали за выбросы и исключали из анализа (в случае Anthurium см. текст).
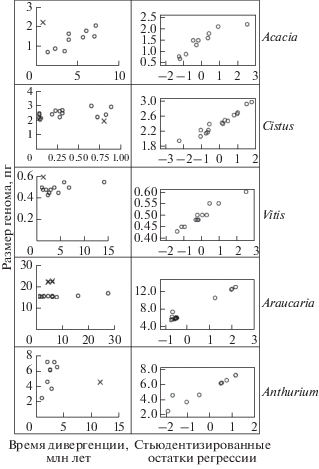
В статистике общепринято считать значения зависимой переменной выбросами, если их стьюдентизированные остатки больше трех сигм. Однако такая интерпретация в значительной степени подразумевает случайные отклонения. Как отмечалось выше, вряд ли правомерно считать исключенные виды результатом случайных ошибок измерения. В связи с этим представляется возможным принять за выбросы значения зависимой переменной (РГ), для которых стьюдентизированные остатки лежат в интервале σ > 2 и таким образом формализовать исключение из анализа видов, не следующих общей внутриродовой динамике РГ.
Род Anthurium представляет особый случай, иллюстрирующий необходимость принимать во внимание также значения временной переменной. Хотя остаток РГ для вида A. clidemioides (обозначен крестиком на рис. 2, слева) находится в интервале σ < 2, его время дивергенции значительно превосходит времена других видов этого рода, и если рассматривать остатки не РГ, а времени дивергенции, точка, соответствующая A. clidemioides, попадает в интервал 2 < σ < 3 и может также считаться выбросом.
РГ видов и их эволюционный возраст также использовали, чтобы вычислить три дополнительных параметра (табл. 2): 1) предполагаемое время внутриродового видообразования, которое представляет собой разность между максимальным и минимальным временем дивергенции видов внутри рода; 2) приобретение или потерю ДНК, что является увеличением или уменьшением РГ внутри рода, выраженных в процентах; 3) скорость приобретения или потери ДНК, выраженную в пикограммах ДНК на миллион лет (пг/млн лет).
Таблица 2.
Параметры внутриродовой потери и приобретения ДНК
| Линия | Порядок | Семейство | Род | Время
видообразования, млн лет |
Приобр./ потеря ДНК, % |
Скорость приобр./ потери ДНК, пг/млн лет | Приобр./ потеря |
|---|---|---|---|---|---|---|---|
| Покрытосеменные | |||||||
| Двудольные | |||||||
| Rosidae | Vitales | Vitaceae | Vitis | 12.9 | 9.1 | 0.004 | Пот. |
| Rosidae I | Rosales | Rosaceae | Crataegus | 13.0 | 20.2 | 0.013 | При. |
| Rosidae I | Rosales | Rosaceae | Malus | 22.1 | 8.2 | 0.003 | При. |
| Rosidae I | Rosales | Rosaceae | Rosa | 3.3 | 24.5 | 0.04 | Пот. |
| Rosidae I | Fabales | Fabaceae | Acacia | 5.7 | 65.9 | 0.24 | Пот. |
| Rosidae II | Malvales | Cistaceae | Cistus | 0.9 | 29.6 | 1.00 | Пот. |
| Asteridae | Caryophyllales | Plumbaginaceae | Limonium | 3.3 | 62.6 | 1.32 | При. |
| Asteridae II | Dipsacales | Caprifoliaceae | Valerianella | 16.8 | 18.5 | 0.003 | При. |
| Asteridae II | Asterales | Asteraceae | Artemisia | 5.9 | 23.3 | 0.88 | Пот. |
| Однодольные | |||||||
| Alismatales | Araceae | Anthurium | 2.8 | 64.3 | 1.7 | Пот. | |
| Liliales | Liliaceae | Lilium | 2.7 | 18.5 | 6.8 | Пот. | |
| Commel. | Poales | Poaceae | Hordeum | 5.0 | 37.6 | 0.41 | Пот. |
| Голосеменные | |||||||
| Pinales | Araucariaceae | Araucaria | 25.8 | 8.6 | 0.06 | Пот. | |
| Pinales | Pinaceae | Pinus | 18.2 | 38.9 | 2.14 | Пот. | |
Эти характеристики динамики РГ варьировали в широких пределах (табл. 2). У покрытосеменных, например, их значения составили соответственно 0.9–22.1 млн лет, 8.2–65.9% и 0.003–6.8 пг/млн лет, а у голосеменных – 18.2–25.8, 8.6–38.9 и 0.06–2.14. Так как период видообразования ограничен числом видов с известными РГ и калиброванными филогенетическими деревьями, характеристики динамики РГ, представленные в табл. 2, могут изменяться в обе стороны по мере того как больше видов будут доступны для анализа. Семейство Rosaceae включало более одного рода, что позволило сравнить разные рода в пределах одного семейства. Сравнение показало, что внутриродовая динамика РГ внутри семейства также весьма изменчива (табл. 2). Согласно критерию Стьюдента, в отношении второго и третьего параметров, достоверных различий между двудольными и однодольными, а также между покрытосеменными и голосеменными растениями не было. Однако значения первого параметра (предполагаемое время видообразования) достоверно различались при сравнении этих групп растений (р = 0.02). К тому же сравнение значений второго и третьего параметров между группами покрытосеменных растений, к одной из которых отнесли рода с увеличивающимся содержанием ДНК, а к другой – с противоположной тенденцией, не выявило статистически значимых различий. Другими словами, ни приобретение ДНК, ни ее потеря не преобладали среди родов покрытосеменных растений, что согласуется с результатами других исследователей [38, 41, 48]. Преобладание родов голосеменных с потерей ДНК, по-видимому, объясняется меньшим объемом данных для этой группы растений (табл. 2).
Линейная зависимость между РГ и временем дивергенции предполагает, что у большинства видов одного рода РГ изменялся в одном направлении и с близкими значениями скорости приобретения или потери ДНК. Тем не менее некоторые виды не следовали внутриродовой линейной динамике изменения РГ. История изучения значений “С” у растений насчитывает не один десяток лет, а используемые методы, такие как микроденситометрия и проточная цитометрия, доказали свою надежность [73, 74]. Таким образом, отличающаяся динамика РГ таких “выпадающих” видов, по-видимому, объясняется специфическими факторами их эволюции, а не случайным отклонением. Так, вариация значений “С” в базе данных для одного и того же вида незначительна. Например, для видов Araucaria cunninghamii, Brachypodium sylvaticum, Hydrangea scandens и Paeonia caucasica база содержит соответственно по две записи: 17.35, 17.85; 0.43, 0.44; 1.84, 2.01 и 15.91, 16.3. Для двух видов, Pinus nigra и Vitis aestivalis, база содержит соответственно семь и три записи в диапазонах 22.25–25.80 и 0.48–0.55. Значения для P. nigra были получены двумя разными методами: микроденситометрией и проточной цитометрией.
Близкие значения скорости изменения количества ДНК у видов одного рода в процессе видообразования, что следует из линейности динамики РГ, позволяют полагать, что у близкородственных видов внутриклеточные факторы вносят более значимый вклад в эту динамику по сравнению с факторами внешней среды. К подобному выводу можно прийти также, принимая во внимание тот факт, что однонаправленный линейный характер динамики РГ не менялся на протяжении миллионов лет эволюции видов. Маловероятно, что окружающая среда вида оставалась стабильной на протяжении такого длительного периода эволюционного времени с однонаправленным действием естественного отбора.
Необходимо отметить, что РГ относится к современным популяциям, а эволюционный возраст, т.е. время дивергенции видов, ко времени их возникновения. Таким образом, линейная зависимость между этими величинами была бы невозможна в случае значительных случайных вариаций РГ в процессе видообразования. Большинство исследований внутривидовых вариаций РГ у растений относятся к культивируемым видам и их близким родственникам. Такие вариации РГ были обнаружены у сои (15%) [75] и кукурузы (36%) [76]. Небольшую нестабильность РГ обнаружили среди сортов кукурузы и популяций ее дикорастущего родственника – теозинта (6.1% в обоих случаях) [77]. Незначительные вариации РГ нашли также среди культивируемых (7.8%) и дикорастущих (8.8%) популяций Artemisia arborescens [78] и у Cocos nucifera (1.7%) [79]. Напротив, РГ гороха, Pisum sativum, оказался стабилен с внутривидовой вариацией, не превышающей 1.054 раза [80].
Взрывной характер размножения LTR-ретротранспозонов трех семейств в геноме Oryza australiensis, дикорастущего родственника O. sativa, свидетельствует о подвижности РГ у растений [81]. Несмотря на то что у некоторых видов рода Anthurium обнаружили значительную внутривидовую вариацию РГ (>20%) [64], восемь из десяти видов, за исключением древнего вида A. clidemioides, следовали сильной линейной зависимости между их РГ и эволюционным возрастом, обнаруженной в настоящей работе (r = 0.61, р = 0.04; табл. 1).
Эти факты вместе с фактом внутриродовой линейной динамики указывают на относительно высокую стабильность содержания ДНК у большинства сформировавшихся видов диплоидных семенных растений. Хотя РГ может колебаться среди популяций, даже 20%-ная вариация, как у видов Anthurium, не влияет на внутриродовую корреляцию.
Наши результаты согласуются с моделью других исследователей, показавших, что изменение РГ может быть результатом постепенного накопления в геноме подвижных элементов и небольших делеций на протяжении многих миллионов лет [82]. В предположении этих авторов маловероятно, чтобы отбор действовал на небольшие изменения РГ, происходящие вследствие видоспецифичных механизмов его регулирования, таких как амплификация и делеции ДНК. В то же время значительные изменения количества ДНК могут служить точкой приложения отбора в то время, когда окружающая среда быстро меняется. В этом отношении наши результаты, т.е. линейность динамики РГ, ее родоспецифичность, сохранение этой линейности на протяжении многих миллионов лет и существование видов с динамикой РГ, отличающейся от линейной, согласуются с этой моделью. Это, соответственно, постепенные изменения РГ, видоспецифичные клеточные механизмы, накопление небольших изменений РГ в течение длительного эволюционного времени и внезапное селективное давление [82]. Нельзя также исключать возможность того, что негативный эффект резкого увеличения или потери ДНК, которые неизбежно приводят к дестабилизации коадаптированного генома, вносит свой вклад в постепенную линейную динамику РГ внутри родов.
Другая интерпретация полученных результатов основывается на гипотезе динамического хаоса в эволюции РГ [83]. Гипотеза предполагает существование положительной и отрицательной обратной связи в изменении количества ДНК в процессе видообразования. В этом случае положительная обратная связь поддерживает тенденцию изменения РГ в ту или другую сторону. Напротив, отрицательная обратная связь приводит к его стабилизации после завершения видообразования. Предполагается, что обратная связь существует в результате действия отбора на ферменты метаболизма ДНК, а через их функцию и на изменение РГ.
В настоящей работе не рассматривали факторы внешней среды, способные действовать на динамику РГ, хотя некоторые известные ассоциации упоминались в начале статьи. Линейный характер внутриродовой динамики РГ можно было бы связать с длительным изменением глобальной температуры, однако вероятность этого не слишком велика вследствие двунаправленных изменений количества ДНК у видов, существовавших примерно в одно и то же эволюционное время.
Некоторое препятствие в проведении подобного исследования состоит в том, что два пула исходной информации, т.е. РГ и филогенетические деревья, неодинаковы по своему объему. Данных по филогении родов с достаточным числом видов существенно меньше, поэтому большее количество такой информации позволит провести более точный и расширенный анализ. Рассматриваемые виды в основном принадлежат к двудольным растениям линий розид и астерид. Однодольные представлены тремя семействами трех порядков, а голосеменные включают только два семейства единственного порядка. Эти явные филогенетические бреши необходимо заполнить новыми данными в будущем. Возможно, что у полиплоидных видов растений динамика РГ более сложная и ее понимание позволило бы расширить наше представление об этом явлении. Сравнение внутриродовой динамики РГ между растениями и животными также представляет значительный интерес.
Таким образом, в результате изучения динамики размера генома внутри родов диплоидных семенных растений обнаружена неожиданно сильная линейная зависимость между размером генома и эволюционным возрастом видов, которая прослеживается на протяжении миллионов лет. Родоспецифичная линейная зависимость предполагает, что в постепенном изменении РГ более существенным фактором являются клеточные механизмы, а не селективное давление окружающей среды, как это обычно принято считать. Клеточные механизмы могут включать, например, видоспецифичность ферментов метаболизма ДНК, отвечающих, в том числе, за рекомбинацию и амплификацию ДНК. Можно предположить, что давление отбора проявляется при совпадении двух событий: значительного изменения количества ДНК и меняющейся окружающей среды. Напротив, те виды, динамика РГ которых отличается от линейной, могли подвергаться резкому изменению этого признака в течение короткого периода эволюционного времени до завершения видообразования или после него и таким образом претерпевать давление движущих сил эволюции. Влияние внешних факторов, возможно, становилось существенным в том случае, если отбор благоприятствовал значительному изменению содержания ДНК.
Работа Л.Г. Немчинова финансировалась United States Department of Agriculture, Agricultural Research Service, Beltsville, Maryland (CRIS project no. 8042-21000-268-00D).
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Bennett M.D., Leitch I.J. Plant Genome Size Research: A Field In Focus // Ann. Bot. 2005. V. 95. P. 1–6.
Greilhuber J., Dolezel J., Lysák M.A., Bennett M.D. The origin, evolution and proposed stabilization of the terms ‘genome size’ and ‘C-value’ to describe nuclear DNA contents // Ann. Bot. 2005. V. 95. P. 255–260.
Petrov D.A. Mutational equilibrium model of genome size evolution // Theor. Popul. Biol. 2002. V. 61. P. 531–544.
Muotri A.R., Marchetto M.C., Coufal N.G., Gage F.H. The necessary junk: new functions for transposable elements // Hum. Mol. Genet. 2007. V. 16. № 2. P. R159–R167.
Jurka J., Kapitonov V.V., Kohany O., Jurka M.V. Repetitive sequences in complex genomes: structure and evolution // Annu. Rev. Genomics Hum. Genet. 2007. V. 8. P. 241–259.
Jurka J. Conserved eukaryotic transposable elements and the evolution of gene regulation // Cell Mol. Life Sci. 2008. V. 65. № 2. P. 201–204.
Palazzo A.F., Gregory T.R. The case for junk DNA // PLoS Genet. 2014. V. 10. P. e1004351. https://doi.org/10.1371/journal.pgen.1004351
Dodsworth S., Leitch A.R., Leitch I.J. Genome size diversity in angiosperms and its influence on gene space // Curr. Opin. Genet. Dev. 2015. V. 35. P. 73–78. https://doi.org/10.1016/j.gde.2015.10.006
Pellicer J., Hidalgo O., Dodsworth S., Leitch I.J. Genome size diversity and its impact on the evolution of land plant // Genes (Basel). 2018. V. 9. № 2. P. E88. https://doi.org/10.3390/genes9020088
Cavalier-Smith T. Skeletal DNA and the evolution of genome size // Annu. Rev. Biophys. Bioeng. 1982. V. 11. P. 273–302.
Cavalier-Smith T. Economy, speed and size matter: evolutionary forces driving nuclear genome miniaturization and expansion // Ann. Bot. 2005. V. 95. P. 147–175.
Gregory T.R. Coincidence, coevolution, or causation? DNA content, cell size, and the C-value enigma // Biol. Rev. 2001. V. 7. P. 65–101.
Vinogradov A.E. Genome size and chromatin condensation in vertebrates // Chromosoma. 2005. V. 113. P. 362–369.
Alonso C., Pérez R., Bazaga P., Herrera C.M. Global DNA cytosine methylation as an evolving trait: phylogenetic signal and correlated evolution with genome size in angiosperms // Front. Genet. 2015. V. 6. P. 4. https://doi.org/10.3389/fgene.2015.00004
Morgan H.D., Westoby M. The relationship between nuclear DNA content and leaf strategy in seed plants // Ann. Bot. 2005. V. 96. P. 1321–1330.
Garnatje T., Vallès J., Garcia S. et al. Genome size in Echinops L. and related genera (Asteraceae, Cardueae): karyological, ecological and phylogenetic implications // Biology Cell. 2004. V. 96. P. 117–124.
Gregory T.R. Genome size and developmental complexity // Genetica. 2002. V. 115. № 1. P. 131–146.
Марков A.В., Анисимов В.А., Коротаев А.В. Взаимосвязь размера генома и сложности организма в эволюционном ряду от прокариот к млекопитающим // Палеонтол. журн. 2010. Т. 4. С. 3–14.
Vinogradov A.E., Anatskaya O.V. Genome size and metabolic intensity in tetrapods: a tale of two lines // Proc. Biol. Sci. 2006. V. 273. P. 27–32.
Chénais B., Caruso A., Hiard S., Casse N. The impact of transposable elements on eukaryotic genomes: from genome size increase to genetic adaptation to stressful environments // Gene. 2012. V. 509. P. 7–15. https://doi.org/10.1016/j.gene.2012.07.042
Leitch A.R., Leitch I.J. Ecological and genetic factors linked to contrasting genome dynamics in seed plants // New Phytol. 2012. V. 194. P. 629–646. https://doi.org/10.1111/j.1469-8137.2012.04105.x
Knight C.A., Molinari N.A., Petrov D.A. The large genome constraint hypothesis: evolution, ecology and phenotype // Ann. Bot. 2005. V. 95. P. 177–190.
Hidalgo O., Pellicer J., Christenhusz M. et al. Is there an upper limit to genome size? // Trends Plant Sci. 2017. V. 22. № 7. P. 567–573. https://doi.org/10.1016/j.tplants.2017.04.005
Guignard M.S., Crawley M.J., Kovalenko D. et al. Interactions between plant genome size, nutrients and herbivory by rabbits, molluscs and insects on a temperate grassland // Proc. Biol. Sci. 2019. V. 286(1899). P. 20182619. https://doi.org/10.1098/rspb.2018.2619
Шереметьев С.Н., Гамалей Ю.В., Слемнев Н.Н. Направления эволюции генома покрытосеменных // Цитология. 2011. Т. 53. № 4. С. 295–312.
Vinogradov A.E. Selfish DNA is maladaptive: evidence from the plant Red list // Trends Genet. 2003. V. 19. P. 609–614.
Petrov D.A. Evolution of genome size: new approaches to an old problem // Trends Genet. 2001. V. 17. P. 23–28.
Vitte C., Panaud O. LTR retrotransposons and flowering plant genome size: emergence of the increase/decrease model // Cytogenet. Genome Res. 2005. V. 110. P. 91–107.
Ågren J.A., Wright S.I. Co-evolution between transposable elements and their hosts: a major factor in genome size evolution? // Chromosome Res. 2011. V. 19. P. 777–786. https://doi.org/10.1007/s10577-011-9229-0
Zhao M., Ma J. Co-evolution of plant LTR-retrotransposons and their host genomes // Protein Cell. 2013. V. 4. P. 493–501. https://doi.org/10.1007/s13238-013-3037-6
Elliott T.A., Gregory T.R. Do larger genomes contain more diverse transposable elements? // BMC Evol. Biol. 2015. V. 5. P. 69. https://doi.org/10.1186/s12862-015-0339-8
Devos K.M., Brown J.K., Bennetzen J.L. Genome size reduction through illegitimate recombination counteracts genome expansion in Arabidopsis // Genome Res. 2002. V. 12. P. 1075–1079.
Bennetzen J.L. Mechanisms and rates of genome expansion and contraction in flowering plants // Genetica. 2002. V. 115. P. 29–36.
Orel N., Puchta H. Differences in the processing of DNA ends in Arabidopsis thaliana and tobacco: possible implications for genome evolution // Plant Mol. Biol. 2003. V. 51. P. 523–531.
Gregory T.R. Is small indel bias a determinant of genome size? // Trends Genet. 2003. V. 19. № 9. P. 485–488.
Puchta H. The repair of double-strand breaks in plants: mechanisms and consequences for genome evolution // J. Exp. Bot. 2005. V. 56. P. 1–14.
Leitch I.J., Chase M.W., Bennett M.D. Phylogenetic analysis of DNA C-values provides evidence for a small ancestral genome size in flowering plants // Annals Bot. 1998. V. 82. P. 85–94.
Soltis D.E., Soltis P.S., Bennett M.D., Leitch I.J. Evolution of genome size in the angiosperms // Am. J. Bot. 2003. V. 90. P. 1596–1603.
Leitch I.J., Soltis D.E., Soltis P.S., Bennett M.D. Evolution of DNA amounts across land plants (Embryophyta) // Ann. Bot. 2005. V. 95. P. 207–217.
Leitch I.J., Beaulieu J.M., Chase M.W. et al. Genome size dynamics and evolution in monocots // J. Botany. 2010. Article ID 862516. https://doi.org/10.1155/2010/862516
Wendel J.F., Cronn R.C., Johnston J.S., Price H.J. Feast and famine in plant genomes // Genetica. 2002. V. 115. P. 37–47.
Johnston J.S., Pepper A.E., Hall A.E. et al. Evolution of genome size in Brassicaceae // Ann. Bot. 2005. V. 95. № 1. P. 229–235.
Price H.J., Dillo S.L., Hodnett G. et al. Genome evolution in the genus Sorghum (Poaceae) // Ann. Bot. 2005. V. 95. P. 219–227.
Leitch I.J., Beaulieu J.M., Cheung K. et al. Punctuated genome size evolution in Liliaceae // J. Evol. Biol. 2007. V. 20. P. 2296–2308.
Lysak M.A., Koch M.A., Beaulieu J.M. et al. The dynamic ups and downs of genome size evolution in Brassicaceae // Mol. Biol. Evol. 2009. V. 26. P. 85–98. https://doi.org/10.1093/molbev/msn223
Leitch J., Kahandawala I., Suda J. et al. Genome size diversity in orchids: consequences and evolution // Ann. Bot. 2009. V. 104. P. 469–481. https://doi.org/10.1093/aob/mcp003
Pellicer J., Kelly L.J., Magdalena C., Leitch I.J. Insights into the dynamics of genome size and chromosome evolution in the early diverging angiosperm lineage Nymphaeales (water lilies) // Genome. 2013. V. 56. P. 437–449. https://doi.org/10.1139/gen-2013-0039
Pellicer J., Kelly L.J., Leitch I.J. et al. A universe of dwarfs and giants: genome size and chromosome evolution in the monocot family Melanthiaceae // New Phytol. 2014. V. 201. P. 1484–1497. https://doi.org/10.1111/nph.12617
Garcia S., Leitch I.J., Anadon-Rosell A. et al. Recent updates and developments to plant genome size databases // Nucl. Acids Res. 2014. V. 42. P. D1159–D1166. https://doi.org/10.1093/nar/gkt1195
Berry P.E., Hahn W.J., Sytsma K.J. et al. Phylogenetic relationships and biogeography of Fuchsia (Onagraceae) based on noncoding nuclear and chloroplast DNA data // Am. J. Bot. 2004. V. 91. P. 601–614.
Lledó M.D. Molecular phylogenetics of Limonium and related genera (Plumbaginaceae): biogeographical and systematic implications // Am. J. Bot. 2005. V. 92. № 7. P. 1189–1198.
Good-Avila S.V. Timing and rate of speciation in Agave (Agavaceae) // Proc. Natl Acad. Sci. USA. 2006. V. 103. P. 9124–9129.
Moore B.R., Donoghue M.J. Correlates of diversification in the plant clade Dipsacales: geographic movement and evolutionary innovations // Am. Nat. 2007. V. 170. Suppl. 2. P. S28–S55. https://doi.org/10.1086/519460
Qiao C.-Y., Ran J.H., Li Y., Wang X.Q. Phylogeny and biogeography of Cedrus (Pinaceae) inferred from sequences of seven paternal chloroplast and maternal mitochondrial DNA regions // Ann. Bot. 2007. V. 100. P. 573–580.
Guzmán B., Lledó M.D., Vargas P. Adaptive radiation in mediterranean Cistus (Cistaceae) // PLoS One. 2009. V. 4. P. e6362. https://doi.org/10.1371/journal.pone.0006362
Blattner F.R., Pleines T., Jakob S.S. Rapid radiation in the barley genus Hordeum (Poaceae) during the pleistocene in the americas // Evolution in Action / Ed. Glaubrecht M. Springer, 2010. P. 17–33. https://doi.org/10.1007/978-3-642-12425-9_2
Carlsen M.M. Understanding the Origin and Rapid Diversification of the Genus Anthurium Schott (Araceae), Integrating Molecular Phylogenetics, Morphology and Fossils // PhD Thesis, Graduate School Univ. Missouri, USA, 2011.
Poczai P. Molecular Genetic Studies on Complex Evolutionary Processes in Archaesolanum (Solanum, Solanaceae) // PhD Thesis, Univ. Pannonia, Hungary, 2011. http://konyvtar.uni-pannon.hu/doktori/2011/ Poczai_Peter_dissertation.pdf.
Sanz M., Schneeweiss G.M., Vilatersana R., Vallès J. Temporal origins and diversification of Artemisia and allies (Anthemideae, Asteraceae) // Collectanea Bot. 2011. V. 30. P. 7–15. https://doi.org/10.3989/collectbot.2011.v30.001
Ikinci N. Molecular phylogeny and divergence times estimates of Lilium section Liriotypus (Liliaceae) based on plastid and nuclear ribosomal ITS DNA sequence data // Turk. J. Bot. 2011. V. 35. P. 319–330. https://doi.org/10.3906/bot-1003-29
Guo Y.Y., Luo Y.B., Liu Z.J., Wang X.Q. Evolution and biogeography of the slipper orchids: eocene vicariance of the conduplicate genera in the old and new world tropics // PLoS One. 2012. V. 7. № 6. P. e38788. https://doi.org/10.1371/journal.pone.0038788
Lo E.Y.Y., Donoghue M.J. Expanded phylogenetic and dating analyses of the apples and their relatives (Pyreae, Rosaceae) // Mol. Phylogenet. Evol. 2012. V. 63. P. 230–243. https://doi.org/10.1016/j.ympev.2011.10.005
Heibl C., Renner S.S. Distribution models and a dated phylogeny for Chilean Oxalis species reveal occupation of new habitats by different lineages, not rapid adaptive radiation // Syst. Biol. 2012. V. 61. № 5. P. 823–834. https://doi.org/10.1093/sysbio/sys034
Bliss B.J., Suzuki J.Y. Genome size in Anthurium evaluated in the context of karyotypes and phenotypes // AoB Plants. 2012. P. pls006. https://doi.org/10.1093/aobpla/pls006
Díez C.M., Gaut B.S., Meca E. et al. Genome size variation in wild and cultivated maize along altitudinal gradients // New Phytol. 2013. V. 199. P. 264–276. https://doi.org/10.1111/nph.12247
Wan Y., Schwaninger H.R., Baldo A.M. et al. A phylogenetic analysis of the grape genus (Vitis L.) reveals broad reticulation and concurrent diversification during neogene and quaternary climate change // BMC Evol. Biol. 2013. V. 13. P. 141. https://doi.org/10.1186/1471-2148-13-141
McLeish M.J., Miller J.T., Mound L.A. Delayed colonisation of Acacia by thrips and the timing of host-conservatism and behavioural specialization // BMC Evol. Biol. 2013. V. 13. P. 188. https://doi.org/10.1186/1471-2148-13-188
Lockwood J.D., Aleksić J.M., Zou J. et al. A new phylogeny for the genus Picea from plastid, mitochondrial, and nuclear sequences // Mol. Phylogenet. Evol. 2013. V. 69. P. 717–727. https://doi.org/10.1016/j.ympev.2013.07.004
Wang B. Hybridization and Evolution in the Genus Pinus // PhD Thesis, Umeå Univ., Sweden, 2013. https://www.diva-portal.org/smash/get/diva2:652236/ FULLTEXT01.pdf.
Sherman-Broyles S., Bombarely A., Grimwood J. et al. Complete plastome sequences from Glycine syndetika and six additional perennial wild relatives of soybean // G3 (Bethesda). 2014. V. 4. P. 2023–2033. https://doi.org/10.1534/g3.114.012690
Kranitz M.L., Biffin E., Clark A. et al. Evolutionary diversification of New Caledonian Araucaria // PLoS One. 2014. V. 29. № 10. P. e110308. https://doi.org/10.1371/journal.pone.0110308
Fougère-Danezan M., Joly S., Bruneau A. et al. Phylogeny and biogeography of wild roses with specific attention to polyploids // Ann. Bot. 2015. V. 115. P. 275–291. https://doi.org/10.1093/aob/mcu245
Zonneveld B.J., Leitch I.J., Bennett M.D. First nuclear DNA amounts in more than 300 angiosperms // Ann. Bot. 2005. V. 96. № 2. P. 229–244.
Bai C., Alverson W.S., Follansbee A., Waller D.M. New reports of nuclear DNA content for 407 vascular plant taxa from the United States // Ann. Bot. 2012. V. 110. № 8. P. 1623–1629. https://doi.org/10.1093/aob/mcs222
Graham M.J., Nickell C.D., Rayburn A.L. Relationship between genome size and maturity group in soybean // Theor. Appl. Genet. 1994. V. 88. P. 429–432.
Realini M.F., Poggio L., Cámara-Hernández J., González G.E. Intra-specific variation in genome size in maize: cytological and phenotypic correlates // AoB Plants. 2015. V. 8. P. plv138. https://doi.org/10.1093/aobpla/plv138
Díez C.M., Gaut B.S., Meca E. et al. Genome size variation in wild and cultivated maize along altitudinal gradients // New Phytol. 2013. V. 199. № 1. P. 264–276. https://doi.org/10.1111/nph.12247
Garcia S., Garnatje T., Twibell J.D., Vallès J. Genome size variation in the Artemisia arborescens complex (Asteraceae, Arthemideae) and its cultivars // Genome. 2006. V. 49. P. 244–253.
Gunn B.F., Baudouin L., Beulé T. et al. Ploidy and domestication are associated with genome size variation in Palms // Am. J. Bot. 2015. V. 102. P. 1625–1633. https://doi.org/10.3732/ajb.1500164
Greilhuber J., Ebert I. Genome size variation in Pisum sativum // Genome. 1994. V. 37. № 4. P. 646–655.
Piegu B., Guyot R., Picault N. et al. Doubling genome size without polyploidization: dynamics of retrotransposition-driven genomic expansions in Oryza australiensis, a wild relative of rice // Genome Res. 2006. V. 16. P. 1262–1269.
Bennetzen J.L., Ma J., Devos K.M. Mechanisms of recent genome size variation in flowering plants // Ann. Bot. 2005. V. 95. P. 127–132.
Vinogradov A.E. Evolution of genome size: multilevel selection, mutation bias or dynamical chaos? // Curr. Opin. Genet. Dev. 2004. V. 14. P. 620–626.
Дополнительные материалы
- скачать ESM.xlsx
- Supplementary Table S1: Species, their c-values and divergence times.


