Генетика, 2020, T. 56, № 6, стр. 690-697
Асимметричная гибридизация калуги Acipenser dauricus Georgi, 1775 и амурского осетра A. sсhrenckii Brandt, 1869 (Acipenseridae) в природе согласно анализу митохондриального и ядерных ДНК-маркеров
С. В. Шедько 1, *, И. Л. Мирошниченко 1, Г. А. Немкова 1
1 Федеральный научный центр Биоразнообразия наземной биоты Восточной Азии Дальневосточного отделения Российской академии наук
690022 Владивосток, Россия
* E-mail: shedko@biosoil.ru
Поступила в редакцию 17.06.2019
После доработки 19.08.2019
Принята к публикации 22.08.2019
Аннотация
Естественная гибридизация симпатрично обитающих видов осетров – сравнительно частое, но малоизученное явление. Уникальная для Acipenseridae картина асимметричной гибридизации сложилась в р. Амур, где обитают два эндемика этого речного бассейна – калуга и амурский осетр. Ранее нами было показано, что доля их гибридов в разные годы составляет в среднем около 2.5% от численности родительских видов. Все проанализированные гибриды несли мтДНК лишь одного типа – калуги. Однако идентификация гибридов и чистых особей основывалась на рассмотрении одних лишь внешних морфологических характеристик. В настоящей работе набор генетических маркеров был расширен за счет включения в анализ шести ядерных локусов: двух интронов рибосомального белка L8 (RP4L8, RP5L8) и микросателлитов An20, Afug41, HLJSX37 и HLJSX350. В результате показано, что все морфологически промежуточные особи, ранее отнесенные на этом основании к гибридам, по каждому из ядерных локусов имели смешанный состав аллелей, характерных и для калуги, и для амурского осетра. Среди особей, по внешней морфологии определенных сборщиками как калуга, были выявлены три особи, которые по генетическим характеристикам должны быть отнесены к разряду гибридов. Кроме того, четыре другие особи были классифицированы как бэккроссы на калугу. Все гибриды и бэккроссы имели калужий тип мтДНК, что подтверждает ранее сделанное заключение об однонаправленной гибридизации калуги и амурского осетра в природе, которая проходит по схеме калуга (♀) × амурский осетр (♂). Использованный набор маркеров обладает необходимой разрешающей способностью и может быть рекомендован для идентификации чистых особей калуги и амурского осетра, а также их гибридов. Минимально достаточный для диагностики набор маркеров включает в себя мтДНК, интроны RP4L8 и RP5L8, а также микросателлит HLJSX37.
Межвидовая гибридизация – одно из заметных явлений, сопровождающих процесс эволюции. Роль гибридизации в видообразовании, адаптации и вымирании тех или иных групп организмов во многих случаях не вполне ясна, дискутируется и требует изучения [1].
Осетровые рыбы (Acipenseridae) – малочисленное (25 видов) семейство рыб, которое вместе с двумя видами семейства веслоносовых (Poly-odontidae) представляет собой рано (около 370 млн лет назад [2]) обособившуюся линию лучеперых (Actinopterygii) рыб. Гибриды между симпатрично обитающими видами осетровых рыб в природе встречаются довольно часто [3]. При этом в гибридизацию могут вступать виды, резко отличающиеся друг от друга как морфологически, так и экологически. К примеру [4], в бассейне Каспийского моря ярко выраженный хищник-ихтиофаг белуга (весом до 1500 кг) образует гибриды со всеми распространенными здесь видами осетров Acipenser, на порядок меньшими по массе и занимающими экологическую нишу бентофага-эврифага: стерлядью A. ruthenus L., 1758, севрюгой A. stellatus Pallas, 1771, шипом A. nudiventris Lovetsky, 1828 и русским осетром A. guelden-staedtii Brandt et Ratzeburg, 1833. Сам феномен легкости естественной гибридизации осетров, включая причины, по которым она происходит, и ее различные следствия, исследован недостаточно.
В бассейне р. Амур, как и в бассейне Каспия, наблюдается естественная гибридизация, с одной стороны, крупного рыбоядного хищника (достигает длины пять и более метров и веса свыше 1000 кг) – калуги Acipenser dauricus Georgi, 1775 и, с другой, намного меньшего по размеру бентофага (до трех метров длины и 150 кг веса) – амурского осетра A. sсhrenckii Brandt, 1869 (первое упоминание – [5], полную библиографию см. в [6]). Их гибриды в природе в разные годы составляют заметную величину – примерно 2.5% от общей численности родительских видов [6]. Сходная оценка доли гибридов найдена для осетров из р. Волга [7]. Среди других случаев естественной межвидовой гибридизации осетров [8–13] этот выделяется важной особенностью. Согласно недавнему исследованию [6], все морфологически промежуточные между калугой и амурским осетром особи (вероятные их гибриды) несли митохондриальную ДНК (мтДНК) лишь первого из двух видов. То есть, судя по этим данным, гибридизация калуги и амурского осетра в природе происходит лишь в одном направлении – калуга (♀) × амурский осетр (♂). Идентификация гибридов в указанной работе производилась на основе анализа нескольких морфологических характеристик. Однако известно [4], что осетровым рыбам в принципе свойствен высокий уровень индивидуальной изменчивости внешних морфологических признаков, а также структурных элементов внутреннего скелета. Данное обстоятельство могло вызывать сомнение в правильности произведенных выводов.
В задачу настоящего исследования входила проверка ранее сделанного заключения о направлении гибридизации калуги и амурского осетра в природе с использованием, в дополнение к мтДНК, шести генетических маркеров, кодируемых локусами ядерной ДНК и считающихся диагностическими для этих видов осетров.
МАТЕРИАЛЫ И МЕТОДЫ
Материалом для работы послужили образцы тотальной ДНК, полученные от 112 амурских осетров, 103 калуг, 16 их гибридов и использованные ранее для секвенирования контролирующего региона мтДНК [6, 14, 15]. К ним добавлены 25 образцов калуги, у которых этот участок мтДНК был секвенирован так, как описано в [6]. Все рыбы, от которых получены образцы, были собраны в ходе научного контрольного лова, проведенного Хабаровским филиалом ТИНРО-центра в нижнем течении р. Амур (район г. Николаевск-на-Амуре, примерно 53°7′ с.ш. 140°40′ в.д.) и Амурском лимане в летне-осенний сезон 2009–2011 гг.
Кроме мтДНК в набор диагностических для калуги и амурского осетра маркеров были включены интроны рибосомального белка L8 [16] и четыре микросателлитных локуса – Afug41 и An20 [17], HLJSX37 [18] и HLJSX350 [19].
Амплификацию фрагментов ДНК проводили на термоциклере Biometra Tprofessional (Biometra, Германия) или MiniAmp Plus (Applied Biosystems, США) в 25 мкл реакционной смеси, включавшей 0.1–0.2 мкг тотальной ДНК, 2.5 мкл буфера 67 мМ трис-HCl (pH 8.8), 16.6 мM (NH4)2SO4, 0.01% Tween-20, 0.75 мкл 50 мМ MgCl2, 0.5 мкл суммы дезокситрифосфатов из 10 мМ раствора (СибЭнзим, Новосибирск), по 2.5 мкл обоих праймеров (Beagle, Санкт-Петербург или Синтол, Москва) из их 2 мкМ растворов, 1 е.а. Taq-полимеразы (СибЭнзим) и деионизированную воду.
Микросателлитный локус Afug41 [20] амплифицировали с помощью праймеров Afug41_F (5-TGACGCACAGTAGTATTATTTATG-3) и Afug41_R (5-TGACGCACAGTAGTATTATTTATG-3) при следующих условиях: начальная денатурация (94°C – 120 с), 30 циклов (94°C – 25 с, 55°C – 60 с), достройка цепей (68°C – 180 с).
Фрагменты интронов рибосомального белка RP4L8 и RP5L8 амплифицировали с помощью праймеров RP4L8_H.dau_F (5-CAAGTTCAGAACACAAACAAAGGA-3) и RP4L8_H.dau_R (5-TGGACTATTTTCTCAAGACAAATGC-3), а также RP5L8_groupA_F (5-TCCGTGGTGTGGCTATGATT-3) и RP5L8_A.sch_R (5-AGTGCAATAAACTACTTCTGTG-3) [16] в одной мультиплексной реакции (добавляли по 1.5 мкл всех четырех праймеров из их 2 мкМ растворов) при тех же условиях, что и выше.
Микросателлитный локус HLJSX37 амплифицировали с помощью праймеров HLJSX37_F (5-TAGGAATCGCATCAGCAGTG-3) и HLJSX37_R (5-GTAAAAACCGCGAGTTCACA-3) [18] при следующих условиях: начальная денатурация (94°C – 120 с), 30 циклов (94°C – 25 с, 48°C – 60 с), достройка цепей (68°C – 180 с).
Микросателлитный локус HLJSX350 амплифицировали с помощью праймеров HLJSX350_F (ROX-5-AGTGAGCGAGGAAGCAAAAA-3) и HLJSX350_R (5-TGCCTGACTTATGGGAAACC-3) [19] при следующих условиях: начальная денатурация (94°C – 120 с), 34 цикла (94°C – 25 с, 68°C – 60 с), достройка цепей (68°C – 30 мин).
Микросателлитный локус An20 амплифицировали с помощью праймеров An20_F (R6G-5-AATAACAATCATTACATGAGGCT-3) и An20_R (5-TGGTCAGTTGTTTTTTTATTGAT-3) [21] при следующих условиях: начальная денатурация (94°C – 240 с), 5 циклов (94°C – 30 с, 65°C – 30 с, 72°C – 45 с) с понижением температуры отжига в каждом цикле на 1.5°C и затем 25 циклов (94°C – 30 с, 57°C – 30 с, 72°C – 45 с), достройка цепей (72°C – 30 мин).
Продукты реакции по локусам Afug41, RP4L8, RP5L8 и HLJSX37 выявляли с помощью рутинного электрофореза в 1.6–1.8%-ном агарозном геле с последующим окрашиванием бромистым этидием и экспозицией под ультрафиолетом.
Определение набора аллелей в локусах HLJSX350 и An20 производили следующим образом. Ампликоны разводили деионизированной водой в 6–9 раз, после чего 1 мкл переносили в смесь 10 мкл Hi-Di формамида и 0.4 мкл размерного стандарта СД-450 (24 одноцепочечных фрагмента ДНК длиной 60–450 пн, меченных флуоресцентным красителем DY632; Синтол). Затем смесь денатурировали и разгоняли на автоматическом лазерном генетическом анализаторе Нанофор-05 (Синтол) из приборной базы ФНЦ Биоразнообразия ДВО РАН (г. Владивосток). Полученные спектры далее обрабатывали в программе GeneMarker v. 3.0 (SoftGenetics, LLC, США).
Анализ различий особей по всему набору маркеров выполняли методом главных координат, реализованным в пакете программ NTSYS-pc [22]. Различия между особями были выражены через расстояние (1 – D)1/2, где D – индекс сходства Дайса [23], равный в нашем случае отношению удвоенного числа идентичных аллелей у рассматриваемой пары особей к общему числу аллелей у них.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Результаты анализа 256 особей осетровых из р. Амур суммированы в табл. 1.
Таблица 1.
Встречаемость аллелей диагностических локусов у особей, идентифицированных по морфологическим признакам как амурский осетр, калуга или их гибриды
| Маркер | Аллели* | Амурский осетр(n = 112) | Гибриды (n = 16) | Калуга (n = 128) |
F-тест (p)** | |||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ядро выборки | x1 | x2 | x3 | x4 | x5 | x6 | x7 | |||||
| мтДНК | DAU | 0 | 16 | 121 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | ⪡0.001 |
| SCH | 112 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ||
| RP4L8 | 213 | 1 | 16 | 121 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | ⪡0.001 |
| RP5L8 | 146 | 112 | 16 | 12 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | ⪡0.001 |
| Afug41 | 185–225 | 112 | 16 | 13 | 1 | 1 | 1 | 0 | 1 | 0 | 0 | ⪡0.001 |
| HLJSX37 | 122 | 112 | 16 | 0 | 1 | 1 | 0 | 1 | 0 | 0 | 0 | ⪡0.001 |
| HLJSX350 | 218 | 14 | 16 | 121 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | ⪡0.001 |
| 235 | 112 | 16 | 19 | 1 | 1 | 1 | 1 | 0 | 1 | 0 | ||
| 239 | 23 | 3 | 18 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ||
| 243 | 2 | 1 | 9 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | ||
| An20 | 137 | 109 | 15 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | ⪡0.001 |
| 150 | 23 | 9 | 79 | 1 | 1 | 0 | 1 | 0 | 1 | 1 | ||
| 154 | 59 | 3 | 9 | 1 | 1 | 0 | 0 | 1 | 0 | 0 | ||
| 156 | 3 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ||
| 158 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ||
| 162 | 14 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | ||
| 166 | 1 | 12 | 84 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | ||
| 170 | 6 | 10 | 112 | 1 | 1 | 1 | 0 | 1 | 0 | 0 | ||
| 174 | 2 | 0 | 11 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ||
| 178 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ||
| 183 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ||
| 195 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ||
По типу мтДНК [6, 14, 15] все образцы распадаются на две группы: образцы амурского осетра, с одной стороны, и калуги с гибридами – с другой (табл. 1).
Микросателлитный локус Afug41 [20] был предложен в качестве диагностического для амурского осетра Барминцевой и Мюге [17]. Проведенное этими авторами исследование 58 амурских осетров из искусственной популяции, 111 калуг (из природы – 48, из рыбоводных хозяйств – 63) показало, что фрагменты ДНК длиной 185–225 пн присутствуют только у амурского осетра. Позднее были получены несколько отличающиеся результаты: положительная амплификация была найдена для восьми из 54 образцов (15%) калуг из природы и для двух из 20 образцов (10%) калуг из рыбоводных хозяйств [16]. В наших выборках положительный сигнал (присутствие указанных фрагментов) наблюдался у всех особей амурских осетров и гибридов, а также у 13.3% особей, определенных по морфологическим признакам как калуга (табл. 1).
Согласно недавнему исследованию [16], интроны рибосомального белка L8 – RP4L8 и RP5L8 – могут рассматриваться как диагностические, соответственно для калуги (положительная амплификация фрагмента длиной 213 пн) и амурского осетра (фрагмент длиной 146 пн). Аллель RP4L8 (213) был обнаружен у 100% калуг и у 20 из 213 (9.4%) диких амурских осетров [16]. В наших выборках он присутствовал у 100% особей, идентифицированных как калуга, у всех гибридов, у одного из 112 амурских осетров (табл. 1). Аллель RP5L8 (146) был обнаружен у 100% диких амурских осетров и у 17 из 54 (31.5%) диких калуг [16]. В наших выборках он присутствовал у 100% особей амурского осетра, у всех гибридов, а также у 14.1% особей, определенных по морфологическим признакам как калуга (табл. 1).
Микросателлитный локус HLJSX37 считается диагностическим для амурского осетра – фрагмент длиной 122 пн присутствует у особей этого вида, но не калуги [18]. Маркер был предложен по результатам анализа небольшой выборки: 8 амурских осетров, 12 калуг и 12 их гибридов [18]. В наших выборках данный фрагмент присутствовал у 100% особей амурского осетра, у всех гибридов, а также у трех из 128 особей, определенных как калуга.
Микросателлитный локус HLJSX350 был предложен в качестве диагностического для амурского осетра (имеется фрагмент длиной 227 пн) и калуги (фрагмент длиной 209 пн) по результатам анализа 15 амурских осетров, 15 калуг и 15 их гибридов [19]. По нашим данным, длина маркерных аллелей в локусе HLJSX350 составляет 218 и 235, а не 209 и 227 пн. Различие вызвано, вероятно, недостаточно высокой точностью методики расчета длин фрагментов, использованной Лиу с соавт. [19]: продукты амплификации в их случае разгонялись с помощью ординарного электрофореза в полиакриламидном геле, а в качестве размерного стандарта использовался маркер с большим шагом (в данном интервале длин его шаг составлял 100 пн). Аллель HLJSX350 (218) наблюдался у всех образцов калуги и 12.5% амурских осетров, а аллель HLJSX350 (235) – у всех образцов амурского осетра и 18.8% особей, определенных как калуга (табл. 1). У всех гибридов были выявлены оба аллеля.
Микросателлитный локус An20 [21] был предложен [17] в качестве диагностического для амурского осетра (имеется фрагмент длиной 137 пн, который у калуги не обнаружен) и калуги (имеется фрагмент длиной 169 пн, у амурского осетра редок) по результатам анализа того же материала, что и по локусу Afug41 (см. выше). По нашим данным, длина маркерного для калуги аллеля в локусе An20 составляет 170, а не 169 пн. В работе [17], к сожалению, не указано какой размерный стандарт был применен, но приведено название использованных систем капиллярного электрофореза – “ABI3100/3130 Genetic analyzer”. Для этих приборов в типичном размерном диапазоне микросателлитов (до 400 пн) обычно применяется стандарт длин LIZ-500. Использованный нами размерный стандарт СД-450, в отличие от LIZ-500, имеет большую плотность маркерных фрагментов (24 против 16) и более равномерное их распределение с шагом в 20 пн (у LIZ-500 в этой части размерного диапазона шаг составляет 10–50 пн – 150, 160, 200, 250 пн). Возможно, именно это и обусловило отличие в оценках длины указанного фрагмента. Диагностический для амурского осетра фрагмент ранее был найден у 99.1% исследованных особей данного вида и 5.6% диких калуг [16]. В наших выборках он выявлен у 97.3% особей амурского осетра и 14.1% калуг. Диагностический для калуги фрагмент ранее был найден у всех 54 диких калуг и 4.2% особей амурского осетра [16]. В наших выборках он обнаружен у 90.6% особей, определенных как калуга, и 5.4% особей амурского осетра (табл. 1). Укажем на то, что по нашим данным диагностическими могут считаться не только пара аллелей 137 и 170, но и несколько других: 150, 166 – для калуги и 154, 162 – для амурского осетра. У гибридов были выявлены все перечисленные аллели (табл. 1).
При сравнении амурского осетра и калуги для всех маркеров было установлено статистически значимое (точный критерий Фишера) отличие в частоте встречаемости аллелей (табл. 1).
Анализ наших выборок осетровых из р. Амур методом главных координат с использованием интегрального показателя – расстояния Дайса, рассчитанного по всем семи использованным маркерам, привел к следующим результатам (рис. 1).
Рис. 1.
Распределение особей, идентифицированных по морфологическим признакам как калуга (◼), амурский осетр (⚫) или их гибриды (◻), в пространстве первых двух главных координат.
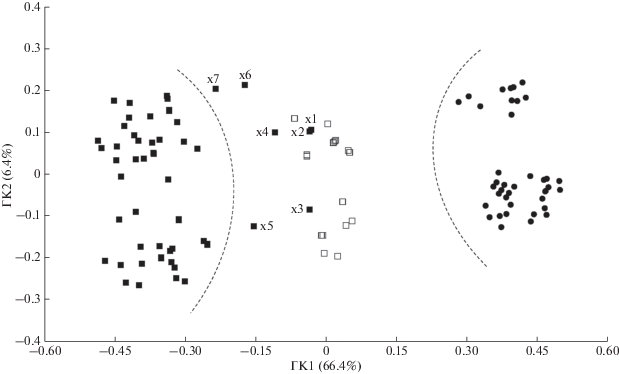
Особи амурского осетра и основная часть выборки калуги по оси первой главной координаты сформировали две группы, условные границы которых на рисунке обозначены пунктирными линиями. Образцы гибридов образовали собственную группу, расположенную примерно посередине между первыми двумя.
Особей, определенных по морфологическим признакам как калуга, можно разделить на ядро выборки и отстоящих от него особей, попадающих в группу гибридов или располагающихся между последними и ядром. Часть таких уклоняющихся особей (x1–x3), по всей видимости, должна быть отнесена к гибридам F1. Остальные (x4–x7), вероятно, являются бэккроссами на калугу (образец x4 отнесен к ним, поскольку располагался за пределами полигона распределения гибридов). Образцы x1–x3 происходят из небольшой выборки образцов калуги, собранной сотрудниками ХфТИНРО-центра (г. Хабаровск) в ночное время. Этих калуг, к сожалению, не удалось фотодокументировать по полному набору морфологических характеристик, необходимых для выявления гибридов [6]. Скорее всего, эти особи были неверно определены. Особи x4–x7 по морфологическим признакам были сходны с типичными калугами.
Ранее нами было установлено, что гибридизация амурского осетра и калуги асимметрична, поскольку все особи, по морфологическим признакам отнесенные к гибридам, имели калужий тип мтДНК [6]. Так как особи x1–x3 также имели разные калужьи мтДНК-гаплотипы (DAU9, DAU6 и DAU23 соответственно), плюс к тому же были разного размера (их длина тела в см – 179, 170, 199), то можно заключить, что произошли они в результате трех независимых актов гибридизации калуга (♀) × амурский осетр (♂), не идентичных тем 16, что установлены ранее [6]. Из этих данных следует, что в настоящий период времени вероятность встретить гибридов F1 амурский осетр (♀) × калуга (♂) в природе составляет примерно 1/20 или менее вероятности встречи гибридов калуга (♀) × амурский осетр (♂). Таким образом, новые данные, полученные при анализе ядерной ДНК, укрепляют нас в правильности сделанного ранее [6] заключения относительно преимущественной однонаправленности гибридизации амурского осетра и калуги. В согласии с ним находятся и результаты анализа двух гибридов калуги и амурского осетра, отловленных в 2001 и 2015 гг. у побережья о-ва Хоккайдо, имевших калужий тип мтДНК и смешанный состав аллелей микросателлитного локуса An20 [24]. Факторы, обусловливающие асимметричную гибридизацию этих видов осетров, не ясны [6] и могут составить предмет будущих исследований.
Тот факт, что у амурского осетра найдены аллели ядерных генетических маркеров, свойственные калуге, и наоборот (табл. 1), может рассматриваться как свидетельство того, что эти виды осетров, тем не менее, могут обмениваться участками ядерного генома в обоих направлениях. Однако аналогичная картина может возникать и в случае сохранения предкового полиморфизма. Чтобы исключить последнее, необходимо исследовать изменчивость этих маркеров у сестренских для амурского осетра и калуги видов – белого осетра A. transmontanus Richardson, 1836, с одной стороны, и зеленого A. medirostris Ayeres, 1854 или сахалинского A. mikadoi Hilgendorf, 1892 осетра, с другой.
Как известно, с конца 90-х гг. прошлого века в китайской части бассейна Амура получило интенсивное развитие промышленное производство гибридов калуги и амурского осетра [25]. Массовый преднамеренный или случайный (в результате, к примеру, наводнения, что не редкость в данном регионе, подверженном воздействию мощных тайфунов) выпуск в природу таких гибридов может представлять угрозу существованию естественных популяций этих видов, численность которых сейчас находится на уровнях их исторических минимумов [14, 15]. В связи с этим мониторинг частоты встречаемости гибридов калуги и амурского осетра в природе с помощью генетических методов представляется актуальной задачей.
Основываясь на полученных результатах (табл. 1, рис. 1), нами был произведен подбор минимального набора маркеров, позволяющего проводить более или менее уверенное разделение амурских осетров и калуг, а также выявлять их гибриды. Таковым оказалась панель, включающая в себя следующие маркеры: мтДНК, RP4L8, RP5L8 и микросателлитный локус HLJSX37. Идентификация образцов с помощью этой панели может производиться с использованием простого индекса. Он рассчитывается для каждой особи отдельно путем кодирования присутствия/отсутствия аллелей DAU и RP4L8 (213) – в виде (1/0), аллелей SCH, RP5L8 (146), HLJSX37 (122) – в виде (–1/0) и последующего арифметического суммирования кодов по всем четырем маркерам. Расчет индекса для наших выборок выявил следующую картину. Для особей амурского осетра описанный индекс был равен –3 во всех случаях за исключением одного, когда он оказался равным –2. Для особей калуги индекс был равным двум (88%), реже единице (11%), как исключение – нулю (особь x4). То есть “чистые” особи амурского осетра и калуги идентифицируются со 100%-ной эффективностью. Для гибридов индекс был равен нулю (как исключение единице – особь x3). Таким образом, ошибка может составлять примерно 1% случаев и только лишь при разделении гибридов и особей калуги. Привлекательной стороной данной панели является то, что для работы с этими маркерами не требуется использования дорогостоящих систем капиллярного электрофореза и выявление изменчивости по ним может производиться с помощью обычной аппаратуры для электрофореза в агарозном геле.
Исследование проводилось в рамках темы НИР № АААА-А17-117062710083-0. Часть работ (анализ микросателлитов) выполнена при финансовой поддержке Программы “Приоритетные научные исследования в интересах комплексного развития Дальневосточного отделения РАН” (грант № 18-4-032).
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Arnold M.L. Natural Hybridization and Evolution. N.Y.: Oxford Univ. Press, 1997. 232 p.
Near T.J., Eytan R.I., Dornburg A. et al. Resolution of ray-finned fish phylogeny and timing of diversification // Proc. Natl Acad. Sci. USA. 2012. V. 109. № 34. P. 13698–13703. https://doi.org/10.1073/pnas.1206625109
Birstein V.J., Hanner R., DeSalle R. Phylogeny of the Acipenseriformes: cytogenetic and molecular approaches // Environ. Biol. Fishes. 1997. V. 48. P. 127–155.
Берг Л.С. Фауна России и сопредельных стран. Рыбы (Marsipobranchii и Pisces). Т. 3. Ostariophysi. Вып. 1. СПб.: Изд-во Имп. акад. наук, 1912. 336 с.
Солдатов В.К. Исследования осетровых Амура // Материалы к познанию русского рыболовства. 1915. Т. 3. Вып. 12. С. 96–415.
Шедько С.В., Шедько М.Б. Однонаправленная гибридизация калуги Acipenser dauricus Georgi, 1775 и амурского осетра A. sсhrenckii Brandt, 1869 по данным мтДНК-типирования их природных гибридов // Генетика. 2016. Т. 52. № 3. С. 332–338. https://doi.org/10.7868/S0016675816020132
Lagunova V.S. The impact of conditions in the Volga River on hybridization, abnormal development and reproduction rate of sturgeon (Acipenseridae) // J. Appl. Ichthyol. 1999. V. 15. № 4–5. P. 291. https://doi.org/10.1111/j.1439-0426.1999.tb00275.x
Tranah G., Campton D.E., May B. Genetic evidence for hybridization of Pallid and Shovelnose sturgeon // J. Hered. 2004. V. 95. № 6. P. 474–480. https://doi.org/10.1093/jhered/esh077
Schrey A.W., Boley R., Heist E.J. Hybridization between Pallid sturgeon Scaphirhynchus albus and Shovelnose sturgeon Scaphirhynchus platorynchus // J. Fish Biol. 2011. V. 79. № 7. P. 1828–1850. https://doi.org/10.1111/j.1095-8649.2011.03123.x
Eichelberger J.S., Braaten P.J., Fuller D.B. et al. Novel single-nucleotide polymorphism markers confirm successful spawning of endangered Pallid sturgeon in the Upper Missouri River Basin // Trans. Am. Fish. Soc. 2014. V. 143. № 6. P. 1373–1385. https://doi.org/10.1080/00028487.2014.935479
Dudu A., Suciu R., Paraschiv M. et al. Nuclear markers of Danube sturgeons hybridization // Int. J. Mol. Sci. 2011. V. 12. № 10. P. 6796–6809. https://doi.org/10.3390/ijms12106796
Chassaing O., Desse-Berset N., Duffraisse M. et al. Palaeogenetics of western French sturgeons spotlights the relationships between Acipenser sturio and Acipenser oxyrinchus // J. Biogeogr. 2013. V. 40. № 2. P. 382–393. https://doi.org/10.1111/j.1365-2699.2012.02785.x
Li J., Liu D., Ma Q. et al. Discriminating Dabry’s sturgeon (Acipenser dabryanus) and Chinese sturgeon (A. sinensis) based on DNA barcode and six nuclear markers // Hydrobiologia. 2015. V. 757. № 1. P. 185–196. https://doi.org/0.1007/s10750-015-2251-z
Шедько С.В., Мирошниченко И.Л., Немкова Г.А. и др. Изменчивость митохондриальной ДНК, историческая демография и популяционная структура амурского осетра Acipenser sсhrenckii Brandt, 1869 // Генетика. 2015. Т. 51. № 2. С. 200–216. https://doi.org/10.7868/S0016675815020125
Шедько С.В., Мирошниченко И.Л., Немкова Г.А., Шедько М.Б. К популяционно-генетическому портрету калуги, Acipenser dauricus Georgi, 1775: анализ изменчивости контролирующего региона митохондриальной ДНК // Генетика. 2015. Т. 51. № 9. С. 1025–1034. https://doi.org/10.7868/S0016675815080093
Boscari E., Barmintseva A., Zhang S. et al. Genetic identification of the caviar-producing Amur and Kaluga sturgeons revealed a high level of concealed hybridization // Food Control. 2017. V. 82. P. 243–250. https://doi.org/10.1016/j.foodcont.2017.07.001
Барминцева А.Е., Мюге Н.С. Использование микросателлитных локусов для установления видовой принадлежности осетровых (Acipenseridae) и выявления особей гибридного происхождения // Генетика. 2013. Т. 49. № 9. С. 1093–1105. https://doi.org/10.7868/S0016675813090038
Hu J., Wang D., Wei Q., Shen L. Molecular identification of Amur sturgeon (Acipenser schrenckii), Kaluga (Huso dauricus) and their reciprocal hybrids // J. Fish. Sci. China. 2010. V. 17. № 1. P. 21–30.
Liu Y., Lu C., Li C. et al. Genetic identification of Amur sturgeon, Kaluga and their hybrids based on microsatellite markers // Chinese J. Fish. 2015. V. 28. № 1. P. 18–23.
Welsh A.B., Blumberg M., May B. Identification of microsatellite loci in lake sturgeon, Acipenser fulvescens, and their variability in green sturgeon, A. medirostris // Mol. Ecol. Notes. 2002. V. 3. № 1. P. 47–55. https://doi.org/10.1046/j.1471-8286.2003.00346.x
Zane L., Patarnello T., Ludwig A. et al. Isolation and characterization of microsatellites in the Adriatic sturgeon (Acipenser naccarii) // Mol. Ecol. Notes. 2002. V. 2. № 4. P. 586–588. https://doi.org/10.1046/j.1471-8286.2002.00328.x
Rohlf F.J. NTSYS-pc Numerical Taxonomy and Multivariate Analysis System Version 2.1. N.Y.: Exeter Publ. Setauket, 2000. 38 p.
Dice L.R. Measures of the amount of ecological association between species // Ecology. 1945. V. 26. № 3. P. 297–302.
Azuma N., Hagihara S., Ichimura M. et al. Genetic characterization of Amur sturgeon Acipenser schrenckii and its hybrid caught around Hokkaido // Ichthyol. Res. 2017. V. 64. № 1. P. 139–144. https://doi.org/10.1007/s10228-016-0544-5
Shen L., Shi Y., Zou Y.C. et al. Sturgeon aquaculture in China: status, challenge and proposals based on nation-wide surveys of 2010–2012 // J. Appl. Ichthyol. 2014. V. 30. № 6. P. 1547–1551. https://doi.org/10.1111/jai.12618
Дополнительные материалы отсутствуют.


