Генетика, 2020, T. 56, № 9, стр. 1065-1074
Y-хромосома как инструмент для ДНК-идентификации и определения популяционной принадлежности
В. Н. Харьков 1, *, А. А. Зарубин 1, К. В. Вагайцева 1, М. О. Раджабов 2, 3, Л. М. Новикова 1, Л. В. Валихова 1, И. Ю. Хитринская 1, В. А. Степанов 1
1 Научно-исследовательский институт медицинской генетики, Томский национальный исследовательский медицинский центр Российской академии наук
634050 Томск, Россия
2 Дагестанский государственный медицинский университет
367000 Махачкала, Россия
3 Институт физики Дагестанского федерального исследовательского центра
Российской академии наук
367015 Махачкала, Россия
* E-mail: vladimir.kharkov@medgenetics.ru
Поступила в редакцию 13.07.2019
После доработки 12.12.2019
Принята к публикации 17.12.2019
Аннотация
Проведен анализ генетического разнообразия большого числа популяций по различным маркерам Y-хромосомы. Генотипирование широкого набора новых высокоинформативных маркеров SNP и YSTR показывает, что в пределах большинства гаплогрупп Y-хромосомы наблюдается разделение не только на этноспецифические линии, но и на более узкие сублинии и кластеры гаплотипов. Показана значительная степень популяционной и межэтнической генетической дифференциации. Большинство этнических генофондов характеризуются преобладанием или даже полным доминированием специфичных SNP по всем основным гаплогруппам, что открывает большой потенциал для использования в этнической идентификации биологических образцов мужчин.
Анализ структуры генофондов популяций человека с использованием диаллельных и микросателлитных маркеров Y-хромосомы является одним из наиболее современных и результативных методов изучения генетического разнообразия различных популяций человека, позволяет проводить комплексную оценку генофонда человечества в целом, отдельных этносов, выполнять филогенетические реконструкции отдельных линий, филогеографические исследования гаплогрупп, миграционных процессов и ряд других задач. Несмотря на бурное развитие методов, позволяющих анализировать генофонды популяций на уровне полных геномов, использование маркеров Y-хромосомы по-прежнему является актуальным для решения целого ряда вопросов популяционной генетики [1–3].
Помимо фундаментальных аспектов результаты таких работ имеют важное значение и для разработки методов ДНК-идентификации биологических образцов человека. Анализ маркеров Y-хромосомы может быть эффективен в криминалистике и судебной медицине и служить альтернативой ДНК-идентификации по аутосомным локусам. Дополнительный анализ гаплотипов Y-хромосомы наиболее эффективен в случаях биологических образцов со смесью мужской и женской ДНК, а также при непрямой идентификации личности, когда сравнительный материал представлен в основном родственниками по мужской линии. Преимущество линий Y-хромосомы заключается в том, что они, обладая достаточно высокой информативностью и для “общей” ДНК-идентификации, передаются строго по отцовской линии, что позволяет напрямую устанавливать родство [4–6].
Для маркеров Y-хромосомы показан наибольший уровень генетических различий между популяциями в сравнении с любыми другими генетическими системами. Это приводит к значительной степени территориальной и межэтнической генетической дифференциации и высокому уровню специфичности гаплотипов. Высокие показатели межпопуляционного и межгруппового разнообразия Y-хромосомы делают ее маркеры наиболее дифференцирующими из всех, применяемых в популяционной генетике и геномике человека, и подчеркивают эффективность анализа гаплотипов Y-хромосомы для выявления различий между популяциями и при изучении миграций [7, 8]. Таким образом, при наличии подробной информации о частотах аллелей и гаплотипов у населения какого-либо региона становится возможным проводить определение популяционного и этнического происхождения индивида по Y-хромосомным маркерам.
В последние годы произошел настоящий прорыв в популяционной генетике, связанный с широким внедрением методов массового параллельного секвенирования (МПС), как для полногеномного генотипирования образцов, так и для поиска новых информативных SNP-маркеров в различных гаплогруппах Y-хромосомы [9–11]. Использование новых высокоинформативных специфичных SNP-маркеров Y-хромосомы является одним из наиболее перспективных инструментов для анализа региональных и этнических популяционных генофондов. Развитие методов массового параллельного секвенирования вывело анализ генофондов популяций на максимально возможный уровень разрешения. Обнаружение огромного числа новых информативных маркеров позволяет исследовать филогенетические взаимосвязи отдельных линий внутри гаплогрупп с невозможной ранее точностью [1, 3, 12]. Генотипирование новых SNP-маркеров может служить не только дополнением, но и альтернативой ДНК-идентификации по аутосомным локусам. При этом возможно создание эффективных тест-систем для определения наиболее вероятного популяционного происхождения индивида.
ОПРЕДЕЛЕНИЕ ПОПУЛЯЦИОННОГО ПРОИСХОЖДЕНИЯ НЕИЗВЕСТНОГО ИНДИВИДА ПО ОБРАЗЦУ ДНК
Современные достижения молекулярной генетики и геномики человека – полное секвенирование большого числа индивидуальных геномов, систематическая каталогизация генов и различных генетических маркеров, выявление полногеномной структуры гаплотипов и новые статистические подходы позволяют проводить очень подробное исследование генофондов. Успехи популяционной генетики в области изучения генетического разнообразия обусловлены как обнаружением различных типов информативных генетических маркеров, так и активным развитием технологий их генотипирования. Новые подходы в детекции различных типов ДНК-полиморфизма (в особенности SNP) разрабатываются очень быстро, и на сегодняшний день существует большое количество протоколов генотипирования разных маркерных систем. В течение двух последних десятилетий произошел настоящий прорыв в исследовании генетической вариабельности человеческих популяций по нейтральным маркерам, где накоплены данные по тысячам индивидуальных геномов представителей различных популяций.
Среди мировых приоритетных направлений в области ДНК-идентификации в настоящее время на первый план выступают разработки тест-систем, направленных на создание высокотехнологичной услуги по определению происхождения индивида по образцу его ДНК. Актуальность этой проблемы подтверждается большим количеством научных работ, посвященных анализу генетического разнообразия популяций различных географических регионов. При этом с каждым годом увеличивается не только число статей, но и число крупных международных проектов, посвященных различным аспектам анализа генетического разнообразия человечества. Разработка и практическая реализация технологий ДНК-идентификации невозможны без фундаментальной научной основы – комплексного и всестороннего анализа структуры генофондов конкретных популяций человека.
Каждая популяция характеризуется своим собственным уникальным составом генофонда: специфичностью имеющихся SNP-маркеров, частот аллелей, гаплогрупп Y-хромосомы и мтДНК, идентичных по происхождению блоков сцепления и т.д. Для эффективной дифференциации генофондов различных этнических групп и ДНК-идентификации индивида необходимо выявление генетических маркеров, наиболее информативных именно для тех популяций и этносов, которые должны быть объектом применения разрабатываемой технологии этнической идентификации, что позволит определять принадлежность индивида к конкретной популяции.
В результате специфичности наследования и отсутствия рекомбинации Y-хромосомы популяционная специфичность различных линий (гаплотипов и субгаплогрупп), при достаточном числе используемых маркеров, позволяет достаточно точно проводить определение популяционного происхождения индивида мужского пола (по отцовской линии) [13]. В настоящее время разработаны и применяются коммерческие наборы ДНК-маркеров Y‑хромосомы, на основе STR, которые рутинно используются для ДНК-идентификации. К сожалению, их использование малоинформативно для определения популяционной принадлежности индивида, поскольку для повышения идентификационного потенциала в состав наборов входят STR, имеющие высокий темп мутирования. Получаемые гаплотипы хорошо дифференцируют разные образцы, но относительно слабо дифференцируют образцы, принадлежащие представителям разных этнических и субэтнических групп.
YSTR
Современные технологии позволяют использовать в целях ДНК-идентификации различные типы маркеров. На настоящий момент известны работы по изучению идентификационного потенциала тест-систем, основанных на SNP, STR, гаплотипах, инсерционно-делеционном полиморфизме [14–16]. Наиболее широкое распространение получили микросателлитные маркеры (STR) за счет высокого темпа мутирования (0.1% и выше, на локус на поколение), высокого уровня гетерозиготности (70–90%) и небольших размеров [17].
Первый коммерческий набор генетических маркеров для идентификации человека был создан в 1988 г. [5], первые информативные микросателлитные локусы (STR) человека были охарактеризованы в 1991 г. [5, 18]. В 1992 г. были описаны первые информативные STR Y-хромосомы и впервые применены в деле об изнасиловании [5, 19].
Для всех трех систем маркеров для ДНК-идентификации (аутосомные STR, Y-хромосомные STR, мтДНК) вплоть до недавнего времени отсутствовали систематические знания о частотах аллелей в популяциях России, которые можно было бы использовать в качестве референсных. Для аутосомных STR стандарта CODIS, применяемого в России, эта проблема начала успешно разрешаться, в том числе и нашими предыдущими работами [20], для маркеров Y-хромосомы и мтДНК она чрезвычайно актуальна.
К настоящему времени на Y-хромосоме идентифицировано несколько сот полиморфных микросателлитных локусов [6], которые позволяют решать популяционные и эволюционные задачи различного масштаба – от характеристики мирового генофонда до высокоразрешающего анализа близких популяций [21].
SNP
Вопрос о возможности замены криминалистических стандартов микросателлитов SNP-маркерами впервые был поставлен в 2000 г. [22]. Поиском ответа на этот вопрос занимались многие научные коллективы, но все они на тот момент пришли к выводу, что SNP не способны стать равнозначной заменой STR-маркерам. Основная причина этого заключалась в том, что за счет низкой полиморфности однонуклеотидных замен для достижения информативности соответствующей панели STR необходимо проводить генотипирование большого числа маркеров (~50), что не выгодно как в плане трудозатрат, так и экономическом [22–28]. Современные технологии генотипирования позволили решить эту проблему, и интерес к возможностям SNP маркеров в сфере криминалистики возобновился [29, 30].
SNP-маркеры, имеющие значение для криминалистики, можно разделить на две группы.
1. Маркеры индивидуальной идентификации. Маркеры, входящие в состав этой группы, представляют собой набор высокополиморфных SNP, которые совместно дают высокое значение вероятности установления различий между двумя неродственными индивидами [31–33].
2. Маркеры для анализа на родство и происхождение. Маркеры данной группы в совокупности помогают определить вопрос о наличии родственной связи с более высокой вероятностью [16]. Так, SNP-маркеры Y-хромосомы и мтДНК, за счет униродительского наследования, передаются от предков к потомкам практически без изменений, что позволяет не только анализировать родственную связь, но и предсказать вероятную этническую принадлежность индивида [34].
Одной из основных проблем в популяционно-генетических исследованиях с применением монофилетических линий Y-хромосомы до недавнего времени оставался дефицит информативных SNP-маркеров для детального анализа филогенетической структуры и происхождения гаплогрупп. Но в последнее время ситуация заметно изменилась в лучшую сторону. Новые SNP Y-хромосомы в различных гаплогруппах все активнее выявляются в ходе работы проектов по полногеномному секвенированию и специальному поиску таких маркеров научными группами и различными компаниями. Количество таких SNP, открытых за один-два последних года, достигает уже многих тысяч. Однако точных данных по их филогенетическим взаимоотношениям в пределах различных гаплогрупп в конкретных популяциях зачастую нет, многие SNP подтверждены на ограниченном наборе образцов и данные о частотах определяемых ими сублиний в реальных этничеких группах отсутствуют или являются весьма приблизительными в силу нерепрезентативности исследованных выборок. Пока отсутствуют результаты генотипирования многих обнаруженных маркеров на материале популяционных выборок, значительно затруднены их филогеографический анализ, достоверные реконструкции возникновения и распространения вновь открытых линий.
SNP-маркеры, в большом количестве обнаруживаемые с помощью методов массового параллельного секвенирования, имеют огромный потенциал для использования в этнической идентификации биологических образцов мужчин. К сожалению, для подавляющего большинства из них отсутствуют систематические сведения о частотах аллелей и гаплотипов в популяциях России, которые можно было бы использовать в качестве референтных. Для разработки новых эффективных тест-систем на основе Y-хромосомных маркеров для определения популяционной принадлежности неизвестного индивида необходимы проведение массового популяционного скрининга, отбор информативных SNP, дифференцирующих генофонды различных популяций, разработка методов их совместного генотипирования и создание референтных баз данных частот генотипов.
Целью нашего исследования была оценка возможности и точности определения этнического происхождения индивидов из популяций населения России с помощью комплексного филогеографического анализа STR-гаплотипов Y-хромосомы и этноспецифичных сублиний гаплогрупп по SNP-маркерам.
ФОРМИРОВАНИЕ СПИСКА МАРКЕРОВ
В рамках работы было проведено генотипирование широкого набора маркеров Y-хромосомы для эффективной дифференциации мужских образцов. Для этого потребовалось провести выбор маркеров, который позволял бы выполнить две основные задачи. Во-первых, приписать исследуемый образец к конкретной гаплогруппе Y-хромосомы (для этого необходимо генотипирование различных SNP, определяющих как корневые, так и концевые ветви современного филогенетического древа Y‑хромосомных линий). Во-вторых, прогенотипировать маркеры, которые проявляли бы максимальную этническую специфичность аллелей (для этого необходимо генотипирование SNP специфичных для конкретных этносов и широкого набора YSTR, определяющих этноспецифичные гаплотипы Y‑хромосомы).
Формирование перечня потенциально информативных SNP-маркеров проводилось на основе работы с генетическими базами данных и литературой. Если 10–15 лет тому назад генотипирование новых SNP и YSTR далеко не всегда приводило к значительному увеличению информативности выбранной тест-системы, то сейчас можно отбирать ДНК-маркеры, широкий полиморфизм которых уже подтвержден. Более того, можно выбирать как быстро мутирующие STR для подробного анализа относительно молодых сублиний, так и маркеры с гораздо более медленным темпом мутирования, выделяющие в пределах отдельных гаплогрупп региональные и этноспецифичные кластеры гаплотипов.
К сожалению, для большинства новых SNP-маркеров отсутствует информация о их частотах в большинстве популяций. Мы выбрали недавно обнаруженные в различных гаплогруппах Y-хромосомы SNP, потенциально информативные для молекулярно-филогенетического анализа гаплогрупп и этнической идентификации в популяциях европейской части России, Сибири и Дагестана. После отбора SNP был проведен подбор условий генотипирования, преимущественно с помощью ПДРФ-анализа, выполнен подбор соответствующих ферментов и олигонуклеотидных праймеров.
Для выбора таких ДНК-маркеров из всех доступных источников (научных публикаций, данных The 1000 Genomes Project, Hap Map, International Society of Genetic Genealogy, Family Tree DNA, YFull и др.) были отобраны недавно обнаруженные в гаплогруппах SNP, потенциально информативные для молекулярно-филогенетического анализа гаплогрупп в популяциях России, которые с высокой долей вероятности позволят эффективно выявлять и дифференцировать как новые крупные сублинии в пределах гаплогрупп, так и более мелкие, специфичные для отдельных этносов и популяций.
Кроме этого, был использован расширенный набор YSTR-маркеров, что позволило перейти на принципиально новый уровень детализации молекулярно-филогенетической структуры гаплогрупп и дифференциации популяций. Уровень информативности гаплотипов по выбранным маркерам должен был достичь значений, практически позволяющих идентифицировать различия между этническими группами. Мы предполагали, что это с высокой долей вероятности позволит эффективно выявлять и дифференцировать как новые крупные сублинии в пределах гаплогрупп, так и более мелкие, специфичные для отдельных этнических групп. В результате генотипирования SNP-маркеров большинство из них подтвердили высокую информативность и этническую специфичность.
МАТЕРИАЛЫ И МЕТОДЫ
Материал для исследования составили образцы ДНК, выделенные из лейкоцитов крови мужчин, принадлежащих к различным этническим группам населения Восточной Европы, Северного Кавказа, Сибири, Дальнего Востока и Средней Азии, не состоящих в родстве, без метисации, принадлежащих к популяциям РФ и сопредельных государств (Банк ДНК “НИИ медицинской генетики”). Охарактеризовано более 70 популяционных выборок, принадлежащих к 34 этническим группам. В исследование были включены 36 микросателлитных локусов (DYS: 19, 385a, 385b, 388, 389I, 389II, 390, 391, 392, 393, 426, 434, 435, 436, 437, 438, 439, 442, 444, 445, 448, 456, 458, 462, 504, 505, 525, 531, 537, 552, 635, YCAIIa, YCAIIb, GATAH4, YGATAA10, GGATT1B07). Генотипирование проводили с помощью капиллярного гель-электрофореза на генетических анализаторах ABI Prism 3130xl и 3730. Также в исследуемых выборках было прогенотипировано более 250 SNP-маркеров. Генотипирование SNP проводили с помощью ПЦР и последующего анализа различными методами.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Специфичность YSTR-гаплотипов
В результате анализа полученных генотипических данных было показано, что гаплотипы, построенные с помощью генотипирования 36 STR-маркеров Y-хромосомы, обладают огромной этнической и популяционной специфичностью. Установлена очень высокая межпопуляционная дифференциация полученных YSTR-гаплотипов, что позволяет эффективно использовать отработанный набор YSTR-маркеров для целей ДНК-идентификации. Степень детализации зависит практически лишь от количества YSTR, используемых для построения гаплотипа: если для высокоразрешающего анализа индивидов при генеалогических реконструкциях требуется от 30 и более маркеров, то для подробного описания популяционного генофонда достаточно 15–20 YSTR. Анализ структуры медианных сетей гаплотипов по различным гаплогруппам показывает, что в пределах большинства линий наблюдается разделение не только на этноспецифичные группы гаплотипов, но и на более узкие кластеры. Так, для исследованных популяций Северного Кавказа в ряде случаев показана возможность установить популяционное происхождение индивида вплоть до населенного пункта. В большинстве случаев отчетливо видны гаплотипы-основатели. Большинство этнических и региональных генофондов характеризуются преобладанием собственных локальных групп гаплотипов, являющихся производными от предкового для конкретной гаплогруппы. Этноспецифичные и разветвленные медианные сети гаплогрупп свидетельствуют о решающей роли географической изоляции популяций в формировании современной картины генетического разнообразия по линиям Y-хромосомы.
Популяции коренных этносов Сибири и Дагестана выделяются на фоне других значительно более высоким уровнем межэтнической генетической дифференциации по YSTR-гаплотипам. По данным о родовом составе алтайцев, хакасов, шорцев, хантов и телеутов и генетической характеристике отдельных родов обнаружено, что генофонд этих групп также демонстрирует высокоспецифичные кластеры YSTR-гаплотипов, что позволяет проводить ДНК-идентификацию на уровне субэтноса или рода. Оценка межпопуляционных различий по YSTR-гаплотипам выявила статистически значимые различия между всеми парами сравниваемых этносов и субэтнических групп. Выбранные микросателлитные маркеры являются достаточно высокоинформативными для большинства популяционных выборок. Таким образом, YSTR-маркеры имеют огромный потенциал для использования в этнической идентификации биологических образцов, что, несомненно, может найти широкое применение для молекулярно-генетического анализа жертв преступлений, катастроф, террористических актов, преступников, контингентов специальных подразделений армии и сил правопорядка.
Некоторые народы и группы этносов, в популяционной истории которых имели место сильные эффекты основателя и длительная изоляция от влияния миграций в силу географической удаленности или труднодоступности горной местности, отличаются очень низким разнообразием по составу гаплогрупп. В Сибири это якуты и буряты, а в европейской части России это малочисленные народы Дагестана, проживающие в южной горной части республики. Популяции горных районов характеризуются значительно более низким уровнем генетического разнообразия по гаплогруппам Y‑хромосомы по сравнению с другими северокавказскими популяциями. При этом внутри гаплогруппы J1 наблюдается огромное разнообразие YSTR-гаплотипов, что свидетельствует о значительной древности J1 на этой территории (рис. 1).
Рис. 1.
Медианная сеть микросателлитных гаплотипов гаплогруппы J1 в популяциях Дагестана, относящихся к Цезской языковой группе. Красным обозначены цезы, зеленым – гунзибцы, сиреневым – бежтинцы, синим – гинухцы.
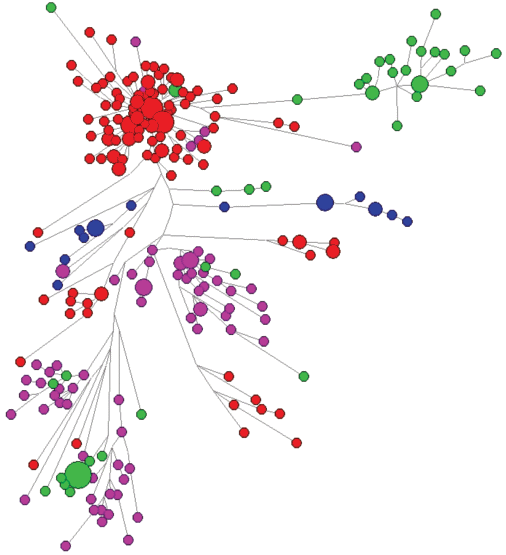
Результаты филогенетического анализа показывают, что состав и структура YSTR-гаплотипов для гаплогруппы J1 и уровень генетического разнообразия и внутриэтнической генетической дифференциации популяций в пределах отдельных линий Y-хромосомы являются полностью этноспецифичными. Наблюдаются значительные недавние эффекты основателя по гаплогруппе J1 в отдельных населенных пунктах для большинства этносов. Практически все этносы демонстрируют следы значительного роста численности, проявляющиеся в присутствии на медианных сетях этноспецифичных звездообразных кластеров гаплотипов с выраженным гаплотипом-основателем, в пределах отдельных населенных пунктов. Наиболее отчетливо это видно на примере цезов, гунзибцев и ахвахцев.
Специфичность сублиний внутри гаплогрупп Y-хромосомы
Большая часть отобранных в исследование новых терминальных SNP, определяющих молодые узкие сублинии в пределах конкретных гаплогрупп, обладает значительной этно-популяционной специфичностью. В большинстве случаев сравнительный анализ случайно выбранных образцов должен позволять провести популяционную привязку образца с точностью до этноса. Это означает возможность эффективно использовать SNP и YSTR-маркеры для разработки тест-систем и ДНК-идентификации. Точность определения будет зависеть от принадлежности образца к конкретной гаплогруппе, особенностей ее распространения в популяциях и частоты SNP у различных этносови специфичности YSTR-гаплотипа.
Ситуация, когда какая-то одна линия Y-хромосомы составляет значительную долю генофонда отдельной популяции или этноса в целом, является довольно редкой. Большинство этносов характеризуются разнообразным по составу пулом Y-хромосомных линий. Но при этом в большинстве случаев эти различные гаплогруппы отличаются по терминальным SNP и структуре гаплотипов даже у территориально близких популяций.
Приведем конкретные примеры этнической специфичности отдельных сублиний по SNP-маркерам (не по всем исследованным гаплогруппам, а по наиболее широко распространенным, достаточно частым и важным для идентификации).
Клада N Y-хромосомы разделяется на две крупные гаплогруппы: N1a1 и N1a2. Якуты, в составе генофонда которых доминирует гаплогруппа N1a1, в большинстве относятся к ветви N1a1-М1979-M1988-M1991. Все остальные исследованные нами образцы клады N1a1, относящиеся к другим этносам, принадлежат к другим ее ветвям. Генетически близкие якутам эвенки Забайкалья имеют только две из этих мутаций и принадлежат сестринской линии N1a1-M1979-M1991хM1988. У бурятов и калмыков обнаружена только линия N1a1-B219, у казахов – только N1a1-B220, у чукчей и коряков – N1a1-B204, у удмуртов и коми – N1a1-B211, у марийцев – N1a1-B194, у карелов и вепсов – N1a1-Z1936 и N1a1-VL29.
У хакасов и шорцев практически все образцы клады N принадлежат к ветви N-B187-B499, а у близких им тувинцев – к сестринской N-B187 × × B499. При этом N-B187-B499 полностью доминирует по частоте в хакасских родах (сеоках) хый и хобый и родственных шорских кый и кобый, что не наблюдается больше не в одном другом сеоке. У хантов это азиатская ветвь N1a2-B170-Y81068, у хакасов – ее сестринская N1a2-B170 × Y81068, у тувинцев – N1a2-B175-PF3415, у удмуртов – N1a2-B226-B528, у марийцев – N1a2-YFC036.
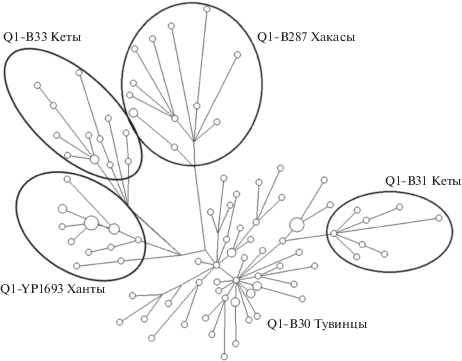
По кладе Q1 также обнаружена практически полная этническая специфичнеость линий. У тувинцев – это Q1-B30, у хантов – Q1-L716 и Q1-YP1693, у хакасов – Q1-B287, у алтайцев и шорцев – Q1-BZ2199, у кетов – две сублинии: Q1-B31 и Q1-B33, у коряков и нивхов – Q1-F746, у калмыков и казахов – Q1-F1340 (рис. 2).
Клада R1a делится на азиатскую (Z93) и европейскую (Z282) ветви. У южных алтайцев (с очень высокой частотой этой гаплогруппы и сильным эффектом основателя) доминирует сублиния R1a-YP1518, а у близкородственных им киргизов – R1a-YP1563, у тубаларов – R1a-Y42090, у хантов – R1a-S7280, у узбеков и уйгуров – R1a-Y30. У южносибирских тувинцев, хакасов и шорцев распространены сразу несколько сублиний, одни из которых, видимо, маркируют ранний европеоидный компонент, а другие – более поздний тюркский. При этом выявлено четкое разделение разных сублиний между разными родовыми группами (сеоками): так, хакасские сеоки пилтыр и чыстар представлены в основном образцами гаплогруппы R1a-Y62155. Представители сеока туран относятся к сублинии R1a-Y43850. Сеок чити-пуур относится к сублинии R1a-Y43109. Мужчины-хакасы из сеока харга и все шорцы родственного сеока карга принадлежат к сублинии R1a-YP1509. Среди популяций европейской части России по линии Z282 сильных межэтнических различий не обнаружено. Возможно, это связано с тем, что было отобрано недостаточно информативных SNP-маркеров, а частично объясняется ассимиляцией различных финно-угорских этносов при славянской экспансии.
Генотипирование новых высокоинформативных маркеров показывает, что в пределах большинства гаплогрупп Y-хромосомы наблюдается разделение не только на этноспецифичные линии, но и на более узкие сублинии и кластеры гаплотипов. Большинство этнических генофондов характеризуются преобладанием или даже полным доминированием специфичных терминальных SNP по большинству концевых сублиний для всех основных гаплогрупп. Степень детализации зависит практически лишь от количества SNP и STR-маркеров, используемых для построения гаплотипа.
Таким образом, возможно с высокой точностью провести определение этнической принадлежности мужчин, из биологического материала которых был получен исследованный образец ДНК. Такое заключение, конечно, не будет являться абсолютно точным, поскольку, во-первых, национальная самоидентификация человека может не соответствовать его биологическому происхождению, например в случае если он является потомком от межнационального брака, во-вторых, он может принадлежать к другой родственной национальности, для которой отсутствуют данные о частотах изучаемых генетических маркеров, в-третьих, его Y‑хромосома может быть унаследована от совершенно неродственного этноса много поколений назад. Принадлежность образца к другим этническим группам при этом полностью не исключается, однако с учетом относительно небольшой доли таких случаев среди большинства популяций принадлежность анализируемого образца к другим популяциям в большинстве случаев намного менее вероятна, а именно это и важно для практических целей.
ЗАКЛЮЧЕНИЕ
Показано, что большая часть новых YSNP, в сочетании с YSTR-гаплотипами, обладает большой этно-популяционной специфичностью. В большинстве случаев сравнительный анализ случайно выбранных образцов позволяет провести популяционную привязку образца с точностью до этноса, субэтноса или рода, а в отдельных случаях и до конкретного населенного пункта и даже небольшого числа фамилий носителей этого спектра сублиний и гаплотипов. Это означает возможность эффективно использовать SNP и YSTR-маркеров для разработки тест-систем и ДНК-идентификации. Точность определения зависит от принадлежности образца к конкретной гаплогруппе, особенностей ее распространения в популяциях и частоты SNP у различных этносови специфичности YSTR-гаплотипа.
Очевидно, что при разработке новых наборов YSTR для идентификации личности помимо дискриминирующего потенциала выбранных маркеров необходимо учитывать и степень представленности информации о генотипах по различным микросателлитным маркерам в доступных генетических базах данных. Поиск совпадающих гаплотипов в базах данных, этнической и географической привязке таких образцов может являться важной составляющей работы по криминалистическому анализу того или иного биологического материала.
Особенность России с точки зрения интерпретируемости данных при ДНК-идентификации состоит в ее многонациональности. Вследствие того, что полиморфизм маркеров Y-хромосомы обладает популяционной и этнической специфичностью, необходимо создание баз данных, содержащих информацию по нескольким референтным популяциям, представляющим все субэтнические и этнические группы.
Полученные результаты исследования могут быть применены для разработки систем и стандартов генетических маркеров для ДНК-идентификации, тестирования отцовского просхождения; а также для разработки референтных баз данных для ДНК-тестирования и геномной регистрации. Применение возможно в области криминалистики, судебной медицины, ДНК-тестирования, разработки услуг геномной медицины.
Работа выполнена в рамках Научно-технической программы Союзного государства “Разработка инновационных геногеографических и геномных технологий идентификации личности и индивидуальных особенностей человека на основе изучения генофондов регионов Союзного государства” (“ДНК-идентификация”), государственный контракт № 011-17 от 26.09.2017, а также поддержана грантом РФФИ № 18-29-13045 “Популяционная геномика и транскриптомика человека: поиск сигналов не-нейтральной эволюции”.
Все процедуры, выполненные в исследовании с участием людей, соответствуют этическим стандартам институционального и/или национального комитета по исследовательской этике и Хельсинкской декларации 1964 г. и ее последующим изменениям или сопоставимым нормам этики.
От каждого из включенных в исследование участников было получено информированное добровольное согласие.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Karmin M., Saag L., Vicente M. et al. A recent bottleneck of Y chromosome diversity coincides with a global change in culture // Genome Res. 2015. P. 459–466. https://doi.org/10.1101/gr.186684.114
Ilumäe A.-M., Reidla M., Chukhryaeva M. et al. Human Y-chromosome haplogroup N: a non-trivial time-resolved phylogeography that cuts across language families // Am. J. Hum. Genet. 2016. V. 99. P. 163–173. https://doi.org/10.1016/j.ajhg.2016.05.025
Hallast P., Batini C., Zadik D. et al. The Y-chromosome tree bursts into leaf: 13 000 high-confidence SNPs covering the majority of known clades // Mol. Biol. Evol. 2015. V. 32(3). P. 661–673. https://doi.org/10.1093/molbev/msu327
Jobling M.A., Tyler-Smith C. The human Y chromosome: an evolutionary marker comes of age // Nat. Rev. Genet. 2003. V. 4. P. 598–612. https://doi.org/10.1038/nrg1124
Jobling M.A., Gill P. Encoded evidence: DNA in forensic analysis // Nat. Reviews. 2004. V. 5. № 10. P. 739–751. https://doi.org/10.1038/nrg1455
Kayser M., Kittler R., Erler A. et al. A comprehensive survey of human Y-chromosomal microsatellites // Am. J. Hum. Genet. 2004. V. 74. P. 1183–1197.https://doi.org/10.1086/421531
Zhivotovsky L.A., Underhill P.A., Cinnioglu C. et al. On the effective mutation rate at Y-chromosome STRs with application to human population divergence time // Am. J. Hum. Genet. 2004. V. 74. P. 50–61. https://doi.org/10.1086/380911
Dupanloup I., Pereira L., Bertorelle G. et al. A recent shift from polygeny to monogamy in humans is suggested by the analysis of worldwide Y-chromosome diversity // Mol. Evol. 2003. V. 57. P. 85–97.https://doi.org/10.1007/s00239-003-2458-x
Rizzo J.M., Buck M.J. Key principles and clinical applications of “next-generation” DNA sequencing // Cancer Prev. Res. 2012. V. 5. № 7. P. 887–900. https://doi.org/10.1158/1940-6207.CAPR-11-0432
Grandell I., Samara R., Tillmar A.O. A SNP panel for identity and kinship testing using massive parallel sequencing // Int. J. Legal. Med. 2016. V. 130. № 4. P. 905–914. https://doi.org/10.1007/s00414-016-1341-4
Amorim A., Pinto N. Big data in forensic genetics // Forensic Sci. Int. Genet. 2018. № 37. P. 102–105. https://doi.org/10.1016/j.fsigen.2018.08.001
Kivisild T. The study of human Y chromosome variation through ancient DNA // Hum. Genet. 2017. V. 136. P. 529–546. https://doi.org/10.1007/s00439-017-1773-z
Харьков В.Н., Степанов В.А. Маркеры Y-хромосомы в ДНК идентификации: достижения, проблемы и перспективы: IX Всерос. научно-практ. конф. с международным участием “Молекулярная диагностика 2017”. Т. 1. С. 423–424.
Boonyarit H., Mahasirimongkol S., Chavalvechakul N. et al. Development of a SNP set for human identification: A set with high powers of discrimination which yields high genetic information from naturally degraded DNA samples in the Thai population // Forensic. Sci. Int. Genet. 2014. V. 11. P. 166–173. https://doi.org/10.1016/j.fsigen.2014.03.010
Ge J., Budowle B., Planz J.V. et al. Haplotype block: a new type of forensic DNA markers // Int. J. Legal. Med. 2010. V. 124. № 5. P. 353–361. https://doi.org/10.1007/s00414-009-0400-5
Da Costa Francez P.A., Rodrigues E.M., Velasco A.M. et al. Insertion-deletion polymorphisms–utilization on forensic analysis // Int. J. Legal. Med. 2012. V. 126. № 4. P. 491–496.https://doi.org/10.1007/s00414-011-0588-z
Dib C., Faure S., Fizames C. et al. A comprehensive genetic map of the human genome based on 5264 microsatellites // Nature. 1996. V. 380. P. 152–154. https://doi.org/10.1038/380152a0
Edwards A., Civitello A., Hammond H.A. et al. DNA typing and genetic mapping with trimeric and tetrameric tandem repeats // Am. J. Hum. Genet. 1991. V. 49. № 4. P. 746–756.
Roewer L., Epplen J.T. Rapid and sensitive typing of forensic stains by PCR amplification of polymorphic simple repeat sequences in case work // Forensic. Sci. Int. 1992. V. 53. № 2. P. 163–171. https://doi.org/10.1016/0379-0738(92)90193-z
Степанов В.А., Балановский О.П., Мельников А.В. и др. Характеристика популяций Российской Федерации по панели пятнадцати локусов, используемых для ДНК-идентификации и в судебно-медицинской экспертизе // Acta Naturae. 2011. Т. 3. № 2. С. 59–71.
Ballantyne K.N., Keerl V., Wollstein A. et al. A new future of forensic Y-chromosome analysis: rapidly mutating Y-STRs for differentiating male relatives and paternal lineages // Forensic. Sci. Int. Genet. 2012. V. 6. P. 208–218. https://doi.org/10.1016/j.fsigen.2011.04.017
Butler J.M., Coble M.D., Vallone P.M. STRs vs SNP: thoughts on the future of forensic DNA testing // Forensic. Sci. Med. Pathol. 2007. V. 3. № 3. P. 200–205. https://doi.org/10.1007/s12024-007-0018-1
Chakraborty R., Stivers D.N., Su B. et al. The utility of short tandem repeat loci beyond human identification: implications for development of new DNA typing systems // Electrophoresis. 1999. V. 20. № 8. P. 682–1696. https://doi.org/10.1002/(SICI)1522-2683(19990101)20:8< 1682::AID-ELPS1682>3.0.CO;2-Z
Gill P. An assessment of the utility of single nucleotide polymorphisms (SNPs) for forensic purposes // Int. J. Lega. l Med. 2001. V. 114. № 4–5. P. 204–210. https://doi.org/10.1007/s004149900117
Gill P., Werrett D.J., Budowle B. et al. An assessment of whether SNPs will replace STRs in national DNA databases-joint considerations of the DNA working group of the European Network of Forensic Science Institutes (ENFSI) and the Scientific Working Group on DNA Analysis Methods (SWGDAM) // Sci. Justice. 2004. V. 44. № 1. P. 51–53.
Budowle B. SNP typing strategies // Forensic Sci. Int. 2004. V. 146. Suppl.: S139–S142.
Amorim A., Pereira L. Pros and cons in the use of SNPs in forensic kinship investigation: a comparative analysis with STRs // Forensic. Sci. Int. 2005. V. 150. № 1. P. 17–21.https://doi.org/10.1016/j.forsciint.2004.06.018
Sobrino B., Bri M., Carracedo A. SNPs in forensic genetics: a review on SNP typing methodologies // Forensic. Sci. Int. 2005. V. 154. P. 181–194. https://doi.org/10.1016/j.forsciint.2004.10.020
Petkovski E., Keyser-Tracqui C., Hienne R. et al. SNPs and MALDI-TOF MS: tools for DNA typing in forensic paternity testing and anthropology // J. Forensic. Sci. 2005. V. 50. № 3. P. 535–541.
Johansen P., Andersen J.D., Morling C. Evaluation of the iPLEX® Sample ID Plus Panel designed for the Sequenom Mass ARRAY® system // Forensic. Sci. Int. Genet. 2013. V. 7. № 5. P. 482–487.https://doi.org/10.1016/j.fsigen.2013.04.009
Dixon L., Dobbins A.E., Pulker H.K. et al. Analysis of artificially degraded DNA using STRs and SNPs-results of a collaborative European (EDNAP) exercise // Forensic. Sci. Int. 2006. V. 164. № 1. P. 33–44. https://doi.org/10.1016/j.forsciint.2005.11.011
Sanchez J.J., Phillips C., Balogh C. et al. A multiplex assay with 52 single nucleotide polymorphisms for human identification // Electrophoresis. 2006. V. 27. № 9. P. 1713–1724. https://doi.org/10.1002/elps.200500671
Pakstis A.J., Speed W.C., Fang R. et al. SNPs for a universal individual identification panel // Hum. Genet. 2010. V. 127. № 3. P. 315–324. https://doi.org/10.1007/s00439-009-0771-1
Mizuno N.A., Kitayama T., Fujii K. et al. Forensic method for the simultaneous analysis of biallelic markers identifying Y chromosome haplogroups inferred as having originated in Asia and the Japanese archipelago // Forensic Sci. Int. Genet. 2010. V. 4. № 2. P. 73–79. https://doi.org/10.1016/j.legalmed.2009.12.004
Дополнительные материалы отсутствуют.