Генетика, 2021, T. 57, № 12, стр. 1415-1422
Ассоциация метилирования генов нейромедиаторных систем мозга с психоэмоциональными характеристиками человека
И. Б. Моссэ 1, *, Н. Г. Седляр 1, А. С. Бабенко 2, К. А. Моссэ 1, Р. С. Шулинский 1, А. В. Кильчевский 1
1 Институт генетики и цитологии Национальной академии наук Беларуси
220072 Минск, Республика Беларусь
2 Белорусский государственный медицинский университет
220116 Минск, Республика Беларусь
* E-mail: I.Mosse@igc.by
Поступила в редакцию 22.06.2021
После доработки 28.06.2021
Принята к публикации 29.06.2021
Аннотация
Эпигенетическая регуляция активности генов вовлечена в развитие ряда психических нарушений, включая нарушения развития головного мозга, личностные расстройства и девиантное поведение. Проведено исследование уровня метилирования 24 избранных CpG-сайтов в группах пациентов с перечисленными нарушениями и контрольной группе. Показано, что CpG-сайты в регуляторных участках трех генов нейромедиаторных систем человека – BDNF, COMT и CACNA1C – гиперметилированы, что может приводить к снижению экспрессии указанных генов. Полученные результаты могут быть основой для разработки диагностических тест-систем и поиска фармакологических мишеней для выявления и коррекции указанных состояний.
Интерес к эпигенетическим механизмам резко возрос после того, как появилось большое количество данных о роли эпигенетических процессов в основополагающих биологических явлениях таких как онтогенез, старение, а также в развитии ряда заболеваний (ожирение, наркомания, алкоголизм, шизофрения, личностные расстройства, депрессивные состояния и т.п.). Изменения экспрессии генов, возникшие под влиянием стрессорных средовых факторов, приобретают устойчивый долговременный характер, т.е. остаются относительно неизменными в течение длительного времени, часто на всю оставшуюся жизнь (поэтому кратковременные адаптивные изменения, связанные с изменением генной экспрессии, под понятие эпигенетических сдвигов не подпадают).
Возникшие изменения активности могут сохраняться на протяжении двух–трех поколений, особенно при продолжении воздействия стрессовых средовых факторов [1]. На существующую генетическую программу “накладываются” эпигенетические метки, индуцированные неблагоприятными жизненными обстоятельствами, в том числе психосоциальными факторами.
Эпигенетические механизмы играют важную роль в стресс-индуцированных нарушениях поведения и психики. Фактически можно говорить о психиатрической и поведенческой эпигенетике как о самостоятельном разделе исследований [2]. Стресс при участии эпигенетических механизмов выступает как один из основных факторов, провоцирующих ряд расстройств психики.
Оценка статуса метилирования специфических локусов геномной ДНК является эффективным способом отслеживания, изучения и контроля уникальных процессов, регулирующих жизнедеятельность человека [3, 4]. Эпигенетические стресс-индуцированные феномены при этом становятся ведущим фактором нарушений психического здоровья, прежде всего депрессии и тревоги, но также личностных расстройств, зависимостей и асоциального поведения [5, 6]. Выявление ключевых генетическихи эпигенетических маркеров риска развития психических расстройств и агрессивного поведения позволяет оценить вероятность наличия анализируемых признаков у индивида по его ДНК в целях диагностики, выявления фармакологических мишеней и возможного применения в криминалистике.
Наиболее изученный вид эпигенетической модификации ДНК у людей – метилирование цитозина в положении C5 в CpG-динуклеотидах. Более 28 миллионов сайтов CpG распределены по всему геному человека и 70–80% из них могут быть метилированы. В большинстве случаев гиперметилирование промоторных областей приводит к снижению экспрессия генов [7].
Интерес ученых к статусу метилирования генов, ответственных за психоэмоциональный статус человека, возрастает. Анализ современного состояния проблемы показал, что необходима оценка целого спектра генетических и эпигенетических локусов генома человека. Для решения таких задач активно применяются технологии секвенирования нового поколения (NGS) и разработка небольших таргетных панелей. Преимуществом такого подхода является, с одной стороны, возможность исследования десятков и сотен локусов в ходе анализа одного образца, с другой – имеется возможность протестировать в рамках одного эксперимента сотни образцов, что сильно снижает стоимость одного анализа для конечного потребителя, делая его более доступным в рутинной практике [9].
В представленном исследовании выявлены гены нейромедиаторных систем мозга, уровень метилирования CpG-сайтов в регуляторных участках которых ассоциирован с нарушениями психического здоровья человека.
МАТЕРИАЛЫ И МЕТОДЫ
Исследование проведено на двух группах представителей белорусской популяции: контрольная группа условно здоровых добровольцев (К) численностью 30 человек и пациенты РНПЦ психического здоровья с органическими нарушениями головного мозга и умственной отсталостью, страдающие личностными расстройствами и девиантным поведением (П) – 61 человек.
От каждого участника было получено информированное согласие на включение в исследование. Дизайн исследования утвержден Биоэтическим комитетом Института генетики и цитологии НАН Беларуси.
В качестве биологического материала использовали буккальный эпителий. Выделение ДНК проводили наборами “Нуклеосорб”, модификация А (ОДО Праймтех).
Метилирование ДНК определяли методом бисульфитной конверсии, при которой каждый неметилированный цитозин превращается в урацил посредством дезаминирования. Бисульфитную конверсию геномной ДНК в количестве 1 мкг выполняли с использованием набора реагентов EpiTect Fast DNA Bisulfite Kit (Qiagen) согласно инструкции производителя. Набор оптимизирован для получения после конверсии участков геномной ДНК длиной 300–500 пн и в некотором количестве более 500 пн.
Для анализа уровня метилирования целевых генов использовали разработанную нами панель специфических олигонуклеотидных праймеров, фланкирующих выбранные для исследования локусы [8]. Амплификацию конвертированной ДНК проводили для 17 целевых участков исследуемых генов, которые содержали 24 специфических CpG-сайта метилирования.
Для контроля качества и определения концентрации полученных фрагментов ДНК использовали прибор для флуориметрического анализа Glomax Explorer (Promega) с набором реагентов QuantiFluor® ONE dsDNA System (Promega).
Нормализацию и пулирование образцов выполняли с помощью системы автоматического дозирования Eppendorf epMotion. Для приготовления NGS-библиотек использовали наборы реагентов QIAseq Methyl Library Kit (Qiagen), позволяющие работать с фрагментированными участками ДНК после бисульфитной конверсии. Конечная концентрация анализируемой библиотеки составила 8 пмоль, концентрация контрольной библиотеки PhiX – 10%.
Высокопроизводительное секвенирование выполнялось одновременно для 24 образцов на приборе IlluminaMiseq. Для запуска использован набор v3 2 × 250 (500 cycles) Illumina – MiSeq Reagent Kit v3.
Оценку качества прочтений проводили в программе FastQC, c последующим триммингом некачественных оснований в Trimmomatic. Локальное картирование прочтений на референс (GRCh37 [GCF_000001405.13]) проводили по алгоритму Burrows-Wheeler-Aligner при помощи программного модуля Bowtie2 в утилите Bismark.
Фильтрацию с последующим преобразованием карты выравнивания прочтений (SAM), также сортировку и индексирование проводили в Samtools. Вызов сайтов метилирования на “изначально верхней цепи” (original top strand) в CpG-островах проведено в пайплайне Bismark. Подсчет отношения деметилированных сайтов к метилированным был произведен на языке программирования Python с использованием библиотеки Рandas.
Сравнение уровней метилирования отдельных CpG-сайтов в группах исследования проводилось с использованием критериев Манна–Уитни. Различия принимались как статистически значимые при р < 0.05.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
На первом этапе исследования анализ результатов высокопроизводительного секвенирования позволил получить данные об уровне метилирования 24 целевых CpG-сайтов в образцах ДНК от 61 пациента и 30 человек из группы контроля.
Наибольшие различия между группой пациентов и контрольной группой были зафиксированы для шести CpG-сайтов, расположенных в промоторных областях трех генов – BDNF, COMT и CACNA1C.
Ген BDNF, локализованный в хромосомном локусе 11р1, кодирует мозговой нейротрофический фактор – цитокин, представляющий собой гомодимерный белок с молекулярной массой 27 кДа. BDNF оказывает уникальное действие на ЦНС, стимулируя в нейронах репаративные процессы, повышая синаптическую пластичность и устойчивость клеток ЦНС к повреждающим факторам [9, 10]. Дефицит BDNF снижает пластичность нейронов, нарушает память и обучаемость, когнитивные способности. Экспрессия BDNF значительно уменьшается у пациентов с депрессией, аффективными расстройствами, при стрессе [11, 12]. Хронический стресс приводит к снижению продукции BDNF, которая участвует во многих клеточных процессах в нервной ткани, особенно связанных с долговременной памятью, путем активизации метилирования гистонов и изменения степени компактизации хроматина.
BDNF – один из наиболее изученных генов у пациентов с психоэмоциональными нарушениями и в том числе с биполярным расстройством (БР). В частности, повышенное метилирование области промотора BDNF (экзон 1) было обнаружено в мононуклеарных клетках периферической крови пациентов с БР второго типа, у которых уровни экспрессии гена BDNF также были значительно снижены по сравнению с контрольной группой [13].
В другом исследовании степень метилирования ДНК различалась у пациентов с БР и контрольной группы для 11 из 36 CpG-сайтов, проанализированных в промоторах 3 и 5 гена BDNF [14]. Несколько дифференциально метилированных сайтов перекрывались с участками связывания факторов транскрипции в промоторе 5 или непосредственно рядом с ними, предполагая возможную роль метилирования ДНК в модуляции экспрессии гена BDNF.
Ген COMT расположен на участке 22q11.21, кодирует фермент катехол-О-метилтрансферазу, участвующую в обмене различных катехоламинов (адреналин, норадреналин, дофамин) [15, 16]. Физиологическая роль фермента заключается в элиминации катехоламинов из синаптической щели путем реакции метилирования. Динамика катехоламинов в разных участках мозга регулирует социальное поведение. Катехол-О-метилтрансфераза регулирует передачу нервного импульса, влияет на особенности эмоциональных реакций, участвует в метаболизме эстрогенов [16]. Существуют две изоформы данного фермента: S-СОМТ (растворимая изоформа, обнаружена в крови и периферических тканях) и МВ-СОМТ (мембран-связанный протеин, основная часть которого локализована в префронтальной коре) [17]. Известно около 300 полиморфных сайтов [17], для некоторых из которых показано клиническое значение [18].
Гиперметилирование сайта COMT_19949901 (cg04856117) было также зафиксировано при изучении степени метилирования ДНК 41-ного сайта CpG в гене COMT у 185 человек с посттравматическим стрессовым расстройством и контрольной группы [19]. По результатам исследования увеличение уровня метилирования только двух сайтов, расположенных в промоторе гена, включая cg04856117, было независимо связано с данным состоянием.
Ген CACNA1C расположен на участке хромосомы 12p13.3, кодирует белок главного потенциал-зависимого кальциевого канала L-типа, Cav1.2 (субъединица альфа-1C). Каналы Cav1.2 являются критическими модуляторами ряда клеточных процессов, которые участвуют в развитии психопатологии [20]. Полногеномные исследования выявили ассоциацию гена CACNA1C с множественными психоневрологическими расстройствами, включая большое депрессивное расстройство, биполярное расстройство и шизофрению, которые проявляются депрессивными симптомами [21].
Метилирование гена CACNA1C также изучалось у пациентов с БР – как одна из возможных причин данного состояния. Среди 5-CpG-островков, расположенных в различных частях гена, вариации в уровнях метилирования были зафиксированы только для сайтов, расположенных в третьем интроне гена. При этом пять из шести сайтов показали значительное гиперметилирование в ДНК пациентов с БР по сравнению с контролем. Также обнаружено, что на уровень метилирования влияет пол и также близлежащие полиморфные сайты [22].
Распределение уровней метилирования шести сайтов, расположенных в промоторных областях генов BDNF, COMT и CACNA1C, в исследованных группах представлено в виде графиков box-plot на рис. 1–3. Для всех шести сайтов выявленные различия между группами были статистически достоверны (р < 0.05). Результаты представлены в табл. 1.
Рис. 1.
Распределение уровней метилирования четырех сайтов гена BDNF в группах пациентов и контроля.
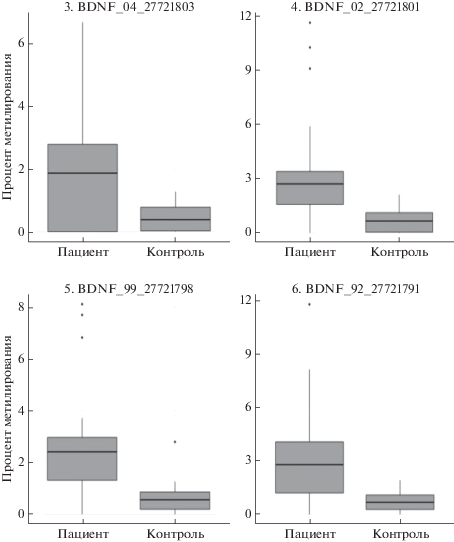
Рис. 2.
Распределение уровней метилирования сайта 2339515 гена CACNA1C в группах пациентов и контроля.
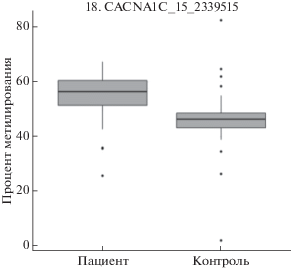
Рис. 3.
Распределение уровней метилирования сайта 19949901 гена COMT в группах пациентов и контроля.
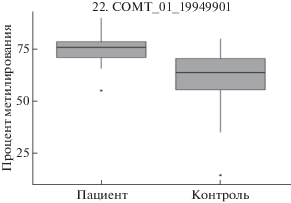
Таблица 1.
Сравнение уровней метилирования целевых сайтов генов BDNF, COMT и CACNA1C в группах пациентов и контроля
| Анализируемые CpG-сайты |
Уровень метилирования | Уровень значимости p | |
|---|---|---|---|
| пациенты | контроль | ||
| BDNF_27721791 | 2.78 | 0.67 | 0.00007816 |
| BDNF_27721798 | 2.41 | 0.55 | 0.00003795 |
| BDNF_27721801 | 2.70 | 0.65 | 0.000003296 |
| BDNF_27721803 | 1.86 | 0.38 | 0.004353 |
| COMT_19949901 | 75.87 | 63.76 | 0.000071 |
| CACNA1C_2339515 | 56.36 | 46.35 | 0.00001048 |
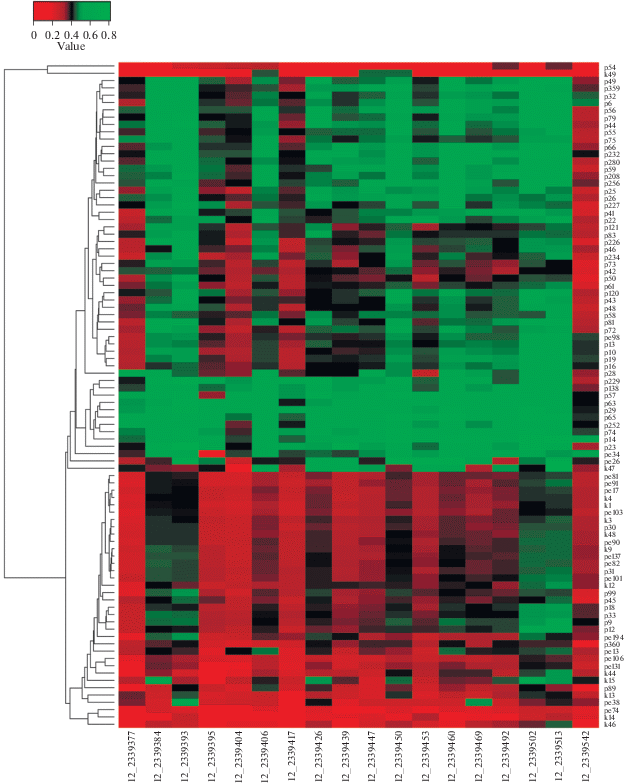
Для того, чтобы оценить, насколько изменения в уровне метилирования целевых сайтов у пациентов с личностными расстройствами и асоциальным поведением отражают общие изменения в метилировании промоторных областей генов BDNF, COMT и CACNA1C, мы дополнительно проанализировали все CpG-сайты, выявленные во фрагментах ДНК генов в исследованных ампликонах. Данные об их метилировании были также получены при проведении NGS. Для сравнительного анализа большого числа дифференциально метилированных сайтов применяли карты интенсивности сигналов (heatmap).
Наиболее значимые результаты получены для 18 сайтов, входящих в состав промотора гена CACNА1C (рис. 4). Визуальное сравнение интенсивности сигналов четко указывает на более высокий уровень метилирования у пациентов по сравнению с контролем. При иерархической кластеризации по уровню метилирования 18 сайтов гена наблюдается четкое объединение образцов ДНК пациентов в отдельные кластеры. Анализ распределения образцов показал, что построение дендрограммы на основании статуса метилирования позволяет выделить в отдельную группу более 85% пациентов.
Рис. 4.
Карта интенсивности сигналов (heatmap) дифференциально метилированных сайтов в промоторной области гена CACNА1C. Справа по вертикали – шифры образцов. По горизонтали – сайты метилирования.
Таким образом, выявлены информативные гены, анализ метилирования регуляторных областей которых может рассматриваться как высокоспецифичный тест для определения психоэмоционального статуса человека как в научных целях, так и в практике здравоохранения и криминалистики. Полученные результаты должны быть подтверждены на независимых выборках.
Работа проведена в рамках реализации мероприятия 5 “Разработка методики определения статуса генетических локусов и прижизненной модификации участков ДНК, влияющих на психоэмоциональный статус человека” Научно-технической программы Союзного государства “Разработка инновационных геногеографических и геномных технологий идентификации личности и индивидуальных особенностей человека на основе изучения генофондов регионов Союзного государства” (“ДНК-идентификация”).
Все процедуры, выполненные в исследовании с участием людей, соответствуют этическим стандартам институционального и/или национального комитета по исследовательской этике и Хельсинкской декларации 1964 г. и ее последующим изменениям или сопоставимым нормам этики.
От каждого из включенных в исследование участников было получено информированное добровольное согласие.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Sasaki A., de Vega W.C., McGowan P.O. Biological embedding in mental health: An epigenomic perspective // Biochem. and Cell. Biol. 2013. V. 9. № 1. P. 14–21. https://doi.org/10.1139/bcb-2012-0070
Provenkal N., Binder E. The neurobiological effects of stress as contributors to psychiatric disorders: Focus on epigenetics // Curr. Opinion in Neurobiol. 2015. V. 30. P. 31–37. https://doi.org/10.1016/j.conb.2014.08.007
Rozanov V.A. Epigenetics: Stress and behavior // Neurophysiology. 2012. V. 44. № 4. P. 332–350.
McEwen B. Brain on stress: How the social environment gets under the skin // Proc. Natl Acad. Sci. USA. 2012. V. 109. № 2. P. 17180–17185.
Campbell K.A., Colacino J.A., Park S.K., Bakulski K.M. Cell types in environmental epigenetic studies: Biological and epidemiological frameworks // Curr. Envir. Health Repot. 2020. V. 7. № 3. P. 185–197. https://doi.org/10.1007/s40572-020-00287-0
Hing B., Gardner C., Potash J.B. Effects of negative stressors on DNA methylation in the brain: Implications for mood and anxiety disorders // Am. J. Med. Genet. Neuropsyshiatric Genet. 2014. V. 165B. № 7. P. 541–554. https://doi.org/10.1002/ajmg.b.32265
Heller D., Grodner D., Tanenhaus M.K. The role of perspective in identifying domains of reference // Cognition. 2008. V. 108. № 3. P. 831–836. https://doi.org/10.1016/j.cognition.2008.04.008
Бабенко А.С., Моссэ К.А., Седляр Н.Г. и др. Разработка панели для анализа профиля метилирования целевых эпигенетических локусов, ассоциированных с психоэмоциональным статусом человека // Мол. и прикл. генетика. 2020. Т. 29. С. 37–48.
Shimizu E., Hashimoto K., Okamura N. et al. Alterations of serum levels of brain-derived neurotrophic factor (BDNF) in depressed patients with or without antidepressants // Biol. Psychiatry. 2003. V. 54. № 1. P. 70–75. https://doi.org/10.1016/s0006-3223(03)00181-1
Bocchio-Chiavetto L., Bagnardi V., Zanardini R. et al. Serum and plasma BDNF levels in major depression: A replication study and meta-analyses // World J. Biol. Psychiatry. 2010. V. 11. № 6. P. 763–773. https://doi.org/10.3109/15622971003611319
Terracciano A., Lobina M., Grazian Piras M. et al. Neuroticism, depressive symptoms, and serum BDNF // Psychosom. Med. 2011. V. 73. № 8. P. 638–642. https://doi.org/10.1097/PSY.0b013e3182306a4f
Minelli A., Zanardini R., Bonvicini C. et al. BDNF serum levels, but not BDNF Val66Met genotype, are correlated with personality traits in healthy subjects // Eur. Arch. Psychiatry Clin. Neurosci. 2011. V. 261. № 5. P. 323–329. https://doi.org/10.1007/s00406-011-0189-3
Dell’Osso B., D’Addario C., Carlotta Palazzo M. et al. Epigenetic modulation of BDNF gene: Differences in DNA methylation between unipolar and bipolar patients // J. Affect Disord. 2014. V. 166. P. 330–333. https://doi.org/10.1016/j.jad.2014.05.020
Strauss J.S., Khare T., De Luca V. Quantitative leukocyte BDNF promoter methylation analysis in bipolar disorder // Int. J. Bipolar Disord. 2013. V. 12. P. 1–28. https://doi.org/10.1186/2194-7511-1-28
Craddock N., Owen M., O’Donovan M. The catechol-O-methyl transferase (COMT) gene as a candidate for psychiatric phenotypes: evidence and lessons // Mol. Psychiatry. 2006. V. 11. P. 446–458. https://doi.org/10.1038/sj.mp.4001808
Chen J., Lipska B.K., Halim N. et al. Functional analysis of genetic variation in catechol-O-methyltransferase (COMT): Effects on mRNA, protein, and enzyme activity in postmortem human brain. // Am. J. Hum. Genet. 2004. V. 75. № 5. P. 807–821. https://doi.org/10.1086/425589
Hu X., Oroszi G., Chun J. et al. An expanded evaluation of the relationship of four alleles to the level of response to alcohol and the alcoholism risk // Alcohol Clin. Exp. Res. 2005. V. 29. № 1. P. 8–16. https://doi.org/10.1097/01.alc.0000150008.68473.62
Зуева И.Б., Улитина А.С., Гораб Д.Н. и др. Роль аллельных вариантов генов ангиотензин-превращающего фермента ACE и серотонинового транспортера SLC6A4 в развитии когнитивного дефицита у лиц с метаболическим синдромом // Артериальная гипертензия. 2012. Т. 18. № 6. С. 531–539.
Norrholm S.D., Jovanovic T., Smith A.K. et al. Differential genetic and epigenetic regulation of catechol-O-methyltransferase is associated with impaired fear inhibition in posttraumatic stress disorder // Front. Behav. Neurosci. 2013. V. 10. P. 7–30. https://doi.org/10.3389/fnbeh.2013.00030
Bhat S., Dao D.T., Terrillion C.E. et al. CACNA1C (Cav1.2) in the pathophysiology of psychiatric disease // Prog. Neurobiol. 2012. V. 99. № 1. P. 1–14. https://doi.org/0.1016/j.pneurobio.2012.06.001
Bagot R.C., Labonte B., Pena C.J., Nestler E.J. Epigenetic signaling in psychiatric disorders: stress and depression // Dialogues Clin. Neurosci. 2014. V. 16. № 3. P. 281–295. https://doi.org/10.31887/DCNS.2014.16.3/rbagot
Starnawska A., Demontis D., Pen A. et al. CACNA1C hypermethylation is associated with bipolar disorder // Trans. Psychiatry. 2016. V. 6. № 6. P. 1–7. https://doi.org/10.1038/tp.2016.99
Дополнительные материалы отсутствуют.


