Генетика, 2021, T. 57, № 2, стр. 165-178
Особенности quorum sensing системы SprIR Serratia proteamaculans 94 и ее участие в регуляции клеточных процессов
Ю. В. Зайцева 1, 2, В. А. Липасова 1, О. А. Кокшарова 1, 3, В. А. Плюта 1, И. В. Демидюк 1, Л. С. Чернин 4, И. А. Хмель 1, *
1 Институт молекулярной генетики Национального исследовательского центра
“Курчатовский институт”
123182 Москва, Россия
2 Ярославльский государственный университет им. П.Г. Демидова
150003 Ярославль, Россия
3 Научно-исследовательский институт физико-химической биологии им. А.Н. Белозерского, Московский государственный университет им. М.В. Ломоносова
119899 Москва, Россия
4 Отдел патологии растений и микробиологии, Иерусалимский университет
76100 Реховот, Израиль
* E-mail: khmel@img.ras.ru
Поступила в редакцию 20.03.2020
После доработки 19.05.2020
Принята к публикации 09.06.2020
Аннотация
Исследована регуляторная quorum sensing система SprIR психротрофного штамма Serratia proteamaculans 94. Сконструирован мутант с инактивированным геном sprR, кодирующим регуляторный рецепторный белок SprR. Показано, что инактивация этого гена влияла на состав жирных кислот, синтезируемых S. proteamaculans 94, и не влияла на синтез N-ацил-гомосеринлактонов (АГЛ), активности внеклеточных протеаз, хитиназ, гемолизинов, на свимминг-миграцию клеток и подавление роста мицелия фитопатогенных грибов летучими соединениями, выделяемыми этим штаммом. Инактивация гена sprI (но не гена sprR) снижала образование биопленок, которое увеличивалось при внесении в культуру экзогенного АГЛ. Проведенный сравнительный протеомный анализ клеток исходного штамма и мутантов с инактивированными генами sprI и sprR показал, что экспрессия 30 белков S. proteamaculans 94 находится под влиянием quorum sensing системы SprIR.
В последнее время растущее внимание исследователей привлекают quorum sensing (QS) системы регуляции экспрессии генов, участвующие в коммуникации бактерий и обеспечивающие скоординированный ответ популяций бактерий на внешние факторы. QS – это особый тип регуляции, функционирующий обычно при высоких плотностях бактериальных популяций. QS системы включают низкомолекулярные сигнальные молекулы различной химической природы, диффундирующие из клеток в культуральную среду, и рецепторные регуляторные белки, с которыми взаимодействуют сигнальные молекулы. QS системы функционируют как глобальные факторы регуляции, они играют важную роль в контроле большого количества клеточных процессов, относящихся к различным сторонам метаболизма бактерий: они участвуют во взаимодействии многих бактерий с высшими организмами, в регуляции вирулентности бактерий, регуляции экспрессии генов, связанных с синтезом различных экзоферментов, токсинов, антибиотиков и др. [1–7].
Больше всего исследованы QS системы LuxI/LuxR-типа грамотрицательных бактерий, использующие в качестве сигнальных молекул N‑ацил-гомосеринлактоны (АГЛ). QS системы этого типа можно разделить на две группы в зависимости от функций и организации экспрессии QS генов.
Первая группа включает классическую QS систему Vibrio fischeri; у этой морской бактерии впервые был открыт феномен QS. В QS-регуляции V. fischeri участвуют два основных регуляторных компонента: белок LuxI – синтаза АГЛ (катализирует синтез АГЛ) и LuxR-белок – он образует димер, присоединяет АГЛ, в результате чего изменяется его конфигурация; затем этот комплекс, связываясь с промотором lux-оперона, активирует его транскрипцию, что приводит к синтезу люциферазы и эмиссии света. При увеличении популяции V. fischeri АГЛ накапливаются до порогового уровня, который достаточен для активации LuxR, связывания его с промоторной областью lux-оперона и индукции этого оперона. Сходные QS системы были обнаружены в клетках различных видов псевдомонад, Agrobacterium tumefaciens, Burkholderia и др. [1, 3–7].
Вторая группа включает QS системы бактерий семейства Enterobacteriaceae (Pantoea, Erwinia, Serratia и др.). Регуляторные R-белки в QS системах этой группы функционируют главным образом как негативные регуляторы; они могут репрессировать транскрипцию своих собственных генов в отсутствие АГЛ.
Количество АГЛ увеличивается с увеличением плотности популяции бактерий по мере роста культуры. При достижении достаточно высокой концентрации АГЛ взаимодействует с белком LuxR-типа (например, с белком EsaR P. stewartii), при этом происходят изменения его конформации, препятствующие связыванию с ДНК, что приводит к дерепрессии [8–12].
Объектом нашего исследования была бактерия Serratia proteamaculans 94 [13]. QS системы бактерий этого вида изучены мало [11]. В предыдущей работе [14] мы показали, что штамм S. proteamaculans 94 синтезирует два главных типа сигнальных молекул АГЛ (N-3-оксо-гексаноил-L-гомосеринлактон и N-3-гидрокси-гексаноил-L-гомосеринлактон) и несколько минорных АГЛ. Гены sprI и sprR этой QS системы, кодирующие соответственно синтазу АГЛ и рецепторный регуляторный белок SprR, были клонированы и секвенированы. Было показано, что гены sprI и sprR транскрибировались конвергентно с частичным перекрыванием их рамок считывания. Изучение полученного нами мутанта с инактивированным QS геном sprI позволило определить роль этого гена в регуляции ряда клеточных процессов. Инактивация гена sprI приводила к отсутствию синтеза АГЛ, уменьшению внеклеточной протеолитической активности, отсутствию хитинолитической активности, свимминг-миграции клеток (swimming motility), отсутствию или сильному снижению подавления роста мицелия фитопатогенных грибов летучими соединениями, выделяемыми штаммом 94, изменению состава клеточных жирных кислот, синтезируемых этим штаммом [14].
В штамме S. proteamaculans 94 нами была обнаружена также сигнальная молекула типа AI-2 и исследована функциональная роль гена luxS, ответственного за синтез AI-2. Инактивация этого гена приводила к действию, сходному с действием инактивации гена sprI на синтез экзоферментов, свимминг-миграцию клеток, и вызывала снижение или отсутствие ингибирования фитопатогенных грибов летучими веществами, выделяемыми S. proteamaculans 94 [15].
В настоящей работе был получен мутант с инактивированным геном sprR QS системы штамма S. proteamaculans 94, кодирующим рецепторный регуляторный белок SprR. Ранее мутации гена, кодирующего R-белок S. proteamaculans, не были получены, и информация о роли этого гена в контроле метаболизма S. proteamaculans отсутствует. В работе изучено влияние инактивации гена sprR на регуляцию клеточных процессов S. proteamaculans 94; исследовано действие инактивации генов sprI и sprR на образование биопленок этого штамма, проведен сравнительный протеомный анализ экспрессии белков исходного и мутантных штаммов. Полученные данные расширяют наше представление о диапазоне клеточных процессов, в регуляции которых участвуют гены QS системы S. proteamaculans.
МАТЕРИАЛЫ И МЕТОДЫ
1. Штаммы бактерий и условия культивирования
Штаммы бактерий, плазмиды и праймеры ПЦР, использованные в работе, приведены в табл. 1, 2. Бактерии выращивали на средах Luria Broth (LB), LA (Sigma-Aldrich Chemie GmbH, Steinheim, Germany) и M9 [16] c необходимыми добавками при температуре 28–30°C. Антибиотики отечественного производства использовали в концентрациях (мкг/мл): ампициллин, 100–200; канамицин, 100; гентамицин 40. Тетрациклин (Sigma) добавляли в среду в концентрации 20 мкг/мл.
Таблица 1.
Штаммы бактерий и плазмиды, использованные в работе
| Штаммы и плазмиды | Характеристика | Источник или ссылка |
|---|---|---|
| Штаммы | ||
| Serratia proteamaculans 94 | Выделен из испорченного в холодильнике мяса | [13] |
| S. proteamaculans 94 rif-r | Спонтанный мутант rif-r | [14] |
| Escherichia coli S17-1 (λ-pir) | thi pro hsdR hsdM recA rpsL RP4-2 (Tcr::Mu) (Kmr::Tn7) (λ -pir) | Коллекция ИМГ РАН |
| E. coli TG-1 | K-12 supE thi-1 Δ(lac-proAB) Δ(mcrB-hsdSM)5, $(r_{K}^{ - }m_{K}^{ - })$ F' [tra D36proAB+ lacIqlac ZΔM15] |
Stratagene |
| Chromobacterium violaceum CV026 | Биосенсор для определения АГЛ на основе продукции виолацеина, Km-r | [17] |
| Agrobacterium tumefaciens NT1/pZLR4 | pZLR4 traG::lacZ. Биосенсор для определения АГЛ на основе индукции синтеза β-галактозидазы, Gmr Cbr | [18] |
| S. proteamaculans 94 rif-r sprI::Gm | Мутант sprI::Gm штамма 94 rif-r | [14] |
| Плазмиды | ||
| p34S-Gm | Источник Gmr-кассеты | [21] |
| pEX18Tc | Вектор для замены генов, Tetr oriT sacB | [20] |
| pAL-TA | Вектор для клонирования продуктов ПЦР, Apr ori pUC Plac | Eurogen |
2. Определение продукции АГЛ
Для определения продукции АГЛ использовали два биосенсора. Сенсор Chromobacterium violaceum CV026 высевали штрихами на поверхность среды LA, пересекали их штрихами тестируемых культур, инкубировали 24–48 ч при 30°С. В случае, когда штамм продуцировал АГЛ, наблюдали окрашивание индикаторного штамма CV026 в фиолетовый цвет. Интенсивность окраски оценивали визуально [17]. Второй биосенсор Agrobacterium tumefaciens NT1/pZLR4 выращивали на среде LB с добавлением ампициллина (100 мкг/мл) и гентамицина в течение ночи при 30°С. Чашку с агаризованной средой М9 с добавленным X-Gal (конечная концентрация 80 мкг/мл) заливали 3 мл M9 c 0.5% агара с добавлением 0.5 мл ночной культуры A. tumefaciens NT1/pZLR4. Тестируемые на продукцию АГЛ штаммы высевали уколами на поверхность агаризованной среды после ее застывания или вносили жидкую ночную культуру в лунки в агаризованной среде и инкубировали при 30°С 24–48 ч. О синтезе АГЛ штаммами судили по появлению голубых зон гидролиза X‑Gal [18].
3. Манипуляции с ДНК
Выделение суммарной и плазмидной ДНК, рестрикцию, агарозный гель-электрофорез, лигирование, трансформацию E. coli проводили согласно методикам, описанным в [19]. Реакцию ПЦР проводили в 20 мкл реакционной смеси, содержащей однократный буфер для Taq ДНК-полимеразы (НПО “СибЭнзим”), по 250 мкМ dGTP, dATP, dCTP и dTTP (НПО “СибЭнзим”), 10 пМ каждого праймера (ЗАО “Синтол”), 0.5 U Taq ДНК-полимеразы (ИМГ РАН). В качестве матрицы для ПЦР использовали плазмидные ДНК или прокипяченные клетки, взятые из свежевыращенных колоний. Реакцию ПЦР-амплификации проводили на четырехканальном программируемом термостате ТП4-ПЦР-01-“Терцик” (ЗАО “НПФ ДНК-Технология”).
4. Получение мутанта S. proteamaculans 94 c инактивированным геном sprR
Мутант с нокаутом гена sprR был получен методом замены генов с использованием sacB-стратегии [20]. Ранее для клонирования генов sprI и sprR и их секвенирования нами был клонирован фрагмент ДНК размером 1660 пн, содержащий оба гена, и была определена нуклеотидная последовательность этих генов (JX901285) [14]. Для получения клонированного гена sprR была проведена ПЦР-амплификация с праймерами для этого гена – SprR-F и SprR-R (табл. 2). ПЦР проводили при 94°С 2 мин, затем 30 циклов при 94°С 20 с, 58°С 20 с, 72°C 40 с, и последняя стадия была при 72°C 4 мин. Продукты ПЦР были выделены из геля (использовали Wizard SV Gel kit и PCR Clean-Up System, Promega) и лигированы с pAL-TA векторной ДНК. Лигазная смесь была трансформирована в E. coli TG1. Клетки высевали на среду LA с добавлением 200 мкг/мл ампициллина, наличие sprR гена в отобранных клонах определяли методом ПЦР, используя универсальные праймеры M13. Сконструированная плазмида была названа pAL-TA-sprR.
Для конструирования sprR::Gm мутанта BamHI‑фрагмент ДНК размером 865 пн плазмиды p34S-Gm [21], несущий ген резистентности к гентамицину, был клонирован в BglII-сайт внутри гена sprR в pAL-TA-sprR плазмиде. Фрагмент ДНК 1700 пн из полученной плазмиды pAL-TA-sprR::Gm был клонирован в EcoRI-сайт вектора pEX18Tc [20], и сконструированная в результате плазмида pEX18Tc-sprR::Gm была трансформирована в E. coli S17-1 (λ-pir), который был использован как донор для передачи этой плазмиды в S. proteamaculans 94 rif-r штамм конъюгацией. Полученные клетки были высеяны на среду LA, содержащую Gm, Rif и 5% (wt/vol) сахарозы, после чего клоны, резистентные к гентамицину, рифампицину и сахарозе и чувствительные к тетрациклину, были тестированы с помощью ПЦР с праймерами Gm-F, GmR и праймерами sprR-F и sprR-R. В результате был получен мутантный штамм S. proteamaculans 94 sprR::Gm.
5. Анализ биопленок
Свежие культуры, выросшие на среде LA, высевали в LB с необходимыми антибиотиками и инкубировали при аэрации в течение 24 ч при температуре 30°C. Затем культуры разбавляли в 300 раз в LB без антибиотиков. Для измерения образования биопленок культуры выращивали в полистироловых планшетах 24 ч с перемешиванием на качалке при температуре 30°C. Образование биопленок измеряли после удаления среды, промывки ячеек водой и окрашивания прикрепленных клеток красителем кристаллический фиолетовый (1 г кристалвиолета, РЕАХИМ, Россия; 2 г кристаллического фенола; 90 мл дистиллированной воды; 10 мл 96%-ного этанола) в течение 45 мин при комнатной температуре. После окрашивания жидкость сливали, краситель из биопленок экстрагировали 96%-ным этанолом, оптическую плотность раствора измеряли при 595 нм. Для измерения уровня образованных биопленок использовали Model 2550 Microplate Reader (Bio-Rad, США). По интенсивности окрашивания раствора судили об уровне образования биопленок. Каждый эксперимент повторяли три раза, при этом в каждом варианте культуру выращивали в 4–8 лунках.
6.1. Определение ферментативных активностей
Для определения активностей внеклеточных ферментов 3 мкл разведенной в 300 раз ночной культуры бактерий помещались на поверхность среды LA (1.5% агара) с необходимыми добавками. Эксперименты со всеми вариантами проводились 3 раза, с не менее чем тремя повторностями в каждом опыте [14].
Определение внеклеточной протеазной активности. Клетки штаммов высевали уколом на агаризованную среду LA с молоком (молоко с 0.5% жира составляло 1/3 часть от общего объема среды), инкубировали 24–48 ч при 30°С. В случае, когда штаммы обладали внеклеточной протеазной активностью, вокруг колоний наблюдали зоны ферментативного гидролиза казеина молока (зоны просветления). О ферментативной активности судили по радиусу зон гидролиза.
Определение липазной активности. Клетки тестируемых штаммов высевали уколом на среду LA с добавлением Tween-20 (1%) и CaCl2 (0.01%). Инкубировали 48 ч при 30°С. В случае, когда штаммы обладали липазной активностью, вокруг колоний наблюдали мутные зоны (Tween-20 гидролизовался с образованием лауриновой кислоты, что приводило к образованию в присутствии кальция нерастворимой соли этой кислоты). О ферментативной активности судили по радиусу непрозрачных зон вокруг колоний и степени помутнения среды в пределах непрозрачной зоны.
Определение хитинолитической активности. Тестируемые штаммы высевали уколом на агаризованную среду (1.5% агара) следующего состава: 0.1% (NH4)2SO4, 0.03% MgSO4 · 7H2O, 0.08% KH2PO4, 0.04% KNO3, 0.05% дрожжевой экстракт (Difco); в среду добавляли 0.2% коллоидного хитина. Инкубировали 72–96 ч при 30°С. В случае, когда штаммы обладали хитинолитической активностью, около колоний наблюдали зоны ферментативного гидролиза (зоны просветления). О ферментативной активности судили по радиусу зон.
Определение гемолитической активности. Клетки тестируемых штаммов высевали уколом на кровяной агар (среда LA с добавлением 5% стерильной крови человека), инкубировали 3–7 суток при 28°С. О гемолитической активности судили по прозрачным зонам гемолиза вокруг колоний.
6.2. Определение действия летучих соединений, выделяемых S. proteamaculans 94
Ранее мы показали, что S. proteamaculans 94 синтезирует летучие вещества (ЛВ), в том числе органические летучие вещества, из которых в наибольшем количестве штамм продуцировал диметилдисульфид (ДМДС). Было показано, что общий пул летучих соединений, выделяемых этим штаммом, и индивидуально ДМДС подавляют рост агробактерий, фитопатогенных грибов, нематод и дрозофил [22]. Для определения влияния инактивации гена sprR на действие ЛВ на грибы были использованы чашки Петри, разделенные на две части перегородками, которые не прилегали к крышке, оставалось свободное пространство для обмена воздухом. Одну половину чашки засевали S. proteamaculans 94 или мутантом sprR::Gm на среде LA (20 мкл ночной культуры, выросшей в LB и содержащей ~4–6 × 107 клеток распределялись микробиологической петлей по поверхности LA). Чашки инкубировали 24 ч при 28°C. После этого на вторую половину чашки помещали блок с мицелием гриба. Блоки грибов диаметром 8 мм вырезали из агаризованной среды Чапека на чашках Петри, засеянных грибами в виде спор петлей с культуры гриба, после роста в течение пяти суток при 25°С. Чашки плотно герметизировали парафильмом (4 раза) (“Parafilm M” Sealing Film (Pechiney Plastic Packaging Company, Chicago, IL, США)). Рост грибов анализировали через 3–6 дней при 25°C [22].
6.3. Анализ состава жирных кислот
Ночные культуры бактерий разводили до OD600 ~ 0.10, и клетки росли в течение 20 ч при 30°C. Пробы сухой биомассы клеток (5 мг) обрабатывали в 0.4 мл 1.2 N хлористого водорода в метаноле при 80°С в течение одного часа (кислый метанолиз). Образовавшиеся метиловые эфиры жирных кислот экстрагировали дважды гексаном. Далее проводили газовую хроматографию и масс-спектрометрию. Параметры хроматографии выбирали как рекомендовано в Microbial Identification System (MIS) [23, 24] с Hewlett-Packard 5890A газовым хроматографом и Hewlett-Packard 7673A автоматическим сэмплером. Хромато-масс-спектрометрический анализ (GS-MS) проводили на хромато-масс-спектрометре AT-5975 Agillent Technologies (США). Для хроматографического разделения пробы использовали капиллярную колонку HP-5ms. Хроматографирование проводили в режиме программирования температуры от 135 до 320°C со скоростью 7 град/мин. Обработку данных проводили с помощью штатных программ прибора. Вещества в хроматографических пиках идентифицировали с помощью библиотечных программ с базой данных масс-спектров NIST.
7. Протеомный анализ
Сравнительный протеомный анализ в клетках S. proteamaculans 94 и двух мутантных штаммов был проведен в НИИ биомедицинской химии РАМН им. Ореховича, Москва.
Экстракция и разделение белков. Культуры бактерий выращивали при 30°С в течение 17 ч. Клетки осаждали центрифугированием при 10 000 об./мин 5 мин, при 4°С, отмывали 3 раза стандартным фосфатным буфером (137 мМ NaCl, 2.7 мМ KCl, 10.1 мМ Na2HPO4, 1.8 мМ KH2PO4, pH 7.4), затем центрифугировали. Супернатант удаляли, а осадок замораживали и хранили при –80°C. Для экстракции белков осадки бактериальных клеток ресуспендировали в буфере, содержащем 7 M мочевины, 2 M тио-мочевины, 2% CHAPS, 0.5% Triton X100, 20 мM Tris-base, 2 мM MgCl2, 5 мM PMSF, 65 мM DTT и 1 мM Na2EDTA, и подвергали обработке ультразвуком. Для каждого образца с целью оценки качества экстракции белков проводился аналитический одномерный, а затем двумерный электрофорез при использовании программного обеспечения Quantity One software (BioRad, США). Стандартное нанесение составляло около 150 мкг на 24 см геля.
Двумерный (2-Д) электрофорез. Для проведения электрофореза в первом направлении осуществляли гидратацию 24 см полосок (IPG Strips) с нанесенным на них гелем с pH 3–10 (Agilent, США) в буфере, содержащем 7 M мочевины, 2 M тиомочевины, 4% CHAPS, 65 мM DTT, 1.25% амфолитов 3-10 (Agilent) в течение ночи. Образцы в этом же буфере наносили на стрипы и проводили форез в первом направлении при 80 кВ на приборе Agilent Technologies 3100 Offgel Fractionator (Agilent). Второе направление электрофореза (SDS-PAGE) осуществлялось в 12%-ном полиакриламидном геле. Буфер для электрофореза содержал: Трис, 25 мМ; глицин, 192 мМ; SDS, 0.1%. После фиксации геля в растворе, содержащем 10% уксусной кислоты и 20% этанола, проводили его окрашивание нитратом серебра [25].
Анализ изображений 2-Д гелей. Окрашенные гели сканировали на сканере Bio-Rad GS-800 Calibrated Densitometer. Интенсивность окрашивания белковых пятен сравнивали, используя программное обеспечение Melanie III. Пятна, различавшиеся между образцами по крайней мере в 1.5 раза по интенсивности окрашивания (представлено в процентах), вырезали из геля и анализировали с помощью MALDI-TOF.
Времяпролетная масс-спектрометрия с матрично-активированной лазерной десорбцией/ионизацией (MALDI-TOF MS анализ) и идентификация белков. Обработка вырезанных из геля окрашенных серебром белковых пятен осуществлялась по методу Shevchenko et al. [26]. Масс-спектры регистрировали с использованием масс-спектрометра Ultraflex MALDI-TOF-TOF (Bruker Daltonics, Германия), снабженного азотным (N2) лазером (О-350 нм). Список пиков пептидов был создан в программном обеспечении Flex Analysis 2.4 (Bruker Daltonics). Идентификация белка в соответствии с содержимым пептида проводилась с использованием программного обеспечения Mascot 2.3 (Matrix Science, США), соответствующего базе данных NCBI для таксонов Eubacteria.
Результаты, полученные в трех независимых экспериментах, были выражены в виде среднего значения с учетом среднеквадратичного отклонения (SD) и рассчитаны с помощью программного обеспечения GraphPad PRISM 5.0 (GraphPad Software Inc.).
РЕЗУЛЬТАТЫ
1. Получение мутанта S. proteamaculans 94 c инактивированным геном sprR
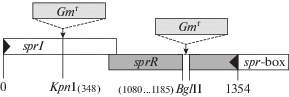
Для изучения особенностей функционирования SprI/SprR QS системы S. proteamaculans 94 нами ранее был получен мутант с инактивированным геном sprI [14]. В настоящей работе мы получили методом замены гена мутант sprR::Gm с нокаутом второго гена SprI/SprR QS системы этой бактерии, кодирующего рецепторный регуляторный белок SprR. Мутация стабильно сохранялась в клетках S. proteamaculans 94, поддерживаемых на среде с гентамицином (40 мкг/мл). Анализ синтеза АГЛ, проведенный с двумя биосенсорами в чашках Петри на среде LA, показал, что мутантный штамм активно продуцирует АГЛ. Локализация мутаций sprR::Gm и sprI::Gm показана на рис. 1.
2. Влияние QS системы SprI/SprR на формирование биопленок у S. proteamaculans 94
В регуляции формирования биопленок различными бактериями важную роль играют QS системы регуляции экспрессии генов [12, 27, 28]. Для представителей вида S. proteamaculans вопрос о взаимодействии QS систем с образованием биопленок не исследовался.
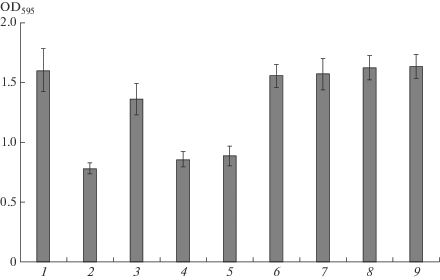
При исследовании влияния инактивации генов sprI и sprR на регуляцию образования биопленок у S. proteamaculans 94 было показано, что мутация в гене sprR не оказывала влияния на уровень образования биопленок (рис. 2). Как было отмечено выше, эта мутация не влияла на синтез АГЛ. Напротив, в мутантном по гену sprI штамме 94 образование биопленок снижалось вдвое по сравнению с уровнем биопленок в исходном штамме. Ранее мы показали, что клетки этого мутанта не продуцировали АГЛ [14]. Добавление экзогенного 3-оксо-С6-ГЛ, одного из двух доминантных АГЛ, синтезируемых штаммом S. proteamaculans 94 дикого типа [14], восстанавливало способность мутанта к формированию биопленки практически до уровня исходного штамма. Добавление других видов АГЛ, синтезируемых клетками S. proteamaculans 94 в значительно меньшем количестве (С4-ГЛ, С6-ГЛ), не давало подобного эффекта (рис. 2). Полученные результаты показывают зависимость образования биопленок у S. proteamaculans 94 от синтеза АГЛ; существенную роль в контроле формирования биопленок играет 3-оксо-С6-ГЛ.
Рис. 2.
Образование биопленок исходным штаммом S. proteamaculans 94 (1), мутантным штаммом sprI::Gm (2), мутантным штаммом sprI::Gm с добавлением: 3-оксо-С6-ГЛ (3), С6-ГЛ (4), С4-ГЛ (5); мутантным штаммом sprR::Gm (6), мутантным штаммом sprR::Gm с добавлением: 3-оксо-С6-ГЛ (7), С6-ГЛ (8), С4-ГЛ (9). Все АГЛ добавляли в конечной концентрации 25 нМ.
Эффект мутации в гене sprI на формирование биопленок у S. proteamaculans 94 проявлялся также при проведении опытов в стеклянных пробирках на среде LB. У исходного штамма S. proteamaculans 94 и мутанта sprR::Gm на стенках пробирок было заметно образование рыхлых биопленок, в то время как биопленки у мутанта S. proteamaculans sprI::Gm на стенках пробирок не образовывались (рис. 3). Мутация в гене sprI изменяла также характер роста S. proteamaculans 94 в жидкой среде. Для штаммов дикого типа и мутанта по гену sprR было характерно образование многоклеточных агрегатов и формирование осадка, а для мутанта S. proteamaculans 94 по гену sprI был характерен диффузный планктонный рост с равномерным помутнением среды.
3. Изучение влияния мутации sprR::Gm на активности экзоферментов, синтезируемых S. proteamaculans 94
Известно, что QS системы являются глобальными регуляторами экспрессии генов бактерий. Поэтому нас интересовала роль QS в регуляции клеточных процессов, относящихся к различным сторонам метаболизма S. proteamaculans 94. Было исследовано, как влияет нокаут гена QS системы sprR на активность некоторых внеклеточных ферментов. Мы показали, что протеазная активность у мутантного штамма sprR::Gm практически не отличалась от активности исходного штамма, в отличие от активности этого фермента у штамма с мутацией sprI::Gm, где протеолитическая активность была снижена [14]. Мутант sprR::Gm не отличался по хитинолитической активности от исходного штамма, в то время как у мутантного штамма sprI::Gm хитинолитическая активность отсутствовала. Липазная и гемолитическая активности клеток мутантов sprR::Gm и sprI::Gm не отличались от активности исходного штамма S. proteamaculans 94 (данные не приводятся).
4. Действие летучих соединений, выделяемых S. proteamaculans 94 и sprR:Gm мутантом, на фитопатогенные грибы
Мы сравнили действие общего пула летучих соединений, выделяемых S. proteamaculans 94 и sprR::Gm мутантом, на рост мицелия двух фитопатогенных грибов – Rhizoctonia solani и Helminthosporium sativum. По данным проведенных экспериментов, мутант sprR::Gm подавляет рост мицелия гриба и обладает ингибирующим действием, сходным с действием S. proteamaculans 94, т.е. мутация sprR::Gm, по-видимому, не затрагивала гены, ответственные за регуляцию синтеза летучих соединений (данные не приводятся).
5. Анализ состава бактериальных жирных кислот (ЖК)
Методом хромато-масс-спектрометрии было проведено сравнение состава жирных кислот у штамма S. proteamaculans 94 дикого типа и мутантного штамма S. proteamaculans sprR::Gm (табл. 3). В образцах исследуемых штаммов были обнаружены насыщенные, ненасыщенные и гидроксикислоты. Всего в спектрах были идентифицированы 22 жирные кислоты с длиной цепи от 12 до 20 атомов углерода.
Таблица 3.
Состав жирных кислот в штаммах S. proteamaculans 94 (A) и мутанте S. proteamaculans sprR::Gm (B)
| № | R.T. | Жирная кислота | Содержание жирных кислот, % от суммы площадей всех хроматографических пиков | |
|---|---|---|---|---|
| A | B | |||
| 1 | 6.983 | Додекановая | 0.5 ± 0.1 | 0.9 ± 0.1 |
| 2 | 9.495 | 9-Тетрадеценовая | 0 | 0.4 ± 0.0 |
| 3 | 9.578 | 11-Тетрадеценовая | 0 | 0.6 ± 0.1 |
| 4 | 9.873 | Тетрадекановая | 2.7 ± 0.3 | 5.5 ± 0.2 |
| 5 | 10.120 | 2-Гидрокси-додекановая | 0 | 0.7 ± 0.1 |
| 6 | 11.305 | Пентадекановая | 0.6 ± 0.1 | 1.3 ± 0.3 |
| 7 | 12.379 | 7-Гексадеценовая | 0.4 ± 0.1 | 0 |
| 8 | 12.449 | 9-Гексадеценовая | 3.4 ± 1.2 | 20.7 ± 0.8 |
| 9 | 12.797 | Гексадекановая | 37.2 ± 1.7 | 36.1 ± 1.6 |
| 10 | 12.839 | 3-Гидрокси-тетрадекановая | 0.6 ± 0.1 | 8.8 ± 0.5 |
| 11 | 13.570 | Изо-гептадекановая | 0.2 ± 0.1 | 0 |
| 12 | 13.694 | Антеизо-гептадекановая | 0.3 ± 0.1 | 0 |
| 13 | 13.764 | Гептадеценовая | 0.2 ± 0.0 | 0 |
| 14 | 13.888 | Циклопропан-гептадекановая | 0.2 ± 0.1 | 10.2 ± 0.4 |
| 15 | 14.071 | Гептадекановая | 0.8 ± 0.1 | 1.9 ± 0.2 |
| 16 | 15.144 | 9-Октадеценовая | 33.2 ± 1.3 | 8.2 ± 0.5 |
| 17 | 15.186 | 11-Октадеценовая | 4.6 ± 0.2 | 0.4 ± 0.1 |
| 18 | 15.427 | Октадекановая | 11.0 ± 0.3 | 3.4 ± 0.2 |
| 19 | 16.170 | Октадекадиеновая, конъюгированная | 1.3 ± 0.1 | 0.9 ± 0.2 |
| 20 | 17.556 | 9-Эйкозеновая | 1.5 ± 0.2 | 0 |
| 21 | 17.597 | 11-Эйкозеновая | 0.9 ± 0.2 | 0 |
| 22 | 17.845 | Эйкозановая | 0.5 ± 0.1 | 0 |
| Коэффициент ненасыщенности жирных кислот | 0.83 | 0.45 | ||
В спектре штамма дикого типа S. proteamaculans 94 идентифицировано 19 жирных кислот. Основными насыщенными кислотами являлись гексадекановая, октадекановая и тетрадекановая. Доминирующими ненасыщенными кислотами были 9-октадеценовая, 11-октадеценовая и 9-гексадеценовая. Коэффициент ненасыщенности жирных кислот, определенный как отношение ∑ненасыщенных ЖК/∑насыщенных ЖК, у штамма S. proteamaculans 94 был 0.83.
Состав жирных кислот мутанта sprR::Gm существенно отличался от состава жирных кислот родительского штамма S. proteamaculans 94 (см., например, № 10, 14, 16, 17, 18). Мутация в этом гене могла приводить как к снижению, так и к увеличению относительного содержания жирных кислот. Содержание жирных кислот мутанта характеризовалось более низким коэффициентом ненасыщенности, сравнительно с исходным штаммом (0.45). Спектр жирных кислот мутанта sprR::Gm отличался от спектра исходного штамма 94 дикого типа резко увеличенным содержанием циклопропан-гептадекановой и 3-гидрокси-тетрадекановой кислот (в 50- и 15 раз соответственно). Несколько жирных кислот (7, 11–13, 20–22) отсутствовали у мутантного штамма, сравнительно с S. proteamaculans 94. Три жирные кислоты (2, 3, 5), отсутствовавшие у штамма 94, были обнаружены у мутантного штамма в количестве меньше 1%. Данные, приведенные выше, показывают, таким образом, что инактивация гена sprR S. proteamaculans 94 значительно влияла на состав жирных кислот бактерии.
6. Сравнение протеомных карт культур S. proteamaculans 94 и мутантов sprI::Gm и sprR::Gm
Для характеристики полученных мутантов использовали метод протеомного анализа синтезируемых ими белков, сравнительно с белками исходного штамма. Более чем 600 белковых пятен выявилось на каждом из 2-Д гелей, среди которых 30 белковых пятен отличались по интенсивности окрашивания, отражающей существенные количественные изменения в протеомах бактерий дикого типа и мутантов (в 1.5 и более раз).
В случае мутанта sprI::Gm экспрессия 20 белков изменялась по сравнению со штаммом дикого типа: экспрессия 11 белков значительно усиливалась, в то время как экспрессия 9 белков снижалась. В случае мутанта S. proteamaculans sprR::Gm экспрессия 14 белков изменялась по сравнению со штаммом дикого типа: экспрессия двух белков заметно усиливалась, а экспрессия 12 белков снижалась (табл. 4).
Таблица 4.
Белковые пятна, демонстрирующие статистически достоверные отличия, и их относительные интенсивности
| № | %V | № | %V | ||
|---|---|---|---|---|---|
| S. prot. 94 | sprI::Gm | S. prot. 94 | sprR::Gm | ||
| 175 | 0.15 ± 0.00 | 0.09 ± 0.02 | 64 | 0.03 ± 0.00 | 0 |
| 187 | 0.37 ± 0.01 | 0.23 ± 0.02 | 131 | 0.11 ± 0.01 | 0.06 ± 0.00 |
| 195 | 1.00 ± 0.11 | 0.36 ± 0.17 | 142 | 0.41 ± 0.06 | 0.18 ± 0.02 |
| 232 | 0.15 ± 0.01 | 0.08 ± 0.00 | 187 | 0.37 ± 0.01 | 0.15 ± 0.05 |
| 304 | 0.06 ± 0.00 | 0 | 190 | 0.31 ± 0.00 | 0.06 ± 0.01 |
| 477 | 0.59 ± 0.07 | 1.05 ± 0.07 | 195 | 1.00 ± 0.11 | 0.09 ± 0.02 |
| 487 | 0.14 ± 0.01 | 0.24 ± 0.02 | 237 | 0.08 ± 0.01 | 0 |
| 495 | 0.46 ± 0.08 | 0.69 ± 0.02 | 376 | 0.06 ± 0.00 | 0 |
| 524 | 0.14 ± 0.00 | 0.22 ± 0.01 | 391 | 0.04 ± 0.01 | 0 |
| 540 | 0.31 ± 0.08 | 0.69 ± 0.06 | 406 | 0.20 ± 0.01 | 0.09 ± 0.01 |
| 546 | 0.07 ± 0.01 | 0.17 ± 0.01 | 408 | 0.14 ± 0.03 | 0 |
| 560 | 0.11 ± 0.03 | 0 | 447 | 0.03 ± 0.01 | 0.94 ± 0.16 |
| 595 | 0.15 ± 0.01 | 0.43 ± 0.01 | 596 | 0 | 1.77 ± 0.41 |
| 596 | 0 | 1.97 ± 0.11 | 635 | 0.04 ± 0.01 | 0 |
| 610 | 0.11 ± 0.00 | 0.23 ± 0.02 | |||
| 617 | 0.26 ± 0.01 | 0.54 ± 0.04 | |||
| 619 | 0.30 ± 0.05 | 1.16 ± 0.27 | |||
| 621 | 0.31 ± 0.02 | 0.08 ± 0.02 | |||
| 631 | 0.14 ± 0.03 | 0 | |||
| 642 | 0.61 ± 0.07 | 0.05 ± 0.07 | |||
Примечание. Растворимые белки из клеток S. proteamaculans 94 и двух мутантов (sprI::Gm и sprR::Gm) были проанализированы путем разделения на двумерных гелях (2-Д гелях); в первом направлении – по суммарному заряду белков в диапазоне рI 4–7, во втором направлении – по относительной молекулярной массе в районе 75–10 кДа. Каждый штамм представлен тремя независимыми биологическими повторностями. Всего было проанализировано девять гелей (по три для каждого образца). Полужирным шрифтом выделены белки, которые удалось идентифицировать. Значение 0 соответствует отсутствию пятна в данных координатах на геле.
Выявленные по разнице в интенсивности белковые пятна были проанализированы при помощи MALDI-TOF масс-спектрометрии. В итоге удалось успешно идентифицировать восемь белков (табл. 5). В обоих мутантах наблюдался повышенный уровень экспрессии белков, связанных с устойчивостью штаммов к антибиотикам. Наличие аминогликозид-(3)-N-ацетилтрансферазы (596) в мутантных штаммах, которая отсутствует в штамме дикого типа, определяет их устойчивость к гентамицину. Аминогликозид-(3)-N-ацетилтрансфераза кодируется геном accC1, который несет гентамициновая кассета из p34S-Gm [21], использованная при получении мутантов. У мутанта sprR::Gm была значительно повышена экспрессия β-лактамазы (447). В обоих мутантах наблюдалось значительное снижение экспрессии ФМН-редуктазы (195). У мутанта sprI::Gm наблюдалось усиление экспрессии белков, участвующих в защите клеток от стрессов (619, 617, 540). У мутантного штамма sprR::Gm происходило снижение уровня экспрессии сукцинил-СoA-синтетазы. Таким образом, мутации в генах, продукты которых были вовлечены в QS-регуляцию, приводили к изменению экспрессии белков, существенных для метаболизма клеток S. proteamaculans.
Таблица 5.
Белки, идентифицированные в S. proteamaculans 94
| № | Название белка | Предполагаемые М.в./pI |
Номер в базе данных NCBI |
|---|---|---|---|
| Цикл трикарбоновых кислот | |||
| 406 | Сукцинил-CoA-синтетаза (α-субъединица) | 29.8/5.9 | 157369513 |
| 190 | Сукцинил-CoA-синтетаза (β-субъединица) | 41.3/5.3 | 157369512 |
| Окислительно-восстановительные реакции | |||
| 195 | ФМН-редуктаза | 26.2/5.0 | 157368508 |
| Белки общего ответа на стресс | |||
| 540 | Белок теплового шока GrpE | 21.4/4.9 | 157371919 |
| Защита от окислительного стресса | |||
| 619 | Тиоредоксин 2 | 15.6/4.9 | 157371981 |
| 617 | Тиоредоксин-зависимая тиолпероксидаза | 17.3/5.0 | 157371748 |
| Устойчивость к антибиотикам | |||
| 596 | Аминогликозид-(3)-N-ацетилтрансфераза | 19.4/5.8 | 239721 |
| 447 | β-Лактамаза | 31.7/5.7 | 46129905 |
ОБСУЖДЕНИЕ
Феномен QS-регуляции вызывает в настоящее время большой интерес исследователей, работающих в различных фундаментальных и прикладных областях биологии, медицины и биотехнологии. Показано, что этот тип регуляции широко распространен у бактерий различных таксономических групп.
Настоящая работа посвящена изучению QS систем грамотрицательных бактерий Serratia proteamaculans. Нас интересовало изучение роли QS систем в регуляции ряда метаболических процессов, существенных для жизнедеятельности этих бактерий. Бактерии этого вида характеризуются приспособленностью к обитанию в различных экологических нишах – они используют различные субстраты для питания, живут в почве, в ризосфере растений, участвуют в порче сельскохозяйственных продуктов [11, 13, 29]. QS системы у бактерий S. proteamaculans изучены мало, до наших работ [14, 15] имелась только одна статья с достаточно изученной системой QS-регуляции S. proteamaculans [11]. В качестве модельного объекта нами был выбран штамм S. proteamaculans 94, выделенный из мяса, испортившегося в холодильной камере мясокомбината. Штамм способен гидролизовать коллаген при низкой температуре и является продуцентом нескольких внеклеточных протеиназ [13].
В настоящей работе мы показали, что синтез АГЛ у S. proteamaculans 94 не зависит от белка SprR. Организация генов SprI/SprR системы была конвергентной [14]. Ранее было показано, что у бактерий с конвергентной организацией QS генов экспрессия гена синтазы luxI -типа, как правило, не зависит от R-белка [8–10, 12]. Это отличается от основной концепции QS-регуляции, где необходима активация АГЛ белка LuxR для экспрессии гена синтазы АГЛ (в случае классической QS системы Vibrio fischeri). Анализ нуклеотидной последовательности промоторной области перед геном sprI не выявил регуляторного элемента типа lux-бокс [14], с которым в системе V. fischeri связывается комплекс белка LuxR с АГЛ, что приводит к дальнейшей активации транскрипции lux-оперона. Таким образом, приведенные выше данные показывают, что аутоиндукция синтеза АГЛ, наблюдаемая у V. fischeri, не характерна для исследуемого штамма S. proteamaculans 94. Как мы показали в предыдущей работе, в промоторном районе гена sprR присутствует гомолог lux-бокса, spr-бокс, который перекрывается с –10 сайтом в этом промоторе. Поэтому связывание белка SprR с spr-боксом может ингибировать транскрипцию sprR гена, мешая связыванию РНК-полимеразы. Такая закономерность была показана для регуляции транскрипции некоторых белков R-типа у Serratia и Pantoea [14]. Полученные данные показывают сходство регуляторной SprI/SprR QS системы S. proteamaculans 94 с QS системами других бактерий семейства Enterobacteriaceae.
В настоящей работе была изучена роль QS систем в регуляции клеточных процессов, относящихся к различным сторонам метаболизма S. proteamaculans 94. Энтеробактерии рода Serratia являются продуцентами большого количества внеклеточных ферментов, используя их для утилизации высокомолекулярных соединений окружающей среды и в качестве факторов вирулентности при инвазии и колонизации других организмов. Мы исследовали влияние мутаций в генах QS системы S. proteamaculans 94 на синтез протеаз, липаз, хитиназ и гемолизина. В предыдущей работе было показано, что инактивация гена sprI приводила к снижению экзопротеазной, хитинолитической активностей и не вызывала изменения липазной и гемолитической активностей [14]. Мутация в гене sprR не оказывала заметного влияния на изученные ферментативные активности.
Представляло интерес исследовать влияние мутаций в QS генах sprI и sprR на регуляцию образования биопленок у S. proteamaculans 94 – процесс, который у многих бактерий зависит от QS-регуляции [12, 27, 28]. Было показано, что мутация в гене sprR не оказывала влияния на уровень образования биопленок. В мутантном по гену sprI штамме образование биопленок снижалось вдвое по сравнению с уровнем биопленок в исходном штамме. При этом добавление экзогенного 3-оксо-С6-АГЛ (доминирующий тип АГЛ у S. proteamaculans 94) восстанавливало способность к формированию биопленки практически до уровня штамма дикого типа. Полученные результаты показывают, что образование биопленок у S. proteamaculans 94 регулируется позитивно 3-оксо-С6-ГЛ-зависимой QS системой. Эти данные согласуются с результатами аналогичных исследований S. liquefaciens и S. marcescens [28, 30]. По данным этих авторов, у АГЛ-дефицитных мутантов способность формировать биопленки снижалась по сравнению со штаммом дикого типа.
Интересно, что у другого исследованного нами вида Serratia – S. plymuthica (штамм HRO-C48) инактивация гена синтазы АГЛ splI вызывала увеличение уровня образованных биопленок в 2–2.5 раза, а введение в клетки этого мутанта плазмиды с клонированными генами splI/splR снижало формирование биопленок до нормального уровня. В этом случае формирование биопленок у S. plymuthica регулировалось негативно SplIR QS системой. У другого штамма этого вида, S. plymuthica IC1270, QS система SplIR также оказывала негативное влияние на образование биопленок. Введение в клетки S. plymuthica IC1270 рекомбинантной плазмиды, несущей клонированные гены splI/splR из S. plymuthica HRO-C48, приводило к снижению вдвое формирования биопленок [31]. Эти данные показывают, что QS-регуляция формирования биопленок у рода Serratia может быть видоспецифичной.
Успешное формирование биопленок связано со способностью бактерий к адгезии. Мы показали, что мутация в гене sprI приводила к изменению характера роста клеток S. proteamaculans 94 в жидкой среде и резко снижала способность клеток адсорбироваться на поверхности стекла. Это могло быть вызвано изменением структуры клеточных стенок бактерий. В связи с этим мы исследовали состав жирных кислот, многие из которых входят в состав клеточных мембран бактерий, у S. proteamaculans 94 и мутантных штаммов [[14] и настоящая работа].
Сравнительный анализ показал значительные изменения состава жирных кислот мутантных штаммов по сравнению со штаммом дикого типа: содержание некоторых жирных кислот изменялось более чем на порядок, существенно изменилось соотношение насыщенных и ненасыщенных жирных кислот. По сравнению со штаммом S. proteamaculans 94 дикого типа жирнокислотный состав мутантов с нокаутом генов sprI [14] и sprR характеризовался меньшей ненасыщенностью, что свидетельствует о более плотной упаковке ЛПС на клеточной поверхности. Возможно, это один из механизмов адаптации клеток к неблагоприятным условиям окружающей среды, позволяющий повысить толерантность мембран к стрессовым факторам. Известно, что соотношение насыщенных и ненасыщенных жирных кислот в составе ЛПС влияет на физические свойства мембран бактерий [32, 33]. Фазовое состояние жидкокристаллической структуры липидного слоя мембраны, степень ее текучести, в свою очередь, оказывают весьма существенное влияние на вязко-эластические свойства клеток, их способность к деформации, на активность мембранно-связанных ферментов, на проницаемость мембран.
Чтобы получить общую оценку количества белков, синтез которых зависел от QS-регуляции, был проведен сравнительный протеомный анализ клеток исходного штамма и мутантов. Полученные результаты демонстрируют, что экспрессия более 30 белков находится под влиянием SprIR QS-системы. Ранее было показано, что экспрессия как минимум 39 белков у S. proteamaculans B5a находилась под контролем 3-оксо-С6-ГЛ-зависимой QS системы [11]. Таким образом, наши данные согласуются с результатами аналогичных исследований других авторов.
В рамках настоящей работы впервые была проведена идентификация некоторых белков, для которых наблюдались отличия в уровне экспрессии в штамме дикого типа и мутантах. В обоих мутантах обнаружены изменения в синтезе существенно важных для метаболизма бактерий белков. Например, мы показали значительное снижение экспрессии ФМН-редуктазы, являющейся одним из компонентов дыхательной цепи бактерий, у обоих мутантов – почти в 3 раза для sprI::Gm мутанта и более чем в 10 раз для мутанта sprR::Gm. У мутантного штамма sprI::Gm наблюдалась усиленная экспрессия белков, участвующих в защите клеток от стрессов: тиоредоксина 2, тиоредоксин-зависимой тиолпероксидазы, белка теплового шока GrpE.
У мутантного штамма sprR::Gm отмечено снижение уровня экспрессии двух субъединиц белка сукцинил-CoA-синтетазы: более чем в 2 раза для α-субъединицы и в 5 раз для β-субъединицы. Сукцинил-СoA-синтетаза – один из ключевых ферментов цикла лимонной кислоты, катализирующий реакцию образования свободного сукцината. При этой реакции происходит образование богатого энергией АТФ (ГТФ) за счет АДФ и минерального фосфата с использованием сукцинил-СоА. Изменения, вызванные мутацией в гене sprR, могли привести к дефициту АТФ и общего уровня энергии в клетке.
Кроме того, было обнаружено, что у мутанта sprR::Gm значительно усиливалась экспрессия β‑лактамазы – более чем в 30 раз по сравнению с ее экспрессией в исходном штамме. Эти ферменты отвечают за устойчивость бактерий к β-лактамным антибиотикам, включая карбапенемы. Синтез карбапенемов обнаружен у бактерий рода Serratia [12]. Хотя мы не имеем данных о синтезе карбапенема у S. proteamaculans 94, можно предположить, что этот антибиотик также продуцируется штаммом 94. В этом случае репрессия синтеза β-лактамазы в штамме дикого типа полезна для бактерии, так как β-лактамаза разлагает карбапенем, который может играть существенную роль в конкуренции с другими бактериями.
Полученные нами данные протеомного анализа показывают, что идентифицированные белки, экспрессия которых изменяется в результате мутаций QS-генов, являются компонентами важных клеточных процессов.
Работа частично финансировалась грантами РФФИ № 18-04-00375, 19-04-00756 и грантом Минобрнауки России, государственное задание ЯрГУ, работа № 0856-2020-0008.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Fuqua C., Winans S.C., Greenberg E.P. Census and consensus in bacterial ecosystems: the LuxR_LuxI family of quorum_sensing transcriptional regulators // Annu. Rev. Microbiol. 1996. V. 50. P. 727–751. https://doi.org/10.1146/annurev.micro.50.1.727
Waters C.M., Bassler B.L. Quorum sensing: cell-to-cell communication in bacteria // Annu. Rev. Cell. Dev. Biol. 2005. V. 21. P. 319–346. https://doi.org/10.1146/annurev.cellbio.21.012704.131001
Хмель И.А., Метлицкая А.З. Quorum Sensing регуляция экспрессии генов – перспективная мишень для создания лекарств против патогенности бактерий // Мол. биология. 2006. Т. 40. С. 195–210.
Веселова М.А. Quorum Sensing регуляция у псевдомонад // Генетика. 2010. Т. 46. № 2. С. 149–158.
Зайцева Ю.В., Попова А.А., Хмель И.А. Регуляция типа Quorum Sensing у бактерий семейства Enterobacteriaceae // Генетика. 2014. Т. 50. № 14. С. 373–391.
Papenford K., Bassler B. Quorum-sensing signal-response systems in gram-negative Bacteria // Nat. Rev. Microbiol. 2016. V. 14. P. 576–588. https://doi.org/10.1038/nrmicro.2016.89
Whiteley M., Diggle S.P., Greenberg E.P. Bacterial quorum sensing: The progress and promise of an emerging research area // Nature. 2017. V. 551(7680). P. 313–320.
Beck von Bodman S., Majerczak D.R., Coplin D.L. A negative regulator mediates quorum-sensing control of exopolysaccharide production in Pantoea stewartii subsp. Stewartii // Proc. Acad. Sci. USA. 1998. V. 95. P. 7687–7692. https://doi.org/10.1073/pnas.95.13.7687
Horng Y.T., Deng S.C., Daykin M. et al. The LuxR family protein SpnR functions as a negative regulator of N‑acylhomoserine lactone-dependent quorum sensing in Serratia marcescens // Mol. Microbiol. 2002. V. 45. P. 1655–1671. https://doi.org/10.1046/j.1365-2958.2002.03117.x
Minogue T.D., Wehland-von Trebra M., Bernhard F., von Bodman S.B. The autoregulatory role of EsaR, a quorum-sensing regulator in Pantoea stewartii ssp. stewartii: evidence for a repressor function // Mol. Microbiol. 2002. V. 44. P. 1625–1635. https://doi.org/10.1046/j.1365-2958.2002.02987.x
Christensen A.B., Riedel K., Eberl L. et al. Quorum-sensing-directed protein expression in Serratia proteamaculans B5a // Microbiology. 2003. V. 149. P. 471–483. https://doi.org/10.1099/mic.0.25575-0
Van Houdt R., Givskov M., Michiels C.W. Quorum sensing in Serratia // FEMS Microbiol. Rev. 2007. V. 31. P. 407–424. https://doi.org/10.1111/j.1574-6976.2007.00071.x
Demidyuk I.V., Kalashnikov A.E., Gromova T.Y. et al. Cloning, sequencing, expression, and characterization of protealysin, a novel neutral proteinase from Serratia proteamaculans representing a new group of thermolysin-like proteases with short N-terminal region of precursor // Protein Expression and Purification. 2006. V. 47. P. 551–561. https://doi.org/10.1016/j.pep.2005.12.005
Zaitseva Y.V., Koksharova O.A., Lipasova V.A. et al. SprI/SprR Quorum Sensing System of Serratia proteamaculans 94 // BioMed. Res. Internat. Volume 2019, Article ID 3865780, 10 p. https://doi.org/10.1155/2019/3865780
Zaitseva Y.V., Lipasova V.A., Plyuta V.A. et al. Effect of inactivation of luxS gene on the properties of Serratia proteamaculans 94 strain // Folia Microbiol. (Praha). 2019. V. 64. P. 265–272. https://doi.org/10.1007/s12223-018-0657-5
Миллер Дж. Эксперименты в молекулярной генетике. М.: Мир, 1976. 436 с.
McClean K.H., Winson M.K., Fish L. et al. Quorum sensing in Chromobacterium violaceum: exploitation of violacein production and inhibition for the detection of N-acylhomoserine lactones // Microbiology. 1997. V. 143. P. 3703–3711. https://doi.org/10.1099/00221287-143-12-3703
Shaw P.D., Ping G., Daly S.L. et al. Detecting and characterizing N-acyl-homoserine lactone signal molecules by thin-layer chromatography // Proc. Natl Acad. Sci. USA. 1997. V. 94. P. 6036–6041. https://doi.org/10.1073/pnas.94.12.6036
Ausubel F.M., Brendt R., Kingston R.E. (Eds) Current Protocols in Molecular Biology. N.Y.: John Wiley & Sons Inc., 1994.
Hoang T.T., Karkhoff-Schweizer R.R., Kutchma A.J., Schweizer H.P. A broad-host-range Flp-FRT recombination system for site-specific excision of chromosomally-located DNA sequences: application for isolation of unmarked Pseudomonas aeruginosa mutants // Gene. 1998. V. 212. P. 77–86. https://doi.org/10.1016/s0378-1119(98)00130-9
Dennis J.J., Zylstra G.J. Plasposons: modular self-cloning minitransposon derivatives for rapid genetic analysis of gram-negative bacterial genomes // Appl. Environ. Microbiol. 1998. V. 64. P. 2710–2715. 0099-2240/98/$04.0010
Popova A.A., Koksharova O.A., Lipasova V.A. et al. Inhibitory and toxic effects of volatiles emitted by strains of Pseudomonas and Serratia on growth and survival of selected microorganisms, Caenorhabditis elegans and Drosophila melanogaster // BioMed Res. Int. 2014. V. 2014. Article ID 125704, 11 p. https://doi.org/10.1155/2014/125704
Microbial ID Inc. Microbial identification system operational manual, version 4. Microbial ID Inc., Newark, Del, 1992.
Stead D.E., Sellwood J.E., Wilson J., Viney I. Evaluation of a commercial microbial identification system based on fatty acid profiles for rapid, accurate identification of plant pathogenic bacteria // J. Appl. Bacteriol. 1992. V. 72. P. 315–321.
Yan J.X., Wait R., Berkelman T. et al. A modified silver staining protocol for visualization of proteins compatible with matrix-assisted laser desorption/ionization and electrospray ionization-mass spectrometry // Electrophoresis. 2000. V. 21. P. 3666–3672. https://doi.org/10.1002/1522-2683(200011)21:17<3666:: AID-ELPS3666>3.0.CO;2-6
Shevchenko A., Chernushevich I., Wilm M., Mann M. De novo peptide sequencing by nanoelectrospray tandem mass spectrometry using triple quadrupole and quadrupole/time-of-flight instruments // Methods Mol. Biol. 2000. V. 146. P. 1–16. https://doi.org/10.1385/1-59259-045-4:1
Davies D.G., Parsek M.R., Pearson J.P. et al. The involvement of cell-to-cell signals in the development of a bacterial biofilm // Science. 1998. V. 280. P. 295–298.
Labbate M., Queck S.Y., Koh K.S. et al. Quorum sensing-controlled biofilm development in Serratia liquefaciens MG1 // J. Bacteriol. 2004. V.186. P. 692–698. https://doi.org/10.1128/jb.186.3.692-698.2004
Ercolini D., Russo F., Nasi A. et al. Mesophilic and psychrotrophic bacteria from meat and their spoilage potential in vitro and in beef // Appl. Environ. Microbiol. 2009. V. 75. P. 1990–2001. https://doi.org/10.1128/AEM.02762-08
Rice S.A., Koh K.S., Queck S.Y. et al. Biofilm formation and sloughing in Serratia marcescens are controlled by quorum sensing and nutrient cues // J. Bacteriol. 2005. V. 187. P. 3477–3485. https://doi.org/10.1128/JB.187.10.3477-3485.2005
Зайцева Ю.В., Волошина П.В., Лиу Х. и др. Участие глобальных регуляторов GrrS, RpoS и SplIRe в формировании биопленок у Serratia plymuthica // Генетика. 2010. Т. 46. № 5. С. 616–621.
Sinensky M. Homeoviscous adaptation – a homeostatic process that regulates the viscosity of membrane lipids in Escherichia coli // Proc. Natl Acad. Sci. USA. 1974. V. 71. P. 522–525. https://doi.org/10.1073/pnas.71.2.522
Cybulski L.E., Albanesi D., Mansilla M.C. et al. Mechanism of membrane fluidity optimization: isothermal control of the Bacillus subtilis acyl-lipid desaturase // Mol. Microbiol. 2002. V. 45. P. 1379–1388. https://doi.org/10.1046/j.1365-2958.2002.03103.x
Дополнительные материалы отсутствуют.