Генетика, 2021, T. 57, № 5, стр. 536-543
Применение SRAP-маркеров для ДНК-идентификации российских сортов люцерны
А. О. Шамустакимова 1, Ю. М. Мавлютов 1, *, И. А. Клименко 1
1 Федеральный научный центр кормопроизводства и агроэкологии им. В.Р. Вильямса ФНЦ “ВИК им. В.Р. Вильямса”
141055 Лобня, Московская обл., Россия
* E-mail: yulian92@mail.ru
Поступила в редакцию 02.06.2020
После доработки 28.07.2020
Принята к публикации 25.08.2020
Аннотация
Проведена оценка 17 сортов и одного сортообразца люцерны с использованием маркерной системы SRAP (sequence-related amplified polymorphism) с целью определения ее эффективности для сортовой идентификации. Из 25 протестированных комбинаций SRAP-маркеров семь оказались информативными для выявления ДНК-полиморфизма в исследуемых сортах. Эти комбинации генерировали 129 ПЦР-продуктов с уровнем полиморфизма от 21 до 50%. Для 14 сортов удалось выявить специфические продукты амплификации с целью последующей разработки генетических паспортов.
Люцерна является важнейшей сельскохозяйственной культурой, которая возделывается в качестве источника высококачественного белкового корма для всех видов скота и птицы [1]. Род люцерны насчитывает более 100 видов, из которых наиболее значимыми на территории Российской Федерации являются четыре: люцерна изменчивая (Medicago varia Mart.), люцерна посевная (M. sativa L.), люцерна желтая (M. falcata L.) и люцерна хмелевидная (М. lupulina L.). На их основе создана система сортов, обладающих высокой урожайностью и устойчивостью к болезням и вредителям [1, 2]. С целью успешного решения практических задач, связанных с использованием новых сортов в кормопроизводстве, для развития семеноводства, а также для обеспечения правовой защиты селекционных достижений необходима надeжная система идентификации [3, 4].
Современные подходы анализа генетического разнообразия, основанные на полимеразной цепной реакции, позволяют идентифицировать виды и сорта различных культур, в частности с целью последующей паспортизации [5]. При этом одной из главных проблем является адекватный подбор маркерной системы, соответствующей основным требованиям и задачам исследования. Среди важнейших характеристик маркерной системы, необходимых при анализе генетического разнообразия, можно выделить уровень полиморфизма, принцип наследования маркера, а также воспроизводимость полученных результатов [6]. Особый интерес в настоящий момент представляет маркерная система SRAP (Sequence-related amplified polymorphism), основанная на амплификации открытых рамок считывания (Open reading Frame, OR), или интрон-экзонных областей генома. Длина SRAP-праймеров, используемых для этого метода, составляет 17–18 нуклеотидов. Коровая последовательность содержит 13–14 оснований, включающих неспецифический участок из 10–11 нуклеотидов на 5'-конце, а также GC-богатый регион в прямом праймере и AT-богатый – в обратном. Вариабельность продуктов ПЦР достигается за счет использования обратного праймера, нацеленного на некодирующую область генома, обладающую низкой консервативностью [7]. SRAP-маркерная система на данный момент успешно используется в ряде исследований: для изучения генетического разнообразия и описания зародышевой плазмы [8], разработки генетических карт [9], идентификации генов, отвечающих за целевой признак [10], контроля гибридизации [11] и сравнительного геномного анализа [12].
Цель исследования – ДНК-идентификация российских сортов люцерны с использованием SRAP-маркеров для последующей паспортизации.
МАТЕРИАЛЫ И МЕТОДЫ
Растительный материал и ДНК-экстракция. В исследовании использовали 17 сортов и один перспективный сортообразец люцерны российской селекции (табл. 1), принадлежащих к разным видам: Medicago varia Mart. (N = 15), Medicago sativa L. (N = 2), Medicago lupulina L. (N = 1). Экспериментальная работа по генотипированию сортов проводилась на семидневных проростках (по 30 шт. на образец), выращенных в чашках Петри. Тотальную геномную ДНК выделяли с помощью модифицированного SDS метода [13] и разбавляли до концентрации 20 нг/мкл.
Таблица 1.
Перечень исследуемых сортов люцерны
| № | Аббревиатура | Сорт | Вид | Оригинатор |
|---|---|---|---|---|
| 1 | ЛГВ | Луговая 67 | Medicagovaria Mart. | ФНЦ ВИК им. В.Р. Вильямса |
| 2 | ВГА | Вега 87 | То же | |
| 3 | ЛДА | Лада | » | |
| 4 | АГН | Агния | ФНЦ ВИК им. В.Р. Вильямса ООО “НТЦ Травы Сибири” |
|
| 5 | КМЛ | Камелия | Пензенский НИИСХ | |
| 6 | ВРЖ | Воронежская 6 | ФНЦ ВИК им. В.Р. Вильямса | |
| 7 | ВЛД | Влада | НИИСХ Юго-Востока | |
| 8 | СЛН | Селена | ФНЦ ВИК им. В.Р. Вильямса | |
| 9 | ТСА | Таисия | То же | |
| 10 | ВЕЛ | Вела | » | |
| 11 | СРГ | Сарга | УрФАНИЦ УрО РАН | |
| 12 | ПСА | Пастбищная 88 | ФНЦ ВИК им. В.Р. Вильямса | |
| 13 | A2 | А2 | То же | |
| 14 | НКА | Находка | » | |
| 15 | БЛД | Благодать | » | |
| 16 | ТМБ | Тамбовчанка | Medicagosativa L. | ОНО “Моршанская селекционная станция” |
| 17 | УЗН | Узень | НИИСХ Юго-Востока | |
| 18 | МРА | Мира | Medicago lupulina L. | ФНЦ ВИК им. В.Р. Вильямса |
SRAP-анализ. В исследовании использовали пять прямых и пять обратных SRAP-праймеров, образующих в общей сложности 25 комбинаций (табл. 2).
Таблица 2.
Нуклеотидные последовательности SRAP-праймеров, использованных для выявления ДНК-полиморфизма в сортах люцерны
| Прямой праймер (F) | Последовательность праймера (5' → 3') |
Обратный праймер (R) | Последовательность праймера (5' → 3') |
|---|---|---|---|
| F9 | GTAGCACAAGCCGGACC | R9 | GACTGCGTACGAATTTCA |
| F13 | CGAATCTTAGCCGGCAC | R7 | GACTGCGTACGAATTGAG |
| Me4 | CGAATCTTAGCCGGAAT | Em2 | GACTGCGTACGAATTCGG |
| F10 | GTAGCACAAGCCGGAAG | R14 | CGCACGTCCGTAATTAAC |
| F11 | CGAATCTTAGCCGGATA | R8 | GACACCGTACGAATTGAC |
Реакционная смесь объемом 25 мкл содержала 10× Taq Turbo buffer, 50× dNTP mix, 5 U Taq-ДНК полимеразы, а также по 0.2 мкМ каждого праймера и 20 нг ДНК. Амплификация осуществлялась на приборе “Bio-Rad iCycler, USA” в соответствии с условиями, предложенными Li и Quiros [7] с внесением некоторых модификаций. Детекция результатов ПЦР проводилась путeм электрофореза в 1.6%-ном агарозном геле. Для оценки размера фрагментов продуктов амплификации использовался молекулярный маркер 1kb DNA Ladder (“Евроген”, Россия). Визуализация фрагментов амплификации проводилась на приборе “Gel Doc™ XR+” (Bio-Rad, USA).
Анализ данных. Размер продуктов амплификации определяли с использованием программы Image Lab. Для каждой праймерной пары были составлены бинарные матрицы. Показатели генетического разнообразия рассчитывали с помощью программы PopGene. Определяли: эффективное число аллелей на локус, индекс Шеннона, индекс генетического разнообразия, индекс генетического сходства и дистанции Нея. На основе данных генетических дистанций Нея составлена дендрограмма методом невзвешенной попарной группировки с усреднением UPGMA.
РЕЗУЛЬТАТЫ
SRAP-анализ. В нашем исследовании семь из 25 протестированных комбинаций SRAP-праймеров генерировали чeткие и воспроизводимые продукты амплификации на большинстве тестируемых сортов люцерны.
Показатели генетического разнообразия сортового материала, полученные с помощью семи информативных праймерных пар, представлены в табл. 3. В общей сложности с использованием SRAP-маркеров удалось выявить 129 воспроизводимых ПЦР-продуктов длиной от 50 до 708 пн, из которых 41 оказался полиморфным. Количество полученных ПЦР-фрагментов на одну комбинацию SRAP-маркеров варьировало от девяти до 27 при среднем показателе 18.4 ампликона на праймерную пару. Число полиморфных бэндов составляло от 3 до 9, в среднем 5.9 – на комбинацию праймеров. Наибольшее количество полиморфных продуктов (9) было получено с праймерами F13-R7. Уровень полиморфизма при использовании данной комбинации составил 50%. Эффективное число аллелей (Ne) находилось в диапазоне от 1.2637 до 1.4367. Коэффициент генетического разнообразия по Нею составил от 0.1941 (F13-R7) до 0.2716 (F13-R9) со средним значением 0.2503. Индекс разнообразия Шэннона варьировал от 0.3357 до 0.4305 со средней 0.4038.
Таблица 3.
Показатели изменчивости при анализе сортов люцерны с использованием SRAP-маркеров
| Праймерая пара | Последовательности праймеров (5'–3') | Размер ПЦР-продукта (пн) | Общее количество ПЦР-продуктов | Количество полиморфных продуктов | Процент полиморфных продуктов | Эффективное число аллелей (Ne) | Индекс генетического разнообразия по Нею (He) | Индекс Шеннона (I) |
|---|---|---|---|---|---|---|---|---|
| F9-R9 | GTAGCACAAGCCGGACC GACTGCGTACGAATTTCA |
105–490 | 25 | 7 | 28 | 1.3498 | 0.2368 | 0.3882 |
| F9-R8 | GTAGCACAAGCCGGACC GACACCGTACGAATTGAC |
125–385 | 14 | 3 | 21 | 1.4102 | 0.2654 | 0.4235 |
| F13-Em2 | CGAATCTTAGCCGGCAC GACTGCGTACGAATTCGG |
125–235 | 9 | 3 | 33 | 1.3925 | 0.2524 | 0.4050 |
| F10-R7 | GTAGCACAAGCCGGAAG GACTGCGTACGAATTGAG |
165–708 | 16 | 6 | 38 | 1.4367 | 0.2685 | 0.4220 |
| F10-R8 | GTAGCACAAGCCGGAAG GACACCGTACGAATTGAC |
145–708 | 27 | 8 | 30 | 1.3934 | 0.2634 | 0.4223 |
| F13-R9 | CGAATCTTAGCCGGCAC GACTGCGTACGAATTTCA |
60–485 | 20 | 5 | 25 | 1.4230 | 0.2716 | 0.4305 |
| F13-R7 | CGAATCTTAGCCGGCAC GACTGCGTACGAATTGAG |
50–305 | 18 | 9 | 50 | 1.2637 | 0.1941 | 0.3357 |
| Среднее | 18.4 | 5.9 | 32 | 1.3813 | 0.2503 | 0.4038 |
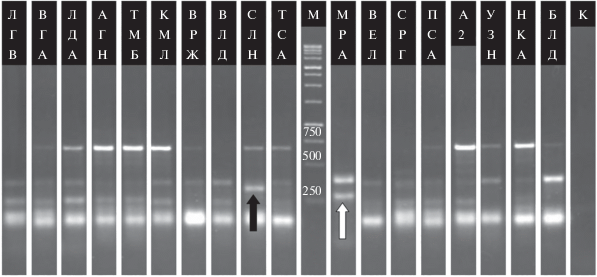
На рис. 1 представлена электрофореграмма ПЦР-продуктов, полученных с использованием пары праймеров F10-R7, где черной стрелкой обозначен специфический ампликон для сорта Селена (316 пн), белой стрелкой – для сорта Мира (275 пн).
Рис. 1.
Электрофореграмма продуктов амплификации ДНК сортов люцерны с комбинацией SRAP-праймеров F10-R7. Расшифровка аббревиатур дана в табл. 1.
На базе бинарных матриц подсчитали индексы генетического сходства и дистанции Нея для выявления филогенетических отношений между сортами (табл. 4). Наименьший коэффициент генетического сходства (0.6667) был обнаружен между сортами Сарга и Лада, а также Пастбищная и Тамбовчанка. Наибольший (0.8837) – между образцами Камелия и Воронежская 9. Наименьшие генетические дистанции (0.1236) наблюдались между сортами Камелия и Воронежская.
Таблица 4.
Индексы генетического сходства (над диагональю) и дистанции Нея (под диагональю)
| ЛГВ | ВГА | ЛДА | АГН | ТМБ | КМЛ | ВРЖ | ВЛД | СЛН | ТСА | МРА | ВЕЛ | СРГ | ПСА | А2 | УЗН | НКА | БЛД | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ЛГВ | 0.7442 | 0.6744 | 0.7287 | 0.6744 | 0.7442 | 0.7364 | 0.7054 | 0.6899 | 0.7054 | 0.7132 | 0.7054 | 0.7287 | 0.7132 | 0.6977 | 0.6977 | 0.6744 | 0.6977 | |
| ВГА | 0.2955 | 0.7287 | 0.7674 | 0.6977 | 0.7209 | 0.7442 | 0.7442 | 0.7132 | 0.7132 | 0.7364 | 0.7287 | 0.6899 | 0.7209 | 0.7674 | 0.6744 | 0.6822 | 0.6589 | |
| ЛДА | 0.3939 | 0.3165 | 0.8217 | 0.8140 | 0.7597 | 0.7829 | 0.7674 | 0.7829 | 0.7519 | 0.7287 | 0.7209 | 0.6667 | 0.6822 | 0.7132 | 0.6822 | 0.6589 | 0.6357 | |
| АГН | 0.3165 | 0.2647 | 0.1964 | 0.7442 | 0.7519 | 0.7752 | 0.7442 | 0.7597 | 0.7287 | 0.7364 | 0.7287 | 0.7364 | 0.7054 | 0.7364 | 0.7054 | 0.6822 | 0.6744 | |
| ТМБ | 0.3939 | 0.3600 | 0.2059 | 0.2955 | 0.7752 | 0.7674 | 0.7054 | 0.7364 | 0.7364 | 0.7287 | 0.7364 | 0.6822 | 0.6667 | 0.7132 | 0.6977 | 0.6589 | 0.6357 | |
| КМЛ | 0.2955 | 0.3272 | 0.2748 | 0.2851 | 0.2546 | 0.8837 | 0.7907 | 0.7752 | 0.7287 | 0.7364 | 0.6822 | 0.7054 | 0.7054 | 0.7364 | 0.7364 | 0.7132 | 0.7364 | |
| ВРЖ | 0.3059 | 0.2955 | 0.2447 | 0.2546 | 0.2647 | 0.1236 | 0.8140 | 0.7984 | 0.7829 | 0.7442 | 0.7209 | 0.7132 | 0.7442 | 0.7287 | 0.7132 | 0.7054 | 0.7132 | |
| ВЛД | 0.3490 | 0.2955 | 0.2647 | 0.2955 | 0.3490 | 0.2348 | 0.2059 | 0.8295 | 0.8760 | 0.7442 | 0.7829 | 0.7597 | 0.7597 | 0.7907 | 0.6822 | 0.7054 | 0.6977 | |
| СЛН | 0.3712 | 0.3380 | 0.2447 | 0.2748 | 0.3059 | 0.2546 | 0.2251 | 0.1870 | 0.8140 | 0.7597 | 0.7674 | 0.7597 | 0.7597 | 0.7597 | 0.6822 | 0.7209 | 0.6822 | |
| ТСА | 0.3490 | 0.3380 | 0.2851 | 0.3165 | 0.3059 | 0.3165 | 0.2447 | 0.1324 | 0.2059 | 0.7752 | 0.8140 | 0.7907 | 0.7597 | 0.7752 | 0.7132 | 0.7054 | 0.7287 | |
| МРА | 0.3380 | 0.3059 | 0.3165 | 0.3059 | 0.3165 | 0.3059 | 0.2955 | 0.2955 | 0.2748 | 0.2546 | 0.7442 | 0.7364 | 0.7209 | 0.7364 | 0.7364 | 0.7287 | 0.7209 | |
| ВЕЛ | 0.3490 | 0.3165 | 0.3272 | 0.3165 | 0.3059 | 0.3825 | 0.3272 | 0.2447 | 0.2647 | 0.2059 | 0.2955 | 0.8372 | 0.7287 | 0.8217 | 0.6977 | 0.7054 | 0.7132 | |
| СРГ | 0.3165 | 0.3712 | 0.4055 | 0.3059 | 0.3825 | 0.3490 | 0.3380 | 0.2748 | 0.2748 | 0.2348 | 0.3059 | 0.1777 | 0.7829 | 0.7984 | 0.7209 | 0.7287 | 0.7209 | |
| ПСА | 0.3380 | 0.3272 | 0.3825 | 0.3490 | 0.4055 | 0.3490 | 0.2955 | 0.2748 | 0.2748 | 0.2748 | 0.3272 | 0.3165 | 0.2447 | 0.8450 | 0.7209 | 0.7287 | 0.7364 | |
| А2 | 0.3600 | 0.2647 | 0.3380 | 0.3059 | 0.3380 | 0.3059 | 0.3165 | 0.2348 | 0.2748 | 0.2546 | 0.3059 | 0.1964 | 0.2251 | 0.1685 | 0.7364 | 0.7597 | 0.7364 | |
| УЗН | 0.3600 | 0.3939 | 0.3825 | 0.3490 | 0.3600 | 0.3059 | 0.3380 | 0.3825 | 0.3825 | 0.3380 | 0.3059 | 0.3600 | 0.3272 | 0.3272 | 0.3059 | 0.8372 | 0.7519 | |
| НКА | 0.3939 | 0.3825 | 0.4172 | 0.3825 | 0.4172 | 0.3380 | 0.3490 | 0.3490 | 0.3272 | 0.3490 | 0.3165 | 0.3490 | 0.3165 | 0.3165 | 0.2748 | 0.1777 | 0.8372 | |
| БЛД | 0.3600 | 0.4172 | 0.4531 | 0.3939 | 0.4531 | 0.3059 | 0.3380 | 0.3600 | 0.3825 | 0.3165 | 0.3272 | 0.3380 | 0.3272 | 0.3059 | 0.3059 | 0.2851 | 0.1777 |
Дендрограмма, полученная по методу UPGMA (рис. 2), позволила выявить пять кластеров и подгрупп. В первую группу вошли сорта Луговая 67 и Вега 87. Вторая группа представлена тремя подгруппами, а именно: 2a – Лада, Агния и Тамбовчанка; 2б – Камелия, Воронежская, 2в – Влада, Таисия и Селена. В третью группу вошел сорт люцерны хмелевидной Мира. Четвертая группа включает такие сорта как Вела и Сарга, выделившиеся в подгруппу 4a, Пастбищная 88 и А2 – подгруппа 4б. Пятая группа представлена тремя сортами – Узень, Находка, Благодать. Проведенный анализ позволил выявить специфические продукты амплификации для 14 из 18 исследуемых сортов (рис. 3).
Рис. 2.
UPGMA-дендрограмма, основанная на значениях генетических дистанций Нея для исследуемых сортов люцерны.
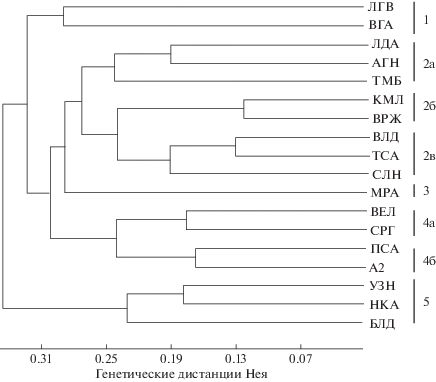
Рис. 3.
Информативные комбинации SRAP-праймеров, выявляющие специфические ампликоны в сортах люцерны.

Так, наибольшим числом комбинаций SRAP-праймеров (N = 5) удалось идентифицировать сорта Мира и Луговая 67. Для сортов Агния, Камелия, Воронежская и сортообразца А2 протестированные комбинации не образовали специфических продуктов. Две пары праймеров (F9-R9 и F10-R8) обнаружили различия в шести исследуемых сортах. Комбинация F13-Em2 позволила идентифицировать два сорта: Луговая 67 и Вега 87. Для некоторых сортов с использованием одной комбинации праймеров одновременно было получено от двух (F13-Em2 и F13-R7 на сорте Луговая 67; F9-R9 на сорте Тамбовчанка; F10-R7 на сорте Благодать; F10-R8 на сорте Лада) до трех специфических продуктов амплификации (F13-R7 на сорте Вега 87).
ОБСУЖДЕНИЕ
В настоящее время исследования с использованием SRAP-маркеров проведены на большом количестве сельскохозяйственных культур [14, 15]. В частности, этот вид ДНК-маркеров применялся для изучения генетического разнообразия зарубежных видов и сортов люцерны [16–20].
По результатам настоящей работы на сортах люцерны российской селекции уровень полиморфизма равнялся 31.7%. При этом его максимальное значение зафиксировано на комбинации праймеров F13-R7 (50%), а минимальное – на F9-R9 (28%), что свидетельствует о различной эффективности праймерных пар на исследуемых сортах. Эти показатели различаются с данными других авторов. В частности, при изучении генетического разнообразия 15 популяций люцерны [16] уровень полиморфизма составил 90%. В исследованиях H.B. Rhouma с соавт. [17] анализировались сорта и естественные популяции из Туниса, Казахстана, Сербии, Австралии и США с помощью SRAP-маркеров. В этом случае процент полиморфизма достигал 100%. При изучении сортов люцерны из Саудовской Аравии уровень полиморфизма равнялся 98.3% [18]. В работах 2011 г., проведенных на иранских [19] и китайских [20] сортах люцерны, уровень полиморфизма составил 49 и 40.93% соответственно. Полученные различия по-видимому могут быть связаны с высоким уровнем генетического сходства большинства исследуемых сортов (оригинатор ФНЦ “ВИК им. В.Р. Вильямса”).
Исходя из значения коэффициента генетического сходства, можно сделать вывод о наибольшей степени ДНК-полиморфизма между сортами Сарга и Лада, а также между Пастбищная 88 и Тамбовчанка (0.6667). При этом наибольшее сходство (0.8837) наблюдалось между сортами Камелия и Воронежская 9.
По значениям генетических дистанций Нея наиболее отдаленными оказались сорта Находка (ФНЦ “ВИК им. В.Р. Вильямса”) и Тамбовчанка (ОНО “Моршанская селекционная станция”). Это может быть обусловлено видовой принадлежностью этих сортов и различиями в происхождении. Наименьшая генетическая дистанция выявлена между сортами Камелия и Воронежская 9 (оба сорта отличаются повышенной зимостойкостью). Из 18 анализируемых образцов с использованием маркерной системы SRAP удалось выявить специфические продукты амплификации для 14 сортов. Это свидетельствует о высокой информативности тестируемой маркерной системы. Таким образом, маркерная система SRAP может использоваться для ДНК-идентификации российских сортов люцерны с целью их последующей паспортизации.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Писковацкий Ю.М., Косолапов В.М., Михалев В.Е. и др. Агротехника возделывания сортов люцерны селекции ВНИИ кормов им. В.Р. Вильямса на семенные и кормовые цели. М.: ФГУ РЦСК, 2008. С. 3–15.
Veronesi F., Brummer E.C., Huyghe C. Alfalfa // Fodder Crops and Amenity Grasses. N.Y.: Springer, 2010. P. 395–437.
Чесноков Ю.В., Косолапов В.М. Генетические ресурсы растений и ускорение селекционного процесса. М.: Угрешская типография, 2016. 172 с.
Писковацкий Ю.М., Ломова М.Г., Соложенцева Л.Ф. и др. Создание перспективного материала люцерны с высокой семенной продуктивностью // Научное обеспечение кормопроизводства и его роль в сельском хозяйстве, экономике, экологии и рациональном природопользовании России: Матер. междунар. науч.-практ. конф., посв. памяти акад. А.А. Жученко (Лобня, 19–20 июня 2013 г.). М.: Угрешская типография, 2013. С. 97–103.
Хлесткина Е.К. Молекулярные маркеры в генетических исследованиях и в селекции // Вавил. журн. генет. и селекции. 2013. Т. 17. № 4/2. С. 1044–1054.
Хлесткина Е.К. Молекулярные методы анализа структурно-функциональной организации генов и геномов высших растений // Вавил. журн. генет. и селекции. 2011. Т. 15. № 4. С. 757–767.
Li G., Quiros C.F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica // Theor. Appl. Genet. 2001. V. 103. № 2–3. P. 455–461. https://doi.org/10.1007/s001220100570
Osman G., Suleyman K., Kazim A. Diversity and relationships among Turkish okra germplasm by SRAP and phenotypic marker polymorphism // Biologia (Bratislava). 2007. V. 62. № 1. P. 41–45. https://doi.org/10.2478/s11756-007-0010-y
Lin Z., Zhang X., Nie Y. et al. Construction of a genetic linkage map for cotton based on SRAP // Chin. Sci. Bull. 2003. V. 48. № 19. P. 2064–2068. https://doi.org/10.1360/03wc0193
Luo Y.X., Du D.Z., Fu G. et al. Inheritance of leaf color and sequence-related amplified polymorphic (SRAP) molecular markers linked to the leaf color gene in Brassica juncea // Afr. J. Biotechnol. 2011. V. 10. № 66. P. 14724–14730. https://doi.org/10.5897/AJB11.1096
Hao Q., Liu Z.A., Shu Q.Y. et al. Studies on Paeonia cultivars and hybrids identification based on SRAP analysis // Hereditas. 2008. V. 145. № 1. P. 38–47. https://doi.org/10.1111/j.0018-0661.2008.2013.x
Guo D.L., Luo Z.R. Genetic relationships of some PCNA persimmons (Diospyros kaki Thumb.) from China and Japan revealed by SRAP analysis // Genet. Resour. Crop. Evol. 2006 V. 53. № 8. P. 1597–1603. https://doi.org/10.1007/s10722-005-8717-5
Dellaporta S.L., Wood J., Hicks J.B. A plant DNA minipreparation: version II // Plant Mol. Biol. Rep. 1983. V. 1. № 4. P. 19–21.
Robarts D.W.H., Wolfe A.D. Sequence-related amplified polymorphism (SRAP) markers: A potential resource for studies in plant molecular biology // Appl. In Plant Sci. 2014. V. 2. №. 7. P. 1400017. https://doi.org/10.3732/apps.1400017
Aneja B., Yadav N.R., Chawla V., Yadav R.C. Sequence-related amplified polymorphism (SRAP) molecular marker system and its applications in crop improvement // Mol. Breeding. 2012. V. 30. № 4. P. 1635–1648. https://doi.org/10.1007/s11032-012-9747-2
Vandemark J.G., Ariss J.J., Bauchan G.A. et al. Estimating genetic relationships among historical sources of alfalfa germplasm and selected cultivars with sequence related amplified polymorphisms // Euphytica. 2006. V. 152. № 1. P. 9–16. https://doi.org/10.1007/s10681-006-9167-7
Rhouma H.B., Taski-Ajdukovic K., Zitouna N. et al. Assessment of the genetic variation in alfalfa genotypes using SRAP markers for breeding purposes // Chil. J. Agric. Res. 2017. V. 77. № 4. P. 332–339. https://doi.org/10.4067/S0718-58392017000400332
Al-Faifi S., Migdadi H., Al-Doss A. et al. Morphological and molecular genetic variability analyses of Saudi Lucerne (Medicago sativa L.) // Crop and Pasture Sci. 2013. V. 64. № 2. P. 137–146. https://doi.org/10.1071/CP12271
Talebi M.B., Hajiahmadi Z., Rahimmalek M. Genetic diversity and population structure of four Iranian alfalfa populations revealed by sequence-related amplified polymorphism (SRAP) markers // J. Crop. Sci. Biotech. 2011. V. 14. № 3. P. 173–178. https://doi.org/10.1007/s12892-011-0030-6
Yuan Q., Gao J., Gui Z. et al. Genetic relationships among alfalfa germplasms resistant to common leaf spot and selected Chinese cultivars assessed by sequence-related amplified polymorphism (SRAP) markers // Afr. J. Biotech. 2011. V. 10. № 59. P. 12527–12534. https://doi.org/10.5897/AJB11.901
Дополнительные материалы отсутствуют.


