Генетика, 2022, T. 58, № 1, стр. 52-67
Полиморфизм митохондриальной ДНК и шести генов ядерного генома в популяции амурских эвенков
М. А. Губина 1, *, В. Н. Бабенко 1, В. А. Бацевич 2, Н. А. Лейбова 3, А. П. Забияко 4
1 Федеральный исследовательский центр Институт цитологии и генетики
Сибирского отделения Российской академии наук
630090 Новосибирск, Россия
2 Московский государственный университет им. М.В. Ломоносова, Научно-исследовательский институт
и Музей антропологии
125009 Москва, Россия
3 Институт этнологии и антропологии им. Н.Н. Миклухо-Маклая Российской академии наук
119334 Москва, Россия
4 Амурский государственный университет
675000 Благовещенск, Россия
* E-mail: marina@bionet.nsc.ru
Поступила в редакцию 21.01.2021
После доработки 17.06.2021
Принята к публикации 23.06.2021
- EDN: FSQQDJ
- DOI: 10.31857/S0016675822010039
Аннотация
Проведен анализ по шести генам ядерного генома ACE (I/D, rs1799752), NOS3 (4b/4a, rs61722009), ADRA2B (I/D, rs28365031), MTHFR (С677Т, rs1801133), TCF7L2 (rs7903146) и CSK (rs1378942), а также митохондриальной ДНК в популяции амурских эвенков. Выявлено, что для эвенков наиболее характерны восточно-евразийские гаплогруппы мтДНК с преобладанием двух: C и D (58%), данные гаплогруппы наиболее характерны для популяций Северной Азии и Сибири. Среди западно-евразийских гаплогрупп самая распространенная – Н (15%). Наименьшие статистически значимые различия по мтДНК выявлены между эвенками, тюрками и самодийцами и наибольшие с финно-угорскими популяциями. Наблюдаемое распределение частот локусов по четырем генам в исследованной популяции не отличалось от теоретически ожидаемого согласно закону Харди–Вайнберга, исключение составили TCF7L2 и CSK. Обнаружена статистически значимая ассоциация по гаметическому неравновесию между четырьмя парами генов (ACE и MTHFR, ACE и TCF7L2, NOS3 и CSK, ADRA2B и MTHFR). Проведена оценка распределения сочетаний аллелей по шести локусам методом максимального правдоподобия. Посчитано число ассоциированных аллелей на каждый псевдо-гаплотип. Обнаружено, что число ассоциированных аллелей пропорционально возрастает с уменьшением частоты встречаемости псевдо-гаплотипов (R2 = 0.5, R = 0.7, d.f. = 16, p < 0.001). Полученные нами данные, возможно, характерны не только для исследованной популяции и вероятно могут отражать такие процессы как гаметическое неравновесие (мейотический драйв).
Огромный интерес для исследователей представляют народы Северной, Центральной и Восточной Азии с их этнической историей и широким ареалом расселения. Коренные этносы Сибири всегда были интересным объектом популяционной генетики человека. Эвенки – один из таких этносов, который занимает обширную территорию от левобережья Енисея на западе до Охотского моря на востоке. Южная граница проходит по левобережью Амура и Ангаре. Амурская область располагается на территории, которая с глубокой древности выступала ареалом расселения и перемещения популяций человека. Бассейн среднего течения Амура находится на стыке Северной, Восточной и Северо-Восточной Азии. Амур связывал обитателей Восточной Сибири и Приморья. Равнины к северу от Амура являлись своеобразным перекрестком переселенческих путей и местом культурных контактов древних обитателей территории современного Китая, Кореи и населения Северо-Восточной Азии [1]. В период палеометалла (2–1 тыс. до н. э.) данный регион населяли племена, которые, вероятно, относились к предкам тунгусо-маньчжурской общности. В эпоху раннего средневековья земли Верхнего и Среднего Амура заселяли племена мохэ, вероятно связанные с тунгусо-маньчжурскими и монголоязычными племенами [2]. Язык – эвенкийский, принадлежит к тунгусо-маньчжурской группе алтайской языковой семьи. Среди северных народностей эвенки – самая многочисленная языковая группа. Численность эвенков, проживающих на территории Российской Федерации – 37 843 чел. [3]. В Амурской области ныне проживает около 1300 человек, большая их часть живет в пяти поселках – Бомнак (Зейского района), Ивановское (Селемджинского района), Усть-Уркима, Первомайское и Усть-Нюкжа (Тындинского района) [4].
В антропологическом отношении современные эвенки неоднородны. Для эвенков Прибайкалья, Якутии и Охотского побережья характерны черты байкальского антропологического типа. Среди западных эвенков таежной зоны Средней Сибири широко распространен катангский антропологический тип, в южных группах эвенков Забайкалья и бассейна Среднего Амура – центрально-азиатский [5–7]. Таким образом, на протяжении многих тысячелетий на данной территории происходили процессы миграции и метисации, которые оказали влияние на популяцию эвенков.
В последнее время в связи с изменением образа жизни и диеты у коренных народов Сибири и Дальнего Востока все чаще выявляется предрасположенность к сердечно-сосудистым заболеваниям и сахарному диабету. Данные заболевания относятся к мультифакториальным, и их развитие обусловлено как взаимодействием факторов внешней среды, образа жизни, так и наследственной предрасположенностью. В формировании наследственной предрасположенности участвует большое количество генов, которые могут работать как самостоятельно, так и путем взаимодействия друг с другом и с факторами внешней среды [8, 9]. Многочисленные исследования позволили определить круг генов-кандидатов, вовлеченных в патогенез этих заболеваний [10–15]. Также было выявлено, что сочетания “неблагоприятных” аллелей с различной частотой встречаются среди больных не только мужского и женского пола, но и национальной принадлежности [16–18]. Ранее было высказано предположение, что в процессе paco- и этногенеза частоты аллелей и генотипов приобрели свою специфику у разных народов и это, в свою очередь, могло внести определенный вклад в наследственную компоненту в разных популяциях [19].
Таким образом, в рамках реализации поставленных задач в работе приведены данные по полиморфизму генов ACE (I/D, rs 1799752), NOS3 (4b/4a rs 61722009), ADRA2B (I/D, rs28365031), MTHFR (С677Т, rs1801133), TCF7L2 (rs7903146) и CSK (rs1378942), а также митохондриальной ДНК в популяции амурских эвенков.
МАТЕРИАЛЫ И МЕТОДЫ
Сбор образцов и выделение ДНК
Материалом для исследования является ДНК представителей амурских эвенков, проживающих в населенных пунктах Усть-Нюкжа Тындинского района и Ивановское Селемджинского района Амурской области. Выборка составила 230 человек, из них 147 женщин и 83 мужчины. Выборка представлена шестью возрастными группами (1939–1960 гг., 8.2%; 1961–1970, 10%; 1971–1980, 12.6%; 1981–1990, 12.2%; 1991–2000, 11.3%; 2001–2011, 45.7%). ДНК была выделена из буккального эпителия, собранного у добровольцев (взрослых и детей старше восьми лет). Забор материала проводили после подписания информационного согласия участниками исследования или их законными представителями. Материал был собран в ходе экспедиционных работ под руководством профессора А.П. Забияко и к. б. н. доцента В.А. Бацевич. Этническую принадлежность людей выясняли методом анкетирования и дополнительным перекрестным опросом, с выяснением национальности родителей как минимум до третьего поколения.
ДНК из буккального эпителия выделяли с помощью набора реагентов РеалБест экстракция 100 (Вектор Бест, Новосибирск).
Анализ мтДНК
Проведен анализ нуклеотидной последовательности гипервариабельных сегментов I и II (ГВС I и II) у 98 представителей эвенкийской национальности. Фрагменты ГВС I и II мтДНК соответствуют 15 898–16 402 пн (и гипервариабельный сайт 16519) и 73-400 пн, согласно референсной последовательности [20]. Амплифицировали методом ПЦР с использованием специфических олигонуклеотидных праймеров, подобранных по референсной последовательности мтДНК [20]. После электрофореза ПЦР-продукты переосаждали c использованием ацетата аммония, проводили прямое секвенирование по методу Сенгера с использованием автоматического секвенатора ABI Prism 3500 Genetic Analyzer (PE Applied Biosystems) и BigDay Terminator v3.1 Cycle Sequencing kit. Полиморфные позиции выявляли методом сопоставления с Кембриджской последовательностью [20]. Гаплогруппы определяли с помощью анализа характерных “диагностических” сайтов рестрикции (ПДРФ).
Анализ ядерных генов
Полиморфизм генов ACE (I/D, rs 1799752), NOS3 (4b/4a, rs 61722009), ADRA2B (I/D, rs28365031), MTHFR (С677Т, rs1801133), TCF7L2 (rs7903146) и CSK (rs1378942) тестировали с помощью ПЦР и рестрикционного анализа (табл. 1).
Таблица 1.
Последовательности праймеров и особенности амплификации полиморфных участков генов
| Ген, полиморфизм | Полиморфный сайт | Последовательность праймеров | Темп. отжига | Рестриктаза | Длина |
|---|---|---|---|---|---|
| ACE, rs1799752 |
I/D | 5'-GCCCTGCAGTGTCTGCAGCATGT-3', 5'-GGATGGCTCTCCCCGCCTTGTCTC-3' |
58°С |
– | 319 пн – делеция, 355 пн – инсерция |
| Для инсерции | |||||
| 5'-TGGGACCACAGCGCCCGCCACTAC-3', 5'-ТCGCCAGCCCTCCCATGCCCATAA-3' |
58°С | ||||
| NOS3, rs61722009 |
4a/4b | 5'-AGGCCCTATGGTAGTGCCTTT-3′, 5′-TCTCTTAGTGCTGTGCTCAC-3′ |
65°С | – | 4bb – 420 пн, 4аа – 393 пн, гетерозиготы 420/393 пн |
| ADRA2B, rs28365031 |
I/D | 5'-AGGGTGTTTGTGGGGCATCTCC-3', 5'-CAAGCTGAGGCCGGAGACACTG-3' |
68°С | 103/112 пн | |
| MTHR, rs1801133 |
C677T | 5'-TGAAGGAGAAGGTGTCTGCGGGA-3', 5'-AGGACGGTGCGGTGAGAGTG-3' |
62°С | HinfI | 198 пн – продукт при наличии аллеля Т: 175 и 23 пн |
| TCF7L2, rs7903146 | C/T | 5'-TAGAGCGCTAAGCACTTTTTAGGA-3', 5'-TTGCCTTCCCTGTAACTGTG-3' |
58°С | RsaI | 96 пн |
| CSK, rs1378942 |
C/A | 5'-TGAATCTGAGCTCATTATAGGGAGG-5', 5'-CCTGCTGAAGAAATAGTGGGTGAT-3' |
60°С | HaeIII | 124 пн |
Статистические методы
Многомерное шкалирование, основанное на частотах гаплотипов мтДНК, было посчитано с помощью программы XLStat (Addinsoft, Inc., 2011).
Генетическое разнообразие h было оценено как:
Генетическую дифференциацию между различными популяциями и ее статистическую значимость оценивали с помощью статистики FST (рассчитанной по частотам гаплогрупп и гаплотипов ГВС I мтДНК). Статистическую значимость межпопуляционных различий по частотам гаплогрупп оценивали с помощью точного теста популяционной дифференциации (количество шагов цепей Маркова – 10 000, уровень значимости P = 0.001). Среднее генетическое разнообразие по локусам считали методом максимального правдоподобия [21]. Расчеты индексов разнообразия для мтДНК, фазирование и подсчет псевдо-гаплотипов по ядерным генам проводились с использованием программы Arlequin 3.01 [22].
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Митохондриальный генофонд амурских эвенков
Исследование нуклеотидной последовательности гипервариабельных сегментов I и II (ГВС I и II) у 98 представителей эвенкийской национальности выявило 61 гаплотип, который был классифицирован в 10 гаплогрупп (табл. 2, 3). Генетическое разнообразие в данной популяции составило 0.984.
Таблица 2.
Частота встречаемости гаплотипов мтДНК в популяции эвенков
| № п/п | ГВС I (–16 000) | ГВС II | Гаплогруппа | % |
|---|---|---|---|---|
| 1 | 134-223-290-319 | 73-152 | A | 1 |
| 2 | 223-290-311-362 | 73-152-263 | A | 1 |
| 3 | 223-290-319-362 | 73-152 | A | 1 |
| 4 | 179-223-290-311-319-362 | 73-152 | A2a3 | 4 |
| 5 | 219-223-298-327 | 73 | C | 1 |
| 6 | 219-223-298-327 | 73-96-124-146 | C | 1 |
| 7 | 223-298-318-519 | 73-263 | C | 1 |
| 8 | 223-298-327 | 73-146 | C | 2 |
| 9 | 223-298-327 | 73 | C | 1 |
| 10 | 223-298-327-519 | 73-146 | C | 3 |
| 11 | 223-298-327-519 | 73 | C | 2 |
| 12 | 223-298-327-519 | 73-146-249d | C | 2 |
| 13 | 223-298-327-519 | 73-146-263 | C | 2 |
| 14 | 129-223-298-327 | 73-124-249d | C4a1 | 1 |
| 15 | 129-223-298-327 | 73-249d-263 | C4a1 | 1 |
| 16 | 129-223-298-327-519 | 73-249d-263 | C4a1 | 1 |
| 17 | 93-129-223-298-327 | 73 | C4a1 | 4 |
| 18 | 93-129-223-298-327-519 | 73 | C4a1 | 1 |
| 19 | 129-150-223-235-298-327-519 | 73-152 | C4a1a4a | 1 |
| 20 | 129-150-223-298-327-519 | 73-195-263 | C4a1a4a | 1 |
| 21 | 51-171-223-198-327-344-357-519 | 73 | C4a2a1 | 1 |
| 22 | 171-223-198-327-344-357-519 | 73-249d-263 | C4a2a1 | 8 |
| 23 | 171-223-241-198-327-344-357 | 73-249d-263 | C4a2a1 | 1 |
| 24 | 223-298-327-344-357-519 | 73-146 | C4a2a1 | 1 |
| 25 | 93-223-261-288-298-519 | 73 | C5a | 1 |
| 26 | 223-261-288-298-519 | 73 | C5a1 | 4 |
| 27 | 223-288-298-327-390-519 | 73-85-152 | C5a1 | 2 |
| 28 | 223-263-288-327-390-519 | 73 | C5d | 1 |
| 29 | 223-362 | D | 2 | |
| 30 | 223-362-519 | 73-263 | D | 1 |
| 31 | 223-362-368 | 85-152 | D4i | 2 |
| 32 | 223-362-368-557-558 | 125-127 | D4i | 1 |
| 33 | 145-223-362 | 152-199 | D4I1a | 2 |
| 34 | 223-291-362 | 73 | D4j2 | 1 |
| 35 | 223-263-362 | 73-150-263 | D4j4a | 1 |
| 36 | 223-263-362-519 | 73-149-254-263 | D4j4a | 2 |
| 37 | 223-263-362-519 | 73-263 | D4j4a | 1 |
| 38 | 172-182-183-189-223-362 | 73-150-263 | D5a2a | 1 |
| 39 | 223-227-274-278-362 | 73-152-263 | G2a | 1 |
| 40 | 223-227-278-362 | 73-152-263 | G2a | 2 |
| 41 | 519 | 73-124 | H | 1 |
| 42 | 519 | 73-146 | H | 1 |
| 43 | CRS | 73 | H | 2 |
| 44 | 519 | 73-151-152-201-249-263 | H | 1 |
| 45 | 362-519 | 73 | H13b | 1 |
| 46 | 362 | 73-152 | H13b | 1 |
| 47 | 162-465-519 | 73-263 | H1a3b | 4 |
| 48 | 357 | 73 | H1af | 1 |
| 49 | 304 | 73 | H5 | 1 |
| 50 | 288-344-362 | 73 | H8 | 2 |
| 51 | 129-172-223-311-391-519 | 73-199 | I1a | 1 |
| 52 | 126-171 | 69-73-150 | J | 1 |
| 53 | 126-241 | 73-150-195-263 | J | 1 |
| 54 | 69-126-143-519 | 73-150-152 | J | 1 |
| 55 | 140-187-209-223-519 | 73-146 | M7a2a3 | 2 |
| 56 | 92-172-182-183-189-266-362 | 73-150-263 | N10a | 4 |
| 57 | 92-172-182-183-189-362 | 73-150-198 | N10a | 1 |
| 58 | 172-182-183-189-296-342-362 | 73-150-263 | N10a | 1 |
| 59 | 172-182-183-189 -362 | 73-150-198 | N10a | 2 |
| 60 | 148-223 | 73 | N | 1 |
| 61 | 223-519 | 73-263 | N | 2 |
| h | 0.984 |
Таблица 3.
Частота встречаемости гаплогрупп мтДНК в популяции амурских эвенков
| № п/п | Гаплогруппа | % |
|---|---|---|
| 1 | A | 7.0 |
| 2 | C | 43.9 |
| 3 | D | 13.3 |
| 4 | G2a | 3.1 |
| 5 | H | 15.3 |
| 6 | I1a | 1.0 |
| 7 | J | 3.1 |
| 8 | M7a2a3 | 2.0 |
| 9 | N10a | 8.2 |
| 10 | N | 3.1 |
Большинство гаплогрупп относятся к восточно-евразийским (A, C, D, G, M), их частота составила 70%. Распространенными являются две: С и D (58%). Обе гаплогруппы характерны для популяций Азии и Восточной Сибири [23].
Самая высокая частота (71.8%) гаплогруппы С выявлена в Центральной Сибири у эвенков: у западных она колеблется от 48.4 до 71.9%, у восточных – 62% [24, 25]. Ранее было показано, что данная галогруппа выявлена в Северо-Восточной Азии у юкагиров (67%), на Дальнем Востоке у тубаларов (19.4%), в Западной Сибири у тундровых ненцев (48%), на Алтае у алтайцев (32.2%), в Восточном Саяне у тофаларов (62.1%), в Западном Саяне у хакасов (27.3%), в районе оз. Байкал у бурят (21.2%), в Волго-Уральском регионе у башкир (12.1%) [23, 25–28]. Таким образом, самая высокая частота данной гаплогруппы обнаружена в Центральной Сибири (52.6%), далее идет Восточный Саян (49.9%), Западная Сибирь (25.6%), Северо-Восточная Азия (22.8%), Алтайский регион и Западный Саян (20%), район оз. Байкал (19%), Дальний Восток (12.3%) и Волго-Уральский регион (3.7%) [23]. В Монголии ее частота достигает 15%, а в популяциях Кореи, Китая, Таиланда, Японии и Индии колеблется от 1 до 5% [23, 28–34]. По частоте данной гаплогруппы эвенки наиболее близки к якутам, тоджинцам и тофаларам [23, 35]. Среди гаплотипов гаплогруппы С наиболее распространенным вариантом является 171-223-198-327-344-357-519 (8%). Ранее данный гаплотип был выявлен в популяциях Средней Азии (тувинцы (3.5%), алтайцы (3.2%), хакасы (2.7%) и шорцы (3.6%)), Дальнего Востока (юкагиры (9.1%), эвенки (5.1%), нганасаны (1.8%)), самодийцев (селькупы (4.2%) и ненцы (1.3)) и кетов (4.5%) [23, 36–38]. Гаплогруппа С, как полагают, возникла между Каспийским морем и оз. Байкал приблизительно 60 000 лет назад, и сейчас наиболее распространена в Северо-Восточной Азии (включая Сибирь) [25, 28, 39].
Второй по распространенности в популяции амурских эвенков является гаплогруппа D (13.3%). Гаплогруппа D широко представлена в популяциях Северной Азии и Алтае-Саянского нагорья. Частота гаплогруппы D у западных эвенков варьирует от 21.1 до 30.4%, у восточных – 24.1% [24, 25]. Самая высокая частота зафиксирована в районе оз. Байкал, от 46.7% у сойотов до 29% у калмыков [23]. На Дальнем Востоке частота этой гаплогруппы – 27.5% (от 0 у удэгейцев до 68.8% у ороков) [25, 40], на Алтае – 18.7% (от 8.9% у алтай-кижи до 26.5% у алтайских казахов) [23], в Западной Сибири – 17% (от 2.9% у кетов до 39.5% у долган) [28, 38], в районе Восточного и Западного Саяна обнаружена у тувинцев (15.6%) и отсутствует у тофаларов [23], в Волго-Уральском регионе – 4.8% (от 0.8% у мордвы до 12.7% у удмуртов) [26]. Гаплогруппа D с низкой частотой найдена и в некоторых северо-восточных популяциях Европы, таких как карелы, скандинавы, популяциях балтийского региона [41, 42]. Наиболее представленным в исследованной популяции является гаплотип 16223-16263-16362 (D4) и его производные (4%) (табл. 2). Данный гаплотип был обнаружен на Дальнем Востоке и в Китае. На Дальнем Востоке его частота колеблется от 0.6% у ульчей до 12.6% у эвенов [43]. Ранее проведенные исследования популяции эвенков также показали высокую частоту данного гаплотипа (7.9%) [36]. В Китае его частота составила 0.96% [44].
Среди западно-евразийских гаплогрупп выявлено пять (N, R, H, I, J), их частота составила 30%. Самая высокая частота гаплогруппы H, (около 48%), выявлена во всех европейских популяциях, а также в Волго-Уральском регионе (42%) [26, 45]. Ранее было показано, что данная гаплогруппа была выявлена у амурских эвенков (2.6%) и западных (1.4%) [24, 36].
В табл. 4 представлены статистически значимые межпопуляционные различия (FST). Наибольшие различия популяции эвенков выявлены с коми, мордвой, финнами, немцами и португальцами (табл. 5), наименьшие различия выявлены у эвенков с шорцами, тувинцами, алтайцами, тофаларами, ненцами и нганасанами. Нами не выявлено никаких статистических различий между алтайцами и шорцами, чувашами и татарами, португальцами и немцами (FST = 0) (табл. 5).
Таблица 4.
Статистически значимые межпопуляционные различия по частотам гаплогрупп мтДНК (FST)
| № п/п | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | 16 | 17 | 18 | 19 | 20 | 21 | 22 | 23 | 24 | 25 | 26 | 27 | 28 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 0.000 | |||||||||||||||||||||||||||
| 2 | 0.033 | 0.000 | ||||||||||||||||||||||||||
| 3 | 0.054 | 0.095 | 0.000 | |||||||||||||||||||||||||
| 4 | 0.085 | 0.124 | 0.020 | 0.000 | ||||||||||||||||||||||||
| 5 | 0.074 | 0.093 | 0.174 | 0.200 | 0.000 | |||||||||||||||||||||||
| 6 | 0.085 | 0.050 | 0.204 | 0.238 | 0.059 | 0.000 | ||||||||||||||||||||||
| 7 | 0.013 | 0.050 | 0.055 | 0.082 | 0.100 | 0.115 | 0.000 | |||||||||||||||||||||
| 8 | 0.032 | 0.078 | 0.044 | 0.049 | 0.154 | 0.177 | 0.039 | 0.000 | ||||||||||||||||||||
| 9 | 0.048 | 0.084 | 0.156 | 0.182 | 0.023 | 0.060 | 0.088 | 0.129 | 0.000 | |||||||||||||||||||
| 10 | 0.006 | 0.043 | 0.064 | 0.099 | 0.066 | 0.082 | 0.003 | 0.061 | 0.055 | 0.000 | ||||||||||||||||||
| 11 | 0.050 | 0.012 | 0.148 | 0.184 | 0.082 | 0.020 | 0.084 | 0.129 | 0.065 | 0.060 | 0.000 | |||||||||||||||||
| 12 | 0.024 | 0.090 | 0.070 | 0.110 | 0.083 | 0.135 | 0.033 | 0.070 | 0.074 | 0.015 | 0.108 | 0.000 | ||||||||||||||||
| 13 | 0.030 | 0.061 | 0.005 | 0.017 | 0.138 | 0.162 | 0.031 | 0.018 | 0.123 | 0.041 | 0.111 | 0.058 | 0.000 | |||||||||||||||
| 14 | 0.054 | 0.064 | 0.154 | 0.181 | 0.006 | 0.030 | 0.085 | 0.133 | 0.010 | 0.047 | 0.045 | 0.080 | 0.122 | 0.000 | ||||||||||||||
| 15 | 0.096 | 0.138 | 0.016 | 0.004 | 0.212 | 0.255 | 0.100 | 0.054 | 0.199 | 0.117 | 0.199 | 0.116 | 0.021 | 0.197 | 0.000 | |||||||||||||
| 16 | 0.008 | 0.028 | 0.059 | 0.103 | 0.079 | 0.078 | 0.017 | 0.050 | 0.057 | 0.008 | 0.053 | 0.040 | 0.036 | 0.058 | 0.117 | 0.000 | ||||||||||||
| 17 | 0.066 | 0.100 | 0.005 | 0.012 | 0.178 | 0.213 | 0.073 | 0.046 | 0.160 | 0.082 | 0.156 | 0.089 | 0.010 | 0.157 | 0.008 | 0.072 | 0.000 | |||||||||||
| 18 | 0.037 | 0.052 | 0.061 | 0.075 | 0.147 | 0.138 | 0.046 | 0.010 | 0.121 | 0.057 | 0.089 | 0.086 | 0.033 | 0.117 | 0.081 | 0.045 | 0.052 | 0.000 | ||||||||||
| 19 | 0.079 | 0.106 | 0.180 | 0.212 | 0.119 | 0.126 | 0.120 | 0.158 | 0.044 | 0.106 | 0.094 | 0.120 | 0.148 | 0.087 | 0.230 | 0.074 | 0.190 | 0.144 | 0.000 | |||||||||
| 20 | 0.032 | 0.060 | 0.139 | 0.173 | 0.024 | 0.047 | 0.070 | 0.116 | 0.000 | 0.039 | 0.041 | 0.056 | 0.108 | 0.010 | 0.189 | 0.041 | 0.147 | 0.105 | 0.042 | 0.000 | ||||||||
| 21 | 0.100 | 0.132 | 0.013 | 0.030 | 0.212 | 0.249 | 0.109 | 0.078 | 0.198 | 0.120 | 0.194 | 0.126 | 0.027 | 0.191 | 0.022 | 0.097 | 0.005 | 0.092 | 0.222 | 0.186 | 0.000 | |||||||
| 22 | 0.028 | 0.082 | 0.074 | 0.137 | 0.134 | 0.149 | 0.031 | 0.080 | 0.101 | 0.019 | 0.111 | 0.029 | 0.062 | 0.110 | 0.148 | 0.014 | 0.103 | 0.089 | 0.107 | 0.078 | 0.123 | 0.000 | ||||||
| 23 | 0.021 | 0.059 | 0.003 | 0.041 | 0.145 | 0.160 | 0.020 | 0.033 | 0.124 | 0.022 | 0.107 | 0.042 | 0.000 | 0.122 | 0.045 | 0.019 | 0.024 | 0.047 | 0.145 | 0.102 | 0.035 | 0.023 | 0.000 | |||||
| 24 | 0.141 | 0.201 | 0.270 | 0.286 | 0.038 | 0.144 | 0.178 | 0.239 | 0.053 | 0.156 | 0.180 | 0.157 | 0.225 | 0.055 | 0.300 | 0.152 | 0.277 | 0.237 | 0.166 | 0.071 | 0.302 | 0.214 | 0.246 | 0.000 | ||||
| 25 | 0.098 | 0.141 | 0.020 | 0.007 | 0.220 | 0.263 | 0.103 | 0.056 | 0.204 | 0.129 | 0.203 | 0.121 | 0.025 | 0.199 | 0.000 | 0.121 | 0.014 | 0.084 | 0.234 | 0.191 | 0.031 | 0.163 | 0.055 | 0.317 | 0.000 | |||
| 26 | 0.083 | 0.051 | 0.185 | 0.219 | 0.161 | 0.095 | 0.125 | 0.169 | 0.114 | 0.120 | 0.033 | 0.161 | 0.147 | 0.117 | 0.238 | 0.086 | 0.196 | 0.138 | 0.092 | 0.084 | 0.228 | 0.138 | 0.142 | 0.238 | 0.237 | 0.000 | ||
| 27 | 0.074 | 0.135 | 0.182 | 0.206 | 0.019 | 0.105 | 0.107 | 0.135 | 0.029 | 0.078 | 0.128 | 0.077 | 0.146 | 0.030 | 0.215 | 0.090 | 0.186 | 0.139 | 0.133 | 0.039 | 0.221 | 0.133 | 0.154 | 0.027 | 0.225 | 0.204 | 0.000 | |
| 28 | 0.063 | 0.060 | 0.185 | 0.232 | 0.038 | 0.012 | 0.082 | 0.151 | 0.045 | 0.043 | 0.037 | 0.083 | 0.146 | 0.020 | 0.248 | 0.054 | 0.203 | 0.123 | 0.110 | 0.030 | 0.241 | 0.092 | 0.130 | 0.114 | 0.259 | 0.121 | 0.062 | 0.000 |
Примечание. 1. т-и татары [47]; 2. казахи [48]; 3. мари [26]; 4. исландцы [49]; 5. эвенки [данное исследование]; 6. буряты [50]; 7. ханты [27]; 8. коми [26]; 9. шорцы [48]; 10. манси [51]; 11. монголы [52]; 12. селькупы [28]; 13. татары [26]; 14. тувинцы [48]; 15. немцы [53, 54]; 16. башкиры [26]; 17. мордва [26]; 18. удмурты [26]; 19. хакасы [48]; 20. алтайцы [48]; 21. фины [42, 55, 56]; 22. кеты [57]; 23. чуваши [26]; 24. тофалары [50]; 25. португальцы [58]; 26. китайцы [31]; 27. ненцы [59]; 28. нганасаны [37].
Таблица 5.
Частота встречаемости генотипов и аллелей изученных генов в популяции эвенков
| Ген, полиморфизм* | DD | ID | II | CC | CT | TT | АА | АС | CC |
|---|---|---|---|---|---|---|---|---|---|
| ACE (230), rs1799752 |
13.5 (31) |
45.7 (105) | 40.9 (94) | ||||||
| NOS3 (228), rs61722009 |
0.0 (0) |
8.3 (19) |
91.7 (209) | ||||||
| ADRA2B (230), rs28365031 |
19.6 (45) | 46.1 (106) | 34.3 (79) | ||||||
| MTHFR (230), rs1801133 |
74.3 (171) | 21.7 (50) | 3.9 (9) | ||||||
| TCF7L2 (229), rs7903146 |
63.8 (146) | 34.5 (79) | 1.7 (4) | ||||||
| CSK (224), rs1378942 |
72.3 (162) | 23.2 (52) | 4.5 (10) | ||||||
| D | I | C | T | A | T | ||||
| ACE (230), rs1799752 |
36.3 (167) |
63.7 (293) |
|||||||
| NOS3 (228), rs61722009 |
4.2 (19) |
95.8 (437) |
|||||||
| ADRA2B (230), rs28365031 |
42.6 (196) |
57.4 (264) |
|||||||
| MTHFR (230), rs1801133 |
85.2 (392) |
14.8 (68) |
|||||||
| TCF7L2 (229), rs7903146 |
81.0 (371) |
19.0 (87) |
|||||||
| CSK (224), rs1378942 |
83.9 (376) |
16.1 (72) |
На рис. 1 представлены результаты многомерного шкалирования попарных генетических расстояний гаплотипов мтДНК в некоторых популяциях. Наблюдается достаточно четкое разграничение популяций. В верхнем поле расположились популяции, в генетическом пуле которых выявлено наибольшее количество западно-евразийских гаплогрупп, – это популяции, относящиеся к финно-угорской языковой семье, а также португальцы, немцы, исландцы, селькупы и кеты. В нижнем поле – популяции, проживающие на территории Алтае-Саянского нагорья, а также эвенки, буряты, монголы, китайцы и две самодийские популяции (ненцы и нганасаны). В генетическом пуле этих популяций в основном присутствуют восточно-евразийские гаплогруппы.
Рис. 1.
Многомерное шкалирование попарных генетических расстояний восточно- и западно-евразийских гаплотипов мтДНК в некоторых популяциях. Мордва, мари, чуваши, татары, коми, удмурты, башкиры [26]; ханты [27]; селькупы [28]; монголы [51]; китайцы [31]; нганасаны [37]; финны [42, 54, 55]; казахи, алтайцы, шорцы, тувинцы, хакасы [47]; т-и татары [46]; тофалары, буряты [49]; кеты [56]; немцы [52, 53]; исландцы [48]; манси [50]; португальцы [57]; ненцы [58].
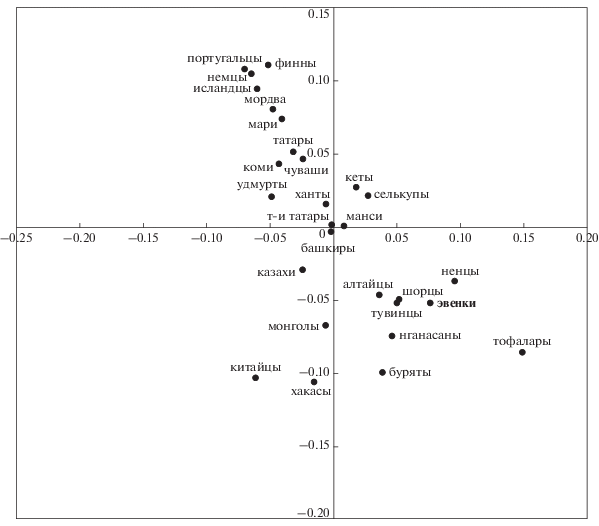
Таким образом, проведенные нами исследования отражают сложную историю населения Сибири. Наличие в генетическом пуле эвенков около 70% восточно-евразийских гаплогрупп, с преобладанием двух С и D (57%), сближает их не только с популяциями Северной Азии и Сибири, но и с некоторыми популяциями Алтаe-Саянского нагорья. Из западно-евразийских гаплогрупп самой распространенной у эвенков является гаплогруппа Н (15%), характерная для европеоидных популяций. Выявленые нами результаты согласуются с ранее полученными данными по эвенкам.
Полиморфизм генов ACE, NOS3, ADRA2B, MTHFR, TCF7L2 и CSK
Ранее было высказано предположение, что в процессе paco- и этногенеза частоты аллелей и генотипов приобрели свою специфику у разных народов, а также с изменением их образа жизни и диеты; дальнейшие исследования должны включать изучение генов некоторых мультифакториальных заболеваний, таких как инсульт, инфаркт, артериальная гипертензия, диабет и т.д. [19]. В настоящей работе проведено исследование популяции амурских эвенков по шести генам ACE (I/D, rs1799752), NOS3 (4b/4a, rs61722009), ADRA2B (I/D, rs28365031), MTHFR (С677Т, rs1801133), TCF7L2 (rs7903146) и CSK (rs1378942) (табл. 5).
Одним из ключевых звеньев ренин-ангиотензиновой системы является ангиотензинпревращающий (АСЕ) фермент, активность которого примерно на 50% находится под генетическим контролем. Этот полиморфизм гена ACE обусловлен присутствием (инсерцией) или отсутствием (делецией) элемента Alu размером 287 пн в интроне 16. Лица, гомозиготные по делеции, имеют более высокий уровень ангиотензинпревращающего фермента в плазме, высокую активность превращения ангиотензина I в ангиотензин II и разрушение вазопротекторного пептида брадикинина [59, 60]. В связи с этим было высказано предположение, что аллель D (делеция) является фактором риска артериальной гипертензии, так как играет важную роль в регуляции кровяного давления и водно-солевого обмена [61, 62]. Анализ гена ACE (I/D) показал, что частота встречаемости варианта D (делеция) в исследованной нами выборке в 2 раза ниже, чем варианта I (инсерция), 36.3 и 63.7% соответственно (табл. 5). По частоте генотипа DD (13.5%) амурские эвенки близки к бурятам и шорцам (14.5 и 10.1% соответственно) [19, 63]. У русских его частота колеблется от 20.8 до 32.6%, у мордвы – 25.4% [19, 64].
Полиморфизм гена NOS3 характеризуется количеством повторов участка в 4-м интроне гена NOS3 (4a/4b) и ассоциирован с изменением уровня NO в плазме крови и, как было показано, является причиной изменения уровней нитритов и нитратов в плазме крови [65, 66]. Тандемные повторы в интроне 4 (4b/4a) являются одним из изученных маркеров гена NOS3. Вариант 4b включает пять повторов (по 27 пн), а редкий вариант 4а связан с делецией одной из трех первых пар оснований. В изученной нами популяции вариант 4b с пятью повторами (95.8%) встречается чаще, чем вариант 4а (4.2%) (табл. 5). В популяциях Восточной Сибири частота гомозиготного генотипа 4а/4а гена NOS3 составила 15.56% [67]. У шорцев выявлено преобладание генотипа 4b/4b (82.5%) у коренного населения и 67.7% у некоренного населения [19]. У русских генотип 4а/4а составил 8.7% [19], в Австралии – 1%, у британцев – 2.9%, у словаков – 5.2% [68, 69]. В азиатских популяциях встречаемость редкого генотипа колеблется от 1.4% у корейцев до 2% у японцев [65, 70]. В исследованной нами выборке редкий генотип не обнаружен.
Активация α2В-адренергических рецепторов (ADRA2B), локализованных в гладкомышечных клетках сосудов, приводит к вазоконстрикции. При исследовании амурских эвенков была выявлена высокая частота аллеля D (делеция) (42.6%), который является важным генетическим маркером развития артериальной гипертензии (табл. 5). Самая высокая частота аллеля D выявлена у русских (47.6%) и финнов (47.5%), а самая низкая – у японцев (35%) и корейцев (36%) [19, 71, 72]. У шорцев частота данного полиморфизма составила 39% [19]. Таким образом, по частоте аллеля D (42.6%) изученная нами выборка занимает промежуточное положение между японской (35%), с одной стороны, финской (47.5%) и русской (47.6%) популяциями, с другой стороны [19, 71, 72].
Ген MTHFR кодирует белок метилентетрагидрофолатредуктазу, участвующий в превращении гомоцистеина в метионин при наличии кофакторов – пиридоксина, цианокобаламина и субстрата – фолиевой кислоты. Гомоцистеин участвует в повреждении эндотелия сосудов: усиливается пролиферация гладкомышечных клеток, в мембранах клеток накапливаются липопротеины низкой и очень низкой плотности, снижается эластичность стенки сосудов [73]. Частота мутантного аллеля Т у амурских эвенков составляет 14.8% (табл. 5). Самая высокая частота аллеля Т обнаружена у испанцев (55.0%) и самая низкая у индонезийцев (2.0%) [74, 75]. По частоте встречаемости аллеля Т эвенки наиболее близки к шорцам (12.8%) [19]. В популяции русских его частота варьирует от 25 до 29%, в Западной Азии – 16.4%, в Восточной Азии – 34.2% и в европейских популяциях средняя частота составляет 28.8% [19, 76–81].
Ген TCF7L2 кодирует Т-клеточный транс-крипционный фактор, участвующий в контроле гомеостаза глюкозы в крови. У амурских эвенков редкий аллель T был выявлен с частотой 19% (табл. 5). Во всем мире его частота варьирует от 6.9% в Восточной Азии до 35% в Южной Азии, в Европе он встречается с частотой 33% [82]. Исследованная нами выборка занимает промежуточное положение между Восточной Азией, с одной стороны, и Южной Азией и Европой, с другой стороны. В популяции русских его частота 23%, а у татар 35% [83, 84].
Различные семейства тирозинкиназы кодирует ген CSK [85]. Тирозинкиназы являются представителями семейства протеинкиназ – натрийуретических пептидов, катализирующих перенос концевого фосфата с молекулы АТР на гидроксильную группу остатка тирозина в молекулах клеток-мишеней, которыми представлены сердечно-сосудистая система, почки, эндокринные органы и центральная нервная система [86, 87]. Частота редкого аллеля T в популяции эвенков не превышает 16.1% (табл. 5). Ранее высокая частота выявлена в выборках хантов и манси (57.2%) [88]. В популяции русских она колеблется от 41% в Красноярском крае до 57% в Ханты-Мансийском автономном округе [88, 89]. У корейцев его частота составила 82.8% [90]. Выявленная нами частота в исследованной популяции является самой низкой из всех исследованных групп.
Наблюдаемое распределение частот локусов по четырем генам в исследованной популяции не отличалось от теоретически ожидаемого согласно закону Харди–Вайнберга, исключение составили TCF7L2 и CSK (табл. 6).
Таблица 6.
Проверка соответствия распределению Харди–Вайнберга для шести локусов изученных генов
| Гетерозиготность | ACE | NOS3 | ADRB2 | MTHFR | TCF7L2 | CSK |
|---|---|---|---|---|---|---|
| Наблюдаемая | 0.47059 | 0.08597 | 0.46154 | 0.22172 | 0.35294 | 0.23529 |
| Ожидаемая | 0.47010 | 0.08246 | 0.48553 | 0.25143 | 0.30855 | 0.27334 |
| P-value | 1.00000 | 1.00000 | 0.49148 | 0.10383 | 0.03008 | 0.04753 |
Генетическую основу различных заболеваний составляют сочетания полиморфных вариантов различных генов. В связи с вышесказанным нами проведен анализ ассоциации аллелей локусов исследуемых генов. Использован метод оценки неравновесия по сцеплению для локусов, находящихся в трансположении. В табл. 7 представлены данные о неравновесии по парному сцеплению в четырех парах генов (ACE и MTHFR, ACE и TCF7L2, NOS3 и CSK, ADRA2B и MTHFR). Как мы видим, эти четыре пары имеют неравновесие по сцеплению с вероятностью Р < 10–5. Нами были взяты эти четыре пары для детальной проверки распределения ди-гетерозигот. В табл. 8 представлены результаты достоверного отклонения от ожидаемого распределения ди-гетерозигот по четырем локусам генов. Выявлено, что наиболее часто встречаются аллели минор–мажор (гетерозиготы) и мажор–мажор (гомозиготы по мажорному аллелю). В каждой паре отмечен определяющий ген, так как он встречается с высокой частотой в сочетании мажор–минор и мажор–мажор. В первых двух парах определяющим геном является ACE, в третьей паре – NOS3 и в четвертой – ADRA2B.
Таблица 7.
Неравновесие по парному сцеплению
| Гены | P-value |
|---|---|
| ACE, NOS3 | 0.45846 ± 0.01353 |
| ACE, ADRA2B | 0.14076 ± 0.01038 |
| ACE, MTHFR | 0.00000 ± 0.00000 |
| ACE, TCF7L2 | 0.00000 ± 0.00000 |
| ACE, CSK | 0.01075 ± 0.00300 |
| NOS3, ADRA2B | 0.89541 ± 0.00780 |
| NOS3, MTHFR | 0.00880 ± 0.00288 |
| NOS3, TCF7L2 | 0.00293 ± 0.00164 |
| NOS3, CSK | 0.00000 ± 0.00000 |
| ADRA2B, MTHFR | 0.00000 ± 0.00000 |
| ADRA2B, TCF7L2 | 0.17107 ± 0.00969 |
| ADRA2B, CSK | 0.18182 ± 0.01201 |
| MTHFR, TCF7L2 | 0.08407 ± 0.00804 |
| MTHFR, CSK | 0.24242 ± 0.01150 |
| TCF7L2, CSK | 0.51613 ± 0.01769 |
Таблица 8.
Достоверное отклонение от ожидаемого распределения ди-гетерозигот
| Гены | Псевдогаплотипы | Число ди-гетерозигот | χ2, P-value | |
|---|---|---|---|---|
| наблюдаемое | ожидаемое | |||
| ACE, MTHFR | DC | 166 | 139 | χ2 = 34.8,P-value = 0.000001343 |
| IC | 211 | 232 | ||
| IT | 65 | 41 | ||
| DT | 0 | 26 | ||
| ACE, TCF7L2 | DC | 161 | 132 | χ2 = 115.7,P-value = 0.0000000001 |
| DT | 5 | 31 | ||
| IC | 197 | 222 | ||
| IT | 79 | 52 | ||
| NOS3, CSK | DG | 7 | 15 | χ2 = 8.8,P-value = 0.31 |
| DT | 12 | 3 | ||
| IG | 363 | 356 | ||
| IT | 60 | 68 | ||
| ADRA2B, MTHFR | DC | 171 | 154 | χ2 = 10.64,P-value = 0.014 |
| DT | 11 | 27 | ||
| IC | 206 | 222 | ||
| IT | 54 | 39 | ||
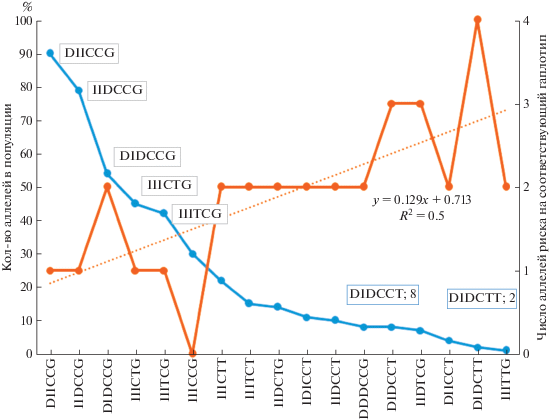
На рис. 2 представлен график частот сочетаний аллелей (псевдогаплотипов), оцененных методом максимального правдоподобия. Как мы видим на графике, из 32 псевдогаплотипов только 16 встретились в исследованной выборке 2 и более раз. Самыми распространенными (более 40) оказались пять. Нами было посчитано число аллелей риска, ассоциированных с неблагоприятным эффектом, на каждый псевдогаплотип. Как мы наблюдаем относительно графика линейной регрессии, число аллелей риска в целом пропорционально возрастает с уменьшением частоты встречаемости гаплотипов (R2 = 0.5, R = 0.7, d.f. = 16, p < 0.001). При том, что все аллели риска являются минорными, данная картина ожидаемая, так как число аллелей риска обратно пропорционально численности соответствующего псевдогаплотипа.
Рис. 2.
График частот псевдогаплотипов, оцененных методом максимального правдоподобия. Число носителей определенного гаплотипа (кол-во человек) – синяя линия; число аллелей, ассоциированных с риском возникновения заболевания, в составе гаплотипа – оранжевая линия.
Таким образом, проведенные нами исследования амурских эвенков выявили восточно-евразийские гаплогруппы мтДНК, характерные для популяций Северной Азии и Сибири. Полученные результаты по мтДНК согласуются с ранее выявленными данными.
Исследованные шесть полиморфных вариантов ядерных генов, связанных с некоторыми мультифакториальными заболеваниями, такими как гипертензия, атеросклероз, ишемическая болезнь сердца и диабет, выявили, что для популяции эвенков не характерно распространение аллелей генов, предрасполагающих к данным заболеваниям. Возможно, это связано с тем, что амурские эвенки в течение продолжительного времени находились не только в благоприятных условиях окружающей среды, но и в относительной изоляции. В результате исследования сочетаний аллелей у индивидов показано, что число аллелей, ассоциированных с заболеваниями, пропорционально возрастает с уменьшением частоты встречаемости псевдогаплотипов. Выявленные нами неслучайные сцепления локусов, вероятно, могут отражать такие процессы как гаметическое неравновесие (мейотический драйв). Для выяснения наших предположений необходимо продолжить изучение данного набора генов на других популяциях.
Авторы выражают благодарность Научно-просветительскому Центру палеоэтнологических исследований (Москва) и лично Д.В. Пежемскому за участие в организации и проведении экспедиции в 2016 г.
Работа выполнена при финансовой поддержке Бюджетного проекта: № 0259-2021-0014, гранта МГУ им. М.В. Ломоносова для поддержки ведущих научных школ МГУ “Депозитарий живых систем Московского университета” в рамках Программы развития МГУ, гранта РФФИ, проект № 20-011-00408, в рамках темы НИР “Эволюционный континуум рода Homo”.
Все процедуры, выполненные в исследовании с участием людей, соответствуют этическим стандартам институционального и/или национального комитета по исследовательской этике и Хельсинкской декларации 1964 г. и ее последующим изменениям или сопоставимым нормам этики.
От каждого из включенных в исследование участников было получено информационное добровольное согласие.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Деревянко А.П., Волков П.В., Ли Х. Селемджинская позднепалеолитическая культура. Новосибирск: Ин-т археологии и этнографии СО РАН, 1998. 336 с.
Нестеров С.П. Народы Приамурья в эпоху раннего средневековья. Новосибирск: Изд-во Ин-та археологии и этнографии СО РАН, 1998. 184 с.
Всероссийская перепись населения. 2010.
Забияко А.П., Аниховский С.Э., Воронкова Е.А. и др. Эвенки Приамурья: оленная тропа истории и культуры / Под ред. Забияко А.П. Благовещенск, 2012. 384 с.
История и культура дальневосточных эвенков: историко-этнографические очерки / Под ред. Тураева В.А. СПб.: Наука, 2010. 334 с.
Дебец Г.Ф. Антропологические исследования в Камчатской области // Тр. Ин-та этнографии АН СССР. Т. XVII. 1951. 264 с.
Левин М.Г. Этническая антропология и проблемы этногенеза народов Дальнего Востока // Тр. Ин-та этнографии АН СССР. Т. XXXVI. 1958. 359 с.
Баранов В.С., Иващенко Т.Э., Баранова Е.В. Генетический паспорт – основа индивидуальной и предиктивной медицины. СПб.: Изд-во Н.-Л., 2009. 528 с.
Горбунова В.Н. Генетика и эпигенетика синтропных заболеваний // Экол. генетика. 2010. Т. 8. № 4. С. 39–43.
Snapir A., Scheinin M., Groop L.C., Orho-Melander M. The insertion/deletion variation in the α2B-adrenoceptor does not seem to modify the risk for acute myocardial infarction, but may modify the risk for hypertension in sib-pairs from families with type 2 diabetes // Cardiovasc. Diabetol. 2003. V. 24. № 2. P. 15–21. https://doi.org/10.1186/1475-2840-2-15
Lima J.J., Feng H., Duckworth L. et al. Association analyses of adrenergic receptor polymorphisms with obesity and metabolic alterations // Metabolism. 2007. V. 56. № 6. P. 757–765. https://doi.org/10.1016/j.metabol.2007.01.007
Salimi S., Firoozrai M., Nourmohammadi I. et al. Endothelial nitric oxide synthase gene intron 4 VNTR polymorphism in patients with coronary artery disease in Iran // Indian J. Med. Res. 2006. V. 124. № 6. P. 683–688.
Lindpaintner K., Pfeffer M.A., Kreutz R. et al. A prospective evaluation of an angiotensin-converting enzyme gene polymorphism and the risk of ischemic heart disease // N. Engl. J. Med. 1995. V. 332. № 11. P. 706–711. https://doi.org/10.1056/NEJM199503163321103
Fujimura H., Kawasaki T., Sakata T. et al. Common C677T polymorphism in the methylenetetrahydrofolate reductase gene increases the risk for deep vein thrombosis in patients with predisposition of thrombophilia // Thromb. Res. 2000. V. 98. № 1. P. 1–8. https://doi.org/10.1016/s0049-3848(99)00231-5
Wang Y., Kikuchi S., Suzuki H. et al. Endothelial nitric oxide synthase gene polymorphism in intron 4 affects the progression of renal failure in non-diabetic renal diseases // Nephrol. Dial. Transplant. 1999. V. 14. № 12. P. 2898–2902. https://doi.org/10.1093/ndt/14.12.2898
Avila-Vanzzini N., Posadas-Romero C., Gonzalez-Salazar M. et al. The ACE I/D polymorphism is associated with nitric oxide metabolite and blood pressure levels in healthy Mexican men // Arch. Cardiol. Mex. 2015. V. 85. № 2. P. 105–110. https://doi.org/10.1016/j.acmx.2014.12.005
Rolim T., Cancino J., Zucolotto V. A nanostructured genosensor for the early diagnosis of systemic arterial hypertension // Biomed. Microdevices. 2015. V. 17. № 1. P. 3–9. https://doi.org/10.1007/s10544-014-9911-z
Hu D.C., Zhao X.L., Shao J.C. et al. Interaction of six candidate genes in essential hypertension // Genet. Mol. Res. 2014. V. 13. № 4. P. 8385–8395. https://doi.org/10.4238/2014
Барабаш О.Л., Воевода М.И., Артамонова Г.В. и др. Генетические детерминанты артериальной гипертонии в двух национальных когортах Горной Шории // Терапев. архив. 2017. Т. 89. № 9. С. 68–77.
Andrews R.M., Kubacka I., Chinnery P.F. et al. Reanalysis and revision of the Cambridge reference sequence for human mitochondrial DNA // Nat. Genet. 1999. V. 23. № 2. P. 147.
Nei M., Tajima F. DNA polymorphism detectable by restriction endonucleases // Genetics. 1981. V. 97. № 1. P. 145–163.
Excoffier L., Laval G., Schneider S. Arlequin (version 3.0): An integrated software package for population genetics dataanalysis // Evol. Bioinform. Online. 2005. № 1. P. 47–50.
Derenko M., Malyarchuk B., Grzybowski T. et al. Origin and post-glacial dispersal of mitochondrial DNA haplogroups C and D in Northern Asia // PLoS One. 2010. V. 5. № 12. P. 1–9. https://doi.org/10.1371/journal.pone.0015214
Derenko M., Malyarchuk B., Grzybowski T. et al. Phylogeographic analysis of mitochondrial DNA in Northern Asian populations // Am. J. Hum. Gene. 2007. V. 81. № 5. P. 1025–1041. https://doi.org/10.1086/522933
Starikovskaya E., Sukernik R., Derbeneva O. et al. Mitochondrial DNA diversity in indigenous populations of the Southern extent of Siberia, and the origins of Native American haplogroups // Ann. of Hum. Genet. 2005. V. 69. № 1. P. 67–89. https://doi.org/10.1046/j.1529-8817.2003.00127.x
Бермишева М., Тамбетс К., Виллемс Р., Хуснутдинова Э. Разнообразие гаплогрупп митохондриальной ДНК у народов Волго-Уральского региона России // Мол. биология. 2002. Т. 36. № 6. С. 990–1001.
Volodko N.V., Starikovskaya E.B., Mazunin I.O. et al. Mitochondrial genome diversity in arctic Siberians, with particular reference to the evolutionary history of Beringia and Pleistocenic peopling of the Americas // Am. J. Hum. Genet. 2008. V. 82. № 5. P. 1084–1100. https://doi.org/10.1016/j.ajhg.2008.03.019
Tanaka M., Cabrera V.M., González A.M. et al. Mitochondrial genome variation in Eastern Asia and the peopling of Japan // Genome Res. 2004. V. 14. № 10A. P. 1832–1850. https://doi.org/10.1101/gr.2286304
Lee H.Y., Yoo J.E., Park M.J. et al. Mitochondrial DNA control region sequences in Koreans: Identification of useful variable sites and phylogenetic analysis for mtDNA data quality control // Int. J. Legal Med. 2006. V. 120. № 1. P. 5–14. https://doi.org/10.1007/s00414-005-0005-6
Kivisild T., Tolk H.-V., Parik Y. et al. The emerging limbs and twigs of the East Asian mtDNA tree // Mol. Biol. Evol. 2002. V. 19. № 10. P. 1737–1751. https://doi.org/10.1093/oxfordjournals.molbev.a003996
Yao Y.-G., Kong Q.-P., Bandelt H.-J. et al. Phylogeographic differentiation of mitochondrial DNA in Han Chinese // Am. J. Hum. Genet. 2002. V. 70. № 3. P. 635–651. https://doi.org/10.1086/338999
Yao Y.G., Kong Q.P., Wang C.Y. et al. Different matrilineal contributions to genetic structure of ethnic groups in the Silk Road region in China // Mol. Biol. Evol. 2004. V. 21. № 12. P. 2265–2280. https://doi.org/10.1093/molbev/msh238
Metspalu M., Kivisild T., Metspalu E. et al. Most of the extant mtDNA boundaries in south and southwest Asia were likely shaped during the initial settlement of Eurasia by anatomically modern humans // BMC Genet. 2004. V. 5. № 26. P. 1–25. https://doi.org/10.1186/1471-2156-5-26
Wen B., Li H., Gao S. et al. Genetic structure of Hmong-Mien speaking populations in East Asia as revealed by mtDNA lineages // Mol. Biol. Evol. 2005. V. 22. № 3. P. 725–734. https://doi.org/10.1093/molbev/msi055
Федорова С.А., Бермишева М.А., Виллемс Р. и др. Анализ гаплотипов митохондриальной ДНК в популяции якутов // Мол. биология. 2003. Т. 37. № 4. С. 643–653.
Губина М.А., Гырголькау Л.А., Бабенко В.Н. и др. Полиморфизм митохондриальной ДНК в популяциях коренных жителей Дальнего Востока // Генетика. 2013. Т. 49. № 7. С. 862–876.
Гольцова Т.В., Осипова Л.П., Жаданов С.И., Виллемс Р. Влияние брачной миграции на генетическую структуру популяции нганасан Таймыра: генеалогический анализ по маркерам митохондриальной ДНК // Генетика. 2005. Т. 41. № 7. С. 954–965.
Derbeneva O., Starikovskaya E., Wallace D., Sukernik R. Traces of early Eurasians in the Mansis of Northwest Siberia revealed by mitichondrial DNA analysis // Am. J. Hum. Genet. 2002. V. 70. № 4. P. 1009–1014. https://doi.org/10.1086/339524
Kong Q.P., Yao Y.G., Sun C. et al. Phylogeny of East Asian mitochondrial DNA linerages inferred from complete sequences // Am. J. Hum. Genet. 2003. V. 73. № 3. P. 671–676. https://doi.org/10.1086/377718
Бермишева М.А., Кутуев И.А., Спицын В.А. и др. Анализ изменчивости митохондриальной ДНК в популяции oроков // Генетика. 2005. Т. 41. № 1. С. 78–84.
Lappalainen T., Laitinen V., Salmela E. Migration waves to the Baltic Sea region // Ann. Hum. Genet. 2008. V. 72. Pt. 3. P. 337–348. https://doi.org/10.1111/j.1469-1809.2007.00429.x
Richards M.B., Macaulay V., Hickey E. et al. Tracing European founder lineages in the Near Eastern mtDNA pool // Am. J. Hum. Genet. 2000. V. 67. № 5. P. 1251–1276.
Sukernik R.I., Volodko N.V., Mazunin I.O. et al. Mitochondrial genome diversity in the Tubalar, Even, and Ulchi: Contribution to prehistory of native Siberians and their affinities to native Americans // Am. J. Phys. Anthropol. 2012. V. 148. № 1. P. 123–138. https://doi.org/10.1002/ajpa.22050
Xu K., Hu S. Population data of mitochondrial DNA HVS-I and HVS-II sequences for 208 Henan Han Chines // Legal Med. 2015. V. 17. № 4. P. 287–294. https://doi.org/10.1016/j.legalmed.2015.02.003
Simoni L., Calafell F., Pettener D. et al. Geographic patterns of mtDNA diversity in Europe // Am. J. Hum. Genet. 2000. V. 66. № 1. P. 262–278. https://doi.org/10.1086/302706
Наумова О.Ю., Рычков С.Ю., Морозова И.Ю. и др. Разнообразие митохондриальной ДНК у тоболо-иртышских сибирских татар // Генетика. 2008. Т. 44. № 2. С. 257–268.
Губина М.А., Дамба Л.Д., Бабенко В.Н. и др. Гаплотипическое разнообразие мтДНК и Y-хромосомы в популяциях Алтае-Саянского региона // Генетика. 2013. Т. 49. № 3. С. 376–391.
Helgason A., Sigurdaroottir S., Gulcher J. et al. mtDNA and the origin of the Icelanders: Deciphering signals of recent population history // Am. J. Hum. Genet. 2000. V. 66. № 3. P. 999–1016. https://doi.org/10.1086/302816
Деренко М.В., Малярчук Б.А., Денисова Г.А. и др. Молекулярно-генетическая дифференциация этнических групп Южной и Восточной Сибири по данным о полиморфизме митохондриальной ДНК // Генетика. 2002. Т. 38. № 10. С. 1409–1416.
Pimenoff V., Cjmas D., Palo J. et al. Northwest Siberian Khanty and Mansi in the junction of West and East Eurasian gene pools as revealed by uniparental markers // Eur. J. Hum. Genet. 2008. V. 16. № 10. P. 1254–1264. https://doi.org/10.1038/ejhg.2008.101
Kolman C., Sambuughin N. Mitochondrial DNA analysis of Mongolian populations and implications for the origin of New World founders // Genetics. 1996. V. 142. № 4. P. 1321–1334.
Lutz S., Weisser H., Heizmann J. et al. Location and frequency of polymorphic positions in the mtDNA control region of individuals from Germany // Int. J. Legal Med. 1998. V. 111. № 2. P. 67–77. https://doi.org/10.1007/s004140050117
Pfeiffer H., Brinkmann B., Huhne J. et al. Expanding the forensic German mitochondrial DNA control region database: Genetic diversity as a function of sample size and microgeography // Int. J. Legal Med. 1999. V. 112. № 5. P. 291–298. https://doi.org/10.1007/s004140050252
Lahermo P., Sajantila A., Sistonen P. et al. The genetic relationship between the Finns and the Finnish Saami (Lapps): Analysis of nuclear DNA and mtDNA // Am. J. Hum. Genet. 1996. V. 58. № 6. P. 1309–1322.
Kittles R., Bergen A., Urbane K. et al. Autosomal, mitochondrial, and Y Chromosome DNA variation in Finland: Evidence for a male specific bottleneck // Am. J. Phys. Anthropol. 1999. V. 108. № 4. P. 381–399. https://doi.org/10.1002/(SICI)1096-8644(199904)-108:4<381::AID-AJPA1>3.0.CO;2-5
Дербенева О.А., Стариковская Е.В., Володько Н.В. и др. Изменчивость митохондриальной ДНК у кетов и нганасан в связи с первоначальным заселением Северной Евразии // Генетика. 2002 Т. 38. № 11. С.1554–1560.
Pereira L., Prata M.J., Amorim A. Diversity of mtDNA lineages in Portugal not a genetic edge of European variation // An. Hum. Genet. 2000. V. 64. № 6. P. 491–506. https://doi.org/10.1046/j.1469-1809.2000.6460491.x
Saillard J., Evseva I., Tranebjaerg L., Norby S. Mitochondrial DNA diversity among nenets. Archaeogenetics: DNA and the population prehistory of Europe. McDonald Institute for archaeological research monograph series / Eds Renfrew C., Boyle K. Cambridge: Cambr. Univ. Press, 2000. P. 255–258.
Danser A.H., Schalekamp M.A., Bax W.A. et al. Angiotensin-converting enzyme in the human heart. Effect of the deletion/insertion polymorphism // Circulation. 1995. V. 92. № 6. P. 1387–1388. https://doi.org/10.1161/01.cir.92.6.1387
Шулутко Б.И. Артериальная гипертензия. СПб.: Сотис, 2001. С. 98–108.
Fox C.S., Heard-Costa N.L., Vasan R.S. et al. Genomewide linkage analysis of weight change in the framingham heart study // J. Clinl. Endocrinol. Metab. 2005. V. 15. № 6. P. 3197–3201. https://doi.org/10.1161/01.cir.92.6.1387
Караулова Ю.Л., Павлова А.В. Моисеев В.С. и др. Изучение клинико-генетических детерминант гипертрофии левого желудочка у больных артериальной гипертонией и гипертрофической кардиомиопатией // Практикующий врач. 2006. Т. 1. № 2 С. 58–63.
Бимбаев А.Б.-Ж., Баирова Т.А., Шадрина Н.А. Хойкова О.У. Системная гипертензия при гломерулонефрите у детей разных этнических групп и ее взаимосвязь с полиморфизмом гена ангиотензинконвертирующего фермента // Бюлл. ВСНЦ СО РАМН. 2005. № 5(43). С. 128–133.
Гончарова Л.Н., Сергутова Н.П., Кузовенкова О.Н. и др. Показатели центральной гемодинамики и полиморфизм генов ренин-ангиотензивной системы у больных артериальной гипертензией в республике Мордовия // Мед. вестник Башкортостана. 2011. Т. 6. № 2. С. 357–361.
Ichihara S., Yamada Y., Fujimura T. et al. Association of a polymorphism of the endothelial constitutive nitric oxide synthase gene with myocardial infarction in the Japanese population // Am. J. Cardiol. 1998. V. 1. № 1. P. 3–6. https://doi.org/10.1016/s0002-9149(97)10806-2
Ignarro L.J. Biological actions and properties of endothelium-derived nitric oxide formed and released from artery and vein // Circ. Res. 1989. V. 65. № 1. P. 1–21. https://doi.org/10.1161/01.res.65.1.1
Баирова Т.А., Долгих В.В., Бимбаев А.Б.-Ж. и др. Взаимосвязь полиморфизма гена эндотелиальной синтазы окиси азота и эссециальной артериальной гипертензии в популяциях Восточной Сибири // Бюл. Восточно-Сибирского науч. центра СО РАМН. 2007. № 3(65). С. 64–65.
Granath B., Taylor R.R., van Bockxmeer F.M., Mamotte C.D. Lack of evidence for association between endothelial nitric oxide synthase gene polymorphisms and coronary artery disease in the Australian Caucasian population // J. Cardiovasc. Risk. 2001. V. 8. № 4. P. 235–241. https://doi.org/10.1177/174182670100800408
Milutinović A., Hruskovicova H. The eNOS gene polymorphism does not have a major impact on lipid parameters and premature coronary artery disease in Slovene men (Caucasians) // Folia Biol. (Praha). 2005. V. 51. № 2. P. 47–49.
Kim I.J., Bae J., Lim S.W. et al. Influence of endothelial nitric oxide synthase gene polymorphisms (786TNC, 4a4b, 894GNT) in Korean patients with coronary artery disease // Thrombosis Res. 2007. V. 119. № 5. P. 579–585. https://doi.org/10.1016/j.thromres.2006.06.005
Suzuki N., Matsunaga T., Nagasumi K. et al. Alpha(2B)-adrenergic receptor deletion polymorphism associates with autonomic nervous system activity in young healthy Japanese // J. Clin. Endocrinol. Metab. 2003. V. 88. № 3. P. 1184–1187. https://doi.org/10.1210/jc.2002-021190
Snapir A., Heinonen P., Tuomainen T.P. et al. An insertion/deletion polymorphism in the alpha2B-adrenergic receptor gene is a novel genetic risk factor for acute coronary events // J. Am. Coll. Cardiol. 2001. V. 37. № 6. P. 1516–1522. https://doi.org/10.1016/s0735-1097(01)01201-3
Weisberg I., Tran P., Christensen B. et al. A second genetic polymorphism in methylenetetrahydrofolate reductase (MTHFR) associated with decreased enzyme activity // Mol. Genet. Metab. 1998. V. 64. № 3. P. 169–172. https://doi.org/10.1006/mgme.1998.2714
Добролюбов А.С., Липин М.А., Поляков А.В., Фетисова И.Н. Полиморфизм генов фолатного обмена и болезни человека // Вестн. новых мед. технологий. 2006. Т. 13. № 4. С. 71–73.
Botto L.D., Yang Q. 5,10-Methylenetetrahydrofolate reductase gene variants and congenital anomalies: A HuGE review // Am. J. Epidemiol. 2000. V. 151. № 9. P. 862–877. https://doi.org/10.1093/oxfordjournals.aje.a010290
Калашникова Е.А., Кокаровцева С.Н. Ассоциация наследственных факторов тромбофилии с невынашиванием беременности у женщин в русской популяции // Мед. генетика. 2005. Т. 4. № 8. С. 386–391.
Al-Shahrani H., Al-Dabbagh N., Al-Dohayan N. et al. Association of the methylenetetrahydrofolate reductase (MTHFR) C677T polymorphism with primary glaucoma in Saudi population // BMC Ophthalmol. 2016. V. 16. № 1. P. 156–163. https://doi.org/10.1186/s12886-016-0337-7
Gupta S., Bhaskar P. K., Bhardwaj R. MTHFR C677T predisposes to POAG but not to PACG in a North Indian population: A case control study // PLoS One. 2014. V. 9. № 7. e103063. https://doi.org/10.1371/journal.pone.0103063
Micheal S., Qamar R., Akhtar F. et al. MTHFR gene C677T and A1298C polymorphisms and homocysteine levels in primary open angle and primary closed angle glaucoma // Mol. Vis. 2009. № 15. P. 2268–2278.
Nilforoushan N., Aghapour S., Raoofian R. et al. Lack of association between the C677T single nucleotide polymorphism of the MTHFR gene and glaucoma in Iranian patients // Acta Med. Iranica. 2012. V. 50. № 3. P. 208–212.
Zhang L., Chen B. Correlation between MTHFR polymorphisms and glaucoma: A meta-analysis // Mol. Genet. Genomic Med. 2019. V. e5387. № 4. P. 1–7. https://doi.org/10.1002/mgg3.538
Lou L., Wang J., Wang J. Genetic associations between transcription factor 7 Like 2 rs7903146 polymorphism and type 2 diabetes mellitus: A meta-analysis of 115,809 subjects // Diabetology & Metabolic Syndrome. 2019. V. 11. № 56. P. 3. https://doi.org/10.1186/s13098-019-0451-9
Самойленко В.А., Петрова Н.В., Бабаджанова Г.Ю. и др. Роль гена-модификатора TCF7L2 в возникновении диабета у взрослых больных муковисцидозом // Пульмонология. 2014. № 2. С. 33–39.
Авзалетдинова Д.Ш., Шарипова Л.Ф., Кочетова О.В. и др. Анализ ассоциаций полиморфного маркера rs7903146 гена TCF7L2 с сахарным диабетом 2 типа в татарской этнической группе, проживающей в Башкортостане // Сахарный диабет. 2016. Т. 19. № 2. С. 119–124.
Naik M.U., Caplan J.L., Naik U.P. Junctional adhesion molecule-A suppresses platelet integrin αIIbβ3 signaling by recruiting Csk to the integrin-c-Src complex // Blood. 2014. V. 123. № 9. P. 1393–1402. https://doi.org/10.1182/blood-2013-04-496232
Tintori C., Fallacara A.L., Radi M. et al. Combining X‑ray crystallography and molecular modeling toward the optimization of pyrazolo[3,4-d]pyrimidines as potent c-Src inhibitors active in vivo against neuroblastoma // J. Med. Chem. 2015. V. 58. № 1. P. 347–361. https://doi.org/10.1021/jm5013159
Meng Y., Roux B. Locking the active conformation of c-Src kinase through the phosphorylation of the activation loop // J. Mol. Biol. 2014. V. 426. № 2. P. 423–435. https://doi.org/10.1016/j.jmb.2013.10.001
Корнеева Е.В., Воевода М.И., Семаев С.Е., Максимов В.Н. Ассоциация rs1378942 гена CSK с артериальной гипертензией у молодых жителей с метаболическим синдромом, проживающих в северных условиях // Совр. пробл. науки и образования. 2019. № 2. URL: http://science-education.ru/ru/article/view?id=28646
Платунова И.М., Никулина С.Ю., Чернова А.А. и др. Роль гена CSK в развитии ишемического инсульта // Совр. пробл. науки и образования. 2016. № 6. URL: http://www.science-education.ru/ru/article/view
Park Y.M., Kwock C.K., Kim K. et al. Interaction between single nucleotide polymorphism and Urinary Sodium, Potassium, and Sodium-Potassium ratio on the risk of hypertension in Korean // Nutrients. 2017. V. 9. № 3. P. 235–249. https://doi.org/10.3390/nu9030235
Дополнительные материалы отсутствуют.


