Генетика, 2022, T. 58, № 1, стр. 35-44
Генетическое разнообразие четырех видов рода Astragalus L. секции Cenantrum Bunge (Fabaceae) в азиатской части России по данным изменчивости trnL–trnF хлоропластной ДНК и изоферментных маркеров
О. С. Корчагина 1, *, Д. А. Кривенко 2, **, А. Ю. Беляев 3
1 Сахалинский филиал ботанического сада-института Дальневосточного отделения
Российской академии наук
693023 Южно-Сахалинск, Россия
2 Сибирский институт физиологии и биохимии растений Сибирского отделения
Российской академии наук
664033 Иркутск, Россия
3 Институт экологии растений и животных Уральского отделения
Российской академии наук
620144 Екатеринбург, Россия
* E-mail: dymshakova@rambler.ru
** E-mail: krivenko.irk@gmail.com
Поступила в редакцию 24.03.2021
После доработки 14.07.2021
Принята к публикации 09.08.2021
- EDN: SYYYPQ
- DOI: 10.31857/S0016675822010052
Аннотация
Изучено генетическое разнообразие и филогенетические связи четырех видов рода Astragalus секции Cenantrum: A. frigidus, A. mongholicus, A. penduliflorus и A. sericeocanus в азиатской части России по данным изменчивости trnL–trnF хпДНК и изоферментных маркеров. Для каждого вида выявлены специфичные наборы хлоропластных гаплотипов, аллелей и частот изоферментов. По данным хпДНК изученные популяции характеризуются низким гаплотипическим (h изменяется от 0.095 до 0.333) и нуклеотидным (π изменяется от 0.00019 до 0.00378) разнообразием. Большинство популяций (20 из 27) были мономорфными. По данным аллозимного анализа показатели полиморфизма для трех видов (исключение составляет мономорфная популяция A. penduliflorus) имеют довольно высокие значения. Для вида в среднем полиморфность (P95) варьирует от 55.5 до 85.5%, число аллелей на локус (А) – от 1.8 до 2.7, наблюдаемая гетерозиготность (Ho) – от 0.134 до 0.215. Географическая приуроченность гаплотипов и полиморфные популяции A. frigidus и A. mongholicus выявлены на Алтае и в Байкальской Сибири. Эти местонахождения располагаются, вероятно, на месте пересечения миграционных путей или рефугиумов.
Род Astragalus L. – крупнейший не только в семействе Fabaceae, но и среди покрытосеменных растений, насчитывает около 2900 видов, из которых в Старом Свете встречаются более 2400 видов, в Новом Свете – около 500 видов [1, 2]. В системе рода выделяют два подрода: Astragalus – растения, опушенные простыми волосками, и Cercidothrix Bunge – в опушении растений присутствуют как простые, так и двуконечные (мальпигиевые) волоски [3], и примерно 200 секций [1].
Секция Cenantrum Bunge относится к типовому подроду Astragalus. Она характеризуется примитивностью морфологических признаков: многолетние поликарпические растения с ассимилирующими (листовыми) прилистниками, парноперистыми черешковыми листьями, многоцветковыми кистевидными соцветиями, желтыми цветками и одногнездными бобами, – занимая, таким образом, базальное положение в системе рода [3, 4]. Эта секция объединяет в своем составе около 40 видов, обитающих преимущественно в Азии, с центром биоразнообразия в восточной ее части (Корейский полуостров, Японские острова, восточная часть Китая). На территории России, в том числе ее азиатской части, известно восемь видов секции Cenantrum [5, 6].
В настоящее исследование включены четыре вида: A. frigidus (L.) A. Gray – европейско-североазиатский вид, A. mongholicus Bunge – вид с ареалом на юге Сибири и Дальнего Востока (Россия, Китай, Япония), северной части Центральной Азии (Казахстан, Монголия), A. penduliflorus Lam. – северо- и центральноевропейский вид с единственным местонахождением на Среднем Урале и A. sericeocanus Gontsch. – эндемик побережий оз. Байкал, внесен в Красную книгу Республики Бурятия, с присвоением третьей (NT) категории статуса редкости – редкий вид [7]. Трактовка видов и объем секции Cenantrum приняты нами в соответствии с последней монографической обработкой рода Astragalus Старого Света [5].
С точки зрения современной систематики, в частности для рода Astragalus, необходимо привлекать молекулярно-генетические исследования (полиморфизм изоферментов и хлоропластной ДНК (хпДНК)), также они необходимы для познания эволюционной истории, организации охраны генофонда редких и эндемичных видов, идентификации сырья лекарственных растений [8–11].
Цель настоящего исследования – охарактеризовать генетическое разнообразие, структуру популяций и степень генетического родства четырех видов рода Astragalus секции Cenantrum (A. frigidus, A. mongholicus, A. penduliflorus и A. sericeocanus) азиатской части России по данным анализов изменчивости изоферментов и хпДНК.
МАТЕРИАЛЫ И МЕТОДЫ
Материалом для изучения изменчивости хпДНК были 437 растений, для изоферментного анализа – 291. Географическое положение исследованных популяций видов рода Astragalus секции Cenantrum представлено на рис. 1 и в табл. 1.
Рис. 1.
Географическое распределение выявленных 15 гаплотипов хпДНК для 27 исследованных популяций четырех видов Astragalus секции Cenantrum.
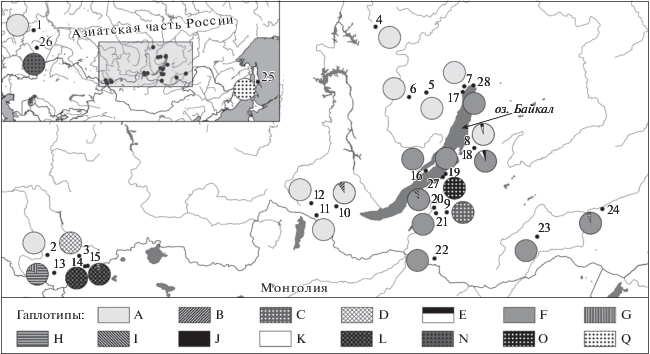
Таблица 1.
Генетический полиморфизм у видов Astragalus секции Cenantrum по данным хпДНК
| Популяция | Координаты | № ваучера | nH | h | π | |
|---|---|---|---|---|---|---|
| с.ш. | в.д. | |||||
| A. frigidus | ||||||
| 1. Ивдель | 60°46′ | 60°15′ | IRK36800 | 27 A | – | – |
| 2. Белый Бом | 50°18′ | 86°58′ | IRK36495 | 14 A | – | – |
| 3. Саратам | 50°16′ | 88°41′ | IRK45181 | 7 D | – | – |
| 4. Игирма | 57°30′ | 104°33′ | IRK24685 | 17 A | – | – |
| 5. Харахикта | 55°33′ | 107°16′ | IRK55549 | 26 A | – | – |
| 6. Орлинга | 55°25′ | 106°20′ | IRK46601 | 20 A | – | – |
| 7. Тыя | 55°45′ | 109°18′ | IRK45554 | 17 A | – | – |
| 8. Улюн-1 | 53°50′ | 109°50′ | IRK45537 | 20 A, 1 E | 0.095 (0.084) | 0.00034 |
| 9. Челутай | 51°45′ | 108°21′ | IRK55479 | 3 С | – | – |
| 10. Охор-Шибирь | 51°56′ | 102°26′ | IRK32809 | 9 A, 1 B | 0.200 (0.154) | – |
| 11. Мойготы | 51°39′ | 101°23′ | IRK29595 | 15 A | – | – |
| 12. Китой | 52°03′ | 101°06′ | IRK16611 | 16 A | – | – |
| Общие показатели для вида | 181 A, 1 B, 3 C, 7 D, 1 E | 0.120 (0.032) | 0.00022 | |||
| A. mongholicus | ||||||
| 13. Джазатор | 49°41′ | 87°20′ | IRK39812 | 1 G, 5 H | 0.333 (0.215) | 0.00060 |
| 14. Кокоря | 49°54′ | 88°59′ | IRK39813 | 12 L | – | – |
| 15. Чуйская степь | 49°55′ | 89°08′ | IRK32832 | 18 L, 1 H | 0.105 (0.092) | 0.00019 |
| 16. Шара-Нур | 53°06′ | 107°14′ | IRK28971 | 24 F | – | – |
| 17. Северобайкальск | 55°34′ | 109°13′ | IRK23043 | 10 F | – | – |
| 18. Улюн-2 | 53°50′ | 109°50′ | IRK55583 | 16 F, 1 J, 1 K | 0.216 (0.124) | 0.00378 |
| 19. Горячинск | 53°00′ | 108°18′ | IRK32252 | 24 F | – | – |
| 20. Улан-Удэ | 51°54′ | 107°41′ | IRK41767 | 25 F, 2 I | 0.143 (0.086) | 0.00077 |
| 21. Николаевский | 51°43′ | 107°47′ | IRK32253 | 16 F | – | – |
| 22. Малая Кудара | 50°11′ | 107°42′ | IRK39814 | 5 F | – | – |
| 23. Ара-Иля | 50°55′ | 113°12′ | IRK16604 | 20 F | – | – |
| 24. Калиновка | 51°51′ | 116°41′ | IRK23897 | 15 F, 1 H | 0.125 (0.106) | 0.00023 |
| 25. Известковый | 49°57′ | 143°22′ | VLA197661 | 6 Q | – | – |
| Общие показатели для вида | 155 F, 1 G, 7 H, 2 I, 1 J, 1 K, 30 L, 6 Q | 0.395 (0.040) | 0.00102 | |||
| A. penduliflorus | ||||||
| 26. Голендухино | 57°27′ | 61°25′ | IRK32794 | 20 N | – | – |
| A. sericeocanus | ||||||
| 27. Турка | 52°54′ | 108°09′ | IRK 23490 | 21 O | – | – |
Геномная ДНК экстрагирована СТАВ-методом [12] из проростков или высушенных в силикагеле листьев. Для амплификации и секвенирования фрагмента trnL–trnF хпДНК использованы праймеры “c” и “d” [13]. Реакцию ПЦР проводили согласно протоколу, описанному в работе Е.В. Хантемировой с соавт. [14]. Секвенирование ПЦР-продукта проведено в обоих направлениях при условиях, описанных J. Shaw et al. [15], с использованием набора BigDye v 3.1 на генетическом анализаторе 3130 DNA Analyzer (Applied Biosystems, США). Нуклеотидные последовательности редактировали в программе BioEdit v 7.0.5.3 [16].
Для анализа популяционной изменчивости был проведен рестриктный анализ с ферментом TaqI, согласно протоколу производителя. При наличии делеций разной длины, а также точечных замен, при которых появлялся/исчезал сайт рестрикции, на электрофореграмме присутствовали фрагменты разной длины или отсутствовали совсем. Для визуализации рестриктных продуктов использовали электрофорез в вертикальной камере S2 (Life Technologies) в 6%-ном денатурирующем полиакриламидном геле и серебряное окрашивание [17]. По совокупности фрагментов разной длины нуклеотидные последовательности относили к тому или иному гаплотипу.
Для выявления генеалогических связей между гаплотипами был использован метод Median-Joining (MJ, программа Network 5.0.1.1 [18]) как наиболее подходящий для исследований на внутривидовом уровне и в группах близкородственных видов. Каждую делецию, вставку или замену кодировали как единичное мутационное событие, обозначая 1/0 как присутствующие/отсутствующие соответственно.
Популяционно-генетический анализ выполнен в программах Arlequin v 3.5 [19] и DnaSP v 6.12.03 [20]. Были рассчитаны следующие параметры: число гаплотипов (nH), гаплотипическое разнообразие (h), нуклеотидное разнообразие (π), генный поток (Nm) и дивергенция нуклеотидных последовательностей (Dxy). С помощью анализа молекулярной дисперсии (AMOVA, Arlequin v 3.5) оценивали распределение генетической изменчивости между популяциями и между видами.
Для определения филогенетической структуры сравнивали индексы NST и GST в программе Permut v 2.0 [21], где показатель GST отражает только частоту гаплотипов в популяции, тогда как NST учитывает родство гаплотипов, а филогенетическая структура определяется при NST значимо (p < 0.05) большем, чем GST [22].
Для реконструкции демографической истории исследуемых популяций использовали показатели отклонения от стандартной нейтральной модели эволюции: тесты F. Tajima (D) [23] и Y.-X. Fu (FS) [24]. Для выявления популяционно-генетических изменений во времени применяли анализ распределения парных различий (Arlequin v 3.5), связывающий число различий между гаплотипами с их частотой, используя значения суммы квадратов отклонений (SSD) между наблюдаемым и ожидаемым значениями распределения и индекс r как тест на уровень значимости. Доверительный 95%-ный интервал демографических параметров оценивали при 1000 повторах.
Протокол для исследования изменчивости изоферментов представлен в нашей ранней работе [25].
РЕЗУЛЬТАТЫ
Анализ изменчивости хпДНК
Для 437 образцов четырех видов рода Astragalus получены нуклеотидные последовательности части фрагмента некодирующей области trnL–trnF (554–563 пн). В сиквенсах этого региона выявлены 24 полиморфных сайта, из них 11 информативны согласно методу максимальной экономии. На основе полученных данных выявлено 15 гаплотипов (А–Q), последовательности которых депонированы в GenBank под номерами доступа MW594292–MW594306.
Для каждого вида характерен специфичный набор гаплотипов (рис. 1, табл. 1), а их генеалогические связи представлены на рис. 2. Наиболее часто встречающийся гаплотип для A. frigidus – A (выявлен у 93.8% образцов), гаплотипы C и D обнаружены у 1.6 и 3.6% растений соответственно, гаплотипы B и E – единичные (менее 1%). Часто встречающийся гаплотип для A. mongholicus – F (обнаружен у 76.4% образцов), алтайский гаплотип L выявлен у 14.8% растений, гаплотипы H и Q обнаружены у 3.5 и 3.0% образцов соответственно, остальные гаплотипы – менее 1%. Для A. penduliflorus и A. sericeocanus характерны специфичные гаплотипы N и O соответственно. На популяционном уровне показатели гаплотипического и нуклеотидного разнообразия были низкими, для A. mongholicus показатели гаплотипического и нуклеотидного разнообразия выше, чем для A. frigidus (табл. 1). Генетические взаимоотношения гаплотипов остались неясными из-за наличия в медианной сети альтернативных связей, свидетельствующих о гомоплазии (рис. 2).
Рис. 2.
Филогенетическая сеть гаплотипов хпДНК. Размер кружка соответствует частоте встречаемости гаплотипа, поперечные штрихи на связывающих линиях соответствуют количеству мутационных событий. Черный кружок – предполагаемый промежуточный гаплотип.
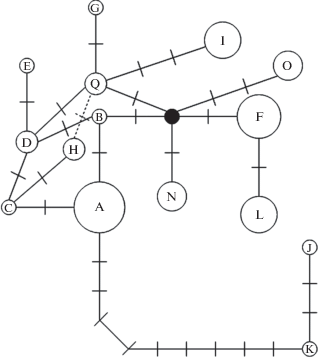
Согласно результатам AMOVA, у A. frigidus и A. mongholicus основная доля всей генетической изменчивости приходится на межвидовые различия – 84.17% (p < 0.01), между популяциями внутри видов и внутри популяций доля генетической изменчивости составляет 9.82 и 6.01% соответственно (p < 0.01).
Дивергенция нуклеотидных последовательностей (Dxy, среднее число нуклеотидных замен на один сайт) между популяциями A. frigidus составила 0.0002 – между “Улюн-1” и остальными популяциями, 0.0018 – между парами, образованными каждой из популяций “Саратам” и “Челутай” с остальными популяциями. Между остальными популяциями A. frigidus Dxy отсутствует. Между популяциями A. mongholicus показатель Dxy варьировал от 0.0000 до 0.0024 (между “Улюн-2” и “Улан Удэ”). В табл. 2 приведены значения дивергенции нуклеотидных последовательностей между исследуемыми видами. Наибольшие значения Dxy определены между парами, образованными каждым из видов A. frigidus и A. mongholicus с A. sericeocanus, наименьшее – между A. frigidus и A. penduliflorus.
Таблица 2.
Нуклеотидная дивергенция (среднее число нуклеотидных замен на один сайт) между видами Astragalus секции Cenantrum по данным хпДНК
| Вид | A. frigidus | A. mongholicus | A. penduliflorus | A. sericeocanus |
|---|---|---|---|---|
| A. frigidus | – | 0.00359 | 0.00193 | 0.00550 |
| A. mongholicus | – | 0.00201 | 0.00555 | |
| A. penduliflorus | – | 0.00361 | ||
| A. sericeocanus | – |
Показатели генетической дифференциации популяций высоки (GST = 0.942; NST = 0.922), значения NST < GST, что указывает на отсутствие филогенетической структуры [21, 22].
Результаты тестов на нейтральность и анализов несоответствия представлены в табл. 3. Значения теста D для всех популяций были отрицательными, для A. frigidus “Улюн-1”, A. mongholicus “Чуйская степь” и “Калиновка” показатели статистически значимые (p < 0.05). Показатели теста FS, кроме A. frigidus “Охор-Шибирь”, – положительные (p > 0.05). Анализ демографических параметров выявил, что все полиморфные популяции находятся в стабильном состоянии (интервалы Θ0 и Θ1 перекрываются).
Таблица 3.
Значения тестов на нейтральность и анализов несоответствия распределения в популяциях видов Astragalus секции Cenantrum по данным хпДНК
| Вид, популяции |
Тест на нейтральность | Анализ несоответствия распределения | |||||
|---|---|---|---|---|---|---|---|
| D | FS | τ (CI) | Θ0 (CI) | Θ1 (CI) | SSD | r | |
| A. frigidus | –0.960 | –3.614* | 3.000 (0.445–3.000) | 0.000 (0.000–0.000) | 0.103 (0.000–∞) | 0.006 | 0.709 |
| 8. Улюн-1 | –1.727* | 0.494 | 3.000 (0.496–3.000) | 0.000 (0.000–0.000) | 0.055 (0.000–∞) | 0.013* | 0.837 |
| 10. Охор-Шибирь | –1.112 | –0.339 | 2.930 (0.000–87.930) | 0.900 (0.000–8.387) | 3.600 (0.136–∞) | 0.331 | 0.400 |
| A. mongholicus | –1.392* | –0.398 | 3.439 (0.000–5.918) | 0.004 (0.000–0.030) | 1.607 (0.190–∞) | 0.021 | 0.098 |
| 13. Джазатор | –1.132 | 0.952 | 2.982 (0.000–87.982) | 0.900 (0.00–6.589) | 3.600 (0.292–∞) | 0.260 | 0.667 |
| 15. Чуйская степь | –1.861* | 1.055 | 3.000 (0.449–3.000) | 0.000 (0.000–0.002) | 0.061 (0.000–∞) | 0.016 | 0.823 |
| 18. Улюн-2 | –1.244 | 3.547 | 3.000 (0.467–3.000) | 0.000 (0.000–0.005) | 0.149 (0.000–∞) | 0.042* | 0.659 |
| 20. Улан-Удэ | –1.192 | 1.842 | 3.000 (0.463–3.000) | 0.000 (0.000–0.000) | 0.087 (0.000–∞) | 0.030* | 0.776 |
| 24. Калиновка | –1696* | 0.769 | 3.000 (0.611–3.000) | 0.000 (0.000–0.000) | 0.076 (0.000–∞) | 0.022* | 0.797 |
Примечание. D – тест Tajima, FS – тест Fu, τ – время экспансии популяции, Θ0 и Θ1 – начальное и конечное значение размера популяции соответственно, SSD – сумма квадратных отклонений между наблюдаемым и ожидаемым значениями распределения, r – индекс несоответствия, * p < 0.05, СI – 95%-ный интервал значимости.
Анализ изменчивости изоферментных маркеров
Значения основных показателей генетического полиморфизма в исследованных популяциях приведены в табл. 4. Популяция A. penduliflorus “Голендухино” является мономорфной. Среднее число аллелей на локус для полиморфных популяций варьирует от 1.3 до 3.0, эффективное число аллелей – от 1.04 до 1.49. Доля полиморфных локусов (P95) изменяется от 22.2 до 88.9%. Средняя наблюдаемая гетерозиготность (Ho) находится в диапазоне от 0.107 до 0.261 и почти во всех популяциях ниже, чем значения ожидаемой гетерозиготности (He), варьирующей от 0.115 до 0.331. Отличие значений Ho и He значимое (р < 0.05), за исключением четырех популяций A. frigidus (“Игирма”, “Орлинга”, “Тыя”, “Харахикта”) и одной популяции A. mongholicus (“Николаевский”).
Таблица 4.
Значения показателей генетического полиморфизма в популяциях видов Astragalus секции Cenantrum по данным аллозимного анализа
| Популяция | Ni | Nl | A | Nе | P95, % | Ho | He |
|---|---|---|---|---|---|---|---|
| A. frigidus | |||||||
| 1. Ивдель | 19 | 19.0 (0.0) | 1.3 (0.2) | 1.13 | 22.2 | 0.111 (0.083) | 0.115 (0.080) |
| 2. Игирма | 30 | 30.0 (0.0) | 2.2 (0.3) | 1.30 | 77.8 | 0.200 (0.072) | 0.232 (0.071) |
| 5. Харахикта | 25 | 24.0 (1.0) | 1.7 (0.3) | 1.20 | 44.4 | 0.136 (0.065) | 0.166 (0.068) |
| 6. Орлинга | 16 | 15.6 (0.3) | 1.9 (0.3) | 1.21 | 66.7 | 0.140 (0.040) | 0.175 (0.050) |
| 7. Тыя | 13 | 12.4 (0.6) | 1.4 (0.2) | 1.04 | 33.3 | 0.026 (0.018) | 0.042 (0.022) |
| 10 Охор-Шибирь | 9 | 8.9 (0.1) | 1.9 (0.3) | 1.23 | 55.6 | 0.123 (0.054) | 0.186 (0.069) |
| 11. Мойготы | 30 | 29.3 (0.2) | 2.2 (0.2) | 1.25 | 88.9 | 0.202 (0.072) | 0.201 (0.057) |
| Среднее | 20.3 | 19.9 (0.3) | 1.8 (0.3) | 1.09 | 55.5 | 0.134 (0.058) | 0.160 (0.060) |
| A. mongholicus | |||||||
| 16. Шара-Нур | 38 | 33.2 (0.8) | 2.8 (0.4) | 1.32 | 88.9 | 0.166 (0.043) | 0.244 (0.067) |
| 19. Горячинск | 20 | 20.0 (0.0) | 3.0 (0.4) | 1.49 | 88.9 | 0.217 (0.040) | 0.331 (0.078) |
| 21. Николаевский | 17 | 16.9 (0.1) | 2.3 (0.3) | 1.40 | 77.8 | 0.261 (0.073) | 0.284 (0.077) |
| Среднее | 24.7 | 23.4 (0.3) | 2.7 (0.4) | 1.40 | 85.2 | 0.215 (0.052) | 0.286 (0.074) |
| A. penduliflorus | |||||||
| 26. Голендухино | 18 | 18.0 (0.0) | 1.0 (0.0) | 1.00 | 0.0 | 0.000 (0.000) | 0.000 (0.000) |
| A. sericeocanus | |||||||
| 27. Турка | 37 | 31.1 (1.2) | 2.7 (0.4) | 1.38 | 80.0 | 0.213 (0.069) | 0.274 (0.071) |
| 28. Ярки | 26 | 26.0 (0.0) | 2.0 (0.4) | 1.20 | 44.4 | 0.107 (0.054) | 0.169 (0.086) |
| Среднее | 31.5 | 28.6 (0.6) | 2.4 (0.4) | 1.29 | 62.2 | 0.160 (0.062) | 0.222 (0.079) |
Примечание. Ni – число исследованных растений, Nl – средний размер выборки на локус, A – среднее число аллелей на локус, Nе – эффективное число аллелей, P95 – полиморфизм с учетом 95%-ного критерия, Ho – средняя наблюдаемая гетерозиготность, He – средняя ожидаемая гетерозиготность. В скобках даны значения стандартной ошибки среднего.
Анализ генетической структуры, выполненный с помощью индексов фиксации Райта, для трех популяций A. mongholicus показал, что каждая особь обнаруживает 25.1%-ный дефицит гетерозигот относительно популяции и 29.0%-ный относительно вида в целом. У A. mongholicus на межпопуляционную изменчивость приходится 5.2% от выявленной генетической изменчивости и 94.8% – на внутрипопуляционную. Этот же анализ для семи популяций A. frigidus показал, что каждая особь обнаруживает 10.0%-ный дефицит гетерозигот относительно популяции и 37.0%-ный относительно вида в целом. У A. frigidus на межпопуляционную изменчивость приходится 30% от выявленной генетической изменчивости и 70% – на внутрипопуляционную.
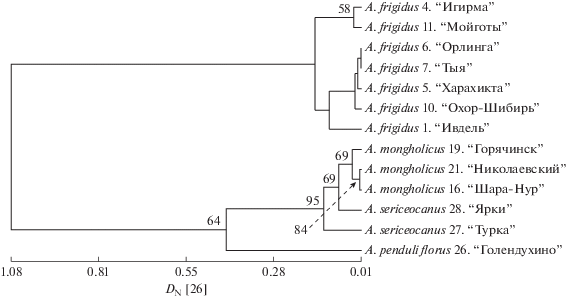
Выявлена большая генетическая дистанция (DN = 1.08) между A. frigidus и остальными изученными видами (рис. 3). Внутри клады A. frigidus выявлены две субклады (DN = 0.15): предбайкальская, объединяющая популяции “Игирма” и “Мойготы”, расположенные на значительном удалении от оз. Байкал, и прибайкальская, объединяющая наиболее приближенные к озеру популяции “Орлинга”, “Тыя”, “Харахикта” и “Охор-Шибирь”, а также включающая краевую уральскую популяцию “Ивдель” (DN = 0.11). Внутри субклад значения DN варьируют от 0.01 до 0.03. Внутри клады A. mongholicus–A. penduliflorus–A. sericeocanus также выявлены две субклады (DN = 0.42): первая объединяет популяции A. mongholicus и эндемичный вид A. sericeocanus, вторая представлена популяцией A. penduliflorus. Следует отметить, что A. sericeocanus не образует самостоятельной ветви – DN = 0.07 между популяцией “Ярки” и популяциями A. mongholicus, DN = 0.12 между популяцией “Турка” и остальными популяциями субклады.
Рис. 3.
Дендрограмма сходства популяций видов Astragalus секции Cenantrum, построенная на основе генетических дистанций DN [26]. Цифры в узлах ветвления – значения бутстрепа.
ОБСУЖДЕНИЕ
Секция Cenantrum – монофилетичная группа, для которой, как и для рода Astragalus в целом, интрогрессия и межвидовая гибридизация считаются крайне редкими событиями [9]. Однако, несмотря на это, ее представители характеризуются морфологическим полиморфизмом, имеют обширные ареалы и разнообразный спектр экологических условий обитания.
По результатам анализа изменчивости хпДНК для A. frigidus выявлена однородность исследуемых популяций, большая часть которых мономорфна. Полиморфные популяции “Улюн-1” и “Охор-Шибирь” обнаружены в области, близкой к центральной части ареала вида – в Прибайкалье. Анализ показателей демографических данных указывает на то, что они находятся в стабильном состоянии, для последней возможен популяционный рост. Показатели теста на нейтральность для популяции “Улюн-1” свидетельствуют о наличии распространенных гаплотипов и относительно недавнем прохождении популяции через “бутылочное горлышко”. В пользу последнего свидетельствует ее местонахождение в поврежденном лесу среди валунов на берегу горной реки. Вероятно, она произошла от единичных особей, сохранившихся после природного разрушительного события. Показатели нуклеотидной дивергенции между популяциями A. frigidus низкие (до 0.00180), хотя такие же низкие значения (0.00193) выявлены между A. frigidus и A. penduliflorus. Показатели генного потока Nm = 0.081, 0.030 и 0.010 между A. frigidus и A. monhgolicus, A. penduliflorus и A. sericeocanus соответственно, указывая на отсутствие заметного обмена генами между ними в настоящее время [27].
Анализ изоферментных данных выявил генетическую дистанцию между A. frigidus и остальными видами секции – 1.08, которая, согласно шкале генетических дистанций, разработанной для растений, соответствует видам разных подсекций (0.12–1.2) [28].
Разнообразные по морфологическим признакам особи A. mongholicus имеются как в генетически мономорфных, так и в полиморфных по хпДНК популяциях. По показателям тестов на нейтральность полиморфные популяции A. mongholicus содержат редкие гаплотипы при равновесии “мутация–дрейф”. Показатели Nm = 0.130 и 0.040 между A. mongholicus и A. penduliflorus, A. sericeocanus соответственно. Для популяций “Чуйская степь” и “Калиновка” вероятно их прохождение через “бутылочное горлышко”. Первая представляет собой локальную популяцию в небольшом понижении рельефа среди степи, вероятно возникшую здесь от единичных особей. Вторая находится близ населенного пункта на опушке леса у автомобильной дороги и вероятно в прошлом подвергалась чрезмерному антропогенному воздействию [29].
Показатели гаплотипического и нуклеотидного разнообразия для видов A. frigidus и A. mongholicus ниже, чем, например, для A. onobrychus [30], что возможно связано с использованием только одного и относительно короткого фрагмента хпДНК. Напротив, показатели изменчивости аллозимов A. frigidus и A. mongholicus выше по сравнению с другими видами рода Astragalus, как и для травянистых бобовых в целом, что уже отмечалось нами ранее [25]. Вероятно, это объясняется их нахождением вблизи центров ареалов видов и небольшим количеством исследуемых локусов. Наиболее низкие значения показателей генетического полиморфизма по изоферментным маркерам получены для популяции A. frigidus “Ивдель”, что обычно характерно для краевых популяций, как правило находящихся под влиянием дрейфа генов и изоляции в течение длительного времени [31].
Эндемик побережий оз. Байкал A. sericeocanus резко отличается от A. mongholicus и A. penduliflorus по морфологическим, экологическим и, как было установлено, генетическим признакам. Растения этого вида имеют характерное серебристо-мохнатое опушение, местообитания приурочены к перевиваемым пескам. Для вида выявлены видоспецифичные признаки: набор изоферментных аллелей и их частот, а также гаплотип O. Значения нуклеотидной дивергенции между A. sericeocanus и остальными видами самые высокие: от 0.00361 до 0.00555. Значения изоферментного полиморфизма высокие для популяции “Турка” и значительно меньше для популяции “Ярки”, что ожидаемо для островной популяции. При этом интересно отметить, что величины показателей полиморфизма даже в этой популяции сопоставимы с таковыми для ряда популяций широкоареального A. frigidus, а в среднем для вида показатели полиморфизма узколокального эндемика A. sericeocanus выше, чем у A. frigidus (табл. 4). Полученные на основе изоферментных маркеров значения генетических дистанций между популяциями A. mongholicus и A. sericeocanus соответствуют значениям для видов одной подсекции (0.032–0.652) [28].
По морфологическим признакам и генетическим показателям A. penduliflorus отличается от других исследованных видов. Для него выявлены специфичный гаплотип N и мономорфность по показателям изменчивости изоферментов и хпДНК. По изоферментным маркерам значение генетической дистанции 0.42 является самой большой для видов клады A. mongholicus–A. penduliflorus–A. sericeocanus, но также соответствует величине дистанции между близкородственными видами одной подсекции [28]. Генетические показатели этой популяции характерны для краевых частей ареала вида. В настоящее время ареал вида разорван [6], его основная часть находится в европейских горных системах (Альпы, Карпаты и Пиренеи) и в Центральной Швеции [5]. Можно предположить, что ранее существовала обширная предковая европейская популяция A. penduliflorus, а сейчас популяция “Голендухино” представляет собой лишь ее фрагмент и является реликтовой.
Обнаружена корреляция между гаплотипами и их географическим положением. Так, для A. mongholicus гаплотип F отмечен только в байкальских популяциях, а гаплотипы G, H и L – в алтайских, гаплотип Q выявлен только на о-ве Сахалин. Для A. frigidus гаплотипы B, C и E характерны для байкальских популяций, а D – для алтайских. Подобная корреляция с использованием ITS, ITS2 и psbA–trnH была выявлена для A. mongholicus в Китае [32]. Вероятно, в прошлом типичные местообитания этих видов неоднократно подвергались изменению, сокращаясь и вновь расширяясь из-за климатических циклов в плейстоцене. Было показано, что такие климатические циклы могли способствовать диверсификации видов растений за счет многократного деления популяций, что привело к аллопатрическому видообразованию [33, 34]. Вероятно, таким же образом в ходе эволюционной дивергенции A. sericeocanus обособился в условиях специфической среды песчаных арен Прибайкалья.
Ранее было показано [35], что для большинства видов покрытосеменных растений присутствие нескольких гаплотипов в популяциях указывает на их расположение в зонах контакта разных материнских линий. Это позволяет проследить пути миграции этих видов и число рефугиумов, а иногда и их предполагаемые местонахождения. Анализируя полученные нами данные, можно предположить, что такие зоны путей миграции и контакта находились к востоку и юго-западу от оз. Байкал и на Алтае.
Выражаем благодарность коллегам А.В. Верхозиной, Е.В. Жмудь, М.А. Полежаевой, Д.В. Санданову, Д.Г. Чимитову и Е.Г. Филиппову за помощь в сборе материала для исследований и при анализе полученных данных.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Zarre Sh., Azani N. Perspectives in taxonomy and phylogeny of the genus Astragalus (Fabaceae): a review // Prog. Biol. Sci. 2013. V. 3. № 1. P. 1–6. https://doi.org/10.22059/PBS.2013.32086
Frodin D.G. History and concepts of big plant genera // Taxon. 2004. V. 53. № 3. P. 753–776. https://doi.org/10.2307/4135449
Podlech D. Revision von Astragalus L. sect. Caprini DC. (Leguminosae) // Mitt. Bot. Staatssamml. München. 1988. V. 25. P. 1–924.
Сытин А.К. Астрагалы (Astragalus L., Fabaceae) Восточной Европы и Кавказа: систематика, география, эволюция: Автореф. дис. … д-ра биол. наук. СПб., 2009. 48 с.
Podlech D., Zarre Sh. A Taxonomic Revision of the Genus Astragalus L. (Leguminosae) in the Old World. Vienna: Naturhistorisches Museum Wien, 2013. V. 1. 822 p.
Беляев А.Ю., Дымшакова О.С., Зимницкая С.А., Князев М.С. Уникальная природная популяция астрагала (Astragalus penduliflorus Lam. s.l.) на Cреднем Урале // Проблемы популяционной биологии: Матер. XII Всеросс. популяционного семинара памяти Николая Васильевича Глотова (1939–2016). Йошкар-Ола: ООО ИПФ “СТРИНГ”, 2017. С. 41–43.
Санданов Д.В., Кривенко Д.А. Астрагал шелковисто-седой – Astragalus sericeocanus Gontsch. // Красная книга Республики Бурятия: редкие и находящиеся под угрозой исчезновения виды животных, растений и грибов. Улан-Удэ: Изд-во БНЦ СО РАН, 2013. С. 518–519.
Karron J.D., Linhart Y.B., Chaulk C.A., Robertson C.A. Genetic structure of populations of geographically restricted and widespread species of Astragalus (Fabaceae) // Amer. J. Bot. 1988. V. 75. № 8. P. 1114–1119. https://doi.org/10.1002/j.1537-2197.1988.tb08823.x
Kazempour Osaloo S., Maassoumi A.A., Murakami N. Molecular systematics of the genus Astragalus L. (Fabaceae): Phylogenetic analyses of nuclear ribosomal DNA internal transcribed spacers and chloroplast gene ndhF sequences // Plant Syst. Evol. 2003. V. 242. № 1–4. P. 1–32. https://doi.org/10.1007/s00606-003-0014-1
Riahi M., Zarre Sh., Maassoumi A.A. et al. Towards a phylogeny for Astragalus section Carpini (Fabaceae) and its allies based on nuclear and plastid DNA sequences // Plant Syst. Evol. 2011. V. 293. № 1–4. P. 119–133. https://doi.org/10.1007/s00606-011-0417-3
Choi I.-S., Kim S.-Y., Choi B.-H. Taxonomic position and genetic differentiation of Korean Astragalus mongholicus Bunge // Korean J. Pl. Taxon. 2013. V. 43. № 1. P. 12–21. https://doi.org/10.11110/kjpt.2013.43.1.12
Devey M.E., Bell J.C., Smith D.N. et al. A genetic linkage map for Pinus radiata based on RFLP, RAPD and microsatellite markers // Theoret. Appl. Genetics. 1996. V. 92. № 6. P. 673–679. https://doi.org/10.1007/BF00226088
Taberlet P., Gielly L., Pautou G., Bouvet J. Universal primers for amplification of three non-coding regions of chloroplast DNA // Plant Mol. Biol. 1991. V. 17. № 5. P. 1105–1109. https://doi.org/10.1007/BF00037152
Хантемирова Е.В., Пименова Е.А., Корчагина О.С. Полиморфизм хлоропластной ДНК и филогеография ольхи зеленой (Alnus alnobetula (Enhr.) K. Koch. s.l.) в Азиатской России // Генетика. 2018. Т. 54. № 1. С. 75–86. https://doi.org/10.7868/S0016675818010058
Shaw J., Lickey E.B., Beck J.T. et al. The tortoise and the hare II: Relative utility of 21 noncoding chloroplast DNA sequences for phylogenetic analysis // Am. J. Bot. 2005. V. 92. № 1. P. 142–166. https://doi.org/10.3732/ajb.92.1.142
Hall T.A. BioEdit: A user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT // Nucl. Acids. Symp. Ser. 1999. V. 41. P. 95–98. https://doi.org/10.14601/Phytopathol_Mediterr-14998u1.29
Bassam B.J., Caetano-Anollés G., Gresshoff P.M. Fast and sensitive silver staining of DNA in polyacrylamide gels // Anal. Biochem. 1991. V. 196. № 1. P. 80–83. https://doi.org/10.1016/0003-2697(91)90120-I
Bandelt H.-J., Forster P., Röhl A. Median-Joining Networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16. № 1. P. 37–48. https://doi.org/10.1093/oxfordjournals.molbev.a026036
Excoffier L., Laval G., Schneider S. Arlequin ver. 3.0: An integrated software package for population genetic data analysis // Evol. Bioinform. Online. 2005. V. 1. P. 47–50.
Rozas J., Sánchez-DelBarrio J.C., Messeguer X., Rozas R. DnaSP, DNA polymorphism analyses by the coalescent and other methods // Bioinform. 2003. V. 19. № 18. P. 2496–2497. https://doi.org/10.1093/bioinformatics/btg359
Petit R.J., Duminil J., Fineschi S. et al. Comparative organization chloroplast, mitochondrial and nuclear diversity in plant populations // Mol. Ecol. 2005. V. 14. № 3. P. 689–701. https://doi.org/10.1111/j.1365-294X.2004.02410.x
Pons O., Petit R.J. Measuring and testing genetic differentiation with ordered versus unordered alleles // Genetics. 1996. V. 144. № 3. P. 1237–1245.
Tajima F. The effect of change in population size on DNA polymorphism // Genetics. 1989. V. 123. № 3. P. 597–601.
Fu Y.-X. Statistical tests of neutrality of mutation against population growth hitchhiking and background selection // Genetics. 1997. V. 147. № 2. P. 915–925.
Дымшакова О.С., Кривенко Д.А., Беляев А.Ю., Верхозина А.В. Генетическая дифференциация трех видов рода Astragalus L. секции Сenantrum Bunge (Fabaceae) // Генетика. 2015. Т. 51. № 8. С. 887–894. https://doi.org/10.7868/S0016675815080020
Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals // Genetics. 1978. V. 89. № 3. P. 583–590.
Wright S. Evolution and the Genetics of Populations. Volume 4: Variability within and among Natural Populations. Chicago: Univ. Chicago Press, 1978. 580 p.
Шурхал А.В., Подогас А.В., Животовский Л.А. Уровни генетической дифференциации жестких сосен род Pinus, подрод Pinus, по данным аллозимной изменчивости // Генетика. 1993. Т. 29. № 1. С. 77–90.
Forman R.T.T., Alexander L.E. Roads and their major ecological effect // Ann. Rev. Ecol. Syst. 1998. V. 29. P. 207–231. https://doi.org/10.1146/annurev.ecolsys.29.1.207
Plenk K., Willnerb W., Deminaet O.N. et al. Phylogeographic evidence for long-term persistence of the Eurasian steppe plant Astragalus onobrychis in the Pannonian region (eastern Central Europe) // Flora. 2020. V. 264. P. 1–10. https://doi.org/10.1016/j.flora.2020.151555
Тимофеев-Ресовский Н.В., Яблоков А.В., Глотов Н.В. Очерк учения о популяции. М.: Наука, 1973. 277 с.
Li L., Zheng S., Brinckmann J. et al. Chemical and genetic diversity of Astragalus mongholicus grown in different eco-climatic region // PLoS One. 2017. V. 12. № 9. e0184791. https://doi.org/10.1371/journal.pone.0184791
Bargheri A., Maasoumi A.A., Rahiminejad M.R. et al. Molecular phylogeny and divergence time of Astragalus section Hymenostegis: An analysis of rapidly diversifying species group in Fabaceae // Scientific Reports. 2017. V. 7. 14033. https://doi.org/10.1038/s41598-017-14614-3
Абрамсон Н.И. Филогеография: итоги, проблемы, перспективы // Информ. вестник ВОГиС. 2007. Т. 11. № 2. С. 307–331.
Soltis D.E., Soltis P.S., Ranker T.A., Ness B.D. Chloroplast DNA variation in a wild plant, Tolmiea menziesii // Genetics. 1989. V. 121. № 4. P. 819–826.
Дополнительные материалы отсутствуют.


