Генетика, 2022, T. 58, № 12, стр. 1431-1441
STR-типирование европейского лося и европейской косули с использованием новых криминалистических наборов выявило различные паттерны генетической структуры двух видов оленевых в Беларуси
К. Рембала 1, Д. Э. Недзвецкая 2, *, С. А. Котова 2, Т. В. Забавская 2, В. И. Рыбакова 2, М. В. Холодова 3, И. С. Цыбовский 4
1 Кафедра судебной медицины Гданьского медицинского университета
80-210 Гданьск, Польша
2 Научно-практический центр Государственного комитета судебных экспертиз
220073 Минск, Беларусь
3 Институт проблем экологии и эволюции им. А.Н. Северцова Российской академии наук
119071 Москва, Россия
4 Республиканское унитарное предприятие “БелЮрОбеспечение”
220069 Минск, Беларусь
* E-mail: Nedzvedskayadiana@gmail.com
Поступила в редакцию 01.02.2022
После доработки 22.03.2022
Принята к публикации 23.03.2022
- EDN: BAOTPS
- DOI: 10.31857/S0016675822120104
Аннотация
Для судебно-экспертной идентификации биологических образцов лосей (Alces alces) и косуль (Capreolus capreolus) разработаны тест-системы для генотипирования, включающие 16 и 12 микросателлитов (STR-локусов) соответственно. Информативность тест-систем проверена генотипированием биологических образцов 386 лосей и 360 косуль из всех шести административных регионов Беларуси. Анализ молекулярной дисперсии выявил генетическую однородность популяции лосей практически по всей территории страны и значительную региональную генетическую дифференциацию популяции косуль. Причиной наблюдаемой популяционно-генетической структуры лосей в Беларуси является “бутылочное горлышко” – задокументированное массовое сокращение популяции в 1920-х гг. с последующим быстрым расширением популяции на основе однородного генетического субстрата, унаследованного от небольшой предковой популяции. Наблюдаемая географическая подразделенность европейской косули, вероятно, является результатом многочисленных генетических дрейфов и/или локального межвидового потока генов между двумя видами косули, обусловленного искусственным расселением сибирской косули (C. pygargus) в Восточной Европе в XX в. Вероятность различения генотипов двух неродственных особей с использованием общебелорусской базы генотипов лосей составляет выше 0.99999999999. В случае использования региональных баз данных генотипов косуль вероятность различения генотипов двух неродственных особей превышает 0.999999999. Это первое исследование, в котором представлены тест-системы для генетической ДНК-идентификации биологических образцов лосей и косуль, статистические параметры которых охарактеризованы исследованием генетической структуры популяций в масштабе всей страны, и с панелью STR-маркеров, достаточной для достижения доказательств уровня чрезвычайно убедительных в судебной экспертизе объектов дикой природы. Судебно-экспертные методики ДНК-идентификации биологических образцов животных вида лось (A. alces) и косуля (C. capreolus) имплементированы в национальную правовую систему Республики Беларусь.
Филогенетические исследования делят оленевых (Cervidae) на два подсемейства: Cervinae (олени Старого Света или плезиометакарпальные олени) и Capreolinae (олени Нового Света или телеметакарпальные олени) [1]. В лесах Беларуси (39% площади страны, более 9.4 млн га) обитают три распространенных вида оленевых: благородный олень (Cervus elaphus), лось (A. alces) и косуля (C. capreolus). Все три вида являются популярными охотничьими млекопитающими во многих странах, на них охотятся как легально, так и нелегально. В 2013 г. в лесах Беларуси обитало около 74 000 косуль, 28 000 лосей и 12 000 благородных оленей [2]. Четвертый вид оленевых – лань (Dama dama) можно встретить в дикой природе, однако в основном разводится как вольерное животное и промыслового значения не имеет.
Ежегодно в Беларуси проводится более тысячи расследований, связанных с правонарушениями в отношении диких животных, и примерно половина из них касается копытных. Всего в 2019 г. Научно-практическим центром Государственного комитета судебных экспертиз Республики Беларусь проведено 221 судебно-экспертное ДНК-типирование биологических образцов диких животных, причем в большинстве дел (81%) фигурировали три вида оленевых – лось, косуля и олень благородный. Судебные разбирательства по делам о незаконной охоте для предоставления неопровержимых доказательств преступления требуют видовой идентификации биологических образцов и их индивидуализации до уровня особи.
Видовая идентификация обычно основана на секвенировании митохондриальных генов цитохрома b (cytb) и цитохромоксидазы I (COI) [3]. Индивидуальная идентификация образцов заключается в генотипировании высокополиморфных ДНК-маркеров, оценке вероятности существования идентичных генетических профилей ДНК в популяции и демонстрации их уникальности в пределах тестируемых образцов. Мультиаллельные микросателлиты (короткие тандемные повторы, STR-локусы) являются маркерами, обычно используемыми в судебно-экспертных лабораториях для генетической идентификации лиц (особей), и их применение в криминалистике требует разработки методов генотипирования, основанных на мультиплексной полимеразной цепной реакции (ПЦР) и капиллярном электрофорезе [4].
Для расследования случаев незаконной коммерциализации и браконьерства в отношении ряда видов диких животных разработаны криминалистические тесты STR-типирования, созданы базы данных частот STR-аллелей для изучаемых локальных популяций [5]. Ранее нами описан метод криминалистической ДНК-идентификации образцов свиней и проведен обширный анализ полиморфизма микросателлитов у диких кабанов, обитающих в Беларуси, и у домашних свиней [6]. Однако для судебно-экспертного генотипирования оленевых известно только несколько таких тестов. Несмотря на то что олени являются очень популярными охотничьими млекопитающими в Европе [7, 8], большинство судебных STR-тестов и соответствующие базы данных частот аллелей были разработаны для видов оленевых, обитающих в Северной Америке [9–12]. В Европе внимание судебных генетиков приковано к благородному оленю (C. elaphus), считающемуся самым ценным охотничьим трофеем в этой части света [13]. Проведено генотипирование микросателлитов и созданы популяционные базы данных оленей (C. elaphus) в графстве Камбрия в Великобритании [14] и в Венгрии [13].
Цель настоящего исследования – разработка метода идентификации биологических образцов двух наиболее многочисленных оленевых Беларуси – лосей (A. alces) и косуль (C. capreolus), основанного на генотипировании STR-локусов методами мультиплексной ПЦР и капиллярного электрофореза, и создание общебелорусской базы данных частот аллелей для статистической оценки доказательств в криминалистической ДНК-экспертизе диких животных в Беларуси. Для генотипирования лосей разработана панель из 17 ДНК-маркеров, включающая 16 микросателлитов и локус амелогенин для установления пола. Соответственно для косуль предложена панель из 13 ДНК-маркеров, в том числе 12 микросателлитов и локус амелогенин.
МАТЕРИАЛЫ И МЕТОДЫ
Коллекции ДНК представлены 386 образцами лосей и 360 образцами косуль, законно добытых во всех административных областях Беларуси: Брестской области (27 лосей, 73 косули), Витебской (213 лосей, 78 косуль), Гомельской (30 лосей, 37 косуль), Гродненской (48 лосей, 94 косули), Минской (40 лосей, 47 косуль) и Могилевской (25 лосей, 29 косуль). 3 образца лося и 2 образца косули были получены в ходе судебной экспертизы, и их региональное происхождение в пределах Беларуси осталось неизвестным. В качестве контрольной популяции, не подвергшейся массовой убыли, в исследование были включены 11 образцов лосей из Сибири, происходящих из трех административных регионов Российской Федерации: Томской области (n = 5), Республики Саха (Якутия, n = 4) и Эвенкийского района Красноярского края (n = 2).
Для отбора STR-маркеров, проявляющих полиморфизм у лосей и косуль, у других оленевых (Rangifer tarandus, C. elaphus, C. canadensis), а также полорогих (Bos taurus, Ovis aries, Capra hircus), был проведен анализ научной литературы [15–26]. Эффективность отобранных микросателлитов на матрицах ДНК лося/косули была проверена в монолокусной и мультилокусной ПЦР, что позволило разработать два мультиплексных ПЦР-анализа, включающих в общей сложности 17 ДНК-маркеров (16 STR-локусов и локус амелогенин в качестве гендерного маркера) для генотипирования лосей и двух мультиплексных ПЦР, включающих в общей сложности 13 ДНК-маркеров (12 STR-локусов и амелогенин) для генотипирования косуль (табл. 1 ). Семь микросателлитов (BL4, BM203, BM848, BM1818, BM6438, T26, T172) использовали в мультиплексных ПЦР для обоих видов оленевых. Для всех мультиплексных систем амплификацию проводили в термоциклере С1000 (Bio-Rad) в конечном объеме 10 мкл, содержащем 10 мМ Трис-HCl, pH 8.8, 50 мМ KCl, 0.08% октилфеноксиполиэтоксиэтанол (Nonidet P-40), 2 мМ MgCl2, 0.2 мМ dNTP, 0.04–0.2 мкМ праймеров, 2 нг ДНК и 0.75 ед. Taq-полимеразы. Программа ПЦР включала начальную денатурацию (3 мин при 95°С), 10 циклов амплификации с отжигом при 60°С (45 с при 94°С, 1 мин при 60°С, 1 мин при 68°С), 21 цикл амплификации с отжигом при 59°С (45 с при 93.5°С, 1 мин при 59°С, 1 мин при 68°С) и терминальную элонгацию (30 мин при 72°С). Продукты ПЦР разделяли электрофоретически на генетическом анализаторе 3500 (Thermo Fisher Scientific). Аллели тестируемых локусов идентифицировали по размеру в парах нуклеотидов (пн) с использованием программного обеспечения GeneMapper ID-X v1.4 (Thermo Fisher Scientific) путем сравнения с внутренним стандартом размера GeneScan 600 LIZ v2.0 (Thermo Fisher Scientific).
Частоты аллелей, значения наблюдаемой и ожидаемой гетерозиготности, а также P-значения тестирований на соответствие равновесию Харди–Вайнберга (HWE) были получены с использованием программного обеспечения Arlequin 3.1 [27]. Это же программное обеспечение использовали для проверки неравновесия по сцеплению (LD) между маркерами с помощью теста отношения правдоподобия. Для проверки различий ожидаемой гетерозиготности между популяциями лосей из Беларуси и Сибири рассчитывали непараметрический критерий знаковых рангов Уилкоксона с использованием программного обеспечения STATISTICA 12 (StatSoft). Вероятность случайного совпадения (MP), определяемая как вероятность того, что два генотипа, выбранных случайным образом из популяции, будут идентичными, и сила дискриминации (PD), определяемая как вероятность того, что два генотипа, выбранные случайным образом из популяции, будут разными, рассчитывали с помощью PowerStats 1.2 [28]. Генетическую дифференциацию популяций по исследованным микросателлитам оценивали методом молекулярной дисперсии (AMOVA) с использованием программы Arlequin 3.1. Многомерное шкалирование (MDS), основанное на линеаризованных попарных значениях FST, выполнено в программе STATISTICA 12.
РЕЗУЛЬТАТЫ
Распределение частот аллелей исследованных STR-локусов, выявленное в локальных (региональных) популяциях лосей и косуль из Беларуси и в контрольной выборке лосей из Сибири, представлено в табл. S1 . Маркер T172, описанный как полиморфный у оленей C. canadensis, C. elaphus [11, 13] и включенный в наши STR-тесты для генотипирования как A. alces, так и C. capreolus, показал полиморфизм только у косуль. У всех лосей локус T172 амплифицируется в виде одного неизменного фрагмента с молекулярным размером 144 пн (табл. 1).
Таблица 1.
Характеристика ДНК-маркеров, использованных в данном исследовании
| Локус | Тип повтора | Вид- источник | Последовательность праймеров | Тест-система* (флуоресцентная метка) | A. alces (в данном исследовании) | C. capreolus (в данном исследовании) | Источник литературы | ||
|---|---|---|---|---|---|---|---|---|---|
| число аллелей | диапазон аллелей, пн | число аллелей | диапазон аллелей, пн | ||||||
| BL4 | 2 | B. taurus | F: AAATTTTTCATCCTTCTTTCTGAC R: TCACCCTGACTGTGAATGC | A1 (ROX), C2 (R6G) |
13 | 149–173 | 15 | 131–171 | [14, 15] |
| BM203 | 2 | B. taurus | F: GGGTGTGACATTTTGTTCCC R: CTGCTCGCCACTAGTCCTTC | A2 (ROX), C1 (ROX) |
12 | 219–255 | 2 | 213–215 | [14, 16] |
| BM757 | 2 | B. taurus | F: TGGAAACAATGTAAACCTGGG R: TTGAGCCACCAAGGAACC | A2 (TMR) | 12 | 180–204 | – | – | [16] |
| BM848 | 2 | B. taurus | F: TGGTTGGAAGGAAAACTTGG R: CCTCTGCTCCTCAAGACAC | A2 (FAM), C2 (FAM) |
11 | 331–361 | 9 | 349–371 | [16, 17] |
| BM888 | 2 | B. taurus | F: AGGCCATATAGGAGGCAAGCTT R: CTCGGTGAGCTCAAAACGAG | A1 (R6G) | 7 | 175–195 | – | – | [16] |
| BM1258 | 2 | B. taurus | F: GTATGTATTTTTCCCACCCTGC R: GAGTCAGACATGACTGAGCCTG | A2 (R6G) | 8 | 89–113 | – | – | [16] |
| BM1818 | 2 | B. taurus | F: AGCTGGGAATATAACCAAAGG R: AGTGCTTTCAAGGTCCATGC | A1 (R6G), C2 (R6G) |
8 | 246–264 | 14 | 235–269 | [16, 18] |
| BM6438 | 2 | B. taurus | F: TTGAGCACAGACACAGACTGG R: ACTGAATGCCTCCTTTGTGC | A1 (TMR), C2 (TMR) |
9 | 263–279 | 10 | 246–294 | [16, 19] |
| BMC1009 | 2 | B. taurus | F: GCACCAGCAGAGAGGACATT R: ACCGGCTATTGTCCATCTTG | C1 (TMR) | – | – | 6 | 279–289 | [14] |
| ILST058 | 2 | B. taurus | F: GCCTTACTACCATTTCCAGC R: CATCCTGACTTTGGCTGTGG | C1 (FAM) | – | – | 15 | 149–179 | [20] |
| MaF46 | 2 | O. aries | F: AAATACCCTATAAGGCACAGTACCAC R: CACCATGGCCACCTGGAATCAGG | A1 (FAM) | 12 | 87–113 | – | – | [21] |
| MaF70 | 2 | O. aries | F: CACGGAGTCACAAAGAGTCAGACC R: GCAGGACTCTACGGGGCCTTTGC | C1 (R6G) | – | – | 14 | 113–153 | [18] |
| NVHRT16 | 2 | R. tarandus | F: ATTCTAAGCCCAAATAATCTT R: TCTAAGGGGTCTGTGTCTT | C1 (TMR) | – | – | 9 | 158–178 | [22] |
| NVHRT21 | 2 | R. tarandus | F: GCAGCGGAGAGGAACAAAAG R: GGGGAGGAGCAGGGAAATC | C2 (ROX) | – | – | 14 | 152–188 | [22] |
| RBP3 | 2 | B. taurus | F: TGTATGATCACCTTCTATGCTTC R: GCTTTAGGTAATCATCAGATAGC | A1 (R6G) | 8 | 135–155 | – | – | [19] |
| RT30 | 2 | R. tarandus | F: CACTTGGCTTTTGGACTTA R: CTGGTGTATGTATGCACACT | A2 (R6G) | 14 | 186–212 | – | – | [23] |
| T26 | 4 | C. canadensis | F: GTTCCAATAGACACGCTCAT R: TGCCATAGTTTTTCCTACCTT | A2 (R6G), C2 (R6G) |
11 | 315–355 | 26 | 328–362 | [9] |
| T172 | 4 | C. canadensis | F: AGCATCTCCCCTTTCAACA R: CTTCCCAACCCAAGTATCG | A2 (FAM), C2 (FAM) |
1 | 144 | 10 | 152–196 | [9] |
| T193 | 4 | C. canadensis | F: AGTCCAAGCCTGCTAAATAA R: CTGCTGTTGTCATCATTACC | A1 (TMR) | 10 | 198–234 | – | – | [24] |
| T268 | 4 | C. canadensis | F: ATTCCCTTCTCCAGTGTATG R: GATGATAACAGCTCAACAGATC | A2 (FAM) | 7 | 215–237 | – | – | [24] |
| T530 | 4 | C. canadensis | F: GTCCTCACAGCAGCTCTATG R: GCATTCTTTAGAACTCCAACTG | A1 (FAM) | 7 | 241–261 | – | – | [9] |
| Amelogenin | – | B. taurus | F: AGTTCCTGGCCAACACTC R: GCTGGCCAAGCTTCCAGA | A1 (ROX), C1 (ROX) |
2 | 234–269 | 2 | 234–278 | [25] |
Анализ ассоциаций аллелей выявил неравновесное сцепление LD (P < 0.05) между 13 и 5 парами тестируемых STR-локусов у лосей и косуль соответственно. Однако после коррекции Бонферрони LD оставалось статистически значимым (P < < 0.00001; табл. S2 ) только для одной пары маркеров (T268 и T530 у лосей). Детальное изучение этих двух маркеров выявило очень похожее распределение частот аллелей в изученных популяциях лосей, при этом подавляющее большинство, но не все, аллели локусов T268 и T530 различаются по размеру на 26 пн. Сравнением последовательностей T268 и T530 C. canadensis, депонированных в GenBank (инвентарные номера: AF192399.1 и AF442817.1 соответственно), установлено, что две последовательности практически идентичны, а различающиеся два нуклеотида расположены за пределами тандемного участка. Таким образом, праймеры локусов T268 и T530 амплифицируют один и тот же STR-локус, а наблюдаемые небольшие различия между двумя маркерами являются результатом вариации фланкирующих областей. Для дальнейшего анализа был выбран локус T530, поскольку он показал несколько более высокую силу исключения у белорусских лосей, чем локус T268 (0.881 против 0.880 соответственно). В конечном итоге, после исключения локусов Т172 (мономорфного у лосей) и Т268 (идентичного с Т530), в исследовании популяций лосей и косуль были использованы суммарно 14 и 12 микросателлитов соответственно.
Оценка молекулярной дисперсии (AMOVA) выявила генетическую дифференциацию между региональными белорусскими популяциями лосей и косуль (табл. 2). Попарные сравнения locus by locus (AMOVA) показали, что наблюдаемая географическая дифференциация является результатом статистически значимых различий в частотах аллелей (P < 0.05) двух STR-локусов (BL4, T193) у лосей и восьми STR-локусов (BM848, BM6438, ILST058, MaF70, NVHRT16, NVHRT21, Т26, Т172) у косуль. Единая для всей территории база данных частот аллелей обеспечила бы возможность статистической оценки доказательств, полученных ДНК-типированием, независимо от географического происхождения образцов. Устранение локусов, ответственных за обнаруженную генетическую дифференциацию (согласно AMOVA), привело к географической однородности популяций лосей и косуль по всей изученной части ареала. Суммарная вероятность различения генотипов двух неродственных особей на основе полученных общеприменимых баз данных (12 микросателлитов у лосей и 4 микросателлита у косуль) составила 0.999999999997 и 0.9993 соответственно (табл. 2 ). Сила дискриминации общеприменимой базы данных для косуль не соответствует уровню доказательства, потенциально интерпретируемого как чрезвычайно убедительное (т.е. с отношением правдоподобия не менее 106) [29].
Таблица 2.
Результаты AMOVA, значения вероятностей случайного совпадения генотипов (MP), значения вероятностей различения генотипов двух неродственных индивидуумов (PD) у лосей и косуль из шести административных регионов для панелей маркеров из 14 и 12 STR-локусов соответственно и данные, в которых гомогенность была достигнута за счет исключения маркеров, демонстрирующих региональную гетерогенность или генетически различающиеся популяции
| Набор данных | AMOVA, FST-индекс | AMOVA, P-значение | MP | PD |
|---|---|---|---|---|
| A. alces, 14 STR в шести регионах* | 0.00254 | 0.00822 | – | – |
| A. alces, 12 STR, демонстрирующих однородность в шести регионах | 0.00092 | 0.21762 | 1 × 3.7 × 10–11 | 0.999999999997 |
| A. alces, 14 STR, демонстрирующих однородность в пяти регионах | 0.00109 | 0.16653 | 1 × 6.0 × 10–13 | 0.99999999999998 |
| C. capreolus, 12 STR в шести регионах* | 0.00891 | 0.00000 | – | – |
| C. capreolus, 4 STR, демонстрирующих однородность в шести регионах | 0.00300 | 0.06941 | 1 × 1.4 × 10–3 | 0.9993 |
| C. capreolus, 12 STR, регион Витебска | – | – | 1 × 1.3 × 10–10 | 0.99999999992 |
| C. capreolus, 12 STR, регион Минска | – | – | 1 × 5.0 × 10–10 | 0.99999999998 |
| C. capreolus, 11 STR**, регион Гомель + Могилев | 0.00140 | 0.31838 | 1 × 6.0 × 10–9 | 0.9999999998 |
| C. capreolus, 11 STR**, регион Бреста | – | – | 1 × 5.9 × 10–9 | 0.9999999998 |
| C. capreolus, 11 STR**, регион Гродно | – | – | 1 × 2.6 × 10–9 | 0.9999999996 |
Отклонения от равновесия Харди–Вайнберга HWE (P < 0.05) в популяциях лосей и косуль в Беларуси выявлены всего в двух и трех локусах соответственно. После применения поправки Бонферрони статистически значимым оставалось только одно отклонение от HWE, которое касалось маркера Т26 у белорусских косуль (P < < 0.00001). Неравновесие Харди–Вайнберга в локусе Т26 у косуль оставалось статистически значимым после поправки Бонферрони также в трех региональных популяциях из Беларуси: гомельской, брестской и гродненской (табл. S3). После исключения локуса Т26 из-за несоответствия равновесию Харди–Вайнберга совокупная сила дискриминации в региональных популяциях косуль превышала 0.999999999 (табл. 2).
Генетические дистанции между региональными популяциями лосей и косуль из Беларуси, визуализированные с помощью MDS, показаны на рис. 1. Для полной панели из 12 микросателлитов, протестированных у косуль, все попарные сравнения между региональными популяциями, кроме двух, были статистически значимыми (P < 0.05; табл. S4 ). Последовательное исключение наиболее дивергентных региональных популяций не позволило сформировать общую базу данных в формате 12 STR-локусов даже для трех наиболее генетически близких популяций косуль – гомельской, могилевской и гродненской (FST = 0.00444; P = 0.01584). В то же время для полной панели из 14 STR-локусов, протестированных у лосей, статистически значимые попарные значения FST для региональных популяций наблюдались только для гомельской популяции, которая оказалась генетически однородной только с лосями из Брестской области. После исключения гомельской выборки из AMOVA остальные региональные популяции показали отсутствие генетической субструктуры в популяции лосей в Беларуси (табл. 2).
Рис. 1.
График MDS генетических расстояний FST между популяциями лосей (верхний график) и косуль (нижний график) из шести административных регионов Беларуси.
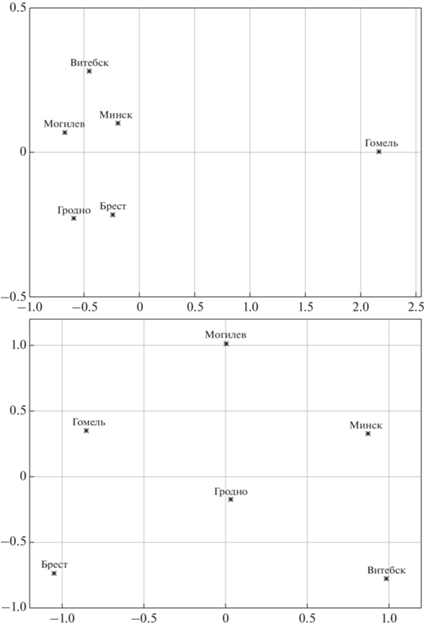
Лоси из Сибири генетически отличались от популяций лосей из Беларуси (P < 0.00001 для всех попарных сравнений) со значениями FST в среднем в 5 раз выше, чем значения FST для внутрибелорусских попарных сравнений. В отношении шести региональных популяций лосей из Беларуси сибирские лоси показали самый низкий индекс FST при сравнении с географически ближайшей популяцией из Могилевской области (табл. S4 ). Хотя в настоящее исследование было включено всего 11 лосей из Сибири по сравнению с 386 белорусскими особями, у сибирских лосей мы выявили суммарно 22 аллеля, которые не наблюдались у лосей из Беларуси (табл. S1 ). Критерий знаковых рангов Уилкоксона показал, что ожидаемая гетерозиготность исследуемых STR-маркеров у белорусских лосей статистически значимо ниже, чем у лосей из Сибири (среднее: 0.689 против 0.743; медиана: 0.733 против 0.848; P = = 0.02).
ОБСУЖДЕНИЕ
Идентификация биологических образцов в судебно-экспертном исследовании ДНК по фактам правонарушений в отношении диких животных может осуществляться путем генотипирования микросателлитов (STR-локусы) или однонуклеотидных полиморфизмов (SNP). Однако применение SNP-маркеров часто ограничивается тем, что эти маркеры нередко проявляют географическую и/или популяционую специфичность [30].
В разработанной нами панели для криминалистической идентификации образцов оленевых используются STR-маркеры, которые имеют высокую частоту мутаций и с большой вероятностью останутся полиморфными в большинстве популяций [31], что обеспечивает универсальность разработанного метода ДНК-идентификации образцов лосей и косуль, обитающих в Беларуси, а также его применимость для генотипирования оленевых в других странах Европы.
Микросателлитный анализ, разработанный для судебно-экспертной идентификации лосей, впервые был использован для отнесения особей A. alces к их исходным популяциям из провинций Новая Шотландия и Нью-Брансуик в Канаде [32], но информативность анализа для проверки генетической идентичности образцов европейских лосей не оценивалась. Совсем недавно была опубликована новая криминалистическая тест-система для STR-типирования A. alces, апробированная на популяциях лосей из провинции Альберта и территории Юкон в Канаде, однако данное исследование не предоставляет базу данных частот аллелей для криминалистических и популяционно-генетических исследований [33]. Другой тест для типирования микросателлитов лося компании “Гордиз” (Россия) недавно стал коммерчески доступным в виде набора COrDIS Alces, но его полезность в судебно-экспертной практике не подтверждена какими-либо оценками параметров полиморфизма или популяционными исследованиями. Аналогично, хотя косули являются наиболее многочисленным видом оленевых [7] и самым добываемым видом оленевых в Европе [34], до недавнего времени было доступно только одно исследование, в котором использовано судебно-экспертное STR-типирование для идентификации и разработки базы данных частот аллелей C. capreolus, населяющей регион Передней Померании на северо-востоке Германии [35]. К тому же данный тест включал только шесть STR-локусов и имел ограниченную способность различения неродственных особей в судебно-экспертной практике, достигающей только 0.995, в связи с чем не мог обеспечить получение доказательств, интерпретируемых как чрезвычайно убедительные (с отношением правдоподобия не менее 106) [29]. Новый криминалистический STR-тест для косули, опубликованный в 2021 г., имеет высокую эффективность идентификации, но протестирован только на одной популяции косуль из Швейцарии [36].
Лось – самый крупный из ныне живущих видов оленевых и одно из крупнейших наземных млекопитающих в Европе. По сравнению с более мелкими млекопитающими крупные млекопитающие всегда непропорционально сильно страдали от чрезмерного промысла человеком [37] по причине более долгого жизненного цикла и потребности в более крупных участках обитания, в связи с чем их популяции, вероятно, подвергались событиям генетического дрейфа [38]. Наши результаты выявили противоположные закономерности генетической структуры двух исследованных видов оленевых в Беларуси. Удивительно, но статистически значимая генетическая дифференциация (FST = 0.00891; P < 0.00001) была обнаружена между региональными популяциями гораздо меньших по размеру косуль, возможно, в результате локальных генетических дрейфов, тогда как популяция лосей, имеющих гораздо большие размеры, показала однородность практически на всей территории Беларуси. Данный феномен можно было бы объяснить значительно большими популяционными изъятиями косули, вызванными охотой, браконьерством, гибелью на дорогах, хищниками и болезнями. Однако в официальной статистике показатели ежегодного изъятия особей охотниками в Беларуси сопоставимы для обоих видов и составляют в среднем 6% для лосей и 7% для косуль. Кроме того, предполагаемая ежегодная общая убыль популяции с учетом теоретического коэффициента воспроизводства у косуль лишь немного выше, чем у лосей (23% против 19%) [2]. Таким образом, наблюдаемые различия в генетической структуре обоих видов нельзя объяснить меньшей эксплуатацией и меньшими общими потерями популяции лосей. На наш взгляд, обнаруженная гомогенность региональных популяций лосей из Беларуси может быть связана с прохождением генетического “бутылочного горлышка”, в результате которого вид восстанавливался из небольшой предковой популяции с однородным генетическим субстратом. В 1920-х гг. лось в Беларуси стал чрезвычайно редким видом, находящимся на грани исчезновения. В 1927 г. было зарегистрировано всего около 150 особей лосей [39], а менее чем за столетие популяция лося в Беларуси выросла примерно в 200 раз. Нашими исследованиями зафиксировано также снижение генетического полиморфизма STR-локусов у белорусских лосей по сравнению с сибирскими лосями, что, по-видимому, является еще одним последствием прохождения популяцией довольно жесткого “бутылочного горлышка”. Следы недавнего генетического узкого места были обнаружены также в популяциях лосей, населяющих соседнюю Польшу, с аналогичной демографической историей [40]. Следует также учитывать, что исследованная выборка сибирских лосей насчитывает всего 11 особей, населяющих обширную территорию Сибири, и представляет другой подвид, к тому же с разнообразным генетическим составом [41, 42]. Наблюдаемая географическая подразделенность европейской косули в Беларуси предположительно может быть также результатом локальной гибридизации с сибирской косулей (C. pygargus), возникшей естественным или искусственным образом в результате крупномасштабного выпуска особей обоих видов косули в Восточной Европе в ХХ в. [43, 44]. Таким образом, наиболее вероятной причиной наблюдаемых противоположных паттернов генетической структуры двух исследованных видов оленевых в Беларуси являются экспансия небольшой предковой популяции лосей после прохождения “бутылочного горлышка” и многочисленные генетические дрейфы и/или межвидовой поток генов в локальных популяциях косуль.
В случаях, если популяционные исследования выявляют генетическую дифференциацию региональных популяций, с целью оценки вероятности встречаемости генетического профиля при статистической интерпретации результатов судебно-экспертных исследований для генетически разнообразных популяций рекомендуется использовать отдельные базы данных частот аллелей [45]. Наши результаты AMOVA показывают, что для лосей в судебной криминалистике в Беларуси следует использовать две базы данных генотипов: одну для всей территории (за исключением Гомельской области) и отдельную базу для локальной юго-восточной популяции, населяющей леса Гомельской области Беларуси. В случае косуль мы рекомендуем проводить оценку вероятностей встречаемости генетических профилей на основе региональных баз данных частот аллелей STR-локусов. Наши результаты также показывают, что для части региональных популяций косуль из Беларуси не следует использовать для статистической оценки достоверности доказательств экспертных исследований локус T26. Установлено, что частоты генотипов в этом локусе отклоняются от HWE в гомельской, брестской и гродненской популяциях косуль, в то время как оценка вероятности наблюдения идентичного ДНК-профиля в судебной практике может быть непосредственно рассчитана по частотам аллелей только в том случае, если распределение аллелей согласуется с законом Харди–Вайнберга [46].
Таким образом это первое исследование, в котором представлены тест-системы для ДНК-идентификации биологических образцов лосей (A. alces) и косуль (C. capreolus) с использованием панели STR-маркеров, обладающей статистическими параметрами, достаточными для получения экспертных доказательств уровня чрезвычайно убедительных в расследовании правонарушений в отношении объектов дикой природы. Судебно-экспертные методики ДНК-идентификации биологических образцов животных видов лось (A. alces) и косуля (C. capreolus) включены в Реестр судебно-экспертных методик и иных методических материалов Государственного комитета судебных экспертиз Республики Беларусь, что соответствует имплементации разработки в национальную правовую систему Беларуси.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Gilbert C., Ropiquet A., Hassanin A. Mitochondrial and nuclear phylogenies of Cervidae (Mammalia, Ruminantia): systematics, morphology, and biogeography // Mol. Phylogenet. Evol. 2006. V. 40. № 1. P. 101–117. https://doi.org/10.1016/j.ympev.2006.02.017
Козорез А.И. Ресурсы Оленьих Беларуси: ученые – практикам // Лесное и охотничье хоз-во. 2014. Т. 136. № 11. С. 42–47.
Tobe S.S., Kitchener A.C., Linacre A.M.T. Reconstructing mammalian phylogenies: A detailed comparison of the cytochrome b and cytochrome oxidase subunit I mitochondrial genes // PLoS One. 2010. V. 5. № 11. e14156. https://doi.org/10.1371/journal.pone.0014156
Butler J.M. Advanced Topics in Forensic DNA Typing: Methodology. Waltham: Elsevier, 2012. 708 p.
Linacre A., Gusmão L., Hecht W. et al. ISFG: recommendations regarding the use of non-human (animal) DNA in forensic genetic investigations // Forensic Sci. Int. Genet. 2011. V. 5. № 5. P. 501–505. https://doi.org/10.1016/j.fsigen.2010.10.017
Rębała K., Rabtsava A.A., Kotova S.A. et al. STR profiling for discrimination between wild and domestic swine specimens and between main breeds of domestic pigs reared in Belarus // PLoS One. 2016. V. 11. № 11. P. 1–14. https://doi.org/10.1371/journal.pone.0166563
Burbaitė L., Csányi S. Roe deer population and harvest changes in Europe // Estonian J. Ecol. 2009. V. 58. № 3. P. 169–180. https://doi.org/10.3176/eco.2009.3.02
Burbaitė L., Csányi S. Red deer population and harvest changes in Europe // Acta Zool. Litu. 2010. V. 20. № 4. P. 179–188. https://doi.org/10.2478/v10043-010-0038-z
Jones K.C., Levine K.F., Banks J.D. DNA-based genetic markers in black-tailed and mule deer for forensic applications // Calif. Fish Game. 2000. V. 86. № 2. P. 115–126.
Jones K.C., Levine K.F., Banks J.D. Characterization of 11 polymorphic tetranucleotide microsatellites for forensic applications in California elk (Cervus elaphus canadensis) // Mol. Ecol. Notes. 2002. V. 2. № 4. P. 425–427. https://doi.org/10.1046/j.1471-8286.2002.00264.x
Meredith E.P., Rodzen J.A., Levine K.F., Banks J.D. Characterization of an additional 14 microsatellite loci in California elk (Cervus elaphus) for use in forensic and population applications // Conserv. Genet. 2005. V. 6. № 1. P. 151–153. https://doi.org/10.1007/s10592-004-7735-8
Smith P.F., DenDanto D., Smith K.T. et al. Allele frequencies for three STR loci RT24, RT09, and BM1225 in northern New England white-tailed deer // J. Forensic Sci. 2002. V. 47. № 3. P. 673–675. https://doi.org/10.1520/JFS15312J
Szabolcsi Z., Egyed B., Zenke P. et al. Constructing STR multiplexes for individual identification of Hungarian red deer // J. Forensic Sci. 2014. V. 59. № 4. P. 1090–1099. https://doi.org/10.1111/1556-4029.12403
Socratous E., Graham E.A.M., Rutty G.N. Forensic DNA profiling of Cervus elaphus species in the United Kingdom // Forensic Sci. Int. Genet. Suppl. Ser. 2009. V. 2. № 1. P. 281–282. https://doi.org/10.1016/j.fsigss.2009.08.127
Vial L., Maudet C., Luikart G. Thirty-four polymorphic microsatellites for European roe deer // Mol. Ecol. Notes. 2003. V. 3. № 4. P. 523–527. https://doi.org/10.1046/j.1471-8286.2003.00499.x
Schmidt J.I., Hundertmark K.J., Bowyer R.T., McCracken K.G. Population structure and genetic diversity of moose in Alaska // J. Hered. 2009. V. 100. № 2. P. 170–180. https://doi.org/10.1093/jhered/esn076
Bishop M.D., Kappes S.M., Keele J.W. et al. A genetic linkage map for cattle // Genetics. 1994. V. 136. № 2. P. 619–639. https://doi.org/10.1093/genetics/136.2.619
Bonnot N., Gaillard J.M., Coulon A. et al. No difference between the sexes in fine-scale spatial genetic structure of roe deer // PLoS One. 2010. V. 5. № 12. P. 1–5. https://doi.org/10.1371/journal.pone.0014436
Kuehn R., Hindenlang K.E., Holzgang O. et al. Genetic effect of transportation infrastructure on roe deer populations (Capreolus capreolus) // J. Hered. 2007. V. 98. № 1. P. 13–22. https://doi.org/10.1093/jhered/esl056
Cronin M.A., Patton J.C., Balmysheva N., MacNeil M.D. Genetic variation in caribou and reindeer (Rangifer tarandus) // Anim. Genet. 2003. V. 34. № 1. P. 33–41. https://doi.org/10.1046/j.1365-2052.2003.00927.x
Kemp S.J., Hishida O., Wambugu J. et al. A panel of polymorphic bovine, ovine and caprine microsatellite markers // Anim. Genet. 1995. V. 26. № 5. P. 299–306. https://doi.org/10.1111/j.1365-2052.1995.tb02663.x
Swarbrick P.A., Dietz A.B., Womack J.E., Crawford A.M. Ovine and bovine dinucleotide repeat polymorphism at the MAF46 locus // Anim. Genet. 1992. V. 23. № 2. P. 182. https://doi.org/10.1111/j.1365-2052.1992.tb00040.x
Røed K.H., Midthjell L. Microsatellites in reindeer, Rangifer tarandus, and their use in other cervids // Mol. Ecol. 1998. V. 7. № 12. P. 1773–1776. https://doi.org/10.1046/j.1365-294x.1998.00514.x
Wilson G.A., Strobeck C., Wu L., Coffin J.W. Characterization of microsatellite loci in caribou Rangifer tarandus, and their use in other artiodactyls // Mol. Ecol. 1997. V. 6. № 7. P. 697–699. https://doi.org/10.1046/j.1365-294x.1997.00237.x
Meredith E.P., Rodzen J.A., Banks J.D. et al. Microsatellite analysis of three subspecies of elk (Cervus elaphus) in California // J. Mammal. 2007. V. 88. № 3. P. 801–808. https://doi.org/10.1644/06-MAMM-A-014R.1
Gurgul A., Radko A., Słota E. Characteristics of X- and Y-chromosome specific regions of the amelogenin gene and a PCR-based method for sex identification in red deer (Cervus elaphus) // Mol. Biol. Rep. 2010. V. 37. № 6. P. 2915–2918. https://doi.org/10.1007/s11033-009-9852-4
Excoffier L., Laval G., Schneider S. Arlequin (version 3.0): An integrated software package for population genetics data analysis // Evol. Bioinform. Online. 2005. V. 1. P. 47–50.
Tereba A. Tools for analysis of population statistics // Profiles DNA. 1999. V. 2. № 3. P. 14–16.
Buckleton J. A framework for interpreting evidence // Forensic DNA Evidence Interpretation. Boca Raton: CRC Press, 2005. P. 27–63.
Ferguson B., Street S.L., Wright H. et al. Single nucleotide polymorphisms (SNPs) distinguish Indian-origin and Chinese-origin rhesus macaques (Macaca mulatta) // BMC Genomics. 2007. V. 8. № 43. https://doi.org/10.1186/1471-2164-8-43
Fan H., Chu J.Y. A brief review of short tandem repeat mutation // Genom. Proteom. Bioinform. 2007. V. 5. № 1. P. 7–14. https://doi.org/10.1016/S1672-0229(07)60009-6
Ball M.C., Finnegan L.A., Nette T. et al. Wildlife forensics: “supervised” assignment testing can complicate the association of suspect cases to source populations // Forensic Sci. Int. Genet. 2011. V. 5. № 1. P. 50–56. https://doi.org/10.1016/j.fsigen.2010.02.002
Sim Z., Monderman L., Hildebrand D. et al. Development and implementation of a STR based forensic typing system for moose (Alces alces) // Forensic Sci. Int. Genet. 2021. V. 53. P. 1–10. https://doi.org/10.1016/j.fsigen.2021.102536
Reimoser F., Reimoser S. Long-term trends of hunting bags and wildlife populations in Central Europe // Beitr. Jagd. Wildforsch. 2016. V. 41. P. 29–43.
Poetsch M., Seefeldt S., Maschke M., Lignitz E. Analysis of microsatellite polymorphism in red deer, roe deer, and fallow deer – possible employment in forensic applications // Forensic Sci. Int. 2001. V. 116. № 1. P. 1–8. https://doi.org/10.1016/s0379-0738(00)00337-6
Morf N.V., Kopps A.M., Nater A. et al. STRoe deer: A validated forensic STR profiling system for the European roe deer (Capreolus capreolus) // Forensic Sci. Int. Anim. Environ. 2021. V. 1. https://doi.org/10.1016/j.fsiae.2021.100023
Bowyer R.T., Boyce M.S., Goheen J.R., Rachlow J.L. Conservation of the world’s mammals: status, protected areas, community efforts, and hunting // J. Mammal. 2019. V. 100. № 3. P. 923–941. https://doi.org/10.1093/jmammal/gyy180
Harris R.B., Wall W.A., Allendorf F.W. Genetic consequences of hunting: what do we know and what should we do? // Wildl. Soc. Bull. 2002. V. 30. № 2. P. 634–643.
Сержанин И.Н. Млекопитающие Беларуси. 2-е изд. Минск: Изд-во Акад. наук Белорусской ССР, 1961. 330 с.
Świsłocka M., Czajkowska M., Duda N., Ratkiewicz M. Admixture promotes genetic variation in bottlenecked moose populations in eastern Poland // Mamm. Res. 2015. V. 60. № 2. P. 169–179. https://doi.org/10.1007/s13364-015-0221-5
Hundertmark K.J., Bowyer R.T. Genetics, evolution, and phylogeography of moose // Alces. 2004. V. 40. P. 103–122.
Рожков Ю.И., Проняев А.В., Давыдов А.В. и др. Лось: популяционная биология и эволюция. М.: Scientific Press. Ltd., 2009. 520 с.
Плахина Д.А., Звычайная Е.Ю., Холодова М.В., Данилкин А.А. Выявление гибридов европейской (Capreolus capreolus L.) и сибирской (C. pygargus Pall.) косуль на основе микросателлитного анализа // Генетика. 2014. Т. 50. № 7. С. 862–867. https://doi.org/10.7868/S0016675814070157
Кашинина Н.В., Данилкин А.А., Звычайная Е.Ю. и др. О генофонде косуль (Capreolus) Восточной Европы: Анализ вариабельности нуклеотидных последовательностей гена cytb // Генетика. 2018. Т. 54. № 7. С. 817–824. https://doi.org/10.1134/S0016675818070044
Zhivotovsky L.A., Malyarchuk B.A., Derenko M.V. et al. Developing STR databases on structured populations: the native south Siberian population versus the Russian population // Forensic Sci. Int. Genet. 2009. V. 3. № 4. P. e111–e116. https://doi.org/10.1016/j.fsigen.2008.08.001
Buckleton J. Population genetic models // Forensic DNA Evidence Interpretation. Boca Raton: CRC Press, 2005. P. 65–122.
Дополнительные материалы
- скачать ESM_1.xls
- Таблица 1. Частоты аллелей исследованных STR-локусов, выявленные у лося из разных регионов Беларуси и Сибири.
- скачать ESM_2.xls
- Таблица 2. P-значение по неравновесному сцеплению между парами STR-маркеров у косули (Capreolus capreolus) из Беларуси.
- скачать ESM_3.xls
- Таблица 3. Лось (Alces alces), P -значения отклонения от равновесия Харди–Вайнберга. Косуля (Capreolus capreolus), P -значения отклонения от равновесия Харди–Вайнберга
- скачать ESM_4.xls
- Таблица 4. Значения Fst (ниже от диагонали) и соответствующие P-значения (выше от диагонали) для 14 STR-маркеров у лося (Alces alces) из шести административных регионов Беларуси и Сибири. Значения Fst (ниже от диагонали) и соответствующие P-значения (выше от диагонали) для 12 STR-маркеров у косули (Capreolus capreolus) из шести административных регионов Беларуси.


