Генетика, 2022, T. 58, № 4, стр. 410-418
Генетическая однородность популяции самцов макак-резус по полиморфизму генов oprm1, npy, maoa, crh, 5-htt, определенная кластерным анализом данных клинического анализа крови
Л. Е. Павлова 1, *, Ал. В. Панченко 1, М. Ф. Тимина 1, Т. Е. Гвоздик 1, В. В. Коваленко 2, А. А. Агумава 1, Ан. В. Панченко 1
1 Научно-исследовательский институт медицинской приматологии
354376 Сочи, Россия
2 Сочинский государственный университет
354000 Сочи, Россия
* E-mail: pavlova_laura@mail.ru
Поступила в редакцию 12.07.2021
После доработки 30.08.2021
Принята к публикации 21.09.2021
- EDN: QOQDGA
- DOI: 10.31857/S0016675822030092
Аннотация
Генетическая неоднородность популяции в экспериментальных исследованиях с использованием живых организмов в качестве модельных объектов является одной из причин плохой воспроизводимости результатов. Приматы – наиболее филогенетически близкая модель к человеку, что позволяет применять ее для изучения генетических основ болезней с наследственной предрасположенностью, однако при содержании в неволе они представляют собой значительно ограниченную численностью популяцию. Нами проведено изучение генетической однородности популяции самцов макак-резус по полиморфизму генов oprm1, npy, maoa, crh, 5-htt. Для оценки использован кластерный анализ (метод K-means) с учетом данных генотипирования, происхождения животных, гематологического анализа и ветеринарного обследования. В результате были выявлены два значимых кластера животных, различающихся значениями показателей периферической крови в пределах референтного диапазона. Выделенные кластеры не были связаны с происхождением, возрастом, условиями содержания и уровнем заболеваемости животных. Кластеризация позволила выявить субпопуляцию животных, среди которых доля носителей полиморфизма гена maoa VNTR с семью повторами на 25% выше, чем в общей популяции. Таким образом, кластерный анализ негенетических данных может быть применен для оценки однородности распределения генетических вариантов в популяции и для отбора генетически более однородной субпопуляции с целью уменьшения проблемы малых выборок в биомедицинских исследованиях.
Прогресс в молекулярной биологии привел к качественно новому уровню понимания сложности организации и функционирования живых систем, используемых для моделирования заболеваний человека, и требует значительных усилий от экспериментатора по стандартизации условий опыта с целью получения достоверных данных. Одним из неполностью решенных вопросов остается воспроизводимость экспериментальных данных в последующих экспериментах. С одной стороны, это является нормальной частью научного процесса, что связано с внутренними вариациями, сложной природой изучаемого объекта, объемом текущих научных знаний и ограничениями существующих технологий. С другой стороны, невозможность воспроизведения результата может привести к открытию новых явлений или новому пониманию изменчивости изучаемой системы [1]. Очевидно, генетическая неоднородность популяции в экспериментальных исследованиях с использованием живых организмов в качестве модельных объектов является одной из причин плохой воспроизводимости данных. Основным подходом при изучении живых объектов в эксперименте является сопоставление данных, получаемых от испытуемых объектов контрольной и опытной групп. При этом зачастую основным ограничением является малый объем выборки экспериментальных объектов [2]. Малые выборки из-за генетически разнородной популяции могут приводить к включению в сравниваемые группы живых объектов с различающимися характеристиками, которые могут влиять как на результат эксперимента, так и на воспроизводимость полученных данных в последующих исследованиях. Традиционным объектом для современных генетических исследований в биомедицине являются мыши, использование которых позволяет наблюдать относительно большие выборки, а наличие тысяч генетически однородных инбредных линий позволяет исключить фактор генетической вариабельности популяции при проведении исследований [3]. Один из ценнейших объектов в биомедицинских исследованиях представляют собой приматы, что обусловлено их филогенетической близостью к человеку. Однако сложность работы с приматами связана с рядом ограничений, один из которых – малый объем выборки. Поэтому актуальным является вопрос изучения генетической однородности ограниченных популяций этих животных, содержащихся в неволе.
В современной медицине важное место занимают методы анализа для обнаружения в массивах данных ранее неизвестных, но полезных с практической точки зрения знаний, необходимых для принятия решений в различных сферах. Одна из задач такого анализа – предсказывать из наборов медицинских или ветеринарных данных возможные болезни. Например, на основании данных клинического анализа крови 524 пациентов был разработан алгоритм предсказания пяти типов заболеваний, включая лейкемию, воспалительные заболевания, бактериальные или вирусные инфекции, ВИЧ-инфекцию и анемию [4]. Получение данных о рисках генетических детерминант заболеваний может иметь важное значение для их профилактики, изучения взаимодействия со средовыми факторами в патогенезе. Влияние каждого генетического варианта на риск заболевания человека может быть незначительным, но выделение всей этой сложной генетической информации в единую полигенную оценку может дать значимый прогноз [5]. Еще одним подходом является поиск возможных кластеров, предсказывающих определенные характеристики пациентов, по данным негенетических анализов. Так, иерархический кластерный анализ применяется для предсказания прогноза выживаемости пациентов с гемодиализом на основании анализов крови и мочи [6]. Выделение популяционных кластеров может быть использовано как в фундаментальных, так и в прикладных целях [7].
Приматы используются в качестве уникальной модели для исследований болезней зависимости, поскольку обладают полиморфизмом ряда генов-ортологов, ассоциированных с этой патологией у человека. В частности, нами проведено изучение такого полиморфизма у макак-резус:
1) ген µ-опиоидного рецептора (oprm1, Arg26Pro), SNP (77 C>G) в кодирующей последовательности [8];
2) ген нейропептида Y (npy), SNP (–1002 T>G) промотора, GRF сайт [9];
3) ген моноаминоксидазы (maoa), полиморфизм VNTR (5, 6, 7–18 пн повторы) в промоторе, Х-хромосома [10];
4) ген кортиколиберина (crh), SNP (–248 C>T) в промоторе, GRF сайт [11];
5) ген транспортера серотонина (5-htt), Indel-полиморфизм 21 пн в промоторе [12].
Для оценки генетической однородности распределения этих генов в изучаемой популяции приматов нами проведен кластерный анализ с учетом данных происхождения животных, гематологического анализа и ветеринарного обследования.
МАТЕРИАЛЫ И МЕТОДЫ
Животные
В исследование включены 70 половозрелых самцов обезьян вида Macaca mulatta возрастом от 3.7 до 21.8 лет, содержащихся в питомнике ФГБНУ “НИИ МП” (г. Сочи, Россия). Животные или их предки поступали в питомник в разное время из Индии, Вьетнама, Китая и других стран или питомников. Для большинства животных известна их родословная, что позволило оценить происхождение как фактор потенциального генетического различия. Обезьяны получали сбалансированное питание в виде полнорационного комбинированного корма (ФГБНУ “НИИ МП”), а также дополнительно хлеб, яйца, свежие овощи, фрукты. Манипуляции с животными выполнялись в соответствии с принципами гуманного обращения с животными, принятыми в институте, и с директивой Европейского сообщества (2010/63/EC) и Хельсинкской декларацией.
Генетический анализ
Во время проведения профилактических ветеринарных осмотров получали пробы крови в пробирки с К2ЭДТА. Выделение ДНК из лейкоцитов периферической крови проводилось модифицированным гуанидиновым методом с адсорбцией на положительно заряженных частицах оксида кремния, с последующей отмывкой и элюацией [13].
Для идентификации полиморфизма был выполнен подбор праймеров к геномной последовательности ДНК макак-резус с помощью программного обеспечения Gene Runner (версия 6.5.51 Beta). В качестве консенсусных использованы геномные последовательности, депонированные в базе данных Ensembl Genome Browser (URL: https://www.ensembl.org/index.html). Для проверки специфичности подобранных праймеров использовался интернет портал Primer Blast (URL: https://www.ncbi.nlm.nih.gov/tools/primer-blast/).
Генотипирование каждого полиморфизма проводилось следующим методом: при помощи аллель-специфической ПЦР в реальном времени идентифицировали SNP генов crh (C–248T), npy (T–1002G), oprm1 (C77G). Программа амплификации включала фазу активации полимеразы (95°С, 10 мин), за которой следовали 45 циклов, включающих последовательно сменяющие друг друга фазы денатурации (95°С, 15 с), отжига (60–68°С, 30 с) и элонгации (72°С, 30 с). В случае VNTR-полиморфизма (maoa-lpr) и Indel-полиморфизма (5-htt-lpr) генотипирование проводилось при помощи ПЦР-амплификации с последующим анализом полиморфизма длины амплифицированных фрагментов ДНК (ПДАФ-анализ) в 2%-ном агарозном геле. Программа амплификации включала фазу активации полимеразы (95°С, 10 мин) и 40–50 циклов, состоящих из фаз денатурации (95°С, 20 с), отжига (60–65°С, 45 с) и элонгации (72°С, 45 с).
В табл. 1 представлены нуклеотидные последовательности праймеров, использовавшихся в работе, а также температуры отжига для каждого из анализов.
Таблица 1.
Последовательности общих и аллель-специфических праймеров, использованных в работе, а также температуры отжига для каждого из анализов
| Ген, полиморфизм, тип | Праймеры | Последовательности | Температура отжига |
|---|---|---|---|
| oprm1 (Arg26Pro) SNP (77C>G) | Аллель-специфические | F: CAAGTTGCTCCCCAGCACG F: CAAGTTGCTCCCCAGCACC |
64°C |
| Общие | F: AGCAATTGCACTGATGCCTT R: ATGGAGTAGAGGGCCATGAT |
63°C | |
| npy SNP (–1002 T>G) |
Аллель-специфические | F: CGCGATGAGCAAATTAATGTG F: CGCGATGAGCAAATTAATGTT |
60°C |
| Общие | F: TCTTGCATATTCATTCAACAGG R: CCAAAGTGACAGTGGTTAAATG R: AATGAGCAACAGTTTCTCTCTT |
60°C | |
| crh SNP (–248 C>T) |
Аллель-специфические | F: ATGGACAAGTCATAAGAAGCC F: ATGGACAAGTCATAAGAAGCT R: ATTTTGCTATCTCAACACTGAAT |
60°C |
| Общие | F: GGCCTTTCATAGTAAGAGGTCAATATGT R: CGCCTCTTGGTGACGTCAA |
60°C | |
| maoa-lpr VNTR (18-нуклеотидный повтор) |
Общие | F: CAGAAACATGAGCACAAACG R: TACGAGGTGTCGTCCAAGTT |
63°C |
| 5-htt-lpr Indel-полиморфизм (21 пн) |
Общие | F: GGCGTTGCCGCTCTGAATGCC R: CAGGGGAGATCCTGGGAGGGA |
67°C |
Для подтверждения специфичности конструированных праймеров полученные продукты амплификации разделяли электрофорезом в 10%-ном полиакриламидном и 2%-ном агарозном гелях. Для подтверждения генотипов, идентифицированных методом аллель-специфической ПЦР, дополнительно были подобраны и синтезированы праймеры, охватывающие участки с исследуемым полиморфизмом.
VNTR-полиморфизм и Indel-полиморфизм анализировались путем ПЦР-амплификации с последующим электрофоретическим разделением фрагментов ДНК в 2%-ном агарозном геле и анализом полиморфизма длины амплифицированных фрагментов ДНК. Продукты амплификации секвенировали для подтверждения специфичности тестируемых в ПЦР фрагментов ДНК.
На основании генетического анализа полиморфизма генов 5-htt, crh, npy и oprm1 было проведено преобразование качественных данных в количественные путем присвоения значения 0 при отсутствии в генотипе мутантного аллеля, значения 1 при наличии одной копии и значения 2 при наличии двух копий. Для VNTR-полиморфизма по гену maoa анализировали число повторов, равное 5, 6 и 7 (табл. 2).
Таблица 2.
Пример анализируемых данных
| Индивидуальный номер животного | 35 353 | 36 540 | 36 554 | 37 247 | … | |
|---|---|---|---|---|---|---|
| Число аллелей гена c полиморфизмом | maoa (VNTR-повторы) | 7 | 7 | 6 | 5 | … |
| 5-htt | 2 | 0 | 1 | 1 | … | |
| crh | 0 | 1 | 0 | 0 | … | |
| npy | 0 | 1 | 1 | 0 | … | |
| oprm1 | 0 | 1 | 0 | 1 | … | |
| Возрастная категория | 1 | 2 | 1 | 2 | … | |
| Усредненные показатели клинического анализа крови | WBC | 10.9 | 9.7 | 7.4 | 17.8 | … |
| LYMP | 2.1 | 1.5 | 1.6 | 4.6 | … | |
| GRA | 15.3 | 6.7 | 5.4 | 10 | … | |
| RBC | 3.6 | 7.72 | 4.4 | 6.37 | … | |
| Ветеринарный анамнез | количество осмотров | 1 | 0 | 2 | 2 | … |
| травмы | 1 | 0 | 0 | 2 | … | |
| заболевания ЖКТ | 0 | 0 | 0 | 2 | … | |
| Страна происхождения | Индия | 0.75 | 0.625 | 0.25 | 0.6875 | … |
| Вьетнам | 0.125 | 0.25 | 0.5625 | 0.15625 | … | |
| другая или не уточнено | 0.125 | 0.125 | 0.1875 | 0.15625 | … | |
Клинический анализ крови
Пробы для гематологического анализа получали от клинически здоровых обезьян при отсутствии каких-либо экспериментальных воздействий во время нахождения животных в индивидуальных клетках из поверхностных вен плеча в пробирки с К2ЭДТА в период с января 2007 по февраль 2021 г.
Подсчет форменных элементов крови проводили ручным методом с использованием стеклянных эритроцитарных/лейкоцитарных меланжеров для разбавления и счетом в камере Горяева или с использованием автоматического гематологического анализатора HumaCount 30TS (Германия) в ветеринарном режиме.
Для статистического анализа использовали четыре показателя: абсолютное содержание в крови лейкоцитов (WBC), гранулоцитов (GRA), лимфоцитов (LYMP) и эритроцитов (RBC). При наличии данных нескольких анализов крови (от одного до трех) для животных одной возрастной категории данные по параметрам усредняли.
Статистические методы
Соответствие распределения генотипов закону Харди–Вайнберга оценивалось при помощи теста Хи-квадрат с одной степенью свободы. Все данные были объединены в единую базу, после чего были проведены предварительный анализ и исключение случаев с неполными данными. В результате для проведения кластерного анализа были отобраны только случаи с полным набором данных по всем изучаемым параметрам. Предварительно исключили влияние различий в методах анализа крови на результаты. Пример анализируемых данных приведен в табл. 2. Анализ данных выполнен в программе SPSS версии 17.0. Построение кластеров выполняли для четырех параметров крови WBC, GRA, LYMP, RBC (метод K-means). Учитывали количество случаев, включенных в каждый кластер и кластерные центры по четырем параметрам. Проводили визуальную оценку распределения исследуемых параметров по кластерам (см. рис. 1,б, рис. 2). Для визуальной оценки непрерывные количественные параметры, такие как возраст и происхождение, были разделены на категории, получившие соответствующий цифровой код. Для возраста: 1 – молодые зрелые от 3 до 10 лет включительно, 2 – зрелые от 11 до 15 лет включительно и 3 – старше 15 лет. Для происхождения данные нормировали в диапазоне от 0 (отсутствие связи со страной импорта) до 1 (полное соответствие стране происхождения). Определяли достоверности распределения животных по кластерам (Т-тест для независимых выборок).
Рис. 1.
Распределение животных по кластерам. а – визуализация кластерного распределения животных при использовании метода K-means по четырем параметрам: WBC – лейкоциты, GRA – гранулоциты, LYMP – лимфоциты, RBC – эритроциты. Выделено четыре кластера (обозначены цифрами 1–4), кластер 3 не учитывали как значимый, так как он содержал единичное наблюдение; б – визуальный анализ результатов распределения возрастных групп животных (код “1” – от 3 до 10 лет, “2” – от 11 до 15 лет, “3” – старше 15 лет, обозначены кругами разного оттенка) в различных кластерах. Границы кластеров выделены сплошной линией.
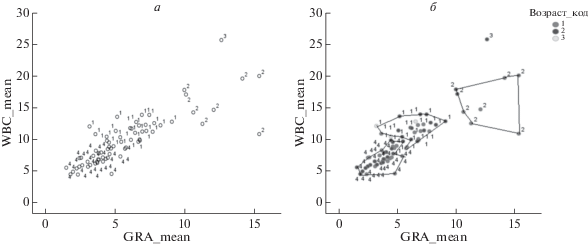
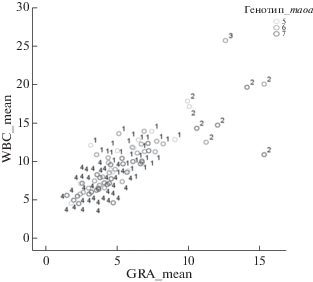
Рис. 2.
Визуальный анализ результатов распределения животных по VNTR-полиморфизму гена maoa c 5, 6 или 7 повторами по различным кластерам (обозначены цифрами 1–4).
Межкластерные сравнения проводили с помощью двустороннего дисперсионного анализа с поправкой на множественные сравнения Туки. Для сравнения частотных признаков использовали двусторонний точный тест Фишера. Наименьший критический уровень значимости принимали как α = 0.05.
РЕЗУЛЬТАТЫ
Для каждого полиморфизма закон Харди–Вайнберга соблюдается (p > 0.05), поскольку нет статистически значимых различий между наблюдаемыми и ожидаемыми генотипами.
Наилучшее распределение животных на кластеры было получено по четырем параметрам: WBC, GRA, LYMP, RBC. Результат кластеризации представлен на рис. 1. Достоверно выделено четыре кластера. Кластер 1 включает 28 животных, кластер 2 – восемь, кластер 3 – одно и кластер 4 – 33. Кластер 3, состоящий из единичного наблюдения, был исключен из дальнейшего анализа как незначимый (рис. 1). Оценка достоверности распределения животных по кластерам проведена с помощью Т-теста для независимых выборок по четырем параметрам. Для параметра WBC_mean × число случаев кластера F = 179.318, p < 0.0001; для параметра LYMP_mean × число случаев кластера F = 25.934, p < 0.0001; для параметра GRA_mean × число случаев кластера F = = 62.267, p < 0.0001 и для параметра RBC_mean × × число случаев кластера F = 0.610, p < 0.438.
Известно, что гематологические показатели макак, содержащихся в неволе, значительно зависят от их пола, возраста и происхождения [14, 15]. Наши данные свидетельствуют о том, что макаки-резус питомника ФГНУ “НИИ МП” значительно различаются по гематологическим показателям и делятся на три основных кластера. Однако подобное деление не связано с происхождением животных (табл. 3).
Таблица 3.
Происхождение животных, распределенных по кластерам
| Номер кластера | Индия, усл. ед. |
Вьетнам, усл. ед. |
Происхождение другое или не уточнено, усл. ед. |
|---|---|---|---|
| 1 (n = 28) | 0.4 ± 0.3 (0.3–0.5) |
0.3 ± 0.3 (0.1–0.4) |
0.3 ± 0.3 (0.2–0.5) |
| 2 (n = 8) | 0.4 ± 0.2 (0.2–0.6) |
0.4 ± 0.2 (0.2–0.6) |
0.3 ± 0.2 (0.1–0.4) |
| 4 (n = 33) | 0.4 ± 0.2 (0.3–0.5) |
0.2 ± 0.2 (0.2–0.3) |
0.4 ± 0.3 (0.3–0.5) |
Животные кластера под номером 2 имели меньший средний возраст и наибольшие значения уровня лейкоцитов и гранулоцитов в крови, которые превышали референтный диапазон. Это может свидетельствовать о наличии субклинического воспалительного процесса, который и привел к выделению животных в данный кластер. Животные двух наибольших кластеров под номерами 1 и 4 не различались по возрастному составу, но имели значимые различия по уровню лейкоцитов, лимфоцитов и гранулоцитов крови (табл. 4). При этом все параметры животных 1 и 4 кластеров соответствовали референтным данным, поэтому отличия не могут быть объяснены воспалительными изменениями в анализе крови и, вероятно, обусловлены другими особенностями.
Таблица 4.
Клиническая характеристика животных, распределенных по кластерам
| Номер кластера | Возраст, годы | Лейкоциты, 109/л | Лимфоциты, 109/л | Гранулоциты, 109/л | Эритроциты, 1012/л |
|---|---|---|---|---|---|
| Референтные данные [13] | 4–8 | 6.3 ± 2.6 (3.1–12.1) |
2.4 ± 1.3 (0.4–5.5) |
3.4 ± 2.0 (1.3–8.9)a |
5.39 ± 0.66 (4.39–7.02) |
| 1 (n = 28) | 11 ± 4.5 (9.2–12.8) |
11.3 ± 1.3 (10.8–11.8) |
3.9 ± 1.4 (3.3–4.4) |
6.2 ± 1.4 (5.6–6.7) |
6.60 ± 1.28 (6.09–7.11) |
| 2 (n = 8) | 7.3 ± 3.6 (4.0–10.6)*** ### |
15.9 ± 3.3 (13.1–18.6)*** ### |
3.1 ± 1.6 (1.8–4.4) |
12.3 ± 2.3 (10.4–14.2)*** ### |
5.35 ± 1.51 (4.09–6.62)# |
| 4 (n = 33) | 10.2 ± 3.5 (8.9–11.4) |
6.9 ± 1.2 (6.4–7.3)*** |
2.5 ± 0.8 (2.2–2.8)*** |
3.7 ± 1.1 (3.3–4.1)*** |
6.85 ± 1.32 (6.39–7.32) |
Примечание. Все параметры приведены в виде среднего значения и стандартного отклонения, в скобках указан 95%-ный доверительный интервал. *** – p < 0.001 по сравнению с кластером 1, ### – p < 0.001 по сравнению с кластером 4, # – p < 0.05 по сравнению с кластером 4, a – приведены значения для нейтрофилов.
В анамнезе у животных имелись заболевания инфекционного и травматического характера. Наиболее часто травмы были связаны с агрессивным поведением, представляли собой рваные раны различных участков тела и нередко сочетались с инфекционной патологией желудочно-кишечного тракта (табл. 5). Однако анализ не показал статистически значимых различий по количеству травм и воспалительных заболеваний желудочно-кишечного тракта в анамнезе у животных разных кластеров. Результаты свидетельствуют об отсутствии сочетания изученных вариантов полиморфизма генов и частотой заболеваемости у животных.
Таблица 5.
Генетическая характеристика животных разных кластеров и анамнестические данные о развитии у них заболеваний
| Номер кластера (число животных в кластере) | 1 (n = 28) | 2 (n = 8) | 4 (n = 33) | |||
|---|---|---|---|---|---|---|
| Генотип maoa, 7 повторов | 7 | 25% | 4 | 50% | 18 | 55%* |
| Генотип 5-htt, носители S-аллеля | 11 | 39% | 4 | 50% | 17 | 52% |
| Генотип crh, носители Т-аллеля | 2 | 7% | 1 | 13% | 2 | 6% |
| Генотип npy, гомозиготы по G-аллелю | 8 | 29% | 2 | 25% | 6 | 18% |
| Генотип oprm1, носители G-аллеля | 10 | 36% | 2 | 25% | 13 | 39% |
| Количество заболеваний в анамнезе | 0.3 ± 0.5 | (0.1–0.5) | 0.6 ± 0.7 | (0–1.2) | 0.6 ± 0.8 | (0.3–0.9) |
| Травмы в анамнезе | 0.3 ± 0.4 | (0.1–0.4) | 0.6 ± 0.7 | (0–1.2) | 0.2 ± 0.5 | (0–0.4) |
| Воспалительные заболевания ЖКТ в анамнезе | 0.1 ± 0.3 | (0–0.2) | 0.3 ± 0.7 | (0–0.8) | 0.2 ± 0.5 | (0–0.4) |
Примечание. Качественные параметры приведены в виде количества случаев, указана частота от количества животных в кластере, количественные параметры приведены в виде среднего значения и стандартного отклонения, в скобках указан 95%-ный доверительный интервал. ЖКТ – желудочно-кишечный тракт. * – p < 0.05 по сравнению с кластером 1 по точному тесту Фишера.
Из изученных генотипов только по VNTR-полиморфизму гена maoa была выявлена на 30% большая частота встречаемости семи повторов в кластере с номером 4 по сравнению со вторым по численности кластером под номером 1. Визуализация результатов распределения животных, носителей генотипа maoa с 5, 6 и 7 повторами VNTR по кластерам представлена на рис. 2.
Общая частота встречаемости 18-нуклеотидного повтора в гене maoa-lpr среди животных питомника для пяти копий составила 21%, для шести копий – 48%, а для семи повторов – 30%. В четвертом кластере животных частота встречаемости семи повторов maoa-lpr составила 55%, что на 25% больше, чем в общей популяции (p < 0.05, критерий Хи-квадрат).
Таким образом, полиморфизм четырех генов oprm1 (Arg26Pro) SNP (77C>G), npy SNP (–1002 T>G), crh SNP (–248 C>T) и 5-htt-lpr Indel-полиморфизм (21 пн) распределен в популяции равномерно, в отличие от полиморфизма гена maoa-lpr VNTR (18-нуклеотидный повтор).
ОБСУЖДЕНИЕ
В результате кластерного анализа нами были выявлены три значимые подгруппы животных. В случае одного из кластеров причиной его выделения могло быть наличие неустановленного субклинического воспалительного процесса, животные этого кластера имели меньший средний возраст. Несмотря на все попытки обеспечить включение в исследование только здоровых животных, этого практически никогда нельзя добиться из-за потенциального наличия какого-либо субклинического заболевания у клинически здоровых субъектов, что представляет определенные трудности при определении референтных диапазонов данных [16]. Два кластера животных имели значения показателей крови, соответствующие референтным диапазонам. У людей различия в референтных интервалах клинического анализа крови связаны с расовыми, этническими и географическими различиями, что требует устанавливать референтные интервалы, специфичные для определенной группы населения [17]. Однако выделенные нами кластеры не были связаны с происхождением животных, возрастом, условиями содержания (питание, температурный режим и др. факторы) и полиморфизмом генов oprm1 (Arg26Pro) SNP (77C>G), npy SNP (–1002 T>G), crh SNP (–248 C>T) и 5-htt-lpr Indel-полиморфизм (21 пн) у животных. Возможно, кластеры могли бы быть ассоциированы с продолжительностью жизни животных и гибелью от определенных заболеваний. Однако анализ связи с продолжительностью жизни провести не представляется возможным, так как на момент его проведения все 70 отобранных животных были живы. Не было выявлено связи кластеров с уровнем заболеваемости животных по данным ветеринарных осмотров. Исследования близнецов свидетельствуют, что порядка 61–96% межиндивидуальной вариабельности в количестве лейкоцитов, эритроцитов и тромбоцитов связаны с генетическими факторами [18]. Полногеномный поиск ассоциаций (GWAS) идентифицировал более 100 локусов, которые влияют на индивидуальные гематологические параметры у людей [19]. Подобная генетическая характеристика макак-резус питомника ФГБНУ “НИИ МП” не проводилась, что может быть предметом для дальнейших исследований.
В нашем случае кластеризация позволила оценить генетическую равномерность встречаемости в популяции полиморфизма нескольких генов-ортологов, ассоциированных с болезнями зависимости у человека. Была выявлена субпопуляция животных, в которой доля носителей полиморфизма гена maoa VNTR с семью повторами на 25% выше, чем в общей популяции. Известна ассоциация регуляторного полиморфизма промотора гена моноаминоксидазы-A (maoa-VNTR) с симптомами депрессии, широким спектром антиобщественного поведения и качеством сна, которая подтверждается эффектами препаратов, угнетающих активность MAOA и обладающих нежелательным действием в отношении качества сна [20, 21]. На основании полученных данных можно сделать вывод о значимой вариабельности изученной популяции макак-резус по некоторым генетическим вариантам. Для анализа генетического разнообразия популяции используются различные методы, в частности определение микросателлитных маркеров, SNP. Однако они требуют анализа тысяч таких маркеров и применения методов высокопроизводительного секвенирования в сочетании с биоинформационным анализом [22]. При изучении отдельных генетических вариантов и наличии данных негенетических исследований, таких как клинический анализ крови, для оценки гомогенности изучаемой популяции альтернативно может быть применен кластерный анализ.
Таким образом, кластерный анализ негенетических данных может быть применен для оценки однородности распределения генетических вариантов в популяции, отбора генетически более однородной субпопуляции с целью уменьшения проблемы малых выборок и улучшения воспроизводимости результатов при различных аспектах моделирования заболеваний человека.
Работа выполнена в рамках госзадания ФГБНУ “НИИ МП” за 2019–2021 гг.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Washington D.C. Reproducibility and Replicability in Science. Washington: Natl Acad. Press, 2019. 256 p. https://doi.org/10.17226/25303
Ledolter J., Kardon R.H. Focus on data: Statistical design of experiments and sample size selection using power analysis // Invest. Ophthalmol. Vis. Sci. 2020. V. 61. P. 11–11. https://doi.org/10.1167/iovs.61.8.11
Vijayarani S., Sudha S. An efficient clustering algorithm for predicting diseases from hemogram blood test samples // Ind. J. Science and Technology. 2015. V. 8. P. 1–8. https://doi.org/10.17485/ijst/2015/v8i17/52123
Phifer-Rixey M., Nachman M.W. Insights into mammalian biology from the wild house mouse Mus musculus // ELife. 2015. V.4. e05959. https://doi.org/10.7554/eLife.05959
Homburger J.R., Neben C.L., Mishne G. et al. Low coverage whole genome sequencing enables accurate assessment of common variants and calculation of genome-wide polygenic scores // bioRxiv. 2019. V. 11. P. 74. https://doi.org/10.1186/s13073-019-0682-2
Komaru Y., Yoshida T., Hamasaki Y. et al. Hierarchical clustering analysis for predicting 1-year mortality after starting hemodialysis // Kidney Intern. Reports. 2020. V. 5. P. 1188–1195. https://doi.org/10.1016/j.ekir.2020.05.007
Nnoaham K.E., Cann K.F. Can cluster analyses of linked healthcare data identify unique population segments in a general practice-registered population? // BMC Publ. Health. 2020. V. 20. P. 798. https://doi.org/10.1186/s12889-020-08930-z
Barr C.S., Schwandt M., Lindell S.G. et al. Association of a functional polymorphism in the µ-opioid receptor gene with alcohol response and consumption in male rhesus macaques // Archives General Psychiatry. 2007. V. 64. № 3. P. 369–376. https://doi.org/10.1001/archpsyc.64.3.369
Lindell S.G., Schwandt M.L., Sun H. et al. Functional NPY variation as a factor in stress resilience and alcohol consumption in rhesus macaques // Archives General Psychiatry. 2010. V. 67. № 4. P. 423–431. https://doi.org/10.1001/archgenpsychiatry.2010.23
Barr C.S., Schwandt M.L., Newman T.K., Higley J.D. The use of adolescent nonhuman primates to model human alcohol intake: neurobiological, genetic, and psychological variables // Annals N.Y. Acad. Sci. 2004. V. 1021. P. 221–233. https://doi.org/10.1196/annals.1308.027
Barr C.S., Dvoskin R.L., Gupte M. et al. Functional crh variation increases stress-induced alcohol consumption in primates // Proc. Natl Acad. Sci. USA. 2009. V. 106. P. 14593–14598. https://doi.org/10.1073/pnas.0902863106
Barr C.S., Newman T.K., Lindell S. et al. interaction between serotonin transporter gene variation and rearing condition in alcohol preference and consumption in female primates // Archives of General Psychiatry. 2004. V. 61. P. 1146–1152. https://doi.org/10.1001/archpsyc.61.11.1146
Agumava A.A., Chikobava M.G., Lapin B.A. Development of PCR test system for detecting primate betaherpes virinae // Mol. Genetics, Microbiology and Virology. 2010. V. 25. P. 132–135. https://doi.org/10.3103/S0891416810030079
Miyabe-Nishiwaki T., MacIntosh A.J.J., Kaneko A. et al. Hematological and blood chemistry values in captive Japanese macaques (Macaca fuscata fuscata) // J. Med. Primatology. 2019. V. 4. P. 338–350. https://doi.org/10.1111/jmp.12434
Koo B.-S., Lee D.-H., Kang P. et al. Reference values of hematological and biochemical parameters in young-adult cynomolgus monkey (Macaca fascicularis) and rhesus monkey (Macaca mulatta) anesthetized with ketamine hydrochloride // Lab. Animal Research. 2019. V. 35. № 7. https://doi.org/10.5607/en20040
Ichihara K. Statistical considerations for harmonization of the global multicenter study on reference values // Clinica Chimica Acta. 2014. V. 432. P. 108–118. https://doi.org/10.1016/j.cca.2014.01.025
Omuse G., Maina D., Mwangi J. et al. Complete blood count reference intervals from a healthy adult urban population in Kenya // PLoS One. 2018. V. 13. e0198444. https://doi.org/10.1371/journal.pone.0198444
Evans D.M., Frazer I.H., Martin N.G. Genetic and environmental causes of variation in basal levels of blood cells // Twin Research: The Official J. Intern. Society for Twin Studies. 1999. V. 2. P. 250–257. https://doi.org/10.1375/136905299320565735
Okada Y., Kamatani Y. Common genetic factors for hematological traits in Humans // J. Human Genet. 2012. V. 57. P. 161–169. https://doi.org/10.1038/jhg.2012.2
Драгович А.Ю., Боринская С.А. Генетическая и геномная основа агрессивного поведения // Генетика. 2019. Т. 55. № 12. С. 1381–1396. https://doi.org/10.1134/S0016675819090054
Brummett B.H., Krystal A.D., Siegler I.C. et al. Associations of a regulatory polymorphism of monoamine oxidase-A gene promoter (MAOA-uVNTR) with symptoms of depression and sleep quality // Psychosomatic Med. 2007. V. 69. P. 396–401. https://doi.org/10.1097/PSY.0b013e31806d040b
Fischer M.C., Rellstab C., Leuzinger M. et al. Estimating genomic diversity and population differentiation – an empirical comparison of microsatellite and SNP variation in Arabidopsis halleri // BMC Genomics. 2017. V. 18. № 69. https://doi.org/10.1186/s12864-016-3459-7
Дополнительные материалы отсутствуют.


