Генетика, 2022, T. 58, № 4, стр. 457-462
Оценка генеалогической структуры вятской породы лошадей (Equus ferus caballus) с использованием анализа ДНК
Л. А. Храброва 1, *, Н. В. Блохина 1, Н. Ф. Белоусова 1, Е. Г. Котран 2
1 Всероссийский научно-исследовательский институт коневодства, Рязанская область
391105 пос. Дивово, Россия
2 Колледж ветеринарной медицины и биомедицинских наук Техасского университета
77843 TX, США
* E-mail: l.khrabrova@yandex.ru
Поступила в редакцию 28.07.2021
После доработки 18.09.2021
Принята к публикации 28.09.2021
- EDN: ICWPEH
- DOI: 10.31857/S0016675822040063
Аннотация
Процесс формирования генеалогической структуры в вятской породе делает актуальной оценку ее дифференциации на генетическом уровне. В результате тестирования 293 вятских лошадей по 17 локусам микросателлитов ДНК было выявлено 136 аллелей (Ае = 4.11, Ho = 0.726) и дана оценка генетической дифференциации десяти линий по ряду показателей, включая индекс фиксации Fst (0.011–0.134). Секвенирование участка D-петли мтДНК (530 пн) показало наличие 11 гаплогрупп при высоком уровне вариабельности гаплотипов в маточных семействах. Высокий уровень генетической дифференциации генеалогической структуры вятской породы лошадей является предпосылкой успешной работы по сохранению этой малочисленной автохтонной породы.
Вятская лошадь создана с помощью метода “народной селекции” и известна с середины XIV в., относится к типу местных лесных пород. Эта уникальная отечественная порода была выведена в Вятской губернии, на современной территории Удмуртской Республики и Кировской обл., в бассейнах рек Вятка, Кама и Обва. В первой половине XIX в. тройки вятских лошадей были основным транспортом, но со временем они стали вытесняться орловскими рысаками. Уже к концу этого столетия вятку признали почти исчезнувшей породой. Попытки восстановления вятской лошади в 1930–1950-х гг. оказались нерезультативными, так как совпали с общей тенденцией сокращения численности лошадей в сельском хозяйстве. Целенаправленная работа по сохранению этой уникальной породы была начата в середине 1980-х гг. В результате обследования десятков хозяйств были отобраны наиболее типичные лошади и налажена система первичного племенного учета. Приоритетным направлением селекционной работы стало чистопородное разведение вятских лошадей на нескольких племенных фермах Удмуртии и Кировской обл. [1]. В настоящее время общая численность поголовья вяток составляет 760 лошадей, включая 250 кобыл. По современным критериям ФАО эта малочисленная порода имеет уязвимый статус, поэтому в программах по ее сохранению целесообразно использовать генетические методы оценки биоразнообразия [2].
Молекулярные методы исследований последних десятилетий существенно расширили наши знания о генетических особенностях пород, их филогенетических связях и процессах породообразования [3–7]. Стандартизация тестирования микросателлитной ДНК лошадей под эгидой международного общества генетики животных (ISAG) позволила сопоставлять результаты тестирования разных лабораторий, необходимые как при контроле происхождения лошадей, так и при паспортизации конских пород [8, 9]. Изучение полиморфизма ядерного и митохондриального геномов у лошадей отечественных пород выявило достаточно высокий уровень генетического разнообразия даже в малочисленных популяциях, включая наличие ряда приватных аллелей [10–14]. ДНК-маркеры подтвердили свою эффективность при оценке внутрипородного генетического разнообразия на уровне популяций, линий и маточных семейств, характеризующего степень дифференциации генеалогической структуры породы [15–18].
Комплексные исследования полиморфизма ядерной и митохондриальной ДНК вятских лошадей ранее не проводились. Задача настоящего исследования – изучение вариабельности микросателлитных локусов и последовательности D-петли митохондриальной ДНК (мтДНК) у лошадей вятской породы, а также характеристика генетических особенностей и оценка дифференциации мужских линий и женских семейств.
Материалом для анализа микросателлитов ДНК служили пробы крови и волос 293 лошадей вятской породы, принадлежащих племенным фермам и частным владельцам. Пробы ДНК амплифицировали с праймерами “StockMarks for Horses” (“Applied Biosystems”, США) и COrDIS S550 (ООО “ГОРДИЗ”, г. Москва); оба набора включают 17 STR-локусов, рекомендованных ISAG. Электрофорез и детекцию продуктов амплификации проводили на генетическом анализаторе ABI 3130 (“Applied Biosystems”, США) с использованием программного обеспечения GeneMapperTM 4.0. Лошадей тестировали в лаборатории генетики Всероссийского НИИ коневодства (n = 253) и Техасского университета США (n = 40).
При характеристике генетических параметров вятской породы и ее линейной структуры учитывали общее число аллелей (Na), среднее число аллелей на локус (MNа), эффективное число аллелей (Ae), степень наблюдаемой (Ho) и ожидаемой (He) гетерозиготности и показатель информационного полиморфизма (PIC) для каждого локуса. Генетические различия между линиями оценивали с учетом коэффициентов генетического сходства и генетических дистанций между ними по М. Nei [19] и методов F-статистики [20]. Обработку данных проводили на базе программ Exсel 2010, Statistica 12 v.10 и FSTAT 1.2 (www2.unil.ch/popgen/fstat.htm).
Для секвенирования D-петли мтДНК были взяты пробы волос 22 лошадей вятской породы из основных маточных семейств. Выделенную ДНК очищали и амплифицировали с праймерами, подобранными в соответствии с референсной последовательностью ископаемой шведской лошади Х79547 [21]. Амплификаты секвенировали с использованием набора BigDyeTM Terminator Cycle Sequencing Kit (PE “Applied Biosystems”, США) на анализаторе ABI 3130xl (PE “Applied Biosystems”, США) в лаборатории ООО “Генетика”, г. Москва. Для сравнительного анализа последовательностей гипервариабельного участка D-петли мтДНК использовали данные GenBank по 18 базовым гаплогруппам (A–R) c номерами доступа JN398377–JN398457, а также результаты генотипирования 18 вятских лошадей (18: DQ328020–DQ328037). При филогенетическом анализе секвенированного участка D-петли мтДНК с 15 471 по 16 000 основание использовали модель максимального комплексного правдоподобия и 50% уровень бутстреп-поддержки достоверности построения дерева на базе программ BioEdit 7.2.1 и MEGA7.
При тестировании вятских лошадей по 17-ти STR-локусам было определено 136 аллелей, что свидетельствует о достаточно высоком уровне вариабельности микросателлитной ДНК в этой малочисленной породе. Число аллелей в локусах варьировало от 5 до 14, наиболее широкий спектр вариантов был представлен в локусах ASB17 (14), AHT4 (10) и HTG10 (10). Для генетической структуры вятской породы была характерна высокая частота встречаемости аллелей: VHL20Q (0.224), АНТ4P (0.386), HTG6O (0.708), ASB2N (0.380), HTG10M (0.389), HMS2H (0.448) и LEX3L (0.232). В двух локусах были выявлены редкие аллели – HTG6L (0.002) и АНТ5P (0.007), не встречающиеся у лошадей Западной Европы [8] (табл. 1).
Таблица 1.
Характеристика полиморфизма 17-ти STR локусов у лошадей вятской породы
| Локус | Na | Ае | Ho | He | Fis | PIC |
|---|---|---|---|---|---|---|
| VHL20 | 9 | 6.312 | 0.835 | 0.842 | 0.008 | 0.824 |
| HTG4 | 6 | 1.973 | 0.522 | 0.493 | –0.058 | 0.444 |
| AHT4 | 10 | 4.128 | 0.768 | 0.758 | –0.013 | 0.729 |
| HMS7 | 7 | 3.672 | 0.731 | 0.728 | –0.004 | 0.700 |
| HTG6 | 7 | 1.861 | 0.463 | 0.463 | –0.002 | 0.426 |
| AHT5 | 8 | 3.901 | 0.785 | 0.744 | –0.055 | 0.709 |
| HMS6 | 5 | 3.932 | 0.778 | 0.746 | –0.043 | 0.707 |
| ASB23 | 9 | 4.533 | 0.801 | 0.779 | –0.027 | 0.758 |
| ASB2 | 9 | 4.222 | 0.784 | 0.763 | –0.027 | 0.724 |
| HTG10 | 10 | 4.695 | 0.798 | 0.787 | –0.014 | 0.757 |
| HTG7 | 5 | 2.669 | 0.655 | 0.625 | –0.048 | 0.569 |
| HMS3 | 7 | 4.782 | 0.760 | 0.791 | 0.039 | 0.767 |
| HMS2 | 7 | 3.370 | 0.718 | 0.703 | –0.021 | 0.651 |
| ASB17 | 14 | 6.972 | 0.895 | 0.857 | –0.045 | 0.838 |
| LEX3 | 9 | 5.816 | 0.593 | 0.628 | 0.000 | 0.803 |
| HMS1 | 7 | 3.275 | 0.703 | 0.695 | –0.012 | 0.663 |
| CA425 | 7 | 3.672 | 0.758 | 0.728 | –0.041 | 0.676 |
| B среднем на локус | 8.0 | 4.105 | 0.726 | 0.714 | –0.017 | 0.691 |
Число эффективных аллелей в локусах (Ае) менялось в широком диапазоне от 1.861 (HTG6) до 6.972 (ASB17), при среднем значении 4.105. В большинстве локусов наблюдался положительный баланс гетерозиготных генотипов, среднее значение Fis составило –0.017. Показатели информационного полиморфизма (PIC), характеризующие число и частоту встречаемости аллелей в локусах, изменялись в интервале 0.426–0.838 (табл. 1). Среднее значение PIC оказалось достаточно высоким (0.691), что свидетельствует об эффективности использования микросателлитных локусов в качестве генетических маркеров.
Оценка генетической дифференциации мужских линий показала, что между ними имеются различия по ряду показателей, включая спектр аллелей, уровень полиморфности, степень гетерозиготности и коэффициент фиксации (табл. 2).
Таблица 2.
Характеристика полиморфизма 17-ти STR локусов в разных линиях вятских лошадей
| Линия | N | Na | MNa | Ае | Ho | He | Fis | Fst |
|---|---|---|---|---|---|---|---|---|
| Боба | 44 | 111 | 6.529 | 3.322 | 0.683 | 0.636 | –0.071 | 0.054 |
| Боцмана-Бубенчика | 6 | 73 | 4.294 | 2.818 | 0.676 | 0.593 | –0.139 | 0.122 |
| Бурана | 34 | 100 | 5.882 | 2.757 | 0.670 | 0.593 | –0.128 | 0.115 |
| Воробья | 6 | 63 | 3.706 | 2.627 | 0.678 | 0.589 | –0.149 | 0.113 |
| Габизона | 38 | 106 | 6.235 | 2.888 | 0.651 | 0.602 | –0.070 | 0.110 |
| Добрика | 67 | 111 | 6.529 | 3.459 | 0.720 | 0.653 | –0.098 | 0.032 |
| Знатока | 28 | 110 | 6.471 | 3.678 | 0.707 | 0.667 | –0.050 | 0.011 |
| Кабура | 12 | 86 | 5.059 | 3.083 | 0.696 | 0.623 | –0.111 | 0.075 |
| Малахита | 11 | 75 | 4.412 | 3.047 | 0.727 | 0.610 | –0.189 | 0.087 |
| Радиуса | 17 | 91 | 5.353 | 3.263 | 0.639 | 0.600 | –0.065 | 0.134 |
| В среднем по линиям | 26 | 92.6 | 5.447 | 3.094 | 0.685 | 0.617 | –0.107 | 0.085 |
Сравнительно высокие показатели уровня полиморфности и наблюдаемой гетерозиготности (Ае = 3.68, Ho = 0.707) были характерны для линии Знатока, тогда как пока еще малочисленная линия Воробья выделялась низкими показателями генетической изменчивости (Ае = 2.63, Ho = = 0.678). Высокие коэффициенты фиксации, характеризующие степень дифференциации линий на фоне породы по STR-локусам, были определены для линий Радиуса (0.134), Бурана (0.115) и Воробья (0.110). Для всех линий и породы в целом были рассчитаны отрицательные значения Fis, что свидетельствует о сохранении равновесия Харди–Вайнберга и отсутствии внутрипородного инбридинга. Коэффициенты генетического сходства между представителями разных линий варьировали в интервале 0.664–0.905, что свидетельствует о существенной линейной дифференциации и эффективности зоотехнической работы по формированию линейной структуры вятской породы.
Изучение полиморфизма гипервариабельного региона D-петли мтДНК у 40 вятских лошадей, относящихся к 18-ти маточным семействам, показало широкую индивидуальную вариабельность гаплотипов, восходящих к гаплогруппам A, B, C, J, L, M, N, Р и Q по классификации A.T. Achilli с соавт. [6]. Дополнительно к этому семейства Кукушки и Малютки продемонстрировали два новых уникальных ответвления гаплогрупп, не описанных ранее (рис. 1).
Рис. 1.
Филогенетическое дерево гаплогрупп мтДНК лошадей вятской породы, построенное по методу Neighbor-Joining при бутстреп-поддержке >50%.
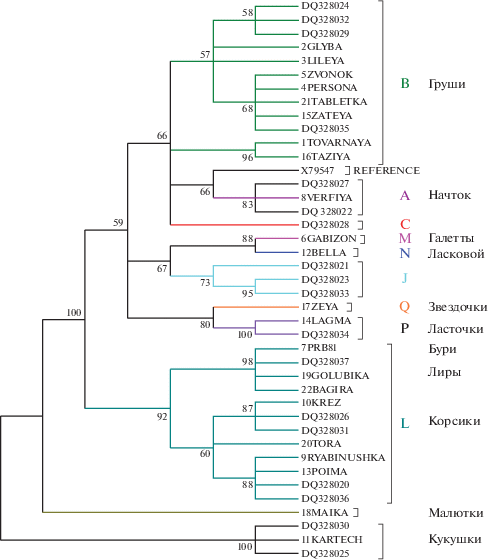
Проанализированный участок митохондриального генома вятских лошадей размером 530 пн включал 69 нуклеотидных замен, представленных преимущественно трансверсиями, при этом коэффициент нуклеотидных замен Rsi/sv составил 0.5. Среднее число замен на сайт достигло 0.13 ± ± 0.0118, что указывает на сравнительно высокий уровень индивидуального разнообразия в этой породе.
Результаты анализа матрилинейной структуры породы свидетельствуют, что 9 из 18 маточных семейств (50%) являются обладателями “фамильной” гаплогруппы мтДНК, которая может служить генетическим маркером конкретной женской линии. Было установлено, что кобылы семейства Ветки, относящиеся к гаплогруппе А, имели отдаленное родство с ископаемой шведской лошадью, представленной референсной последовательностью Х79547 [21]. В отличие от лошадей владимирской породы [22], сформированной в центральной части России, у вяток были дополнительно определены C и Q гаплогруппы мтДНК, указывающие на наличие и азиатских корней в их происхождении.
В структуре митохондриального генома вяток преобладала гаплогруппа L (27.8%), объединяющая пять маточных семейств (Бури, Зуры, Корсики, Лиры и Пумы), при этом было определено близкое родство кобыл-основательниц Зуры и Пумы по материнской линии. Кластер гаплогруппы B включал семейства Груши и Начток, представленные двумя отдельными подгруппами (рис. 1 ).
Попытка использования анализа мтДНК для уточнения происхождения трех типичных вятских кобыл с неправильно указанным происхождением оказалась результативной и позволила установить их принадлежность к конкретному семейству. У кобылы Тора был определен гаплотип, типичный для семейства Корсики и гаплогруппы L. Кобыла Таблетка вошла в кластер, образованный семейством Груши (гаплогруппа В). Кобыла Багира (гаплогруппа L) примкнула к семейству Лиры при высоком уровне бутстреп-поддержки (98%).
В настоящее время сформирована база данных генетических маркеров лошадей вятской породы, что позволяет эффективно вести контроль происхождения по микросателлитам ДНК и оценивать ресурсы внутрипородного разнообразия. Проведенный молекулярно-генетический анализ вятской породы лошадей на базе 17-ти микросателлитных локусов и мтДНК свидетельствует о достаточно высоком уровне геномного разнообразия и отсутствии внутрипородного инбридинга. Установленные различия между лошадьми десяти разных линий по спектру аллелей, уровню гетерозиготностии и коэффициентам генетического сходства, свидетельствуют о продуктивной зоотехнической работе по формированию генеалогической структуры, являющейся основой внутрипородного гетерозиса. Изучена матрилинейная структура породы, представленная 40 гаплотипами и 11 гаплогруппами мтДНК, свидетельствующими об участии в процессе образования вятской лошади женских предков азиатского происхождения.
Мы благодарим канд. с.-х. наук С.И. Сорокина (Лаборатория HorseGene, Москва) за помощь в лабораторных анализах мтДНК.
Исследования выполнены при поддержке Российского научного фонда (проект № 19-76-20058).
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Белоусова Н.Ф. История, современность и проблемы северных лесных пород лошадей России // Аборигенные породы лошадей: их роль и место в коневодстве РФ. I Всерос. науч.-практ. конф. с междунар. участием. Ижевск: Ижевская ГСХА, 2016. С. 8–22.
The Second Report on the State of the World’s Animal Genetic Recourses for Food and Agriculture Organization of the United Nations. Rome: 2015. 784 p.
Jansen T., Foster P., Levine M.A. et al. Mitochondrial DNA and the origin of the domestic horse // Proc. Natl Acad. Sci. USA. 2000. V. 99. № 16. P. 10905–10910. https://doi.org/10.1073/pnas.152330099
McGahern M.A., Bower A., Edwards C.J. et al. Evidence for biogeographic patterning of mitochondrial DNA in eastern horse populations // Animal Genet. 2006. V. 37. № 5. P. 494–497.
Cieslak M., Pruvost M., Benecke N. et al. Origin and history of mitochondrial DNA lineages in domestic horses // PLoS One. 2010. V. 5. № 12. e15311. https://doi.org/10.1371/jornal.pone.0015311.t002
Achilli A. Olivieri A., Soares P. et al. Mitochondrial genomes from modern horses reveal the major haplogroups that underwent domestication // PNAS. 2012. V. 109. № 7. P. 2449–2454.
Khanshour A.M., Cothran E.G. Maternal phylogenetic relationships and genetic variation among Arabian horse populations using whole mitochondrial DNA D-loop sequencing // BMC Genetics. 2013. V. 1. P. 14–83. https://doi.org/10.1186/1471-2156-14-83
Van de Goor L.H.P., van Haeringen W.A. A proposal for standardization in forensic equine DNA typing: Allele nomenclature for equine-specific STR loci // Animal Genet. 2010. V. 41. № 2. P. 122–127. https://doi.org/10.1111/j.1365-2052.2009.01975.x
Warmuth V., Manica A., Eriksson A. et al. Autosomal genetic diversity in non-breed horses from eastern Eurasia provides insights into historical population movements // Animal Genet. 2012. V. 44. № 1. P. 53–61. https://doi.org/10.1111/j.1365-2052.2012.02371.x
Калашников В.В., Храброва Л.А., Зайцев А.М. и др. Изучение полиморфизма сателлитной ДНК лошадей заводских и местных пород // Докл. РАСХН. 2010. № 6. С. 48–50.
Khrabrova L.A. Characterization of Genetic Horse Breeding Resources in Russia. Saarbruchen, Germany. Lap Laambert Acad. Publ., OmniScriptum GmbH& Co. KG, 2015. 68 p.
Воронкова В.Н., Столповский Ю.А. Оценка генетического разнообразия аборигенных пород Саяно-Алтайского региона с использованием ядерных и митохондриальных ДНК-маркеров // Аборигенное коневодство России: история, современность, перспективы. Сб. науч. тр. Архангельск: 2018. С. 60–69.
Юрьева И.Б., Свищева Г.Р., Вдовина В.Н. и др. Генетическое разнообразие мезенской породы лошадей (Equus ferus caballus) по микросателлитной ДНК // Генетика. 2018. Т. 54. Прилож. С. 564–569.
Khaudov A.D., Duduev A.S., Kokov Z.A. et al. Genetic analysis of maternal and paternal lineages in Kabardian horses by uniparental molecular markers // Open Vet. J. 2018. V. 8. № 1. P. 40–46. https://doi.org/10.4314/ovj.v8iI.7
Hill E.W., Bradley M., Al-Barody M. et al. History and integrity of Thoroughbred dam lines revealed in equine mtDNA variation // Anim. Genet. 2002. V. 33. № 4. P. 287–294.
Lopes M.S., Mendonca D., Cymbron T. et al. The Lusitano horse maternal lineage based on mitochondrial D‑loop sequence variation // Anim. Genet. 2006. V. 36. № 3. P. 196–202. https://doi.org/10.1111/j.1365-2052.01279x
Храброва Л.А., Блохина Н.В., Сулейманов О.И. и др. Оценка линейной дифференциации в чистокровной верховой породе лошадей с использованием локусов микросателлитов ДНК // Вавилов. журн. генет. и селекции. 2019. Т. 23. № 5. С. 569–574. https://doi.org/10.18699/VJ19.526
Khrabrova L.A., Zaitsev M.A., Vikulova L.L. et al. MtDNA haplotype analysis in dam families of the Thoroughbred riding horses // Proc. Conf. Modern Trends in Agricultural Production in the World Economy. 2020. P. 34–42. https://doi.org/10.32743/kuz.agri.2020.34-42
Nei M. Molecular Evolutionary Genetics. N.Y.: Columbia Univ. press, 1987. 512 p.
Вейр Б. Анализ генетических данных. М.: Мир, 1995. 399 с.
Xu X., Arnason U. The complete mitochondrial DNA sequence of the horse Equus caballus: Extensive heteroplasmy of control region // Gene. 1994. V. 1480. P. 357–362.
Сорокин С.И. Молекулярно-генетический анализ петли митохондриальной ДНК представителей маточных семейств владимирской породы // Коневодство и конный спорт. 2015. № 6. С. 27–29.
Дополнительные материалы отсутствуют.


