Генетика, 2022, T. 58, № 5, стр. 570-580
Однородность генофонда западной и восточной популяций даурского журавля Antigone vipio на разных пролетных путях
Е. А. Мудрик 1, *, О. А. Горошко 2, 3, С. Г. Сурмач 4, Т. А. Кашенцева 5, А. В. Нечаева 1, С. М. Смиренский 6, Д. В. Политов 1
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
2 Государственный природный биосферный заповедник “Даурский”
674495 Забайкальский край, Нижний Цасучей, Россия
3 Институт природных ресурсов, экологии и криологии Сибирского отделения
Российской академии наук
672014 Чита, Россия
4 Федеральный научный центр биоразнообразия наземной биоты Восточной Азии Дальневосточного отделения Российской академии наук
690022 Владивосток, Россия
5 Питомник редких видов журавлей Окского государственного природного биосферного заповедника
391072 Рязанская область, Брыкин Бор, Россия
6 Муравьевский парк устойчивого природопользования
676964 Амурская область, Муравьевка, Россия
* E-mail: mudrik@vigg.ru
Поступила в редакцию 29.11.2021
После доработки 16.12.2021
Принята к публикации 28.12.2021
- EDN: HSGHJB
- DOI: 10.31857/S001667582205006X
Аннотация
Представлены первые данные о генетической структуре даурского журавля Antigone vipio – редкого мигрирующего вида птиц Северо-Восточной Азии. С использованием семи полиморфных микросателлитных локусов и секвенирования полной последовательности контрольного региона мтДНК (1132 пн) установлена генетическая гомогенность пространственно разобщенных западной и восточной популяций, использующих разные пролетные пути. Обнаруженные высокие уровни наблюдаемой (HO = 0.696 ± 0.033) и ожидаемой (HE = 0.707 ± 0.037) гетерозиготности и гаплотипического разнообразия (Hd = 0.973) даурского журавля сопоставимы с этими показателями у широкоареальных видов журавлей с большой численностью. Отсутствие генетической дифференциации по микросателлитным локусам (FST = 0.013, P = 0.369) и низкий уровень генетических различий по контрольному региону (FST = 0.041, P = 0.05), невыраженность внутривидовой пространственной структурированности даурского журавля по гаплотипам и индивидуальным многолокусным генотипам могут быть обусловлены отсутствием репродуктивной изоляции между особями из разных популяций и изменениями миграционных маршрутов неполовозрелыми птицами.
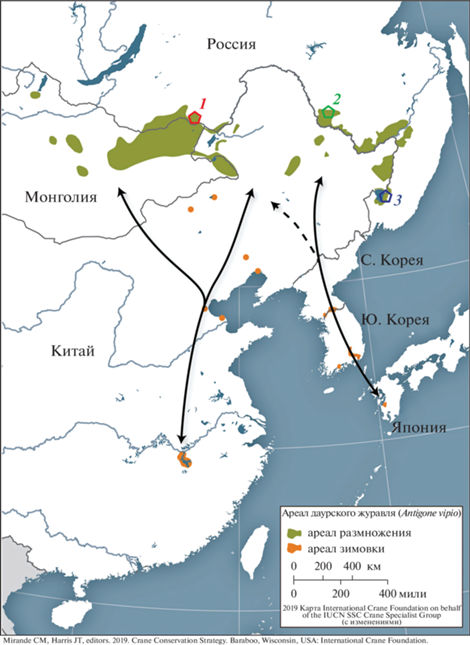
Даурский журавль Antigone vipio Pallas, 1881 – редкий в мировом масштабе вид птиц, занесенный в категорию уязвимых Красного списка Международного союза охраны природы [1]. Гнездовая часть его ареала приурочена к водно-болотным биотопам степной и лесостепной зон в бассейне Амура на территории России, Монголии и Китая [2, 3]. В видовой структуре даурского журавля выделяют западную и восточную популяции, использующие разные места гнездования и зимовки и соответственно разные пролетные пути. Птицы западной популяции гнездятся в России (на юго-востоке Забайкальского края) и Монголии и частично на сопредельных территориях Китая, а зимуют в Китае на озере Поянг. Птицы из восточной популяции гнездятся на Дальнем Востоке России (на юге Амурской области, Еврейской АО, Хабаровского края и в Приморье) и в Северо-Восточном Китае, а зимуют в Японии и демилитаризованной зоне Корейского полуострова [3] (рис. 1).
Рис. 1.
Ареал даурского журавля [3] и места происхождения изучаемых птиц: 1 – западная популяция; 2 – восточная популяция (север); 3 – восточная популяция (юг).
Глобальная численность даурского журавля оценивается в 7000–7800 особей [3]. На долю западной популяции приходится 500–1000 птиц, она сократилась в 4 раза за последние 20 лет; тогда как в восточной популяции насчитывают 6200–6500 особей и ее численность продолжает увеличиваться [3, 4]. Такие изменения могут объясняться как снижением уровня воспроизводства западной популяции в связи с многолетней засухой, так и вероятным оттоком даурских журавлей в восточную часть ареала и оттуда на восточные зимовки, ставшие более привлекательными благодаря практикуемой там искусственной подкормке. Тренды внутригнездовых ареалов популяций на территории России в последние 20–25 лет также разнонаправлены: местообитания и численность западной популяции значительно сократились, а восточной – существенно увеличились. Общая современная численность даурского журавля в России составляет около 610 территориальных пар (90 – в западной популяции, 520 – в восточной), а с учетом негнездящихся птиц – около 2200 особей [5].
Географическая разобщенность двух популяций A. vipio может способствовать их генетической дифференциации, так же как снижение численности западной и рост восточной группировки могут отражаться на их генетическом разнообразии. Никогда ранее популяционно-генетические работы по изучению видовой структуры даурского журавля не проводились ни в одной из стран, где он обитает. Наше исследование представляет первые данные о генетической структуре этого вида c территории России.
Цель работы – анализ генетической изменчивости и дифференциации западной и восточной популяций даурского журавля в России по данным ядерных микросателлитных локусов и секвенирования контрольного региона митохондриальной ДНК.
МАТЕРИАЛЫ И МЕТОДЫ
Биологический материал
В качестве биологического материала использовали индивидуальные образцы от 51 даурского журавля из мест гнездования западной (20 особей) и восточной (31 особь) популяций (рис. 1). В западной популяции образцы (растущие покровные перья из оперения груди) были получены от птенцов 1–1.5-месячного возраста во время их мечения кольцами и передатчиками в Ононском, Агинском и Борзинском районах Забайкальского края (разрешения Росприроднадзора № 50 от 30.06.2015, № 44 от 27.04.2016 и № 7 от 07.02.2019), а также от взрослых птиц, происходящих из природы юго-востока Забайкалья и содержащихся в Питомнике редких видов журавлей Окского заповедника (кровь, взятая во время плановых диспансеризаций). Также использовали линные перья с гнездовых участков даурского журавля в Забайкалье. В восточной популяции источником для выделения ДНК служили линные перья и подскорлуповые оболочки (аллантоисы) с гнездовых участков A. vipio в Тамбовском районе Амурской области, конфискат добытых браконьерами особей из Спасского района Приморского края (мышечные ткани) и полученная во время плановых диспансеризаций кровь птиц природного происхождения (Архаринский район Амурской области), содержащихся на Станции редких видов журавлей Хинганского заповедника и в Питомнике редких видов журавлей Окского заповедника.
Выделение ДНК
Экстракцию ДНК из линных перьев проводили с помощью набора для выделения ДНК innuPREP Forensic Kit (Analytik Jena, Германия), из растущих перьев и крови – с применением набора DIAtom™ DNAPrep100 Kit (ООО “Лаборатория Изоген”, Россия) по протоколам производителей. Полученную геномную ДНК разводили до концентрации 50–100 нг/мкл (Qubit 2.0, Termo Fisher Scientific, США) и использовали для последующей амплификации микросателлитных фрагментов ядерной ДНК и контрольного региона митохондриальной ДНК (мтДНК) методом ПЦР.
Микросателлитный анализ
Поскольку специфические ядерные микросателлитные локусы для даурского журавля не разработаны, мы провели тестирование более чем 20 пар праймеров для микросателлитов, охарактеризованных у других видов журавлей. В результате мы отобрали семь локусов, подходящих для наших целей и полиморфных у A. vipio: Gram22, Gram30 [6], Gpa12, Gpa38, Gpa39 [7] и Gj4066, Gj2298 [8] (табл. 1). ПЦР проводили с помощью набора реагентов GenPak PCR Core (OOO “Лаборатория Изоген”) согласно рекомендациям производителя по режимам отжига праймеров из первоисточников [6–8] на приборе Dyad Peltier Thermal Cycler (Bio-Rad, США). Для определения длин амплифицированных фрагментов проводили вертикальный электрофорез в 6%-ном полиакриламидном геле в трис-ЭДТА-боратной буферной системе с использованием камер модели VE-20 (ООО “Компания Хеликон”, Россия). Окрашенные бромистым этидием ПЦР-продукты на геле визуализировали УФ-светом в системе гель-регистрации Kodak Edas 290 (США). Размеры полученных фрагментов ДНК (аллелей микросателлитных локусов) определяли с помощью программы GelAnalyzer 19.1 [9].
Таблица 1.
Характеристика микросателлитных локусов у даурского журавля
| Локус | Мотив | A | Аллели | HO | HE | Равновесие Харди–Вайнберга |
|---|---|---|---|---|---|---|
| Gram22 | (AAAC)9 | 6 | 160, 164, 168, 172, 176, 180 | 0.627 | 0.753 | ns |
| Gram30 | (AAGG)7 | 10 | 158, 162, 166, 170, 174, 178, 182, 186, 190, 194 | 0.745 | 0.796 | ns |
| Gpa12 | (GATA)11 | 8 | 202, 210, 214, 218, 222, 226, 230, 234 | 0.843 | 0.841 | ns |
| Gpa38 | (CTAT)13 | 5 | 186, 190, 194, 198, 202 | 0.706 | 0.736 | ns |
| Gpa39 | (GA)2 (GATA)13 |
7 | 100, 108, 112, 116, 120, 124, 128 | 0.804 | 0.741 | ns |
| Gj4066 | (ATAG)9 | 5 | 141, 145, 149, 153, 157 | 0.608 | 0.736 | *(0.026) |
| Gj2298 | (CTG)13 | 2 | 142, 145 | 0.510 | 0.398 | *(0.046) |
Секвенирование контрольного региона мтДНК
Амплификацию всего контрольного региона размером 1132 пн осуществляли с использованием прямого LC16575 (5'-ACA AAA GAA ACC CCC AAA CTC A-3') и обратного HC01342 (5'-AAG AAT TCT GCG GAT ACT TGC ATG T-3') [10] праймеров по следующему протоколу: первичная денатурация 3 мин при 94°C, затем 29 циклов амплификации, включающих денатурацию при 94°C – 45 с, отжиг праймеров при 58°C – 1 мин и элонгацию при 72°C – 1 мин, и финальная элонгация 10 мин при 72°C. Так же, как и в случае микросателлитных локусов, ПЦР контрольного региона проводили с помощью реагентов GenPak PCR Core. Фрагменты амплификации проверяли на электрофорезе в 1.5%-ном агарозном геле и секвенировали в прямом и обратном направлениях на генетическом анализаторе ABI 3130 (Applied Biosystems, США) в ЗАО Евроген (Москва, Россия). Нуклеотидные последовательности идентифицированных гаплотипов были депонированы в Генбанк под номерами MN929113–MN929130.
Статистический анализ
Расчеты параметров внутрипопуляционного разнообразия, соответствия распределения генотипов микросателлитных локусов согласно равновесию Харди–Вайнберга, F-статистик межпопуляционной генетической дифференциации, потока генов (Nm) и анализ молекулярной дисперсии (AMOVA) проводили с помощью надстройки для электронной таблицы MS Excel–GenAlEx 6.5 [11]. Для выявления популяционной структуры использовали алгоритм байесовской кластеризации в программе STRUCTURE 2.3.4 [12]. Оценку вероятного числа генетических кластеров К по методу Эванно, структуризацию и визуализацию результатов осуществляли в CLUMPAK [13]. Для каждого предполагаемого значения K (от 2 до 4) проводили пять независимых повторов симуляций с 100 000 итерациями и предшествующим периодом разогрева в 10 000 итераций. В расчетах использовали опции популяционной привязки данных LOCPRIOR = 1, смешанную модель (admixture model) и независимые частоты аллелей между выборками. Для кластеризации индивидуальных многолокусных генотипов по методу главных компонент (Principal Component Analysis, PCA) пользовались пакетами adegenet [14] и ggplot2 [15] в RStudio 2021.09.1.372 [16] на основе R 4.1.2 [17]. Выравнивание, редактирование и сборку сиквенсов после прямого и обратного прочтений проводили относительно друг друга и единственного референса из Генбанка (FJ769852) по алгоритму MAFFT [18] в программе Geneious 9.1.8 [19]. Гаплотипическое и нуклеотидное разнообразие, попарные сравнения нуклеотидных различий, тесты селективной нейтральности Таджимы, Фу и Ли, число мигрантов на поколение (Nm) и коэффициент генетической дифференциации FST рассчитывали в программе DnaSP 6.11.01 [20]. Сеть гаплотипов была построена по TCS-алгоритму [21] в PopART 1.7 [22].
РЕЗУЛЬТАТЫ
Генетическая изменчивость и дифференциация по микросателлитным локусам
В совокупной выборке (51 особь) даурских журавлей по семи полиморфным локусам идентифицировано от 2 (Gj2298) до 10 (Gram30) аллелей (табл. 1). Общее число аллелей составило 43. Пять локусов находились в равновесном состоянии генотипов согласно закону Харди–Вайнберга, а по двум наблюдались отклонения, значимые на уровне вероятности менее 0.05%, в сторону недостатка (по Gj4066) и избытка (по Gj2298) гетерозигот. Значения наблюдаемой (HO) и ожидаемой (HE) гетерозиготности варьировали от наименьших по локусу Gj2298 (HO = 0.510, HЕ = 0.398) до наибольших по локусу Gpa12 (HO = 0.843, HЕ = 0.841).
Среднее число аллелей на локус для обеих популяций составило 5.8, уровни гетерозиготности в среднем оказались высокими: HO = 0.696 ± 0.033, HЕ = 0.707 ± 0.037. Несмещенные оценки ожидаемой гетерозиготности (uHE) в обеих популяциях были близкими (табл. 2). Из 43 аллелей общими для западной и восточной популяций оказались 38; уникальные аллели обнаружены только в восточной популяции (Gram30190, Gram30194, Gpa12202, Gpa39100 и Gpa39108).
Таблица 2.
Генетическое разнообразие западной и восточной популяций даурского журавля по микросателлитным локусам
| Популяция | NA | HO | HE | uHE | FIS | FST |
|---|---|---|---|---|---|---|
| Западная | 5.4 | 0.714 ± 0.030 | 0.710 ± 0.044 | 0.728 ± 0.045 | –0.029 | |
| Восточная | 6.1 | 0.677 ± 0.010 | 0.705 ± 0.064 | 0.716 ± 0.065 | 0.017 | |
| В среднем | 5.8 | 0.696 ± 0.033 | 0.707 ± 0.037 | 0.722 ± 0.030 | –0.006 | 0.013 ± 0.004 |
Значения коэффициента инбридинга FIS указывают на небольшой избыток гетерозигот в западной популяции (2.9%) и небольшой их недостаток в восточной (1.7%), однако в целом для вида этот показатель был близок к нулю (FIS = –0.006). Межпопуляционная генетическая дифференциация даурского журавля по микросателлитным локусам оказалась низкой и достоверно не отличалась от нуля (FST = 0.013, P = 0.369, несмещeнная оценка GST = 0.001, P = 0.359) (табл. 2). По данным AMOVA 96% генетической изменчивости вида сосредоточено внутри особей и 4% – между ними. Поток генов (Nm) между западной и восточной популяциями A. vipio измерялся 29.09 мигрантами на поколение.
Структурированность популяций по микросателлитным локусам
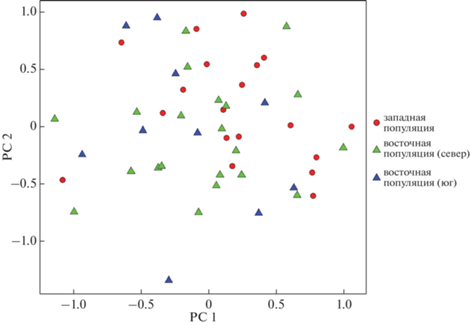
Результаты кластеризации байесовскими методами в программе STRUCTURE для наиболее вероятных значений числа генетических кластеров K от двух до четырех при оптимальном K = 3 не обнаружили различий в популяционной структуре западной и восточной популяций (рис. 2). Фактически данные байесовского анализа показали существование единой популяции вида на территории России. Такая же однородная генетическая структура даурского журавля выявлена и методом РСА. Поскольку птицы из северной и южной частей восточной популяции используют один пролетный путь, но географически разобщены (рис. 1), для кластеризации методом главных компонент мы выделили их в отдельные группы. Однако это тоже не отразилось на пространственном распределении генотипов – совокупность точек, обозначающих многолокусные индивидуальные генотипы западной, северной и южной восточной групп даурских журавлей, перекрывается на координатной плоскости PC1–PC2 (рис. 3).
Генетическое разнообразие гаплотипов контрольного региона мтДНК
В анализ были включены нуклеотидные последовательности всего контрольного региона (1132 пн) 10 и 15 особей из западной и восточной популяций даурского журавля соответственно. Среди этих 25 особей идентифицировано 18 гаплотипов (табл. 3). Только один из них (h6) был общим, а остальные встречались либо в одной, либо в другой популяции. Всего к западной популяции относилось восемь гаплотипов, а к восточной – 11.
Таблица 3.
Распределение 18 гаплотипов контрольного региона в популяциях даурского журавля
| Гаплотип | Номер в Генбанке | Популяция | Всего особей | |
|---|---|---|---|---|
| западная | восточная | |||
| h1 | MN929113 | 1 | 1 | |
| h2 | MN929114 | 2 | 2 | |
| h3 | MN929115 | 2 | 2 | |
| h4 | MN929116 | 1 | 1 | |
| h5 | MN929117 | 1 | 1 | |
| h6 | MN929118 | 1 | 2 | 3 |
| h7 | MN929119 | 1 | 1 | |
| h8 | MN929120 | 2 | 2 | |
| h9 | MN929121 | 1 | 1 | |
| h10 | MN929122 | 1 | 1 | |
| h11 | MN929123 | 1 | 1 | |
| h12 | MN929124 | 1 | 1 | |
| h13 | MN929125 | 1 | 1 | |
| h14 | MN929126 | 2 | 2 | |
| h15 | MN929127 | 1 | 1 | |
| h16 | MN929128 | 1 | 1 | |
| h17 | MN929129 | 1 | 1 | |
| h18 | MN929130 | 2 | 2 | |
| 10 | 15 | 25 | ||
Обе популяции характеризовались высоким гаплотипическим и низким нуклеотидным разнообразием. Средние значения этих показателей составили соответственно Hd = 0.973 ± 0.018 и π = 0.00662 ± 0.00064 (табл. 4), что указывает на высокое сходство гаплотипов даурского журавля. Среднее число нуклеотидных замен в гаплотипах (k) равнялось 7.487. Распределение нуклеотидных различий при попарных сравнениях гаплотипов соответствовало модели популяционной экспансии (рис. 4), однако тесты отклонения от селективной нейтральности Таджимы (D = –0.81269) и Фу и Ли (F = –0.8890) не были достоверно значимыми. Уровень генетической дифференциации восточной и западной популяций по контрольному региону превышал таковой по микросателлитным локусам и достоверно отличался от нуля (FST = 0.041, P = 0.05), а число мигрантов на поколение по мтДНК, соответственно, было ниже: Nm = 5.83. По данным AMOVA 97.4% генетической изменчивости даурских журавлей сконцентрировано внутри особей и 2.6% – между ними.
Таблица 4.
Показатели изменчивости контрольного региона и тесты на селективную нейтральность в популяциях даурского журавля
| Популяция | Hd | π | k | D | F |
|---|---|---|---|---|---|
| Западная | 0.956 ± 0.059 | 0.00635 ± 0.00065 | 7.178 | –0.36410 ns | –0.46458 ns |
| Восточная | 0.962 ± 0.034 | 0.00655 ± 0.00659 | 7.410 | –0.30614 ns | –0.00950 ns |
| В среднем | 0.973 ± 0.018 | 0.00662 ± 0.00064 | 7.487 | –0.81269 ns | –0.8890 ns |
Рис. 4.
График распределения нуклеотидных различий гаплотипов контрольного региона даурского журавля согласно модели недавнего увеличения численности вида. По оси абсцисс приводится число нуклеотидных замен при попарном сравнении гаплотипов, по оси ординат – частота. Пунктирная линия соответствует ожидаемому распределению, сплошная – наблюдаемому.

Сеть гаплотипов, рассчитанная по TCS-алгоритму, демонстрирует кольцевую структуру из западных и восточных вариантов без формирования каких-либо географических кластеров (рис. 5). Северо-восточный гаплотип h4 – единственный из всех обнаруженных образовывал узел, связанный как с другими восточными северными, так и с восточными южными и с западными вариантами.
Рис. 5.
Сеть гаплотипов контрольного региона мтДНК даурского журавля, построенная по TCS-алгоритму [21]. Размер кружков пропорционален числу особей, засечки на соединяющих линиях соответствуют числу мутационных событий между гаплотипами, сектора диаграмм представляют частоты гаплотипов в популяциях.

ОБСУЖДЕНИЕ
Полученные нами данные о генетической структуре двух популяций даурского журавля, использующих разные места гнездования и зимовки и соответственно разные пролетные пути, указывают на однородность генофонда этого редкого вида птиц. Обе популяции – как сокращающаяся западная, так и находящаяся в фазе роста восточная – характеризуются высоким уровнем генетического разнообразия по ядерным микросателлитным локусам и контрольному региону мтДНК, сопоставимым с таковым у широкоареальных видов журавлей с большой численностью – канадского Antigone canadensis [23], серого Grus grus [24], красавки Anthropoides virgo [25, 26], но превышающим данный показатель у гораздо более редких американского Grus americana [6, 27] и японского G. japonensis [28] журавлей.
Низкая генетическая дифференциация, отсутствие четко выраженной популяционной структуры и достаточно интенсивный поток генов между западной и восточной группировками даурского журавля указывают на отсутствие репродуктивной изоляции между популяциями, использующими разные пролетные пути. Применение дистанционных методов слежения позволило выявить случай изменения мест зимовки самкой даурского журавля из западной группировки: свою первую зиму она провела в традиционном месте на озере Поянг в Китае (2016 г.), а последующие (2017–2020 гг.) – на местах зимовки восточной популяции в Корее и Японии, однако свое место гнездования в Юго-Восточном Забайкалье из года в год она не меняла (https://savingcranes.org/the-journey-of-white-naped-crane-borzya). Брачную пару эта самка сформировала на местах зимовки восточной популяции, следовательно ее партнер, вероятно принадлежавший к восточной популяции, изменил место гнездования. Известны и другие достоверные случаи смены даурскими журавлями мест зимовки и путей миграции. Скорее всего, изменения маршрутов больше присущи неполовозрелым особям в ходе их первой самостоятельной, не связанной с родителями, миграции. Таким образом, объединение птиц на общих зимовках с представителями другого миграционного потока может привести к кардинальной смене места гнездования.
На состояние западной популяции, обитающей в аридных условиях, огромное влияние оказывают климатические циклы продолжительностью около 30 лет с чередованием влажных и засушливых периодов, в процессе которых условия обитания птиц могут радикально меняться от благоприятных до крайне неблагоприятных [29–33]. Это вызывает масштабные перемещения особей и изменение пространственной структуры населения как внутри западной популяции даурского журавля, так, вероятно, и внутри вида в целом [31]. Стремительное падение численности западной группировки в 2000–2017 гг. произошло в неблагоприятный засушливый период, когда подавляющая часть ее местообитаний высохла, что позволяет предположить перемещение части западных даурских журавлей на места гнездования восточной популяции, где условия обитания в то время были относительно благоприятны, а численность довольно быстро росла [5, 31]. Подобные перемещения особей, безусловно, способствуют гомогенизации генетической структуры вида.
Слабые или средние генетические различия показаны и для других мигрирующих видов журавлей – как широко распространенных, так и редких. Так, по микросателлитным локусам значение FST между популяциями западного подвида серого журавля составляет 0.012 [34], а между выборками западного и восточного подвидов 0.011 [24]. Такая же низкая (0.3–1.1%) генетическая дифференциация между подвидами серого журавля и по контрольному региону мтДНК [35]. У красавки – вида с большим, но фрагментированным в России ареалом – различия в генетической структуре по микросателлитным локусам (FST = = 0.052 [25]) выше, чем у более многочисленного и распространенного серого журавля и как показано в настоящей работе редкого даурского журавля, населяющего более компактную территорию. По контрольному региону уровень генетической дифференциации красавки на всем ареале (FST = 0.075 [26]) также превышает таковой у даурского журавля. Генетические различия между зимующими популяциями черного журавля G. monacha [36], а также между континентальной мигрирующей и оседлой островной популяциями японского журавля [10], судя по распределению гаплотипов контрольного региона в медианных сетях и филогенетических деревьях, практически отсутствуют.
Полученный нами график распределения попарных различий нуклеотидов, свидетельствующий об увеличении численности вида в недавнем прошлом, не подкрепляется достоверными тестами на селективную нейтральность. Однако эти тесты очень чувствительны к размеру выборок, которые у нас недостаточно велики. При этом в TCS-сети гаплотипов контрольного региона отсутствуют звездообразные структуры, характерные для популяций, прошедших через “бутылочное горлышко”, что также свидетельствует в пользу преждевременности выводов о популяционной экспансии даурского журавля в прошлом.
В целом высокие показатели генетической изменчивости даурского журавля по изученным молекулярно-генетическим маркерам указывают на стабильное состояние его генофонда на разных пролетных путях, однако в связи с сокращением численности западной популяции и в целях поддержания восточной охранные меры для этого редкого вида журавлей актуальны и необходимы.
Авторы благодарят Е.Ю. Гаврикову и Н.В. Кузнецову за биологический материал от даурских журавлей, содержащихся на Станции реинтродукции редких видов журавлей Хинганского государственного природного заповедника.
Работа выполнена при финансовой поддержке гранта РФФИ № 17-04-01287, в рамках Государственного задания 0112-2019-0001, а также с частичной поддержкой Комплексной международной научно-производственной программы Евроазиатской региональной ассоциации зоопарков и аквариумов “Сохранение журавлей Евразии” (2012–2019 и 2021 гг.).
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
BirdLife International. 2018. Antigone vipio. The IUCN Red List of threatened species 2018: e.T22692073A131927305. https://doi.org/10.2305/IUCN.UK.2018-2.RLTS.T22-692073A131927305.en
Meine C.D., Archibald G.W. The Cranes: Status Survey and Conservation Action Plan. IUCN, Gland, Switzerland, and Cambridge, UK, 1996. 282 p.
Mirande C.M., Harris J.T. (eds) // Crane conservation strategy. Baraboo, Wisconsin, USA: Intern. Crane Foundation, 2019. 454 p.
Арчибальд Д. Статус журавлей мира в 2021 г. // Журавли Евразии (распространение, биология). 2021. Вып. 6. С. 31–67.
Горошко О.А., Сурмач С.Г. Даурский журавль // Красная книга Российской Федерации. Животные. 2-е изд. М.: ФГБУ ВНИИ “Экология”. 2021. С. 684–686.
Jones K.L., Henkel J.R., Howard J.J. et al. Isolation and characterization of 14 polymorphic microsatellite DNA loci for the endangered Whooping crane (Grus americana) and their applicability to other crane species // Conserv. Gen. Res. 2010. V. 2. № 1. P. 251–254. https://doi.org/10.1007/s12686-010-9196-3
Meares K., Dawson D., Horsburgh G.J. et al. Characterisation of 14 blue crane Grus paradisea (Gruidae, AVES) microsatellite loci for use in detecting illegal trade // Conserv. Genet. 2008. V. 9. P. 1363–1367. https://doi.org/10.1007/s10592-007-9490-0
Zou H.F., Dong H.Y., Kong W.Y. et al. Characterization of 18 polymorphic microsatellite loci in the red-crowned crane (Grus japonensis), an endangered bird // Anim. Sci. J. 2010. V. 81. № 4. P. 519–522. https://doi.org/10.1111/j.1740-0929.2010.00779.x
GelAnalyzer 19.1 (www.gelanalyzer.com) by Istvan Lazar Jr., PhD and Istvan Lazar Sr., PhD, CSc.
Hasegawa O., Takada S., Yoshida M.C., Abe S. Variation of mitochondrial control region sequences in three crane species, the red-crowned crane Grus japonensis, the common crane G. grus and the hooded crane G. monacha // Zool. Sci. 1999. V. 16. P. 685–692.
Peakall R., Smouse P.E. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research – an update // Bioinformatics. 2012. V. 28. № 19. P. 2537–2539. https://doi.org/10.1093/bioinformatics/bts460
Pritchard J.K., Matthew S., Peter D. Inference of population structure using multilocus genotype data: Linked loci and correlated allele frequencies // Genetics. 2000. V. 164. № 4. P. 1567–1587. https://doi.org/10.3410/f.1015548.197423
Kopelman N.M., Mayzel J., Jakobsson M. et al. Clumpak: A program for identifying clustering modes and packaging population structure inferences across K // Mol. Ecol. Res. 2015. V. 15. № 5. P. 1179–1191. https://doi.org/10.1111/1755-0998.12387
Jombart. T. adegenet: a R package for the multivariate analysis of genetic markers // Bioinformatics. 2008. V. 24. P. 1403–1405. https://doi.org/10.1093/bioinformatics/btn129
Wickham H. ggplot2: Elegant Graphics for Data Analysis. N.Y.: Springer-Verlag, 2016.
RStudio Team. RStudio: Integrated Development Environment for R. RStudio, PBC, Boston, MA, 2021. http://www.rstudio.comhttp://www.rstudio.com
R Core Team. R: A Language and Environment for Statistical Computing. R Foundation for Statistical Computing. Austria: Vienna, 2021. https://www.R-project.org
Katoh K., Misawa K., Kuma K., Miyata T. MAFFT: A novel method for rapid multiple sequence alignment based on fast Fourier transform // Nucl. Acids Res. 2002. V. 30. № 14. P. 3059–3066. https://doi.org/10.1093/nar/gkf436
Kearse M., Moir R., Wilson A. et al. Geneious Basic: An integrated and extendable desktop software platform for the organization and analysis of sequence data // Bioinformatics. 2012. V. 28. № 12. P. 1647–1649. https://doi.org/10.1093/bioinformatics/bts199
Librado P., Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data // Bioinformatics. 2009. V. 25. № 11. P. 1451–1452. https://doi.org/10.1093/bioinformatics/btp187
Clement M., Snell Q., Walke P. et al. TCS: estimating gene genealogies // Proc. 16th Int. Parallel Distrib. Process Symp. 2002. V. 2. P. 184.
Leigh J.W., Bryant D. PopART: Full-feature software for haplotype network construction // Methods Ecol. Evol. 2015. V. 6. № 9. P. 1110–1116. https://doi.org/10.1111/2041-210X.12410
Rhymer J.M., Fain M.G., Austin J.E. et al. Mitochondrial phylogeography, subspecific taxonomy, and conservation genetics of sandhill cranes (Grus canadensis; Aves: Gruidae) // Conserv. Gen. 2001. V. 2. P. 203–218.
Мудрик Е.А., Кашенцева Т.А., Редчук П.С., Политов Д.В. Данные по микросателлитной изменчивости подтверждают низкую генетическую дифференциацию западного и восточного подвидов серого журавля (Grus grus L.) // Мол. биология. 2015. Т. 49. № 2. С. 297–304. https://doi.org/10.7868/S002689841502010X
Mudrik E.A., Ilyashenko E.I., Goroshko O.A. et al. The Demoiselle crane (Anthropoides virgo) population genetic structure in Russia // Vavilov J. Genetics and Breeding. 2018. V. 22. № 5. P. 586–592. https://doi.org/10.18699/VJ18.398
Mudrik E.A., Ilyashenko E.I., Ilyashenko V.Y. et al. Genetic diversity and differentiation of the widespread migratory Demoiselle Crane, Grus virgo, on the northern edge of the species’ distribution // J. Ornithol. 2022. V. 163. № 1. P. 291–299. https://doi.org/10.1007/s10336-021-01919-4
Glenn T.C., Wolfgang S., Braun M.J. Effects of a population bottleneck on whooping crane mitochondrial DNA variation // Conserv. Biol. 1999. V. 13. № 5. P. 1097–1107.
Sugimoto T., Hasegawa O., Azuma N. et al. Genetic structure of the endangered red-crowned cranes in Hokkaido, Japan and conservation implications // Conserv. Genet. 2015. V. 16. P. 1395–1401. https://doi.org/10.1007/s10592-015-0748-7
Горошко О.А., Цэвээнмядаг Н. Данные о влиянии засух на популяцию даурских журавлей // Наземные позвоночные Даурии: Сб. науч. трудов Гос. природного биосферного заповедника “Даурский”. Вып. 3. Чита: Поиск, 2003. С. 121–130.
Горошко О.А. Даурский журавль // Красная книга Забайкальского края. Животные. Новосибирск: Новосиб. изд. дом, 2012. С. 120–122.
Горошко О.А. Динамика популяций и качества местообитаний птиц в Даурии в условиях многолетних климатических циклов // Ареалы, миграции и другие перемещения диких животных. Владивосток: ООО “Рея”, 2014. С. 74–80.
Горошко О.А. Динамика и современное состояние популяций журавлей в Даурии (Забайкалье, Монголия) // Журавли Евразии: биология, охрана, управление. 2015. Вып. 5. С. 116–134.
Горошко О.А. Результаты учетов журавлей в Юго-Восточном Забайкалье, Россия, в 2016–2020 гг. // Журавли Евразии (распространение, биология). 2021. Вып. 6. С. 82–105.
Haase M., Holtje H., Blahy B. et al. Shallow genetic population structure in an expanding migratory bird with high breeding site fidelity, the Western Eurasian Crane Grus grus grus // J. Ornithol. 2019. V. 160. P. 965–972. https://doi.org/10.1007/s10336-019-01688-1
Haase M., Ilyashenko V. A glimpse on mitochondrial differentiation among four currently recognized subspecies of the common crane Grus grus // Ardeola. 2012. V. 59. № 1. P. 131–136. https://doi.org/10.13157/arla.59.1.2012.131
Zhang L., Zhou L., Dai Y. Genetic structure of wintering Hooded Cranes (Grus monacha) based on mitochondrial DNA D-loop sequences // Chinese Birds. 2012. V. 3. № 2. P. 71–81. https://doi.org/10.5122/cbirds.2012.0012
Дополнительные материалы отсутствуют.