Генетика, 2022, T. 58, № 6, стр. 685-699
Генетическая идентификация и анализ родственных отношений содержащихся в неволе соколов
М. М. Белоконь 1, *, Ю. С. Белоконь 1, А. В. Нечаева 1, Н. А. Сильвестров 2, Е. И. Сарычев 3, И. Р. Бёме 2
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
2 Московский государственный университет им. М.В. Ломоносова
119991 Москва, Россия
3 ООО “Питомник редких видов птиц ВИТАСФЕРА”
140170 д. Слободино, Московская область, Россия
* E-mail: belokon@vigg.ru
Поступила в редакцию 02.12.2021
После доработки 14.01.2022
Принята к публикации 18.01.2022
- EDN: DBHMJQ
- DOI: 10.31857/S0016675822060029
Аннотация
Для трех видов содержащихся в неволе соколов – кречета Falco rusticolus, балобана F. cherrug и сапсана F. peregrinus апробирована универсальная система генетической идентификации на основании многолокусных микросателлитных генотипов. Проанализированы коэффициенты родства в видовых группах птиц из питомников. Набор из 13 ядерных микросателлитных локусов позволяет надежно идентифицировать отдельных особей, устанавливать принадлежность особей к родственным группам, в отдельных случаях дает возможность распознавать межвидовые гибриды. Результаты данного исследования могут использоваться в племенной работе, а также быть внедрены в практику судебной молекулярно-генетической экспертизы.
История содержания пернатых хищников в неволе и использования их для охоты насчитывает, согласно археологическим данным, по меньшей мере 5000 лет [1]. В 2016 г. соколиная охота была занесена в список всемирного нематериального культурного наследия ЮНЕСКО [2]. Во многих странах Ближнего Востока, Северной Африки и степных регионах Азии соколиная охота практикуется по сегодняшний день как вид спорта и хобби. Сохраняется высокий коммерческий спрос на ловчих птиц. Три вида крупных соколов – кречет (Falco rusticolus L., 1758), балобан (F. cherrug Gray, 1834) и сапсан (F. peregrinus Тunstall, 1771) – пользуются большой популярностью у современных сокольников. Потребность в птицах для охотничьих целей удовлетворяется как за счет разведения в питомниках, так и за счет незаконного изъятия из природы. Основные регионы отлова соколов – Сибирь, Дальний Восток, Казахстан, Монголия, Афганистан и ряд других стран Центральной Азии. Незаконный отлов птиц остается на стабильно высоком уровне, о чем свидетельствует многолетняя статистика задержаний браконьеров и контрабандистов [3]. В то же время естественные популяции соколов находятся в уязвимом положении из-за сокращения площади пригодной для их обитания среды, химического загрязнения хлорорганическими соединениями и оскудения кормовой базы. В целях охраны балобан был внесен в Красный список угрожаемых видов МСОП, все три вида занесены в Приложение 1 Конвенции о международной торговле видами дикой фауны и флоры, находящимися под угрозой исчезновения (СИТЕС) [4–6]. Торговля этими видами требует наличия документов, подтверждающих происхождение птиц из питомников. Нелегальные попытки вывоза птиц нередко сопровождаются подделкой сертификатов о происхождении и маркировочных колец. Это обстоятельство делает особенно актуальным разработку и внедрение системы генетической идентификации и паспортизации соколов.
Разведение соколов в питомниках преследует две основные цели – удовлетворение спроса любителей соколиной охоты и восстановление численности природных популяций [7–9]. При разведении птиц в питомниках с целью восполнения природных популяций очень важно иметь генетически довольно разнообразную выборку производителей высокой численности с учетом места их происхождения и последующего выпуска птенцов. Также следует избегать скрещиваний между близкородственными особями, которые ведут к появлению инбредного потомства. С другой стороны, разведение птиц для приручения и охоты преследует другие цели – красивый внешний вид, выдающиеся ловчие качества, необычные цветовые морфы. Для этих целей допустимо скрещивать родственных птиц, а также выводить гибридов разных видовых сочетаний. В каждом из этих случаев необходимо осуществлять контроль разведения не только путем маркировки птиц кольцами и записями родословных в племенных книгах, но и ведением генетической паспортизации. Генетический паспорт птицы, наряду с выпиской из племенной книги, мог бы служить доказательством ее происхождения при продаже или смене владельца.
В Российской Федерации успешно осуществляются программы генетической паспортизации редких и исчезающих видов журавлей [10–12]. Недавно стали разрабатываться подходы к оценке генетического разнообразия выпускаемых в природу хищных птиц [13, 14].
Наиболее удобными маркерами для генетической паспортизации являются ядерные микросателлитные локусы. По сравнению с другими маркерами они обладают рядом положительных свойств, таких как нейтральность и высокая скорость мутирования, что обеспечивает наличие полиморфизма в популяциях; кодоминантное наследование, позволяющее выявлять оба аллеля в генотипе и определять генетический вклад обоих родителей; небольшой размер амплифицируемых фрагментов и, следовательно, возможность использования в качестве источника ДНК неинвазивных или даже деградированных образцов; относительная дешевизна и простота в использовании [15, 16].
Целью нашей работы был подбор и тестирование универсального набора ядерных микросателлитных локусов для возможностей генетической идентификации особей и оценки степени родства среди соколов.
МАТЕРИАЛЫ И МЕТОДЫ
Образцы для исследования были предоставлены ООО “Питомник редких видов птиц ВИТАСФЕРА” (Московская обл.), Зоопарком “Роев Ручей” (г. Красноярск), а также Следственным отделом по г. Раменское ГСУ СК РФ по Московской области (46 образцов были предоставлены для проведения судебной молекулярно-генетической экспертизы в рамках расследования по уголовному делу о незаконном отлове и содержании птиц). Всего в нашем распоряжении оказался 161 образец: 80 кречетов, 72 балобана и девять сапсанов.
Выборка кречетов из питомника “Витасфера” состояла из 49 особей, часть из которых представлена изъятыми в природе птенцами по разрешению Росприроднадзора в рамках формирования искусственной резервной популяции, а другая часть, преимущественно состоящая из молодых самок, – птицами, переданными в питомник Таможенной службой РФ после препятствия незаконному вывозу из страны. Следственным отделом предоставлены образцы от девяти кречетов, как неродственных, так и находящихся в разной степени родства согласно документам. Образцы подскорлуповых оболочек 22 птенцов кречета от восьми пар родителей предоставлены питомником “Витасфера”.
Выборка балобанов состояла из предоставленных Следственным отделом 35 птиц, неродственных и находящихся в разной степени родства согласно документам, и 37 птенцов балобана от 10 пар родителей из питомника “Витасфера”.
Сапсаны в нашем исследовании представлены пятью птенцами от одной пары родителей из питомника “Витасфера”, двумя не родственными друг другу взрослыми птицами из зоопарка “Роев Ручей” и двумя предположительно родственными птицами, предоставленными Следственным отделом.
В качестве источников ДНК использовали кровь, собранную у птиц прижизненно (фиксированную в спирте или высушенную на фильтровальной бумаге), фиксированные в спирте фрагменты мышц от погибших птиц и сосуды аллантоиса из подскорлуповых оболочек после вылупления птенцов. Для выделения ДНК из крови и мышц использовали наборы Diatom™ DNA Prep 100 (ООО “Лаборатория Изоген”, Россия) согласно приложенной инструкции. Выделение проводили из 3–5 мкл цельной крови, или эквивалентного количества крови из высушенного образца. Образцы мышц массой 15–20 мг предварительно измельчали путем растирания в лизирующем буфере. Время инкубации при лизисе мышечной ткани было увеличено до 3 ч. Экстракцию ДНК из кровеносных сосудов аллантоиса проводили с помощью ионообменной смолы Chelex100 (20%-ная суспензия в бидистиллированной воде) [17]. Образцы инкубировали при 99°C в течение 1–3 ч, периодически встряхивая, затем центрифугировали, жидкую фракцию переносили в чистые пробирки и в дальнейшем использовали для ПЦР. Полученные образцы ДНК хранили при температуре –20°C.
Пол птиц определяли путем ПЦР со специфическими праймерами P2/P8 для участка интрона гена хромохеликазы (CHD), локализованного на половых хромосомах [18, 19]. Для сбора реакции использовали наборы для ПЦР-амплификации фрагментов ДНК GenePak® PCR Core (ООО “Лаборатория Изоген”). ПЦР включала: начальную денатурацию – 3 мин при 94°С; 35 циклов: 30 с при 94°С, 30 с при 50°С, 45 с при 72°С; финальную элонгацию – 3 мин при 72°С; по окончании реакции смесь охлаждалась до 4°С. Амплифицированные фрагменты разделяли электрофоретически в 2.5%-ном агарозном геле с использованием ТАЕ-буфера.
Для установления многолокусных генотипов птиц использовали 15 ядерных микросателлитных локусов, разработанных для сапсана и кречета: NVH fp5, NVH fp13, NVH fp31, NVH fp46-1, NVH fp54, NVH fp79-1, NVH fp79-4, NVH fp82-2, NVH fp86-2, NVH fp89, NVH fp92-1 (GenBank Accession no. AF118420–AF118423; AF118425–AF118431) [20]; NVH fp347 (GenBank Accession no. AF448412) [21]; NVH fr34, NVH fr142, NVH fr164-1 (GenBank Accession no. AF200200, AF200201, AF200204) [22]. ПЦР-амплификацию фрагментов ДНК проводили с использованием наборов GenePak® PCR Core (ООО “Лаборатория Изоген”). Программа амплификации для группы локусов fp5, fp31, fp46-1, fp54, fp79-1, fp82-2, fp86-2, fp89, fp347, fr34, fr142 включала начальную денатурацию при температуре 94°С – 4 мин, затем 30 циклов: 94°С – 40 с, 55°С – 40 с, 72°С – 40 с и финальную элонгацию при 72°С в течение 5 мин, по окончании реакции смесь охлаждалась до 4°С. Для группы локусов fp13, fp79-4, fp92-1, fr164-1: начальная денатурация при температуре 94°С – 4 мин, затем 35 циклов: 94°С – 40 с, 52°С – 40 с, 72°С – 40 с, финальная элонгация при 72°С в течение 5 мин, по окончании реакции смесь охлаждалась до 4°С. Продукты ПЦР хранились при температуре 4°С. Амплифицированные фрагменты разделяли электрофоретически в 6%-ном полиакриламидном геле, в буферной системе ТБЕ, в течение 2–2.5 ч, в зависимости от длин исследуемых фрагментов. Гели окрашивали раствором бромистого этидия. Визуализацию и документирование электрофоретического разделения продуктов ПЦР проводили в ультрафиолетовом свете при длине волны 312 нм с помощью системы гель-документирования. Длину полученных фрагментов определяли в программе Photo-Capt V 12.4 (Vilber Luormat). В качестве маркера длины фрагментов использовали ДНК плазмиды pBR322 E. coli, обработанную эндонуклеазой рестрикции HpaII.
По результатам генотипирования птиц были составлены таблицы многолокусных генотипов. В программе GenAlEx [23, 24] были получены параметры популяционно-генетической изменчивости для каждого из трех видов. Рассчитаны частоты аллелей, среднее число аллелей на локус (NA), среднее эффективное число аллелей на локус (NE), значения наблюдаемой (HO) и ожидаемой (HE) гетерозиготностей, индекс фиксации (F) и доля полиморфных локусов (P). Соответствие соотношения распределения генотипов равновесию Харди–Вайнберга проверяли при помощи критерия χ2. Также определяли значимость отклонения индекса фиксации от нулевого значения. В случае недостатка гетерозигот проводили проверку на наличие нуль-аллелей с помощью программы Micro-Checker по алгоритмам Oosterhout (2004) и Brookfield (1996) [25].
Для определения вероятности возникновения в выборке двух особей с идентичными генотипами и достаточного минимального набора микросателлитных локусов для точной индивидуальной идентификации при свободном скрещивании особей в программе GenAlEx рассчитывали параметры PI (Probability of Identity) и PIsibs при допущении родственных отношений между особями. Также рассчитывали вероятности исключения родительства (Probability of Exclusion): P1 – в случае, когда известны генотипы обоих предполагаемых родителей; P2 – в случае, когда известен генотип одного из предполагаемых родителей; P3 – в случае, когда генотипы родителей неизвестны.
Для анализа родственных отношений в программе GenAlEx по полиморфным локусам для каждого вида были рассчитаны коэффициенты родства RI (Ritland, 1996) [26], LRM (Lynch & Ritland, 1999) [27], QGM (Queller & Goodnight, 1989) [28]. Значение коэффициента LRM было умножено на два, чтобы его максимальное значение равнялось единице, и значения всех трех коэффициентов находились в пределах от –1 до 1.
РЕЗУЛЬТАТЫ
Пол исследованных образцов
Выборка взрослых кречетов из питомника “Витасфера” состояла из 11 самцов и 38 самок. Среди птенцов соотношение полов было равным – 11 самцов и 11 самок. В выборке кречетов, предоставленной Следственным отделом, семь птиц оказались самцами, а две – самками.
Взрослые балобаны в нашем исследовании представлены 17 самцами и 18 самками, предоставленными Следственным отделом. Среди птенцов из питомника “Витасфера” 2015 г. рождения определены пять самцов и три самки, 2017 г. рождения – 13 самцов и 16 самок. Общая выборка балобанов представлена в равной мере самцами и самками.
Взрослые сапсаны представлены одним самцом и тремя самками. Среди птенцов 2017 г. рождения выявлены один самец и четыре самки. Ввиду того, что использованные в нашей работе микросателлитные локусы имеют аутосомную локализацию [20, 21, 29–31], соотношение полов в выборках не влияет на оценку параметров генетической изменчивости.
Анализ генетического разнообразия
На основании многолокусных генотипов были рассчитаны частоты аллелей микросателлитных локусов для тотальных выборок каждого из видов (табл. 1). Локусы fr142 и fr164-1 оказались мономорфными у всех трех видов с общими аллелями, поэтому они не являются информативными ни для идентификации отдельных птиц, ни для различения видов. Локусы fp5, fp31, fp86-2 и fp89 за счет несовпадения аллельных спектров позволяют четко дифференцировать сапсана от кречета и балобана, остальные локусы также отличают эти виды за счет различий в частотах аллелей. Кречет и балобан мало отличаются по аллельным спектрам и частотам аллелей большинства локусов.
Таблица 1.
Частоты аллелей микросателлитных локусов трех видов соколов
| Локус | Аллель | Falco rusticolus | Falco cherrug | Falco peregrinus | Локус | Аллель | Falco rusticolus | Falco cherrug | Falco peregrinus |
|---|---|---|---|---|---|---|---|---|---|
| fp5 | 103 | 0.000 | 0.042 | 0.000 | fp82-2 | 134 | 0.000 | 0.000 | 0.056 |
| 105 | 1.000 | 0.958 | 0.000 | 136 | 0.000 | 0.007 | 0.000 | ||
| 107 | 0.000 | 0.000 | 1.000 | 138 | 0.125 | 0.035 | 0.000 | ||
| fp13 | 96 | 0.000 | 0.000 | 0.222 | 140 | 0.038 | 0.139 | 0.944 | |
| 100 | 0.000 | 0.014 | 0.000 | 142 | 0.413 | 0.076 | 0.000 | ||
| 102 | 0.519 | 0.660 | 0.611 | 144 | 0.213 | 0.132 | 0.000 | ||
| 104 | 0.475 | 0.306 | 0.111 | 146 | 0.000 | 0.035 | 0.000 | ||
| 106 | 0.006 | 0.021 | 0.000 | 148 | 0.006 | 0.160 | 0.000 | ||
| 108 | 0.000 | 0.000 | 0.056 | 150 | 0.163 | 0.215 | 0.000 | ||
| fp31 | 145 | 0.000 | 0.069 | 0.000 | 152 | 0.025 | 0.104 | 0.000 | |
| 147 | 1.000 | 0.910 | 0.000 | 154 | 0.000 | 0.056 | 0.000 | ||
| 149 | 0.000 | 0.021 | 0.000 | 158 | 0.019 | 0.035 | 0.000 | ||
| 155 | 0.000 | 0.000 | 0.111 | 166 | 0.000 | 0.007 | 0.000 | ||
| 157 | 0.000 | 0.000 | 0.222 | fp86-2 | 143 | 1.000 | 1.000 | 0.000 | |
| 159 | 0.000 | 0.000 | 0.111 | 145 | 0.000 | 0.000 | 0.500 | ||
| 161 | 0.000 | 0.000 | 0.556 | 149 | 0.000 | 0.000 | 0.444 | ||
| fp46-1 | 120 | 0.025 | 0.014 | 0.000 | 151 | 0.000 | 0.000 | 0.056 | |
| 122 | 0.000 | 0.000 | 0.167 | fp89 | 123 | 0.019 | 0.014 | 0.056 | |
| 124 | 0.013 | 0.021 | 0.833 | 125 | 0.819 | 0.917 | 0.000 | ||
| 126 | 0.963 | 0.931 | 0.000 | 127 | 0.163 | 0.042 | 0.000 | ||
| 128 | 0.000 | 0.035 | 0.000 | 129 | 0.000 | 0.028 | 0.000 | ||
| fp54 | 92 | 0.069 | 0.028 | 0.000 | 131 | 0.000 | 0.000 | 0.389 | |
| 96 | 0.025 | 0.035 | 0.000 | 133 | 0.000 | 0.000 | 0.222 | ||
| 102 | 0.044 | 0.007 | 0.000 | 135 | 0.000 | 0.000 | 0.167 | ||
| 104 | 0.231 | 0.118 | 0.000 | 139 | 0.000 | 0.000 | 0.111 | ||
| 106 | 0.063 | 0.104 | 0.778 | 143 | 0.000 | 0.000 | 0.056 | ||
| 108 | 0.138 | 0.438 | 0.167 | fp92-1 | 106 | 0.006 | 0.000 | 0.000 | |
| 110 | 0.106 | 0.181 | 0.000 | 108 | 0.013 | 0.000 | 0.000 | ||
| 112 | 0.013 | 0.028 | 0.000 | 112 | 0.900 | 0.785 | 0.000 | ||
| 114 | 0.138 | 0.000 | 0.056 | 114 | 0.000 | 0.007 | 0.000 | ||
| 116 | 0.019 | 0.035 | 0.000 | 116 | 0.000 | 0.063 | 0.000 | ||
| 120 | 0.063 | 0.000 | 0.000 | 118 | 0.044 | 0.076 | 0.111 | ||
| 122 | 0.031 | 0.000 | 0.000 | 120 | 0.038 | 0.035 | 0.333 | ||
| 124 | 0.025 | 0.000 | 0.000 | 122 | 0.000 | 0.035 | 0.444 | ||
| 128 | 0.019 | 0.007 | 0.000 | 134 | 0.000 | 0.000 | 0.056 | ||
| 130 | 0.013 | 0.000 | 0.000 | 142 | 0.000 | 0.000 | 0.056 | ||
| 140 | 0.006 | 0.000 | 0.000 | fp347 | 135 | 0.000 | 0.000 | 0.778 | |
| 0 | 0.000 | 0.021 | 0.000 | 139 | 0.081 | 0.160 | 0.056 | ||
| fp79-1 | 145 | 0.975 | 0.764 | 1.000 | 141 | 0.100 | 0.368 | 0.056 | |
| 147 | 0.025 | 0.236 | 0.000 | 143 | 0.050 | 0.222 | 0.056 | ||
| fp79-4 | 147 | 0.006 | 0.069 | 0.000 | 145 | 0.088 | 0.069 | 0.056 | |
| 151 | 0.006 | 0.000 | 0.000 | 147 | 0.044 | 0.000 | 0.000 | ||
| 153 | 0.019 | 0.111 | 0.000 | 153 | 0.025 | 0.000 | 0.000 | ||
| 155 | 0.513 | 0.403 | 0.056 | 155 | 0.138 | 0.028 | 0.000 | ||
| 157 | 0.338 | 0.146 | 0.056 | 157 | 0.400 | 0.111 | 0.000 | ||
| 159 | 0.100 | 0.076 | 0.056 | 159 | 0.075 | 0.042 | 0.000 | ||
| 161 | 0.019 | 0.188 | 0.167 | fr34 | 148 | 0.525 | 0.722 | 0.000 | |
| 165 | 0.000 | 0.007 | 0.000 | 154 | 0.475 | 0.278 | 0.556 | ||
| 167 | 0.000 | 0.000 | 0.278 | 156 | 0.000 | 0.000 | 0.444 | ||
| 171 | 0.000 | 0.000 | 0.278 | fr142 | 192 | 1.000 | 1.000 | 1.000 | |
| 173 | 0.000 | 0.000 | 0.111 | fr164-1 | 128 | 1.000 | 1.000 | 1.000 |
Для оценки информативности полученных данных мы рассчитали параметры генетической изменчивости трех видовых групп соколов (табл. 2). В выборке кречета выявлено 63 аллеля. Доля полиморфных локусов составила 66.67%. Среднее число аллелей на локус – 4.20, а среднее число аллелей на локус с частотой более 5% – 2.33. Пять из 15 локусов оказались мономорфными. В десяти полиморфных локусах число аллелей варьировало от 2 до 16. Максимальное число аллелей обнаружено в локусах fp54, fp79-4, fp82-2, fp347. Эти локусы наиболее информативны для идентификации отдельных особей. Наблюдаемая гетерозиготность по ним находилась в пределах от 0.563 до 0.700. По всем полиморфным локусам HO – в пределах от 0.050 до 0.700; HE – от 0.049 до 0.880. Значимый дефицит гетерозигот наблюдался в локусах fp13 и fp82-2. При этом отклонения от равновесного распределения генотипов обнаружены в локусах fp13, fp54, fp82-2, fp347. Статистическая проверка частот генотипов выявила вероятное присутствие нуль-аллелей в данных локусах с частотами: fp13 – 0.155 (по Oosterhout, 2004) и 0.111 (по Brookfield, 1996); fp54 – 0.106 и 0.096; fp82-2 – 0.124 и 0.102; fp347 – 0.091 и 0.076.
Таблица 2.
Показатели генетической изменчивости трех видов соколов по микросателлитным локусам
| Локус | NA | NE | HO | HE | F | χ2 | p |
|---|---|---|---|---|---|---|---|
| Falco rusticolus, n = 80 | |||||||
| fp5 | 1 | 1.000 | 0.000 | 0.000 | – | – | – |
| fp13 | 3 | 2.021 | 0.338 | 0.505 | 0.332* | 10.118* | 0.018 |
| fp31 | 1 | 1.000 | 0.000 | 0.000 | – | – | – |
| fp46-1 | 3 | 1.079 | 0.075 | 0.073 | –0.030 | 0.121 | 0.989 |
| fp54 | 16 | 8.312 | 0.700 | 0.880 | 0.204 | 252.218*** | 0.000 |
| fp79-1 | 2 | 1.051 | 0.050 | 0.049 | –0.026 | 0.053 | 0.819 |
| fp79-4 | 7 | 2.582 | 0.600 | 0.613 | 0.021 | 8.451 | 0.993 |
| fp82-2 | 8 | 3.850 | 0.563 | 0.740 | 0.240* | 70.363*** | 0.000 |
| fp86-2 | 1 | 1.000 | 0.000 | 0.000 | – | – | – |
| fp89 | 3 | 1.434 | 0.325 | 0.303 | –0.073 | 1.383 | 0.710 |
| fp92-1 | 5 | 1.229 | 0.175 | 0.186 | 0.062 | 8.164 | 0.613 |
| fp347 | 9 | 4.677 | 0.650 | 0.786 | 0.173 | 64.007** | 0.003 |
| fr34 | 2 | 1.995 | 0.550 | 0.499 | –0.103 | 0.845 | 0.358 |
| fr142 | 1 | 1.000 | 0.000 | 0.000 | – | – | – |
| fr164-1 | 1 | 1.000 | 0.000 | 0.000 | – | – | – |
| Среднее | 4.200 ± | 2.215 ± | 0.268 ± | 0.309 ± | 0.080 ± | – | – |
| Ст. ош. | ± 1.092 | ± 0.524 | ± 0.071 | ± 0.085 | ± 0.038 | – | – |
| Falco cherrug, n = 72 | |||||||
| fp5 | 2 | 1.087 | 0.083 | 0.080 | –0.043 | 0.136 | 0.712 |
| fp13 | 4 | 1.890 | 0.333 | 0.471 | 0.292* | 10.157 | 0.118 |
| fp31 | 3 | 1.201 | 0.167 | 0.167 | 0.003 | 3.586 | 0.310 |
| fp46-1 | 4 | 1.152 | 0.139 | 0.132 | –0.050 | 0.401 | 0.999 |
| fp54 | 11 | 3.948 | 0.514 | 0.747 | 0.312* | 124.185*** | 0.000 |
| fp79-1 | 2 | 1.564 | 0.333 | 0.361 | 0.076 | 0.415 | 0.519 |
| fp79-4 | 7 | 4.137 | 0.625 | 0.758 | 0.176 | 41.708** | 0.005 |
| fp82-2 | 12 | 7.573 | 0.792 | 0.868 | 0.088 | 71.840 | 0.291 |
| fp86-2 | 1 | 1.000 | 0.000 | 0.000 | – | – | – |
| fp89 | 4 | 1.186 | 0.153 | 0.157 | 0.027 | 10.959 | 0.090 |
| fp92-1 | 6 | 1.592 | 0.306 | 0.372 | 0.179 | 21.695 | 0.116 |
| fp347 | 7 | 4.347 | 0.806 | 0.770 | –0.046 | 33.072* | 0.045 |
| fr34 | 2 | 1.670 | 0.472 | 0.401 | –0.177 | 2.254 | 0.133 |
| fr142 | 1 | 1.000 | 0.000 | 0.000 | – | – | – |
| fr164-1 | 1 | 1.000 | 0.000 | 0.000 | – | – | – |
| Среднее | 4.467 ± | 2.290 ± | 0.315 ± | 0.352 ± | 0.070 ± | – | – |
| Ст. ош. | ± 0.904 | ± 0.488 | ± 0.071 | ± 0.080 | ± 0.038 | – | – |
| Falco peregrinus, n = 9 | |||||||
| fp5 | 1 | 1.000 | 0.000 | 0.000 | – | – | – |
| fp13 | 4 | 2.282 | 0.556 | 0.562 | 0.011 | 4.965 | 0.548 |
| fp31 | 4 | 2.613 | 0.889 | 0.617 | –0.440 | 5.760 | 0.451 |
| fp46-1 | 2 | 1.385 | 0.333 | 0.278 | –0.200 | 0.360 | 0.549 |
| fp54 | 3 | 1.573 | 0.222 | 0.364 | 0.390 | 3.327 | 0.344 |
| fp79-1 | 1 | 1.000 | 0.000 | 0.000 | – | – | – |
| fp79-4 | 7 | 4.909 | 1.000 | 0.796 | –0.256 | 21.480 | 0.430 |
| fp82-2 | 2 | 1.117 | 0.111 | 0.105 | –0.059 | 0.031 | 0.860 |
| fp86-2 | 3 | 2.219 | 0.667 | 0.549 | –0.213 | 1.840 | 0.606 |
| fp89 | 6 | 4.050 | 0.667 | 0.753 | 0.115 | 9.903 | 0.826 |
| fp92-1 | 5 | 3.057 | 0.556 | 0.673 | 0.174 | 22.563* | 0.012 |
| fp347 | 5 | 1.620 | 0.333 | 0.383 | 0.129 | 18.184 | 0.052 |
| fr34 | 2 | 1.976 | 0.444 | 0.494 | 0.100 | 0.090 | 0.764 |
| fr142 | 1 | 1.000 | 0.000 | 0.000 | – | – | – |
| fr164-1 | 1 | 1.000 | 0.000 | 0.000 | – | – | – |
| Среднее | 3.133 ± | 2.053 ± | 0.385 ± | 0.372 ± | –0.023 ± | – | – |
| Ст. ош. | ± 0.506 | ± 0.306 | ± 0.086 | ± 0.075 | ± 0.061 | – | – |
Примечание. NA – число аллелей; NE – эффективное число аллелей; HO – наблюдаемая гетерозиготность; HE – ожидаемая гетерозиготность; F – индекс фиксации; χ2 – значение критерия χ2 в тесте на соответствие соотношения генотипов равновесию Харди–Вайнберга; p – уровень значимости отклонения от χ2. Звездочками отмечены уровни значимости отклонения индекса фиксации от нулевого значения и отклонения от генотипического равновесия Харди–Вайнберга: * p < 0.05; ** p < 0.005; *** p < 0.001.
В выборке балобана выявлено 67 аллельных вариантов. Доля полиморфных локусов составила 80%. Число аллелей в 12 полиморфных локусах варьировало от 2 до 12 (табл. 2). Максимальное число аллелей обнаружено в локусах fp54, fp79-4, fp82-2, fp347. Среднее число аллелей на локус составило 4.47, а среднее число аллелей на локус с частотой более 5% – 2.60. Наблюдаемая гетерозиготность по этим локусам находилась в пределах от 0.514 до 0.806. По всем полиморфным локусам HO – от 0.083 до 0.806; HE – от 0.080 до 0.868. Недостаток гетерозигот наблюдался в локусах fp13, fp54, fp79-4, fp92-1. При этом значимые отклонения от равновесного распределения генотипов в сторону дефицита гетерозигот выявлены в локусах fp54, fp79-4 и fp347. Проверка на наличие нуль-аллелей показала вероятное их присутствие с частотами: fp13 – 0.135 (по Oosterhout, 2004) и 0.093 (по Brookfield, 1996); fp54 – 0.151 и 0.137; fp79-4 – 0.097 и 0.076; fp92-1 – 0.071 и 0.000. В генотипах локуса fp54 у трех птенцов из одного выводка выявлен только аллель 110, а еще у одного птенца из этого же выводка не обнаружено амплифицируемых фрагментов этого локуса (по результатам тройной проверки). На основании этого было сделано предположение о вероятном наличии нуль-аллеля в генотипах обоих родителей.
Довольно высокое генетическое разнообразие обнаружено в выборке сапсанов численностью в девять особей, пять из которых являются сибсами (табл. 2). Всего выявлено 47 аллелей. Доля полиморфных локусов составила 73.33%, среднее число аллелей на локус с частотой более 5% – 3.13. В 11 полиморфных локусах число аллелей составляло от двух до семи. По пять и более аллелей обнаружено в локусах fp79-4, fp89, fp92-1, fp347. Наблюдаемая гетерозиготность по этим локусам составила от 0.333 до 1.000. По всем полиморфным локусам HO находилась в пределах от 0.111 до 1.000; HE – от 0.105 до 0.796. Дефицит гетерозигот наблюдался в локусах fp54, fp92-1. Проверку на наличие нуль-аллелей не проводили ввиду малой численности выборки сапсанов.
Оценка вероятности случайного совпадения генотипов двух особей и исключение родительства
Среди 80 особей кречета, в том числе и сибсов, не обнаружено особей с идентичным генотипом. Зависимость вероятности случайного совпадения генотипов двух особей кречета от числа используемых микросателлитных локусов (рис. 1,а) показывает, что при использовании десяти полиморфных локусов в случае наличия родственных связей между особями вероятность случайного совпадения генотипов довольно мала PIsibs = 4.4 × 10–3, а вероятность случайного совпадения генотипов при отсутствии родства еще ниже – PI = 1.4 × 10–6. Таким образом, генотипирование группы из 80 кречетов по 10 полиморфным локусам позволяет надежно отличать каждую особь. Данный набор локусов позволяет с высокой вероятностью определять отношения родитель–потомок. Вероятность исключения родительства составила: P1 = 0.99 (известные генотипы родителей), P2 = 0.92 (известен генотип одного из родителей) и P3 = 1 (генотипы родителей неизвестны).
Рис. 1.
Зависимость вероятности случайного совпадения генотипов от кумулятивного числа используемых микросателлитных локусов: а – кречеты, б – балобаны, в – сапсаны. PI – при отсутствии родства, PIsibs – среди родственников. Порядок локусов приведен в соответствии с табл. 1.
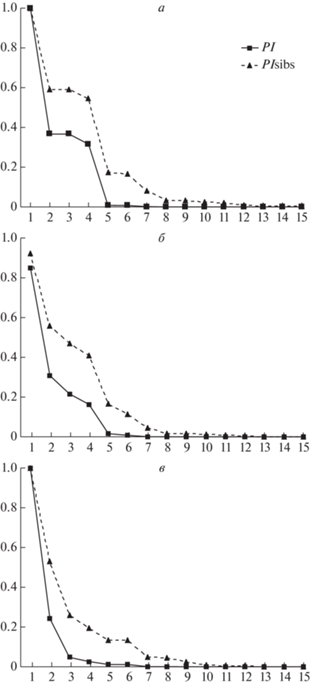
Для выборки балобанов, при использовании 12 полиморфных микросателлитных локусов значение вероятности совпадения генотипов неродственных особей PI = 2.1 × 10–7, в случае наличия родственных связей между особями PIsibs = 2.1 × 10–3 (рис. 1,б). Однако среди 72 исследованных особей балобана были выявлены две группы с повторяющимися идентичными генотипами (по две и три особи). Первая пара – разнополые птенцы из одного выводка 2017 г. рождения (♂1749 и ♀1751), во втором случае – пара птенцов из того же выводка (♂1747, ♀1750) и птенец (♂1519) 2015 г. рождения. Поскольку все они являются потомками одной пары, данный факт указывает на высокое генетическое сходство родителей, которые, по-видимому, являются близкими родственниками. Вероятность исключения родительства при использовании 12 полиморфных локусов составила: P1 = 0.99 (известные генотипы родителей), P2 = 0.93 (известен генотип одного из родителей) и P3 = 1 (генотипы родителей неизвестны).
Зависимость вероятности случайного совпадения генотипов двух особей сапсана от числа используемых микросателлитных локусов (рис. 1,в) показывает, что при использовании 11 полиморфных локусов вероятность случайного совпадения генотипов в выборке сапсанов минимальна как при отсутствии родственных связей между особями (PI = 4.7 × 10–7), так и в случае наличия родственных связей между ними (PIsibs = 1.7 × 10–3). Вероятность исключения родительства при использовании набора из 11 локусов составила: P1 = = 0.99 (известные генотипы родителей), P2 = 0.89 (известен генотип одного из родителей) и P3 = 1 (генотипы родителей неизвестны).
Анализ родственных отношений
Для оценки степени родства между особями мы использовали попарные коэффициенты генетического сходства, которые рассчитали как для тотальных выборок по каждому из видов, так и отдельно для групп сибсов (табл. 3). В нашем материале сибсы представлены четырьмя группами птенцов кречета 2017 г. рождения от трех до восьми особей в каждой, шестью группами птенцов балобана 2017 г. рождения от четырех до пяти особей и двумя парами сибсов 2015 г. рождения, а также одной группой из пяти птенцов сапсана 2017 г. рождения.
Таблица 3.
Значения коэффициентов генетического сходства в группах особей с различной степенью родства
| Статистики | Вся выборка | Группы сибсов | ||||
|---|---|---|---|---|---|---|
| RI | LRM | QGM | RI | LRM | QGM | |
| Falco rusticolus | ||||||
| Число пар | 3160 | 3160 | 3160 | 40 | 40 | 40 |
| Среднее | –0.007 | –0.013 | –0.013 | 0.196 | 0.383 | 0.464 |
| Медиана | –0.022 | –0.052 | –0.029 | 0.175 | 0.309 | 0.447 |
| Ст. откл. | 0.075 | 0.170 | 0.281 | 0.169 | 0.274 | 0.236 |
| Ст. ош. | 0.001 | 0.003 | 0.005 | 0.027 | 0.043 | 0.037 |
| Минимум | –0.152 | –0.376 | –0.905 | –0.029 | –0.090 | 0.017 |
| Максимум | 0.756 | 0.895 | 0.915 | 0.756 | 0.851 | 0.798 |
| Falco cherrug | ||||||
| Число пар | 2556 | 2556 | 2556 | 50 | 50 | 50 |
| Среднее | –0.007 | –0.014 | –0.014 | 0.195 | 0.578 | 0.654 |
| Медиана | –0.026 | –0.062 | –0.042 | 0.166 | 0.588 | 0.695 |
| Ст. откл. | 0.085 | 0.209 | 0.306 | 0.122 | 0.228 | 0.196 |
| Ст. ош. | 0.002 | 0.004 | 0.006 | 0.017 | 0.032 | 0.028 |
| Минимум | –0.166 | –0.411 | –0.924 | 0.028 | 0.101 | 0.324 |
| Максимум | 0.658 | 1.000 | 1.000 | 0.599 | 1.000 | 1.000 |
| Falco peregrinus | ||||||
| Число пар | 36 | 36 | 36 | 10 | 10 | 10 |
| Среднее | –0.059 | –0.125 | –0.125 | 0.056 | 0.261 | 0.411 |
| Медиана | –0.093 | –0.231 | –0.170 | 0.062 | 0.258 | 0.434 |
| Ст. откл. | 0.092 | 0.294 | 0.411 | 0.044 | 0.212 | 0.140 |
| Ст. ош. | 0.015 | 0.049 | 0.068 | 0.014 | 0.067 | 0.044 |
| Минимум | –0.203 | –0.516 | –0.842 | –0.005 | –0.021 | 0.237 |
| Максимум | 0.122 | 0.612 | 0.608 | 0.122 | 0.612 | 0.608 |
В анализе тотальных данных без учета родства средние значения всех коэффициентов близки к нулевому (рис. 2). Средние значения коэффициентов родства (генетического сходства) в пределах групп сибсов во всех случаях оказались выше, чем в тотальной выборке особей. Наибольшие различия показывает коэффициент QGM [28], который не зависит от численности выборок и придает максимальный вес локусам с числом аллелей больше двух. Распределение попарных коэффициентов родства QGM в тотальных выборках кречета и балобана соответствует теоретически ожидаемому нормальному с максимумом вблизи 0 и разбросом значений от –1 до 1 (рис. 3,а, в). Для пар сибсов разброс значений у кречета от 0 до 0.8 (четыре группы попарных сравнений), а у балобана от 0.3 до 1 (восемь групп попарных сравнений) (рис. 3,б, г).
Рис. 2.
Средние значения коэффициентов родства RI, LRM и QGM в тотальных выборках и между сибсами: а – кречеты, б – балобаны, в – сапсаны.
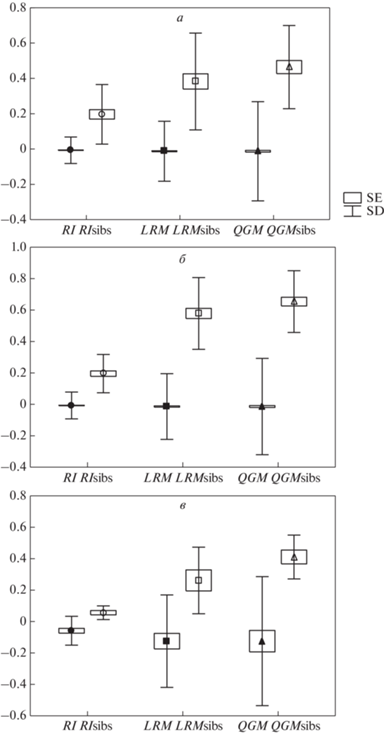
Рис. 3.
Распределение попарных коэффициентов родства в тотальных видовых группах и в диадах сибсов: а, б – кречеты; в, г – балобаны.

Теоретически ожидаемые значения коэффициентов родства между сибсами должны находиться в пределах от 0 до 1. Различия в этих показателях между выборками кречета и балобана хоть и незначительны, но могут свидетельствовать о различиях в степени родства между птицами в родительских парах. Кречеты более разнообразны, а родительские пары балобанов отчасти представлены родственными особями. Среди потомства одной из пар мы обнаружили очень высокие значения коэффициентов родства, от 0.6 до 1.0, и полное совпадение генотипов у двух пар птенцов.
ОБСУЖДЕНИЕ
Микросателлитные локусы применяются уже более 20 лет для изучения генетической изменчивости, анализа системы скрещивания и родственных отношений в популяциях разных видов соколов [20, 21, 29–43]. Однако генетическая изменчивость естественных популяций соколов, обитающих на территории Российской Федерации, остается малоизученной. Все данные об уровнях их генетической изменчивости получены на материале из питомников и поэтому могут считаться только приближенными к показателям генетического разнообразия естественных популяций. Статья А.В. Нечаевой и соавт. [43], посвященная генетической изменчивости чукотско-камчатской популяции кречета, частично заполняет пробел в данных по генетической структуре популяций кречета. Показатели генетического разнообразия этой популяции оказались такими же высокими, как и в естественной популяции Аляски, и выше, чем в популяциях Гренландии и Норвегии [34].
Исследования нуклеотидных последовательностей D-петли митохондриальной (мт) ДНК алтайского балобана и сравнения их с данными зарубежных исследователей позволили выявить специфические для данной формы паттерны [14]. Однако более подробные, базирующиеся на полиморфизме ядерных маркеров, сведения об изменчивости популяций вида с территории РФ по-прежнему отсутствуют. Согласно исследованиям Ф. Ниттингер и соавт. [21, 37] балобан обладает более высоким генетическим разнообразием, чем кречет.
Существующие трудности в различении кречета и балобана при помощи микросателлитных локусов обусловлены, по мнению ряда исследователей, их происхождением от единого предка, а также вероятной естественной гибридизацией восточной ветви балобана с кречетом [21, 32, 34, 37]. Последовательности мтДНК (Cyt b, COI) разделяют эти два вида и указывают на два кластера у балобана (западный и восточный) и единый кластер у кречета, который близок к восточному балобану и, вероятно, отражает общность происхождения или недавнюю гибридизацию между ними [32, 34]. Подробный биоинформатический анализ гена Cyt b показал, что отличие в нуклеотидных последовательностях у этих видов приводит к функционально-структурным различиям белков и носит адаптивный характер [44]. Вовлечение в анализ наследуемой по материнской линии мтДНК наряду с ядерными маркерами может помочь в выявлении гибридов между кречетом и балобаном.
Сапсан – вид-космополит, населяющий все континенты, кроме Антарктиды. Согласно современным классификациям его разделяют на 19 подвидов [45]. Генетическое разнообразие естественных популяций сапсана хорошо изучено, выявлены региональные особенности генетической изменчивости подвидов [20, 30, 33, 35, 36, 40–42, 46, 47]. Применение микросателлитных и минисателлитных локусов позволило оценить изменения в североевропейских популяциях сапсана, вызванные сокращением численности вследствие воздействия химического загрязнения ДДТ на экосистему и последующим ее восстановлением [33, 46]. Изучена гнездовая структура вида в естественных условиях на территории Финляндии, при помощи анализа коэффициентов родства птенцов на гнездовых участках выявлен довольно высокий уровень возвратов взрослых птиц к своим прошлогодним гнездам [40]. На территории России обитают три подвида сапсана – F. p. peregrinus, F. p. calidus и F. p. japonensis. Однако ввиду отсутствия данных о генетическом разнообразии и дифференциации подвидов с территории РФ нет возможности определять принадлежность птиц к локальным популяциям или подвидам, хотя эта информация необходима для восстановления естественных популяций за счет птиц, выращенных в питомниках.
Представленные в нашем исследовании выборки состоят из птиц, содержащихся в неволе, и поэтому не отражают всю полноту генетической изменчивости естественных популяций видов. Аутосомная локализация и независимая сегрегация использованных нами микросателлитных локусов делают их удобным инструментом для генетической паспортизации соколов. Локусы fp5, fp31, fp86-2, fp89 пригодны для идентификации гибридов сапсана с кречетом или балобаном. За счет несовпадения аллельных спектров они позволяют четко дифференцировать сапсана от кречета с балобаном. Остальные локусы также отличают эти виды за счет различий в частотах аллелей. Кречет и балобан мало отличаются по аллельным спектрам и частотам аллелей большинства локусов. Высокоизменчивые локусы с большим числом аллелей (от трех и более) создают максимально информативную базу для индивидуальной идентификации соколов и определения родства между птицами. Низкие оценки вероятности возникновения идентичных генотипов по представленному в нашем исследовании набору локусов достаточны для исключения случайных совпадений при генетической идентификации птиц. В литературе приводятся подобные оценки. Например, в работе Н. Дони и соавт. [48] при использовании 12 микросателлитных локусов для сапсана PI = 9.52 × 10–7. Исследование гнездовой структуры сапсана в Финляндии по десяти микросателлитным локусам, девять из которых совпадают с нашим набором, показало еще более низкие оценки PI = 6.68 × 10–8, а PIsibs = = 1.37 × 10–3 [40]. В исследовании популяционной структуры степной пустельги, Falco naumanii, с использованием восьми гетерологичных локусов от сапсана и кречета PI = 6.21 × 10–6 [29]. У дербника, Falco columbarius, оценки по пяти высокополиморфным локусам составили PI = 5.69 × 10–7 и PIsibs = 1.67 × 10–4 [49]. Получить еще более низкие оценки вероятности совпадения генотипов и тем самым увеличить точность индивидуальной идентификации птиц, в том числе в случае близкого родства, можно с привлечением других полиморфных микросателлитных локусов, в том числе и гетерологичных. Десять высокополиморфных микросателлитных локусов для балобана разработаны группой исследователей из Китая [50]. Из набора новых микросателлитных локусов, разработанных для дербника, по девять локусов полиморфны у сапсана и кречета [49].
В настоящем исследовании продемонстрированы возможности генетической идентификации соколов по универсальному набору микросателлитных локусов. Индивидуальные генотипы позволяют с высокой точностью отличать родственных особей от неродственных. Использование различных коэффициентов родства дает сопоставимые результаты. Средние коэффициенты родства у сибсов находятся около значения 0.5, что соответствует теоретически ожидаемым значениям. Коэффициенты родства между родителями и потомством находятся в пределах от 0.5 до 1. Прямое сопоставление генотипов предполагаемых родителей и потомков позволяет точно установить или отвергнуть факт родительства. Аналогично можно определять принадлежность конкретной птицы к пулу родственников. Анализ сочетания генотипов следует проводить с учетом возможности наличия нуль-аллелей по отдельным локусам или вероятности их возникновения de novo (обычно эта вероятность не превышает 1 × 10–3 случаев на локус на поколение).
Внедрение системы генетической паспортизации в работу питомников может помочь не только в идентификации отдельных особей, но и проверке происхождения птиц от заявленных родителей (контроль результатов скрещивания). Введение данных методик в практику криминалистической генетической экспертизы [51, 52] может быть востребованным при создании доказательной базы для принятия решений в судебной практике.
Работа выполнена при частичной финансовой поддержке по теме Государственного задания Министерства науки и высшего образования РФ для Института общей генетики им. Н.И. Вавилова РАН № 0092-2019-0007.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Negro J.J. Raptors and people: An ancient relationship persisting today // Birds of Prey. Cham, Switzerland: Springer, 2018. P. 161–176. https://doi.org/10.1007/978-3-319-73745-4_7
UNESCO. 2016. Falconry, a living human heritage (Electronic resource) // URL: https://ich.unesco.org/en/RL/falconry-a-living-human-heritage-01209 (accessed: 14.04.2021).
Николенко Э.Г. Обзор “соколиных” задержаний и уголовных дел по ним в России и странах ближнего зарубежья // Пернатые хищники и их охрана. 2018. Т. 36. С. 24–43.
BirdLife International. 2017. Falco rusticolus (amended version of 2016 assessment). The IUCN Red List of Threatened Species 2017: e.T22696500A110639833. Downloaded on 22 November 2021.https://doi.org/10.2305/IUCN.UK.2017-1.RLTS.T22-696500A110639833.en
BirdLife International. 2017. Falco cherrug (amended version of 2016 assessment). The IUCN Red List of Threatened Species 2017: e.T22696495A110525916. Downloaded on 22 November 2021.https://doi.org/10.2305/IUCN.UK.2017-1.RLTS.T2-2696495A110525916.en
BirdLife International. 2019. Falco peregrinus (amended version of 2016 assessment). The IUCN Red List of Threatened Species 2019: e.T45354964A155500538. Downloaded on 22 November 2021.https://doi.org/10.2305/IUCN.UK.2016-3.RLTS.T4-5354964A155500538.en
Михайлова Н.Н. Вольерное разведение соколов как инструмент возрождения охоты с ловчими птицами // Ежегодник: Хищные птицы и совы в зоопарках и питомниках. Вып. 20. М.: Московский зоопарк, 2011. С. 96–102.
Остапенко В.А., Сорокин А.Г., Беме И.Р. О необходимости сохранения кречета (Falco rusticolus) // Ежегодник: Хищные птицы и совы в зоопарках и питомниках. Вып. 21. М.: Московский зоопарк, 2012. С. 12–19.
Сорокин А.Г. Материалы по деятельности питомников редких видов хищных птиц в Российской Федерации // Ежегодник: Хищные птицы и совы в зоопарках и питомниках. Вып. 20. М.: Московский зоопарк, 2011. С. 50–69.
Мудрик Е.А., Кашенцева Т.А., Гамбург Е.А., Политов Д.В. Генетическая паспортизация и идентификация стерхов (Grus leucogeranus Pallas) в искусственно созданных условиях // Изв. РАН. Серия Биол. 2014. № 3. С. 219–227. https://doi.org/10.7868/S0002332914030096
Мудрик Е.А., Кашенцева Т.А., Политов Д.В. Интеграция молекулярных подходов в программу создания резервного генофонда редкого вида журавлей стерха (Grus leucogeranus Pallas) // Успехи соврем. биологии. 2015. Т. 135. С. 139–147.
Мудрик Е.А., Кашенцева Т.А., Постельных К.А. и др. Генетическое разнообразие и родство в разных поколениях искусственной популяции стерха (Grus leucogeranus Pallas) // Генетика. 2014. Т. 50. № 11. С. 1345–1353. https://doi.org/10.7868/S0016675814100105
Зиневич Л.С., Рожкова Д.Н., Николенко Э.Г. и др. Определение пола и другие рутинные ПЦР-анализы в исследованиях хищных птиц // Пернатые хищники и их охрана. 2018. Спецвып. 1. С. 208–210.
Рожкова Д.Н., Зиневич Л.С., Николенко Э.Г. и др. Молекулярно-генетическое сопровождение проекта по восстановлению популяции сокола балобана в Алтае-Саянском регионе // Пернатые хищники и их охрана. 2018. Спецвып. 1. С. 225–227.
Selkoe K.A., Toonen R.J. Microsatellites for ecologists: A practical guide to using and evaluating microsatellite markers // Ecol. Letters. 2006. V. 9. № 5. P. 615–629. https://doi.org/10.1111/j.1461-0248.2006.00889.x
Martínez-Cruz B., Camarena M.M. Conservation genetics in raptors // Birds of Prey. Springer Intern. Publ. AG, part of Springer Nature, 2018. P. 339–371. https://doi.org/10.1007/978-3-319-73745-4_15
Walsh P.S., Metzger D.A., Higuchi R. Chelex-100 as a medium for simple extraction of DNA for PCR-based typing from forensic material // Biotechniques. 1991. V. 10. № 4. P. 506–513.
Griffiths R., Daan S., Dijkstra C. Sex identification in birds using two CHD genes // Proc. R. Soc. Lond. B. 1996. V. 263. P. 1251–1256.
Griffiths R., Double M.C., Orr K., Dawson R.J.G. A DNA test to sex most birds // Mol. Ecology. 1998. V. 7. P. 1070–1075.
Nesje M., Røed K.H., Lifjeld J.T. et al. Genetic relationships in the peregrine falcon (Falco peregrinus) analysed by microsatellite DNA markers // Mol. Ecology. 2000. V. 9. P. 53–60. https://doi.org/10.1046/j.1365-294x.2000.00834.x
Nittinger F., Gamauf A., Pinsker W. et al. Phylogeography and population structure of the saker falcon (Falco cherrug) and the influence of hybridization: mitochondrial and microsatellite data // Mol. Ecology. 2007. V. 16. P. 1497–1517. https://doi.org/10.1111/j.1365-294X.2007.03245.x
Nesje M., Røed K.H. Microsatellite DNA markers from the gyrfalcon (Falco rusticolus) and their use in other raptor species // Mol. Ecology. 2000. V. 9. P. 1433–1449. https://doi.org/10.1046/j.1365-294x.2000.00999-4.x
Peakall R., Smouse P.E. GenAIEx V6: Genetic analysis in Excel. Population genetic software for teaching and research // Mol. Ecology Notes. 2006. V. 6. № 1. P. 288–295. https://doi.org/10.1111/j.1471-8286.2005.01155.x
Peakall R., Smouse P.E. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research-an update // Bioinformatics. 2012. V. 28. P. 2537–2539. https://doi.org/10.1093/bioinformatics/bts460
Van Oosterhout C., Hutchinson W.F., Wills D.P.M., Shipley P. Micro-Checker: software for identifying and correcting genotyping errors in microsatellite data // Mol. Ecology Notes. 2004. V. 4. P. 535–538. https://doi.org/10.1111/j.1471-8286.2004.00684.x
Ritland K. Estimators for pairwise relatedness and individual inbreeding coefficients // Genet. Research. 1996. V. 67. P. 175–185. https://doi.org/10.1017/S0016672300033620
Lynch M., Ritland K. Estimation of pairwise relatedness with molecular markers // Genetics. 1999. V. 152. P. 1753–1766. https://doi.org/10.1093/genetics/152.4.1753
Queller D.C., Goodnight K.F. Estimating relatedness using genetic markers // Evolution. 1989. V. 43. № 2. P. 258–275. https://doi.org/10.1111/j.1558-5646.1989.tb04226.x
Alcaide M., Negro J.J., Serrano D. et al. Captive breeding and reintroduction of the lesser kestrel Falco naumanni: A genetic analysis using microsatellites // Conserv. Genetics. 2010. V. 11. P. 331–338. https://doi.org/10.1007/s10592-009-9810-7
Brown J.W., Van Coeverden de Groot P.J., Birt T.P. et al. Appraisal of the consequences of the DDT-induced bottleneck on the level and geographic distribution of neutral variation in Canadian peregrine falcons, Falco peregrinus // Mol. Ecology. 2007. V. 16. P. 327–343. https://doi.org/10.1111/j.1365-294X.2007.03151.x
Bryndova M., Putnova L., Bartonova P. et al. Genetic differences between wild and captive populations of the Peregrine falcon (Falco peregrinus) and Saker falcon (Falco cherrug) living in the Czech Republic // J. Agricultural Sci. and Technology B 2. 2012. P. 642–651.
Dawnay N., McEwing R., Thorpe R.S., Ogden R. Preliminary data suggests genetic distinctiveness of gyr and saker falcons // Conserv. Genetics. 2008. V. 9. P. 703–707. https://doi.org/10.1007/s10592-007-9392-1
Jacobsen F., Nesje M., Bachmann L., Lifjeld J.T. Significant genetic admixture after reintroduction of peregrine falcon (Falco peregrinus) in Southern Scandinavia // Conserv. Genetics. 2008. V. 9. P. 581–591. https://doi.org/10.1007/s10592-007-9373-4
Johnson J.A., Burnham K.K., Burnham W.A., Mindell D.P. Genetic structure among continental and island populations of gyrfalcons // Mol. Ecology. 2007. V. 16. P. 3145–3160. https://doi.org/10.1111/j.1365-294X.2007.03373.x
Johnson J.A., Talbot S.L., Sage G.K. et al. The use of genetics for the management of a recovering population: Temporal assessment of migratory Peregrine Falcons in North America // PLoS One. 2010. V. 5. № 11. P. 1–15. e14042. https://doi.org/10.1371/journal.pone.0014042
Nesje M., Røed K.H., Bell D.A. et al. Microsatellite analysis of population structure and genetic variability in peregrine falcons (Falco peregrinus) // Animal Conservation. 2000. V. 3. № 3. P. 267–275. https://doi.org/10.1111/j.1469-1795.2000.tb00112.x
Nittinger F., Haring E., Pinsker W. et al. Out of Africa? Phylogenetic relationships between Falco biarmicus and the other hierofalcons (Aves: Falconidae) // JZS. 2005. V. 43. № 4. P. 321–331.
Nittinger F., Haring E., Pinsker W., Gamauf A. Are escaped hybrid falcons a threat to the Pannonian population of the Saker Falcon (Falco cherrug)? // Greifvögel & Eulen in Österreich. Wien. 2006. V. 1. P. 21–26.
Ortego J., Gonzalez E.G., Sanchez-Barbudo I. et al. Novel highly polymorphic loci and cross-amplified microsatellites for the Lesser Kestrel Falco naumanni // Ardeola. 2007. V. 54. № 1. P. 101–108.
Ponnikas S., Ollila T., Kvist L. Turnover and post-bottleneck genetic structure in a recovering population of peregrine falcons Falco peregrinus // Ibis. 2017. V. 159. P. 311–323. https://doi.org/10.1111/ibi.12460
Talbot S.L., Palmer A.G., Sage G.K. et al. Lack of genetic polymorphism among peregrine falcons Falco peregrinus of Fiji // J. Avian Biology. 2011. V. 42. № 5. P. 415–428. https://doi.org/10.1111/j.1600-048X.2011.05280.x
Talbot S.L., Sage G.K., Sonsthagen S.A. et al. Intraspecific evolutionary relationships among peregrine falcons in western North American high latitudes // PLoS One. 2017. V. 12. № 11. e0188185. https://doi.org/10.1371/journal.pone.0188185
Нечаева А.В., Белоконь М.М., Белоконь Ю.С. и др. Генетическая изменчивость чукотско-камчатской популяции кречета (Falco rusticolus, Falconiformes, Falconidae) на сновании анализа ядерных микросателлитных локусов // Зоол. журн. 2018. Т. 97. № 3. С. 337–342. https://doi.org/10.7868/S0044513418030078
Рожкова Д.Н., Зиневич Л.С., Карякин И.В. и др. Ненейтральная изменчивость цитохрома b у балобана Falco cherrug Grey, 1834 и кречета Falco rusticolus L. // Генетика. 2021. Т. 57. № 4. С. 454–463. https://doi.org/10.31857/S0016675821040123
White C.M. Peregine falcon // Handbook of Birds of the World. V. 2 (New World Vultures to Guineafowl). Barcelona: Lynx Editions, 1994. P. 274–275.
Lifjeld J.T., Bjørnstad G., Steen O.F., Nesje M. Reduced genetic variation in Norwegian peregrine falcons Falco peregrinus indicated by minisatellite DNA fingerprinting // Ibis. 2002. V. 144. P. E19–E26. https://doi.org/10.1046/j.0019-1019.2001.00029.x
White C.M., Sonsthagen S.A., Sage G.K. et al. Genetic relationships among some subspecies of the peregrine falcon (Falco peregrinus L.), inferred from mitochondrial DNA control-region sequences // The Auk. 2013. V. 130. № 1. P. 78–87. https://doi.org/10.1525/auk.2012.11173
Dawnay N., Ogden R., Wetton J.H. et al. Genetic data from 28 STR loci for forensic individual identification and parentage analyses in 6 bird of prey species // Forensic Sci. Intern.: Genetics. 2009. V. 3. P. e63–e69. https://doi.org/10.1016/j.fsigen.2008.07.001
Hull J.M., Sage G.K., Sonsthagen S.A. et al. Isolation and characterization of microsatellite loci in merlins (Falco columbarius) and cross-species amplification in gyrfalcons (F. rusticolus) and peregrine falcons (F. peregrinus) // Mol. Biol. Reports. 2020. V. 47. P. 8377–8383. https://doi.org/10.1007/s11033-020-05842-4
Hou X., Xu P., Lin Z. et al. Integrated tool for microsatellite isolation and validation from the reference genome and their application in the study of breeding turnover in an endangered avian population // Integrative Zool. 2018. V. 13. P. 553–568. https://doi.org/10.1111/1749-4877.12305
Linacre A., Gusmaõ L., Hecht W. et al. ISFG: Recommendations regarding the use of non-human (animal) DNA in forensic genetic investigations // Forensic Sci. Intern.: Genetics. 2011. V. 5. P. 501–505. https://doi.org/10.1016/j.fsigen.2010.10.017
Смирнова С.А., Омельянюк Г.Г., Стороженко И.В. и др. Судебная молекулярно-генетическая экспертиза объектов биологического происхождения – новое направление судебно-экспертной деятельности Минюста России // Теория и практика судебной экспертизы. 2021. Т. 16. № 1. С. 6–18. https://doi.org/10.30764/1819-2785-2021-1-6-18
Дополнительные материалы отсутствуют.


