Генетика, 2022, T. 58, № 6, стр. 661-667
Первые данные о генетической структуре видов рода Alyssum L. секции Odontarrhena на Урале, способных к накоплению никеля
Д. Р. Юнусова 1, *, А. Ю. Тептина 2, В. Л. Семериков 1, М. А. Полежаева 1
1 Институт экологии растений и животных Уральского отделения
Российской академии наук
620144 Екатеринбург, Россия
2 Уральский федеральный университет им. первого Президента России Б.Н. Ельцина
620002 Екатеринбург, Россия
* E-mail: dianaiunusova@mail.ru
Поступила в редакцию 18.01.2021
После доработки 20.01.2022
Принята к публикации 02.02.2022
- EDN: LUJJLO
- DOI: 10.31857/S0016675822060145
Аннотация
Впервые рассматриваются филогенетические отношения между видами Alyssum obovatum (C.A. Mey.) Turcz., A. tortuosum Willd. и A. litvinovii Knjaz. секции Odontarrhena на Урале, имеющими способность к накоплению никеля. Анализ изменчивости двух фрагментов хлоропластной ДНК позволил выявить 15 гаплотипов, распределение которых структурированно географически. Для некоторых популяций A. obovatum и A. tortuosum обнаружены видоспецифичные гаплотипы, однако большая часть близкородственных гаплотипов встречается у географически близких популяций обоих видов. Эндемичный вид A. litvinovii оказался мономорфным и генетически близким к двум другим видам.
Гипераккумуляторы – растения, способные накапливать в своих тканях тяжелые металлы в концентрациях в сотни и тысячи раз больших, чем другие растения [1]. На сегодняшний день около 25% известных растений-гипераккумуляторов обнаружены в семействе Brassicaceae [2]. В основном это представители родов Alyssum L., Noccaea Moench., Thlaspi L., чаще всего накапливающих никель. Уникальная особенность этих растений используется при фиторемедиации – стратегии удаления тяжелых металлов из окружающей среды без использования химических или механических методов [3]. Многие растения-гипераккумуляторы в пределах Brassicaceae обладают большим запасом генетической изменчивости, что делает их перспективными для отбора в сельскохозяйственных и рекультивационных целях [4]. Однако помимо биотехнологического приложения, растения-гипераккумуляторы представляют интерес в качестве модельного объекта для изучения процессов микроэволюции и адаптации к экстремальным условиям среды. Так, в некоторых работах отмечается корреляция между уровнем генетической изменчивости и способностью к накоплению тяжелых металлов [5, 6], что обусловливает актуальность изучения генетического разнообразия и филогении данных видов.
В роде Alyssum, содержащем более сотни видов, в настоящее время проводится таксономическая ревизия. В отношении многих европейских видов выявлено несоответствие существующих таксономических концепций и филогенетических реконструкций, основанных на молекулярно-генетических маркерах, плоидности, а также морфологических параметрах [7–9]. Более того, ряд таксонов, до недавнего времени относившихся к роду Alyssum в ранге секции Odontarrhena, в настоящий момент предлагается выделить в отдельный род Odontarrhena [10]. Систематика данной секции в Северной Евразии не упорядочена. Спектр взглядов на таксономическое разнообразие Odontarrhena сводится к нескольким подходам, согласно которым следует выделять как единственный полиморфный вид Alyssum obovatum (=Odontarrhena obovata) [11], так и два–три вида [12], и даже восемь видов только для азиатской части России и Монголии [13]. Таким образом, границы и объем A. obovatum дискуссионы [14].
Урал – зона совместного произрастания трех видов Alyssum секции Odontarrhena. Виды A. obovatum и A. tortuosum имеют широкий, но дизьюнктивный евроазиатский ареал и проявляют себя как облигатный и факультативный гипераккумуляторы никеля соответственно [15]. Третий вид, A. litvinovii, является эндемиком Урала [16] и имеет способность к гемиаккумуляции никеля [15]. Таким образом, на данной территории произрастает комплекс морфологически трудно различимых видов, которые никогда не исследовались ранее. Поэтому на начальном этапе исследования необходимо отработать методику молекулярно-генетических исследований для решения вопросов о генетической структуре и динамике ареалов видов Alyssum на Урале; о генетическом разнообразии популяций и существовании репродуктивных барьеров между видами.
Цель исследования – оценка эффективности использования маркеров хлоропластной ДНК (хпДНК) для выяснения генетических отношений между видами А. obovatum, А. tortuosum и А. litvinovii на территории Среднего и Южного Урала с особым вниманием к вопросу о происхождении А. litvinovii.
Исследуемые виды: A. obovatum, A. tortuosum и A. litvinovii – многолетники, представители петрофитно-степной флоры; в качестве внешней группы был выбран A. lenense Adams из секции Alyssum – вид, приуроченный к лесным сообществам. Все исследуемые виды секции Odontarrhena имеют перекрывающиеся морфологические характеристики и представляют собой стелющиеся полукустарнички с мелкими листьями, сложными соцветиями и односемянными локулами стручочков. А. obovatum имеет широчайший для рода естественный ареал распространения: в Евразии – от Восточной Европы до севера Центральной Азии, а также на Северо-Востоке Америки [10]. А. tortuosum распространен в Юго-Восточной Европе, России (Кавказ, Урал, Сибирь). На Урале А. obovatum встречается на севере Башкирии, Свердловской и Челябинской областей, а А. tortuosum южнее – в Оренбургской и на юге Челябинской области. А. litvinovii – редкий вид, известный из единственного местонахождения – гора Дюртель Оренбургской обл. [16]. Ареал A. lenense включает Восточную Европу, Россию, Северный Китай. Экологические ниши, занимаемые видами, разнятся: А. obovatum встречается на остепненных скалах, каменистых склонах, в петрофитных степях [17]. А. tortuosum встречается на выходах известняков, песчаников, в каменистых степях, на щебнистых степных склонах [18]. А. litvinovii произрастает на карбонатизированных серпентинитах в единственном локалитете. A. lenense встречается на травянистых склонах, в лесах на разных типах пород.
МАТЕРИАЛЫ И МЕТОДЫ
Всего изучено 148 образцов из районов Южного и Среднего Урала (Оренбургская, Челябинская области, Республика Башкортостан), включающих шесть ценопопуляций A. obovatum, три ценопопуляции A. tortuosum, одну ценопопуляцию A. litvinovii и одну ценопопуляцию A. lenense. Список и номера ценопопуляций представлены в табл. 1, их географическое расположение на рис. 1,а; для удобства далее по тексту они будут упоминаться как популяции.
Таблица 1.
Географические координаты мест сбора и выявленные гаплотипы хпДНК в изученных выборках A. obovatum, A. tortuosum, A. litvinovii, A. lenense
| № | Популяция | Коорд., с.ш./в.д. | N | Nh | Ns |
|---|---|---|---|---|---|
| 1 | A. litvinovii Орен. обл., Новокиевка | 51°28′/58°10′ | 17 | c:17 | c1:1 |
| 2 | A. obovatum Башк., Шигаево | 53°48′/58°11′ | 17 | d:8 c:9 | d2:1 |
| 3 | A. obovatum Башк., Калкан | 54°25′/59°20′ | 9 | d:1 c:8 | d1:1 |
| 4 | A. obovatum Чел. обл., Егоза | 55°45′/60°26′ | 12 | b:12 | b1:1 |
| 5 | A. obovatum Чел. обл., Вишневогорск | 55°58′/60°38′ | 2 | c:1 e:1 | c4:1 |
| 6 | A. obovatum Чел. обл., Горн. воздух | 53°42′/58°39′ | 13 | c:13 | c2:2 |
| 7 | A. obovatum Чел. обл., Карабаш | 55°46′/60°19′ | 10 | b:10 | b2:2 |
| 8 | A. tortuosum Орен. обл., Новокиевка | 51°28′/58°10′ | 24 | c:4 f:12 e:7 d:1 |
f1:2 c3.1:1 f2:1 c3.2:1 |
| 9 | A. tortuosum Орен. обл., Аккермановка | 51°11′/58°08′ | 23 | a:23 | a:1 |
| 10 | A. tortuosum Орен. обл., Хабарное | 51°06′/58°06′ | 18 | a:18 | a:3 |
| 11 | A.lenense Орен. обл., Новокиевка | 51°28′/58°10′ | 3 | g:3 | g:2 |
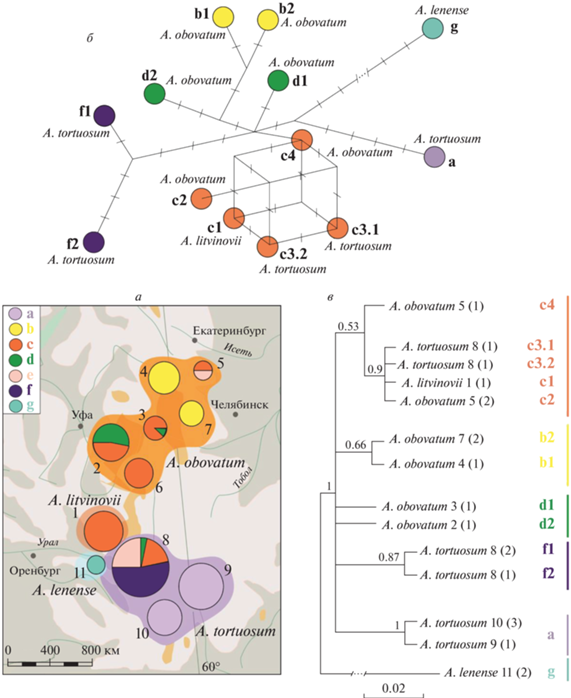
Рис. 1.
а – карта местоположения изученных выборок, распределение частот обнаруженных гаплотипов хпДНК, основанное на ПДРФ анализе; размер окружностей пропорционален размеру выборки; популяции одного вида объединены тонированной областью. б – сеть гаплотипов, построенная на основании последовательностей 20 образцов в программе Network; гаплотипы обозначены цветом и индексом (см. объяснение в тексте); мутации отмечены штрихами (для аутгруппы учтены не все мутации). в – филогенетическое древо, построено с помощью байесовского метода; над ветвями указано значение апостериорной вероятности; латинскими буквами обозначены гаплотипы; цифры соответствуют номерам популяций в табл. 1; в скобках указано количество отсеквенированных образцов.
ДНК выделяли по стандартному протоколу для растительных тканей (СТАВ-метод) из свежего материала [19]. При отборе маркеров проводили амплификацию с наиболее часто используемыми в филогенетических и популяционных исследованиях фрагментами хпДНК с универсальными праймерами trnH-trnK, trnK1-trnK2, psaA-trnS и trnC-trnD [20]; trnF-trnVr [21], trnT-trnF [22], trnS-trnG [23] при температурном профиле, рекомендуемом авторами. Для межгенного спейсера psbA-trnH de novo были разработаны родоспецифичные праймеры на основе полного хлоропластного генома Alyssum desertorum (номер в GenBank KY498535.1) с использованием онлайн-программы Primer3Web (version 4.1.0) [24]: psbAa – GAACGACGGGAATTGAACC; trnHa – TAACCGCGCTAACCTTGGTA. Амплификацию проводили при следующих условиях: предварительная денатурация при 94°C 5 мин, в последующих циклах: денатурация 45 с, отжиг праймеров при 60.5°C в течение 1 мин, элонгация при 72°C 2 мин, затем финальная элонгация 10 мин при 72°C. Всего 35 циклов реакции.
Далее продукты амплификации подвергались рестрикции с пятью ферментами: HaeIII, HinfI, Kzo9I, TaqI, Tru9I. Наиболее изменчивыми хлоропластными фрагментами оказались trnS-trnG и psbAa-trnHa, позволяющими определить наибольшее количество мутаций. Эти фрагменты в сочетании с перечисленными эндонуклеазами рестрикции использовались для анализа методом ПДРФ (полиморфизм длин рестриктных фрагментов) всех 148 образцов. Для 20 образцов всех четырех видов получены полные нуклеотидные последовательности по этим двум фрагментам с использованием генетического анализатора ABI 3130 (Applied Biosystems, USA). Выравнивание последовательностей проводили вручную в программе BioEdit [25]. Филогенетический анализ проведен только с использованием полных последовательностей байесовским методом в программе MrBayes v.3.1.2 [26] на основе модели нуклеотидных замен GTR + G + I. Инсерции, инверсии и делеции рассматривались как одиночные события и были закодированы в виде бинарной матрицы данных, состоящей из нулей и единиц. Визуализировались консенсус-деревья с помощью программы Fig Tree v1.4.3 [27]. Сеть гаплотипов построена на основании метода Median Joining (MJ) в программе Network v.5.0.0.3 [28].
РЕЗУЛЬТАТЫ
На первом этапе отбора маркеров на 24 образцах четырех видов большинство анализируемых фрагментов хпДНК проявили себя как мономорфные, с низкой изменчивостью, либо с нестабильной амплификацией. Высокополиморфными оказались два фрагмента – межгенный спейсер trnS-trnG и межгенный спейсер trnHа-psbAа. На втором этапе проведен рестриктный анализ (ПДРФ) двух выбранных фрагментов с пятью рестриктазами на всем объеме материала (148 образцов). В результате выявлено семь гаплотипов: a, b, c, d, e, f и g. По распределению гаплотипов и их частот (рис. 1,а) можно заключить, что изменчивость структурирована географически и не носит строгий видоспецифичный характер. Так, видоспецифичным гаплотипом a объединены самые южные популяции A. tortuosum (поп. 9 и 10) из Аккермановки и Хабарного; видоспецифичный гаплотип b характерен для северных популяций A. obovatum (4 и 7) из Егозы и Карабаша. Однако у всех трех видов секции Odontarrhena распространен общий гаплотип c, который встретился у A. tortuosum в популяции из Новокиевки (8) и у A. obovatum сразу в нескольких популяциях – из Вишневогорска (5), Горного воздуха (6), Калкана (3) и Шигаево (2), а локальный эндемик A. litvinovii (популяция 1) оказался мономорфным по этому гаплотипу. Гаплотип d встретился у обоих видов – у A. obovatum из Шигаево (2) и Калкана (3), у A. tortuosum в Новокиевке (8). Редкий гаплотип e также обнаружен у A. obovatum из Вишневогорска (5) и у A. tortuosum из Новокиевки (8). Популяция A. tortuosum из Новокиевки оказалась самой разнообразной: в нее вошли четыре гаплотипа (c, d, e, f), причем большая часть образцов в популяции имела уникальный гаплотип f. Образцы A. lenense (популяция 11), взятые в качестве внешней группы имеют видоспецифичный гаплотип g, далеко отстоящий от остальных (95 мутаций). Таким образом, из семи гаплотипов три являются общими.
Более детальное представление об изменчивости уральских популяций Alyssum удалось получить методом секвенирования. Последовательности фрагментов для каждого гаплотипа размещены в Генбанке (GenBank) под номерами доступа (OK329970–OK329993). Длина объединенной последовательности составила 1196 пар нуклеотидов (trnS-trnG 1–688 пн; trnHа-psbAа 689–1196 пн). В исследованных образцах выявлено большое количество точковых мутаций (87), инделей (34) и небольшое количество инверсий (3). Всего обнаружено 124 изменчивых сайта, из них только 8 парсимонистически информативных. Обнаружена внутривидовая и внутрипопуляционная изменчивость. Таким образом, секвенирование позволило обнаружить бóльшую изменчивость, чем ПДРФ-анализ – количество обнаруженных гаплотипов возросло с 7 до 15. При этом общая картина распределения изменчивости осталась прежней – с ярко выраженной географической структурой, а установленные ранее гаплотипы образовали кластеры близких гаплотипов, отличающихся на 1–3 мутации.
Медианная сеть генеалогических связей между гаплотипами (рис. 1,б) выявила пул из пяти гаплотипов (c1, c2, c3.1, c3.2 и c4), на которые распался гаплотип с, общий для образцов из близлежащих популяций Оренбургской и Челябинской областей. Так, на этапе ПДРФ-анализа образцы A. obovatum из популяции Горный воздух (6) имели общий с образцами A. litvinovii (1) гаплотип с. Секвенирование позволило разделить эти образцы на два отдельных гаплотипа: c1 для A. litvinovii и c2 для A. obovatum. У A. tortuosum из Новокиевки (8) гаплотип с проявил внутрипопуляционную изменчивость, распавшись на с3.1 и с3.2. У A. obovatum из Вишневогорска (5) выделился гаплотип c4.
Специфичный для A. tortuosum гаплотип a далее не разделился, а специфичный для A. obovatum гаплотип b разделился на гаплотипы b1 Егоза (4) и b2 Карабаш (7). У A. obovatum гаплотип d распался на d1 в Калкане (3) и d2 в Шигаево (2), у A. tortuosum его секвенировать не удалось, также как и гаплотип е. В уникальном для A. tortuosum гаплотипе f выделилось два: f1 и f2. Таким образом, секвенирование нуклеотидных последовательностей позволило углубить представление об изменчивости, существующей в уральских популяциях рода Alyssum.
На филогенетическом древе, построенным баесовым методом (рис. 1,в) выделяются четыре клады, соответствующие основным гаплотипам, детектированным еще на стадии ПДРФ-анализа. С высокой статистической поддержкой (PPvalue 0.87–1.00) выделяются клады образцов A. tortuosum с видоспецифичными гаплотипами a и f1, f2. Клада пула гаплотипов c объединила три вида, в ее пределах субклады соответствуют разным популяциям. Другие клады выделены с меньшими поддержками, однако, с четкой структурой по регионам в пределах Урала и содержат по одному виду.
ОБСУЖДЕНИЕ
Наблюдаемая картина соответствует ситуации, когда предполагаемые виды еще не обрели репродуктивную изоляцию, активно образуют гибриды и связаны существенным генетическим потоком. Исследователи [29] склонны рассматривать современный ареал распространения видов из секции Odontarrhena как следствие феномена быстрой экспансии видов после эпохи оледенения, поэтому можно предполагать, что в данной таксономической группе в настоящий момент идет постепенная фиксация предкового полиморфизма [30–32]. По этой причине уральские виды, хотя уже и имеют специфичные гаплотипы (a, b, f), но благодаря неполному “lineage sorting” у разных видов с высокой частотой встречаются общие или близкие варианты гаплотипов. В дальнейшем редкие варианты могут быть утеряны, а в пределах подгруппы близких гаплотипов может накопиться значительное количество мутаций, что приведет к закреплению специфичных для видов хлоропластных линий. Например, в группе гаплотипов c уже заметно обособление хлоропластных вариантов: гаплотипы c1, c2 и c3 соответствуют трем видам – A. litvinovii, A. obovatum, A. tortuosum. Особое внимание нужно уделить уральскому эндемику A. litvinovii. Наличие у него варианта гаплотипа из группы с, объединяющей все три вида, свидетельствует о недавнем происхождении и близком родстве с двумя другими видами. Однако на данном этапе невозможно сделать заключение, действительно ли этот вид обладает сниженной изменчивостью или это объясняется малым количеством исследованных образцов (всего 17). Интересная особенность A. litvinovii заключается в его повышенной плоидности [А.Ю. Тептина, неопубл.], что позволяет предполагать гибридную природу происхождения этого вида – обычное явление для рода Alyssum [33].
В целом, выявленная в настоящей работе картина распределения изменчивости согласуется с данными, полученными ранее для европейских видов Alyssum. В ряде работ [29, 34–36] использование различных типов маркеров (хлоропластной ДНК, ядерной – ITS-маркеры, или мультилокусный анализ) не дает филогении, которая бы согласовалась с классическим таксономическим видовым делением. Часто виды с небольшими ареалами или эндемики оказываются внутри клад широкоареальных видов на уровне межпопуляционной внутривидовой дифференциации.
Представители семейства Крестоцветные часто входят в состав флор, сформировавшихся на почвах с высоким содержанием тяжелых металлов. При этом у растений, произрастающих на таких почвах, наблюдается повышенное генетическое разнообразие [6, 37, 38]. Непосредственно связь между генетическим разнообразием растений и содержанием тяжелых металлов в почве или способностью их накопления в частях растений была изучена только для нескольких видов секции Odontarrhena – итальянского A. bertolonii и двух видов с северного Кавказа – A. murale и A. daghestanicum [5, 6]. Так, для A. bertolonii показана положительная корреляция между варьированием содержания Ni в почве и величиной показателя генетического разнообразия популяции по маркерам микросателлитов хпДНК [38]. Популяционный полиморфизм A. murale по ITS-маркеру разделил изученные образцы соответственно их аккумулирующей способности к Ni. Авторы показали, что гипераккумулирующая и негипераккумулирующая популяции вида A. murale генетически различаются (пятью мутациями и 16 полиморфными сайтами), в то время как другой вид, A. daghestanicum, не гипераккумулятор, оказался генетически мономорфным [6]. В описанные закономерности укладывается и выявленная в данном исследовании изменчивость уральских видов: способные к облигатной или факультативной аккумуляции виды A. obovatum и A. tortuosum оказались более изменчивыми, чем гемиаккумулятор A. litvinovii. Однако данные по изменчивости маркеров хлоропластной ДНК могут лишь косвенно отражать адаптационные возможности видов. Для выявления подобных закономерностей более подходящим будет использование мультилокусных ядерных маркеров. Например, на видах секции Odontarrhena в Албании [36] было показано различие между видами, облигатно произрастающими на серпентинитах и на других типах почв по количеству “outlier”-локусов, т.е. локусов выходящих за рамки распределения изменчивости при условии их нейтральности, а следовательно потенциально сцепленных с адаптационными признаками.
В ходе работы отобраны маркеры хпДНК, перспективные для выявления общей пространственно-генетической структуры исследуемых видов. Установлено близкое родство всех трех видов, генетическое разнообразие и наличие специфичных для видов A. obovatum и A. tortuosum гаплотипов, отсутствие генетического разнообразия у A. litvinovii. Увеличение выборки растений, больший территориальный охват их сбора и привлечение новых маркеров поможет составить более полное представление о филогении исследуемых видов. Относительно применения маркеров хпДНК можно говорить об эффективности их использования только при анализе методом секвенирования, выявляющим весь диапазон изменчивости. Несмотря на то, что ПДРФ-метод имеет преимущество за счет более низкой стоимости, в большинстве случаев он подходит для популяционных исследований только в качестве предварительной оценки генетической изменчивости и ее структуры.
Работа выполнена в рамках государственного задания Института экологии растений и животных УрО РАН № 122021000090-5, проведение молекулярно-генетического анализа поддержано грантом РФФИ № 16-04-01346.
Благодарим лабораторию молекулярной генетики ИЕНиМ за помощь в проведении сиквенсного анализа.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Van der Ent A., Baker A.J.M., Reeves R.D. et al. Hyperaccumulators of metal and metalloid trace elements: Facts and fiction // Plant and Soil. 2013. V. 362. № 1. P. 319–334. https://doi.org/10.1007/s11104-012-1287-3
Krämer U. Metal hyperaccumulation in plants // Annual Review of Plant Biol. 2010. V. 61. P. 517–534. https://doi.org/10.1146/annurev-arplant-042809-112156
Reeves R.D. Hyperaccumulation of trace elements by plants // Phytoremediation of Metal-Contaminated Soils. 2005. V. 68. P. 25–52.
Warwick S.I., Francis A., Gugel R.K. Guide to wild germplasm: Brassica and allied crops (tribe Brassiceae, Brassicaceae) Ottawa: Agriculture Agri-Food Research Canada, 2009. 19 p. http://www.brassica.info/info/publications/guide-wild-germplasm.php
Galardi F., Mengoni A., Pucci S. et al. Intra-specific differences in mineral element composition in the Ni-hyperaccumulator Alyssum bertolonii: A survey of populations in nature // Environmental and Exp. Botany. 2007b. V. 60. № 1. P. 50–56. https://doi.org/10.1016/j.envexpbot.2006.06.010
Drozdova I.V., Machs E., Kalimova I. et al. Accumulation of potentially toxic elements by plants of North Caucasian Alyssum species and their molecular phylogenetic analysis // Environ Geochem. Health. 2021. V. 43. № 4. P. 1617–1628. https://doi.org/10.1007/s10653-020-00674-4
Španiel S., Marhold K., Filová B., Zozomová-Lihová J. Genetic and morphological variation in the diploid-polyploid Alyssum montanum in Central Europe: taxonomic and evolutionary considerations // Plant Syst. and Evol. 2011. V. 294. № 1. P. 24–27. https://doi.org/10.1007/s00606-011-0438-y
Španiel S., Kempa M., Salmerón-Sánchez E. et al. AlyBase: Database of names, chromosome numbers, and ploidy levels of Alysseae (Brassicaceae), with a new generic concept of the tribe // Plant Systematics and Evolution. 2015. V. 301. № 10. P. 2463–2491. https://doi.org/10.1007/s00606-015-1257-3
Zozomová-Lihová J., Melichárková A., Svitok M., Španiel S. Pleistocene range disruption and postglacial expansion with secondary contacts explain the genetic and cytotype structure in the western Balkan endemic Alyssum austrodalmaticum (Brassicaceae) // Plant Syst. and Evol. 2020. V. 306. № 2. P. 1–25. https://doi.org/10.1007/s00606-020-01677-5
German D.A. (2058) Proposal to conserve Odontarrhena obovatum (Alyssum obovatum), nom. cons. prop., against Alyssum fischerianum (Cruciferae) // Taxon. 2012a. V. 61. № 2. P. 470. https://doi.org/10.1002/tax.612023
Буш Н.А. Cruciferae // Флора Сибири и Дальнего Востока. 1913. Т. 1. № 34. С. 491–714.
Толмачёв А.И. Сем. Cruciferae Juss. – Крестоцветные // Флора Забайкалья. 1949. № 5. С. 419–471.
Nyárády E.J. Synopsis specierum, variatiorum et formarum sectionis Odontarrhenae generis Alyssum // Anal. Acad. Rep. Pop. Române. 1949. Ser. A. V. 1. Mem. 3. P. 67–199.
German D.A. Taxonomical confusions in the Cruciferae of North and Central Asia. I. Alyssum fischerianum and Alyssum canescens // Turczaninowia. 2011b. V. 14. № 4. P. 18–28.
Teptina A.Y., Paukov A.G. Nickel accumulation by species of Alyssum and Noccaea (Brassicaceae) from ultramafic soils in the Urals, Russia // Austr. J. Botany. 2015. V. 63. № 2. P. 78–84. https://doi.org/10.1071/bt14265
Князев М.С. Заметки о некоторых видах крестоцветных (Brassicaceae) на Урале и сопредельных территориях // Новости сист. высш. растений. 2011. Т. 42. С. 136–146.
Горчаковский П.Л., Шурова Е.А., Князев М.С. и др. Определитель сосудистых растений Среднего Урала. М.: Наука, 1994. 525 с.
Рябинина З.Н., Князев М.С. Определитель сосудистых растений Оренбургской области. М.: Товарищество научных изданий КМК, 2009. 758 с.
Devey M.E., Bell J.C., Smith D.N. et al. A genetic linkage map for Pinus radiata based on RFLP, RAPD and microsatellite markers // Theor. Appl. Genet. 1996. V. 92. P. 673–679. https://doi.org/10.1007/BF00226088
Demesure B., Sodzi N., Petit R.J. A set of universal primers for amplification of polymorphic non-coding regions of mitochondrial and chloroplast DNA in plants // Mol. Ecol. 1995. V. 4. № 1. P. 129–134. https://doi.org/10.1111/j.1365-294X.1995.tb00201.x
Dumolin-Lapegue S., Pemonge M.H., Petit R.J. An enlarged set of consensus primers for the study of organelle DNA in plants // Mol. Ecol. 1997. V. 6. № 4. P. 393–397. https://doi.org/10.1046/j.1365-294x.1997.00193.x
Taberlet P., Gielly L., Pautou G., Bouvet J. Universal primers for amplification of three non-coding regions of chloroplast DNA // Plant Mol. Biol. 1991. V. 17. № 5. P. 1105–1109.https://doi.org/10.1007/BF00037152
Shaw J., Lickey E.B., Beck J.T. et al. The tortoise and the hare II: Relative utility of 21 noncoding chloroplast DNA sequences for phylogenetic analysis // Am. J. Botany. 2005. V. 92. № 1. P. 142–166. https://doi.org/10.3732/ajb.92.1.142
Rozen S., Skaletsky H.J. Primer 3 on the WWW for general users and for biologist programmers // Bioinformatics Methods and Protocols: Methods in Molecular Biology. N.J.: Humana Press, 2000. P. 365–386.
Hall T.A. Bioedit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT // Nucl. Acids Symp. Ser. 1999. V. 41. P. 95–98. https://doi.org/10.1111/jbi.12867
Ronquist F., Huelsenbeck J.P. MrBAYES 3: Bayesian phylogenetic inference under mixed models // Bioinformatics. 2003. V. 19. № 12. P. 1572–1574. https://doi.org/10.1093/bioinformatics/btg180
Rambaut A. FigTree v1.3.1: Tree figure drawing tool // Mol. Evol., Phylogen. and Epidem. 2009. http://tree.bio.ed.ac.uk
Bandelt H.J., Forster P., Rohl A. Median-joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16. P. 37–48.https://doi.org/10.1093/oxfordjournals.molbev.a026036
Mengoni A., Baker A.J.M., Bazzicalupo M. et al. Evolutionary dynamics of nickel hyperaccumulation in Alyssum revealed by ITS nrDNA analysis // New Phytologist. 2003. V. 159. № 3. V. 691–699. https://doi.org/10.1046/j.1469-8137.2003.00837.x
Maddison W.P. Gene trees in species trees // Systematic Biology. 1997. V. 46. № 3. P. 523–536. https://doi.org/10.1093/sysbio/46.3.523
Flagel L., Udall J., Nettleton D., Wendel J. Duplicate gene expression in allopolyploid Gossypium reveals two temporally distinct phases of expression evolution // BMC Biology. 2008. V. 6. № 1. P. 16–19. https://doi.org/10.1186/1741-7007-6-16
Gurushidze M., Fritsch R.M., Blattner F.R. Species-level phylogeny of Allium subgenus Melanocrommyum: Incomplete lineage sorting, hybridization and trnF gene duplication // Taxon. 2010. V. 59. № 3. P. 829–840. https://doi.org/10.1002/tax.593012
Warwick S.I., Al-Shehbaz I.A. Brassicaceae: chromosome number index and database on CD-Rom // Plant Syst. and Evol. 2006. V. 259. № 2. P. 237–248. https://doi.org/10.1007/s00606-006-0421-1
Li Y., Kong Y., Zhang Z. et al. Phylogeny and biogeography of Alyssum (Brassicaceae) based on nuclear ribosomal ITS DNA sequences // J. Genet. 2014. V. 93. № 2. P. 313–323. https://doi.org/10.1007/s12041-014-0362-3
Zozomová-Lihová J., Marhold K., Španiel S. Taxonomy and evolutionary history of Alyssum montanum (Brassicaceae) and related taxa in southwestern Europe and Morocco: Diversification driven by polyploidy, geographic and ecological isolation // Taxon. 2014. V. 63. № 3. P. 562–591. https://doi.org/10.12705/633.18
Coppi A., Baker A.J., Bettarini I. et al. Population genetics of Odontarrhena (Brassicaceae) from Albania: The effects of anthropic habitat disturbance, soil, and altitude on a Ni-Hyperaccumulator plant group from a major serpentine hotspot // Plants. 2020. V. 9. № 12. P. 1686. https://doi.org/10.3390/plants9121686
Федоренко О.М., Зарецкая М.В., Лебедева О.Н., Титов А.Ф. Генетическое разнообразие природных популяций Arabidopsis thaliana (L.), расположенных на северной периферии ареала вида // Тр. Карельского науч. центра РАН. 2014. № 2. С. 36–42.
Galardi F., Corrales I., Mengoni A. et al. Intra-specific differences in nickel tolerance and accumulation in the Ni-hyperaccumulator Alyssum bertolonii // Envir. and Exp. Botany. 2007a. V. 60. № 3. P. 377–384. https://doi.org/10.1016/j.envexpbot.2006.12.011
Дополнительные материалы отсутствуют.


