Генетика, 2022, T. 58, № 9, стр. 1021-1028
Микробиом кишечника человека – резервуар аминогликозид-N-ацетилтрансфераз – генов лекарственной устойчивости
М. Г. Алексеева 1, *, Н. В. Захаревич 1, А. В. Ратькин 1, В. Н. Даниленко 1
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
* E-mail: Alekseevamg@mail.ru
Поступила в редакцию 22.03.2022
После доработки 31.03.2022
Принята к публикации 12.04.2022
- EDN: TMFMHV
- DOI: 10.31857/S0016675822090028
Аннотация
Микробиом кишечника человека представляет собой сложную экосистему, играющую ключевую роль в поддержании общего гомеостаза, и является динамическим резервуаром генов устойчивости к антибиотикам, которые могут передаваться между бактериями в смешанных популяциях. Одними из основных ферментов, осуществляющих ацетилирование широкого спектра субстратов, являются аминогликозид-N-ацетилтрансферазы (ААС). Ранее у Enterococcus и Staphylococcus был идентифицирован и охарактеризован бифункциональный фермент AAC(6')-Ie/APH(2'')-Ia, являющийся клинически важным. В рамках данного исследования в секвенированных геномах штаммов, относящихся к Bacteroides и Lactobacillus, выявлены гены, кодирующие AAC(3). В геномах штаммов, относящихся к большинству видов Bacteroides, Enterococcus и Lactobacillus, выявлены гены, кодирующие Eis (от англ. Enhsnced intracellular survival). Последовательности всех выявленных AAC(3) и Eis не имеют сходства с последовательностями известных ферментов. AAC(3) и Eis у данных бактерий могут иметь разные функции в организме человека, в том числе участвовать в ацетилировании пептидов, белков и других субстратов.
Микробиом кишечника человека представляет собой сложную экосистему, играющую ключевую роль в поддержании общего гомеостаза, и является динамическим резервуаром генов устойчивости к антибиотикам. В кишечнике здоровых людей обнаружено около 500 видов микроорганизмов, из них 66 видов являются доминирующими [1, 2]. Лечение с использованием антибактериальных агентов оказывает значительное влияние на резистом кишечника и приводит к увеличению горизонтального переноса и селекции устойчивых форм. Мощный методологический подход к изучению кишечной микробиоты обеспечивает современный уровень технологии секвенирования геномов [3]. Сравнение последовательностей генов устойчивости показывает, что комменсальные кишечные бактерии могут быть резервуарами генов устойчивости для других видов бактерий (в том числе патогенных для человека) [4].
Устойчивость к аминогликозидным антибиотикам обусловлена наличием в геномах бактерий модифицирующих ферментов, относящихся к трем подклассам: аминогликозид-N-ацетилтрансферазам (AAC), аминогликозиднуклеотидилтрансферазам (АNТ) и аминогликозидфосфотрансферазам (APH). Аминогликозид-N-ацетилтрансферазы (AAC) катализируют ацетилирование групп NH2 в молекуле аминогликозидов с использованием ацетилкофермента A. В зависимости от аминогруппы антибиотика, модифицируемой ферментом, различают четыре подсемейства ААС: AAC(1), AAC(3), AAC(2') и AAC(6') [5–7].
Наиболее распространенными и исследованными являются ферменты AAC(6'), они присутствуют у грамотрицательных и грамположительных бактерий. Гены, кодирующие эти ферменты, были обнаружены на плазмидах и хромосомах и/или являются частью мобильных генетических элементов [6, 8]. Ферменты AAC(6') могут существовать в виде слитых белков, занимающих N- или C-концевую область композитного белка. Интересно, что белки, содержащие ферменты подкласса AAC(6')-I, были обнаружены слитыми с APH, ANT или другими AAC. Наиболее хорошо исследован фермент AAC(6')-Ie/APH(2'')-Ia, идентифицированный у Enterococcus faecalis и стафилококков [9].
Недавно у Мycobacterium tuberculosis был идентифицирован белок, повышающий внутриклеточную выживаемость микобактерий в макрофагах, названный Eis (от англ. Enhanced intracellular survival), который является новым классом AAC, контролирующим выработку провоспалительных цитокинов и способным ацетилировать несколько аминогрупп различных аминогликозидных антибиотиков, используя не только ацетил-КоА, но и другие производные этого кофактора [10].
Аминогликозид-N-ацетилтрансферазы, и в частности AAC и Eis, относятся к moonlighting-белкам, т.е. проявляют полифункциональные свойства. Так, у микобактерий ААС вносят частичный вклад в устойчивость к аминогликозидам и связаны с другими бактериальными функциями и способствуют ацетилированию белков клеточной стенки и пептидогликана [10]. Белки Eis обладают способностью ацетилировать не только антибиотики, но и широкий спектр пептидов и белков, включая эукариотические гистоны (в том числе N3-концевой пептид гистона человека H3) [11].
Eis у бактерий представляют собой новый тип арилалкиламин-N-ацетилтрансфераз, поскольку они способны ацетилировать арилалкиламины (например, гистамин, дофамин, октопамин, тирамин, триптамин, норэпинефрин, метокситриптамин, серотонин и 5-гидрокситриптамин), которые являются биологически активными соединениями с различными физиологическими функциями в нервной, нейроэндокринной и иммунной системах человека. Установлено, что Eis M. tuberculosis ацетилирует гистамин и октопамин, а Eis M. smegmatis использует тирамин и октопамин в качестве субстратов. Ферменты Eis обнаружены также у других бактерий, в основном грамположительных – у Bacillales, Lactobacillales и Clostridiales. У многих бактерий также аннотированы белки с другими функциями, содержащие домены Eis [12].
Известно, что бактерии-комменсалы способны продуцировать различные классы веществ, в том числе ферменты с нейромодулирующей и иммуномодулирующей активностью, поэтому очевидно, что ферменты ААС (в том числе Eis) у бактерий-комменсалов могут играть важную роль во взаимодействиях с организмом хозяина. Однако в современной литературе отсутствуют публикации по идентификации и изучению функций данных ферментов у бактерий-комменсалов микробиоты кишечника человека.
Цель настоящего исследования – биоинформатический анализ распространения генов aac в секвенированных геномах основных таксонов бактерий-комменсалов микробиоты кишечника человека Enterococcus, Bacteroides и более детальный анализ для представителей рода Lactobacillus; сравнение выявленных генов между собой и поиск ближайших гомологов из геномов других бактерий (в том числе патогенных).
МАТЕРИАЛЫ И МЕТОДЫ
Биоинформатический анализ. Последовательности генов и белков аминогликозид-N-ацетилтрансфераз Bacteroides, Enterococcus и Lactobacillus, обсуждаемые в статье, были получены из базы данных NCBI (http://www.ncbi.nlm.nih.gov/). Для сравнения их с известными по литературе использовали базу данных неповторяющихся белковых последовательностей (“Non-redundant protein sequences”, NCBI) и программы Blastp [13] и Clustal Omega [14], последнюю для множественного выравнивания белковых последовательностей.
РЕЗУЛЬТАТЫ
Анализ распространения генов аминогликозид-N-ацетилтрансфераз у Bacteroides
Род Bacteroides – одна из преобладающих групп в желудочно-кишечном тракте (ЖКТ) человека. Важность Bacteroides дополнительно иллюстрируется тем фактом, что этот род бактерий является наиболее стабильным компонентом желудочно-кишечной микробиоты с течением времени у здоровых взрослых людей.
Первый вид рода Bacteroides – Bacteroides fragilis был выделен в 1898 г. как человеческий патоген, связанный с аппендицитом. B. fragilis является частью нормальной микробиоты толстой кишки человека, однако нарушение поверхности слизистой оболочки способствует распространению B. fragilis в кровоток, что приводит к клинически значимой инфекции [15].
Наиболее часто и в заметном количестве в кишечнике здорового человека обнаруживают следующие виды Bacteroides: B. vulgatus (Phocaeicola vulgatus), B. dorei (Phocaeicola dorei), B. uniformis и B. ovatus [16].
Биоинформатический анализ секвенированных геномов штаммов Bacteroides для данных видов показал наличие генов, кодирующих аминогликозид-N-ацетилтрансферазы, относящиеся к двум классам: AAC(3) и Eis (табл. 1).
Таблица 1.
Наличие аминогликозид-N-ацетилтрансфераз у видов рода Bacteroides (Phocaeicola)
| Вид | N-ацетилтрансфераза семейства AAC(3) | Eis (N-ацетилтрансфераза семейства GNAT) | ||||
|---|---|---|---|---|---|---|
| название штамма | размер, а/к | ссылка на GenBank | название штамма | размер, а/к | ссылка на GenBank | |
| Phocaeicola dorei | Отсутствуют гены, кодирующие ААС | RJX1045 | 336 | TDB22695 E1J03_20575 |
||
| Bacteroides fragilis | 3783N1-6 | 305 | EYB10660 M119_1197 |
3783N1-6 | 339 | EYB10962 M119_0490 |
| S24L34 | 300 | EYA78531 M134_4032 |
S24L34 | 339 | EYA82370 M134_0485 |
|
| CL03T12C07 | 283 | QUU02560 INE73_00840 |
CL03T12C07 | 339 | QUU05501 INE73_03854 |
|
| Bacteroides ovatus | Отсутствуют гены, кодирующие ААС | am_0171 | 338 | RYT75558 EAJ07_03790 |
||
| Bacteroides uniformis | AM50-4 | 308 | RGZ49523 DW988_07985 |
AM50-4 | 338 | RGZ50150 DW988_06155 |
| Phocaeicola vulgatus | Отсутствуют гены, кодирующие ААС | ATCC 8492 | 336 | PQL54778 C5Z04_02675 |
||
Штаммы, относящиеся к видам Phocaeicola dorei, Bacteroides ovatus и Phocaeicola vulgatus, содержат только по одному гену, кодирующему ацетилтрансферазы Eis. Штаммы, относящиеся к видам Bacteroides uniformis и Bacteroides fragilis, содержат одновременно по одному гену, кодирующему ацетилтрансферазы AAC(3), и по одному гену, кодирующему ацетилтрансферазы Eis, расположенные в разных генных кластерах.
Штаммы, относящиеся к виду Bacteroides fragilis, по наличию гена, кодирующего AAC(3), можно разделить на три группы, кодирующие ферменты размером 305, 300 и 283 а/к. Аминокислотные последовательности Eis из разных штаммов Bacteroides fragilis идентичны.
Мы сравнили между собой аминокислотные последовательности Eis из пяти различных видов Bacteroides: Phocaeicola dorei, B. fragilis, B. ovatus, B. uniformis и Phocaeicola vulgatus. Идентичность ацетилтрансферазы Eis из P. dorei и из P. vulgatus составила 97%. Идентичности ацетилтрансфераз из B. fragilis, B. ovatus и B. uniformis лежат в диапазоне от 55 до 60%. В то же время идентичность между последовательностями ацетилтрансфераз из видов Phocaeicola и Bacteroides не превышает 43%.
При поиске ацетилтрансфераз, сходных с ацетилтрансферазами из P. dorei и P. vulgatus, наиболее близкой (идентичность 98–99%) оказалась ацетилтрансфераза из Niabella sp. и с более низким процентом идентичности (39–45) были ацетилтрансферазы из видов Oscillospiraceae bacterium, Catenibacterium mitsuokai, Escherichia coli и Enterobacter hormaechei.
Niabella sp. также относится к группе Bacteroides, Oscillospiraceae bacterium – анаэробная бактерия из микробиома человека, Catenibacterium mitsuokai – грамположительная анаэробная бактерия, выделенная из фекалий человека, относится к подтипу Clostridium. Бактерии Escherichia coli и Enterobacter hormaechei являются условно-патогенными для человека.
Для ацетилтрансфераз из B. fragilis, B. ovatus и B. uniformis наиболее близкими были те же пять ацетилтрансфераз, только идентичность была чуть выше для четырех (53–68%), а для ацетилтрансферазы из Niabella sp. – ниже (42%). При этом последовательность ацетилтрансферазы из E. hormaechei была идентична последовательности ацетилтрансферазы из B. ovatus, а последовательность ацетилтрансферазы из C. mitsuokai была идентична последовательности ацетилтрансферазы из B. uniformis.
Анализ распространения генов аминогликозид-N-ацетилтрансфераз у Enterococcus
Энтерококки – многочисленная группа условно-патогенных бактерий, в микробиоте ЖКТ человека в основном встречаются виды Enterococcus faecium и Enterococcus faecalis [17].
Ранее у Enterococcus был идентифицирован и охарактеризован бифункциональный фермент AAC(6')-Ie/APH(2'')-Ia, являющийся клинически важным, ответственным за высокий уровень устойчивости к аминогликозидным антибиотикам. Этот уникальный фермент с широкой субстратной специфичностью содержит N-концевой домен AAC(6') и С-концевой домен APH(2''); оба домена могут функционировать независимо и посредством ацетилирования и/или фосфорилирования инактивировать большинство аминогликозидных антибиотиков [9].
Нами проведен биоинформатический анализ последовательностей секвенированных геномов штаммов Е. faecalis и Е. faecium.
Анализ показал, что все штаммы Е. faecalis содержат ген, кодирующий бифункциональный фермент AAC(6')-Ie/APH(2'')-Ia, а также по два гена, кодирующих аминогликозид-N-ацетилтрансферазы класса Eis. Например, геном штамма Е. faecalis 1207/14 содержит гены, кодирующие: бифункциональный фермент AAC(6')-Ie/APH(2'')-Ia размером 479 аминокислот (а/к) (GenBank: QWW17314.1), Eis размером 403 а/к (GenBank: QWW17706.1) и Eis размером 406 а/к (GenBank: QWW15801.1). Все гены, обнаруженные в штаммах Е. faecalis, расположены в разных генных кластерах.
Анализ геномов Е. faecium показал, что все штаммы содержат ген, кодирующий бифункциональный фермент AAC(6')-Ie/APH(2'')-Ia, а также один ген, кодирующий AAC(6), и два гена, кодирующих Eis. Все обнаруженные гены расположены в разных генных кластерах в геномах штаммов Е. faecium. Так, например, геном штамма Е. faecium U-1313438 содержит гены, кодирующие: бифункциональный фермент AAC(6')-Ie/APH(2'')-Ia размером 479 а/к (GenBank: KGQ77505.1), AAC(6) размером 182 а/к (GenBank: KGQ77679.1), Eis размером 393 а/к (GenBank: KGQ77148.1) и Eis размером 406 а/к (GenBank: KGQ76559.1).
Аминокислотные последовательности бифункциональных ферментов AAC(6')-Ie/APH(2'')-Ia штаммов E. faecium и E. faecalis оказались идентичны. Последовательность дополнительного AAC(6) E. faecium не имеет сходства с последовательностью бифункционального фермента AAC(6').
Аминокислотные последовательности бифункциональных ферментов AAC(6')-Ie/APH(2'')-Ia E. faecium и E. faecalis также оказались практически идентичны (99%) последовательностям бифункциональных ферментов AAC(6')-Ie/APH(2'')-Ia, идентифицированным у патогенных бактерий рода Staphylococcus.
Анализ распространения генов аминогликозид-N-ацетилтрансфераз у Lactobacillus
Лактобациллы – это широко распространенные бактерии, включающие многие виды и являющиеся одними из наиболее важных компонентов кишечной микробиоты человека, включающие множество пробиотических штаммов, обеспечивающих защиту кишечной флоры от патогенов и стимуляцию иммунной системы. Их широкое использование в пищевой и фармацевтической промышленности продиктовано длительной историей безопасного применения. Однако наличие генов вирулентности и устойчивости к антибиотикам, а также их потенциальная передача патогенным микроорганизмам представляют риск, который следует учитывать [18].
Известно, что лактобациллы более устойчивы к аминогликозидам и ванкомицину и чувствительны к эритромицину, бета-лактамным антибиотикам, хлорамфениколу и тетрациклину. Результаты анализа бактериальных геномов показали наличие у Lactobacillus генов устойчивости к тетрациклину tet(W), наибольшее количество генов устойчивости к антибиотикам было перенесено с мобильными генетическими элементами [19].
Проведенный нами биоинформатический анализ секвенированных геномов штаммов лактобацилл показал наличие генов, кодирующих аминогликозид-N-ацетилтрансферазы, относящиеся к двум классам: AAC(3) и Eis (табл. 2). Идентичность последовательностей ААС из разных штаммов одного вида составляет 98–100%.
Таблица 2.
Наличие аминогликозид-N-ацетилтрансфераз у лактобацилл
| Лактобациллы | N-ацетилтрансфераза семейства AAC(3) | Eis (N-ацетилтрансфераза семейства GNAT) | ||||
|---|---|---|---|---|---|---|
| название штамма | размер, а/к | ссылка на GenBank | название штамма | размер, а/к | ссылка на GenBank | |
| Lactobacillus acidophilus | DSM 20079 | 274 | AVW87698 LA20079_08190 |
DSM 20079 | 388 | AVW86996 LA20079_04400 |
| Lacticaseibacillus casei | Отсутствуют гены, кодирующие ААС | DS1_13 | 405 | PTU95687 DB330_06535 |
||
| Lactobacillus crispatus | ATCC 33820 | 271 | QWW28104 J6L97_05725 |
ATCC 33820 | 388 | QWW29972 J6L97_04615 |
| Lactobacillus gasseri | ATCC 33323 | 272 | ABJ60414 LGAS_1039 |
ATCC 33323 | 386 | ABJ59439 LGAS_0024 |
| Lactobacillus helveticus | LMG 22465 | 268 | GFP05459 LMG22465_14720 |
LMG 22465 | 388 | GFP05532 LMG22465_15450 |
| Lactobacillus jensenii | DSM 20557 | 269 | KRM51187 FC45_GL000617 |
Отсутствуют гены, кодирующие аминогликозид-N-ацетилтрансферазу Eis | ||
| Lactobacillus johnsonii | ATCC 33200 | 269 | KRK56013 FC22_GL000158 |
ATCC 33200 | 387 | KRK54121 FC22_GL000316 |
| Lacticaseibacillus paracasei | Отсутствуют гены, кодирующие ААС | DTA72 | 405 | MXI84121 GRZ59_10270 |
||
| Lactobacillus paragasseri | JCM 11657 | 275 | GIL32313 PGA11657_02420 |
JCM 11657 | 346 | GIL32979 PGA11657_09080 |
| Lactiplantibacillus plantarum | Отсутствуют гены, кодирующие ААС | 90sk | 393 | KIN20067 SC12_09630 |
||
| Lacticaseibacillus rhamnosus | Отсутствуют гены, кодирующие ААС | K32 | 405 | KFC37507 LRK_00405 |
||
| Latilactobacillus sakei | Отсутствуют гены, кодирующие ААС | NBRC 3541 | 403 | GEA76977 LSA01_10560 |
||
| 392 | GEA76583 LSA01_06620 |
|||||
| Lactobacillus ultunensis | Kx293C1 (DSM 16048) | 239 | QQP29480 H4B44_05395 |
Kx293C1 (DSM 16048) | 388 | QQP29371 H4B44_04780 |
Штаммы, относящиеся к видам L. acidophilus, L. crispatus, L. gasseri, L. helveticus, L. johnsonii, L. paragasseri и L. ultunensis, содержат одновременно по одному гену, кодирующему ацетилтрансферазы AAC(3), и по одному гену, кодирующему ацетилтрансферазы Eis. Данные гены расположены в разных генных кластерах.
Штаммы, относящиеся к виду L. jensenii, содержат только по одному гену, кодирующему ацетилтрансферазы AAC(3). Штаммы, относящиеся к видам L. casei, L. paracasei, L. plantarum, L. rhamnosus и L. sakei, содержат только гены, кодирующие ацетилтрансферазы Eis, причем штаммы L. sakei содержат два гена, кодирующих Eis размером 403 и 392 а/к.
Штаммы, относящиеся к видам L. fermentum, L. reuteri, L. ruminis и L. salivarius, не содержат генов, кодирующих аминогликозид-N-ацетилтрансферазы.
Для подсемейства AAC(3) было идентифицировано одиннадцать подклассов ферментов AAC(3)-I–AAC(3)-XI, гены которых были обнаружены в основном на плазмидах или мобильных генетических элементах грамотрицательных клинических изолятов и у продуцентов аминогликозидных антибиотиков. Подкласс AAC(3)-V был исключен после подтверждения того, что единственный фермент в этой группе идентичен AAC(3)-II [5, 7].
Нами не было обнаружено сходства между рассматриваемыми в настоящей работе генами, кодирующими аминогликозид-N-ацетилтрансферазы класса AAC(3), и описанными ранее в литературе генами подсемейства AAC(3). Поэтому мы провели сравнение анализируемых аминогликозид-N-ацетилтрансфераз класса AAC(3) между собой, а также поиск наиболее похожих по аминокислотной последовательности ферментов по базе данных NCBI.
При сравнении ацетилтрансфераз друг с другом была выявлена довольно высокая консервативность последовательностей по всей длине (идентичность 70–75%). Исключением стала последовательность L. jensenii – при сравнении с другими анализируемыми последовательностями идентичность для нее не превысила 33%. Стоит также отметить, что идентичность между последовательностями видов L. casei и L. parcasei составила 99%.
При поиске наиболее близких по аминокислотной последовательности ацетилтрансфераз класса AAC(3) в других бактериальных родах наибольший процент идентичности был отмечен для последовательностей ацетилтрансфераз из видов Bifidobacterium vespertilionis и Scardovia wiggsiae – 41 и 42 соответственно. B. vespertilionis – данный вид бифидобактерий выделен из микробиома млекопитающих. Scardovia wiggsiae – патогенная бактерия микробиома полости рта, тесно связанная с кариесом зубов.
Представители нового класса ацетилтрансфераз Eis – ферменты с многофункциональными свойствами, обнаружены относительно недавно и охарактеризованы лишь для ряда патогенных бактерий класса Mycobacterium, в том числе для Mycobacterium tuberculosis.
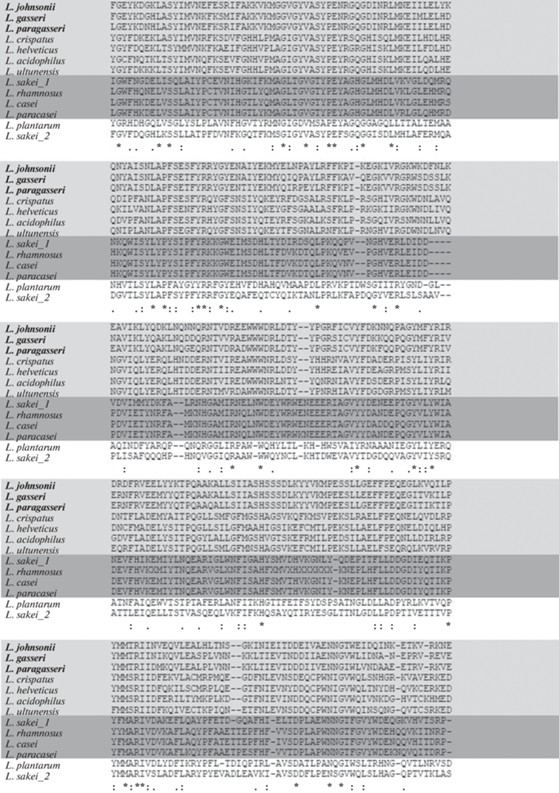
Сравнительный анализ ацетилтрансфераз Eis лактобацилл позволил разделить их на пять групп (на основании сходства аминокислотных последовательностей) (рис. 1). Ацетилтрансферазы L. sakei_1, L. rhamnosus, L. casei и L. paracasei довольно сильно похожи друг на друга и отличаются от остальных, их можно выделить в отдельную группу. Следующие две группы ацетилтрансфераз можно рассматривать либо как самостоятельные, либо как подгруппы одной группы – это L. johnsonii, L. gasseri, L. paragasseri и L. crispatus, L. helveticus, L. acidophilus, L. ultunensis (одна из предполагаемых подгрупп выделена полужирным шрифтом). Две оставшиеся ацетилтрансферазы L. plantarum и L. sakei_2 отличаются от всех остальных, но и между собой не так близки, чтобы относить их к одной группе. Сходства с изученными и описанными последовательностями Eis микобактерий найдено не было (идентичность <30% и выравнивание не по всей длине).
ОБСУЖДЕНИЕ
Ферменты с ацетилтрансферазной активностью представляют собой большой класс белков, широко распространенных у организмов из различных экосистем. Происхождение AAC можно проследить по микробным видам, представляющим огромный резервуар для новых, появляющихся ферментов устойчивости, которые в настоящее время недостаточно изучены. Присутствие антибиотик-модифицирующих AAC-ферментов в микробиоме человека необходимо учитывать при разработке новых лекарственных препаратов [18, 19].
Аминогликозид-N-ацетилтрансферазы патогенных бактерий человека зачастую способны проникать в кровяное русло и клетки человека, осуществляя ацетилирование широкого спектра субстратов (т.н. moonlighting-функции). Представители такого класса ацетилтрансфераз Eis – ферменты с многофункциональными свойствами, обнаружены и охарактеризованы относительно недавно и лишь для ряда патогенных бактерий класса Mycobacterium, в том числе для M. tuberculosis. В то же время первичный биоинформатический анализ показал, что гены, аннотированные как eis, широко представлены у других бактерий, в основном грамположительных – у Bacillales, Lactobacillales и Clostridiales [12]. Проведенный нами биоинформатический анализ показал, что у многих бактерий (в том числе бактерий микробиома человека) также аннотированы белки с другими функциями, содержащие домены Eis (неопубликованные данные).
В представленном исследовании был проведен анализ распространения генов аминогликозид-N-ацетилтрансфераз в секвенированных геномах основных таксонов бактерий комменсалов микробиоты кишечника человека. В геномах штаммов, относящихся к Bacteroides и Lactobacillus, выявлены гены, кодирующие AAC(3). В геномах штаммов, относящихся к большинству видов Bacteroides, Enterococcus и Lactobacillus, выявлены гены, кодирующие Eis.
Последовательности всех выявленных AAC(3) у штаммов Bacteroides и Lactobacillus не имеют сходства с последовательностями известных ферментов AAC(3)-I–AAC(3)-XI из грамотрицательных клинических изолятов и продуцентов аминогликозидных антибиотиков. Последовательности всех выявленных ферментов Eis у штаммов Bacteroides, Enterococcus и Lactobacillus не имеют сходства с последовательностями Eis патогенных бактерий (M. tuberculosis). Наибольшее сходство данные ферменты показывают с ферментами из других комменсальных и условно-патогенных бактерий микробиома человека и млекопитающих (например, с ацетилтрансферазами из Enterococcus).
Принимая во внимание тот факт, что бактерии микробиома способны продуцировать различные классы веществ, в том числе с нейромодулирующей и иммуномодулирующей активностью, ферменты AAC(3) и Eis у данных бактерий могут иметь разные функции в организме человека и участвовать в ацетилировании пептидов, белков и других субстратов. В связи с этим изучение функций выявленных AAC(3) и Eis ферментов у Lactobacillus и выяснение их возможной роли в коммуникации с организмом человека является темой дальнейших исследований.
Работа выполнена в рамках Государственного задания № 0092-2022-003; тема “Механизмы генетических процессов у микроорганизмов, растений, животных и человека”: “Микробиом кишечника человека: иммуномодулирующий и антиоксидантный потенциал”, подтема “Распространение и функции генов аминогликозид-N-ацетилтрансфераз в микробиоме почв и человека”.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Schloissnig S., Arumugam M., Sunagawa S. et al. Genomic variation landscape of the human gut microbiome // Nature. 2013. V. 493. № 7430. P. 45–50. https://doi.org/10.1038/nature11711
Rajilić-Stojanović M., de Vos W.M. The first 1000 cultured species of the human gastrointestinal microbiota // FEMS Microbiol. Rev. 2014. V. 38. № 5. P. 996–1047. https://doi.org/10.1111/1574-6976.12075
Heintz-Buschart A., Wilmes P. Human gut microbiome: function matters // Trends Microbiol. 2018. V. 26. № 7. P. 563–574. https://doi.org/10.1016/j.tim.2017.11.002
Willmann M., El-Hadidi M., Huson D.H. et al. Antibiotic selection pressure determination through sequence-based metagenomics // Antimicrob. Agents. Chemother. 2015. V. 59. № 12. P. 7335–7345. https://doi.org/10.1128/AAC.01504-15
Ramirez M.S., Tolmasky M.E. Aminoglycoside modifying enzymes // Drug. Resist. Updat. 2010. V. 13. № 6. P. 151–171. https://doi.org/10.1016/j.drup.2010.08.003
Ogawara H. Comparison of antibiotic resistance mechanisms in antibiotic-producing and pathogenic bacteria // Molecules. 2019. V. 24. № 19. P. 3430. https://doi.org/10.3390/molecules24193430
Favrot L., Blanchard J.S., Vergnolle O. Bacterial GCN5-related N-acetyltransferases: from resistance to regulation // Biochemistry. 2016. V. 55. № 7. P. 989–1002. https://doi.org/10.1021/acs.biochem.5b01269
Xu Z., Stogios P.J., Quaile A.T. et al. Structural and functional survey of environmental aminoglycoside acetyltransferases reveals functionality of resistance enzymes // ACS Infect. Dis. 2017. V. 3. № 9. P. 653–665. https://doi.org/10.1021/acsinfecdis.7b00068
Frase H., Toth M., Vakulenko S.B. Revisiting the nucleotide and aminoglycoside substrate specificity of the bifunctional aminoglycoside acetyltransferase (6')-Ie/aminoglycoside phosphotransferase(2'')-Ia enzyme // J. Biol. Chem. 2012. V. 287. № 52. P. 43262–43269. https://doi.org/10.1074/jbc.M112.416453
Sanz-García F., Anoz-Carbonell E., Pérez-Herrán E. et al. Mycobacterial aminoglycoside acetyltransferases: A little of drug resistance, and a lot of other roles // Front. Microbiol. 2019. V. 10. P. 46. https://doi.org/10.3389/fmicb.2019.00046
Houghton J.L., Biswas T., Chen W. et al. Chemical and structural insights into the regioversatility of the aminoglycoside acetyltransferase Eis // Chembiochem. 2013. V. 14. № 16. P. 2127–2135. https://doi.org/10.1002/cbic.201300359
Pan Q., Zhao F.L., Ye B.C. Eis, a novel family of arylalkylamine N-acetyltransferase (EC 2.3.1.87) // Sci. Rep. 2018. V. 8. № 1. P. 2435. https://doi.org/10.1038/s41598-018-20802-6
Altschul S.F., Madden T.L., Schaffer A.A. et al. Gapped BLAST and PSI-BLAST: A new generation of protein database search programs // Nucl. Acids Res. 1997. V. 25. № 17. P. 3389–3402. https://doi.org/10.1093/nar/25.17.3389
Sievers F., Wilm A., Dineen D. et al. Fast, scalable generation of high-quality protein multiple sequence alignments using Clustal Omega // Mol. Syst. Biol. 2011. V. 7. P. 539. https://doi.org/10.1038/msb.2011.75
Elsaghir H., Reddivari A.K.R. Bacteroides Fragilis // 2021 Jun 4. In: StatPearls [Internet]. Treasure Island (FL): StatPearls Publ., 2022 Jan. PMID: 31971708.
Lück R., Deppenmeier U. Genetic tools for the redirection of the central carbon flow towards the production of lactate in the human gut bacterium Phocaeicola (Bacteroides) vulgates // Appl. Microbiol. Biotechnol. 2022. V. 106. № 3. P. 1211–1225. https://doi.org/10.1007/s00253-022-11777-6
Shirvani-Rad S., Ejtahed H.S., Ettehad Marvasti F. et al. The role of gut microbiota-brain axis in pathophysiology of ADHD: A systematic review // Atten. Disord. 2022. V. 20. P. 10870547211073474. Online ahead of print.https://doi.org/10.1177/10870547211073474
Colautti A., Arnoldi M., Comi G. et al. Antibiotic resistance and virulence factors in lactobacilli: something to carefully consider // Food Microbiol. 2022. May. 103:103934. https://doi.org/10.1016/j.fm.2021.103934
Fatahi-Bafghi M., Naseri S., Alizehi A. Genome analysis of probiotic bacteria for antibiotic resistance genes // Antonie Van Leeuwenhoek. 2022. Jan 6. Epub ahead of print.https://doi.org/10.1007/s10482-021-01703-7
Дополнительные материалы отсутствуют.