Генетика, 2022, T. 58, № 9, стр. 1008-1020
Видоформирующий PFNA-оперон бифидобактерий: модули сенсорных белков Pkb2 и FN3, структура и распределение среди разных видов и штаммов бифидобактерий в микробиоме кишечника человека
В. Н. Даниленко 1, *, М. Г. Алексеева 1, Т. А. Кошенко 1, А. С. Ковтун 1, В. З. Незаметдинова 1
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
* E-mail: valerid@vigg.ru
Поступила в редакцию 22.03.2022
После доработки 31.03.2022
Принята к публикации 05.04.2022
- EDN: YURJJD
- DOI: 10.31857/S0016675822090077
Аннотация
Недавно нами был открыт и исследован видоспецифический (видоформирующий) оперон PFNA бифидобактерий. Гены pkb2 и fn3 оперона кодируют белки, которые могут участвовать в восприятии сигналов организма хозяина. Было показано, что ген fn3 кодирует FN3-белок, содержащий два фибронектиновых домена с мотивами цитокиновых рецепторов. Фрагмент этого белка способен селективно связываться с TNFα. Предполагается, что PFNA-оперон Bifidobacterium longum subsp. longum GT15 контролирует двунаправленную коммуникацию с элементами (цитокинами) иммунной системы человека. В данной работе с использованием баз данных секвенированных геномов видов бифидобактерий – обитателей микробиоты желудочно-кишечного тракта человека, исследовали внутривидовой полиморфизм сенсорного С-концевого домена Pkb2 (SCD Pkb2 – sensor C-end domain) и фрагмента белка FN3, содержащего два FN3-домена (2D FN3). В изучаемых доменах каждого из восьми исследованных видов бифидобактерий различного географического происхождения обнаружены значимые неповторяющиеся аминокислотные замены, позволяющие выделить от двух до пяти групп у каждого вида бифидобактерий. С использованием программы trRosetta показано, что обнаруженные замены аминокислот могут приводить к изменению 3D-структур анализируемых доменов. Предполагается, что обнаруженная кластеризация штаммов может отражать гетерогенность в популяциях бифидобактерий у людей, из которых они были выделены, по иммунному статусу. Сформированы каталоги белков SCD Pkb2 и 2D FN3, включающие значимые замены аминокислот у восьми исследуемых видов, относящихся к четырем филогенетическим группам: B. adolescentis, B. bifidum, B. longum и B. pseudolongum. С использованием ранее разработанных алгоритмов и созданного каталога установили встречаемость исследуемых групп штаммов в метагеномах здоровых жителей Москвы двух возрастных групп: дети 3–5 лет и взрослые старше 18 лет. Разработанный подход предполагается использовать для изучения корреляции различного иммунного статуса и наличия в микробиоте штаммов бифидобактерий, принадлежащих к конкретным группам, кластеризованным по заменам аминокислот в SCD Pkb2 и 2D FN3.
Бифидобактерии – анаэробные бактерии, существовавшие на планете Земля до появления на ней кислорода и после его появления, заселившие анаэробные полости практически всех представителей животного мира от насекомых до человека [1–3]. Род Bifidobacterium насчитывает 99 видов, одиннадцать из которых встречаются в ЖКТ человека [3, 4]. Человеческие виды бифидобактерий относятся к четырем филогенетическим группам: B. adolescentis, B. bifidum, B. longum, B. pseudolongum [3]. Частоты встречаемости разных видов у человека существенно различаются. Также различаются частоты встречаемости одного и того же вида у детей и взрослых [5–7]. Бифидобактерии участвуют в формировании иммунной системы своих хозяев [8–10]. Хорошо известно, что подвид B. longum subsp. infantis и вид B. bifidum первыми после рождения заселяют кишечник человека и формируют иммунную систему ребенка [7, 11]. Интенсивно исследуется роль бифидобактерий и их компонентов (метаболитов) в коммуникации кишечник–мозг и кишечник–иммунная система [12, 13]. Относительно хорошо изучены метаболиты и компоненты клеток бифидобактерий, модулирующие с помощью тех или иных механизмов активность иммунной системы [14, 15]. Вместе с тем, практически ничего не известно о сигнальных системах (белках, рецепторах и др.), способных воспринимать сигналы иммунной системы, в том числе осуществлять взаимодействия с цитокинами [9, 16, 17].
Ученые лаборатории генетики микроорганизмов ИОГен РАН открыли и исследовали видоспецифический оперон PFNA бифидобактерий, ответственный за такие взаимодействия [18, 19]. Оперон PFNA встречается в геномах большинства видов бифидобактерий [13], включая 11 видов, встречающихся у человека. В состав оперона входят пять основных генов: pkb2, fn3, aaa-atp, duf58, tgm. Серин-треониновая протеин киназа Pkb2, кодируемая геном pkb2, является белком сигнальной трансдукции и состоит из N-концевого внутриклеточного каталитического домена, трансмембранного домена и C-концевого сенсорного внеклеточного домена (SCD), способного связываться с сигналом внешней среды (лигандом). На данный момент лиганд неизвестен, и его идентификация является важной научной и практической задачей. Ген fn3 кодирует FN3-белок, содержащий два фибронектиновых домена с мотивами цитокиновых рецепторов. Впервые в мире нами было показано, что фрагмент FN3-белка способен селективно связываться с фактором некроза опухоли TNFα [17].
В статье представлены результаты исследований полиморфизма SCD Pkb2 и фрагмента белка FN3, содержащего два FN3-домена (2D FN3), с целью выявить основные группы аминокислотных замен, получить прогноз влияния замен на пространственные структуры доменов, а также определить встречаемость различных групп аминокислотных замен в метагеномах российских жителей (детей и взрослых).
МАТЕРИАЛЫ И МЕТОДЫ
Поиск последовательностей доменов сенсорных белков SCD Pkb2 и 2D FN3 в метагеномных данных
Полные метагеномы для проведения биоинформатического анализа были получены нами и внесены в базу данных NCBI SRA ранее, в рамках реализации проектов РНФ № 17-15-01488 и № 20-14-00132. Всего в анализируемое множество вошло 23 метагенома микробиоты кишечника здоровых детей в возрасте 3–5 лет (идентификационный номер в базе данных NCBI SRA – PRJNA516054) и 38 метагеномов микробиоты кишечника здоровых взрослых людей (PRJNA762199). Отбор здоровых детей и взрослых проводился среди жителей г. Москвы. В группы включались добровольцы, соответствующие следующим критериям: отсутствие психиатрического заболевания, отсутствие острых инфекционных заболеваний и хронических аутоиммунных заболеваний, отсутствие соматических заболеваний, способных повлиять на результаты молекулярного тестирования (например, ВИЧ, рак, диабет), прием курса антибиотиков не позднее трех месяцев до даты взятия фекальных проб, хронический прием антибиотиков. Оценка психологического здоровья детей проводилась в соответствии с диагностическим и статистическим руководством по психическим расстройствам пятого издания (DSM-V), а взрослых – в соответствии с опросником центра эпидемиологических исследований (CES-D) и опросником генерализированного тревожного расстройства (ГТР-7). Оценка качества метагеномных чтений была проведена при помощи программы FastQC версии 0.11.9 [20], а исправление ошибок секвенирования и удаление адаптеров – при помощи Trimmomatic версии 0.39 [21]. Далее чтения были собраны в контиги с использованием metaSPAdes версии 3.14.1 [22]. Поиск аминокислотных последовательностей доменов сенсорных белков SCD Pkb2 и 2D FN3 из соответствующих каталогов выполнялся в контигах программой BLASTx версии 2.10.1 [23]. Отбор результатов проводился по критериям: гомология между определенной последовательностью и референсом не менее 90%, а длина – не менее 70% от длины референса.
Биоинформатический анализ
Последовательности генов и белков бифидобактерий, обсуждаемые в статье, были получены из базы данных NCBI (http://www.ncbi.nlm.nih.gov/). В работе использовали следующие программы: BLAST (http://blast.ncbi.nlm.nih.gov/Blast.cgi) для выявления полиморфизма аминокислотных последовательностей; Clustal Omega (http://www.ebi.ac.uk/ Tools/msa/clustalo/) для множественных выравниваний аминокислотных последовательностей; SMART (http://smart.embl-heidelberg.de/) для идентификации доменов белков FN3; Phobius (https:// phobius.sbc.su.se/) для идентификации трансмембранных доменов белка Pkb2 (ТМ-домен); trRosetta (https://yanglab.nankai.edu.cn/trRosetta/) для предсказания пространственных структур фрагментов белков [24]. В качестве референтных штаммов конкретных видов бифидобактерий для распределения по группам аминокислотных замен использовались либо типовые штаммы, либо выделенные нами лабораторные штаммы с секвенированным геномом: B. longum subsp. longum GT15 [25] и B. angulatum GT102 [26]. Позиции аминокислот и аминокислотных замен указаны по полной последовательности Pkb2.
РЕЗУЛЬТАТЫ
1. Анализ сенсорных последовательностей белков Pkb2 бифидобактерий человека
1.1. Идентификация коровых аминокислот в последовательностях SCD Pkb2 у видов бифидобактерий, выделенных из микробиоты человека. Протяженность белка Pkb2 у изучаемых видов бифидобактерий человека составляет от 376 до 492 а.о.; N-концевой каталитический домен от 314 до 348 а.о.; ТМ-домен 20–25 а.о.; SCD 120-135 а.о. (Приложение, табл. 1). Аминокислотные последовательности Pkb2 практически идентичны у штаммов внутри вида и сильно дивергированы между видами [13, 19].
Таблица 1.
Наличие аминокислотных замен в последовательности SCD Pkb2 у видов Bifidobacterium, встречающихся в микробиоте человека (по базе данных NCBI)
| Вид бифидобактерий (количество секвенированных геномов) | Группа | Количество геномов | Штамм | Pkb2, номер белкового продукта | Наличие замен аминокислот |
|---|---|---|---|---|---|
| 1. Bifidobacterium longum subsp. longum (478 геномов) | 1.1 | 353 | GT15 | WP_007057781.1 | Нет |
| 1.2 | 123 | ATCC 55813 | WP_032746814.1 | 400 T→A | |
| 1.3 | 8 | JCM 7053 | WP_101026033.1 | 363 N→T | |
| 2. Bifidobacterium longum subsp. infantis (48 геномов) | 2.1 | 29 | 157F | WP_007057781.1 | Нет |
| 2.2 | 1 | BT1 | WP_060620037.1 | 357 G→R*; 469 A→S (+) | |
| 2.3 | 1 | LH_665 | WP_174774163.1 | 376 A→V; 388 G→S* | |
| 2.4 | 1 | IN-07 | WP_065436081.1 | 461 V→A; 466 A→T*; 468 A→E* | |
| 3. B. adolescentis (102 генома) | 3.1 | 4 | ATCC 15703 | WP_011743628.1 | Нет |
| 3.2 | 20 | AM34-11 | WP_117805832.1 | 397 S→G | |
| 3.3 | 31 | Km4; 150 | WP_038444944.1 | 353 A→S (+); 397 S→G | |
| 3.4 | 8 | MSK.20.2 | WP_217717171.1 | 366 A→T; 397 S→G | |
| 3.5 | 10 | 70B | WP_085407849.1 | 366 A→T; 397 S→G; 437 S→R | |
| 4. B. angulatum (6 геномов) | 4 | 6 | GT102 | WP_052946515.1 | Нет |
| 5. B. animalis subsp. lactis (118 геномов) |
5.1 | 94 | DSM 10140 | WP_004218258.1 | Нет |
| 5.2 | 11 | ATCC 25527 | WP_014698127.1 | от 9 до 16 спорадических замен | |
| 6. B. bifidum (131 геном) | 6.1 | 28 | PRL 2010 | WP_013389588.1 | Нет |
| 6.2 | 17 | ca_0067 | WP_129901282.1 | 348 T→A | |
| 6.3 | 22 | YIT 10347 | WP_230094024.1 | 463 N→D (+) | |
| 6.4 | 11 | LMG 11583 | WP_047298710.1 | 424 G→V | |
| 6.5 | 7 | 85B | WP_047285264.1 | 406 K→R (+); 433 A→V; 463 N→D (+) | |
| 7. B. pseudocatenulatum (140 геномов) |
7.1 | 35 | JCM 1200 | WP_004221354.1 | Нет |
| 7.2 | 23 | YIT12232 | WP_226595471.1 | 399 S→G | |
| 7.3 | 7 | YIT11027 | WP_226590582.1 | 363 K→E (+) | |
| 7.4 | 9 | CA-C29 | WP_065435345.1 | 399 S→G; 443 K→D |
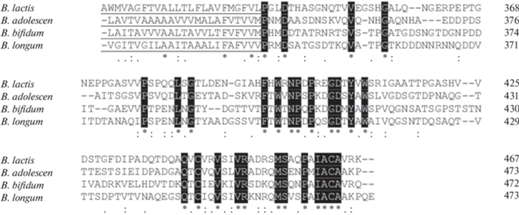
Для сравнения последовательностей ТМ и SCD Pkb2 были выбраны по одному виду бифидобактерий из каждой филогенетической группы, которые часто встречаются у человека и для которых секвенировано не менее 50 геномов: B. animalis subsp. lactis, B. adolescentis, B. bifidum, B. longum (Приложение, табл. 2). Проведенное множественное выравнивание аминокислотных последовательностей ТМ и SCD Pkb2 позволило выявить коровые аминокислоты, имеющиеся у SCD Pkb2 всех видов, встречающихся у человека (рис. 1).
Таблица 2.
Характеристика аминокислотных замен, обнаруженных в сенсорных последовательностях 2D FN3 бифидобактерий из ЖКТ человека
| Вид Bifidobacterium | Группы по амино-кислотным заменам | Название штамма | Наличие замен аминокислот | Ссылка на GenBank | |
|---|---|---|---|---|---|
| первый домен | второй домен | ||||
| B. longum subsp. longum | 1.1 | GT15 | Нет | Нет | AIW43408 |
| 1.2 | DSM 20219 | 43 A→V | SEB43038 | ||
| 1.3 | MCC10014 | 43 A→V 51 A→T |
TCD94273 | ||
| 1.4 | MCC10099 | Нет | 111 T→I | TCF41702 | |
| B. longum subsp. infantis | 2.1 | ATCC 15697 | Нет | Нет | BAJ69696 |
| 2.2 | LH_23 | 15 N→D | VWQ35166 | ||
| B. adolescentis | 3.1 | ATCC 15703 | Нет | Нет | BAF40092 |
| 3.2 | BIOML-A186 | 154 F→S | KAB5750345 | ||
| 3.3 | BIOML-A135 | 123 N→D 154 F→S |
KAB5842581 | ||
| 3.4 | BIOML-A120 | 123 N→D 150 N→D 154 F→S |
KAB5869187 | ||
| B. angulatum | 4.1 | GT102 | Нет | Нет | AMK57067 |
| 4.2 | LMG 11039 | 97 Q→H 170 S→A 181 S→G |
KFI41277 | ||
| B. dentium | 5.1 | JCM 1195 | Нет | Нет | BAQ27672 |
| 5.2 | ATCC 27679 | Нет | 132 D→G | EFM42500 | |
Рис. 1.
Выравнивание последовательностей ТМ и SCD Pkb2 между филогенетическими группами с использованием одного часто встречающегося в микробиоте человека представителя каждой группы. ТМ-домен отмечен шрифтом с подчеркиванием. Коровые аминокислоты SCD Pkb2 указаны белыми буквами на черном фоне и звездочкой под строкой. Обозначения видов: B. lactis – B. animalis subsp. lactis DSM10140; B. adolescen – B. adolescentis ATCC 15703; B. bifidum – B. bifidum PRL 2010; B. longum – B. longum subsp. longum GT15.
1.2. Полиморфизм аминокислотных последовательностей SCD Pkb2 различных видов бифидобактерий, выделенных из микробиоты человека. Анализ проводился для часто встречающихся в микробиоте человека видов и подвидов бифидобактерий: B. longum subsp. longum, B. adolescentis, B. animalis subsp. lactis, B. bifidum, B. pseudocatenulatum, и для редко встречающегося у человека вида B. angulatum, а также для B. longum subsp. infantis, который часто встречается только у младенцев [27, 28]. Учитывались только замены, часто встречающиеся в базе данных секвенированных геномов (5 и более раз). У большинства штаммов подвидов B. longum subsp. infantis и B. longum subsp. longum изучаемая последовательность Pkb2 идентична референтной последовательности штамма GT15 [25] (группы 1.1 и 2.1) (табл. 1). Большая группа штаммов B. longum subsp. longum имеют SCD Pkb2, содержащий замену 400 T→A (группа 1.2). Кроме того, у B. longum subsp. longum обнаружена небольшая группа из восьми штамов, содержащая последовательность с заменой 363 N→T (группа 1.3). Девятнадцать штаммов подвида B. longum subsp. infantis имеют спорадические замены. Три такие штамма содержат замены четырех коровых аминокислот в сочетании с заменами некоровых аминокислот (табл. 1; группы 2.2, 2.3, 2.4). У других изученных видов замены коровых аминокислот не обнаружены. Последовательности SCD Pkb2 штаммов B. adolescentis можно разделить на четыре группы: группа 3.1 – штаммы не содержат аминокислотных замен по сравнению с референтным штаммом ATCC 15703; группа 3.2 – содержат замену 397 S→G; группа 3.3 – содержат замены 353 A→S(+); 397 S→G; группа 3.4 – содержат замены 366 A→T; 397 S→G. Замена 397 S→G, таким образом, встречается у всех штаммов B. adolescentis, имеющих замены (группы 3.2, 3.3, 3.4). Последовательности SCD Pkb2 во всех секвенированных геномах B. angulatum являются идентичными (группа 4). Штаммы B. animalis subsp. lactis можно разделить на две группы: у подавляющего большинства штаммов последовательности идентичны референтной последовательности штамма DSM10140 (группа 5.1); также выделяется небольшая группа из 11 штаммов (группа 5.2), имеющая в SCD от 9 до 16 спорадических замен. Вид B. bifidum разделяется на пять групп: самая многочисленная группа не имеет замен в изучаемом домене (группа 6.1); три небольшие группы – по 10–20 штаммов с одиночными заменами (группы 6.2, 6.3, 6.4) и маленькая группа из семи штаммов, имеющая комбинацию из трех замен (группа 6.5). Штаммы B. pseudocatenulatum делятся на группу без замен (группа 7.1) и на три группы (7.2, 7.3, 7.4), имеющие одиночные или двойные замены, самая частая из которых 399 S→G.
Составлен каталог SCD Pkb2 из геномов разных видов бифидобактерий, включающий последовательности, содержащие выявленные аминокислотные замены (Приложение, табл. 3 ).
1.3. Предполагаемая пространственная структура ТМ и SCD Pkb2 у видов бифидобактерий, выделенных из микробиоты человека. Предсказанные программой trRosetta 3D структуры ТМ и SCD Pkb2 трех основных видов бифидобактерий – обитателей ЖКТ человека: B. adolescentis, B. bifidum и B. longum, оказались похожи. ТМ-домен представляет собой спиральную структуру, а SCD представляет собой комбинацию 5–6 антипараллельных β-слоев (рис. 2,а). SCD Pkb2 у редкого для человека вида B. angulatum очень короткий (Приложение, табл. 1), и его пространственная структура существенно отличается (рис. 2,а).
Рис. 2.
а – предполагаемая пространственная структура ТМ и SCD Pkb2 у видов бифидобактерий, выделенных из микробиоты человека. б – предполагаемая пространственная структура ТМ и SCD Pkb2 B. longum subsp. infantis со спорадическими аминокислотными заменами, совпадающими с коровыми аминокислотами Pkb2.
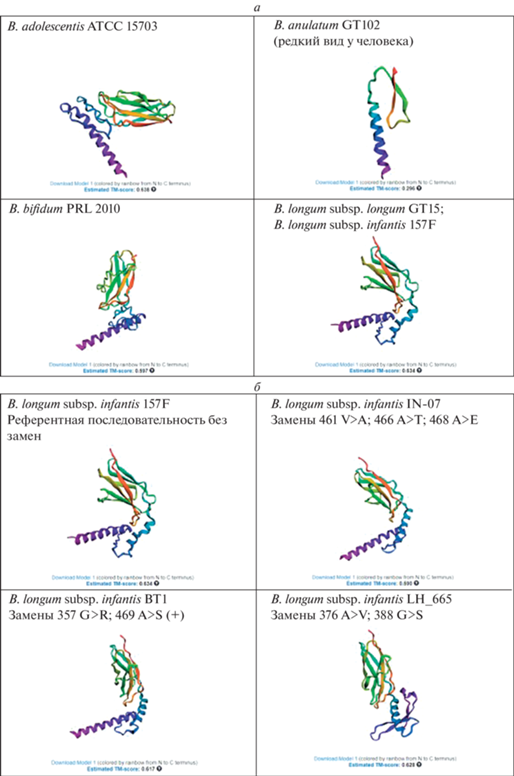
У трех штаммов у B. longum subsp. infantis обнаружены четыре замены в SCD Pkb2, совпадающие с коровыми аминокислотами Pkb2 у человеческих видов бифидобактерий (табл. 1). Замены коровых аминокислот не встречаются по одиночке, а только в комбинации с другими заменами не коровых аминокислот. Были предсказаны 3D структуры С‑концевых доменов этих трех штаммов. Предполагаемая пространственная структура существенно не изменялась (рис. 2,б). Попытка моделировать пространственную структуру SCD Pkb2 у гипотетического штамма с одиночной заменой коровой аминокислоты показала, что структура домена при этом существенно нарушается, что, предположительно, может привести к потере функции.
2. Анализ сенсорных последовательностей 2D FN3-белков PFNA-оперона бифидобактерий – обитателей ЖКТ человека
2.1. Фибронектин-содержащие домены FN3-белков бифидобактерий. FN3-белок, кодируемый геном fn3 PFNA-оперона бифидобактерий (рис. 3,а), содержит два фибронектиновых домена типа 3 (домены FN3) [19], локализованных в С-концевой области. Объектами исследования являются белки, содержащие только два фибронектиновых домена, обозначенные нами как 2D FN3. Сенсорная последовательность 2D FN3 штамма B. longum subsp. longum GT15 состоит из 184 аминокислотных остатков.
Рис. 3.
а – белок FN3 штамма B. longum subsp. longum GT15. Размер белка – 1994 а.о. Доменная структура белка: сигнальный пептид – 1–45 а.о.; трансмембранный регион – 29–51 а.о.; регион RPT1 (1) – 468–885 а.о.; регион RPT1 (2) – 951–1373 а.о.; первый домен FN3 – 1494–1581 а.о.; второй домен FN3 – 1586–1671 а.о.; С-терминальный регион – 1672–1994 а.о. Белок 2D FN3 – 1494–1671 а.о. б – гипотетическая схема механизма взаимодействия SCD Pkb2 и 2D FN3 с иммунной системой человека.

2.2. Идентификация коровых аминокислот в сенсорных последовательностях 2D FN3 у референтных штаммов бифидобактерий. Проведено выравнивание последовательностей 2D FN3 из девяти видов бифидобактерий, относящихся к трем филогенетическим группам: группа B. adolescentis (B. adolescentis, B. catenulatum subsp. catenulatum, B. catenulatum subsp. kashiwanohense, B. dentium, B. pseudocatenulatum); B. longum (B. longum, B. breve, B. angulatum) и группа B. bifidum (B. bifidum) [3]. В качестве референтных штаммов использовались: B. adolescentis ATCC 15703, B. bifidum LMG 11041, B. breve LMG 13208, B. catenulatum LMG 11043, B. catenulatum subsp. kashiwanohense DSM 21854, B. dentium JCM 1195, B. pseudocatenulatum LMG 10505, B. longum subsp. infantis ATCC 15697 [3], B. longum subsp. longum GT15 [25] и B. angulatum GT102 [26].
Выравнивание последовательностей (рис. 4) 2D FN3 показало, что последовательности первого домена имеют бóльшую гомологию, чем последовательности второго домена, выявлены коровые (консервативные) аминокислоты. В первом домене выявлена 31 коровая аминокислота, во втором домене – 10 коровых аминокислот.
Рис. 4.
Выравнивание последовательностей двух FN3-доменов бифидобактерий из ЖКТ человека. Коровые аминокислоты выделены белыми буквами на черном фоне.
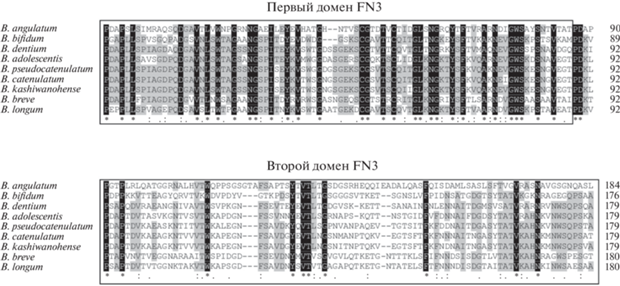
Гомология последовательностей 2D FN3 B. longum subsp. longum GT15 и B. adolescentis ATCC 15703 составляет: 62.4% идентичности, 84.5% сходства.
Последовательности 2D FN3 у B. bifidum и у B. angulatum имеют более существенные отличия от последовательностей других видов бифидобактерий из ЖКТ человека. Гомология данных последовательностей B. longum subsp. longum GT15 и B. bifidum LMG 11041 составляет: 54.6% идентичности, 77.0% сходства; гомология последовательностей 2D FN3 B. longum subsp. longum GT15 и B. angulatum GT102 составляет: 41.1% идентичности, 68.6% сходства.
Ранее нами было выявлено, что мотивы цитокиновых рецепторов были различны у трех видов бифидобактерий: у B. longum: WSХPS и WSХES, у B. bifidum: WSХPS и EG-PS, а у B. angulatum: WSХYS и SGХQA, что может быть связано с взаимодействием с различными цитокинами [19, 26]. Таким образом, полученные данные совпадают.
2.3. Распределение по группам штаммов внутри каждого из видов бифидобактерий, обитающих в ЖКТ человека. Проведен биоинформатический анализ наличия полиморфизма в сенсорных последовательностях 2D FN3 в секвенированных геномах штаммов бифидобактерий из разных коллекций (в основном зарубежных), выделенных из ЖКТ человека. По результатам данного сравнительного анализа в секвенированных геномах штаммов B. longum subsp. longum можно выделить четыре группы (табл. 2): группа 1.1 – не содержат аминокислотных замен по сравнению с референтным штаммом GT15; группа 1.2 – содержат замену 43 A→V; группа 1.3 – содержат две замены 43 A→V и 51 A→T; группа 1.4 – содержат замену 111 T→I.
Последовательности 2D FN3 штаммов B. longum subsp. infantis можно разделить на две группы: группа 2.1 – не содержат аминокислотных замен по сравнению с референтным штаммом ATCC 15697; группа 2.2 – содержат замену 15 N→D.
Последовательности 2D FN3 штаммов B. adolescentis можно разделить на четыре группы: группа 3.1 – не содержат аминокислотных замен по сравнению с референтным штаммом ATCC 15703; группа 3.2 – содержат замену 154 F→S; группа 3.3 – содержат две замены 123 N→D и 154 F→S; группа 3.4 – содержат три замены 123 N→D, 150 N→D и 154 F→S.
Последовательности 2D FN3 штаммов B. angulatum можно разделить на две группы: группа 4.1 – не содержат аминокислотных замен по сравнению с референтным штаммом GT102; группа 4.2 – содержат три замены 97 Q→H, 170 S→A и 181 S→G. Последовательности 2D FN3 штаммов B. dentium можно разделить на две группы: группа 5.1 – не содержат аминокислотных замен по сравнению с референтным штаммом JCM 1195; группа 5.2 – содержат замену 132 D→G. Замены в коровых (консервативных) аминокислотах не обнаружены.
Последовательности 2D FN3 штаммов, относящиеся к видам B. bifidum, B. breve, B. catenulatum, B. catenulatum subsp. kashiwanohense, B. gallicum и B. pseudocatenulatum, не имели аминокислотных замен по сравнению с референтными штаммами LMG 11041, LMG 13208, LMG 11043, DSM 21854, LMG 11596 и LMG 10505 соответственно.
Составлен каталог сенсорных последовательностей 2D FN3 из геномов разных видов бифидобактерий, включающий последовательности, содержащие выявленные аминокислотные замены (табл. 2, Приложение, табл. 4 ).
2.4. Предсказание структур белков 2D FN3 B. longum subsp. longum GT15, B. bifidum 791, B. angulatum GT102 и B. adolescentis ATCC 15703. С использованием Программы trRosetta были предсказаны 3D-структуры 2D FN3-белков штаммов B. longum subsp. longum GT15, B. bifidum 791, B. angulatum GT102 и B. adolescentis ATCC 15703 (рис. 5,а). Из рисунка видно, что все фрагменты белков образуют по два структурных элемента для первого и второго доменов FN3, в которых преобладают антипараллельные β-слои. Выявлены структурные отличия для 2D FN3-белков B. bifidum 791 и B. angulatum GT102 в сравнении со структурой 2D FN3-белка штамма B. longum GT15.
Рис. 5.
Предсказание структур фрагментов 2D FN3-белков у бифидобактерий. а – структуры 2D FN3-белков штаммов B. longum subsp. longum GT15, B. bifidum 791, B. angulatum GT102 и B. adolescentis ATCC 15703; б – структуры 2D FN3-белков из четырех групп штаммов B. longum subsp. longum.

Также были выявлены структурные отличия для 2D FN3-белков из четырех групп штаммов B. longum subsp. longum (рис. 5,б).
3. Поиск и идентификация в российских метагеномах микробиоты здоровых детей и взрослых последовательностей, кодирующих SCD Pkb2 и 2D FN3
Последовательности, кодирующие SCD Pkb2 и 2D FN3, были выявлены в 11 из 23 метагеномов детей и в 34 из 38 метагеномов взрослых. Результаты анализа представлены на рис. 6 (а и б) и в Приложении, табл. 5 и 6 . В изучаемых метагеномах не были обнаружены последовательности, соответствующие следующим видам бифидобактерий человека: B. angulatum, B. breve, B. catenulatum subsp. kashiwanohense, B. dentium, B. gallicum, B. longum subsp. infantis. В метагеномах детей, кроме того, отсутствовали последовательности, характерные для B. bifidum и B. pseudocatenulatum.
Рис. 6.
Результаты поиска последовательностей SCD Pkb2 (а) и 2D FN3-сенсора (б) в микробиоте кишечника здоровых детей (“CHILD_HC”) и взрослых (“ADULT_HC”). На рисунках отражены только образцы, в которых нашлась хотя бы одна исследуемая последовательность, и белки, гомологи которых были выявлены как минимум в двух образцах. Цветовой градиент отображает процент идентичности между оригинальной последовательностью из каталога и найденным гомологом.

Анализ встречаемости групп штаммов (табл. 1), несущих аминокислотные замены в SCD Pkb2, в метагеномах российских жителей (рис. 6,а, Приложение, табл. 5 ) показал, что у подвида B. longum subsp. longum группа 1.1 очень редко встречается в изучаемых метагеномах и детей, и взрослых, хотя это самая распространенная группа у этого подвида (353 штамма из 535, имеющих секвенированные геномы); группа 1.2 не встречается в изучаемых метагеномах детей и очень редкая у взрослых; очень редкая у иностранных штаммов (8 штаммов из 535) группа 1.3 оказалась самой распространенной (около 55–65%) в изучаемых метагеномах российских жителей (детей и взрослых). У вида B. adolescentis группа 3.1 не встречается в исследуемых метагеномах ни у детей, ни у взрослых; является самой редкой группой среди всех известных в мире штаммов B. adolescentis (около 4%). Группа 3.2 встречается примерно в 30% изучаемых метагеномов детей и взрослых и у 20% иностранных штаммов; группы 3.3 и 3.4 не встречаются в исследуемых метагеномах детей; группа 3.3 встречается в 20–30% изучаемых метагеномов взрослых россиян и в геномах иностранных штаммов; группа 3.4 довольно редко встречается в метагеномах взрослых и в базах данных (менее 20%); группа 3.5 встречается в изучаемых метагеномах у детей и взрослых, а также у иностранных штаммов, редко (менее 15%). Группа 5.1 B. animalis subsp. lactis обнаружена в одном метагеноме ребенка и в двух метагеномах взрослых; группа 5.2 не встречается в изучаемых метагеномах. Группы 6.1, 6.2, 6.3 вида B. bifidum редко встречаются у взрослых, а группы 6.4 и 6.5 не обнаружены. У вида B. pseudocatenulatum редко встречаются группы 7.2 и 7.3 у взрослых; группы 7.1 и 7.4 не обнаружены у взрослых.
Результат анализа встречаемости групп штаммов (табл. 2), несущих различные аминокислотные замены в сенсорном домене 2D FN3, у различных видов бифидобактерий показан на рис. 6,б и в Приложении, табл. 6 . У детей и взрослых были выявлены гены, кодирующие 2D FN3 B. adolescentis (группа 3.3, часто встречается в базе данных и в 40% изучаемых метагеномов), B. longum subsp. longum (группа 1.4, встречается в 25% метагеномов и у 15–20% иностранных штаммов), B. animalis subsp. lactis и B. catenulatum и B. pseudocatenulatum. Только у взрослых были выявлены гены, кодирующие 2D FN3 B. bifidum, B. adolescentis (группа 3.2, встречается в 25% метагеномов, часто встречается в базе данных), B. longum subsp. longum (группы 1.1 и 1.3). Группа 1.1 встречается в 15% метагеномов взрослых россиян и среди иностранных штаммов. Группа 1.3 обнаружена у 50% штаммов в базе данных и в 30% метагеномов взрослых россиян. В исследуемых метагеномах здоровых детей и взрослых отсутствуют гены, кодирующие 2D FN3 B. adolescentis (группы 3.1 и 3.4), B. longum subsp. longum (группа 1.2).
ОБСУЖДЕНИЕ
Представленная работа является частью исследований по изучению роли оперона PFNA и кодируемых им белков в процессе взаимодействия бифидобактерий с иммунной системой человека.
Анализ аминокислотных последовательностей SCD Pkb2 и 2D FN3 изучаемых видов бифидобактерий человека подтвердил ранее полученные данные [17, 18], что последовательности очень консервативны внутри вида и сильно отличаются у разных видов. Несмотря на сильную межвидовую дивергенцию, у изучаемых видов бифидобактерий человека в последовательностях SCD Pkb2 и 2D FN3 обнаружены консервативные коровые аминокислоты (рис. 1 и 4). По-видимому, коровые аминокислоты важны для образования правильной пространственной структуры доменов, что обеспечивает нормальное функционирование доменов при связывании с лигандом (SCD Pkb2) или компонентами иммунной системы человека – цитокинами (2D FN3).
Изучение полиморфизма последовательностей SCD Pkb2 и 2D FN3 показало существование часто встречающихся аминокислотных замен, позволяющих внутри каждого изучаемого вида бифидобактерий разделить штаммы на несколько групп (кластеров) (табл. 1 и 2). В случае SCD Pkb2 у подвида B. longum subsp. longum выявились существенные популяционные различия в частоте встречаемости разных групп аминокислотных замен в российских метагеномах и у штаммов иностранного происхождения (рис. 6,а). Очень редкая в российских метагеномах группа 1.1 (табл. 1) является самой распространенной группой у этого подвида (353 штамма из 535, имеющих секвенированные геномы); очень редкая у иностранных штаммов (8 штаммов из 535) группа 1.3 оказалась самой распространенной (около 55–65%) в метагеномах российских жителей (детей и взрослых). Возможно, наличие устойчивых кластеризующих замен и частоты их распределения в разных популяциях бифидобактерий связаны со специфичностью взаимодействия SCD Pkb2 с лигандом и 2D FN3 с цитокином и отражают гетерогенность популяции человека по иммунному статусу. Гипотетическая схема механизма взаимодействия SCD Pkb2 и 2D FN3 с иммунной системой человека представлена на рис. 3,б.
Предполагаемые пространственные структуры ТМ и SCD Pkb2 у трех основных видов бифидобактерий – обитателей ЖКТ человека: B. adolescentis, B. bifidum и B. longum, оказались похожи, в то время как у редкого для человека вида B. angulatum пространственная структура существенно отличается (рис. 2), что может указывать на возможность связывания с различными лигандами. Выявлены структурные отличия для 2D FN3-белков B. bifidum и B. angulatum в сравнении со структурой 2D FN3-белка B. longum (рис. 5). Прогностическое построение пространственной структуры 2D FN3 из различных видов бифидобактерий позволило обнаружить в них потенциальные цитокин-связывающие карманы. Аминокислотные замены, особенно замены коровых аминокислот, могут приводить к изменению 3D-структуры доменов, что вызовет изменение специфичности связывания с различными лигандами или изменение эффективности связывания.
Полученные научные результаты помимо фундаментальной значимости представляют большой практический интерес. Различные виды бифидобактерий потенциально могут быть источником белков, селективно связывающихся с про- и противовоспалительными цитокинами человека. Это открывает огромные возможности для создания панели препаратов белков, способных регулировать уровень цитокинов в различных органах и тканях человека, подвергшегося воспалительным процессам.
Работа выполнена в рамках Государственного задания № 0092-2022-003, тема “Механизмы генетических процессов у микроорганизмов, растений, животных и человека”; “Микробиом кишечника человека: иммуномодулирующий и антиоксидантный потенциал”.
Все процедуры, выполненные в исследовании с участием людей, соответствуют этическим стандартам институционального и/или национального комитета по исследовательской этике и Хельсинкской декларации 1964 г. и ее последующим изменениям или сопоставимым нормам этики.
От каждого из включенных в исследование участников было получено информированное добровольное согласие.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Gao B., Gupta R.S. Phylogenetic framework and molecular signatures for the main clades of the phylum Actinobacteria // Microbiol. Mol. Biol. Rev. 2012. V. 76(1). P. 66–112. https://doi.org/10.1128/MMBR.05011-11
Sun Z., Zhang W., Guo C. et al. Comparative genomic analysis of 45 type strains of the genus Bifidobacterium: A snapshot of its genetic diversity and evolution // PLoS One. 2015. V. 10(2). https://doi.org/10.1371/journal.pone.0117912
Duranti S., Longhi G., Ventura M. et al. Exploring the ecology of bifidobacteria and their genetic adaptation to the mammalian gut // Microorganisms. 2020. V. 9(1). P. 8. https://doi.org/10.3390/microorganisms9010008
Lugli G.A., Milani C., Duranti S. et al. Tracking the taxonomy of the genus Bifidobacterium based on a phylogenomic approach // Appl. Environ. Microbiol. 2018. V. 84(4). https://doi.org/10.1128/AEM.02249-17
Turroni F., Peano C., Pass D.A. et al. Diversity of bifidobacteria within the infant gut microbiota // PLoS One. 2012. V. 7(5). P. e36957. https://doi.org/10.1371/journal.pone.0036957
Turroni F., Milani C., Duranti S. et al. Glycan utilization and cross-feeding activities by bifidobacteria // Trends. Microbiol. 2018. V. 26(4). P. 339–350. https://doi.org/10.1016/j.tim.2017.10.001
Wong C., Sugahara H., Odamaki T., Xiao J. Different physiological properties of human-residential and non-human-residential bifidobacteria in human health // Benef. Microbes. 2018. V. 9(1). P. 111–122. https://doi.org/10.3920/BM2017.0031
Averina O.V., Ermolenko E.I., Yu A. et al. Influence of probiotics on cytokine production in the in vitro and in vivo systems // Med. Immunology. 2015. V. 17(5). P. 443–454. https://doi.org/10.15789/1563-0625-2015-5-443-454
Moriel D.G., Heras B., Paxman J.J. et al. Molecular and structural characterization of a novel Escherichia coli interleukin receptor mimic protein // mBio. 2016. V. 7(2). https://doi.org/10.1128/mBio.02046-15
Kumar H., Collado M.C., Wopereis H. et al. The bifidogenic effect revisited ecology and health perspectives of bifidobacterial colonization in early life // Microorganisms. 2020. V. 8(12). P. 1855. https://doi.org/10.3390/microorganisms8121855
Rabe H., Lundell A.-C., Sjöberg F. et al. Neonatal gut colonization by Bifidobacterium is associated with higher childhood cytokine responses // Gut Microbes. 2020. V. 12(1). P. 1–14. https://doi.org/10.1080/19490976.2020.1847628
Dogra S.K., Dore J., Damak S. Gut microbiota resilience: definition, link to health and strategies for intervention // Front. Microbiol. 2020. V. 11. https://doi.org/10.3389/fmicb.2020.572921
Shang J., Wan F., Zhao L. et al. Potential immunomodulatory activity of a selected strain Bifidobacterium bifidum H3-R2 as evidenced in vitro and in immunosuppressed mice // Front. Microbiol. 2020. V. 11. https://doi.org/10.3389/fmicb.2020.02089
Wang Ya., Wu Y., Wang Yu. et al. Antioxidant properties of probiotic bacteria // Nutrients. 2017. V. 9(5). P. 521. https://doi.org/10.3390/nu9050521
Högbom M., Ihalin R. Functional and structural characteristics of bacterial proteins that bind host cytokines // Virulence. 2017. V. 8. P. 1592–1601. https://doi.org/10.1080/21505594.2017.1363140
Dyakov I.N., Mavletova D.A., Chernyshova I.N. et al. FN3 protein fragment containing two type III fibronectin domains from B. longum GT15 binds to human tumor necrosis factor alpha in vitro // Anaerobe. 2020. V. 65. https://doi.org/10.1016/j.anaerobe.2020.102247
Nezametdinova V.Z., Zakharevich N.V., Alekseeva M.G. et al. Identification and characterization of the serine/threonine protein kinases in Bifidobacterium // Arch. Microbiol. 2014. V. 196. P. 125–136. https://doi.org/10.1007/s00203-013-0949-8
Nezametdinova V.Z., Mavletova D.A., Alekseeva M.G. et al. Species-specific serine-threonine protein kinase Pkb2 of Bifidobacterium longum subsp. longum: Genetic environment and substrate specificity // Anaerobe. 2018. V. 51. P. 26–35. https://doi.org/10.1016/j.anaerobe.2018.03.003
Nezametdinova V.Z., Yunes R.A., Dukhinova M.S. et al. The role of the PFNA operon of bifidobacteria in the recognition of host’s immune signals: Prospects for the use of the FN3 protein in the treatment of COVID-19 // Int. J. Mol. Sci. 2021. V. 22(17). https://doi.org/10.3390/ijms22179219
Andrews S. FastQC: A quality control tool for high throughput sequence data. 2010. Available at http://www.bioinformatics.babraham.ac.uk/projects/fastqc
Bolger A.M., Lohse M., Usadel B. Trimmomatic: A flexible trimmer for Illumina sequence data // Bioinformatics. 2014. V. 30(15). P. 2114–P.2120. https://doi.org/10.1093/bioinformatics/btu170
Nurk S., Meleshko D., Korobeynikov A., Pevzner P. AmetaSPAdes: A new versatile metagenomic assembler // Genome Res. 2017. V. 27(5). P. 824–834. https://doi.org/10.1101/gr.213959.116
Boratyn G.M., Camacho C., Cooper P.S. et al. BLAST: A more efficient report with usability improvements // Nucl. Acids Res. 2013. V. 41. https://doi.org/10.1093/nar/gkt282
Du Z., Su H., Wang W. et al. The trRosetta server for fast and accurate protein structure prediction // Nat. Protoc. 2021. V. 16(12). P. 5634–5651. https://doi.org/10.1038/s41596-021-00628-9
Zakharevich N.V., Averina O.V., Klimina K.M. et al. Complete genome sequence of Bifidobacterium longum GT15: Identification and characterization of unique and global regulatory genes // Microb. Ecol. 2015. V. 70(3). P. 819–834. https://doi.org/10.1007/s00248-015-0603-x
Zakharevich N.V., Nezametdinova V.Z., Averina O.V. et al. Complete genome sequence of Bifidobacterium angulatum GT102: Potential genes and systems of communication with host // Rus. J. Genet. 2019. V. 55(7). P. 847–864. https://doi.org/10.1134/S1022795419070160
Turroni F., van Sinderen D., Ventura M. Bifidobacteria: insights into the biology of a key microbial group of early life gut microbiota // Microorganisms. 2021. V. 7(11). P. 544. https://doi.org/10.3390/microorganisms7110544
Dogra S.K., Chung C.K., Wang D. et al. Nurturing the early life gut microbiome and immune maturation for long term health // Microorganisms. 2021. V. 9(10). P. 2110. https://doi.org/10.3390/microorganisms9102110
Дополнительные материалы
- скачать ESM.docx
- Приложения. Видоформирующий PFNA-оперон бифидобактерий


