Генетика, 2022, T. 58, № 9, стр. 1029-1041
Сравнительный геномный, транскриптомный и протеомный анализ штамма Limosilactobacillus fermentum U-21, перспективного для создания фармабиотика
Е. У. Полуэктова 1, *, Д. А. Мавлетова 1, М. В. Одорская 1, М. В. Марсова 1, К. М. Климина 1, 2, Т. А. Кошенко 1, Р. А. Юнес 1, **, В. Н. Даниленко 1
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
2 Федеральный научно-клинический центр физико-химической медицины
Федерального медико-биологического агентства России
119435 Москва, Россия
* E-mail: epolu@vigg.ru
** E-mail: romanyunes@gmail.com
Поступила в редакцию 22.03.2022
После доработки 07.04.2022
Принята к публикации 12.04.2022
- EDN: UXPQMV
- DOI: 10.31857/S0016675822090120
Аннотация
В настоящее время в мире происходят революционные изменения в области разработки и использования фармакологических препаратов на основе бактерий и их биологически активных компонентов. Наиболее перспективным становится разработка фармабиотиков – живых биотерапевтических препаратов и/или их метаболитов и компонентов с установленными фармакологическими ингредиентами, механизмом действия и направленных на лечение конкретных нозологий. При создании фармабиотиков кроме традиционных микробиологических и биотехнологических подходов используется комплекс омиксных технологий, геномных, транскриптомных и протеомных. В представленной работе данные технологии были использованы для характеристики отобранного ранее по ряду уникальных актиоксидантных свойств штамма Limosilactobacillus fermentum U-21. Геномный анализ штамма позволил выявить 29 генов, продукты которых могут проявлять антиоксидантные свойства, в том числе в отношении организма исследуемых животных. Наиболее важными могут быть гены тиоредоксинового комплекса и метаболизма и транспорта тяжелых металлов. В качестве индуктора оксидантивного стресса использована перекись водорода. 380 генов демонстрировали увеличение экспрессии и 370 генов демонстрировали снижение экспрессии более чем в 2 раза. Наибольшее увеличение экспрессии (в 14–24 раза) показали гены предполагаемого оперона карбоксилазы мочевины. Важными для последующих исследований являются изменения экспрессии генов транспорта, в том числе ионов металлов Fe2+ и Cu2+, а также синтеза и катаболизма некоторых аминокислот. Протеомный анализ экзопротеома штамма позволил идентифицировать белок шаперонного комплекса ClpB, который может играть ключевую роль в рефолдинге неправильно собранных в результате оксидативного стресса белков в различных тканях и органах организма животных. Использование комплекса омиксных технологий для характеристики терапевтических свойств и механизма действия штамма L. fermentum U-21 является одним из первых примеров в этом направлении.
В последние годы в мире происходят революционные изменения в области разработки и использования фармакологических препаратов на основе бактерий – обитателей микробиоты человека и животных – и их биологически активных ингредиентов. Такие препараты все чаще называются фармабиотиками в противовес пробиотикам, используемым в основном как биологические активные добавки и употребляемым здоровыми людьми. Фармабиотики — это живые биотерапевтические препараты и/или их метаболиты и компоненты с установленными фармакологическими ингредиентами, механизмом действия и направленные на лечение конкретных нозологий [1–5]. В связи с этим сформировался консенсус о необходимости изучения конкретных свойств штаммов и генов, определяющих проявление нужного эффекта [6–13]. Для создания фармабиотиков кроме традиционных микробиологических и биотехнологических подходов используется комплекс омиксных технологий, геномных, транскриптомных и протеомных.
Лактобациллы являются важнейшим компонентом микробиоты человека и благодаря активному синтезу биологически активных соединений и двунаправленной связи с организмом хозяина могут влиять на состояние и антиоксидантный (АО) статус организма [14–16]. АО активность отдельных штаммов лактобацилл подтверждается многими исследованиями, использующими различные модели in vitro и in vivo [11, 17, 18].
Исследуемый штамм Limosilactobacillus fermentum U-21 проявил высокую АО активность в исследованиях на моделях in vitro и in vivo с использованием индуктора окислительного стресса (ОС) параквата. Выявленная АО активность была подтверждена стандартными химическими методами. В частности, в биолюминесцентной системе E. coli K-12 было установлено, что культуральная жидкость штамма L. fermentum U-21 подавляет активность оксиданта параквата на 25% [17]. На модели свободноживущей нематоды Caenorhabditis elegans штамм L. fermentum U-21 увеличивал медианную продолжительность жизни нематоды в условиях ОС [18]. На модели индуцированного паркинсонизма у грызунов введение L. fermentum U-21 параллельно с инъекциями параквата предотвращало деградацию допаминергических нейронов мозга [18] и патологические изменения внутренних органов [19]. В совокупности полученные данные демонстрируют уникальные АО свойства штамма, позволяющие позиционировать его в качестве перспективного кандидата для разработки препаратов для лечения и профилактики различных воспалительных заболеваний, включая кардиологические, аутоиммунные и паркинсонизм [5, 11, 12, 19].
Цель данной работы – использовать комбинацию геномных, транскриптомных и протеомных технологий для выявления генов и белков, потенциально определяющих уникальные АО свойства штамма L. fermentum U-21.
МАТЕРИАЛЫ И МЕТОДЫ
Бактериальный штамм и условия роста
Бактериальные штаммы выделены из организма людей – жителей центрально-европейской части РФ. Штаммы L. fermentum U-21, L. fermentum 279 были выделены из фекалий, штамм L. fermentum 103 выделен из слепой кишки. Штамм L. fermentum U-21 депонирован в международной коллекции ВКПМ (Москва) под номером B-12075. Штаммы выращивали на среде MRS (HiMedia) при 37°С в частично анаэробных условиях (в эксикаторе, где кислород был выжжен горением свечи). Для транскриптомного анализа бактериальную культуру выращивали до экспоненциальной фазы роста (OD 600 = 0.6). Культуру центрифугировали, ресуспендировали в среде MRS (контроль) и в среде MRS с 10 мM Н2О2 (опыт), инкубировали 30 мин при 37°С, центрифугировали и ресуспендировали в том же объеме свежей среды. Отбирали по 1 мл суспензии в трех повторностях для контроля и в трех – для опыта. Жизнеспособность бактериальной культуры при такой обработке Н2О2 не изменялась. Для протеомного анализа бактериальные культуры выращивали до стационарной фазы роста (OD 600 = 2.5).
Секвенирование, сборка и аннотация геномов штаммов лактобацилл
Геномная ДНК штаммов L. fermentum была получена с помощью набора GenElute™ Bacterial Genomic DNA Kit (Merck) в соответствии с инструкцией производителя. Секвенирование геномов проводилось в компании “Евроген” на приборе Illumina MiSeq набор v2 с двух сторон, длина прочтений 2*250 нуклеотидов, покрытие более 100.
Сборка геномов de novo осуществлялась с помощью программы SPAdes-3.11.0, адаптеры были отрезаны с помощью программы Trimmomatic-0.36. Нуклеотидные последовательности ДНК штаммов были депонированы в базе данных GenBank NCBI (WGS PNBB01, PGGI01, PGGE01). Аннотация геномов осуществлялась программой NCBI Prokaryotic Genomes Automatic Annotation Pipeline (PGAAP) (http://www.ncbi.nlm.nih.gov/genomes/ static/Pipeline.html).
Геномный анализ
Биоинформатический поиск генов, проявляющих АО свойства, в геноме L. fermentum U-21 проводили с помощью созданного нами на основе литературных источников референсного каталога генов белков-антиоксидантов, встречающихся в различных видах и штаммах лактобацилл [11; https://github.com/Alexey-Kovtun/Catalog], и алгоритма их поиска [20]. Использовали также сравнительный анализ нуклеотидных последовательностей исследуемого штамма со штаммами сравнения и анализ ближайшего окружения найденных генов при помощи Sequence Set Browser. Гипотетическим опероном считали совокупность генов, располагающихся на одной цепи ДНК не далее чем в 30 пн друг от друга и изменявших экспрессию в условиях окислительного стресса в одном направлении.
Выделение РНК L. fermentum U-21
Культуру промывали RNAprotect Bacteria Reagent (Qiagen). Разрушение клеток проводили в пробирках Lysing Matrix B на приборе MagNA Lyzer (Roche) в течение 30 с. Далее РНК выделяли на автоматической станции King Fisher (Thermo Fisher Scientific) набором MagMAX™ mirVana™ Total RNA Isolation Kit (Thermo Fisher Scientific). Выделенную РНК обрабатывали набором TURBO DNA-free kit (Thermo Fisher Scientific). Дополнительную очистку РНК проводили Agencourt RNA Clean XP kit (Beckman Coulter). Количество и качество выделенной тотальной РНК оценивали на приборе Qubit набором Quant-it RiboGreen RNA assay (Thermo Fisher Scientific) и на приборе Bioanalyzer 2100 (Agilent Technologies) набором RNA 6000 Pico chip (Agilent Technologies).
Приготовление транскриптомных библиотек и секвенирование РНК
Тотальную РНК (500 нг) использовали для приготовления библиотек. Рибосомальная РНК удалялась с помощью набора Ribo-Zero Plus rRNA Depletion Kit (Illumina). Библиотеки были подготовлены при помощи набора NEBNext® Ultra II Directional RNA Library Prep Kit (NEB). Очистку РНК проводили при помощи магнитных частиц RNA Clean XP, а окончательная чистка библиотек проводилась Agencourt AMPure XP (Beckman Coulter). Размер и качество библиотек оценивали при помощи высокочувствительного ДНК-чипа (Agilent Technologies). Концентрацию библиотек измеряли набором Quant-iT DNA Assay Kit, High Sensitivity (Thermo Fisher Scientific). Библиотеки смешивали эквимолярно и разводили до конечной концентрации 12 пМ. Секвенирование готовых библиотек проводили на Illumina HiSeq 2500 с добавлением в качестве контроля 1% Phix (Illumina).
Обработка и анализ транскриптомных данных
Контроль качества необработанных чтений проводили с помощью FASTQC v0.11.5. Адаптеры обрезали с помощью программы Trimmomatic v0.33. Программное обеспечение Kallisto v0.46.0 использовалось для картирования чтений и оценки обилия транскриптов. Дифференциальный анализ экспрессии проводили с помощью пакета edgeR v3.26.8, интегрированного в веб-инструмент Degust v4.1.1. Далее анализировались только гены с количеством на миллион (CPM) ≥ 1. Гены были отфильтрованы на основе отсечения частоты ложных обнаружений (FDR) ≤ 0.05 и минимального изменения складки экспрессии (FC) ≥ 1. Выделение РНК и RNAseq были сделаны в Федеральном научно-клиническом центре физико-химической медицины Федерального медико-биологического агентства России.
Выделение белков культуральной жидкости
Бактериальные клетки отделяли от культуральной жидкости центрифугированием, затем культуральную жидкость фильтровали через PEC мембрану (0.22 мкм), добавляли PMSF до конечной концентрации 1 мМ. Белки осаждали смесью 80% ацетон, 10% ТХУ, 20 мМ ДТТ в соотношении образец : осадитель – 1 : 5 в течение 20 ч при температуре –20°С. Осадок отделяли центрифугированием и трижды промывали 90%-ным ацетоном. Для удаления остатков растворителя осадок подсушивали при комнатной температуре. Полученный осадок растворяли в буфере для нанесения SDS-ПААГ, центрифугировали при 10 000 g в течение 10 мин. Концентрацию белков, содержащихся в полученном супернатанте, определяли на флуориметре Qubit 2.0. Белки разделяли в 12.5%-ном SDS-ПААГ, с последующим окрашиванием CBB-G 250.
Масс-спектрометрический анализ
Для масс-спектрометрического анализа окрашенные белковые полосы вырезали из ПААГ. Пептиды были разделены с помощью системы Ultimate 3000 Nano LC (Thermo Fischer Scientific), связанной с Q Exactive HF – масс-спектрометром (Thermo Fischer Scientific) через источник наноэлектроспрея (Thermo Fischer Scientific) на базе группы масс-спектрометрии ИБХ им. М.М. Шемякина и Ю.А. Овчинникова РАН. Анализ данных LC-MS/MS проводился с использованием программного обеспечения PEAKS Studio 8.0 build 2016-0908. Первичные структуры пептидов, генерируемых программным обеспечением PEAKS Studio, были проанализированы на основе базы данных белковых последовательностей UNIPROT KB (07.2016). Допустимая частота ложных обнаружений была установлена на уровне 0.01 и определена путем корреляции набора данных MS/MS с обратной базой данных последовательности белка, сгенерированной студией PEAKS. Идентификацию пептидов проводили при допустимом отклонении массы исходного иона-предшественника до 10 ppm и допустимом отклонении массы фрагмента 0.05 Da.
РЕЗУЛЬТАТЫ
Идентификация и характеристика генов L. fermentum U-21, потенциально определяющих антиоксидантные свойства
В геноме штамма L. fermentum U-21 были выявлены 29 генов, определяющих белки с АО свойствами. Они перечислены в табл. S1 в Приложении. Важную роль в АО защите бактерий играют белки, содержащие тиольные группы, прежде всего глутатион и тиоредоксин. Штамм L. fermentum U-21 имеет гены, определяющие только первый из двух этапов синтеза глутатиона и, вероятно, не способен его продуцировать. В геноме отсутствуют и гены глутатионпероксидазы, глутатионредуктазы, глутатион-S-трансферазы, однако штамм способен импортировать глутатион из среды роста. Транспорт глутатиона через цитоплазматическую мембрану осуществляют белки CydD/СydC. Соответствующие гены входят в состав оперона, включающего также гены субъединиц I и II цитохром-убихинол оксидазы, осуществляющей векторный перенос протонов через мембрану и являющейся компонентом аэробной дыхательной цепи.
Наиболее важную роль в защите от окислительного стресса играют белки тиоредоксиновой системы. Тиоредоксин может восстанавливать дисульфидные связи других белков и участвует в обезвреживании активных форм кислорода, передавая электроны различным пероксидазам. Восстановление тиоредоксина осуществляет тиоредоксинредуктаза [21]. В геноме штамма были идентифицированы четыре гена тиоредоксина (из них три определяют белки с каноническим для тиоредоксина активным сайтом WCGDC), расположенных в разных контигах; тиоредоксинредуктаза, а также глутаредоксин, имеющий сходную с тиоредоксином активность. Штамм имеет два гена тиолпероксидаз, а также еще одну тиолпероксидазу – пероксиредоксин (субъединицу С щелочной гидропероксидредуктазы); его восстановление осуществляет субъединица F щелочной гидропероксидредуктазы. Штамм содержит также гены пероксиредоксина семейства OsmC, обладающего пероксидазной активностью по отношению к органическим гидропероксидам.
Другой важный механизм АО защиты у лактобацилл – хелатирование ионов металлов, Fe2+ и Cu2+. Ионы металлов способны инициировать образование реактивных форм кислорода и запускать перекисное окисление липидов. В геноме штамма идентифицированы два гена, кодирующих белки транспорта двухвалентного железа; выведение ионов железа из клетки снижает вероятность реакции Фентона и образования реактивных форм кислорода. Dps-белок катализирует окисление ионов Fe2+ перекисью водорода. В геноме также идентифицирован ряд генов транспорта меди. Соответствующие опероны изображены на рис. 1,а–в. Это гены белков-переносчиков тяжелых металлов – АТФаз Р-типа, медь-транслоцирующих АТФаз Р-типа, белков, содержащих купредоксиновый домен, репрессоров транспорта меди. Идентифицирован также отдельный ген медь-транслоцирующей АТФазы Р-типа (табл. S1 Приложения). Известные модели описывают ферменты-переносчики тяжелых металлов АТФазы Р-типа – как ответственные за перенос ионов меди и кадмия (и других двухвалентных тяжелых металлов, таких как кобальт, ртуть, свинец и цинк) через биологические мембраны. Эти переносчики содержатся в прокариотах и растениях и экспериментально охарактеризованы. Кластеры данных генов характерны для лактобацилл и встречаются у многих представителей семейства. Белки с купредоксиновым доменом относятся к семейству маленьких белков, способных связывать один атом меди и активно влиять на проявление АО свойств. Купредоксины играют ключевую роль в биологическом переносе электронов и способны реагировать с широким спектром окислительно-восстановительных партнеров. Например, купредоксина зурин Pseudomonas aeruginosa замедляет одноэлектронный перенос между ферментами, связанными с цепью цитохрома, является многоцелевым противоопухолевым агентом, влияющим на сигнальный путь р53 и сигнальный путь нерецепторных тирозинкиназ [22].
Рис. 1.
Предполагаемые (гипотетические) опероны, содержащие гены белков связывания и транспорта тяжелых металлов в геноме штамма L. fermentum U-21. а – схема оперона из контига PNBB01000020.1, содержащего гены белков транслоцирующей тяжелые металлы АТФазы Р-типа (C0965_05525), транспортера катионов (C0965_05530), ДНК-связывающего белка (C0965_05535), транскрипционного регулятора Crp/Fnr (C0965_05540); б – схема оперона из контига PNBB01000024.1, содержащего гены белков с купредоксиновым доменом (C0965_06085, C0965_06090) и медь-транспортирующую ATФазу P-типа (C0965_06095); в – схема оперона из контига PNBB01000074.1, содержащего гены белков транслоцирующей тяжелые металлы АТФазы Р-типа (C0965_09625) и белка, содержащего ассоциированный с тяжелыми металлами домен (C0965_09630); г – схема оперона, уникального для штамма L. fermentum U-21, включающего гены гем-зависимой пероксидазы (C0965_09505), MFS-транспортера (C0965_09510) и феррохелатазы (C0965_09515).
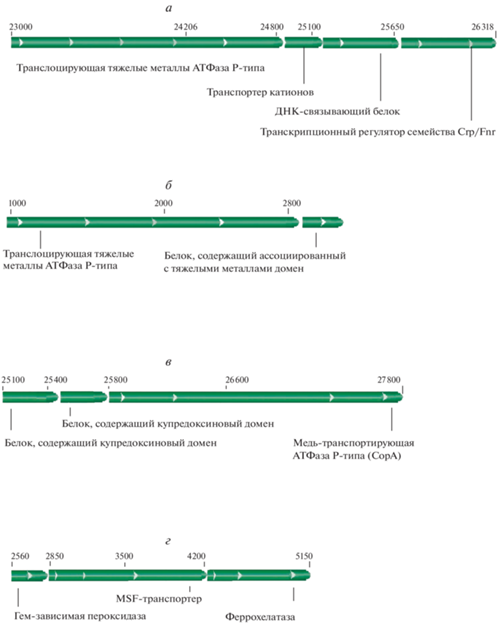
Сравнение штамма L. fermentum U-21 со штаммами L. fermentum 279 и L. fermentum 103, не проявляющими антиоксидантных свойств, выявило 138 уникальных генов. Среди них семь генов были связаны с АО активностью и метаболизмом ионов тяжелых металлов (табл. 1). Особенно стоит отметить оперон, расположенный в контиге PNBB01000070.1 и состоящий из генов, кодирующих MFS-транспортер, феррохелатазу и гем-зависимую пероксидазу (рис. 1,г). MFS-транспортеры – мембранные транспортные белки, осуществляющие перемещение небольших растворенных соединений через клеточные мембраны; феррохелатаза – конечный фермент синтеза гема, катализирующий включение двухвалентного железа. Данный кластер является крайне редким у L. fermentum и обнаруживается только в семи геномах штаммов этого вида, депонированных в GenBank.
Таблица 1.
Белки, связанные с метаболизмом тяжелых металлов и обнаруженные в геноме штамма L. fermentum U-21 и отсутствующие в геномах штаммов L. fermentum 103 и L. fermentum 279
| Контиг | Идентификатор гена | Идентификатор белка | Название фермента | Предполагаемая функция фермента |
|---|---|---|---|---|
| PNBB01000011.1 | C0965_03855 | WP_048339862.1 | Оксидаза, содержащая три купредоксиновых домена | Связывание и транспорт ионов меди/железа |
| PNBB01000040.1 | C0965_07955 | WP_056959392.1 | Оксидаза, содержащая три купредоксиновых домена | Связывание и транспорт ионов меди/железа |
| PNBB01000040.1 | C0965_08000 | WP_102116156.1 | Белок, определяющий устойчивость к кадмию | Экспорт или связывание ионов кадмия |
| PNBB01000060.1 | C0965_09170 | WP_035437569.1 | Репрессор переноса меди семейства CopY/TcrY | Взаимодействие с ДНК, регуляция экспрессии генов |
| PNBB01000070.1 | C0965_09505 (фрагмент гена) |
WP_003680658.1 | Гем-зависимая пероксидаза | Расщепление перекиси |
| PNBB01000070.1 | C0965_09510 | WP_035435948.1 | MFS-транспортер | Мембранный транспортер |
| PNBB01000070.1 | C0965_09515 | WP_021349248.1 | Феррохелатаза | Конечный фермент синтеза гема |
Анализ уникальных и редких генов у L. fermentum U-21 по сравнению со 107 штаммами L. fermentum, депонированными в базе данных NCBI на 28.02.2022, выявил 15 генов (табл. S2 Приложения). Наибольшее число подобных генов локализуется в контигах PNBB01000040.1 и PNBB01000051.1 (по 6 генов). Первая группа содержит гены транскрипционного регулятора, белка, определяющего резистентность к кадмию, токсина YafQ семейства токсин–антитоксинов II типа, резольвазы и ряда гипотетических белков. Вторая группа генов является исключительно уникальной для штамма L. fermentum U-21. Среди белков, кодируемых генами этой группы, есть три гликозилтрансферазы, серинацетилтрансфераза, белок метаболизма фосфорилхолина.
Полученные данные являются первым этапом в идентификации генов, определяющих свойства штамма L. fermentum U-21. Последующие исследования транскриптома и протеома, проводимые в лаборатории генетики микроорганизмов ИОГен РАН, позволят получить больше информации о генах и их продуктах, определяющих свойства штамма L. fermentum U-21.
Транскриптомный анализ
Нас интересовало, какие гены/белки штамма L. fermentum U-21, кроме перечисленных в предыдущем разделе, определяют ответ штамма на окислительный стресс. В качестве индуктора стресса была выбрана перекись водорода. С ней бактериальные клетки сталкиваются в процессе своей жизнедеятельности, так как она наряду с другими реактивными формами кислорода продуцируется клетками иммунной системы макроорганизма при инфекции и воспалении [23]. Использовали концентрацию перекиси, не влияющую на жизнеспособность клеток штамма в данный отрезок времени (10 мM, 30 мин).
Экспрессия генов после действия перекиси резко изменилась, 380 генов демонстрировали увеличение экспрессии в 2 и более раз, 370 демонстрировали уменьшение в 2 и более раз (табл. 2), эти гены составляли 42.56% всех исследованных генов. Численность генов с увеличенной и уменьшенной экспрессией была приблизительно одинакова, однако максимальные значения дифференциальной экспрессии генов (ДЭГ) были больше для уменьшенной экспрессии (70–28 раз), чем для увеличенной экспрессии (24–22 раза).
Таблица 2.
Численность генов, изменивших экспрессию под действием перекиси водорода в 2 и более раз
| Изменение экспрессии генов в N раз | Число генов
с увеличенной экспрессией + |
Число генов
с уменьшенной экспрессией – |
|---|---|---|
| 70 | – | 1 |
| 37 | – | 1 |
| 32 | – | 1 |
| 30 | – | 1 |
| 28 | – | 1 |
| 24 | 1 | – |
| 23 | – | 2 |
| 22 | 1 | – |
| 19–11 | 12 | 12 |
| 10–8 | 16 | 13 |
| 7–4 | 91 | 59 |
| 3 | 81 | 79 |
| 2 | 178 | 200 |
| Всего | 380 | 370 |
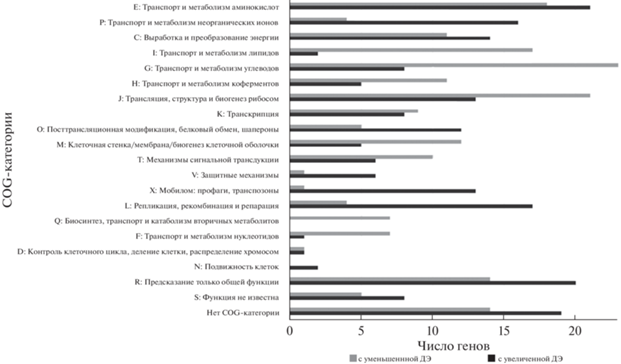
Далее ДЭГ были проанализированы по функциональной активности соответствующих им белков (COG). На рис. 2 представлено распределение по COG-категориям белков, соответствующих генам с измененной в более чем 2 раза экспрессией. Из 26 стандартных COG-категорий (https://www.ncbi.nlm.nih.gov/research/cog/) рассматриваемые белки распределились в 20 категорий. Белки, соответствующие генам с увеличенной и уменьшенной экспрессией, входили примерно в одинаковом соотношении в категории E (транспорт и метаболизм аминокислот), K (транскрипция) и C (выработка и преобразование энергии). Для большей части категорий были различия для генов с увеличенной и уменьшенной экспрессией. Белки, соответствующие генам только или преимущественно с увеличенной экспрессией, были представлены в категориях V (защитные механизмы), X (мобилом), P (транспорт и метаболизм неорганических ионов), L (репликация, рекомбинация, репарация), O (посттрансляционная модификация, шапероны), N (подвижность клеток). Категории F (транспорт и метаболизм нуклеотидов), Q (синтез, транспорт и метаболизм вторичных метаболитов), I (транспорт и метаболизм липидов), G (транспорт и метаболизм углеводов), H (транспорт и метаболизм коферментов), T (механизмы сигнальной трансдукции), M (биогенез клеточной стенки и мембран) включали белки, соответствующие генам только или преимущественно с уменьшенной экспрессией.
Рис. 2.
Распределение белков, соответствующих генам со значительно измененной экспрессией (больше чем в 2 и меньше чем в 2 раза по сравнению с контролем), по функциональным категориям Clusters of Orthologous Groups (COG).
Однако функциональная характеристика белков, соответствующих генам с ДЭ, не дает полного представления о процессах, происходящих в бактериальной клетке при окислительном стрессе. Так, гены, C0965_01070 и C0965_08155, аннотированные как кодирующие биотин-карбоксильный белок-носитель ацетил-КоА-карбоксилазы, изменяли экспрессию в –7 и +16 раз соответственно. По-разному реагировали на стресс увеличением или уменьшением экспрессии гены пероксиредоксина OsmC-семейства (C0965_08900 и C0965_05480) и репрессора транспорта меди CopY/TcrY (C0965_09170 и C0965_08735). Вероятно, более точное представление можно получить при анализе не отдельных генов, а оперонов. Характеристика оперонов и отдельных генов, не входящих в опероны, демонстрирующих наибольшее отклонение экспрессии от контроля, приведены в табл. S3 Приложения.
Наибольшее увеличение экспрессии (в 24–14 раз) демонстрировали гены C0965_08140–C0965_08165, предположительно составляющие один оперон карбоксилазы мочевины (UCA). Комплекс 5.5 тпн шести генов L. fermentum U-21 является видоспецифичным для L. fermentum, он кодирует UCA-аллофанатгидролазный путь деградации мочевины. Уреазная активность штамма Limosilactobacillus reuteri 100-23 в желудке мышей повышала кислотоустойчивость штамма [24]. UCA может использоваться бактериями для ассимиляции источников азота [25]. Значительно увеличивалась экспрессия оксидоредуктаз, ферментов биосинтеза пролина, катаболизма дисахаридов, белков транспорта витаминов.
Перекись водорода повреждает различные клеточные структуры – ДНК, белки, клеточные мембраны. Экспрессия генов репарационных белков – метионинсульфоксидредуктаз, mrsA и mrsB, C0965_06645 и C0965_01540 также возрастала в 7–8 раз. Эти ферменты обеспечивают дисульфидный путь восстановления поврежденных в результате окисления остатков метионинсульфоксида в белках до метионина [26]. Увеличивается экспрессия генов полифосфаткиназы (C0965_06785), осуществляющей фосфорилирование нуклеозидов и обеспечивающей устойчивость к стрессу [27]. Увеличена экспрессия гена рекомбиназы recA.
Максимальное уменьшение экспрессии (в 18–70 раз) демонстрировали опероны и отдельные гены синтеза компонентов ABC-транспортеров, аргинина и орнитина, фосфотрансферазных систем транспорта сахаров, синтеза жирных кислот.
Подавляющее большинство генов, определяющих белки с АО, изменяли экспрессию при действии перекиси водорода, увеличивая ее в 2–4 раза. Увеличение экспрессии отмечено для генов, кодирующих субъединицы алкилгидропероксидредуктазы – F и C (пероксиредоксин), тиоредоксин (4 гена), медь-транслоцирующие АТФазы (2 гена), пероксидазу, глутаредоксин, феррохелатазу. Наибольшее увеличение экспрессии генов этой группы отмечено для оперона, содержащего гены, кодирующие тиоредоксинредуктазу и цистеинсинтазу (табл. S3 Приложения) (в 8–10 раз), и оперона, содержащего гены переносчика тяжелых металлов АТФазы Р-типа и белка, содержащего ассоциированный с тяжелыми металлами домен (рис. 1,в) (в 3.9–5.6 раз). Уменьшение экспрессии отмечено только для нескольких генов этой группы (менее чем в 2 раза). Более значительное уменьшение экспрессии (в 3–4 раза) отмечено только для генов, кодирующих глутаматцистеинлигазу, катализирующую первый этап синтеза глутатиона (табл. S1 Приложения).
Увеличивалась также экспрессия генов, определяющих стрессовые белки, – небольшого белка теплового шока Lo18, свойственного лактобациллам; АТФ-зависимой протеолитической субъединицы ClpP протеазы Clp; шаперонов GroEL и DnaJ; мембранных белков щелочного стресса. Увеличивалась экспрессия генов системы токсин–антитоксин YafQ/RelB и резольваз IS-элементов.
Протеомный анализ
Задачей данной части работы было выявление потенциальных белков, определяющих пробиотические и антиоксидантные свойства штамма L. fermentum U-21. Наиболее важные для взаимодействия с макроорганизмом белки бактерий находятся в культуральной жидкости. Методом масс-спектрометрии были идентифицированы основные белки, присутствующие в супернатанте стационарной культуры штамма L. fermentum U-21. Полученные результаты представлены в табл. 3 и на рис. 3,а. Выявленные белки относятся к различным функциональным группам. Наиболее многочисленной среди идентифицированных белков является группа гомологов ферментов, участвующих в метаболизме углеводов, – глицеральдегид-3-фосфатдегидрогеназа, фосфокетолаза, фосфоглюконатдегидрогеназа, глюкоза-6-фосфатизомераза. Были идентифицированы два белка, обладающих шаперонной активностью, – шаперонин GroEL и ClpB, а также два фермента, участвующих в метаболизме ацетил-КоА – альфа-субъединица цитратлиазы и бета-субъединица цитратлиазы (pro-3S). Следует отметить, что гомологи идентифицированных белков были обнаружены при изучении экзопротеомов других видов лактобацилл [28, 29]. Кроме того, большинство этих белков относят к мунлайт-белкам, т.е. многофункциональным белкам, в которых одна полипептидная цепь выполняет две или более физиологически значимые биохимические или биофизические функции, в частности цитоплазматические белки выполняют вторую функцию на клеточной поверхности, часто в качестве адгезинов [30].
Таблица 3.
Белки, идентифицированные в экзопротеоме штамма L. fermentum U-21
| № | Идентификатор гена | Идентификатор белка | Название белка |
|---|---|---|---|
| 1 | C0965_08340 | PLY17634.1 | Белок семейства фосфокетолаз |
| 2 | C0965_04780 | PLY18262.1 | ClpB, АТФ-связывающая субъединица Clp протеазы |
| 3 | C0965_01125 | PLY18973.1 | Шаперонин GroEL |
| 4 | C0965_07280 | PLY17803.1 | Альфа-субъединица цитратлиазы |
| 5 | C0965_07940 | PLY17705.1 | НАДФ-зависимая фосфоглюконатдегидрогеназа |
| 6 | C0965_10225 | PLY17327.1 | Глюкозо-6-фосфатизомераза |
| 7 | C0965_04525 | PLY18352.1 | Глицеральдегид-3-фосфатдегидрогеназа |
| 8 | C0965_07285 | PLY17804.1 | Бета-субъединица (про-3S)-цитратлиазы |
| 9 | C0965_04410 | PLY18330.1 | LysM белок, содержащий пептидогликан-связывающий домен |
Рис. 3.
Электрофореграмма белков культуральной жидкости штаммов L. fermentum. а – белки штамма L. fermentum U-21. Указаны молекулярные массы стандартных маркерных белков. Цифрами справа (1–9) отмечены идентифицированные методом масс-спектрометрии белки, перечисленные в табл. 3; б – белки различных штаммов L. fermentum: 1 – L. fermentum U-21, 2 – L. fermentum 103, 3 – L. fermentum 279. Стрелками слева отмечены белки ClpB и LysM.

Далее был проведен сравнительный анализ белков культуральной жидкости штамма L. fermentum U-21 и двух других штаммов того же вида, не проявляющих антиоксидантных свойств, – L. fermentum 103 и L. fermentum 279. Состав белковых компонентов данной фракции у штаммов обнаружил качественные и количественные различия (рис. 3,б). Наиболее существенные различия в белках экзопротеома штаммов были связаны с двумя белками – белком, содержащим LysM-домен связывания с пептидогликаном, и ClpB-белком. LysM-белок есть и у двух других исследуемых штаммов L. fermentum, однако у штаммов L. fermentum 103 и L. fermentum 279 он имеет меньшую молекулярную массу. Различия связаны со вставкой короткого полипептида из восьми аминокислот у штамма L. fermentum U-21. Гены, соотвествующие белку ClpB, также есть в геномах всех трех исследуемых штаммов L. fermentum. Однако в культуральной жидкости этот белок обнаруживается только у штамма L. fermentum U-21.
ОБСУЖДЕНИЕ
Анализ генома L. fermentum U-21 выявил 29 генов, являющихся классическими примерами антиоксидантных генов бактерий. Многие из генов были объединены в опероны. Вероятно, основной защитой от окислительного стресса является тиоредоксиновая система, функционирующая посредством регуляции дитиол/дисульфидного баланса. Способность штамма L. fermentum U-21 нейтрализовать супероксид анион в биолюминесцентной тест-системе E. coli по-видимому определяется, во всяком случае отчасти, тиоредоксиновой системой. Хотя эта система широко распространена у лактобацилл, ее эффект может варьировать за счет полиморфизма генов, кодирующих белки, входящие в ее состав, и за счет изменения регуляции активности генов. Экспрессия большинства генов, составляющих тиоредоксиновую систему у L. fermentum U-21, была увеличена в результате экспозиции к перекиси водорода (см. табл. S1 ). Также были выявлены четыре группы генов, связанные с метаболизмом тяжелых металлов и потенциально образующие опероны (см. табл. S1, S2 ). Наличие значительного количества генов транспорта ионов меди, а также транспорта двухвалентного железа, гена dps ДНК-защищающего белка и феррохелатазы свидетельствует о том, что значительный вклад в антиоксидантные свойства штамма вносит процесс хелатирования металлов. Стоит отметить, что составляющие оперон гены гем-зависимой пероксидазы, MFS-транспортера и феррохелатазы отсутствуют в геноме двух штаммов сравнения L. fermentum 103 и L. fermentum 279, не проявлявших сопоставимый уровень антиоксидантной активности. Экспрессия всех трех генов увеличилась в 2 раза в реакции на перекись водорода, что указывает на вероятность связи между активностью этих генов и проявленными антиоксидантными свойствами штамма L. fermentum U-21.
Получасовое действие перекиси водорода резко изменило экспрессию почти половины генов штамма L. fermentum U-21. Гены, вовлеченные в инактивацию перекиси и других реактивных форм кислорода, увеличивали экспрессию, но незначительно. Незначительно активировались и многие шаперонные системы. Наибольшее увеличение экспрессии отмечалось у генов, кодирующих белки, участвующие в продукции аммония как источника азота, катаболизме дисахаридов, кодирующих различные оксидоредуктазы, стрессовые белки, общие для разных видов стресса, а также ферменты, защищающие белки и нуклеиновые кислоты от окисления. Увеличивалась экспрессия генов, кодирующих элементы мобилома, – IS-элементы, системы токсин–антитоксин II типа. Резко уменьшалась экспрессия генов, кодирующих субъединицы АТФ-связывающих кассетных транспортеров (ABC-транспортеров), фосфотрансферазной системы транспорта сахаров, биосинтеза жирных кислот.
Результаты окислительного стресса, вызванного перекисью водорода, зависят от многих факторов, от рода, вида и штамма бактерий, концентрации окислителя, времени его действия, присутствия кислорода в среде роста бактерий, метода исследования [31–33]. Поэтому сравнение с другими немногочисленными работами по транскриптомному анализу результатов окислительного стресса, вызванного перекисью водорода у лактобацилл и близких родов бактерий [33, 34], дает самые общие заключения. Отмечается активация систем защиты белков и ДНК от окисления, изменение активности генов синтеза и транспорта аминокислот и сахаров, индукция стрессовых белков, общих для разных видов стресса. Во всех работах отмечается увеличение экспрессии генов тиоредоксинредуктазы, белков синтеза цистеина и метионинсульфоксидредуктазы – ферментов, обеспечивающих стабильность белков путем дисульфидной редукции. Протеомный анализ Lactobacillus acidophilus NCFM также показал увеличение в количестве белков, связанных с метаболизмом углеводов и энергии, синтезом цистеина и стрессом [35]. У L. rhamnosus hsryfm 1301 транскриптомный RNAseq анализ выявил перекрестную адаптацию при температурном стрессе и при действии перекиси водорода. 154 гена изменяли свою активность в условиях обоих стрессов. Эти гены кодировали белки-транспортеры аминокислот и олигопептидов, белки метаболизма аминокислот и quorum sensing [36].
Результаты транскриптомного анализа штамма лактобацилл в условиях окислительного стресса позволяют значительно расширить круг ферментов бактерий, вовлеченных в ответ на стресс. Эти данные могут быть использованы для отбора пробиотических штаммов с антиоксидантной активностью, а также для разработки дополнительных компонентов в пробиотических препаратах, направленных на лечение заболеваний, связанных с окислительным стрессом, прежде всего воспалительных заболеваний.
Поиск белков, которые могут определять пробиотические и антиоксидантные свойства штамма L. fermentum U-21, выявил два белка, LysM и ClpB. LysM-домен обнаруживается во многих внеклеточных белках бактерий. Считается, что он обеспечивает прикрепление к полисахаридам клеточной стенки бактерий, таким как пептидогликан [37]. Пептид в составе белка LysM у L. fermentum U-21, отсутствующий у двух других штаммов L. fermentum, имеет состав Thr-Ala-Ala-Thr-Thr-Thr-Ser; если учесть еще и окружающие пептид остатки серина, то он будет выглядеть так: Ser-Thr-Ala-Ala-Thr-Thr-Thr-Ser-Ser-Ser. У штамма Lactiplantibacillus plantarum BMCM12 серин-треониновый пептид STp проявляет иммуномодулирующую активность на дендритных клетках кишечника, он является фрагментом секретируемого белка и образуется при действии кишечных протеаз [38]. Серин-треониновые белки других микроорганизмов способны связываться с компонентами эукариотических клеток [39]. Мы полагаем, что данный белок и входящий в его состав пептид могут осуществлять взаимодействие с клетками макроорганизма и определять пробиотические свойства штамма.
Бактериальный белок ClpB – гомолог казеинолитической пептидазы B – играет определяющую роль при выживании бактерий в условиях различных форм стресса. Белок ClpB также регулирует секрецию бактериальных эффекторных молекул VI системы секреции. Белок принадлежит к суперсемейству ААА+ белков (АТФазы, ассоциированные с различными клеточными активностями), является представителем семейства шаперонов Hsp100. Совместно с системой шаперонов DnaK/DnaJ/GrpE солюбилизирует и реактивирует агрегированные белки. В присутствии АТФ ClpB, содержащий два нуклеотидсвязывающих домена, подвергается олигомеризации, образуя гомогексамерную кольцевую структуру, через центральную пору которой транслоцируются полипептиды, которые затем могут быть подвергнуты рефолдингу посредством шаперонной системы HSP70 [40, 41]. Наличие белка ClpB в культуральной жидкости штамма L. fermentum U-21 может способствовать рефолдингу поврежденных белков и определять антиоксидативные свойства штамма. У белка ClpB E. coli было установлено еще одно необычное свойство – он является миметиком ключевого анорексигенного гормона меланотропина человека; это позволило понять механизм взаимодействия бактерий с системой передачи сигналов насыщения в кишечнике. Полагают, что этот бактериальный белок участвует в индукции чувства насыщения у человека [42]. Белок ClpB может играть ключевую роль в рефолдинге неправильно собранных в результате ОС белков в различных тканях и органах макроорганизма.
Использование комплекса омиксных технологий для продвижения в качестве фармабиотика штамма L. fermentum U-21 является одним из первых примеров в этом направлении. Полученные результаты требуют детального анализа и осмысления для выбора ключевых элементов, которые предстоит использовать при создании фармабиотиков с заданными свойствами, противовоспалительными, иммуномодулирующими и нейромодулирующими. Включение метаболомного анализа в тетраду омиксных технологий является необходимым условием.
Работа выполнена при финансовой поддержке Государственного задания № 0092-2022-003.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Sleator R.D., Hill C. Engineered pharmabiotics with improved therapeutic potential // Human Vaccines. 2008. V. 4. № 4. P. 271–274. https://doi.org/10.4161/hv.4.4.6315
O’Toole P.W., Marchesi J.R., Hill C. Next-generation probiotics: the spectrum from probiotics to live biotherapeutics // Nature Microbiol. 2017. V. 2. № 5. 17057. https://doi.org/10.1038/Nmicrobiol.2017.57
Patterson E., Cryan J.F., Fitzgerald G.F. et al. Gut microbiota, the pharmabiotics they produce and host health // Proc. Nutr. Soc. 2014. V. 73. № 4. P. 477–489. https://doi.org/10.1017/S0029665114001426
Cordaillat-Simmons M., Rouanet A., Pot B. Live biotherapeutic products: the importance of a defined regulatory framework // Exp. Mol. Med. 2020. V. 52. № 9. P. 1397–1406. https://doi.org/10.1038/s12276-020-0437-6
Danilenko V.N., Devyatkin A.V., Marsova M.V. et al. Common inflammatory mechanisms in COVID-19 and Parkinson’s diseases: the role of microbiome and probiotics in their prevention // J. Inflamm. Res. 2021. V. 14. P. 6349–6381. https://doi.org/10.2147/JIR.S333887
Shanahan F., Collins S.M. Pharmabiotic manipulation of the microbiota in gastrointestinal disorders, from rationale to reality // Gastroenterol. Clin. North. Am. 2010. V. 39. № 3. P. 721–726. https://doi.org/10.1016/ j.gtc.2010.08.006
Gazerani P. Probiotics for Parkinson’s disease // Intern. J. Mol. Sci. 2019. V. 20. № 17. 4121. https://doi.org/10.3390/ijms20174121
De Luca F., Shoenfeld Y. The microbiome in autoimmune diseases // Clin. Exp. Immunol. 2019. V. 195. № 1. P. 74–85. https://doi.org/10.1111/cei.13158
Yunes R.A., Poluektova E.U., Vasileva E.V. et al. A multi-strain potential probiotic formulation of GABA-producing Lactobacillus plantarum 90sk and Bifidobacterium adolescentis 150 with antidepressant effects // Probiotics Antimicrob. Proteins. 2020. V. 12. № 3. P. 973–979. https://doi.org/10.1007/s12602-019-09601-1
Maria Remes Troche J., Coss Adame E., Angel Valdovinos Diaz M. et al. Lactobacillus acidophilus LB: A useful pharmabiotic for the treatment of digestive disorders // Therap. Adv. Gastroenterol. 2020. V. 13. 1756284820971201. https://doi.org/10.1177/1756284820971201
Averina O.V., Poluektova E.U., Marsova M.V., Danilenko V.N. Biomarkers and utility of the antioxidant potential of probiotic lactobacilli and bifidobacteria as representatives of the human gut microbiota // Biomedicines. 2021. V. 9. № 10. 1340. https://doi.org/10.3390/biomedicines9101340
Poluektova E., Yunes R., Danilenko V. The putative antidepressant mechanisms of probiotic bacteria: relevant genes and proteins // Nutrients. 2021. V. 13. № 5. 1591. https://doi.org/10.3390/nu13051591
Tan A.H., Lim S.-Y., Chong K.K. et al. Probiotics for constipation in Parkinson disease: A randomized placebo-controlled study // Neurology. 2021. V. 96. № 5. e772–e782. https://doi.org/10.1212/WNL.0000000000010998
Mishra V., Shah C., Mokashe N. et al. Probiotics as potential antioxidants: a systematic review // J. Agric. Food Chem. 2015. V. 63. № 14. P. 3615–3626. https://doi.org/10.1021/jf506326t
Nowak A., Paliwoda A., Błasiak J. Anti-proliferative, pro-apoptotic and anti-oxidative activity of Lactobacillus and Bifidobacterium strains: A review of mechanisms and therapeutic perspectives // Crit. Rev. Food Sci. Nutr. 2019. V. 59. № 21. P. 3456–3467. https://doi.org/10.1080/10408398.2018.1494539
Wang Y., Wu Y., Wang Y. et al. Antioxidant properties of probiotic bacteria // Nutrients. 2017. V. 9. № 5. P. 521. https://doi.org/10.3390/nu9050521
Marsova M.V., Abilev S.K., Poluektova E.U., Danilenko V.N. A bioluminescent test system reveals valuable antioxidant properties of Lactobacillus strains from human microbiota // World J. Microbiol. Biotechnol. 2018. V. 34. № 2. 27. https://doi.org/10.1007/s11274-018-2410-2
Marsova M., Poluektova E., Odorskaya M. et al. Protective effects of Lactobacillus fermentum U-21 against paraquat-induced oxidative stress in Caenorhabditis elegans and mouse models // World J. Microbiol. Biotechnol. 2020. V. 36. № 7. 104. https://doi.org/10.1007/s11274-020-02879-2
Даниленко В.Н., Ставровская А.В., Воронков Д.Н. и др. Использование фармабиотика на основе штамма Lactobacillus fermentum U-21 с целью модуляции нейродегенеративного процесса при экспериментальном паркинсонизме // Анналы клинической и экспериментальной неврологии. 2020. Т. 14. № 1. С. 62–69. https://doi.org/10.25692/ACEN.2020.1.7
Ковтун А.С., Аверина О.В., Захаревич Н.В. и др. In silico определение метагеномной сигнатуры, отражающей нейрометаболический потенциал микробиоты кишечника человека в норме // Генетика. 2018. Т. 54. № 9. С. 1081–1091. https://doi.org/0.1134/S0016675818090084
Lu J., Holmgren A. The thioredoxin antioxidant system. Review // Free Radic. Biol. Med. 2014. V. 66. № 8. P. 75–87. https://doi.org/10.1016/j.freeradbiomed.2013.07.036
Gao M., Zhou J., Su Z., Huang Y. Bacterial cupredoxin azurin hijacks cellular signaling networks: Protein-protein interactions and cancer therapy. Review // Protein Sci. 2017. V. 26. № 12. P. 2334–2341. https://doi.org/10.1002/pro.3310
Rivera-Chávez F., Lopez C.A., Bäumler A.J. Oxygen as a driver of gut dysbiosis. Review // Free Radic. Biol. Med. 2017. V. 105. P. 93–101. https://doi.org/10.1016/j.freeradbiomed.2016.09.022
Wilson C.M., Loach D., Lawley B. et al. Lactobacillus reuteri 100-23 modulates urea hydrolysis in the murine stomach // Appl. Environ. Microbiol. 2014. V. 80. № 19. P. 6104–6113. https://doi.org/10.1128/AEM.01876-14
Kanamori T., Kanou N., Atomi H., Imanaka T. Enzymatic characterization of a prokaryotic urea carboxylase // J. Bacteriol. 2004. V. 186. № 9. P. 2532–2539. https://doi.org/10.1128/JB.186.9.2532-2539.2004
Ezraty B., Aussel L., Barras F. Methionine sulfoxide reductases in prokaryotes. Review // Biochim. Biophys. Acta. 2005. V. 1703. № 2. P. 221–229. https://doi.org/10.1016/j.bbapap.2004.08.017
Alcántara C., Coll-Marqués J.M., Jadán-Piedra C. et al. Polyphosphate in Lactobacillus and its link to stress tolerance and probiotic properties // Front. Microbiol. 2018. V. 9. 1944. https://doi.org/10.3389/fmicb.2018.01944
Celebioglu H.U., Svensson B. Exo- and surface proteomes of the probiotic bacterium Lactobacillus acidophilus NCFM // Proteomics. 2017. V. 17. 11. https://doi.org/10.1002/pmic.201700019
Savinova O.S., Glazunova O.A., Moiseenko K.V. et al. Exoproteome analysis of antagonistic interactions between the probiotic bacteria Limosilactobacillus reuteri LR1 and Lacticaseibacillus rhamnosus F and multidrug resistant strain of Klebsiella pneumonia // Int. J. Mol. Sci. 2021. V. 22. 10999. https://doi.org/10.3390/ijms222010999
Jeffery C.J. Intracellular/surface moonlighting proteins that aid in the attachment of gut microbiota to the host // AIMS Microbiology. 2019. V. 5. № 1. P. 77–86. https://doi.org/10.3934/microbiol.2019.1.77
Oberg T.S., Ward R.E., Steele J.L., Broadbent J.R. Transcriptome analysis of Bifidobacterium longum strains that show a differential response to hydrogen peroxide stress // J. Biotechnol. 2015. V. 212. P. 58–64. https://doi.org/10.1016/j.jbiotec.2015.06.405
Basu T.P., Long A.R., Nelson B.J. et al. Complex responses to hydrogen peroxide and hypochlorous acid by the probiotic bacterium Lactobacillus reuteri // mSystems. 2019. V. 4. № 5. e00453-19. https://doi.org/10.1128/mSystems.00453-19
Zhai Z., Yang Y., Wang H. et al. Global transcriptomic analysis of Lactobacillus plantarum CAUH2 in response to hydrogen peroxide stress // Food Microbiol. 2020. V. 87. 103389. https://doi.org/10.1016/j.fm.2019.103389
Yan X., Budin-Verneuil A., Verneuil N. et al. Transcriptomic response of Enterococcus faecalis V583 to low hydrogen peroxide levels // Curr. Microbiol. 2015. V. 70. № 2. P. 156–168. https://doi.org/10.1007/s00284-014-0691-8
Calderini E., Celebioglu H.U., Villarroel J. et al. Comparative proteomics of oxidative stress response of Lactobacillus acidophilus NCFM reveals effects on DNA repair and cysteine de novo synthesis // Proteomics. 2017. V. 17. № 5. https://doi.org/10.1002/pmic.201600178
Zhang C., Gui Y., Chen X. et al. Transcriptional homogenization of Lactobacillus rhamnosus hsryfm 1301 under heat stress and oxidative stress // Appl. Microbiol. Biotechnol. 2020. V. 104. № 6. P. 2611–2621. https://doi.org/10.1007/s00253-020-10407-3
Vermassen A., Leroy S., Talon R. et al. Cell wall hydrolases in bacteria: insight on the diversity of cell wall amidases, glycosidases and peptidases toward peptidoglycan // Front. Microbiol. 2019. V. 10. 331. https://doi.org/10.3389/fmicb.2019.00331
Al-Hassi H.O., Mann E.R., Sanchez B. et al. Altered human gut dendritic cell properties in ulcerative colitis are reversed by Lactobacillus plantarum extracellular encrypted peptide STp // Mol. Nutr. Food Res. 2014. V. 58. № 5. P. 1132–1143. https://doi.org/10.1002/mnfr.201300596
Siboo I.R., Chambers H.F., Sullam P.M. Role of SraP, a serine-rich surface protein of Staphylococcus aureus, in binding to human platelets // Infect. Immun. 2005. V. 73. P. 2273–2280. https://doi.org/10.1128/IAI.73.4.2273-2280.2005
Schramm F., Schroeder K., Jonas K. Protein aggregation in bacteria // FEMS Microbiol. Rev. 2020. V. 44. P. 54–72. https://doi.org/10.1093/femsre/fuz026
Alam A., Bröms J.E., Kumar R., Sjöstedt A. The role of ClpB in bacterial stress responses and virulence // Front. Mol. Biosci. 2021. V. 8. 668910. https://doi.org/10.3389/fmolb.2021.668910
Фетисов С.О. О роли кишечных бактерий в физиологической регуляции аппетита и энергетического обмена // Интегративная физиология. 2021. Т. 2. № 1. С. 21–32. https://doi.org/10.33910/2687-1270-2021-2-1-21-32
Дополнительные материалы
- скачать ESM.docx
- Таблица S1. Гены штамма L. fermentum U-21, кодирующие ферменты с антиоксидантными свойствами.
Таблица S2. Редкие белки, встречающиеся у L. fermentum U-21 и отдельных штаммов вида L. fermentum (по данным базы NCBI).
Таблица S3. Функциональная активность белков, соответствующих генам (отдельным и в составе предполагаемых оперонов) с максимально измененной экспрессией.


