Генетика, 2023, T. 59, № 1, стр. 89-96
Исследование генетического разнообразия в популяции кыргызского горного мериноса с использованием микросателлитных локусов
Ж. Т. Исакова 1, *, А. Б. Бектуров 2, Т. Д. Чортонбаев 2, В. Н. Кипень 3, С. Б. Мукеева 1, У. А. Шергазиев 2, К. А. Айтбаев 1
1 Научно-исследовательский институт молекулярной биологии и медицины
720040 Бишкек, Кыргызская Республика
2 Кыргызский национальный аграрный университет им. К.И. Скрябина
720005 Бишкек, Кыргызская Республика
3 Институт генетики и цитологии Национальной академии наук Беларуси
220072 Минск, Республика Беларусь
* E-mail: jainagul@mail.ru
Поступила в редакцию 22.02.2022
После доработки 08.06.2022
Принята к публикации 20.07.2022
- EDN: CMMVBJ
- DOI: 10.31857/S001667582301006X
Аннотация
Проведена оценка генетической структуры и генетического разнообразия кыргызского горного мериноса с использованием микросателлитных локусов ДНК. В исследование включены 109 овец данной породы, разводимых в трех государственных племенных заводах (ГПЗ): ГПЗ “Оргочор” (Иссык-Кульская обл.), ГПЗ “Катта-Талдык” (Ошская обл.) и ГПЗ им. М.Н. Лущихина (Таласская обл.). В 12-ти исследованных локусах (McM042, INRA006, McM527, ETH152, CSRD247, OarFCB20, INRA172, INRA063, MAF065, MAF214, INRA005, INRA023) было идентифицировано 126 аллелей. Число аллелей в каждом локусе варьировало от 6 до 16, при среднем значении 10.500 ± 0.957 аллелей на локус. Определены 67 редких аллелей (с частотой встречаемости менее 5.0%), что составляет 53.2% от общего количества выявленных аллелей. Наибольшее число аллелей наблюдалось в аутосомных локусах: INRA023 (12 аллелей), INRA005 (13 аллелей), OarFCB20 и INRA063 (по 14 аллелей), CSRD247 (16 аллелей). Результаты оценки популяционных параметров, включая эффективное число аллелей (Ne = 4.556 ± 0.394), уровень наблюдаемой (Ho = 0.731 ± 0.023) и ожидаемой (He = = 0.761 ± 0.021) гетерозиготности позволяют сделать заключение о высоких значениях генетического разнообразия исследуемой выборки овец породы кыргызский горный меринос и ее значительном генетическом потенциале.
В Кыргызской Республике 65% коренного населения проживает в сельской местности в условиях средне- и высокогорья и в силу климатических условий занимается животноводством, в основном – разведением овец. По последним статистическим данным, поголовье овец и коз в республике составляет более 6.1 млн голов, или 42.5% от всех видов поголовья скота и домашней птицы, и занимает первое место по количеству поголовья среди домашних сельскохозяйственных животных [1]. Среди различных пород овец по направлению продуктивности, поголовье тонкорунных овец составляет более 110 тыс. голов. В стадах племенных хозяйств по республике насчитывается более 16 тыс. племенных особей породы кыргызский горный меринос (КГМ). Сохранившийся генофонд представляет собой уникальную ценность и является государственным достоянием Кыргызской Республики. Кыргызский горный меринос уникален тем, что является продолжением кыргызской тонкорунной породы, которая была создана в 1956 г. и за все последующие годы проходила этапы своего дальнейшего совершенствования (рис. 1).
Рис. 1.
Баран-производитель из Государственного племенного завода им. М.Н. Лущихина (сверху); оценка руна тонкорунной шерсти (внизу, слева); белый цвет жиропота у баранчика (внизу, справа).

Завоз в республику из Австралии баранов-производителей и ярок породы австралийский меринос и их использование в стадах фермерских племенных заводов в 2005 г. позволило создать стадо нового качественного уровня, которое и получило название кыргызский горный меринос. В селекционном процессе применялись методы чистопородного разведения, поглотительного и реципрокного скрещивания. Разведение овец желательного типа в племенных стадах велось с применением не только традиционного генеалогического метода, но и за счет отбора по фенотипическим свойствам. В результате, через селекционный отбор прошло более 16 тыс. овец, сохранились только те особи, которые соответствовали требуемым показателям по своим адаптивным свойствам, настригу шерсти, качеству шерстного покрова и по живой массе [2]. Овцы данной породы производят тонкорунную шерсть и мясо высочайшего качественного уровня, оставаясь почти полностью на круглогодовом пастбищном содержании благодаря адаптивной приспособленности к высокогорным экстремальным условиям [2].
Высокое качество шерсти и ее ценность достигаются в первую очередь благодаря правильно выстроенной селекционной программе, а также использованию в этих целях новейших эффективных технологий. В Кыргызстане же селекция сельскохозяйственных животных проводится, в основном, старыми традиционными методами. При этом современный и высокоэффективный методы – оценка генотипа животных с использованием ДНК-маркеров – начал применяться относительно недавно и до сих пор не является широко распространенной практикой [3]. Обобщение и анализ данных научной литературы показывают, что в настоящее время для решения селекционно-племенных задач, связанных с определением породной принадлежности, происхождения потомков, их генетического полиморфизма и ассоциации с продуктивностью животных активно используются микросателлитные локусы STR (Short Tandem Repeat) [3–8].
В этой связи, оценка внутрипопуляционного генетического разнообразия кыргызского горного мериноса на основании анализа STR будет способствовать определению основ самобытности данной породы овец, т. к. ранее подобные исследования не проводились. Кроме того использование в селекции современных молекулярно-генетических методов может предоставить важную информацию, которая ляжет в основу алгоритмов и схем для поддержания внутрипородного генетического разнообразия, а также сохранения генетической идентичности данной породы и корректировки в селекционно-племенных программах. В этих целях в перспективе планируется проведение ряда мероприятий по совершенствованию племенных качеств овец породы кыргызский горный меринос. Поэтому работа по оценке внутрипопуляционного генетического разнообразия кыргызского горного мериноса характеризуется высоким уровнем оригинальности и является передовой.
Таким образом, цель данного исследования – оценка генетической структуры и генетического разнообразия кыргызского горного мериноса с использованием микросателлитных локусов ДНК.
МАТЕРИАЛЫ И МЕТОДЫ
Биологическим материалом для молекулярно-генетического исследования послужили образцы крови овец породы КГМ, взятые у взрослого поголовья – 109 животных, разводимых в трех государственных племенных заводах (ГПЗ): “Оргочор” (село Оргочор, Джеты-Огузский р-н, Иссык-Кульская обл.) – 29 животных, “Катта-Талдык” (село Баш-Булак, Карасуйский р-н, Ошская обл.) – 35 животных, ГПЗ им. М.Н. Лущихина (село Джоон-Тюбе, Кара-Буринский р-н, Таласская обл.) – 45 животных.
Выделение ДНК проводилось методом фенол-хлороформной экстракции [9]. Образцы были генотипированы по 12-ти микросателлитным локусам, рекомендованным Международным обществом генетики животных (ISAG, International Society for Animal Genetics): McM042, INRA006, McM527, ETH152, CSRD247, OarFCB20, INRA172, INRA063, MAF065, MAF214, INRA005, INRA023, – и полспецифичному локусу AMEL.
Генотипирование проводилось с использованием набора реагентов для мультиплексного анализа COrDIS Sheep (ООО “ГОРДИЗ”, РФ) согласно рекомендациям производителя. Для корректного определения генотипа у исследуемых животных (физического размера ампликона в пн) был использован образец с контрольным генотипом, включенным в набор COrDIS Sheep. Анализ результатов ПЦР проводился методом капиллярного высокоразрешающего электрофореза с использованием автоматического генетического анализатора 3500 Applied Biosystems (ThermoFisher, США).
Статистическую обработку данных проводили с использованием программы GenAIEx v.6.503 [10]. Были рассчитаны следующие показатели: среднее число аллелей на локус (Na), эффективное число аллелей (Ne), уровни ожидаемой (He) и наблюдаемой (Ho) гетерозиготности, коэффициент FIS [11]. Коэффициенты информационного полиморфизма PIC (рolymorphism information content) были рассчитаны с использованием веб-приложения Gene-Calc [12].
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Современная порода овец КГМ обнаруживает высокий уровень внутрипородной генетической вариабельности – в 12-ти исследованных микросателлитных локусах, расположенных на аутосомах, было идентифицировано 126 аллелей. Число аллелей в каждом локусе варьировало от 6 до 16, при среднем значении 10.500 ± 0.957 аллелей на локус. Наибольшее число аллелей наблюдалось в аутосомных локусах: INRA023 (12 аллелей), INRA005 (13 аллелей), OarFCB20 и INRA063 (по 14 аллелей), CSRD247 (16 аллелей), табл. 1.
Таблица 1.
Аллели, идентифицированные в экспериментальной выборке овец породы КГМ
| STR | Локализация# (хромосома) |
Аллели |
|---|---|---|
| CSRD247 | 14 | 209*/211*/213/215*/217*/223/225* 227**/229/231/233*/235*/237*/239 241*/243* |
| ETH152 | 3 | 186**/188/190/192/198*/200* |
| INRA005 | 10 | 113*125/127/129*/131/133*/135/ 137*/139*/141/143*/145/147* |
| INRA006 | 1 | 110**/112*/114*/116/118*/120*/ 124*/126*/132/134* |
| INRA023 | 3 | 192*/198/200/202/204/206/208*/ 210*/212*/214*/216/218* |
| INRA063 | 14 | 167*/169/171/173*/175**/177/179*/ 183/187*/189/195*/197*/199*/201* |
| INRA172 | 22 | 126/144*/154**/156*/158*/160/162*/ 164/166*/168* |
| MAF065 | 15 | 123*/125**/127**/129/131*/135/137* |
| MAF214 | 16 | 183*/187/189**/191/221*/223*/225*/ 255*/261*/269* |
| McM042 | 9 | 81/87**/89/95/97*/99/103* |
| McM527 | 5 | 158*/164/166**/168/170/172/176* |
| OarFCB20 | 2 | 77*/83*/87/89**/91/93/95*/99/101/ 103*/105/107*/111*/113* |
Определены 67 редких аллелей (с частотой встречаемости менее 5.0%), что составляет 53.2% от общего количества выявленных аллелей, табл. 1. Наибольшее количество редких аллелей выявлено для STR: CSRD247 (10 аллелей, суммарный процент распространенности – 18.3%), INRA023 (8 аллелей, 12.8%), INRA005 (7 аллелей, 21.1%), INRA006 (7 аллелей, 12.8%), MAF214 (7 аллелей, 12.8%) и OarFCB20 (7 аллелей, 11.9%).
Установлено, что для некоторых STR-локусов с большим числом наблюдаемых аллелей характерны относительно невысокие значения такого показателя, как число эффективных аллелей (Ne), табл. 2. Данное явление объясняется наличием в локусах редких аллелей с частотой встречаемости менее 5.0%. В частности, более половины всех идентифицированных аллелей относилось к данному типу. Число эффективных аллелей значительно варьировало – от 2.831 (INRA172) до 6.673 (INRA023), при среднем значении 4.556 ± 0.394 аллелей на локус.
Таблица 2.
Генетическая характеристика овец породы КГМ по 12-ти STR-локусам
| STR | Na | Ne | Ho | He | PIC | FIS |
|---|---|---|---|---|---|---|
| CSRD247 | 16 | 5.690 | 0.807 | 0.824 | 0.806 | 0.021 |
| ETH152 | 6 | 3.375 | 0.706 | 0.704 | 0.654 | –0.004 |
| INRA005 | 13 | 6.384 | 0.835 | 0.843 | 0.828 | 0.010 |
| INRA006 | 10 | 3.236 | 0.661 | 0.691 | 0.650 | 0.044 |
| INRA023 | 12 | 6.673 | 0.752 | 0.850 | 0.835 | 0.115 |
| INRA063 | 14 | 5.322 | 0.807 | 0.812 | 0.799 | 0.006 |
| INRA172 | 10 | 2.831 | 0.661 | 0.647 | 0.624 | –0.021 |
| MAF065 | 7 | 3.443 | 0.679 | 0.710 | 0.656 | 0.043 |
| MAF214 | 10 | 3.100 | 0.606 | 0.677 | 0.638 | 0.106 |
| McM042 | 7 | 4.420 | 0.697 | 0.774 | 0.742 | 0.099 |
| McM527 | 7 | 4.489 | 0.706 | 0.777 | 0.746 | 0.091 |
| OarFCB20 | 14 | 5.708 | 0.853 | 0.825 | 0.809 | –0.034 |
| Среднее значение: | 4.556 | 0.731 | 0.761 | 0.732 | 0.040 | |
Показатель наблюдаемой гетерозиготности Ho находился в диапазоне от 0.606 (MAF214) до 0.853 (OarFCB20), при среднем значении 0.731 ± 0.023. Для локусов ETH152, INRA172 и OarFCB20 показатель Ho был выше предполагаемого He (табл. 2).
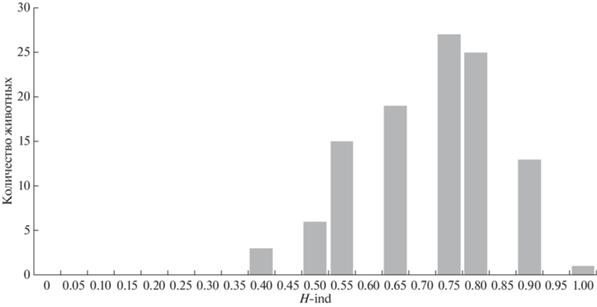
Распределение показателя H-ind, характеризующего совокупный процент (частоту) гетерозиготных генотипов для особей по исследуемым 12-ти STR представлено на рис. 2. Значение показателя H-ind, которое составило в среднем 0.731 ± 0.130, позволяет заключить, что исследуемые локусы являются высокополиморфными для данной выборки животных. Нарушение генетического равновесия выявлено по микросателлитным локусам INRA006 (ChiSq = 72.00, DF = 45, p < 0.01) и MAF214 (ChiSq = 67.90, DF = 45, p < 0.05). Однако при введении поправки на множественные сравнения данные различия нивелировались. Для остальных STR статистически значимых отклонений от равновесия Харди–Вайнберга (HWE) не выявлено (p > 0.05).
Величина PIC определяется способностью STR-локуса устанавливать полиморфизм в популяции в зависимости от числа обнаруживаемых аллелей и распределения их частот [13]. Таким образом, PIC зависит от числа известных аллелей, а также распределения их частот и, тем самым, эквивалентен генетическому разнообразию. Результаты расчетов показали, что для всех локусов показатель PIC превышает значение 0.5, что свидетельствует об их высокой информативной ценности для выявления генетических различий между животными. Наибольшие показатели PIC имели STR: CSRD247 (0.806), OarFCB20 (0.809), INRA005 (0.828) и INRA023 (0.835). Наименьшие значения показателя информативности отмечались у локусов INRA172 (0.624) и MAF214 (0.638).
FIS – индивидуальный индекс фиксации, который указывает на редукцию гетерозиготности из-за неслучайного спаривания и означает меру отклонения генотипических частот от таковых при HWE внутри субпопуляций с точки зрения недостатка или избытка гетерозигот. При FIS > 0 имеет место дефицит гетерозиготных особей (родственное спаривание) – показано для 9 из 12 STR-локусов – CSRD247, INRA005, INRA006, INRA023, INRA063, MAF065, MAF214, McM042 и McM527, а при FIS < 0 – избыток гетерозигот (неродственное спаривание) – показано для ETH152, INRA172 и OarFCB20. Наибольшие рассчитанные значения коэффициента FIS были показаны для STR McM527 (0.091), McM042 (0.099), MAF214 (0.106) и INRA023 (0.115), при среднем значении 0.040 ± ± 0.015. Среднее значение FIS позволяет сделать заключение о наличии незначительного сдвига в сторону процессов инбридинга.
Нами проведен сравнительный анализ параметров Na и He для КГМ и овец тонкорунных пород, разводимых в Казахстане [14], России [15], Пакистане [16] и Польше [17], табл. 3.
Таблица 3.
Генетическая характеристика выборок овец тонкорунных пород по результатам генотипирования STR-локусов
| Порода (n) | STR | Na | Ho | Ссылка |
|---|---|---|---|---|
| Казахстан | ||||
| казахский архарский меринос* (15) | 12 | 7.08 ± 0.64 | 0.678 ± 0.051 | [14] |
| казахская тонкошерстная* (15) | 7.92 ± 0.56 | 0.744 ± 0.048 | ||
| Россия | ||||
| грозненская* (30) | 11 | 9.00 ± 1.14 | 0.540 ± 0.089 | [15] |
| ставропольская* (32) | 9.20 ± 0.92 | 0.575 ± 0.061 | ||
| манычский меринос* (30) | 8.20 ± 0.90 | 0.647 ± 0.055 | ||
| советский меринос* (23) | 8.00 ± 0.75 | 0.651 ± 0.060 | ||
| сальская* (30) | 8.50 ± 0.92 | 0.512 ± 0.089 | ||
| волгоградская* (30) | 8.90 ± 1.22 | 0.525 ± 0.082 | ||
| дагестанская горная* (30) | 9.00 ± 1.07 | 0.560 ± 0.079 | ||
| забайкальская тонкорунная** (30) | 8.90 ± 0.77 | 0.891 ± 0.018 | ||
| кулундинская* (30) | 7.20 ± 0.98 | 0.489 ± 0.095 | ||
| Пакистан | ||||
| каил* (47) | 11 | 5.27 ± 1.49 | 0.766 ± 0.248 | [16] |
| Польша | ||||
| старопольский меринос* (93) | 11 | 7.18 ± 1.94 | 0.663 ± 0.167 | [17] |
| олкуска** (88) | 5.64 ± 1.29 | 0.689 ± 0.138 | ||
| великопольская* (100) | 7.82 ± 2.23 | 0.710 ± 0.065 | ||
| Кыргызстан | ||||
| кыргызский горный меринос* (109) | 12 | 10.50 ± 0.96 | 0.731 ± 0.023 | – |
Установлено, что для КГМ среднее значение параметра Na (в контексте исследуемых в данной работе STR-локусов) было максимальным в сравнении с результатами других исследователей. Данный факт может свидетельствовать как о значительной полиморфности анализируемых нами локусов, так и о существенно высоком генетическом разнообразии изучаемой выборки КГМ. Высокие показатели генетического разнообразия КГМ напрямую связаны с многоступенчатыми селекционными процессами, которым подверглась данная порода на протяжении конца XX в.–начала XIX в. Предположительно, существенный качественный вклад в совершенствование овец КГМ внес процесс углубленной внутрипородной селекции и скрещивания кыргызской тонкорунной овцы с импортными австралийскими мериносами. На данный момент порода КГМ включает пять заводских типов и 23 заводские линии, различающихся по некоторым хозяйственно полезным признакам (живая масса, мясные свойства, технологические свойства шерсти, длина волокон, густота волокон), а также на основании комплексного сочетания признаков [2]. Таким образом, значительное генетическое разнообразие КГМ – следствие грамотных селекционных процессов на протяжении длительного времени.
Рассчитанный показатель Ho также оказался одним из самых больших и был сопоставим со значениями, полученными для пород Великопольская (Польша), Олкуска (Польша), Каил (Пакистан) и Казахская тонкошерстная (Казахстан) [14, 16, 17]. В то же время порода овец КГМ не стала исключением в контексте среднего значения FIS – для 12 из 15 тонкорунных пород овец (не включая КГМ) показан сдвиг в сторону процессов инбридинга. Однако для КГМ это смещение не носит такого серьезного характера как, например, для пород Кулундинская и Сальская (Россия) [15].
Проблема сохранения генетических ресурсов домашних животных – одна из самых актуальных в мире. Для сохранения и поддержания на высоком уровне генетического разнообразия и эффективного использования малочисленных стад предложены различные алгоритмы. Установлено, что при разделении крупной популяции на субпопуляции, среди которых сохраняется обмен единичными особями (например баранами-производителями или ремонтными баранами), устойчивость системы в целом повышается, и потери аллелей не происходит [18]. В контексте проводимых исследований важным и актуальным их продолжением должен стать анализ субпопуляционной подразделенности данной выборки КГМ, т. к. своевременный обмен биологическим материалом между племенными заводами и специлистами, профессионально занимающимися разведением данной породы овец, будет способствовать поддержанию генетического разнообразия породы в целом.
Таким образом, по результатам оценки среднего числа аллелей на локус (Na = 10.500 ± 0.957), эффективного числа аллелей (Ne = 4.556 ± 0.394), уровней наблюдаемой (Ho = 0.731 ± 0.023) и ожидаемой (He = 0.761 ± 0.021) гетерозиготностей можно сделать вывод о высоких значениях генетического разнообразия исследуемой выборки овец породы кыргызский горный меринос и ее значительном генетическом потенциале. Однако положительные значения индекса фиксации FIS для девяти из 12-ти STR-локусов указывают на незначительное смещение генетического равновесия в сторону процессов инбридинга (FIS = 0.040 ± 0.021), не имеющего на данный момент критического значения. В то же время коэффициенты информационного полиморфизма (PIC = 0.732 ± 0.082) для всех локусов имели высокие значения, более 70% всех генотипов по анализируемым локусам находились в гетерозиготном состоянии.
Сравнительный анализ результатов молекулярно-генетического исследования кыргызского горного мериноса, разводимого в государственных племенных заводах Кыргызстан, с данными аналогичных исследований других тонкорунных пород овец, позволяют сделать заключение о его значительном генетическом разнообразии.
По результатам проведенного исследования очевидно, что для сохранения кыргызского горного мериноса и поддержания оптимального уровня генетического разнообразия должен быть проведен контроль параметров генетической структуры субпопуляций. Данные исследования будут проведены в ближайшей перспективе.
Исследование выполнено при финансовой поддержке Министерства образования и науки Кыргызской Республики в рамках Научного проекта “Использование молекулярно-генетических методов исследований и создание информационных ресурсов для интенсификации селекции овец в Кыргызской Республике”.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Об итогах единовременного пересчета скота и домашней птицы в Кыргызской Республике. http://www.stat.kg/ru/news/ob-itogah-edinovremennogo-perescheta-skota-i-domashnej-pticy-v-kyrgyzskoj-respublike/
Лущихина Е.М., Чебодаев Д.В. Кыргызский горный меринос // НАН КР-Б., 2014. 204 с.
Исакова Ж.Т., Исаев М.А., Кипень В.Н и др. Оценка генетического разнообразия кыргызской породы лошадей с использованием микросателлитных маркеров – расширенное геногеографическое исследование // Генетика. 2021. Т. 57. № 4. С. 420–428. https://doi.org/10.31857/S0016675821040032
Денискова Т.Е., Доцев А.В., Охлопков И.М. и др. Характеристика генетической структуры снежного барана (Ovis nivicola lydekkeri) Верхоянской горной страны // Генетика. 2018. Т. 54. № 3. С. 342–348. https://doi.org/10.7868/S0016675818030074
Kharzinova V.R., Zinovieva N.A. Pattern of genetic diversity in local and commercial pig breeds based on microsatellite analysis // Vavilov J. Genet. and Breeding. 2020. V. 24(7). P. 747–754. https://doi.org/10.18699/VJ20.669
Lemesh V.A., Ageets V.Yu., Nosonova A.Yu. et al. Genetic structure of the carp (Cyprinus carpio carpio) population grown in aquaculture in the Republic of Belarus // Rep. Natl Acad. Sci. of Belarus. 2021. V. 65. № 1. P. 68–75. https://doi.org/10.29235/1561-8323-2021-65-1-68-75
Носова А.Ю., Кипень В.Н., Царь А.И., Лемеш В.А. Дифференциация гибридного потомства белого (Hypophthalmichthys molitrix val.) и пестрого (H. nobilis rich.) толстолобиков на основании полиморфизма микросателлитных локусов // Генетика. 2020. Т. 56. № 3. С. 313–320. https://doi.org/10.31857/S0016675820030121
Исакова Ж.Т., Тонкосунов Б.И., Кипень В.Н. и др. Филогенетический анализ для кыргызской породы лошадей по 17 микросателлитным маркерам // Генетика. 2019. Т. 55. № 1. С. 94–99. https://doi.org/10.1134/S0016675819010077
Sambrook J., Russell D.W. Molecular Cloning: A Laboratory Manual. New York: Cold Spring Harbor Laboratory Press, 2001. 2344 p.
Peakall R., Smouse P.E. GenAlEx 6.5: Genetic analysis in Excel. Population genetic software for teaching and research – an update // Bioinformatics. 2012. V. 28(19). P. 2537–2539. https://doi.org/10.1093/bioinformatics/bts460
Excoffier L., Smouse P.E., Quattro J.M. Analysis of molecular variance inferred from metric distances among DNA haplotypes: Application to human mitochondrial DNA restriction data // Genetics. 1992. V. 131(2). P. 479–491. https://doi.org/10.1093/genetics/131.2.479
Polymorphic information content & Heterozygosity [Электронный ресурс]. Режим доступа: https://www.gene-calc.pl/pic. Дата доступа: 10.02.2021.
Botstein D., White R.L., Skalnick M.H., Davies R.W. Construction of a genetic linkage map in man using restriction fragment length polymorphism // Am. J. Hum. Genet. 1980. V. 32. P. 314–331.
Dossybayev K., Orazymbetova Z., Mussayeva A. et al. Genetic diversity of different breeds of Kazakh sheep using microsatellite analysis // Arch. Anim. Breed. 2019. V. 62(1). P. 305–312. https://doi.org/10.5194/aab-62-305-2019
Денискова Т.Е., Селионова М.И., Гладырь Е.А. и др. Изменчивость микросателлитов в породах овец, разводимых в России // С.-хоз. биология. 2016. Т. 51. № 6. С. 801–810. https://doi.org/10.15389/agrobiology.2016.6.801rus
Ahmed Z., Babar M.E., Hussain T. et al. Genetic diversity analysis of Kail Sheep by using microsatellite markers // J. Anim. Plant Sci. 2014. V. 24(5). P 1329–1333.
Szumiec A., Radko A., Koseniuk A. et al. Application of 11 STR markers for the evaluation of genetic variation in sheep. The global standard for livestock date // ICAR Technical Series. 2018. V. 23. P. 141–145.
Столповский Ю.А., Захаров-Гезехус И.А. Проблема сохранения генофондов доместицированных животных // Вавиловский журн. генет. селекции. 2017. Т. 21. № 4. С. 477–486. https://doi.org/10.18699/VJ17.266
Дополнительные материалы отсутствуют.