Генетика, 2023, T. 59, № 1, стр. 106-114
Митохондриальный генофонд украинцев в контексте изменчивости целых митогеномов у славянских народов
Б. А. Малярчук 1, *, М. В. Деренко 1
1 Институт биологических проблем Севера Дальневосточного отделения
Российской академии наук
685000 Магадан, Россия
* E-mail: malyarchuk@ibpn.ru
Поступила в редакцию 14.03.2022
После доработки 24.04.2022
Принята к публикации 23.06.2022
- EDN: CMXSQZ
- DOI: 10.31857/S0016675823010083
Аннотация
Проанализированы данные об изменчивости нуклеотидных последовательностей целых митохондриальных геномов у украинцев. Сравнительный анализ параметров генетического разнообразия показал, что украинцы, как и другие европейские народы, характеризуются высоким уровнем разнообразия мтДНК. Однако межпопуляционные различия в Европе очень низки (FST = 0.36%, p < 0.00001). По результатам FST-анализа украинцы проявляют наибольшее сходство со словаками, русскими, поляками, сербами и эстонцами. На графике многомерного шкалирования FST-дистанций украинцы располагаются вместе с западными и восточными славянами, занимая среди них центральное положение. Результаты анализа динамики эффективной численности (Ne) популяций показали различия по долговременной динамике Ne между севером и югом Восточной Европы. Для украинцев и юго-западного русского населения период резкого роста численности зарегистрирован в неолитическое время (примерно 8.2 тыс. лет назад), в то время как у эстонцев и северо-западных русских рост численности популяций наблюдается значительно позже – в эпоху бронзового века (примерно 4.3 тыс. лет назад). Анализ данных об изменчивости мтДНК в славянских популяциях показал, что частота этноспецифичных гаплотипов мтДНК достаточно широко изменяется в различных этнических группах – от 1.3 у словаков до 10.3% у поляков. Доля славяно-специфичных гаплотипов мтДНК также варьирует: меньше всего такого рода гаплотипов обнаружено у чехов и сербов (менее 10%), а больше всего – у украинцев (23.6%).
В последние десятилетия существенно увеличилось число научных исследований, посвященных анализу изменчивости нуклеотидных последовательностей целых митохондриальных геномов (митогеномов) в популяциях человека. Это позволяет на значительно более детальном уровне реконструировать филогенетические связи между материнскими линиями митохондриальной ДНК (гаплотипами мтДНК) в сравнении с более ранними исследованиями, базировавшимися на анализе полиморфизма отдельных гипервариабельных участков мтДНК (ГВС1 и ГВС2). Результаты исследований изменчивости целых митогеномов способствуют дальнейшему развитию целого ряда направлений, таких как генетика популяций человека, криминалистика и судебная генетика, палеогеномика и этнология. Высокая информативность анализа полиморфизма целых молекул мтДНК находит применение в реконструкции генетической истории народов [1–3]. В последние годы довольно интенсивно развиваются в данном направлении исследования разнообразия митохондриальных генофондов славян. Уже получены популяционные данные об изменчивости митогеномов в различных группах славян: у русских, поляков, сербов, чехов, словаков и болгар [4–7]. Однако некоторые (важные в плане исследования этногенеза славян) этнические группы – например, украинцы – очень мало задействованы в исследованиях генетической истории славян.
Целью настоящей работы, таким образом, является исследование структуры и разнообразия митохондриального генофонда украинцев, основываясь на данных об изменчивости целых молекул мтДНК. Необходимо отметить, что ранее исследования полиморфизма мтДНК у украинцев проводились, главным образом, с помощью анализа нуклеотидных последовательностей ГВС1 (иногда с дополнительным включением ГВС2), а также анализа распределения частот гаплогрупп мтДНК в популяциях [8–13]. Результаты генокартографических исследований показали, что по распределению вариантов полиморфизма мтДНК восточнославянские популяции проявляют наибольшую степень сходства между собой; при этом генофонд украинцев более всего сходен с населением Польши, Беларуси и юга России [10, 12]. Также получены данные о возможном некотором своеобразии западных популяций украинцев [9] и их большем генетическом сходстве с западными славянами [13]. Данные об изменчивости нуклеотидных последовательностей целых митогеномов украинцев использовались лишь в филогеографических исследованиях некоторых гаплогрупп мтДНК [11, 14–17] и поэтому анализ структуры и разнообразия митохондриального генофонда украинцев, основанный на данных об изменчивости целых митогеномов, представляется вполне актуальным.
МАТЕРИАЛЫ И МЕТОДЫ
В настоящей работе проанализированы нуклеотидные последовательности целых митохондриальных геномов 144 индивидуумов украинского происхождения, опубликованных в работах [11, 14–21], а также базах данных GenBank (www.ncbi.nlm.nih.gov), mtDNA Community (www.mtdnacommunity.org), Family Tree DNA project (https://www.familytreedna.com) и Logan DNA Project (http://www.ianlogan.co.uk) (табл. 1 дополнительных материалов). Этническая принадлежность исследованных образцов установлена на основании сведений, представленных в базах данных. Кроме этого, определены нуклеотидные последовательности целых митогеномов у трех индивидуумов украинского происхождения. Для этого использованы методы и подходы, подробно описанные нами ранее [2]. Нуклеотидные последовательности мтДНК депонированы в GenBank под номерами OM802873, OM802874 и OM802875.
Таблица 1.
Генетическое разнообразие в популяциях Европы по данным об изменчивости целых митохондриальных геномов
| Популяция | N | k | Hd (s.e.) | π (s.e.) | mpd | D (p) |
|---|---|---|---|---|---|---|
| Украинцы | 144 | 141 | 0.999 (0.001) | 0.0018 (0.0001) | 30.15 | –2.42 (p < 0.01) |
| Русские | 376 | 361 | 0.999 (0.000) | 0.0018 (0.0001) | 29.03 | –2.57 (p < 0.001) |
| Чехи | 150 | 140 | 0.999 (0.001) | 0.0017 (0.0001) | 28.52 | –2.48 (p < 0.001) |
| Словаки | 139 | 133 | 0.999 (0.001) | 0.0017 (0.0001) | 27.93 | –2.45 (p < 0.01) |
| Татары поволжские | 73 | 68 | 0.998 (0.003) | 0.0021 (0.0001) | 35.2 | –2.33 (p < 0.01) |
| Эстонцы | 114 | 106 | 0.999 (0.0014) | 0.0017 (0.0001) | 27.93 | –2.33 (p < 0.01) |
| Сардинцы | 63 | 50 | 0.992 (0.004) | 0.0015 (0.0001) | 24.31 | –1.8 (p < 0.05) |
| Тосканцы | 110 | 109 | 0.999 (0.0013) | 0.0019 (0.0001) | 30.68 | –2.58 (p < 0.001) |
| Венгры | 80 | 78 | 0.999 (0.002) | 0.0018 (0.0001) | 29.82 | –2.36 (p < 0.01) |
| Сербы | 228 | 207 | 0.999 (0.0005) | 0.0017 (0.0001) | 27.57 | –2.53 (p < 0.001) |
| Поляки | 300 | 287 | 0.999 (0.0003) | 0.0019 (0.0001) | 30.85 | –2.56 (p < 0.001) |
| Болгары | 126 | 119 | 0.999 (0.0011) | 0.0018 (0.0001) | 28.99 | –2.42 (p < 0.01) |
| Датчане | 175 | 167 | 0.999 (0.0007) | 0.0017 (0.0001) | 28.39 | –2.4 (p < 0.01) |
| Суммарно | 2073 | 1841 | 0.999 (0.0001) | 0.0017 (0.0001) | 27.8 | –2.62 (p < 0.001) |
Филогенетические взаимоотношения между нуклеотидными последовательностями мтДНК реконструировали с помощью метода максимальной экономии, реализованного в пакете компьютерных программ mtPhyl v4.015 (http://eltsov.org). Полиморфизм длины в повторяющихся участках мтДНК 16180–16193, 309–315 и 514–524, а также замены в гипервариабельной нуклеотидной позиции 16519 при построении деревьев не учитывали. Для определения эволюционного возраста монофилетических кластеров мтДНК использовали заложенные в программе mtPhyl молекулярные часы, основанные на коррекции долговременной филогенетической скорости мтДНК человека (одна нуклеотидная замена в целом митогеноме за 3624 года, или 1.665 × 10–8 замен на сайт в год) с учетом действия отрицательного отбора [22].
За основу в работе использована филогенетическая классификация гаплогрупп мтДНК, предложенная разработчиками ресурса PhyloTree (www.phylotree.org). Для филогеографического анализа гаплотипов мтДНК, включая анализ популяционной специфичности кластеров мтДНК, применяли информацию об изменчивости целых митогеномов у представителей различных популяций человека, согласно данным GenBank, PhyloTree, mtDNA Community, Logan DNA Project, проекта 1000 Genomes (http://www.internationalgenome.org), Family Tree DNA project. По состоянию на конец 2021 г. в GenBank содержится более 52 000 митохондриальных геномов от представителей различных этнических групп мира (https://www.mitomap.org). Монофилетические кластеры мтДНК учитывались нами как этноспецифичные, если не менее 75% гаплотипов мтДНК в кластере были характерны только для представителей определенной этнической группы (украинцы) или этнической общности (славяне).
Для исследования демографической истории украинцев использовали байесовский анализ эффективной численности популяций (Ne), основанный на данных об изменчивости целых митохондриальных геномов. Для данного анализа применяли пакет программ BEAST 1.10.2 [23]. Наиболее оптимальная модель нуклеотидных замен (HKY + G + I) была выбрана с помощью пакета программ MEGA 5.05 [24]. В анализе использовали строгие молекулярные часы и мутационную скорость, равную 2.041 × 10–8 замен на сайт в год. Это значение является оптимальным для изучения динамики Ne в последние 30 тыс. лет истории согласно данным об изменениях значений мутационной скорости мтДНК человека во времени [22, 25]. Протяженность байесовского анализа динамики эффективной численности популяций составляла от 100 до 400 млн циклов для выборок соответственно меньшего и большего размера. Для анализа данных, сгенерированных BEAST 1.10.2, применяли пакет программ Tracer 1.4. Стабилизацию марковских цепей MCMC (Markov Сhain Monte Carlo) считали завершенной, если значение параметра ESS (Effective Sample Size) для всех статистик было более 200. Изменения эффективной численности популяций оценивали не в абсолютных значениях, а с помощью пропорционального Ne параметра Ne μ (произведение эффективной численности популяций и мутационной скорости), вычисляемого непосредственно по данным об изменчивости мтДНК с помощью пакета программ BEAST 1.10.2.
Для проведения сравнительного анализа полногеномной изменчивости мтДНК на популяционном уровне использовали, кроме украинской выборки, опубликованные данные для русских [4], поволжских татар [26], эстонцев [27], венгров [28], сербов [7], поляков [4–6], чехов (данные GenBank), словаков (данные GenBank), болгар (Family Tree DNA project), датчан [29], тосканцев (1000 Genomes Project), сардинцев [30]. Параметры генетического разнообразия в популяциях рассчитывали с помощью пакета программ DnaSP 5.10.01 [31]. Анализ молекулярной изменчивости (AMOVA, FST-анализ), основанный на попарных нуклеотидных различиях между митогеномами, проводили с помощью пакета программ Arlequin 3.5.1.2 [32]. Расположение популяций в двухмерном пространстве исследовали с помощью метода многомерного шкалирования межпопуляционных FST-различий, реализованного в пакете программ STATISTICA10 package (StatSoft Inc., Tulsa, OK, USA).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Анализ разнообразия мтДНК в европейских популяциях
Анализ параметров разнообразия митохондриальных генофондов европейских популяций показал, что украинский генофонд не отличается от генофондов других европейских народов ни по уровню разнообразия гаплотипов (Hd), ни по показателям нуклеотидного разнообразия (π и mpd) (табл. 1). Европейские митохондриальные генофонды характеризуются высоким генетическим разнообразием и очень низкими значениями D‑статистики Таджимы, указывающими на избыток вариантов полиморфизма, распространенных в популяциях с низкой частотой. Это может свидетельствовать о недавнем росте численности популяций или о воздействии отрицательного отбора на полиморфизм мтДНК [32].
Периоды популяционного роста численности обнаруживаются и при анализе динамики эффективного размера популяций по данным об изменчивости целых митохондриальных геномов у украинцев. Первый из этих периодов связан с медленным и продолжительным увеличением численности от примерно 50 тыс. лет тому назад, за которым последовал медленный спад численности в ледниковое время (20–30 тыс. лет тому назад) (график не показан). Следующий рост численности населения наблюдается в послеледниковый период, начиная от 10 тыс. лет назад (95%-ный доверительный интервал (ДИ): 9.5–10.6 тыс. лет назад). Наиболее резкий (10-кратный) рост численности произошел в период от 10 до 5.3 тыс. лет назад.
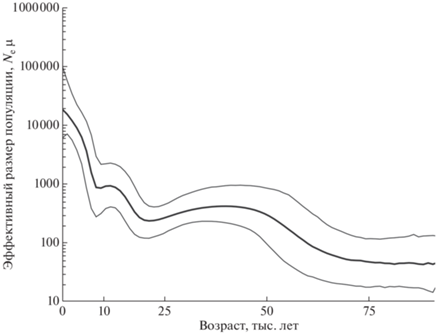
Между тем предыдущие исследования показали, что более детальная картина динамики Ne популяций может быть получена в выборках большего размера, чем исследованная выборка украинцев (N = 144) [3, 33]. Поэтому мы добавили к выборке украинских митогеномов аналогичные данные для географически близких русских популяций Белгородской, Орловской и Тульской областей, увеличив размер выборки до 315 митогеномов. Результаты анализа показали увеличение численности населения южной части Восточной Европы от минимума Ne примерно 21 тыс. лет назад (что соответствует максимуму последнего оледенения) до максимума примерно 11.8 тыс. лет назад с последующим снижением до 9.4 тыс. лет назад и затем резким (20-кратным) ростом численности, начавшимся примерно 8.2 тыс. лет назад (95% ДИ: 7.1–9.4 тыс. лет назад) (рис. 1).
Рис. 1.
Байесовский график динамики эффективной численности популяций во времени, построенный по данным об изменчивости целых митохондриальных геномов украинцев и русских Белгородской, Орловской и Тульской областей. На графике показаны значения медианы и 95%-ного доверительного интервала с наивысшей апостериорной вероятностью.
Поскольку демографическая история населения юга и севера Восточной Европы может различаться [34], нами проведен анализ динамики Ne в популяциях псковско-новгородского русского населения и их соседей – эстонцев (рис. 2). Анализ 246 митогеномов показал, что эффективная численность населения Северо-Запада начала расти лишь примерно 18 тыс. лет тому назад до максимальных значений Ne примерно 8.6 тыс. лет назад (95% ДИ: 8.0–9.2 тыс. лет назад). После этого периода популяционный размер снижался, однако примерно 4.3 тыс. лет назад (95% ДИ: 3.7–4.9 тыс. лет назад) произошел довольно резкий (почти 7-кратный) популяционный рост.
Рис. 2.
Байесовский график динамики эффективной численности популяций во времени, построенный по данным об изменчивости целых митохондриальных геномов русских Псковской и Новгородской областей и эстонцев. На графике показаны значения медианы и 95%-ного доверительного интервала с наивысшей апостериорной вероятностью.
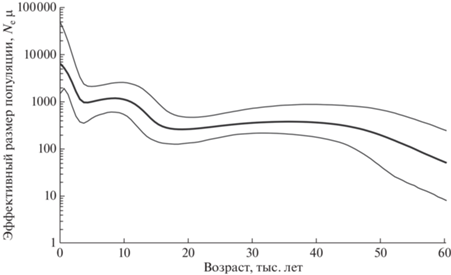
Таким образом, результаты проведенного анализа свидетельствуют о различиях по долговременной динамике Ne между севером и югом Восточной Европы. Ранее по данным о распределении частот гаплогрупп мтДНК у населения Восточной Европы был сделан вывод о том, что внутрипопуляционное разнообразие мтДНК снижается к северу [34]. Это объяснялось усилением дрейфа в северных популяциях вследствие снижения продуктивности территорий и падения плотности населения. По мере возрастания географической широты предполагалось также уменьшение эффективного размера популяций, увеличение степени изоляции субпопуляций, а также возрастание случайного инбридинга и генетического груза популяции [34]. Между тем результаты исследования изменчивости целых митохондриальных геномов показывают, что популяции севера и юга Восточной Европы не различаются по разнообразию гаплотипов мтДНК, однако нуклеотидное разнообразие, измеряемое по числу попарных нуклеотидных различий между митогеномами, действительно несколько выше на юге (табл. 2). Трудно судить, насколько это связано с инбридингом и генетическим грузом популяций. Однако исходя из результатов анализа динамики Ne, вполне возможно, что более высокое разнообразие мтДНК в популяциях юга Восточной Европы может быть связано с такими долговременными эффектами, как более длительное присутствие популяций человека на юге Европы (например, в рефугиумах в ледниковое время) и более раннее начало демографических экспансий – еще в неолитическое время, что могло быть связано с распространением носителей технологий агрокультуры, вышедших из Анатолии. В популяциях севера Восточной Европы выявляется только экспансия эпохи бронзового века, связанная, возможно, с миграциями носителей курганной культуры. В популяциях юга сигналы этой экспансии также присутствуют – и даже в более выраженной форме, поскольку рост численности населения на юге Восточной Европы в эпоху бронзы в несколько раз превышал таковой на севере (по результатам анализа графиков на рис. 1 и 2).
Таблица 2.
Генетическое разнообразие в популяциях севера и юга Восточной Европы по данным об изменчивости целых митохондриальных геномов
| Популяция | N | k | Hd (s.e.) | π (s.e.) | mpd |
|---|---|---|---|---|---|
| Север | 246 | 225 | 0.999 (0.0005) | 0.00166 (0.00005) | 27.55 |
| Юг | 315 | 311 | 0.999 (0.0003) | 0.0018 (0.00006) | 29.7 |
Примечание. Обозначения как в табл. 1. В региональную группу популяций “Север” вошли русские Псковской и Новгородской областей и эстонцы; в группу “Юг” – русские Белгородской, Орловской и Тульской областей и украинцы.
Межпопуляционные различия по данным об изменчивости целых митогеномов
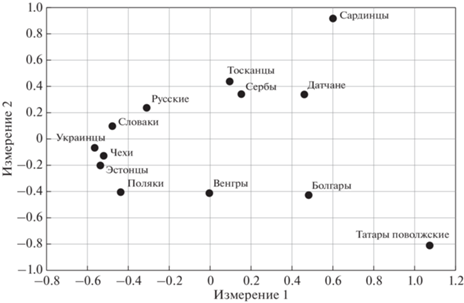
Все изученные европейские популяции характеризуются достаточно высоким уровнем разнообразия гаплотипов мтДНК, однако, как показало исследование F-статистик, межпопуляционные различия, основанные на анализе попарных нуклеотидных различий между гаплотипами мтДНК, очень низки (FST = 0.36%, p < 0.00001). По результатам этого анализа украинцы проявляют наибольшее сходство со словаками, русскими, поляками, сербами, эстонцами (значения FST изменяются в диапазоне от 0 до 0.1%), а максимальные отличия демонстрируют поволжские татары и сардинцы (примерно 1.3%) (табл. 3). Последние популяции в целом занимают наиболее удаленное положение (по результатам многомерного шкалирования FST-различий) как друг от друга, так и от остальных европейских популяций, проанализированных в настоящей работе (рис. 3). В случае сардинцев это может объясняться их изолированным островным положением и сохранением некоторых генетических особенностей еще с эпохи позднего неолита [35], а генофонд поволжских татар имеет другую особенность – наличие митохондриальных линий восточноазиатского происхождения [26]. Важно отметить, что на графике многомерного шкалирования украинцы располагаются вместе с западными и восточными славянами, занимая центральное положение между поляками, чехами, словаками и русскими, а также эстонцами. Между тем южные славяне (сербы и болгары) удалены от этой группы популяций, а также друг от друга. При этом сербы проявляют большее генетическое сходство с южными европейцами, а болгары – с восточными европейцами.
Таблица 3.
FST-различия (в %) между европейскими популяциями по данным об изменчивости нуклеотидных последовательностей целых митогеномов
| Популяция | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1. Русские | 0 | + | + | – | – | – | + | – | + | + | – | – | – |
| 2. Татары | 1.68 | 0 | + | + | + | + | + | + | + | + | + | + | + |
| 3. Сардинцы | 1.109 | 3.746 | 0 | + | + | + | + | + | + | + | + | + | + |
| 4. Эстонцы | 0 | 1.024 | 1.037 | 0 | – | – | – | – | – | + | – | – | – |
| 5. Тосканцы | 0.155 | 1.93 | 0.775 | 0.113 | 0 | – | – | – | – | – | – | – | – |
| 6. Венгры | 0.452 | 1.052 | 1.577 | 0.214 | 0.077 | 0 | – | – | – | – | – | – | – |
| 7. Сербы | 0.398 | 1.868 | 1.372 | 0.157 | 0.264 | 0.227 | 0 | + | + | + | – | – | – |
| 8. Поляки | 0.005 | 0.83 | 1.171 | 0 | 0.104 | 0.122 | 0.309 | 0 | – | + | – | – | – |
| 9. Болгары | 0.371 | 0.931 | 1.991 | 0.29 | 0.236 | 0 | 0.67 | 0.036 | 0 | + | – | + | – |
| 10. Датчане | 0.274 | 2.097 | 1.567 | 0.547 | 0.19 | 0.076 | 0.48 | 0.319 | 0.395 | 0 | – | + | + |
| 11. Чехи | 0.04 | 1.337 | 1.251 | 0.076 | 0.182 | 0 | 0.262 | 0.018 | 0.21 | 0 | 0 | – | – |
| 12. Словаки | 0.047 | 1.447 | 1.138 | 0 | 0.359 | 0.18 | 0.112 | 0.115 | 0.544 | 0.427 | 0.009 | 0 | – |
| 13. Украинцы | 0.047 | 1.283 | 1.318 | 0.077 | 0.306 | 0.149 | 0.068 | 0.05 | 0.369 | 0.404 | 0.125 | 0 | 0 |
Филогеографический анализ полиморфизма мтДНК
Митохондриальный генофонд исследованной выборки украинцев представлен преимущественно (97.2%) гаплогруппами западноевразийского происхождения: гаплогруппами H (42.2%), HV (2.1%), V (4.9%), J (9.7%), T (7.6%), K (3.5%), U2e (2.8%), U3 (0.7%), U4 (4.9%), U5 (6.9%), I (5.6%), W (2.1%), X (4.2%). Восточноазиатские гаплогруппы C, D и Z присутствуют в выборке с суммарной частотой 2.8%. Следует отметить, что восточноазиатский компонент был выявлен со сходными частотами и в других исследованиях полиморфизма мтДНК у украинцев (1.6% в работе [12], 2.5% в работе [11]). Структура западноевразийского компонента у украинцев очень сложная и представлена не менее 100 подгруппами различных гаплогрупп мтДНК (табл. 1 дополнительных материалов).
Результаты филогеографического анализа показывают, что митохондриальные гаплотипы, выявленные у украинцев, относятся к подгруппам мтДНК, распространенным у представителей различных народов Европы. В анализе были использованы более 52 000 митохондриальных геномов от представителей различных этнических групп мира. Исследование показало, что 16% украинских гаплотипов мтДНК идентичны таковым в других европейских популяциях – в основном, славянских и германоязычных. На долю этноспецифичных гаплотипов мтДНК приходится всего лишь 3.5% украинских образцов (подруппы H10a1b и U4c1a-2905-16355). Гораздо выше частота славяно-специфичных гаплотипов мтДНК – 23.6% (табл. 2 дополнительных материалов). Результаты молекулярного датирования показывают, что возраст такого рода подгрупп мтДНК у украинцев изменяется в широком диапазоне – от 1.3 до 6.6 тыс. лет. В среднем эволюционный возраст этого генетического компонента составляет 2.7 ± 0.2 тыс. лет.
Анализ опубликованных данных об изменчивости мтДНК у славян показывает, что частота этноспецифичных гаплотипов мтДНК достаточно широко изменяется в различных этнических группах – от 1.3% у словаков до 10.3% у поляков (табл. 4). Доля гаплотипов, обнаруженных, главным образом, только у славян, также варьирует: меньше всего славяно-специфичных гаплотипов мтДНК обнаружено у чехов и сербов, а больше всего – у украинцев. Насколько этот результат проясняет вопрос о происхождении славян, пока непонятно, поскольку для получения более надежных результатов необходимо больше данных об изменчивости мтДНК на полногеномном уровне как в различных славянских группах, так и у их соседей. А в настоящее время популяции особенно восточной части Европы исследованы очень фрагментарно.
Таблица 4.
Популяционная специфичность гаплотипов мтДНК в различных этнических группах славян
| Популяция | N | Популяционная специфичность (%) | |
|---|---|---|---|
| на этническом уровне | на уровне славянской общности | ||
| Украинцы | 144 | 3.5 | 23.6 |
| Русские | 466 | 9.7 | 12.2 |
| Поляки | 815 | 10.3 | 15.6 |
| Чехи | 150 | 5.3 | 9.3 |
| Словаки | 139 | 1.3 | 11.5 |
| Сербы | 226 | 10.2 | 8.0 |
В целом результаты исследований показывают, что лишь в относительно изолированных популяциях фракция этноспецифичных гаплотипов мтДНК может достигать больших объемов. Так, одно из первых исследований в этом направлении продемонстрировало, например, что у населения Сардинии примерно 80% митогеномов (а всего проанализировано около 3.5 тыс. митогеномов) формируют специфичные для сардинцев подгруппы мтДНК, возникшие в разное время на острове на основе различных предковых линий мтДНК [35]. Анализ полиморфизма 843 митогеномов финнов, история формирования которых также сопровождалась периодами изоляции, позволил установить, что 33% митогеномов финнов относятся к финно-специфичным подгруппам мтДНК [36]. Аналогично у басков – еще одного европейского народа-изолята обнаружена высокая частота (44%) специфичных для басков гаплотипов мтДНК [37]. Примерно треть (27%) митохондриального генофонда приходится на этноспецифичные подгруппы мтДНК и у армян, у которых широко распространены эндогамные браки [38].
Проведенное нами исследование показало, таким образом, что в славянских популяциях, формировавшихся в условиях смешения со своими соседями, частота этноспецифичных компонентов митохондриальных генофондов ожидаемо низка, однако фракция следующего иерархического уровня – общности славян – может достигать довольно высоких частот, как, например, у украинцев. Дополнительные исследования славяно-специфичных фракций гаплотипов мтДНК позволят в перспективе получить сведения о разнообразии и эволюционном возрасте собственно славянских компонентов в генофондах народов и особенностях динамики эффективной численности предковых популяций.
Все процедуры, выполненные в исследовании с участием людей, соответствуют этическим стандартам институционального и/или национального комитета по исследовательской этике и Хельсинкской декларации 1964 г. и ее последующим изменениям или сопоставимым нормам этики.
От каждого из включенных в исследование участников было получено информированное добровольное согласие.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Kivisild T. Maternal ancestry and population history from whole mitochondrial genomes // Investig. Genet. 2015. V. 6. e3. https://doi.org/10.1186/s13323-015-0022-2
Деренко М.В., Малярчук Б.А. Молекулярная филогеография населения Северной Евразии по данным об изменчивости митохондриальной ДНК. Магадан: СВНЦ ДВО РАН, 2010. 376 с.
Малярчук Б.А., Деренко М.В. Разнообразие и структура митохондриальных генофондов славян в этногенетическом аспекте // Успехи соврем. биологии. 2020. Т. 140. № 4. С. 333–346. https://doi.org/10.31857/S0042132420040109
Malyarchuk B., Litvinov A., Derenko M. et al. Mitogenomic diversity in Russians and Poles // Forensic Sci. Int. Genet. 2017. V. 30. P. 51–56. https://doi.org/10.1016/j.fsigen.2017.06.003
Skonieczna K., Malyarchuk B., Jawień A. et al. Mitogenomic differences between the normal and tumor cells of colorectal cancer patients // Hum. Mut. 2018. V. 39. P. 691–701. https://doi.org/10.1002/humu.23402
Piotrowska-Nowak A., Kosior-Jarecka E., Schab A. et al. Investigation of whole mitochondrial genome variation in normal tension glaucoma // Exp. Eye Res. 2019. V. 178. P. 186–197. https://doi.org/10.1016/j.exer.2018.10.004
Davidovic S., Malyarchuk B., Grzybowski T. et al. Complete mitogenome data for the Serbian population: The contribution to high quality forensic databases // Int. J. Legal Med. 2020. V. 134. P. 1581–1590. https://doi.org/10.1007/s00414-020-02324-x
Malyarchuk B.A., Derenko M.V. Mitochondrial DNA variability in Russians and Ukrainians: Implication to the origin of the Eastern Slavs // Ann. Hum. Genet. 2001. V. 65. P. 63–78. https://doi.org/10.1046/j.1469-1809.2001.6510063.x
Nikitin A.G., Kochkin I.T., June C.M. et al. Mitochondrial DNA sequence variation in the Boyko, Hutsul, and Lemko populations of the Carpathian highlands // Hum. Biol. 2009. V. 81. P. 43–58. https://doi.org/10.3378/027.081.0104
Балановский О.П., Пшеничнов А.С., Фролова С.А. и др. Основные черты митохондриального генофонда восточных славян // Мед. генетика. 2010. Т. 9. № 1. С. 29–37.
Mielnik-Sikorska M., Daca P., Malyarchuk B. et al. The history of Slavs inferred from complete mitochondrial genome sequences // PLoS One. 2013. V. 8 e54360. https://doi.org/10.1371/journal.pone.0054360
Pshenichnov A., Balanovsky O., Utevska O. et al. Genetic affinities of Ukrainians from the maternal perspective // Am. J. Phys. Anthropol. 2013. V. 152. P. 543–550. https://doi.org/10.1002/ajpa.22371
Kushniarevich A., Utevska O., Chuhryaeva M. et al. Genetic heritage of the Balto-Slavic speaking populations: A synthesis of autosomal, mitochondrial and Y-chromosomal data // PLoS One. 2015. V. 10. e0135820. https://doi.org/10.1371/journal.pone.0135820
Malyarchuk B., Grzybowski T., Derenko M. et al. Mitochondrial DNA phylogeny in Eastern and Western Slavs // Mol. Biol. Evol. 2008. V. 25. P. 1651–1658. https://doi.org/10.1093/molbev/msn114
Pala M., Olivieri A., Achilli A. et al. Mitochondrial DNA signals of late glacial recolonization of Europe from near eastern refugia // Am. J. Hum. Genet. 2012. V. 90. P. 915–924. https://doi.org/10.1016/j.ajhg.2012.04.003
Olivieri A., Pala M., Gandini F. et al. Mitogenomes from two uncommon haplogroups mark late glacial/postglacial expansions from the near east and neolithic dispersals within Europe // PLoS One. 2013. V. 8. e70492. https://doi.org/10.1371/journal.pone.0070492
Derenko M., Malyarchuk B., Denisova G. et al. Western Eurasian ancestry in modern Siberians based on mitogenomic data // BMC Evol. Biol. 2014. V. 14. 217. https://doi.org/10.1186/s12862-014-0217-9
Oleksyk T.K., Wolfsberger W.W., Weber A.M. et al. Genome diversity in Ukraine // Gigascience. 2021. V. 10. giaa159. https://doi.org/10.1093/gigascience/giaa159
Behar D.M., van Oven M., Rosset S. et al. A “Copernican” reassessment of the human mitochondrial DNA tree from its root // Am. J. Hum. Genet. 2012. V. 90. P. 675–684. https://doi.org/10.1016/j.ajhg.2012.03.002
Bianco E., Laval G., Font-Porterias N. et al. Recent common origin, reduced population size, and marked admixture have shaped European Roma genomes // Mol. Biol. Evol. 2020. V. 37. P. 3175–3187. https://doi.org/10.1093/molbev/msaa156
Starikovskaya E., Shalaurova S., Dryomov S. et al. Mitochondrial DNA variation of Leber’s hereditary optic neuropathy in Western Siberia // Cells. 2019. V. 8. 1574. https://doi.org/10.3390/cells8121574
Soares P., Ermini L., Thomson N. et al. Correcting for purifying selection: An improved human mitochondrial molecular clock // Am. J. Hum. Genet. 2009. V. 84. P. 740–759. https://doi.org/10.1016/j.ajhg.2009.05.001
Drummond A.J., Suchard M.A., Xie D. et al. Bayesian phylogenetics with BEAUti and the BEAST 1.7 // Mol. Biol. Evol. 2012. V. 29. P. 1969–1973. https://doi.org/10.1093/molbev/mss075
Tamura K., Peterson D., Peterson N. et al. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods // Mol. Biol. Evol. 2011. V. 28. P. 2731–2739. https://doi.org/10.1093/molbev/msr121
Soares P.A., Trejaut J.A., Rito T. et al. Resolving the ancestry of Austronesian-speaking populations // Hum. Genet. 2016. V. 135. P. 309–326. https://doi.org/10.1007/s00439-015-1620-z
Malyarchuk B., Derenko M., Denisova G., Kravtsova O. Mitogenomic diversity in Tatars from the Volga-Ural region of Russia // Mol. Biol. Evol. 2010. V. 27. P. 2220–2226. https://doi.org/10.1093/molbev/msq065
Stoljarova M., King J.L., Takahashi M. et al. Whole mitochondrial genome genetic diversity in an Estonian population sample // Int. J. Legal Med. 2016. V. 130. P. 67–71. https://doi.org/10.1007/s00414-015-1249-4
Malyarchuk B., Derenko M., Denisova G. et al. Whole mitochondrial genome diversity in two Hungarian populations // Mol. Genet. Genomics. 2018. V. 293. P. 1255–1263. https://doi.org/10.1007/s00438-018-1458-x
Raule N., Sevini F., Li S. et al. The co-occurrence of mtDNA mutations on different oxidative phosphorylation subunits, not detected by haplogroup analysis, affects human longevity and is population specific // Aging Cell. 2014. V. 13. P. 401–407. https://doi.org/10.1111/acel.12186
Fraumene C., Belle E.M., Castri L. et al. High resolution analysis and phylogenetic network construction using complete mtDNA sequences in Sardinian genetic isolates // Mol. Biol. Evol. 2006. V. 23. P. 2101–2111. https://doi.org/10.1093/molbev/msl084
Librado P., Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data // Bioinformatics. 2009. V. 25. P. 1451–1452. https://doi.org/10.1093/bioinformatics/btp187
Excoffier L., Lischer H.E. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Resour. 2010. V. 10. P. 564–567. https://doi.org/10.1111/j.1755-0998.2010.02847.x
Малярчук Б.А., Литвинов А.Н., Деренко М.В. Структура и формирование митохондриального генофонда русского населения Восточной Европы // Генетика. 2019. Т. 55. № 5. С. 574–582. https://doi.org/10.1134/S0016675819050102
Балановский О.П., Кошель С.М., Запорожченко В.В. и др. Эколого-генетический мониторинг в популяциях человека: гетерозиготность, гаплотипическое разнообразие мтДНК и генетический груз // Генетика. 2011. Т. 47. № 11. С. 1523–1535.
Olivieri A., Sidore C., Achilli A. et al. Mitogenome diversity in Sardinians: A genetic window onto an Island’s past // Mol. Biol. Evol. 2017. V. 34. P. 1230–1239. https://doi.org/10.1093/molbev/msx082
Översti S., Onkamo P., Stoljarova M. et al. Identification and analysis of mtDNA genomes attributed to Finns reveal long-stagnant demographic trends obscured in the total diversity // Sci. Rep. 2017. V. 7. 6193. https://doi.org/10.1038/s41598-017-05673-7
García Ó., Alonso S., Huber N. et al. Forensically relevant phylogeographic evaluation of mitogenome variation in the Basque Country // Forensic Sci. Int. Genet. 2020. V. 46. 102260. https://doi.org/10.1016/j.fsigen.2020.102260
Derenko M., Denisova G., Malyarchuk B. et al. Insights into matrilineal genetic structure, differentiation and ancestry of Armenians based on complete mitogenome data // Mol. Genet. Genomics. 2019. V. 294. P. 1547–1559. https://doi.org/10.1007/s00438-019-01596-2
Дополнительные материалы
- скачать ESM.xlsx
- Приложение 1.
Таблица 1 Дополнительных материалов. Филогенетическая идентификация митохондриальных геномов украинцев и эволюционный.
Таблица 2 Дополнительных материалов. Славяно-специфичные гаплогруппы мтДНК, выявленные в генофонде украинцев.