Генетика, 2023, T. 59, № 3, стр. 308-315
Сайты связывания архитектурного белка Su(Hw) стимулируют рекрутирование эпигенетических регуляторов PcG/TrxG на хроматин: CRISPR/Cas9-тест
М. М. Ерохин 1, *, Ф. В. Горбенко 1, Д. В. Ломаев 1, Д. А. Четверина 1, **
1 Институт биологии гена Российской академии наук
119334 Москва, Россия
* E-mail: yermaxbio@yandex.ru
** E-mail: daria.chetverina@gmail.com
Поступила в редакцию 17.04.2022
После доработки 29.05.2022
Принята к публикации 09.06.2022
- EDN: INYMCX
- DOI: 10.31857/S0016675823030049
Аннотация
Белки групп Polycomb и Trithorax (PcG и TrxG) являются эпигенетическими факторами, отвечающими за репрессию и активацию транскрипции соответственно. У Drosophila белки PcG/TrxG рекрутируются на специализированные ДНК-элементы, называемые PRE (Polycomb response elements). В зависимости от контекста, данные элементы могут репрессировать, активировать или быть нейтральными по отношению к промотору гена-мишени. Ранее в трансгенных исследованиях с использованием PhiC31 сайт-специфической системы интеграции мы продемонстрировали, что расположенные рядом с PRE сайты для архитектурных белков могут индуцировать репрессорную активность bxdPRE, стимулируя связывание факторов PcG/TrxG с данным элементом. Однако этот эффект может зависеть от дополнительных ДНК-элементов, присутствующих в месте интеграции после PhiC31-зависимой инсерции трансгенов. В настоящем исследовании, используя альтернативную систему интеграции на основе CRISPR/Cas9-катализируемой гомологичной репарации, мы доказали, что сайты связывания архитектурного белка Su(Hw) действительно способны индуцировать репрессорную активность bxdPRE и привлечение белков PcG/TrxG вне зависимости от дополнительных ДНК-элементов, присутствующих в месте интеграции после PhiC31-зависимой инсерции трансгенов.
Эпигенетический контроль активности генов является необходимым для дифференцировки клеток и поддержания правильных паттернов экспрессии на взрослой стадии у многоклеточных организмов. Ключевые регуляторы данного процесса – белки групп Polycomb и Trithorax – являются репрессорами и активаторами транскрипции соответственно [1–3]. Нарушение работы данных факторов приводит к различным патологиям, включая онкологические заболевания, что определяет необходимость исследований, направленных на изучение механизма их действия [4–8].
Одним из нерешенных на сегодняшний день вопросов, является понимание механизма рекрутирования белков PcG/TrxG на хроматин. Репрессоры PcG представлены тремя основными белковыми комплексами – PRC1, PRC2 и PhoRC, тогда как активаторы TrxG более разнообразны и включают метилтрансферазные комплексы COMPASS и COMPASS-like, ремоделеры хроматина SWI/SNF и другие (подробнее см. [4]). У Drosophila белки PcG/TrxG привлекаются на специализированные ДНК-регуляторные элементы, названные Polycomb response elements (PRE) [9, 10]. Многочисленные эксперименты с трансгенными конструкциями, интегрированными в геном, показали, что PRE могут подавлять экспрессию репортерных генов и поддерживать репрессию во время развития.
Характерной чертой PRE-зависимой репрессии является эффект, названный pairing-sensitive silencing (PSS), который заключается в том, что репрессия становится намного сильнее у мух, гомозиготных по трансгену [11, 12]. Считается, что усиление репрессии у гомозигот связано с транс-взаимодействиями между двумя копиями PRE, находящихся на гомологичных хромосомах. PSS-эффект также наблюдался для PREs в их эндогенном контексте [13].
Кроме того показано, что активность PRE-элементов в составе трансгенов сильно зависит от окружающего хроматина. В зависимости от сайта интеграции PREs могут репрессировать или активировать транскрипцию репортерных генов, либо же быть нейтральным элементом, не оказывая влияния на экспрессию гена [11, 14–17]. Это указывает на то, что активность PRE модулируется хромосомным контекстом. Однако детали данного процесса и факторы влияния на активность PRE практически не изучены.
Ранее мы показали, что сайты связывания для архитектурных белков, расположенные рядом с PRE-элементом, могут усиливать PRE-опосредованную репрессию и, более того, индуцировать репрессионную активность неактивного PRE [16]. В частности, данный эффект был показан на модельной системе, содержащей четыре сайта связывания для архитектурного белка Su(Hw), PRE-элемента bxdPRE из регуляторной области гена Ubx, репортерный ген white и attB-сайт. Сайт attB необходим для интеграции конструкции в геном с помощью направленной сайт-специфической рекомбинации PhiC31 [18]. При PhiC31-опосредуемой интеграции происходит рекомбинация между сайтом attB трансгена и встроенным в определенное место генома сайтом attP. При этом при встраивании происходит интеграция как тестируемых последовательностей ДНК, так и дополнительных последовательностей, необходимых для наработки плазмиды в бактериях. Кроме того, в геноме рядом с трансгеном присутствуют фрагменты трансгенного элемента Mariner, за счет которого был интегрирован сайт attP [18]. Таким образом, способность сайтов связывания архитектурных белков активировать репрессионную активность PRE потенциально может зависеть от участков ДНК, необходимых для PhiC31-зависимой интеграции конструкции в геном.
Настоящее исследование направлено на проверку способности сайтов связывания архитектурных белков стимулировать активность PRE независимо других ДНК-регуляторных элементов. Для этого была проведена CRISPR/Cas9-зависимая инсерция только внутренних элементов конструкции в геномную область без последовательностей элемента Mariner и attP. В результате анализа было показано, что стимулирование репрессионной активности bxdPRE сайтами Su(Hw) сохраняется при CRISPR/Cas9-зависимой интеграции конструкции. Более того, репрессия сопровождается рекрутированием ДНК-связывающего фактора PRE – Combgap, и белками PcG/TrxG на PRE. Таким образом, стимуляция PRE-опосредованной репрессионной активности bxdPRE сайтами Su(Hw) не зависит от присутствующих в геноме после PhiC31-зависимой интеграции элементов.
МАТЕРИАЛЫ И МЕТОДЫ
Для интеграции трансгенной конструкции в геном (область 96E на третьей хромосоме) мы использовали систему гомологичной рекомбинации, индуцируемой созданием двуцепочечных разрывов с помощью CRISPR/Cas9 системы [19]. Для этого были сделаны следующие плазмиды: 1) CRISPR-96E – плазмида, экспрессирующая гидовую РНК (gRNA) к геномной области 96E; 2) HR-Su(Hw)-bxd – конструкция, содержащая исследуемые ДНК-регуляторные элементы и участки для гомологичной рекомбинации.
Создание плазмиды 96E-CRISPR
Для выбора оптимальных сайтов-мишеней gRNA к геномной области 96E использовалась онлайн-программа поиска мишеней flyCRISPR (https://flycrispr.org/) [20]. Для более эффективной интеграции были выбраны две пары gRNA: первая пара – 5'-96E-CRISPR1 – 5'-aaagcagcatggataccacc-3' и 3'-96E-CRISPR1 – 5'-aagtatgtcgcaaaacggct-3'; вторая пара – 5'-96E-CRISPR2 – 5'-accacctggcccattggatt-3' и 3'-96E-CRISPR2 – 5'-atggataccacctggcccat-3'. 5'- и 3'-96E-CRISPR были одновременно с помощью ПЦР вставлены в вектор для двойной экспрессии CRISPRs – prAc_yiless_U6:1:3_attB [21], под контроль промоторов U6:1 и U6:3 соответственно (см. далее рис. 1). Вектор prAc_yiless_U6:1:3_attB [21] основан на плазмиде pCFD4-U6:1_U6:3tandemgRNAs (Addgene # 49411 [19]) с дополнительной вставкой гена yellow-intron-less-attB под контролем промотора Actin5C. Ген yellow и attB в данной работе не использовались, в плазмиде они присутствуют для возможности стабильной интеграции плазмид с gRNA в геном.
Рис. 1.
Дизайн эксперимента. а – схема создания векторов для экспрессии гидовых РНК (gRNA), подробное описание см. в тексте. б – область 96E, использованная для встройки полученной конструкции. Вертикальной стрелкой указано место после окончания транскрипции гена LpR2, в которое производилась интеграция трансгена. Справа и слева – области, использованные в качестве плеч гомологий. в – схемы конструкций bxd, и Su-bxd и HR-Su-bxd. * – из [16]. Обозначения: “attB” – сайт attB, необходимый для PhiC31-зависимой интеграции конструкции в геном; “bxdPRE” – сайленсер bxdPRE; “4xSu” – четыре сайта связывания белка Su(Hw); white – репортерный ген; “Е” – энхансер гена white; “Т” – терминаторы транскрипции; 5'-96E-HA и 3'-96E-HA – плечи гомологии, необходимые для CRISPR/Cas9-зависимой интеграции конструкции в геном. Внутренние функциональные элементы конструкции HR-Su-bxd полностью повторяют элементы конструкции Su-bxd, но четыре сайта связывания белка Su(Hw) (4xSu) фланкированы сайтами lox (указаны стрелками снизу), для их делеции in vivo и получения аналога конструкции bxd.

Для одновременного клонирования 5'- и 3'-96E-CRISPR были подобраны прямой и обратный праймеры, которые также содержат гомологию с вектором prAc_yiless_U6:1:3_attB для ПЦР-амплификации с данного участка. Праймеры для первой пары для создания плазмиды 96E-CRISPR1: 5'-gagaagacctTTCGgAAAGCAGCATGGATACCACCGTTTTAGAGCTAGAAATAGCAAG-3' и 5'-gagaagacctAAACAGCCGTTTTGCGACATACTTcGACGTTAAATTGAAAATAGGTC-3'. Праймеры для второй пары для создания плазмиды 96E-CRISPR2: 5'-gagaagacctTTCGgACCACCTGGCCCATTGGATTGTTTTAGAGCTAGAAATAGCAAG-3' и 5'-gagaagacctAAACATGGGCCAGGTGGTATCCATcGACGTTAAATTGAAAATAGGTC-3'.
Кроме gRNA, праймеры содержат сайты для эндонуклеазы рестрикции BbsI и спейсеры, необходимые для клонирования. BbsI распознает 5'-GAAGACN2↓-3'/3'-CTTCTGN6↑-5', присутствующий в 5'-части праймера. Сайты для BbsI также присутствуют в векторе prAc_yiless_U6:1:3_attB. Продукты ПЦР, обработанные эндонуклеазой рестрикции BbsI, были вставлены в вектор prAc_yiless_U6:1:3_attB, расщепленный по BbsI. Правильное лигирование последовательностей в результирующих векторах 96E-CRISPR1 и 96E-CRISPR2 было подтверждено секвенированием.
Создание плазмиды HR-Su-bxd
Для создания плазмиды HR-Su-bxd BamHI-ApaI фрагмент 4xSu из плазмиды lox(Sx4) [22] был встроен в вектор bxd [16], обработанный сайтами эндонуклеаз рестрикции SpeI и XhoI (конструкция Su-lox-bxd). Плечи гомологии области 96E, окружающие сайты-мишени 96E-CRISPR, были амплифицированы методом ПЦР, используя в качестве матрицы геномную ДНК D. melanogaster дикого типа и следующие праймеры:
1) 5'-GGTACCCTAACCTTGTGTGCATGT-3' и 5'-GGTACCACCAAATGGCAAAACACT-3' для плеча гомологии 5'-96E-HA с сайтами для эндонуклеазы рекстрикции KpnI (итоговая длина области гомологии 1170 пн),
2) 5'-CCGCGGGGATCATATCTAAATTGA-3' и 5'-CCGCGGTGAAACAAAATATTAGCT-3' для плеча гомологии 3'-96E-HA с сайтами для эндонуклеазы рекстрикции SacII (итоговая длина области гомологии 980 пн).
Для получения итоговой конструкции HR-Su-bxd полученные фрагменты плечей гомологии 5'- и 3'-96E-HA последовательно вставляли в плазмиду Su-lox-bxd. ПЦР-фрагмент, содержащий плечо 5'-96E-HA, был встроен по сайту KpnI. ПЦР-фрагмент, содержащий плечо 3'-96E-HA, был встроен по сайту SacII, в результате чего удалялась функциональная область участка attB.
Инъекция эмбрионов и получение трансгенных линий
Смесь плазмид (10 : 1 – HR-Su-bxd : 96E-CRISPR, общая концентрация 500 нг/мкл) вводили в эмбрионы линии #58492 Bloomington Drosophila Stock Center, экспрессирующие ген Cas9 под контролем промотора Actin5C. Инъецированные эмбрионы выращивали до зрелого возраста и скрещивали с мухами линии y-w1118. Мух с встройками конструкции HR-Su-bxd идентифицировали по цвету глаз за счет экспрессии гена white. Найденных трансформантов скрещивали с линией, несущей балансер TM3,Sb на третьей хромосоме и в дальнейшем переводили в гомозиготное состояние по трансгену.
Правильность гомологичной рекомбинации была подтверждена с помощью ПЦР с использованием ДНК, выделенной из трансгенных мух. Для получения производной линии HR-bxd с делецией сайтов связывания архитектурного белка Su(Hw), мух с конструкцией HR-Su-bxd скрещивали с линией y1w1; Cyo, P(w+,cre)/Sco; +, экспрессирующей Cre-рекомбиназу [23]. Фенотипы трансгенных линий мух HR-Su-bxd и HR-bxd анализировали в геми- (P/+) и гомозиготном (P/P) состояниях. Всех мух содержали при 25°С на стандартной среде.
Иммунопреципитация хроматина (X-ChIP)
Метод иммунопреципитации хроматина проводился как описано в [16]. Хроматин был выделен из голов взрослых мух (150–200 мг) и иммунопреципитирован с помощью специфичных либо неспецифичных антител. В качестве специфичных антител были использованы ранее полученные кроличьи поликлональные антитела к белкам Su(Hw) (C-конец) [24], Ph, Sfmbt, Trx-N [25, 26], Combgap [16]. В качестве неспецифичных антител были использованы IgG не иммунизированного животного. Анализ обогащения был проведен с помощью ПЦР в реальном времени (qPCR) в присутствии SYBR Green с использованием следующих пар праймеров: специфичные к трансгену пары праймеров точка 1 (5'-CCGACCACTACCAGCAGAAC-3' и 5'-GTCCATGCCGAGAGTGATCC-3'), точка 2 (5'-AGCTCCTCGCCCTTGCTCACCAT-3' и 5'-CCATAATGGCTGCGCCGTAAAG-3'), точка 3 (5'-GCCACTACGACGCTGAGGTCAA-3' и 5'-CGTTGTGGGAGGTGATGTCC-3'). В качестве отрицательного контроля использовалась пара к кодирующей области гена Ras64B (5'-GAGGGATTCCTGCTCGTCTTCG-3' и 5'-GTCGCACTTGTTACCCACCATC-3'). В качестве положительного контроля использовались пары к области 62D (область высокого обогащения белка Su(Hw), 5'-GAGGGATTCCTGCTCGTCTTCG-3' и 5'-GTCGCACTTGTTACCCACCATC-3') и к области, соседствующей с bxdPRE в геноме, но не входящей в состав трансгена (участок высокого обогащения PcG/TrxG-факторов, 5'-AAGAGCAAGGCGAAAGAGAGC-3' и 5'-CGTTTTAAGTGCGACTGAGATGG-3').
РЕЗУЛЬТАТЫ
Для интеграции трансгенной конструкции, содержащей сайты связывания архитектурного белка Su(Hw), bxdPRE и гена white, была выбрана область 96E (рис. 1). Согласно ранее полученным нами данным [16], в этой области трансгенный bxdPRE в отсутствие сайтов связывания архитектурных белков (Su(Hw), CTCF либо Pita) находится в нейтральном статусе – не репрессирует транскрипцию и не связывает белки групп PсG и TrxG. В то же время в присутствии сайтов связывания для архитектурных белков Su(Hw), CTCF либо Pita происходила индукция репрессии трансгенного гена white, которая усиливалась в гомозиготе (эффект PSS). Сайты связывания архитектурных белков обладают инсуляторной активностью – в частности, способностью блокировать PRE-опосредованную репрессию гена мишени. Поэтому в данной системе PRE находится между сайтами связывания архитектурных белков и промотором гена white. В таком положении PRE не блокируется сайтами связывания архитектурных белков и способен репрессировать ген white.
Фенотипический анализ трансгенной линии HR-Su-bxd и ее производной HR-bxd с делецией сайтов Su(Hw) (см. Материалы и методы) проводился в сравнении с ранее полученными трансгенами Su-bxd и bxd ([16] и рис. 2). В результате было показано, что фенотипы линий HR-Su-bxd и HR-bxd полностью совпадают с фенотипами линий Su-bxd и bxd соответственно. В отсутствие сайтов связывания для архитектурного белка Su(Hw), окраска глаз в геми- и гомозиготе красная, что свидетельствует о неспособности bxdPRE репрессировать маркерный ген white. В то же время, в присутствии сайтов Su(Hw), окраска глаз гораздо светлее как у гемизигот (коричневый), так и у гомозигот (светло-желтый). Это свидетельствует о репрессии гена white. Более сильная репрессия в гомозиготном состоянии согласуется с эффектом PSS, характерным для PRE-элементов. Таким образом, сайты связывания для архитектурного белка Su(Hw) индуцируют репрессию, опосредованную сайленсером bxdPRE. Кроме того, для индукции активности PRE сайтами Su(Hw) не требуется наличия присутствующих после PhiC31-зависимой интеграции конструкции в геноме элементов, так как при CRISPR/Cas9-зависимой инсерции происходит встраивание только внутренних элементов конструкции.
Рис. 2.
Сайты связывания для архитектурного белка Su(Hw) индуцируют репрессионную активность bxdPRE вне зависимости от использованной системы интеграции трансгена. а – общая схема интеграции конструкции HR-Su-bxd в область 96E с использованием системы CRISPR/Cas9. Последующая Cre-зависмая рекомбинация приводит к удалению сайтов связывания белка Su(Hw) (4xSu) и получению производной линии HR-bxd. б – фенотипы мух трансгенных линий HR-bxd, HR-Su-bxd, Su-bxd, bxd. В конструкциях HR-bxd и bxd, в отсутствие сайтов связывания для Su(Hw) рядом с bxdPRE, окраска глаз в геми- (P/+) и гомозиготе (P/P) R – красная, что свидетельствует об отсутствии (–) репрессии. В конструкциях HR-Su-bxd и Su-bxd, в присутствии 4xSu рядом с bxdPRE, окраска глаз в геми- (P/+, Br – коричневая) и гомозиготе (P/P, pY – светло-желтая), что свидетельствует о репрессии в гемизиготе (+), усиливающейся в гомозиготе (++).

На следующем этапе методом X-ChIP с последующей количественной ПЦР (qPCR) мы проверили состав белковых комплексов, рекрутирующихся на элемент bxdPRE в присутствии и в отсутствие сайтов связывания для белка Su(Hw). Сначала мы протестировали связывание белка Su(Hw) (рис. 3,а). В полном соответствии с ожиданиями Su(Hw) связывается со своими сайтами в созданной трансгенной конструкции. При этом делеция сайтов связывания для Su(Hw) приводит к потере его ассоциации в области трансгена с bxdPRE.
Рис. 3.
Рекрутирование белков PcG/TrxG на bxdPRE в полученных трансгенах зависит от сайтов связывания для архитектурного белка Su(Hw). Результаты X-ChIP-анализа с использованием антител к белкам Su(Hw) (а), Ph (б), Sfmbt (в), Combgap (г), Trx (д). Цифры на оси абсцисс 1, 2, 3 обозначают ампликоны, использованные в количественной ПЦР (qPCR) и указаны на схеме конструкций на рис. 2 соответствующими цифрами. Ось ординат – обогащения при иммунопреципитации хроматина показаны в виде доли от исходного образца (Input) с нормировкой на эндогенный позитивный контроль – область эффективного связывания Su(Hw) 62D (а) или рядом с bxdPRE в геноме (б–д). Кодирующая область гена Ras64B использовалась как отрицательный контроль (ras). На диаграммах черные столбцы обозначают обогащение для линии HR-bxd, заштрихованные столбцы – для линии HR-Su-bxd, серые столбцы – обогащение, полученное при использовании неспецифических антител (IgG). Вертикальными отрезками указаны стандартные отклонения.
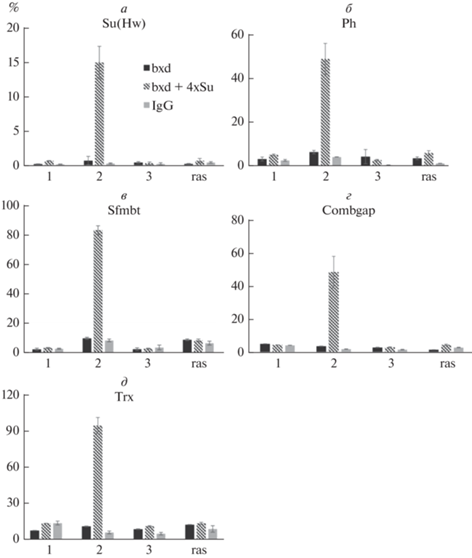
Далее было исследовано связывание репрессоров группы Polycomb – факторов Ph (субъединица комплекса PRC1) и Sfmbt (субъединица комплекса PhoRC). В результате X-ChIP-анализа было установлено, что данные факторы активно рекрутируются на область bxdPRE в составе трансгена только в случае наличия рядом расположенных сайтов связывания для Su(Hw) (рис. 3,б, в). Этот факт полностью согласуется с полученными фенотипическими данными по уровню экспрессии гена white. Важно, что PRE-ассоциированный ДНК-связывающий фактор Combgap [27] также рекрутируется на трансген-расположенный bxdPRE только в присутствие сайтов Su(Hw) (рис. 3,г). В полном соответствии с полученными нами ранее данными, стимуляция PRE-зависимой репрессии сайтами связывания Su(Hw) связана не только с привлечением репрессоров группы PcG, но также и белков семейства TrxG, в частности, белка Trithorax (рис. 3,д).
Таким образом, архитектурный белок Su(Hw) вне зависимости от системы интеграции способен стимулировать рекрутирование эпигенетических регуляторов PcG/TrxG на хроматин и переводить PRE-элемент из неактивного в активное состояние.
ОБСУЖДЕНИЕ
Белки PcG/TrxG являются ключевыми транскрипционными регуляторами, однако вопрос об их специфичном привлечении в строго определенные места генома остается открытым. В данных процессах большую роль, по крайней мере у Drosophila, отводят PRE-ДНК-связывающим факторам, которые взаимодействуют с белками PcG/TrxG и направленно рекрутируют данные комплексы на соответствующие хроматиновые мишени. Однако детали данных процессов не известны. Недавно нами было показано, что расположенные рядом с PRE-сайты связывания архитектурных белков Su(Hw), CTCF или Pita способны стимулировать, а в отдельных случаях и инициировать, привлечение PcG/TrxG факторов и, как следствие, усиливать или индуцировать Polycomb-зависимую репрессию гена-мишени. В качестве объяснения наблюдаемого феномена можно выдвинуть две гипотезы. Согласно первой, связывание архитектурных белков локально дестабилизирует взаимодействие нуклеосом с хроматином, что делает сайты связывания для PcG/TrxG-ассоциированных ДНК-связывающих белков более доступными. Согласно второй гипотезе, архитектурные белки способны создавать и поддерживать стабильные пространственные взаимодействия в ядре [28, 29], что способствует взаимодействую между PRE-элементами и, как следствие, приводит к усилению репрессии. Ранее нами была использована система интеграции PhiC31, в которой для встройки конструкции в геном использовались дополнительные ДНК-регуляторные элементы, потенциально способные так же, как и сайты связывания для архитектурных белков, оказывать влияние на рекрутирование PcG/TrxG факторов на PRE. В представленном исследовании мы протестировали возможную роль таких дополнительных последовательностей в стимуляции активности PRE-элемента. Для этого мы осуществили встройку трансгенной конструкции, содержащей все функциональные элементы ранее использованного вектора, в то же самое место генома (область 96E), но с применением технологии CRISPR/Cas9. В результате было установлено, что сайты связывания для архитектурного белка Su(Hw) вне зависимости от используемой системы интеграции способствуют привлечению как канонических комплексов PcG (PRC1, PhoRC), так и белка Trx из группы TrxG. Важно отметить, что привлечение ДНК-связывающего фактора Combgap также наблюдалось только в присутствии сайтов Su(Hw). Полученные результаты подтверждают, что в рекрутировании белков групп PcG/TrxG на хроматин важную регуляторную роль могут играть архитектурные факторы, такие как белок Su(Hw) у Drosophila.
Работа выполнена при поддержке гранта РНФ № 20-74-10099. В работе была использована инфраструктура Центра высокоточного редактирования и генетических технологий для биомедицины ИБГ РАН.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Chetverina D.A., Elizar’ev P.V., Lomaev D.V. et al. Control of the gene activity by Polycomb and Trithorax group proteins in Drosophila // Russian J. Genetics. 2017. V. 53. № 2. P. 133–154.
Kassis J.A., Kennison J.A., Tamkun J.W. Polycomb and Trithorax group genes in Drosophila // Genetics. 2017. V. 206. № 4. P. 1699–1725. https://doi.org/10.1534/genetics.115.185116
Schuettengruber B., Bourbon H.M., Di Croce L., Cavalli G. Genome regulation by Polycomb and Trithorax: 70 years and counting // Cell. 2017. V. 171. № 1. P. 34–57. https://doi.org/10.1016/j.cell.2017.08.002
Chetverina D.A., Lomaev D.V., Erokhin M.M. Polycomb and Trithorax group proteins: The long road from mutations in Drosophila to use in medicine // Acta Naturae. 2020. V. 12. № 4. P. 66–85. https://doi.org/10.32607/actanaturae.11090
Четверина Д.А., Ломаев Д.В., Георгиев П.Г., Ерохин М.М. Генетические нарушения активности PRC2 при онкологии: проблемы и перспективы // Генетика. 2021. Т. 57. № 3. С. 235–289. https://doi.org/10.31857/S0016675821030048
Erokhin M., Chetverina O., Gyorffy B. et al. Clinical correlations of Polycomb repressive complex 2 in different tumor types // Cancers (Basel). 2021. V. 13. № 13.https://doi.org/10.3390/cancers13133155
Piunti A., Shilatifard A. The roles of Polycomb repressive complexes in mammalian development and cancer // Nat. Rev. Mol. Cell. Biol. 2021. V. 22. № 5. P. 326–345. https://doi.org/10.1038/s41580-021-00341-1
Parreno V., Martinez A.M., Cavalli G. Mechanisms of Polycomb group protein function in cancer // Cell Res. 2022. V. 32. № 3. P. 231–253. https://doi.org/10.1038/s41422-021-00606-6
Erokhin M., Georgiev P., Chetverina D. Drosophila DNA-binding proteins in Polycomb repression // Epigenomes. 2018. V. 2. № 1. P. 1. https://doi.org/10.3390/epigenomes2010001
Kuroda M.I., Kang H., De S., Kassis J.A. Dynamic competition of Polycomb and Trithorax in transcriptional programming // Annu. Rev. Biochem. 2020. V. 89. P. 235–253. https://doi.org/10.1146/annurev-biochem-120219-103641
Kassis J.A. Unusual properties of regulatory DNA from the Drosophila engrailed gene: Three “pairing-sensitive” sites within a 1.6-kb region // Genetics. 1994. V. 136. № 3. P. 1025–1038. https://doi.org/10.1093/genetics/136.3.1025
Kassis J.A. Pairing-sensitive silencing, polycomb group response elements, and transposon homing in Drosophila // Adv. Genet. 2002. V. 46. P. 421–438. https://doi.org/10.1016/s0065-2660(02)46015-4
Mihaly J., Hogga I., Gausz J. et al. In situ dissection of the Fab-7 region of the bithorax complex into a chromatin domain boundary and a Polycomb-response element // Development. 1997. V. 124. № 9. P. 1809–1820. https://doi.org/10.1242/dev.124.9.1809
De S., Cheng Y., Sun M.A. et al. Structure and function of an ectopic Polycomb chromatin domain // Sci. Adv. 2019. V. 5. № 1. P. eaau9739. https://doi.org/10.1126/sciadv.aau9739
De S., Mitra A., Cheng Y. et al. Formation of a Polycomb-domain in the absence of strong Polycomb response elements // PLoS Genet. 2016. V. 12. № 7. P. e1006200. https://doi.org/10.1371/journal.pgen.1006200
Erokhin M., Gorbenko F., Lomaev D. et al. Boundaries potentiate Polycomb response element-mediated silencing // BMC Biol. 2021. V. 19. № 1. P. 113. https://doi.org/10.1186/s12915-021-01047-8
Horard B., Tatout C., Poux S., Pirrotta V. Structure of a Polycomb response element and in vitro binding of Polycomb group complexes containing GAGA factor // Mol. Cell. Biol. 2000. V. 20. № 9. P. 3187–3197. https://doi.org/10.1128/MCB.20.9.3187-3197.2000
Bischof J., Maeda R.K., Hediger M. et al. An optimized transgenesis system for Drosophila using germ-line-specific phiC31 integrases // Proc. Natl Acad. Sci. USA. 2007. V. 104. № 9. P. 3312–3317. https://doi.org/10.1073/pnas.0611511104
Port F., Chen H.M., Lee T., Bullock S.L. Optimized CRISPR/Cas tools for efficient germline and somatic genome engineering in Drosophila // Proc. Natl Acad. Sci. USA. 2014. V. 111. № 29. P. E2967–2976. https://doi.org/10.1073/pnas.1405500111
Gratz S.J., Ukken F.P., Rubinstein C.D. et al. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila // Genetics. 2014. V. 196. № 4. P. 961–971. https://doi.org/10.1534/genetics.113.160713
Zolotarev N., Maksimenko O., Kyrchanova O. et al. Opbp is a new architectural/insulator protein required for ribosomal gene expression // Nucl. Ac. Res. 2017. V. 45. № 21. P. 12285–12300. https://doi.org/10.1093/nar/gkx840
Erokhin M., Davydova A., Kyrchanova O. et al. Insulators form gene loops by interacting with promoters in Drosophila // Development. 2011. V. 138. № 18. P. 4097–4106. https://doi.org/10.1242/dev.062836
Siegal M.L., Hartl D.L. Application of Cre/loxP in Drosophila. Site-specific recombination and transgene coplacement // Methods Mol. Biol. 2000. V. 136. P. 487–495. https://doi.org/10.1385/1-59259-065-9:487
Melnikova L., Elizar’ev P., Erokhin M. et al. The same domain of Su(Hw) is required for enhancer blocking and direct promoter repression // Sci. Rep. 2019. V. 9. № 1. P. 5314. https://doi.org/10.1038/s41598-019-41761-6
Erokhin M., Davydova A., Parshikov A. et al. Transcription through enhancers suppresses their activity in Drosophila // Epigenetics Chromatin. 2013. V. 6. № 1. P. 31. https://doi.org/10.1186/1756-8935-6-31
Erokhin M., Elizar’ev P., Parshikov A. et al. Transcriptional read-through is not sufficient to induce an epigenetic switch in the silencing activity of Polycomb response elements // Proc. Natl Acad. Sci. USA. 2015. V. 112. № 48. P. 14930–14935. https://doi.org/10.1073/pnas.1515276112
Ray P., De S., Mitra A. et al. Combgap contributes to recruitment of Polycomb group proteins in Drosophila // Proc. Natl Acad. Sci. USA. 2016. V. 113. № 14. P. 3826–3831. https://doi.org/10.1073/pnas.1520926113
Chetverina D., Aoki T., Erokhin M. et al. Making connections: Insulators organize eukaryotic chromosomes into independent cis-regulatory networks // Bioessays. 2014. V. 36. № 2. P. 163–172. https://doi.org/10.1002/bies.201300125
Chetverina D., Fujioka M., Erokhin M. et al. Boundaries of loop domains (insulators): Determinants of chromosome form and function in multicellular eukaryotes // Bioessays. 2017. V. 39. № 3. https://doi.org/10.1002/bies.201600233
Дополнительные материалы отсутствуют.


