Генетика, 2023, T. 59, № 3, стр. 294-307
Генетическая предрасположенность к кетозу у крупного рогатого скота: современное состояние
О. В. Соколова 1, *, М. В. Бытов 1, А. И. Белоусов 1, Н. А. Безбородова 1, В. Д. Зубарева 1, Н. А. Мартынов 1, О. С. Зайцева 1, И. А. Шкуратова 1
1 Уральский федеральный аграрный научно-исследовательский центр
Уральского отделения Российской академии наук
620142 Екатеринбург, Россия
* E-mail: nauka_sokolova@mail.ru
Поступила в редакцию 27.04.2022
После доработки 27.06.2022
Принята к публикации 12.07.2022
- EDN: IQMSXQ
- DOI: 10.31857/S0016675823030116
Аннотация
Высокая молочная продуктивность коров связана с интенсивным функционированием всех органов и систем, что предрасполагает к развитию различных форм нарушений обменных процессов. Формирование энергетического дисбаланса у высокопродуктивных коров в лактирующий период способствует развитию комплексных метаболических нарушений, отрицательно влияющих на продуктивное здоровье и репродуктивный потенциал. Интерес к разведению крупного рогатого скота, более устойчивого к кетозу, является глобальным, а поиск мутаций, аллельных вариантов генов и изучение молекулярно-генетических процессов, формирующих тот или иной фенотип, являются ключевыми этапами в понимании этиологии, степени предрасположенности к заболеванию и разработке успешных селекционных программ. В настоящем обзоре представлены результаты исследований, направленных на поиск генетических маркеров развития кетоза крупного рогатого скота на основе молекулярно-генетических методов. В обзоре представлены локализация SNPs по данным метаанализа GWAS, протеин–протеин взаимодействия ассоциированных с ними генов-кандидатов при помощи STRING, а также аннотация SNPs по ключевым биологическим процессам с их участием. Приведен профиль экспрессии генов-кандидатов для ассоциированных с кетозом тканей на основе известных данных по человеку с применением GTEx.
Современная селекция крупного рогатого скота, направленная исключительно на повышение производства молока без внимания к другим чертам фенотипа, приводит к увеличению частоты развития различных патологий: мастита, заболеваний конечностей, дисфункций яичников и других болезней репродуктивной системы [1, 2], а также снижению генетического разнообразия и адаптивности животных [3]. В структуре заболеваний незаразной этиологии у высокопродуктивных коров особое место занимают метаболические нарушения, характеризующиеся повышенным образованием и резким увеличением содержания кетоновых тел в крови, молоке и моче животных. Развитие первичного кетоза, вызванного энергетическим дисбалансом, сопровождается возникновением целого ряда физиологических реакций, наиболее важными из которых можно считать синдром трофической недостаточности, интоксикационный синдром и местную воспалительную реакцию [4]. Использование новейших достижений в области клинической ветеринарной биохимии позволяет расширить диагностические возможности, повысить своевременность и эффективность лечебно-профилактических мероприятий [5].
Несмотря на успехи клинической биохимии, поиск генетических маркеров, ассоциированных с кетозом [6], остается актуальной проблемой современной ветеринарии. Стоит отметить, что даже в одних и тех же условиях содержания и при схожем уровне молочной продуктивности диапазон адаптационных возможностей у коров различается. Это дает основание предполагать, что адаптационный процесс во многом зависит от генетических особенностей организма животного [7, 8]. Необходимо учесть, что метаболические заболевания по своей природе многофакторны, невозможно выделить конкретный ген или полиморфизм, который бы являлся ответственным за развитие данных заболеваний [9, 10]. Основные направления в изучении генетической этиологии кетоза включают в себя полногеномный анализ ассоциаций (ПГАА, GWAS) [11, 12], исследование экспрессии генов с помощью как RNA-seq [13, 14], так и realtime qRT-PCR [15], кДНК-чипов [16], а также анализ ассоциаций генов-кандидатов [10].
Полногеномный анализ ассоциаций в медицине уже имеет свою весьма успешную историю [17–19]. При исследовании кетоза у коров с помощью ПГАА индикатором субклинической и клинической стадий болезни служит высокая концентрация β-гидроксимасляной кислоты как в молоке, так и в крови [12] или фенотипом для ПГАА может служить диагноз животного в целом. Так, S. Nayeri с соавт. [12] был проведен полногеномный анализ ассоциаций, в результате которого были обнаружены ранее неидентифицированные гены, связанные с субклиническим кетозом как во время первой лактации, так и для последующих. В целом авторы указывают о статистически значимых SNPs (single nucleotide polymorphisms) в генах, имеющих тесную взаимосвязь с ключевыми регуляторами иммунной системы: TNF (для субклинического кетоза в первую и последующие лактации), IFNG (для субклинического кетоза в первую лактацию), LEP (для субклинического кетоза во вторую и последующие лактации). Большая часть обнаруженных в данном исследовании транскриптов кандидатных генов играет роль в процессах иммунной системы животных, также часть генов кодирует транспортеры различных ионов. Например, IL-8 (ген CXCL8) – это один из главных медиаторов воспалительной реакции, секретируемый, главным образом нейтрофилами, действующими в месте инфекционного воспаления. Исследователи указывают на предыдущее исследование связи кетоновых тел и врожденного иммунного ответа эпителиальных клеток молочной железы (усиленная экспрессия IL-8) на инфицирование Escherichia coli [20]. В целом результаты S. Nayeri и соавт. [12] не противоречат результатам предыдущих исследований, в которых отмечается взаимосвязь метаболических заболеваний и иммунной системы [21].
В цели ПГАА не входит биологическая интерпретация: каким именно образом конкретные мутации влияют на молекулярные процессы в клетках – изменяется ли уровень экспрессии гена-кандидата, влияет ли мутация на эффективность работы транслируемого белка и т.д. Также не ясна общая картина взаимодействия генов при кетозе: в какой момент заболевание приводит к тому, что мутации в таких генах как CXCL-8 и TNF начинают играть ключевую роль в его развитии, и в каких именно тканях и клетках генетические нарушения являются значимыми при воспалении [22]. Поскольку имеются исследования о том, что ряд генов и метаболических путей вносит значительный вклад в развитие кетоза в разные временные промежутки, следует проводить функциональные исследования на основе результатов ПГАА на животных в течение всего транзитного периода [23]. Одним из первых и доступных вариантов анализа результатов ПГАА может стать аннотация локализации обнаруженных SNP относительно близлежащих генов или генов, непосредственно в которых данные SNP и были обнаружены.
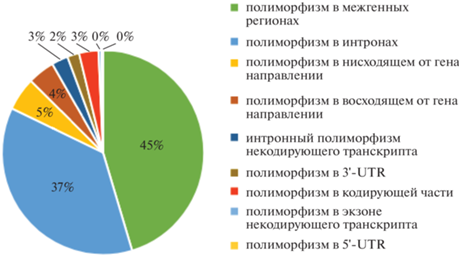
R.A.N. Soares и соавт. был проведен метаанализ данных, приведенных в статьях по экспрессии генов при кетозе, а также метаанализ исследований ПГАА, в которых был определен список генов-кандидатов и ряд SNP, имеющих высокую статистическую значимость в формировании синдрома [24]. Локализация SNPs относительно генов/ближайших генов представлена на рис. 1. Аннотация проведена при помощи https://www.ensembl.org/Bos_taurus/Tools/VEP сборка ARS-UCD1.2 [25].
Мутации в межгенных участках не приводят к заменам аминокислот в белке, однако возможно имеют значение в формировании ДНК-протеин-комплекса (например, места связывания с гистонами или транскрипционными факторами) [26], могут иметь как нисходящий или восходящий эффект на прилежащий ген (например, воздействие на открытые рамки считывания) [27], так и иметь эффект на пространственно близкие, но дистальные по нуклеотидной последовательности гены. Также мутации в межгенных участках могут оказывать воздействие на регуляторные механизмы (например, энхансеры и сайленсеры) [28], связанные с активацией экспрессии дискретных наборов генов на расстоянии нескольких тысяч нуклеотидов. Изменения в связывании протеина с энхансером перепрограммируют экспрессию генов и влияют на фенотип клетки [29, 30]. Также межгенные регионы могут включать в себя еще не обнаруженные гены, такие как регуляторные некодирующие РНК. Несмотря на то что информации о них не так много, известно, что они выполняют регуляторные функции. Уже почти два десятка лет проект ENCODE проводит подробные исследования межгенных регионов у человека [31].
Обнаруживаемые в результате ПГАА SNP в интронах могут стать отправной точкой в изучении зависимости между полиморфизмом в интронах и уровнем экспрессии гена-кандидата, поскольку известно, что полиморфизм в сайтах узнавания сплайсосом пре-мРНК влияет на процесс сплайсинга и следовательно на уровень экспрессии конечного транскрипта гена [27, 32, 33].
Мутации в кодирующих частях могут происходить в кодонах, соответствующая аминокислота которых у мутантного типа ослабляет функциональную активность кодируемого транскрипта или приводит к синонимичным заменам (миссенс, сеймсенс мутации) [34, 35], транкирует его (нонсенс-мутации) [36]. Биологическая интерпретация возможных эффектов полиморфизма является ключевым этапом в понимании молекулярно-генетической этиологии кетоза по результатам GWAS [37].
Про UTRs (нетранслируемые регионы) известно, что они имеют ключевые функции в посттранкрипционной регуляции экспрессии генов, включая направление транспорта мРНК из ядра, эффективность трансляции [38], внутриклеточную локализацию [39] и стабильность [40]. Исследования по влиянию полиморфизмов в UTR-участках проведены для некоторых генов [41, 42], однако анализ комплекса генов-кандидатов, ассоциированных с кетозом, при помощи такого подхода еще не проведен.
Значимые функции, выполняемые некодирующими транскриптами, экспрессируемыми от гибридных генов (генов, экспрессирующих как мРНК, так и некодирующие транскрипты), включают регуляцию сплайсинга мРНК и мРНК процессинга, активность “микроРНК-губок”, конкурентную ассоциацию/диссоциацию белка, связывающего РНК, экспрессию микропептидов/микропротеинов [43].
Биологическая интерпретация результатов GWAS представляется проще, когда статистически значимые SNP находятся вблизи друг от друга и в кодирующей последовательности. Например, в ходе работы S. Nayeri и соавт. при анализе ассоциаций с субклиническим кетозом во время первой лактации были выявлены четыре SNP в первом интроне гена GC [12]. Ген GC кодирует витамин D, связывающий протеин, который является основным переносчиком витамина D3. И у людей, и у крупного рогатого скота во время инфекции моноциты и макрофаги экспрессируют энзим, который конвертирует витамин D в его активную форму (25(OH)D3), играющую важную роль в функционировании иммунной системы [44]. Локализация SNP в интронах, в том числе первом, вероятно не имеет значения в функциональной активности или конформации белка, однако может играть ключевую роль в уровне экспрессии гена [45, 46]. Представленные в настоящей работе и обнаруживаемые в дальнейшем гены, ассоциированные с функциями иммунной системы, согласуются с ролью местной воспалительной реакции при кетозе, как упомянуто выше. Таким образом, подробное изучение уровня экспрессии гена GC в зависимости от метаболического статуса крупного рогатого скота может стать одним из шагов в понимании изменений в иммунном статусе при кетозе.
Схожим примером могут служить результаты работы V. Kroezen и соавт. [10] по поиску генов-кандидатов для кетоза во время первой лактации. Получены статистически значимые SNPs для гена CPT1A (карнитинпальмитоилтрансфераза формы 1А), локализующиеся в 3'-UTR гена. Митохондриальное окисление длинноцепочечных жирных кислот инициируется последовательными реакциями карнитинпальмитоилтрансферазы I (локализованной на внешней мембране митохондрии) и карнитинпальмитоилтрансферазы II (локализованной на внутренней мембране) вместе с карнитинацилкарнитинтранслоказой. CPT1 является ключевым энзимом в карнитин-зависимом транспорте по внутренней мембране митохондрии и его дефицит приводит к снижению β-окисления жирных кислот [47]. При увеличении экспрессии CPT1A оксидация жирных кислот может быть усилена и в некоторой степени может способствовать инсулинрезистентности Накопление жирных кислот в клетках может привести к формированию инсулинрезистентности, способной сформировать развитие диабета 2-го типа и гиперинсулинемии [48]. 3'-UTRs сигнальных РНК хорошо изучены с точки зрения регуляции процессов мРНК, таких как локализация мРНК, их стабильность и трансляция. Дополнительно 3'-UTRs опосредуют протеин–протеин взаимодействия, таким образом передавая генетическую информацию, закодированную в 3'-UTRs до протеинов. Показано, что данная функция регулирует разнообразные свойства протеинов, включая сложные конформации белков и посттрансляционные модификации. Таким образом, 3'-UTR опосредованный информационный трансфер может регулировать свойства белков, которые не закодированы в аминокислотной последовательности [49–51].
Полученные исследователями результаты подают большие надежды, однако стоит учитывать ограничения, которые накладывает низкая наследуемость такого изучаемого явления как кетоз (о наследуемости комплексных фенотипов далее). Сами исследователи указывают на то, что требуются дальнейшие валидационные исследования для полученных ими результатов, в том числе и для того, чтобы определить эффекты потенциальных редких аллелей, которые вероятно не были ими обнаружены. Идентификация и понимание генетических маркеров предрасположенности к кетозу пойдут на пользу всей молочной индустрии, предоставляя хозяйствам, племенным организациям и ветеринарным специалистам инструмент для прогнозирования степени предрасположенности коров к данному заболеванию [10].
Пересекающиеся в GWAS-исследованиях гены-кандидаты, представленные в уже упомянутом метаанализе [24], имеют некоторые протеин–протеин взаимодействия, определенные нами на рис. 2 при помощи веб-приложения STRING c настройкой без “майнинга текста” [52] (SLCO1B1, SLCO10A1 не найдены в базе STRING для Bos taurus).
Рис. 2.
Протеин–протеин взаимодействия, определенные при помощи веб-приложения STRING для генов, обозначенных в метаанализе ПГАА.

На рис. 2 представлены три группы связанных генов. Дальнейшее описание проведено на основе статей о генетике человека. CHRNG и CHRNB1 являются генами, кодирующими субъединицы гамма-рецептора ацетилхолина. Рецептор ацетилхолина меняет конформацию при связывании с ацетилхолином, приводя к открытию ионного канала на поверхности плазмалеммы. Мутации в гене рецептора ассоциируются с замедленной работой канала, врожденным миастеническим синдромом [53]. Предполагаемые гомологи CHRNG и CHRNB1 имеют корреляционные отношения по уровню экспрессии с PTK2-геном, однако такие отношения установлены только для других организмов. Протеин PTK2 (protein tyrosine kinase 2, протеинкиназа тирозина 2) кодируется геном PTK2. PTK2 является ключевым адгезин-ассоциированным белком, участвующим в клеточном сцеплении (как клетки адгезируются друг к другу и окружению) и передвижении (как клетки передвигаются) [54]. Схожие взаимоотношения PTK2 установлены для других организмов с EDIL3 и FN1. Egf-подобные повторы и дискоидиновые домены протеин 3 кодируются геном EDIL3 и являются интегриновым лигандом Del-1-рецептора, играющего ключевую роль в опосредовании ангиогенеза и возможно участвующего в перестройке и развитии стенок сосудов. Он также оказывает влияние на поведение эндотелиальных клеток [55]. FN1 (фибронектин 1) связывает клеточные поверхности и различные компоненты, включая коллаген, фибрин, гепарин, ДНК и актин. Фибронектины вовлечены в клеточную адгезию, способность клеток к передвижению, опсонизацию, ранозаживление и поддержание формы клетки [56]. О схожем отношении с PTK2, обнаруженном у других организмов, также можно сказать про TRIP6, который расположен на ключевых сайтах адгезии и вдоль стрессовых волокон актина. Накопление данного протеина у плазмалеммы происходит в ЛФК-зависимой манере (лизофосфатидная кислота) и регулирует ЛФК-индуцируемую миграцию клеток. Известны альтернативно сплайсированные варианты, кодирующие разные изоформы TRIP6, однако некоторые из них не до конца описаны [57].
В следующей группе в середине рис. 2 представлено три тесно связанных гена CPT1A, CPT1B, CPT1C (все три изоформы CPT1) [58], являющихся изоформами карнитин-О-пальмитоилтрансферазы: печеночной, мышечной и мозговой соответственно, и их связь в результате анализа очевидна [47]. Были обнаружены альтернативно сплайсированные транскрипты, кодирующие разные изоформы гена [59]. Каждый из них связан и имеет корреляционные отношения по уровню экспрессии с геном ACACB, однако такие отношения установлены только для других организмов. Acetyl-CoA carboxylase (ацетил-КоА карбоксилазы) представляют собой систему мультифункциональных ферментов. ACC является геном, кодирующим биотин-содержащий энзим, который катализирует карбоксилирование ацетил-КоА до малонил-КоА (лимитирующая реакция в синтезе жирных кислот). ACC-β (или АСС2) предположительно контролирует оксидацию жирных кислот посредством способности малонил-КоА ингибировать работу карнитинпальмитоилтрансферазы I, участвующей в лимитирующей реакции потребления жирных кислот и их оксидации в митохондриях. ACC2 может быть скорее всего вовлечен в регуляцию окисления жирных кислот [60]. В свою очередь, ACACB имеет связь с PC (у других организмов обнаружены коэкспрессионные отношения, соседство по расположению в геноме). Пируваткарбоксилаза (РС) [61] катализирует карбоксилирование пирувата до аксалоацетата. РС участвует в глюконеогенезе, липогенезе, секреции инсулина и синтезе нейротрансмиттера глутамата [62]. С тесной связью на рис. 2 также представлены ACSL1, ACSL3, ACSL5, ACSL6 (long-chain acyl-CoA synthetase), которые кодируют представителей семейства лигаз длинноцепочечных жирных кислот коэнзима А. И хотя они обладают субстратной специфичностью, внутриклеточной локализацией, распределением по тканям, все изозимы этого семейства конвертируют свободные длинные жирные кислоты до эфиров ацил-КоА и таким образом играют ключевую роль в биосинтезе липидов и деградации жирных кислот [63]. Интересно, что 1, 3, 5, 6 изоформы ацил-КоА-синтетазы жирных кислот с длинной цепью имеют взаимосвязь с изоформами карнитин-О-пальмитоилтрансферазы, существует предположительная модель комплекса этих протеинов на внешней мембране митохондрий [64]. В анализе STRING была обнаружена пара генов, имеющих разные функции: ген рецептора, активируемого пероксисомным пролифератором (PPARG, PPARD) и ретиноидный X-рецептор (RXR). Однако проведены исследования, в которых показано, что они вовлечены в развитие рака эндометрия, ассоциированного как с ожирением, так и с резистентностью к инсулину [65].
Третья получившаяся в результате анализа группа – пара SLCO1A2 и SLCO1B3 – члены семейств 1B2 и 1А2, являющихся переносчиками растворенных носителей органических анионов в печени, а также влияющих на поглощение стероидов [66]. Стоит отметить, что данные представители семейства переносчиков растворенных носителей органических анионов играют ключевую роль в метаболизме лекарственных препаратов [67].
Общая картина взаимодействия почти всех представленных в настоящей работе генов описана в другой работе автора метаанализа [68]. Необходимо подчеркнуть, что данных по взаимодействию (в том числе коэкспрессионных отношений) генов по результатам исследований человека намного больше, чем схожих исследований по протеин–протеин и ген–ген взаимодействиям у Bos taurus. Получение таких знаний представляется крайне актуальным для понимания природы сложных заболеваний крупного рогатого скота, в том числе метаболических.
В целом процессы, ассоциированные с генами по результатам метаанализа [24], можно аннотировать при помощи Generic Gene Ontology Term Finder [69] для биологических процессов. Полученные результаты визуализированы при помощи REVIGO (рис. 3) [70].
Аннотация генов по биологическим процессам не противоречит общим представлениям о кетозе, характеризующемся гиперкетонемией [71]. Кетоновые тела (β-гидроксимасляная кислота ацетоацетат, ацетон) – группа органических соединений, являющихся промежуточными метаболитами жиров, представляющих собой альтернативный продукт окисления свободных жирных кислот в печени [72]. Кетоз ассоциирован с тяжелым отрицательным энергетическим балансом: поступление неэффективных источников энергии, не удовлетворяющих высоким требованиям организма для производства молока в раннем лактационном периоде, дефицит в рационе легкоусвояемых сахаров [6]. Отрицательный энергетический баланс является триггером процесса оксидации жира, запасаемого в адипозных тканях, и приводит к тому, что большие количества свободных жирных кислот в крови окисляются до кетоновых тел. Липопротеины низкой плотности связываются со своими рецепторами в целевых клетках, эфиры холестерина гидролизуются лизосомами до свободного холестерина и жирных кислот. Ацетил-КоА после реакции окисления жирных кислот становится источником энергии в цикле Кребса. Когда цикл Кребса заторможен, чрезмерное количество ацетил-КоА может приводить к образованию кетоновых тел [73, 74]. Таким образом, свободные жирные кислоты преобразуются печенью в кетоновые тела и используются в качестве альтернативного источника энергии для многих тканей организма. Тем самым достигается главный биологический смысл – восстановление энергетического баланса [75].
Печень как один из главных органов, в котором протекают ключевые биохимические реакции, в том числе метаболизм углеводов и липидов [76, 77], представляет наибольший интерес с точки зрения экспрессии ключевых для патогенеза кетоза генов [16, 78]. Однако исследование крови методологически более удобно и представляется возможным вариантом изучения зависимости экспрессионной активности генов от физиологического состояния животного [13, 14]. Данные по экспрессии рассматриваемых нами генов в человеческих тканях представлены на рис. 4 (для SLCO10A1 данных нет) при помощи веб-сервиса GTEx [79]. Уровень экспрессии генов может быть промежуточным показателем между генотипом и сложным признаком и является модификатором предрасположенности к заболеваниям [80–82].
Рис. 4.
Данные об уровне экспрессии в тканях человека генов-кандидатов, обозначенных в метаанализе ПГАА. тнм – транскриптов на миллион.

Не менее важным аспектом в изучении кетоза является оценка уровня экспрессии генов в зависимости от стадий отрицательного энергетического баланса, которые проходит животное. Имеются данные о сравнении уровня экспрессии генов клеток печени при помощи микрочипов у животных с индуцированным кетозом [16], у животных с разной диетой [83]. Проведены исследования индуцированного кетоза в сравнении с контролем при помощи RNA-Seq [84], а также сравнение экспрессии генов жировой ткани между коровами с кетозом и здоровыми животными при помощи количественной real-time RT-PCR [15]. Все перечисленные исследования основаны на единомоментном сравнении групп между собой. Однако исследования, направленные на определение динамики изменений экспрессионного профиля в зависимости от физиологического состояния животного (например, в транзитный период) [85], могут представлять особый интерес для определения ключевых значений уровня экспрессии генов-кандидатов, формируя более ясную картину их коэкспрессионных отношений.
Низкая концентрация инсулина на фоне инсулинорезистентности способствует независимому поглощению питательных веществ молочной железой, тогда как инсулинзависимые ткани увеличивают окисление жирных кислот и уменьшают использование глюкозы [86]. С учетом этого дальнейшие исследования в области экспрессионной активности тканей и органов, в частности эндокринных, могут представлять особый интерес.
Ключевым аспектом в изучении гиперкетонемии является наследуемость данного фенотипа. Проведен ряд исследований наследуемости метаболических заболеваний у коров молочных пород, в том числе для кетоза, однако ее степень оценивается низкой [1, 72, 87, 88]. Стоит отметить, что коэффициенты наследуемости, полученные при линейном регрессионном анализе бинарных данных (“0” – животное не болеет, “1” – животное болеет), зависят от частоты встречаемости болезни в исследовательской выборке, в связи с чем результаты отдельных исследований не могут сравниваться между собой [88]. В других исследованиях о наследуемости уровня β-гидроксимасляной кислоты в молоке коров, основанных на имеющихся в хозяйствах данных, она оценивается как низкая и варьирует от 0.02 до 0.14 [89].
В целом характерная для кетоза низкая наследуемость может быть связана с проблемой утерянной наследуемости, характерной для явлений сложной природы. Патогенез мультифакторных заболеваний часто зависит от промежуточных фенотипов с количественным наследованием. А. Blanco-Gómez и соавт. утверждали, что недетектированные промежуточные фенотипы могут объяснить большую часть утерянной наследуемости [90], соответственно использование непрерывных величин для описания подтипов кетоза как фенотипа (например, ИК-Фурье спектроскопия крови для определения концентрации β-гидроксимасляной кислоты) позволит определять субклинические случаи болезни, более высокие частоты распространения и более высокие значения наследуемости фенотипа [91].
Несмотря на то что было выдвинуто много гипотез для решения проблемы утерянной наследуемости явлений, реальные причины этой проблемы остаются неизвестными. Многие характеристики, такие как размер тела, имеют высокую степень наследуемости. Уже доказано, что 80–90% изменчивости размера тела объясняются наследственностью. Несмотря на то что уже идентифицировано много аллелей, ассоциированных с данным фенотипом, они объясняют только около 5–10% изменчивости фенотипа. Это один из случаев сложных, комплексных фенотипов, и в целом существует большой разрыв между тем, какую долю изменчивости сложного фенотипа ожидается объяснить генетическими изменениями (“ожидаемая наследуемость”), и тем, какую степень наследуемости действительно получилось связать с аллельными вариантами. Эта разница и получила название “утерянная наследуемость” [90].
Вопрос о том, почему не у всех коров с высокими показателями молочной продуктивности наблюдается клиническая картина кетоза, все еще остается открытым, несмотря на значительный технический прогресс в транскриптомике, протеомике и геномике [10]. Наследуемость комплексных фенотипов также зависит от вклада факторов окружающей среды [92]. Таким образом, чем больше фенотипической изменчивости объясняется внешними факторами, тем меньше фенотипической изменчивости объясняется генетическими предикторами, и vice versa. Следовательно, точность фенотипической изменчивости может значительно варьировать от популяции к популяции, находящихся под воздействием разных факторов окружающей среды. Примером могут служить внешние факторы, способствующие развитию кетоза, например качество и полноценность корма, а также нормирование рациона в зависимости от физиологического состояния [74].
Существует предположение о том, что проблема утерянной наследуемости может быть решена через генетические взаимодействия или, по-другому, эпистазис. Утерянная наследуемость в узком понимании базируется на представлении, что не существует взаимодействий между аллелями и что их эффекты имеют только аддитивную природу. Но такое понимание проблемы не совсем верно, поскольку эпистатические взаимодействия также оказывают влияние на наследуемость в широком смысле этого слова. Таким образом, существует предположение, что значительная часть утерянной наследуемости может объясняться не аллельными вариантами, которые еще предстоит открыть, а эпистатическими взаимодействиями между аллелями, которые уже открыты. Хотя может быть и так, что предположительно все аллельные формы, ответственные за изменчивость фенотипа, уже изучены, однако их аддитивный эффект не объясняет всей изменчивости комплексного фенотипа. Поэтому объясненная наследуемость всегда будет меньше всей наследуемости [93]. В любом случае исследование эпистатических явлений в генетике является очень трудной задачей, требующей большой выборки. Степень вклада генетических взаимодействий в явление утерянной наследуемости еще предстоит исследовать, как, например, еще предстоит исследовать предположение о том, что эпигенетические изменения имеют эффект на экспрессию генов от индивидуума к индивидууму, что, в свою очередь, оказывает влияние на изменчивость фенотипа. Как бы то ни было, все эти попытки объяснить причины проблемы утерянной изменчивости еще предстоит проверить экспериментальными исследованиями [94], и такой “пробел в знаниях” отмечается не только в ветеринарии, но и в разных областях медицины [95–97].
Если кетоз – многофакторное заболевание по своей этиологии, то можно сказать, что поиск генов-кандидатов, ассоциированных с повышенным уровнем бета-гидроксимасляной кислоты, является задачей только для одного из промежуточных фенотипов. Целесообразен поиск, например, генов, ассоциированных с резистентностью к инсулину, генов, мутации в которых нарушают стабильность окисления жирных кислот (а также поиск аллельных вариантов, ассоциированных с высокой скоростью окисления). Тогда будут обнаружены промежуточные фенотипы, имеющие значительный вклад в такой сложный признак как предрасположенность к кетозу.
В последние годы разрабатываются новые статистические модели для определения ассоциации между генетическим локусом или аллелем и несколькими признаками, чтобы идентифицировать плейотропию [98], а также вклад промежуточных фенотипов [99]. Исследователями в этой области отмечается интересное явление: некоторые плейотропные локусы не имеют общих SNP, но вместо этого различные SNP являются казуальными для разных признаков. Эти плейотропные локусы ассоциированы с несколькими признаками, однако они не имеют общих генетических аллелей для разных ассоциаций. Существует две модели плейотропии в таких случаях [100]. Первая называется моделью горизонтальной плейотропии, когда генетические аллели имеют прямой эффект на два или более признака. Вторая модель – модель вертикальной плейотропии, когда аллель имеет прямой эффект на какой-либо признак и эффект на второй признак опосредованно через первый [101]. Таким образом проводятся исследования с целью обнаружить ассоциации комплексного признака с некоторыми аллельными вариантами предположительно ключевых генов в патогенезе кетоза: гены лептина и его рецептора [9, 102, 103], ген 5'АМФ-активируемой протеинкиназы [104], гены аполипопротеинов и их рецепторов [105, 106] и других. Однако исследовать аллельные варианты не менее значимых в возможной этиологии кетоза генов, таких как ген грелина, еще предстоит.
ЗАКЛЮЧЕНИЕ
Несмотря на длительную историю исследования кетоза с биохимической точки зрения, задача детерминировать генетические факторы, ассоциированные с развитием данного метаболического синдрома у коров, остается актуальной. Количество исследований генетической предрасположенности к кетозу с применением ПГАА недостаточно для однозначных выводов и требуется их дальнейшая валидация на разных выборках. Результаты уже проведенных исследований нуждаются в экспериментальной проработке как с точки зрения связи аллельный вариант–фенотип, так и с точки зрения молекулярных взаимодействий в клетке (например, экспрессионная активность генов в зависимости от аллельного варианта – функциональное аннотирование). Для высокого разрешения получаемых в ПГАА результатов возможно применение комбинации метода с ПЦР-ПДРФ и/или секвенирования отдельных генов, в которых, например, обнаружены SNPs с высокой статистической значимостью. Также необходимы исследования эпигенетических особенностей и эпистатических взаимодействий для экспериментального изучения молекулярно-генетических процессов, ассоциированных с кетозом. Такой комплексный подход позволит лучше понять патогенез заболевания и идентифицировать аллели, которые вносят вклад в предрасположенность к развитию заболевания.
Работа выполнена в рамках Государственного задания Минобрнауки России по теме № 0532-2022-0004 “Разработка технологии для маркер-ориентированной селекции крупного рогатого скота по генам, ассоциированным с устойчивостью к заболеваниям”.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Berry D.P., Bermingham M.L., Good M.,More S.J. Genetics of animal health and disease in cattle // Irish Veterinary J. 2011. V. 64. № 5. P. 1–10. https://doi.org/10.1186/2046-0481-64-5
Zinovieva N.A. Haplotypes affecting fertility in holstein cattle // Sel’skokhozyaistvennaya Biologiya. 2016. V. 51. P. 423–435. https://doi.org/10.15389/agrobiology.2016.4.423eng
Brito L.F., Bedere N., Douhard F. et al. Review: Genetic selection of high-yielding dairy cattle toward sustainable farming systems in a rapidly changing world // Animal. 2021. V. 15. P. 1–14. https://doi.org/10.1016/j.animal.2021.100292
Белоусов А.И., Красноперов А.С., Опарина О.Ю., Суздальцева М.А. Метаболические признаки алиментарного кетоза у высокопродуктивных коров // Труды ВИЭВ. 2018. Т. 80. № 1. С. 88–100. https://doi.org/10.30917/ATT-PRINT-2018-1
Белоусов А.И., Соколова О.В., Беспамятных Е.Н. Применение биохимического скрининга при оценке продуктивного здоровья высокопродуктивных коров в Свердловской области // Вопр. нормативно-правового регулирования в ветеринарии. 2018. Т. 4. С. 278–280. https://doi.org/10.17238/issn2072-6023.2018.4.278
Михайлова И.И., Евглевская Е.П., Михайлова О.И. и др. Патобиохимические изменения в метаболическом статусе высокопродуктивных коров // Ветеринарная патология. 2016. Т. 1. № 55. С. 75–80.
Kessel S., Stroehl M., Meyer H.H.D. et al. Individual variability in physiological adaptation to metabolic stress during early lactation in dairy cows kept under equal conditions // J. Animal Sci. 2008. V. 86. № 11. P. 2903–2912. https://doi.org/10.2527/jas.2008-1016
van Dorland H.A., Richter S., Morel I. et al. Variation in hepatic regulation of metabolism during the dry period and in early lactation in dairy cows // J. Dairy Sci. 2009. V. 92. № 5. P. 1924–1940. https://doi.org/10.3168/jds.2008-1454
Ковалюк Н.В., Якушева Л.И., Кузьминова Е.В. и др. Связь полиморфизмов гена лептина с предрасположенностью крупного рогатого скота к кетозу // Генетика и разведение животных. 2020. Т. 3. С. 20–26. https://doi.org/10.31043/2410-2733-2020-3-20-26
Kroezen V., Schenkel F.S., Miglior F. et al. Candidate gene association analyses for ketosis resistance in Holsteins // J. Dairy Sci. 2018. V. 101. № 6. P. 5240–5249. https://doi.org/10.3168/jds.2017-13374
Huang H., Cao J., Hanif Q. et al. Genome-wide association study identifies energy metabolism genes for resistance to ketosis in Chinese Holstein cattle // Anim. Genet. 2019. V. 50. № 4. P. 376–380. https://doi.org/10.1111/age.12802
Nayeri S., Schenkel F., Fleming A. et al. Genome-wide association analysis for β-hydroxybutyrate concentration in Milk in Holstein dairy cattle // BMC Genetics. 2019. V. 20. № 58. P. 1–17. https://doi.org/10.1186/s12863-019-0761-9
Yan Z., Huang H., Freebern E. et al. Integrating RNA-Seq with GWAS reveals novel insights into the molecular mechanism underpinning ketosis in cattle // BMC Genomics. 2020. V. 21. № 489. P. 1–12. https://doi.org/10.1186/s12864-020-06909-z
Wu Z.L., Chen S.Y., Qin C. et al. Clinical ketosis-associated alteration of gene expression in Holstein cows // Genes (Basel). 2020. V. 11. № 219. P. 1–11. https://doi.org/10.3390/genes11020219
Mohsin M.A., Yu H., He R. et al. Differentiation of subclinical ketosis and liver function test indices in adipose tissues associated with hyperketonemia in postpartum dairy cattle // Front. Vet. Sci. 2021. V. 8. P. 1–14. https://doi.org/10.3389/fvets.2021.796494
Loor J.J., Everts R.E., Bionaz M. et al. Nutrition-induced ketosis alters metabolic and signaling gene networks in liver of periparturient dairy cows // Physiol. Genomics. 2007. V. 32. № 1. P. 105–116. https://doi.org/10.1152/physiolgenomics.00188.2007
Visscher P.M., Wray N.R., Zhang Q. et al. 10 Years of GWAS discovery: biology, function, and translation // Am. J. Hum. Genet. 2017. V. 101. № 1. P. 5–22. https://doi.org/10.1016/j.ajhg.2017.06.005
Giacomini K.M., Yee S.W., Mushiroda T. et al. Genome-wide association studies of drug response and toxicity: An opportunity for genome medicine // Nat. Rev. Drug Discov. 2017. V. 16. № 1. P. 1–3. https://doi.org/10.1038/nrd.2016.234
Shu L., Blencowe M., Yang X. Translating GWAS findings to novel therapeutic targets for coronary artery disease // Front. Cardiovasc. Med. 2018. V. 5. P. 1–9. https://doi.org/10.3389/fcvm.2018.00056
Hillreiner M., Flinspach C., Pfaffl M.W., Kliem H. Effect of the ketone body beta-hydroxybutyrate on the innate defense capability of primary bovine mammary epithelial cells // PLoS One. 2016. V. 11. № 6. P. 1–18. https://doi.org/10.1371/journal.pone.0157774
Esposito G., Irons P.C., Webb E.C., Chapwanya A. Interactions between negative energy balance, metabolic diseases, uterine health and immune response in transition dairy cows // Animal Reproduction Sci. 2014. V. 144. № 3. P. 60–71. https://doi.org/10.1016/j.anireprosci.2013.11.007
Zhang S., Liu G., Xu C. et al. Perilipin 1 mediates lipid metabolism homeostasis and inhibits inflammatory cytokine synthesis in bovine adipocytes // Front. Immunol. 2018. V. 9. P. 1–14. https://doi.org/10.3389/fimmu.2018.00467
Ha N.T., Gross J.J., van Dorland A. et al. Gene-based mapping and pathway analysis of metabolic traits in dairy cows // PLoS One. 2015. V. 10. № 3. P. 1–15. https://doi.org/10.1371/journal.pone.0122325
Soares R.A.N., Vargas G., Muniz M.M.M. et al. Differential gene expression in dairy cows under negative energy balance and ketosis: A systematic review and meta-analysis // J. Dairy Sci. 2021. V. 104. № 1. P. 602–615. https://doi.org/10.3168/jds.2020-18883
McLaren W., Gil L., Hunt S.E. et al. The Ensembl Variant Effect Predictor // Genome Biology. 2016. V. 17. № 1. P. 1–14. https://doi.org/10.1186/s13059-016-0974-4
Huang D., Ovcharenko I. Identifying causal regulatory SNPs in ChIP-seq enhancers // Nucl. Acids Res. 2015. V. 43. № 1. P. 225–236. https://doi.org/10.1093/nar/gku1318
Calvo S.E., Pagliarini D.J., Mootha V.K. Upstream open reading frames cause widespread reduction of protein expression and are polymorphic among humans // Proc. Natl Acad. Sci. USA. 2009. V. 106. № 18. P. 7507–7512. https://doi.org/10.1073/pnas.0810916106
Chen J., Tian W. Explaining the disease phenotype of intergenic SNP through predicted long range regulation // Nucl. Acids Res. 2016. V. 44. № 18. P. 8641–8654. https://doi.org/10.1093/nar/gkw519
Schmidt S.F., Larsen B.D., Loft A. et al. Acute TNF-induced repression of cell identity genes is mediated by NFκB-directed redistribution of cofactors from super-enhancers // Genome Research. 2015. V. 25. № 9. P. 1281–1294. https://doi.org/10.1101/gr.188300.114
Vlahopoulos S.A. Aberrant control of NF-κB in cancer permits transcriptional and phenotypic plasticity, to curtail dependence on host tissue: molecular mode // Cancer Biology & Medicine. 2017. V. 14. № 3. P. 254–270. https://doi.org/10.20892/j.issn.2095-3941.2017.0029
Birney E., Stamatoyannopoulos J.A., Dutta A. et al. Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project // Nature. 2007. V. 447. P. 799–816. https://doi.org/10.1038/nature05874
Mucaki E.J., Shirley B.C., Rogan P.K. Expression changes confirm genomic variants predicted to result in allele-specific, alternative mRNA splicing // Front. Genet. 2020. V. 11. P. 1–16. https://doi.org/10.3389/fgene.2020.00109
Kalsotra A., Cooper T.A. Functional consequences of developmentally regulated alternative splicing // Nat. Rev. Genet. 2011. V. 12. № 10. P. 715–729. https://doi.org/10.1038/nrg3052
Chasman D., Adams R.M. Predicting the functional consequences of non-synonymous single nucleotide polymorphisms: Structure-based assessment of amino acid variation // J. Mol. Biol. 2001. V. 307. № 2. P. 683–706. https://doi.org/10.1006/jmbi.2001.4510
Dakal T.C., Kala D., Dhiman G. et al. Predicting the functional consequences of non-synonymous single nucleotide polymorphisms in IL8 gene // Scientific Reports. 2017. V. 7. № 1. P. 1–18. https://doi.org/10.1038/s41598-017-06575-4
Sharma J., Keeling K.M., Rowe S.M. Pharmacological approaches for targeting cystic fibrosis nonsense mutations // Eur. J. Med. Chem. 2020. V. 200. P. 1–11. https://doi.org/10.1016/j.ejmech.2020.112436
Petersen G.M., Parmigiani G., Thomas D. Missense mutations in Disease Genes: A Bayesian Approach to Evaluate Causality // Am. J. Human Genet. 1998. V. 62. № 6. P. 1516–1524. https://doi.org/10.1086/301871
van der Velden A.W., Thomas A.A. The role of the 5' untranslated region of an mRNA in translation regulation during development // Int. J. Biochem. Cell Biol. 1999. V. 31. № 1. P. 87–106. https://doi.org/10.1016/s1357-2725(98)00134-4
Jansen Ralf-P. mRNA localization: message on the move // Nature Reviews Mol. Cell Biol. 2001. V. 2. № 4. P. 247–256. https://doi.org/10.1038/35067016
Bashirullah A., Cooperstock R.L., Lipshitz H.D. Spatial and temporal control of RNA stability // Proc. Natl Acad. Sci. USA. 2001. V. 98. № 13. P. 7025–7028. https://doi.org/10.1073/pnas.111145698
Jiang H., Lucy M.C. Variants of the 5'-untranslated region of the bovine growth hormone receptor mRNA: Isolation, expression and effects on translational efficiency // Gene. 2001. V. 265. № 1. P. 45–53. https://doi.org/10.1016/s0378-1119(01)00356-0
Hu L., Ma Y., Liu L. et al. Detection of functional polymorphisms in the hsp70 gene and association with cold stress response in Inner-Mongolia Sanhe cattle // Cell Stress and Chaperones. 2019. V. 24. № 2. P. 409–418. https://doi.org/10.1007/s12192-019-00973-5
Dhamija S., Menon M.B. Non-coding transcript variants of protein-coding genes – what are they good for? // RNA Biology. 2018. V. 15. № 8. P. 1025–1031. https://doi.org/10.1080/15476286.2018.1511675
Nelson C.D., Reinhardt T.A., Thacker T.C. et al. Modulation of the bovine innate immune response by production of 1α,25-dihydroxyvitamin D3 in bovine monocytes // J. Dairy Sci. 2010. V. 93. № 3. P. 1041–1049. https://doi.org/10.3168/jds.2009-2663
Yu-fei S., Liu J., Wang X. et al. Essential role of the first intron in the transcription of hsp90β gene // FEBS Letters. 1997. V. 413. № 1. P. 92–98. https://doi.org/10.1016/S0014-5793(97)00883-1
Jo B., Choi S.S. Introns: The functional benefits of introns in genomes // Genomics Inform. 2015. V. 13. № 4. P. 112–118. https://doi.org/10.5808/GI.2015.13.4.112
Huang D., Chowdhury S., Wang H. et al. Multiomic analysis identifies CPT1A as a potential therapeutic target in platinum-refractory, high-grade serous ovarian cancer // Cell Reports Med. 2021. V. 2. № 12. P. 1–32. https://doi.org/10.1016/j.xcrm.2021.100471
Ren Q., Guo M., Yang F. et al. Association of CPT1A gene polymorphism with the risk of gestational diabetes mellitus: A case-control study // J. Assist. Reprod. Genet. 2021. V. 38. № 7. P. 1861–1869. https://doi.org/10.1007/s10815-021-02143-y
Mayr C. What are 3' UTRs doing? // Cold Spring Harb. Perspect. Biol. 2019. V. 11. № 10. P. 1–17. https://doi.org/10.1101/cshperspect.a034728
Chekulaeva M., Landthaler M. Eyes on translation // Mol. Cell. 2016. V. 63. № 6. P. 918–925. https://doi.org/10.1016/j.molcel.2016.08.031
Mayr C. Regulation by 3'-untranslated regions // Annu. Rev. Genet. 2017. V. 51. P. 171–194. https://doi.org/10.1146/annurev-genet-120116-024704
Szklarczyk D., Gable A.L., Nastou K.C. et al. The STRING database in 2021: Customizable protein–protein networks, and functional characterization of user-uploaded gene/measurement sets // Nucl. Acids Res. 2021. V. 49. № D1. P. 605–612. https://doi.org/10.1093/nar/gkaa1074
Freed A.S., Schwarz A.C., Brei B.K. et al. CHRNB1-associated congenital myasthenia syndrome: Expanding the clinical spectrum // Am. J. Med. Genetics. Part A. 2021. V. 185. № 3. P. 827–835. https://doi.org/10.1002/ajmg.a.62011
Andre E., Beckerandre M. Expression of an N-terminally truncated form of human focal adhesion kinase in brain // Biochem. Biophys. Res. Communications. 1993. V. 190. № 1. P. 140–147. https://doi.org/10.1006/bbrc.1993.1022
Li M., Zhong Di, Li G. Regulatory role of local tissue signal Del-1 in cancer and inflammation: a review // Cellular & Mol. Biol. Letters. 2021. V. 26. № 1. P. 1–12. https://doi.org/10.1186/s11658-021-00274-9
Hsiao C.T., Cheng H.W., Huang C.M. et al. Fibronectin in cell adhesion and migration via N-glycosylation // Oncotarget. 2017. V. 8. № 41. P. 70653–70668. https://doi.org/10.18632/oncotarget.19969
Dutta S., Mana-Capelli S., Paramasivam M. et al. TRIP6 inhibits Hippo signaling in response to tension at adherens junctions // EMBO Reports. 2018. V. 19. № 2. P. 337–350. https://doi.org/10.15252/embr.201744777
Warfel J.D., Vandanmagsar B., Dubuisson O.S. et al. Examination of carnitine palmitoyltransferase 1 abundance in white adipose tissue: Implications in obesity research // Am. J. Physiol. Regul. Integr. Comp. Physiol. 2017. V. 312. № 5. P. 816–820. https://doi.org/10.1152/ajpregu.00520.2016
Price N.T., Jackson V.N., Müller J. et al. Alternative exon usage in the single CPT1 gene of Drosophila generates functional diversity in the kinetic properties of the enzyme: Differential expression of alternatively spliced variants in Drosophila tissues // J. Biol. Chemistry. 2010. V. 285. № 11. P. 7857–7865. https://doi.org/10.1074/jbc.M109.072892
Riancho J.A., Vázquez L., García-Pérez M.A. et al. Association of ACACB polymorphisms with obesity and diabetes // Mol. Genet. Metabolism. 2011. V. 104. № 4. P. 670–676. https://doi.org/10.1016/j.ymgme.2011.08.013
Hellwege J.N., Stallings S., Torstenson E.S. et al. Heritability and genome-wide association study of benign prostatic hyperplasia (BPH) in the eMERGE network // Sci. Rep. 2019. V. 9. № 1. P. 1–10. https://doi.org/10.1038/s41598-019-42427-z
Lao-On U., Cliff T.S., Dalton S., Jitrapakdee S. Pyruvate carboxylase supports basal ATP-linked respiration in human pluripotent stem cell-derived brown adipocytes // Biochem. Biophys. Res. Communications. 2021. V. 569. P. 139–146. https://doi.org/10.1016/j.bbrc.2021.06.096
Rossi S.M., Konstantinidou G. Targeting long chain acyl-СоА synthetases for cancer therapy // Int. J. Mol. Sci. 2019. V. 20. № 15. https://doi.org/10.3390/ijms20153624
Lee K., Kerner J., Hoppel C.L. Mitochondrial carnitine palmitoyltransferase 1a (CPT1a) is part of an outer membrane fatty acid transfer complex // J. Biol. Chem. 2011. V. 286. № 29. P. 25655–25662. https://doi.org/10.1074/jbc.M111.228692
Nickkho-Amiry M., McVey R., Holland C. Peroxisome proliferator–activated receptors modulate proliferation and angiogenesis in human endometrial carcinoma // Mol. Cancer Res. 2012. V. 10. № 3. P. 441–453. https://doi.org/10.1158/1541-7786.MCR-11-0233
Mostaghel E.A., Cho E., Zhang A. et al. Association of tissue abiraterone levels and SLCO genotype with intraprostatic steroids and pathologic response in men with high-risk localized prostate cancer // Clin. Cancer Res. 2017. V. 23. № 16. P. 4592–4601. https://doi.org/10.1158/1078-0432.CCR-16-2245
Lu X., Chan T., Cheng Z. et al. The 5'-AMP-activated protein kinase regulates the function and expression of human organic anion transporting polypeptide 1A2 // Mol. Pharmacology. 2018. V. 94. № 6. P. 1–9. https://doi.org/10.1124/mol.118.113423
Nayeri S., Stothard P. Tissues, metabolic pathways and genes of key importance in lactating dairy cattle // Springer Sci. Rev. 2016. V. 4. № 2. P. 49–77. https://doi.org/10.1007/s40362-016-0040-3
Boyle E.I., Weng S., Gollub J. et al. GO::TermFinder – open source software for accessing Gene Ontology information and finding significantly enriched Gene Ontology terms associated with a list of genes // Bioinformatics. 2004. V. 20. № 18. P. 3710–3715. https://doi.org/10.1093/bioinformatics/bth456
Supek F., Bošnjak M., Škunca N., Šmuc T. REVIGO summarizes and visualizes long lists of Gene Ontology terms // PLoS One. 2011. V. 6. № 7. P. 1–9. https://doi.org/10.1371/journal.pone.0021800
Zhang G., Hailemariam D., Dervishi E. et al. Dairy cows affected by ketosis show alterations in innate immunity and lipid and carbohydrate metabolism during the dry off period and postpartum // Res. Veterinary Sci. 2016. V. 107. P. 246–256. https://doi.org/10.1016/j.rvsc.2016.06.012
Gulinski P. Ketone bodies – causes and effects of their increased presence in cows’ body fluids: A review // Vet. World. 2021. V. 14. № 6. P. 1492–1503. https://doi.org/10.14202/vetworld.2021.1492-1503
Wang Y., Gao Y., Xia C. et al. Pathway analysis of plasma different metabolites for dairy cow ketosis // Italian J. Animal Sci. 2016. V. 15. № 3. P. 545–551. https://doi.org/10.1080/1828051X.2016.1180643
Zhang G., Ametaj B.N. Ketosis an old story under a new approach // Dairy. 2020. V. 1. № 1. P. 42–60. https://doi.org/10.3390/dairy1010005
Dufour D.R., Lott J.A., Nolte F.S. et al. Diagnosis and monitoring of hepatic injury. II. Recommendations for use of laboratory tests in screening, diagnosis, and monitoring // Clin. Chem. 2000. V. 46. № 12. P. 2050–2068.https://doi.org/10.1093/clinchem/46.12.2050
Tsukamoto K., Teramoto T. Carbohydrate and lipid metabolism in liver cirrhosis // Nihon Rinsho. 1994. V. 52. № 1. P. 150–158.
Natesan V., Kim S.J. Lipid metabolism, disorders and therapeutic drugs – review // Biomol. Ther. (Seoul). 2021. V. 29. № 6. P. 596–604. https://doi.org/10.4062/biomolther.2021.122
McCabe M., Waters S., Morris D. et al. RNA-seq analysis of differential gene expression in liver from lactating dairy cows divergent in negative energy balance // BMC Genomics. 2012. V. 13. P. 1–11. https://doi.org/10.1186/1471-2164-13-193
Carithers L.J., Moore H.M. The Genotype-Tissue Expression (GTEx) project // Biopreserv. Biobank. 2015. V. 13. № 5. P. 307–308. https://doi.org/10.1038/ng.2653
Goldinger A., Henders A.K., McRae A.F. et al. Genetic and nongenetic variation revealed for the principal components of human gene expression // Genetics. 2013. V. 195. № 3. P. 1117–1128. https://doi.org/10.1534/genetics.113.153221
Genin E., Feingold J., Clerget-Darpoux F. Identifying modifier genes of monogenic disease: strategies and difficulties // Hum. Genet. 2008. V. 124. № 4. P. 357–368. https://doi.org/10.1007/s00439-008-0560-2
Wright G.E.B., Caron N.S., Ng B. et al. Gene expression profiles complement the analysis of genomic modifiers of the clinical onset of Huntington disease // Hum. Mol. Genet. 2020. V. 29. № 16. P. 2788–2802. https://doi.org/10.1093/hmg/ddaa184
Shahzad K., Lopreiato V., Liang Y. et al. Hepatic metabolomics and transcriptomics to study susceptibility to ketosis in response to prepartal nutritional management // J. Anim. Sci. Biotechnol. 2019. V. 10. P. 96. https://doi.org/10.1186/s40104-019-0404-z
Pralle R.S., Li W., Murphy B.N. et al. Novel facets of the liver transcriptome are associated with the susceptibility and resistance to lipid-related metabolic disorders in periparturient Holstein cows // Animals (Basel). 2021. V. 11. № 9. P. 1–22. https://doi.org/10.3390/ani11092558
Mezzetti M., Cattaneo L., Passamonti M.M. et al. The transition period updated: A review of the new insights into the adaptation of dairy cows to the new lactation // Dairy. 2021. V. 2. № 4. P. 617–636. https://doi.org/10.3390/dairy2040048
Cuiyu Z., Chang Z., Jiang Z. et al. The relationship between insulin resistance and type II ketosis in dairy cows // Acta Scientiae Veterinariae. 2019. V. 47. № 1. P. 1–8. https://doi.org/10.22456/1679-9216.93425
Klein S.L., Scheper C., Brügemann K. et al. Phenotypic relationships, genetic parameters, genome-wide associations, and identification of potential candidate genes for ketosis and fat-to-protein ratio in German Holstein cows // J. Dairy Sci. 2019. V. 102. № 7. P. 6276–6287. https://doi.org/10.3168/jds.2019-16237
Heringstad B., Chang Y.M., Gianola D., Klemetsdal G. Genetic analysis of clinical mastitis, milk fever, ketosis, and retained placenta in three lactations of Norwegian red cows // J. Dairy Sci. 2005. V. 88. № 9. P. 3273–3281. https://doi.org/10.3168/jds.S0022-0302(05)73010-1
Koeck A., Jamrozik J., Schenkel F.S. et al. Genetic analysis of milk β-hydroxybutyrate and its association with fat-to-protein ratio, body condition score, clinical ketosis, and displaced abomasum in early first lactation of Canadian Holsteins // J. Dairy Sci. 2014. V. 97. № 11. P. 7286–7292. https://doi.org/10.3168/jds.2014-8405
Blanco-Gómez A., Castillo-Lluva S., Del Mar S.F.M. et al. Missing heritability of complex diseases: Enlightenment by genetic variants from intermediate phenotypes // BioEssays. 2016. V. 38. № 7. P. 664–673. https://doi.org/10.1002/bies.201600084
Belay T.K., Svendsen M., Kowalski Z.M., Ådnøy T. Genetic parameters of blood β-hydroxybutyrate predicted from milk infrared spectra and clinical ketosis, and their associations with milk production traits in Norwegian Red cows // J. Dairy Sci. 2017. V. 100. № 8. P. 6298–6311. https://doi.org/10.3168/jds.2016-12458
Oreland L., Lagravinese G., Toffoletto S. et al. Personality as an intermediate phenotype for genetic dissection of alcohol use disorder // J. Neural. Transm. (Vienna). 2018. V. 125. № 1. P. 107–130. https://doi.org/10.1007/s00702-016-1672-9
Zuk O., Hechter E., Sunyaev S.R., Lander E.S. The mystery of missing heritability: Genetic interactions create phantom heritability // Proc. Natl Acad. Sci. USA. 2012. V. 109. № 4. P. 1193–1198. https://doi.org/10.1073/pnas.1119675109
Marian A.J. Elements of missing heritability // Curr. Opin. Cardiol. 2012. V. 27. № 3. P. 197–201. https://doi.org/10.1097/HCO.0b013e328352707d
van Calker D., Serchov T. The “missing heritability”–problem in psychiatry: Is the interaction of genetics, epigenetics and transposable elements a potential solution? // Neurosci. Biobehav. Rev. 2021. V. 126. P. 23–42. https://doi.org/10.1016/j.neubiorev.2021.03.019
Manolio T.A., Collins F.S., Cox N.J. et al. Finding the missing heritability of complex diseases // Nature. 2009. V. 461. № 7265. P. 747–753. https://doi.org/10.1038/nature08494
Genin E. Missing heritability of complex diseases: case solved? // Hum. Genet. 2020. V. 139. № 1. P. 103–113. https://doi.org/10.1007/s00439-019-02034-4
Wagner G.P., Zhang J. The pleiotropic structure of the genotype-phenotype map: the evolvability of complex organisms // Nat. Rev. Genet. 2011. V. 12. № 3. P. 204–213. https://doi.org/10.1038/nrg2949
Li Y., Huang J., Amos C.I. Genetic association analysis of complex diseases incorporating intermediate phenotype information // PLoS One. 2012. V. 7. № 10. P. 1–9. https://doi.org/10.1371/journal.pone.0046612
Hackinger S., Zeggini E. Statistical methods to detect pleiotropy in human complex traits // Open Biol. 2017. V. 7. № 11. P. 1–13. https://doi.org/10.1098/rsob.170125
Bone W.P., Siewert K.M., Jha A. et al. Multi-trait association studies discover pleiotropic loci between Alzheimer’s disease and cardiometabolic traits // Alzheimers Res. Ther. 2021. V. 13. P. 1–14. https://doi.org/10.1186/s13195-021-00773-z
Якушева Л.И., Абрамов А.А., Ковалюк Н.В., Сацук В.Ф. Связь полиморфизмов R25C и A80V гена лептина быков-производителей с оценкой их дочерей на предрасположенность к возникновению кетоза // Сб. науч. трудов Краснодарского науч. центра по зоотехнии и ветеринарии. 2019. Т. 8. № 3. С. 24–27. https://doi.org/10.34617/y47d-6h82
Komisarek J. Impact of LEP and LEPR gene polymorphisms on functional traits in Polish Holstein-Friesian cattle // Anim. Sci. Pap. Rep. 2010. V. 28.
Mahmoudi A., Zargaran A., Amini H.R. et al. A SNP in the 3'-untranslated region of AMPKgamma1 may associate with serum ketone body and milk production of Holstein dairy cows // Gene. 2015. V. 574. № 1. P. 48–52. https://doi.org/10.1016/j.gene.2015.07.077
Yang L., Bai J., Ju Z. et al. Effect of functional single nucleotide polymorphism g.-572 A>G of apolipoprotein A1 gene on resistance to ketosis in Chinese Holstein cows // Res. Vet. Sci. 2021. V. 135. P. 310–316. https://doi.org/10.1016/j.rvsc.2020.10.006
Tetens J., Heuer C., Heyer I. et al. Polymorphisms within the APOBR gene are highly associated with milk levels of prognostic ketosis biomarkers in dairy cows // Physiol. Genomics. 2015. V. 47. № 4. P. 129–137. https://doi.org/10.1152/physiolgenomics.00126.2014
Дополнительные материалы отсутствуют.