Вопросы ихтиологии, 2023, T. 63, № 4, стр. 427-441
Генетическая изменчивость азиатской корюшки Osmerus dentex (Osmeridae) Японского и Охотского морей по данным анализа микросателлитных локусов и митохондриальной ДНК
А. В. Семенова 1, 2, *, Е. В. Пономарева 1, О. В. Вилкина 3
1 Московский государственный университет
Москва, Россия
2 Институт общей генетики РАН – ИОГен РАН
Москва, Россия
3 Хабаровский филиал Всероссийского научно-исследовательского института
рыбного хозяйства и океанографии – ХабаровскНИРО
Хабаровск, Россия
* E-mail: seman2000@yandex.ru
Поступила в редакцию 03.10.2022
После доработки 05.12.2022
Принята к публикации 12.12.2022
- EDN: ROLXDS
- DOI: 10.31857/S0042875223040240
Аннотация
Исследовано генетическое разнообразие азиатской корюшки Osmerus dentex Охотского и Японского морей по девяти микросателлитным локусам и контрольному региону митохондриальной ДНК (940 пар нуклеотидов). Самые низкие значения всех параметров генетического разнообразия митохондриальной ДНК наблюдались в группе выборок с северного побережья Охотского моря. Наибольшее значение гаплотипического разнообразия было выявлено в выборке с о-ва Сахалин и у корюшки Японского моря. Звёздообразная топология генеалогических структур указывает на быструю экспансию численности и происхождение всех популяций вида от единого общего предка из основного рефугиума. По результатам анализа ядерных маркеров обнаружена достоверно значимая генетическая структурированность корюшки в пределах её тихоокеанского ареала (коэффициент генетической дифференциации (FST) равен 0.033), которая удовлетворительно описывается моделью изоляции расстоянием. Наибольшие генетические отличия по двум типам маркеров от остальных выборок обнаружены у корюшки рек Шкотовка и Ботчи бассейна Японского моря и р. Найба (Сахалин). Наблюдаемые паттерны генетической дифференциации корюшки по ядерным и митохондриальным маркерам хорошо дополняют друг друга и, вероятнее всего, ассоциированы с различиями в распространении личинок корюшки от мест нереста из-за дифференциации океанографических условий.
Азиатская корюшка Osmerus dentex Steindachner and Kner, 1870 (Nellbring, 1989) – анадромная рыба семейства корюшковых (Osmeridae) – широко распространена в эстуариях и прибрежных водах арктического побережья от Белого до Берингова морей и по азиатскому побережью Тихого океана от Берингова пролива до Кореи и Северной Японии (Клюканов, 1975; Черешнев и др., 2002; Kottelat, Freyhof, 2007). В водах российского Дальнего Востока этот вид является одним из наиболее важных для коммерческого и любительского рыболовства (Бугаев и др., 2014).
Нерестовые миграции корюшки в зависимости от водотока обычно проходят с марта по июнь, вверх по течению рек корюшка поднимается на небольшие расстояния, до нескольких километров, за исключением некоторых крупных водотоков. Например, в р. Амур протяжённость её нерестовых миграций может достигать 300 км. Корюшка созревает в возрасте 2–4 лет, икру откладывает на каменисто-галечный грунт, реже на растительность; после вылупления личинки переносятся течением вниз по реке и развиваются в приустьевых пространствах. Взрослые особи возвращаются в устья рек после нереста и могут перемещаться на морское мелководье, однако дальних миграций они не совершают (Василец, 2000; Гриценко, 2002; Черешнев и др., 2002).
Современный ареал корюшки формировался после окончания последнего оледенения при расселении вида из основного рефугиума в северо-западной части Тихого океана (Японского и южной части Охотского морей). Предполагают, что экспансия началась не ранее 15 тыс. лет назад (Гладенков, Гладенков, 2004; Скурихина и др., 2015; Skurikhina et al., 2018).
Изучение генетической изменчивости корюшек вод Тихого океана (Берингова, Охотского и Японского морей) проводили на основании разных фрагментов митохондриальной ДНК (мтДНК). Была показана слабая пространственная генетическая структурированность корюшки в пределах её тихоокеанского ареала при довольно чётком обособлении выборок из Японского моря (Ковпак и др., 2011; Скурихина и др., 2015; Skurikhina et al., 2018). Исследования полиморфизма ядерных маркеров ДНК позволили выявить дифференциацию корюшки на большой пространственной шкале между популяциями Берингова моря, западного побережья Камчатки, северным побережьем Охотского моря и выборками из южной части Охотского и Японского морей (Семенова и др., 2019а; Semenova et al., 2021).
В настоящей работе мы дополняем область исследования генетической структуры азиатской корюшки в пределах её тихоокеанского ареала за счёт южных районов обитания вида – Японского моря, о-вa Сахалин и материкового побережья Охотского моря. Генетическую изменчивость в популяциях оценивали на основании изменчивости микросателлитных локусов ядерной ДНК (STR) и контрольного региона (CR) мтДНК, содержащего D-петлю.
МАТЕРИАЛ И МЕТОДИКА
Исследованный материал, районы сбора проб
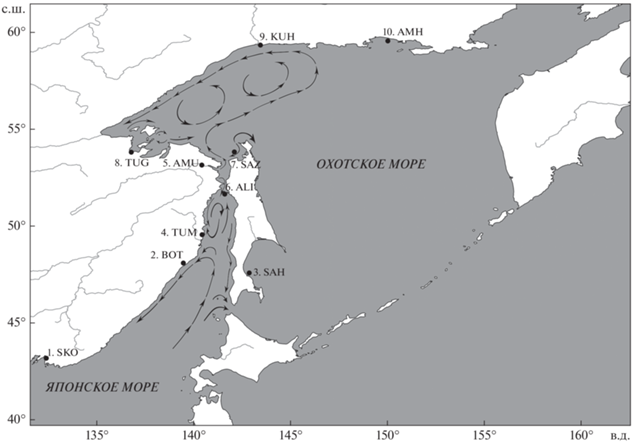
Материал для анализа генетической изменчивости корюшки из 10 локальностей в бассейнах Японского и Охотского морей (n = 464) был собран в 2016–2020 гг. Обозначения выборок и их географическая локализация представлены в табл. 1 и на рис. 1. Все сборы проводили во время нерестового хода корюшки в приустьевых пространствах морей или устьях рек, за исключением выборок из Амурского лимана и Сахалинского залива Охотского моря, представленных молодью из нагульных скоплений.
Таблица 1.
Характеристика исследованного материала
| Выборка | Локальность | Период сбора проб | Географическое положение | Координаты | Объём выборки, экз. | ||
|---|---|---|---|---|---|---|---|
| с.ш. | в.д. | микросател-литный анализ | анализ мтДНК | ||||
| 1. SKO | р. Шкотовка | Апрель 2020 | Уссурийский залив, северо-западная часть Японского моря | 43°30′ | 132°34′ | 32 | 5 |
| 2. BOT | р. Ботчи | Май 2020 | Татарский пролив, Японское море | 47°97′ | 139°52′ | 50 | 4 |
| 3. SAH | р. Найба | Февраль 2019 | Восточное побережье о. Сахалин, Охотское море | 47°41′ | 142°81′ | 37 | 9 |
| 4. TUM | р. Тумнин | Апрель 2019 | Татарский пролив, Японское море | 49°27′ | 140°33′ | 30 | 5 |
| 5. AMU | р. Амур | То же | Устье р. Амур, Охотское море | 53°21′ | 140°33′ | 49 | 7 |
| 6. ALI | Амурский лиман | Май 2020 | Охотское море | 51°70′ | 141°20′ | 46 | 4 |
| 7. SAZ | Сахалинский залив | Август 2019 | То же | 53°60′ | 141°96′ | 29 | 5 |
| 8. TUG | р. Тугур | Май 2020 | Западное побережье Охотского моря | 53°75′ | 136°77′ | 50 | 4 |
| 9. KUH | р. Кухтуй | Март 2020 | Северное побережье Охотского моря | 59°39′ | 143°24′ | 50 | 5 |
| 10. AMH* | Амахтонский залив | Июль 2016, Апрель 2018 | Северное побережье Охотского моря, Тауйская губа | 59°53′ | 150°35′ | 91 | 6 |
Методика генетического анализа
Микросателлитные локусы. Методики выделения ДНК, параметры амплификации ДНК и анализа продуктов полимеразной цепной реакции (ПЦР) при изучении STR-локусов приведены в работе Семеновой с соавторами (2019а), в которой также опубликованы основные характеристики исследованных маркеров.
Митохондриальная ДНК. Вариабельность мтДНК исследовали на основании изменчивости CR мтДНК. Для амплификации были сконструированы праймеры OsmCR_96F: 5'-TCGAACTCCCACCCTTAACT-3' и OsmCR-1.227R: 5'-ATAAAGGGCATTCTCACGGG-3'.
ПЦР проводили с использованием 10 мкл готовых смесей GenePak PCR Core (“ИзоГен”, Россия), с добавлением 5 мкл смеси праймеров (0.5 мкМ) и 5 мкл ДНК. Амплификацию проводили при следующем режиме: 2 мин при 94°С, 35 циклов: 30 с – 94°С, 40 с – 52°С, 1 мин – 72°С; 10 мин при 72°С. ПЦР-продукт очищали с использованием набора Clean-UP (“ИзоГен”) и секвенировали по Сэнгеру (Sanger et al., 1977) в коммерческой лаборатории Центра коллективного пользования “Геном” (Институт молекулярной биологии РАН, Москва). Для секвенирования использовали прямой праймер OsmCR_96F, а также дополнительно разработаны праймеры OsmF1: 5'-CATGAATGATAGGGTCAGGGAC-3' и Osm(int1)R: 5'-GTCCCTGACCCTATCATTCATG-3'.
Статистический анализ
Микросателлитные локусы. В программе Micro-Checker 2.2.3 данные исследовали на поиск возможных ошибок генотипирования, а также присутствия нуль-аллелей (Van Oosterhout et al., 2004). В случае обнаружения нуль-аллелей проводили оценку их частот и с поправкой на их присутствие вычисляли значения коэффициента генетической дифференциации (FST) (Weir, Cockerham, 1984) в программе FreENA (Chapuis, Estoup, 2007).
Программу GDA (http://lewis.eeb.unconn.edu/lewishome/software.html) использовали для оценки частот аллелей, аллельного разнообразия (A), ожидаемой и наблюдаемой гетерозиготности (HE, HO), числа приватных аллелей. Отклонения от равновесия Харди–Вайнберга тестировали с использованием коэффициента инбридинга в программе GENEPOP (http://genepop.curtin.edu.au), достоверность оценивали с применением точных тестов Фишера. Оценки аллельного разнообразия, скорректированные по минимальному размеру выборки (AR), получены в программе FSTAT 2.9.3 (http://www2.unil.ch/popgen/softwares/fstat.htm). Однофакторный дисперсионный анализ для оценки различий AR и HE между выборками провели в программе Excel, для попарных сравнений применяли U-критерий Манна–Уитни. GENEPOP использовали для оценки общей и попарной дифференциации популяций на основании коэффициентов FST. Уровень статистической значимости для множественных тестов корректировали с использованием процедуры Бонферрони (Rice, 1989). Значимость корреляции генетических (FST/(1 – – FST)) и географических (по кратчайшему водному пути, км) дистанций между группировками проверяли с применением теста Мантела в программе IBD 1.52 (http://ibdws.sdsu.edu/).
Анализ популяционной структуры проводили методом Байеса (Rannala, Mountain, 1997) в программе STRUCTURE (Pritchard et al., 2000). Использовали модель, допускающую генетическое смешение и корреляцию аллельных частот среди кластеров, с 105 первоначальными (впоследствии обрезанными) и 106 MCMC (цепь Маркова по методу Монте-Карло) итерациями, для К от 1 до 10, по 10 повторов для каждой величины К. Для оценки наиболее вероятного числа кластеров применяли метод ∆К (Evanno et al., 2005) с использованием Интернет-ресурса STRUCTURE HARVESTER (https://taylor0.biology.ucla.edu/structureHarvester/), а также оценки MedMeaK, MaxMeaK, MedMedK и MaxMedK (Puechmaille, 2016), полученные в программе STRUCTURE SELECTOR (https://lmme.ac.cn/StructureSelector/). Визуализацию разбиений по кластерам проводили на сервисе CLUMPAK (http://clumpak.tau.ac.il/).
Для определения возможного прохождения популяций корюшки через “горлышко бутылки”, т.е. сильного снижения эффективной численности в относительно недавнем прошлом, использовали программу BOTTLENECK 1.2.02 (Piry et al., 1999).
Визуализацию матрицы попарных FST проводили в программе STATISTIСA 6.0 (StatSoft) методом многомерного шкалирования.
Митохондриальная ДНК. Первичную обработку и множественное выравнивание нуклеотидных последовательностей проводили в программе Geneious 6.0.5 (“Biomatters Ltd.”, Новая Зеландия), определение числа гаплотипов (H), разнообразия гаплотипов (Hd) и нуклеотидов (Pi) проводили в программах FaBox (Villesen, 2007) и DnaSP v.5 (Librado, Rozas, 2009).
Анализ методом главных компонент (PCA) на основе ковариационной матрицы генетических дистанций проводили в программе GenAIEx 6.502 (Peakall, Smouse, 2012). Алгоритм minimum spanning network (MSN) (Bandelt et al., 1999) использовали для построения сети гаплотипов на основе дистанций Хамминга в программе PopArt (Leigh, Bryant, 2015).
Вычисление индексов попарной фиксации выборок (ΦPT) (Peakall et al., 1995), потока генов (Nm), анализ молекулярной дисперсии (AMOVA) (Meirmans, 2006) проводили в программе GenAIEx 6.502 (Peakall, Smouse, 2012). Были выделены три иерархических уровня дифференциации: между группами выборок, между выборками и внутри выборок. Полученные последовательности гаплотипов были депонированы в GenBank (www.ncbi.nlm.nih.gov/genbank) под номерами OP795783–OP795802 (Приложение) .
РЕЗУЛЬТАТЫ
Анализ микросателлитных локусов ядерной ДНК
Программа Micro-Checker не обнаружила возможные ошибки генотипирования ни в одном из локусов. В трёх выборках (SKO, TUG, KUH) показана возможность присутствия нуль-аллелей в локусе Tpa119. После проверки влияния возможных нуль-аллелей на оценки генетической дифференциации оказалось, что показатели FST изменяются не более чем на 0.3%. Статистически значимые отклонения от равновесия Харди–Вайнберга в распределении генотипов не обнаружены (после проведения коррекции Бонферрони).
Тестирование в программе BOTTLENECK не выявило прохождение корюшками этапа “горлышка бутылки” в недавнем прошлом (0.829 < p < 1.000).
Всего было выявлено 90 аллелей, число аллелей в каждом локусе варьировало от пяти (HtrG107) до 17 (MGPL-Omo5), в среднем 10 аллелей на локус. Приватные аллели с частотой встречаемости >5% не обнаружены. Средние оценки ожидаемой гетерозиготности (HE) и аллельного разнообразия (AR) в отдельных локальностях варьировали соответственно от 0.488 (ALI) до 0.582 (SKO) и от 5.7 (ALI, SAH) до 6.2 (BOT, KUH) (табл. 2). Однофакторный дисперсионный анализ не показал достоверные различия между выборками ни по HE, ни по AR. Попарное сравнение выборок не выявило различия по показателям аллельного разнообразия, однако в ряде сравнений были обнаружены достоверные различия между выборкой из Амурского лимана и выборками из рек Тумнин, Амур, Тугур и Кухтуй (р = 0.007).
Таблица 2.
Генетическая изменчивость популяций азиатской корюшки Osmerus dentex по микросателлитным локусам
| Локус | Показатель | Выборка | В среднем | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| SKO | BOT | SAH | TUM | AMU | ALI | SAZ | TUG | KUH | AMH | |||
| MGPL-Omo1 | n | 32 | 50 | 37 | 30 | 49 | 46 | 29 | 50 | 50 | 84 | 45.7 |
| A/Ar | 9/8.7 | 9/8 | 9/8.7 | 8/8 | 9/8.5 | 8/7.6 | 8/8 | 10/9.5 | 10/9.8 | 12/9.6 | 9.2/9.1 | |
| HE | 0.839 | 0.802 | 0.826 | 0.826 | 0.830 | 0.818 | 0.868 | 0.864 | 0.877 | 0.865 | 0.841 | |
| HO | 0.781 | 0.800 | 0.837 | 0.800 | 0.816 | 0.804 | 0.896 | 0.840 | 0.860 | 0.928 | 0.836 | |
| MGPL-Omo2 | n | 32 | 50 | 37 | 30 | 49 | 45 | 29 | 50 | 50 | 89 | 46.1 |
| A/Ar | 5/4.9 | 8/6.5 | 7/6.7 | 6/5.9 | 8/6.8 | 7/6.4 | 7/7 | 8/7.1 | 6/5.5 | 8/5.1 | 7.0/6.4 | |
| HE | 0.399 | 0.426 | 0.420 | 0.451 | 0.406 | 0.403 | 0.340 | 0.516 | 0.425 | 0.335 | 0.412 | |
| HO | 0.406 | 0.500 | 0.405 | 0.500 | 0.387 | 0.400 | 0.310 | 0.500 | 0.400 | 0.325 | 0.421 | |
| MGPL-Omo3 | n | 32 | 50 | 37 | 30 | 49 | 46 | 29 | 50 | 50 | 88 | 46.1 |
| A/Ar | 7/6.9 | 8/7.6 | 5/4.9 | 6/5.9 | 6/5.2 | 6/5.3 | 6/6 | 7/6.2 | 8/6.7 | 8/5.9 | 6.7/6.5 | |
| HE | 0.781 | 0.749 | 0.632 | 0.536 | 0.475 | 0.451 | 0.543 | 0.594 | 0.596 | 0.504 | 0.586 | |
| HO | 0.687 | 0.860 | 0.567 | 0.500 | 0.530 | 0.369 | 0.655 | 0.560 | 0.580 | 0.466 | 0.577 | |
| MGPL-Omo4 | n | 32 | 50 | 35 | 30 | 49 | 46 | 29 | 49 | 50 | 89 | 45.9 |
| A/Ar | 5/4.9 | 6/5.2 | 6/5.9 | 7/6.9 | 9/7.9 | 8/7.5 | 7/7 | 7/6.9 | 7/6.4 | 8/6.5 | 7.0/6.8 | |
| HE | 0.660 | 0.656 | 0.766 | 0.742 | 0.748 | 0.728 | 0.740 | 0.735 | 0.712 | 0.717 | 0.720 | |
| HO | 0.718 | 0.760 | 0.742 | 0.667 | 0.693 | 0.760 | 0.724 | 0.632 | 0.640 | 0.741 | 0.708 | |
| MGPL-Omo5 | n | 32 | 50 | 36 | 30 | 49 | 46 | 29 | 50 | 50 | 85 | 45.7 |
| A/Ar | 11/10.7 | 12/11.3 | 12/10.8 | 12/11.9 | 11/10.3 | 13/11.3 | 12/12 | 10/9.4 | 14/12.6 | 12/10.7 | 11.9/11.3 | |
| HE | 0.812 | 0.867 | 0.693 | 0.806 | 0.813 | 0.763 | 0.796 | 0.825 | 0.846 | 0.801 | 0.802 | |
| HO | 0.750 | 0.960 | 0.694 | 0.833 | 0.795 | 0.739 | 0.862 | 0.860 | 0.860 | 0.788 | 0.814 | |
| MGPL-Omo15 | n | 32 | 50 | 37 | 30 | 49 | 45 | 29 | 50 | 50 | 90 | 46.2 |
| A/Ar | 5/4.8 | 3/2.9 | 3/2.9 | 3/3 | 5/4.7 | 5/4.2 | 3/3 | 4/3.9 | 4/3.8 | 5/3.2 | 4.0/3.9 | |
| HE | 0.283 | 0.266 | 0.323 | 0.558 | 0.578 | 0.547 | 0.532 | 0.569 | 0.538 | 0.523 | 0.472 | |
| HO | 0.250 | 0.300 | 0.351 | 0.433 | 0.571 | 0.600 | 0.586 | 0.560 | 0.560 | 0.611 | 0.482 | |
| MGPL-Omo16 | n | 32 | 50 | 37 | 30 | 49 | 45 | 29 | 50 | 49 | 89 | 46.0 |
| A/Ar | 6/5.9 | 8/7.6 | 5/4.9 | 6/5.9 | 6/5.2 | 5/4.6 | 6/6 | 6/5.6 | 7/6.2 | 8/5.8 | 6.3/6.5 | |
| HE | 0.776 | 0.771 | 0.607 | 0.536 | 0.450 | 0.441 | 0.543 | 0.582 | 0.580 | 0.510 | 0.580 | |
| HO | 0.656 | 0.860 | 0.540 | 0.500 | 0.530 | 0.377 | 0.655 | 0.580 | 0.571 | 0.483 | 0.575 | |
| Tpa119 | n | 31 | 50 | 34 | 30 | 49 | 46 | 29 | 33 | 50 | 90 | 44.2 |
| A/Ar | 4/4 | 4/3.6 | 3/3 | 4/4 | 4/3.5 | 2/2 | 3/3 | 2/2 | 3/2.8 | 5/3.8 | 3.4/3.8 | |
| HE | 0.659 | 0.237 | 0.509 | 0.300 | 0.173 | 0.160 | 0.303 | 0.167 | 0.248 | 0.267 | 0.302 | |
| HO | 0.483 | 0.260 | 0.392 | 0.233 | 0.142 | 0.130 | 0.206 | 0.061 | 0.160 | 0.266 | 0.231 | |
| HtrG107 | n | 32 | 50 | 37 | 30 | 49 | 46 | 29 | 50 | 50 | 91 | 46.4 |
| A/Ar | 2/1.9 | 3/2.9 | 3/2.8 | 2/2 | 2/2 | 2/1.9 | 2/2 | 2/2 | 2/1.9 | 4/2.8 | 2.4/2.4 | |
| HE | 0.031 | 0.133 | 0.219 | 0.126 | 0.151 | 0.084 | 0.099 | 0.243 | 0.131 | 0.154 | 0.137 | |
| HO | 0.031 | 0.140 | 0.189 | 0.133 | 0.163 | 0.086 | 0.103 | 0.240 | 0.140 | 0.164 | 0.139 | |
| В среднем | n | 31.8 | 50.0 | 36.3 | 30.0 | 49.0 | 45.6 | 29.0 | 48.0 | 49.8 | 88.3 | 45.8 |
| A/Ar | 6.0/5.9 | 6.7/6.2 | 5.8/5.7 | 6.0/6.0 | 6.7/6.0 | 6.2/5.7 | 6.0/6.0 | 6.2/5.8 | 6.7/6.2 | 7.7/5.9 | 6.4/6.3 | |
| HE | 0.582 | 0.545 | 0.555 | 0.542 | 0.514 | 0.488 | 0.529 | 0.566 | 0.550 | 0.519 | 0.539 | |
| HO | 0.529 | 0.504 | 0.523 | 0.511 | 0.514 | 0.474 | 0.555 | 0.546 | 0.530 | 0.529 | 0.531 | |
Общая оценка генетической дифференциации корюшки из исследованного ареала была достоверна (FST = 0.033 c 95%-м доверительным бутстреп-интервалом (0.015–0.058)). Индивидуальные локусспецифичные оценки изменялись от 0.005 (MGPL-Omo2) до 0.114 (MGPL-Omo15) и были достоверно значимы для каждого локуса.
Достоверная дифференциация между выборками была обнаружена и при их попарном сравнении в 31 случае из 45 (табл. 3). Наибольшие различия выявлены между выборками SKO, BOT и SAH и всеми остальными. Выборка AMH значимо отличается от всех выборок, кроме TUG и KUH. Также достоверны различия при сравнении TUG и AMU, и KUH с AMU и ALI. После коррекции Бонферрони достоверны отличия SKO, BOT и SAH от остальных, а также различия между AMU и AMH. Максимально по оценкам FST дифференцирована от остальных выборка из Японского моря SKO.
Таблица 3.
Коэффициенты генетической дифференциации (FST) по микросателлитным локусам между выборками азиатской корюшки Osmerus dentex при их попарном сравнении (под диагональю) и наличие (+) статистически достоверных различий между выборками после коррекции Бонферрони (над диагональю)
| Выборка | SKO | BOT | SAH | TUM | AMU | ALI | SAZ | TUG | KUH | AMH |
|---|---|---|---|---|---|---|---|---|---|---|
| SKO | + | + | + | + | + | + | + | + | + | |
| BOT | 0.0313 | + | + | + | + | + | + | + | + | |
| SAH | 0.0323 | 0.0298 | + | + | + | + | + | + | + | |
| TUM | 0.0704 | 0.0409 | 0.0113 | |||||||
| AMU | 0.0954 | 0.0549 | 0.0309 | 0.0003 | + | |||||
| ALI | 0.1076 | 0.0691 | 0.0369 | 0.0015 | −0.0037 | |||||
| SAZ | 0.0822 | 0.0571 | 0.0220 | −0.0002 | 0.0005 | −0.0029 | ||||
| TUG | 0.0946 | 0.0655 | 0.0431 | 0.0061 | 0.0080 | 0.0070 | 0.0074 | |||
| KUH | 0.0897 | 0.0612 | 0.0427 | 0.0061 | 0.0096 | 0.0072 | 0.0076 | −0.0032 | ||
| AMH | 0.1094 | 0.0758 | 0.0456 | 0.0072 | 0.0102 | 0.0062 | 0.0069 | 0.0034 | 0.0018 |
Результаты кластеризации в программе STRUCTURE показывают наиболее вероятное число кластеров К = 2 по оценкам MedMeaK, MaxMeaK, MedMedK и MaxMedK (рис. 2), которые сформированы выборками: 1) SKO, BOT, SAH и 2) TUM, AMU, ALI, SAZ, TUG, KUH, AMH. Индивидуальные генотипы корюшки Сахалина (SAH) распределены между кластерами практически поровну (51 : 49%). Максимальное значение ∆К выявлено при кластеризации на три группы, при этом чёткая структура не наблюдается, и к третьему кластеру нельзя отнести ни одну из выборок.
Рис. 2.
Кластерный анализ выборок Osmerus dentex на основании программы STRUCTURE. Разная заливка соответствует разным кластерам и отображает вероятность принадлежности к ним особей. Первый раунд кластеризации: а – K = 2 по оценкам MedMeaK, MaxMeaK, MedMedK и MaxMedK; б – К = 3 по оценке ∆К; второй раунд кластеризации: в – К = 2 по оценкам MedMeaK, MaxMeaK, MedMedK и MaxMedK; г – К = 3 по оценке ∆К.
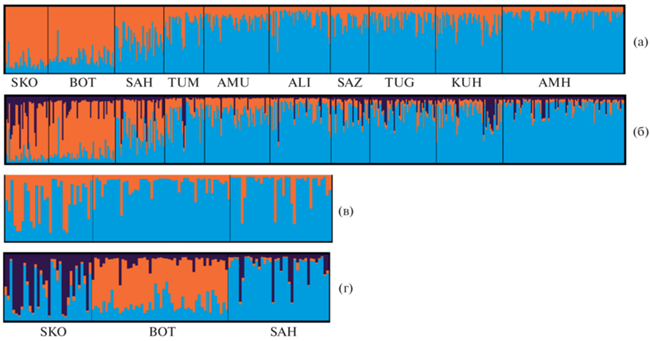
Дальнейший анализ в пределах двух выявленных кластеров не позволяет обнаружить генетическую структурированность во втором кластере. А в кластере SKO–BOT–SAH выявляется подразделение на две группы на основании оценок MedMeaK, MaxMeaK, MedMedK и MaxMedK и на три группы − по ∆К. При этом выборка из р. Ботчи наиболее обособлена (рис. 2). Таким образом, структурированность корюшки из исследованного ареала можно описать четырьмя кластерами, сформированными выборками из рек Шкотовка (1), Ботчи (2) и Найба (Сахалин) (3), а также всеми остальными выборками (4).
Анализ на наличие изоляции расстоянием показал, что корреляция между географическими расстояниями и генетической дифференциацией корюшек достоверна для всей совокупности выборок (Мантел-тест: R2 = 0.52, р = 0.001).
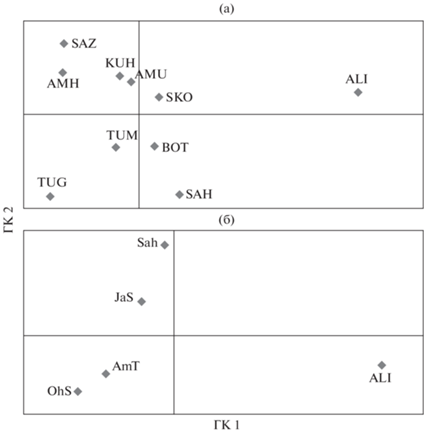
Многомерное шкалирование на основе матрицы попарных дистанций FST (табл. 3) представлено на рис. 3. На диаграмме выделяются кластеры генетически близких выборок из рек Тумнин, Амур, Амурского лимана и Сахалинского залива, а также кластер Амахтонского залива и двух наиболее близких выборок из рек Тугур и Кухтуй. Отдельные популяции рек Шкотовка, Ботчи и Найба (Сахалин) наиболее дифференцированы от остальных. В целом расположение точек, соответствующих выборкам, на матрице соответствует их географическому положению.
Анализ контрольного региона мтДНК
После выравнивания 54 последовательностей в проанализированном участке длиной 940 пар нуклеотидов, содержащем полную последовательность CR мтДНК, было выявлено 16 вариабельных сайтов, из них 11 транзиций и 5 трансверсий. Всего было выделено 20 гаплотипов с инделами и 18 без учёта инделов, все гаплотипы были выявлены впервые, кроме Hap_1, который совпадал с последовательностями CR из Генбанка (MH370836, MH370837), принадлежащими азиатской корюшке.
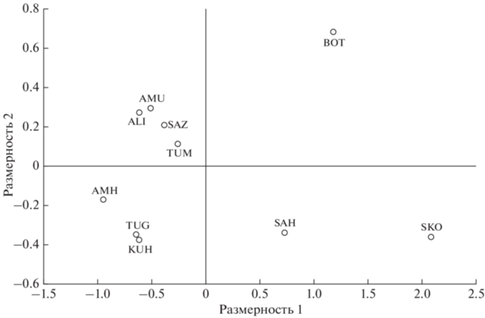
По расположению точек, соответствующих выборкам корюшки, на диаграмме многомерного шкалирования (рис. 4) прослеживается обособленность выборки ALI (Амурского лимана), причиной чего могут быть случайные факторы, связанные с её небольшим объёмом. Для проведения дальнейшего анализа выборки корюшки были объединены в три группы согласно их географическому положению, с учётом результатов многомерного шкалирования на основании микросателлитных локусов (рис. 3), а также применения метода главных компонент (рис. 4): OhS – группа северного побережья Охотского моря (TUG, KUH, AMH), AmT – группа западного материкового побережья Охотского моря (AMU, SAZ, TUM), JaS – выборки из Японского моря (BOT, SKO). Отдельно оценивали изменчивость выборки из р. Найба юго-восточного побережья Сахалина (SAH) и выборки из Амурского лимана (ALI).
Рис. 4.
График анализа главных компонент (ГК) на основе ковариационной матрицы попарных генетических дистанций контрольного региона (CR) мтДНК Osmerus dentex: а – расположение выборок азиатской корюшки в пространстве двух первых осей; ГК 1 описывает 52.2, ГК 2 – 27.5% общей изменчивости; б – расположение групп выборок азиатской корюшки по региону происхождения в пространстве двух первых осей по отношению к выборке ALI; ГК 1 – 80.9, ГК 2 – 18.5% общей изменчивости. Группы: OhS, AmT – соответственно северное (выборки TUG, KUH, AMH) и западное материковое (AMU, SAZ, TUM) побережья Охотского моря, JaS – Японское море (BOT, SKO), Sah – о. Сахалин (SAH).
Самые низкие значения всех параметров генетического разнообразия наблюдались в группе OhS (табл. 4). Наибольшее значение гаплотипического разнообразия (Hd) было выявлено в группе Японского моря (JaS), несколько меньшие значения Hd у корюшки о-ва Сахалин (SAH), остальные значения генетического разнообразия (h, Pi, k) были максимальными в выборке ALI и в группе AmT.
Таблица 4.
Характеристика генетического разнообразия в группах Osmerus dentex на основе гаплотипов контрольного региона мтДНК
| Группа, выборка | n | h | hu | S | k | Pi | Hd ± SD |
|---|---|---|---|---|---|---|---|
| OhS | 15 | 5 | 2 | 3 | 0.933 | 0.00100 | 0.695 ± 0.109 |
| AmT | 17 | 10 | 5 | 8 | 1.412 | 0.00151 | 0.864 ± 0.070 |
| SAH | 9 | 6 | 1 | 4 | 1.500 | 0.00160 | 0.889 ± 0.091 |
| JaS | 9 | 7 | 2 | 6 | 1.500 | 0.00160 | 0.917 ± 0.092 |
| ALI | 4 | 3 | 2 | 17 | 3.167 | 0.00338 | 0.833 ± 0.222 |
| Все выборки | 54 | 18 | 16 | 1.533 | 0.00164 | 0.842 ± 0.043 |
Примечание. n – число исследованных образцов, h – число гаплотипов, hu – число уникальных гаплотипов, S – число сегрегирующих сайтов, k – среднее число нуклеотидных различий, Pi – нуклеотидное разнообразие, Hd ± SD – гаплотипическое разнообразие и стандартное отклонение. Здесь и в табл. 5 показаны группы по региону происхождения: OhS, AmT – соответственно северное (выборки TUG, KUH, AMH) и западное материковое (AMU, SAZ, TUM) побережья Охотского моря, JaS – Японское море (BOT, SKO).
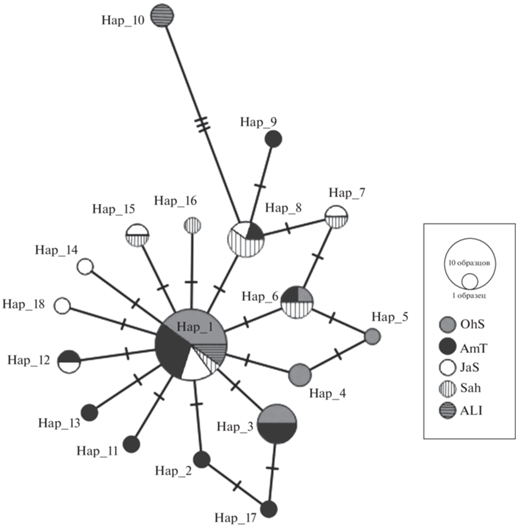
В структуре сети гаплотипов выявляется звёздообразная топология (рис. 5) с центральным, наиболее часто встречающимся, гаплотипом Hap_1, который представлен во всех регионах, и 12 гаплотипами, отличающимися от Hap_1 на одну замену.
Рис. 5.
Сеть minimum spanning network гаплотипов CR мтДНК Osmerus dentex (размер кружков соответствует числу образцов): Hap_1–Hap_18 – номера гаплотипов, число штрихов на линиях соединения гаплотипов – число нуклеотидных замен, разной заливкой обозначены регионы происхождения. Ост. обозначения см. на рис. 4.
Наибольшие значения дифференциации ΦPT наблюдались между группами выборок корюшки OhS и SAH, наименьшие – между JaS и SAH (табл. 5). При проведении данного анализа выборка ALI была исключена, как малочисленная и сильно отклоняющаяся по результатам PCA. На внутривыборочную изменчивость (ΦPT) приходилось 92% общей изменчивости (p < 0.05), на изменчивость между выборками внутри групп (ΦPR) и между группами (ΦRT) – по 4% (p > 0.05). Для последующего анализа AMOVA группы были объединены в две: “север” (Ohs–AmT) и “юг” (JaS–SAH), в этом случае на внутривыборочную изменчивость (ΦPT) приходилось 90.6% общей (p < 0.05), на изменчивость между выборками внутри регионов (ΦPR) и между регионами (ΦRT) – соответственно 6.7 и 2.7% (p > 0.05).
Таблица 5.
Генетическая дифференциация (ΦPT) (под диагональю) и значения потока генов (Nm) (над диагональю) между группами выборок Osmerus dentex на основе контрольного региона мтДНК
| Группа, выборка | OhS | AmT | JaS | SAH |
|---|---|---|---|---|
| OhS | 84.25 | 7.93 | 3.13 | |
| AmT | 0.006 | >100 | 5.07 | |
| JaS | 0.059 | 0.005 | >100 | |
| SAH | 0.138* | 0.090* | 0.001 |
Поток генов (Nm) (табл. 5) в большинстве попарных сравнений между группами был > 4, что соответствует критерию высокого. Так, критерии для значений потока генов определяются как низкие для Nm < 1, средние − для 1 < Nm < 4 и высокие − для Nm > 4 (Boivin et al., 2004). Только в одной паре сравнений между OhS и SAH поток генов можно полагать средним – 3.13. При этом оценка Nm между “севером” и “югом” – 6.37, что также соответствует высоким значениям потока генов.
ОБСУЖДЕНИЕ
Высокие показатели гаплотипического разнообразия в сочетании с низкими значениями нуклеотидного разнообразия выборок азиатской корюшки, выявленные в нашем исследовании на основании CR мтДНК, в целом соответствуют показателям молекулярной изменчивости других участков мтДНК этого вида рыб (Скурихина и др., 2015; Skurikhina et al, 2018).
Звёздообразная топология генеалогических структур (рис. 4) указывает на характерную для многих бореальных видов рыб быструю экспансию численности и происхождение всех популяций азиатской корюшки от единого общего предка из основного рефугиума, которые совершились после периода сокращения численности популяции, связанной с плейстоценовыми оледенениями (Slatkin, Hudson, 1991; Hewitt, 2000). Низкое нуклеотидное разнообразие также может быть свидетельством быстрой популяционной экспансии (Hirase, 2022).
Наши данные показали, что распространение корюшки осуществлялось, наиболее вероятно, из области в районе Амура, поскольку большинство показателей генетического разнообразия и число гаплотипов максимальны у выборок из этого района (табл. 4). Как установлено ранее (Costello et al., 2003; Stamford, Taylor, 2004), наибольшее генетическое разнообразие видов встречается в регионах, расположенных в местах предполагаемых предковых рефугиумов.
Не исключено существование и других рефугиумов на о-ве Сахалин или в Японском море. На это указывают общие гаплотипы для этих выборок. При этом в сахалинской выборке доминируют гаплотипы, общие с выборкой из Японского моря, а не гаплотип Hap_1, как в других регионах. Для подтверждения этого предположения требуются более обширные геногеографические исследования юго-восточных популяций острова и Японского моря.
Исследование митохондриальных генов Cyt b и COI (Skurikhina et al., 2018) позволило предположить основным районом, из которого происходило распространение корюшки, южную часть Охотского моря и Японское море. Также было показано (Брыков и др., 2003; Олейник и др., 2005), что эти географические локации могли служить рефугиумом в период оледенения и для некоторых видов лососёвых рыб.
Современная популяционно-генетическая структура корюшки соответствует модели изоляции расстоянием (Wright, 1943). То есть в целом генетическая дифференциация растёт с увеличением географической дистанции между популяциями. При этом выявлены зоны ограниченного генного потока, свидетельствующие о том, что степень связи популяций азиатской корюшки между собой неодинакова на разных участках ареала.
Для многих видов рыб, в том числе и для корюшковых, показано, что формирование структуры популяции в большей степени ограничено процессами, влияющими на расселение организмов ранних стадий развития, чем миграционными способностями взрослых рыб (Iles, Sinclair, 1982; Baby et al., 1991; Bernatchez, Martin, 1996). На степень распространения личинок рыб от устьевых пространств главным образом оказывают воздействие комплексные гидрографические факторы среды, прежде всего, направление и сила течений и ветров, физические барьеры, особенности рельефа (O’Donnell, 1993; Bradbury et al., 2006, 2008). Региональные различия в действии таких факторов могут приводить к существенным различиям в популяционной структуре рыб в пределах их ареала. Наши предыдущие исследования позволили предположить значительную роль течений и других гидрофизических свойств водоёмов в формировании популяционно-генетической структуры азиатской корюшки в водах Камчатки и Белого моря (Семенова и др., 2019а, 2019б; Semenova et al., 2021).
Несмотря на значительные географические расстояния, обмен генами между популяциями корюшки северного и северо-западного побережий Охотского моря и Татарского пролива Японского моря весьма значителен, что подтверждается результатами анализа двух типов маркеров. При этом кластеризация методом Байеса по STR-локусам свидетельствует о генетическом единстве корюшки в этом ареале (рис. 2). Подтверждают этот вывод и данные генетической дифференциации (ΦPT) по частотам гаплотипов CR мтДНК (табл. 5). Однако классические методы дифференциации на основании показателей FST обнаруживают достоверные различия между наиболее географически удалённой выборкой из Амахтонского залива (северное побережье Охотского моря) и выборками из рек Амур, Тумнин, Амурского лимана и Сахалинского залива. Отличаются от последней группы в некоторых случаях и другие выборки материкового побережья Охотского моря – из рек Тугур и Кухтуй (табл. 3, рис. 2). Кроме того, выборка из Амурского лимана отличается от остальных на основании последовательностей мтДНК (рис. 4), однако для выявления причин этого требуется анализ выборок большего объёма.
По данным акустических съёмок и анализа биологических особенностей корюшки, северная часть Татарского пролива (севернее 49° с.ш.) и Амурский лиман – это зона нагула так называемой “амурской” группировки – комплексного стада азиатской корюшки, основу которого составляет популяция Амура, а также множество популяций отдельных рек, которые впадают в Амурский лиман и северную часть Татарского пролива как сахалинского, так и материкового побережий (Щукина, 1999). Наши данные по генетической дифференциации корюшки по STR-локусам хорошо согласуются с выводами автора о единстве популяции рек северной части Татарского пролива и Амурского лимана материкового побережья. Отсутствие в нашем анализе выборок из рек Западного Сахалина не позволяет оценить генетическую изменчивость этих нерестовых стад. Однако можно предположить, что представленные в данном исследовании выборки из Амурского лимана и Сахалинского залива (нагульные скопления молоди двух–четырёхлетнего возраста) могут быть сформированы корюшкой из многих рек, в том числе и Западного Сахалина.
Генетическому единству корюшки данной области может способствовать широкое распространение личинок и мальков благодаря выносу вод Амура как в Охотское море через Сахалинский залив, так и частично на юг, через пролив Невельского в северную часть Татарского пролива Японского моря. К сожалению, нет научной информации о распространении личинок корюшки в исследуемом районе, но исследования течений косвенно свидетельствуют о потенциальной возможности пассивного разноса ихтиопланктона в Охотском и Японском морях. Показано, что в весенне-летний период в северной части Татарского пролива (севернее 49° с.ш.) действуют как циркуляционные циклонические течения, так и антициклонические (Пищальник и др., 2010), формируемые в том числе и водами Амура. Эти течения могут способствовать распространению личинок корюшки из Охотского моря через пролив Невельского на довольно значительные расстояния в Татарском проливе Японского моря (рис. 1). Полное репродуктивное единство популяции р. Тумнин, впадающей в северную часть Татарского пролива, и р. Амур свидетельствует в пользу этой гипотезы и предполагает существование единого личиночного ареала для популяций корюшки северной части Татарского пролива и Амура.
Очевидно, что зона распространения амурской группировки простирается и на север, в Сахалинский залив, а также вдоль материкового побережья Охотского моря. Воды р. Амур через Амурский лиман в весенне-летний период распространяются преимущественно в Сахалинский залив и далее могут достигать северного побережья Охотского моря, формируя вместе с многочисленными реками материкового побережья (Уда, Охота, Тугур, Иня, Кухтуй) прибрежное течение в направлении с севера на юг (Рогачев, Шлык, 2015), совпадающее с направлением циклонического Северо-Охотского течения (Власова и др., 2008) (рис. 1). Такая система течений может способствовать переносу личинок корюшки на значительные расстояния, как, например, это показано для личинок камчатского краба Paralithodes camtschaticus (Черниенко, 2010).
Наблюдаемые генетические различия не позволяют предполагать абсолютного единства личиночного ареала для корюшки из Охотского моря и амурской комплексной группировки. Однако наличие постоянных течений, особенно в весенне-летний период, по всей вероятности, способствует пассивным миграциям корюшки ранних стадий развития, частично нивелирующим репродуктивную обособленность локальных популяций. Прибрежное течение у северного побережья Охотского моря начинается от п-ова Лисянского (район г. Охотск) и продолжается до Шантарского архипелага (Рогачев, Шлык, 2015). При этом Тауйская губа, частью которого является Амахтонский залив, находится восточнее и не затрагивается этим течением, что может быть объяснением бóльших генетических отличий выборки AMH от остальных выборок из Охотского моря.
Наряду с генетическим единообразием корюшки на протяжённом ареале в Охотском море и в северной части Татарского пролива наблюдается довольно значительный уровень генетической обособленности популяции корюшки из р. Ботчи. Река расположена на небольшом расстоянии от р. Тумнин (~160 км), однако популяции корюшки этих рек различаются с высокой степенью достоверности (FST > 4%, p < 0.0001). При этом географическое расстояние между генетически едиными популяциями из рек Тумнин и Амур более чем в два раза больше. Помимо классических оценок дифференциации обособленность р. Ботчи подтверждается и байесовскими методами анализа (табл. 3, рис. 2).
Можно предположить, что гидрологические факторы играют первостепенную роль в наблюдаемой картине пространственной дифференциации корюшки. Устье р. Ботчи относится к южной части Татарского пролива, более глубоководной по сравнению с северной. Условная граница между северной и южной частями Татарского пролива проходит в области 48°03′ с.ш. Основной фактор, формирующий циркуляцию вод в южной части Татарского пролива, – это Цусимское течение, проходящее с юга на север вдоль Сахалина. На уровне 47°–48° с.ш. из-за резкого уменьшения глубины моря в этом районе основная часть потока Цусимского течения поворачивает на запад и, достигая материкового побережья, движется вдоль берега на юг как Приморское течение, в результате формируя мощный циклонический круговорот вод (Пищальник и др., 2010) (рис. 1).
Вероятно, что данный круговорот препятствует обмену личинок и молоди корюшки между южной и северной частями Татарского пролива. Интересно, что и для горбуши из р. Ботчи была отмечена обособленность от выборок как из Охотского, так и из Японского морей (Шпигальская и др., 2012).
Наибольшая степень генетической дивергенции показана для выборки азиатской корюшки из Уссурийского залива Японского моря (SKO) (табл. 3, рис. 2). Такая дифференциация кажется вполне обоснованной в связи с пространственно обособленным положением выборки на исследованном ареале, поскольку корюшка практически отсутствует в реках южной части Северного Приморья (от м. Поворотный до м. Егорова) (Парпура, Колпаков, 2001).
Также значимая степень генетической дивергенции выявлена для выборки из р. Найба Юго-Восточного Сахалина (SAH). По данным акустических съёмок и биологическим показателям, локальная популяция корюшки р. Найба является частью комплексной популяции, основной нагульный ареал которой находится в зал. Терпения (Щукина, 1999). Основу этой популяции составляют рыбы крупной “материнской” р. Поронай, в состав её входят представители ещё до десятка небольших речек Восточного Сахалина вплоть до 47.03° с.ш. Кластеризация выборки из р. Найба свидетельствует о генетических связях этой группировки как с рыбами амурской комплексной группировки, так и с представителями Японского моря (рис. 2).
Полученным данным о генетической обособленности выборок из рек Шкотовка, Ботчи и Найба от выборок из Охотского моря по STR-маркерам не противоречат результаты иерархического анализа по частотам гаплотипов CR мтДНК (табл. 5). Наиболее вероятным является разделение азиатской корюшки на группу выборок Японского моря и Сахалина и остальные выборки из Охотского моря и северной части Татарского пролива. При этом между ними существует высокий обмен генами.
Дифференциация азиатской корюшки Японского моря от охотоморских выборок была показана ранее по другим маркерам мтДНК (Ковпак и др., 2011; Скурихина и др., 2015; Skurikhina et al., 2018).
Общий уровень генетической дифференциации корюшки по микросателлитным локусам (FST = = 0.033), выявленный в настоящем исследовании, сходен с дифференциацией корюшки из водоёмов восточного и западного побережий Камчатки, т.е. Берингова и Охотского морей (FST = 0.028), и более чем в два раза меньше дифференциации корюшки в водах Белого моря (FST = 0.078) (Семенова и др., 2019а, 2019б). При сравнении генетической дифференциации азиатской корюшки и близкородственных видов рыб с аналогичной жизненной историей, например O. mordax из вод США и Канады, наблюдаются сходные паттерны генетической дивергенции, варьирующие в зависимости от исследованного ареала (Bradbury et al., 2006, 2011; Kovach et al., 2013).
При этом очевидно, что за исключением зон ограниченного обмена у азиатской корюшки на довольно большом расстоянии поток генов весьма значителен. Так, генетические различия в пределах кластеров STRUCTURE (FST ≈ 0.004) сравнимы с дифференциацией в пределах своего ареала у Thaleichthys pacificus – рыбы семейства корюшковых, для которой отмечен очень низкий уровень генетической дифференциации (McLean, Taylor, 2001; Beacham et al., 2005; Flannery et al., 2013).
Список литературы
Андреев А.Г. 2018. Особенности циркуляции вод в южной части Татарского пролива // Исслед. Земли из космоса. № 1. С. 3–11. https://doi.org/10.7868/S0205961418010013
Брыков В.А., Полякова Н.Е., Прохорова А.В. 2003. Филогеографический анализ кеты Oncorhynchus keta (Walbaum) в азиатской части ареала, основанный на изменчивости митохондриальной ДНК // Генетика. Т. 39. № 1. С. 75–82.
Бугаев А.В., Амельченко Ю.Н., Липнягов С.В. 2014. Азиатская зубастая корюшка Osmerus mordax dentex в шельфовой зоне и внутренних водоемах Камчатки: состояние запасов, промысел и биологическая структура // Изв. ТИНРО. Т. 178. С. 3–24. https://doi.org/10.26428/1606-9919-2014-178-3-24
Василец П.М. 2000. Корюшки прибрежных вод Камчатки: Автореф. дис … канд. биол. наук. Владивосток: ИБМ ДВО РАН, 24 с.
Власова Г.А., Васильев А.С., Шевченко Г.В. 2008. Пространственно-временная изменчивость структуры и динамики вод Охотского моря. М.: Наука, 359 с.
Гладенков А.Ю., Гладенков Ю.Б. 2004. Начало формирования межокеанических связей Пацифики и Арктики через Берингов пролив в неогене // Стратиграфия и геол. корреляция. Т. 12. № 2. С. 72–89.
Гриценко О.Ф. 2002. Проходные рыбы острова Сахалин (систематика, экология, промысел). М.: Изд-во ВНИРО, 248 с.
Дьяков Б.С. 2006. О циркуляции вод в Татарском проливе в весеннее время // Изв. ТИНРО. Т. 146. С. 205–212.
Клюканов В.А. 1975. Систематика и родственные отношения корюшек родов Osmerus и Hypomesus (Osmeridae) и их расселение // Зоол. журн. Т. 54. Вып. 4. С. 590–595.
Ковпак Н.Е., Скурихина Л.А., Кухлевский А.Д. и др. 2011. Генетическое разнообразие и родственные взаимоотношения корюшек рода Osmerus российских вод // Генетика. Т. 47. № 8. С. 1081–1096.
Олейник А.Г., Скурихина Л.А., Брыков В.А. и др. 2005. Дифференциация мальмы Salvelinus malma Азии и Северной Америки по данным ПЦР-ПДРФ-анализа митохондриальной ДНК // Там же. Т. 41. № 5. С. 626–634.
Парпура И.З., Колпаков В.Н. 2001. Биология и внутривидовая дифференциация корюшек Приморья // Чтения памяти В.Я. Леванидова. Вып. 1. С. 284–295.
Пищальник В.М., Архипкин В.С., Леонов А.В. 2010. О циркуляции вод в Татарском проливе // Вод. ресурсы. Т. 37. № 6. С. 657–670.
Рогачев К.А., Шлык Н.В. 2015. Исключительное опреснение прибрежных вод северо-западной части Охотского моря в 2013 г. // Вестн. ДВО РАН. № 2 (180). С. 118–125.
Семенова А.В., Строганов А.Н., Бугаев А.В. и др. 2019а. Анализ изменчивости микросателлитных локусов в популяциях азиатской зубастой корюшки Osmerus dentex Восточной и Западной Камчатки // Генетика. Т. 55. № 1. С. 70–80. https://doi.org/10.1134/S0016675819010041
Семенова А.В., Строганов А.Н., Пономарева Е.В., Афанасьев К.И. 2019б. Микросателлитная изменчивость азиатской корюшки Osmerus dentex Белого моря // Там же. Т. 55. № 6. С. 723–727. https://doi.org/10.1134/S001667581906016X
Скурихина Л.А., Олейник. А.Г., Кухлевский А.Д. и др. 2015. Генетическая дифференциация тихоокеанской корюшки Osmerus mordax dentex по данным анализа митохондриальной ДНК // Там же. Т. 51. № 12. С. 1410–1422. https://doi.org/10.7868/S0016675815120115
Хен Г.В., Ванин Н.С., Фигуркин А.Л. 2002. Особенности гидрологических условий в северной части Охотского моря во второй половине 90-х гг. // Изв. ТИНРО. Т. 130. С. 24–43.
Черешнев И.А., Волобуев В.В., Шестаков А.В., Фролов С.В. 2002. Лососевидные рыбы Северо-Востока России. Владивосток: Дальнаука, 496 с.
Черниенко И.С. 2010. Элементы пространственно-функциональной структуры аяно-шантарской популяции камчатского краба (Paralithodes camtschaticus) // Изв. ТИНРО. Т. 163. С. 172–184.
Шпигальская Н.Ю., Пильганчук О.А., Савенков В.В. и др. 2012. Результаты исследований внутривидовой структуры промысловых видов рыб методами популяционной генетики // Исслед. вод. биол. ресурсов Камчатки и сев.-зап. части Тихого океана. Вып. 25. С. 69–97.
Щукина Г.Ф. 1999. Биологические основы внутривидовой дифференциации азиатской корюшки Osmerus mordax dentex в водах Сахалина // Сб. науч. тр. СахНИРО. Т. 2. Южно-Сахалинск: Изд-во СахНИРО. С. 74–84.
Baby M.C., Bernatchez L., Dodson J.J. 1991. Genetic structure and relationships among anadromous and landlocked populations of rainbow smelt, Osmerus mordax, Mitchill, as revealed by mtDNA restriction analysis // J. Fish Biol. V. 39. № sA. P. 61–68. https://doi.org/10.1111/j.1095-8649.1991.tb05068.x
Bandelt H.J., Forster P., Röhl A. 1999. Median-joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. V. 16. № 1. P. 37–48. https://doi.org/10.1093/oxfordjournals.molbev.a026036
Beacham T.D., Hay D.E., Le K.D. 2005. Population structure and stock identification of eulachon (Thaleichthys pacificus), an anadromous smelt, in the Pacific Northwest // Mar. Biotechnol. V. 7. № 4. P. 363–372. https://doi.org/10.1007/s10126-004-4075-0
Bernatchez L., Martin S. 1996. Mitochondrial DNA diversity in anadromous rainbow smelt, Osmerus mordax Mitchill: a genetic assessment of the member-vagrant hypothesis // Can. J. Fish. Aquat. Sci. V. 53. № 2. P. 424–433. https://doi.org/10.1139/f95-180
Boivin T., Bouvier J.C., Beslay D. et al. 2004. Variability in diapause propensity within populations of a temperate insect species: interactions between insecticide resistance genes and photoperiodism // Biol. J. Lin. Soc. V. 83. № 3. P. 341–351. https://doi.org/10.1111/j.1095-8312.2004.00392.x
Bradbury I.R., Coulson M.W., Campana S.E., Bentzen P. 2006. Morphological and genetic differentiation in anadromous smelt Osmerus mordax (Mitchill): disentangling the effects of geography and morphology on gene flow // J. Fish Biol. V. 69. № sc. P. 95–114. https://doi.org/10.1111/j.1095-8649.2006.01263.x
Bradbury I.R., Campana S.E., Bentzen P. 2008. Low genetic connectivity in an estuarine fish with pelagic larvae // Can. J. Fish. Aquat. Sci. V. 65. № 2. P. 147–158. https://doi.org/10.1139/f07-154
Bradbury I.R., Coulson M.W., Campana S.E. et al. 2011. Contemporary nuclear and mitochondrial genetic clines in a north temperate estuarine fish reflect Pleistocene vicariance // Mar. Ecol. Prog. Ser. V. 438. P. 207–218. https://doi.org/10.3354/meps09286
Chapuis M.P., Estoup A. 2007. Microsatellite null alleles and estimation of population differentiation // Mol. Biol. Evol. V. 24. № 3. P. 621–631. https://doi.org/10.1093/molbev/msl191
Costello A.B., Down T.E., Pollard S.M. et al. 2003. The influence of history and contemporary stream hydrology on the evolution of genetic diversity within species: an examination of microsatellite DNA variation in bull trout, Salvelinus confluentus (Pisces: Salmonidae) // Evolution. V. 57. № 2. P. 328–344. https://doi.org/10.1111/j.0014-3820.2003.tb00267.x
Evanno G., Regnaut S., Goudet J. 2005. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study // Mol. Ecol. V. 14. № 8. P. 2611–2620. https://doi.org/10.1111/j.1365-294X.2005.02553.x
Flannery B.G., Spangler R.E., Norcross B.L. et al. 2013. Microsatellite analysis of population structure in Alaska Eulachon with application to mixed-stock analysis // Trans. Am. Fish. Soc. V. 142. № 4. P. 1036–1048. https://doi.org/10.1080/00028487.2013.790841
Hewitt G. 2000. The genetic legacy of the Quaternary ice ages // Nature. V. 405. № 6789. P. 907–913. https://doi.org/10.1038/35016000
Hirase S. 2022. Comparative phylogeography of coastal gobies in the Japanese Archipelago: future perspectives for the study of adaptive divergence and speciation // Ichthyol. Res. V. 69. № 1. P. 1–16. https://doi.org/10.1007/s10228-021-00824-3
Iles T.D., Sinclair M. 1982. Atlantic herring: stock discreteness and abundance // Science. V. 215. № 4533. P. 627–633. https://doi.org/10.1126/science.215.4533.627
Kottelat M., Freyhof J. 2007. Handbook of European Freshwater Fishes. Cornol; Berlin: Kottelat and Freyhof, 646 p.
Kovach A.I., Breton T.S., Enterline C., Berlinsky D.L. 2013. Identifying the spatial scale of population structure in anadromous rainbow smelt (Osmerus mordax) // Fish. Res. V. 141. P. 95–106. https://doi.org/10.1016/j.fishres.2012.07.008
Leigh J.W., Bryant D. 2015. PopART: Full-feature software for haplotype network construction // Methods Ecol. Evol. V. 6. № 9. P. 1110–1116. https://doi.org/10.1111/2041-210X.12410
Librado P., Rozas J. 2009. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data // Bioinformatics. V. 25. № 11. P. 1451–1452. https://doi.org/10.1093/bioinformatics/btp187
McLean J., Taylor E. 2001. Resolution of population structure in a species with high gene flow: microsatellite variation in the eulachon (Osmeridae: Thaleichthys pacificus) // Mar. Biol. V. 139. № 3. P. 411–420. https://doi.org/10.1007/s002270100483
Meirmans P.G. 2006. Using the AMOVA framework to estimate a standardized genetic differentiation measure // Evolution. V. 60. № 11. P. 2399–2402. https://doi.org/10.1111/j.0014-3820.2006.tb01874.x
Nellbring S. 1989. The ecology of smelts (genus Osmerus): A literature review // Nord. J. Freshw. Res. V. 65. P. 116–145.
O’Donnell J. 1993. Surface fronts in estuaries: a review // Estuaries. V. 16. № 1. P. 12–39. https://doi.org/10.2307/1352761
Peakall R., Smouse P.E. 2012. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research an update // Bioinformatics. V. 28. № 19. P. 2537–2539. https://doi.org/10.1093/bioinformatics/bts460
Peakall R., Smouse P.E., Huff D.R. 1995. Evolutionary implications of allozyme and RAPD variation in diploid populations of dioecious buffalograss Buchloë dactyloides // Mol. Ecol. V. 4. № 2. P. 135–147. https://doi.org/10.1111/j.1365-294X.1995.tb00203.x
Piry S., Luikart G., Conuet J.M. 1999. Bottleneck: A computer program for detecting recent reduction in the effective population size using allele frequency data // J. Hered. V. 90. № 4. P. 502–503. https://doi.org/10.1093/jhered/90.4.502
Pritchard J., Stephens M., Donnelly P. 2000. Inference of population structure using multilocus genotype data // Genetics. V. 155. № 2. P. 945–959. https://doi.org/10.1093/genetics/155.2.945
Puechmaille S.J. 2016. The program structure does not reliably recover the correct population structure when sampling is uneven: subsampling and new estimators alleviate the problem // Mol. Ecol. Resour. V. 16. № 3. P. 608–627. https://doi.org/10.1111/1755-0998.12512
Rannala B., Mountain J.L. 1997. Detecting immigration by using multilocus genotypes // Proc. Natl. Acad. Sci. V. 94. № 17. P. 9197–9201. https://doi.org/10.1073/pnas.94.17.9197
Rice W.R. 1989. Analyzing tables of statistical tests // Evolution. V. 43. № 1. P. 223–225. https://doi.org/10.2307/2409177
Sanger F., Nicklen S., Coulson A.R. 1977. DNA sequencing with chain-terminating inhibitors // Proc. Natl. Acad. Sci. V. 74. № 12. P. 5463–5467. https://doi.org/10.1073/pnas.74.12.5463
Semenova A.V., Stroganov A.N., Ponomareva E.V. et al. 2021. Large-scale genetic structure and diversity of Arctic rainbow smelt Osmerus dentex Steindachner et Kner, 1870 throughout its distributional range based on microsatellites // Polar Biol. V. 44. № 5. P. 927–940. https://doi.org/10.1007/s00300-021-02848-x
Skurikhina L.A., Oleinik A.G., Kukhlevsky A.D. et al. 2018. Phylogeography and demographic history of the Pacific smelt Osmerus dentex inferred from mitochondrial DNA variation // Ibid. V. 41. № 5. P. 877–896. https://doi.org/10.1007/s00300-018-2250-4
Slatkin M., Hudson R.R. 1991. Pairwise comparisons of mitochondrial DNA sequences in stable and exponentially growing populations // Genetics. V. 129. № 2. P. 555–562. https://doi.org/10.1093/genetics/129.2.555
Stamford M.D., Taylor E.B. 2004. Phylogeographical lineages of Arctic grayling (Thymallus arcticus) in North America: divergence, origins and affinities with Eurasian Thymallus // Mol. Ecol. V. 13. № 6. P. 1533–1549. https://doi.org/10.1111/j.1365-294X.2004.02174.x
Van Oosterhout C., Hutchinson W.F., Wills D.P., Shipley P. 2004. MICRO-CHECKER: software for identifying and correcting genotyping errors in microsatellite data // Mol. Ecol. Notes. V. 4. № 3. P. 535–538. https://doi.org/10.1111/j.1471-8286.2004.00684.x
Villesen P. 2007. FaBox: an online toolbox for fasta sequences // Ibid. V. 7. № 6. P. 965–968. https://doi.org/10.1111/j.1471-8286.2007.01821.x
Weir B.S., Cockerham C.C. 1984. Estimating F-statistics for the analysis of population structure // Evolution. V. 38. № 6. P. 1358–1370. https://doi.org/10.2307/2408641
Wright S. 1943. Isolation by distance // Genetics. V. 28. № 2. P. 114–138. https://doi.org/10.1093/genetics/28.2.114
Дополнительные материалы
- скачать ESM.docx
- Supplement 1. Distribution of Osmerus dentex haplotypes by sites with sequence numbers deposited in GenBank (NCBI)
Инструменты
Вопросы ихтиологии