Известия РАН. Серия биологическая, 2020, № 5, стр. 482-490
Полиморфизм и филогения гомологов гена ингибитора вакуолярной инвертазы INH2 у видов Solanaceae
М. А. Слугина 1, *, Е. З. Кочиева 1, 2, А. В. Щенникова 1
1 Институт биоинженерии, Федеральный исследовательский центр “Фундаментальные основы биотехнологии” РАН
119071 Москва, Ленинский просп., 33, корп. 2, Россия
2 Московский государственный университет им. М.В. Ломоносова
119991 Москва, Ленинские горы, 1, Россия
* E-mail: mashinmail@mail.ru
Поступила в редакцию 20.02.2019
После доработки 05.11.2019
Принята к публикации 05.11.2019
Аннотация
Определены последовательности и изучен полиморфизм генов-гомологов INH2, кодирующих ингибитор вакуолярной инвертазы, у 37 видов сем. Solanaceae. Выявлен значительный межвидовой полиморфизм кодирующей последовательности INH2 внутри секций Petota и Lycopersicon, что свидетельствует о высокой скорости накопления мутаций, сопровождающей эволюцию генов INH2. Проанализирована структура аминокислотных последовательностей у всех исследуемых гомологов INH2, что позволило обнаружить консервативные мотивы и остатки аминокислот, нетрансмембранный сигнальный пептид и домен PMEI. Отмечена возможность применения последовательности INH2 для определения филогении родов и секций Solanaceae.
Пасленовые (Solanaceae) – одно из крупнейших и наиболее разнообразных семейств двудольных растений, объединяющее 3000–4000 видов, сгруппированных примерно в 90 родов (Gebhardt, 2016). Семейство включает в себя как многолетние деревья, так и однолетние травянистые виды, а география распространения его представителей обширна – от пустынь до тропических лесов (Knapp et al., 2004).
Solanaceae содержит много ценных видов, среди которых можно выделить декоративные (петуния, цеструм, физалис) и лекарственные (белладонна, белена, скополия) растения. Отдельного внимания заслуживают сельскохозяйственно-ценные виды Solanaceae (картофель, томат, перец, баклажан, табак), которые являются одновременно модельными видами для многих клеточных и молекулярно-генетических исследований. Основная цель этих исследований – решение различных агрономических проблем (Gebhardt, 2016).
Обширное генетическое разнообразие видов сем. Solanaceae делает его в целом хорошей модельной системой для изучения эволюции генов, определяющих хозяйственно-ценные признаки (Machida-Hirano, 2015). К таким признакам относятся питательная ценность плодов и клубней, сроки их хранения, особенности созревания, а также устойчивость растений к биотическим и абиотическим стрессам на стадии формирования урожая. Значимые факторы, определяющие данные признаки, – ферменты углеводного обмена, уровень активности которых контролируется специфичными белками-ингибиторами, в частности ингибитором вакуолярной инвертазы INH2 (Brummell et al., 2011).
Ингибитор INH2 относится к семейству небольших белков размером ~17 кДа – пектинметилэстераз (PMEI-RP, PF04043) (Hothorn et al., 2004). INH2 связывается с одним из важнейших ферментов – вакуолярной инвертазой (vacInv/TAI/PAIN-1) (Pressey, 1967; Greiner et al., 1999). Образуется комплекс, в составе которого при определенном значении pH среды вакуолярная инвертаза становится неспособной катализировать осуществляемую ею важнейшую реакцию углеводного обмена – гидролиз сахарозы до редуцирующих сахаров (Di Matteo et al., 2005).
Гены ингибиторов вакуолярной инвертазы INH2 идентифицированы у ряда растений: арабидопсиса (Sherson et al., 2003; Link et al., 2004), кукурузы (Bate et al., 2004), сои, батата, риса (Tymowska-Lalanne, Kreis, 1998; Rausch, Greiner, 2004). У Solanaceae гомологи INH2 охарактеризованы лишь у Nicotiana tabacum, Solanum tuberosum и S. lycopersicum (Greiner et al., 1999; Brummell et al., 2011; Qin et al., 2016). Для многочисленных родственных видов табака, картофеля и томата, а также для других видов пасленовых, отличающихся разнообразием ареала обитания и морфофизиологии, данные о генах INH2 отсутствуют. При этом изучение полиморфизма и филогении генов-гомологов ингибитора вакуолярной инвертазы INH2 может быть важным для прояснения их функций, связей с хозяйственно-ценными признаками и роли в эволюции растений.
Цель работы – идентификация новых гомологов гена INH2, оценка их структурного полиморфизма, а также реконструкция возможной эволюции гена у видов сем. Solanaceae.
МАТЕРИАЛЫ И МЕТОДЫ
В качестве растительного материала использован паслен маврикийский Solanum mauritianum, ДНК которого была выделена из гербарного материала, любезно предоставленного С.Р. Майоровым (кафедра высших растений МГУ им. М.В. Ломоносова). С помощью набора ZR-96 Plant/Seed DNA Kit (Zymo Research, США) выделяли геномную ДНК.
Гомологичные INH2 последовательности у видов Solanaceae определяли как биоинформационными, так и молекулярно-биологическими методами. Идентификация in silico заключалась в проведении выравнивания с помощью программы NCBI-BLAST (https://blast.ncbi.nlm.nih.gov/ Blast.cgi) геномной последовательности гена INH2 картофеля S. tuberosum с доступными в NCBI неаннотированными данными секвенирования геномов (WGS), транскриптомов (TSA) и аннотированными кДНК/генами (NC) представителей Solanaceae. Отбирали полноразмерные гомологичные последовательности с учетом уровня их перекрывания (>60%) и гомологии (>80%). Последовательности, найденные в геномах и транскриптомах, были аннотированы.
Для молекулярной идентификации (амплификации, клонирования и секвенирования) генов-гомологов INH2 подбирали локус-специфичные праймеры на основе выравнивания известных геномных последовательностей у отдаленных видов Solanaceae, относящихся к различным родам, секциям и подсемействам. Ген амплифицировали с помощью разработанной пары праймеров (INH2F: 5'-AACACCTTTAATAATATTCCATTAGG-3'; INF2R: 5'-CTCGTATATATGACGAGAGC-3'). Амплификацию осуществляли при 94°C в течение 10 мин, 35 циклов [94°C 30 с, 55°C 30 с, 72°C 1 мин] и 72°C в течение10 мин. Продукт ПЦР очищали (QIAEX® II Gel Extraction kit, Qiagen, Германия), клонировали в вектор pGEM®-T Easy (Promega, США) и секвенировали (2–4 клона) на ABI Prism 377 DNA Sequencer (Applied Biosystems, США). Последовательность гена была депонирована в NCBI.
Структурный анализ, включая выравнивание последовательностей, поиск полиморфизмов в генах и кодируемых ими белках, проводили с использованием пакета программ MEGA 7.0 (Kumar et al., 2016). Вторичную и третичную структуры белков предсказывали с помощью программы PHYRE2 (Kelley et al., 2015) и визуализировали, используя Chimera-1.11.2 (http://www.cgl.ucsf.edu/ chimera/download.html). Наличие сигнального пептида определяли с использованием SignalP 4.1 (http://www.cbs.dtu.dk./services/SignalP/). Возможное влияние замещений аминокислотных остатков (а. о.) оценивали с помощью PROVEAN (Choi et al., 2012). Консервативные домены и мотивы в белковых последовательностях идентифицировали с помощью NCBI-CDD (https://www.ncbi.nlm.nih.gov/cdd) и MEME 5.0.3 (Bailey, Elkan, 1994; http://meme-suite.org/tools/meme). Филогенетический анализ гомологов белков INH2 проводили с использованием программы MEGA 7.0 методом максимального правдоподобия (ML) с применением модели замен (JTT+G) и бутстрэпа 1000 (Kumar et al., 2016).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Сахароза, основной конечный продукт фотосинтеза у высших растений, распространяется из листьев по всему растению, обеспечивая энергией метаболические процессы и снабжая запасающие органы (Wan et al., 2018). Естественно предположить, что изменения механизма контроля содержания сахарозы в органах и тканях могут лежать в основе эволюционной диверсификации растений. Конечное содержание сахарозы регулируется главным образом балансом активностей инвертаз и сахарозосинтаз, расщепляющих сахарозу, а также ингибиторов инвертаз, препятствующих ее распаду (Wan et al., 2018). Дупликация и последующая функциональная диверсификация предшественников генов, кодирующих данные ферменты, могут составлять основу эволюции высших растений. Изучение полиморфизма данных генов, таким образом, способно найти объяснение морфофизиологическим различиям у исследуемых растений.
Ключевое влияние на состав/содержание сахаров в запасающих органах Solanaceae, таких как клубни (картофель) или плод (томат, перец, баклажан, физалис), должны оказывать в первую очередь вакуолярные ферменты углеводного обмена и их ингибиторы, поскольку главная функция вакуоля – запасающая/накопительная. Поэтому объектом данного исследования стал ген ингибитора вакуолярной инвертазы INH2 у видов сем. Solanaceae.
Идентификация гомологов INH2 у видов Solanaceae. В качестве референса для поиска гомологов INH2 была выбрана наиболее полно охарактеризованная на данный момент последовательность гена INH2 культивируемого картофеля S. tuberosum cv. DM 1-3 516 R44 (INH2a, XM_006349784.2) (табл. 1). Известно, что данный ген представлен одной копией на хромосоме XII (Brummell et al., 2011). Проведенный анализ нуклеотидных коллекций NC (аннотированные последовательности), WGS (данные полногеномного секвенирования) и TSA (данные транскриптомного секвенирования) базы данных NCBI (https://www.ncbi.nlm.nih.gov) выявил у 37 видов Solanaceae последовательности, гомологичные INH2 (84–98% идентичности при перекрытии 66–95% у аминокислотной последовательности), 8 из которых были идентифицированы нами ранее (табл. 1). Часть найденных последовательностей (WGS, TSA) до настоящего времени не была аннотирована, но высокая степень гомологии кодируемых ими продуктов с INH2 S. tuberosum позволила предположить, что именно они кодируют ингибитор вакуолярной инвертазы у исследуемых видов Solanaceae. Полученные последовательности представляли культивируемые и дикорастущие виды томата и картофеля, перца, табака, петунии, физалиса (включая ашваганду) и паслена (включая баклажан) (табл. 1). Кроме того, для проведения сравнительного анализа были идентифицированы in silico и использованы в качестве внешней группы гомолог INH2 из отдаленного вида отдела астеридов – Artemisia annua (сем. Asteraceae), а также гомолог INH1 (SlCIF1) из S. lycopersicum (табл. 1).
Таблица 1.
Характеристика гомологов INH2 использованных в работе видов растений
| Вид/образец | Описание | Размер INH2, кДНК (=ген)/ белок, н./ а.о. | № доступа в базах данных NCBI (WGS, TSA, NC*) или Solgenomics** |
|---|---|---|---|
| Сем. Solanaceae, род Solanum, секция Petota | |||
| S. tuberosum cv. DM 1-3 516 R44 | Культивируемый картофель | 537/178 | WGS: NW_006239038.1:c626714-625903 |
| S. tuberosum ssp. andigenum | То же | 537/178 | ***MK405609 |
| S. verrucosum | Дикорастущие виды картофеля | 543/180 | WGS: FYAG01000235.1:137629-138171 |
| S. oplocense (S. brevicaule)**** | То же | 540/179 | ***MK405610 |
| S. vernei | "" | 537/179 | ***MK405611 |
| S. pampasense (S. candolleanum)**** | "" | 537/179 | ***MK405613 |
| S. demissum | "" | 543/180 | ***MK405614 |
| S. stoloniferum | "" | 531/176 | ***MK405615 |
| S. commersonii cv. cmm1t | "" | 534/177 | WGS: JXZD01120979.1:56187-56720 |
| S. chacoense | "" | 540/179 | ***MK405616 |
| S. pinnatisectum | "" | 537/178 | ***MK405617 |
| Сем. Solanaceae, род Solanum, секция Lycopersicon | |||
| S. lycopersicum cv. Heinz | Культивируемые виды томата | 528/175 | WGS: AEKE02021110.1:369768-371295 (***Solyc12g099190.1 SL2.50 (GCF_000188115.3)) |
| S. lycopersicum var. cerasiforme LA01448 | То же | 528/175 | ** |
| S. pimpinellifolium LA0722 | "" | 528/175 | ** |
| S. cheesmaniae LA0429 | Дикорастущие виды томата | 528/175 | ** |
| S. galapagense LA0528 | То же | 528/175 | ** |
| S. neorickii LA2133 | "" | 528/175 | ** |
| S. chilense LA1969 | "" | 528/175 | ** |
| S. arcanum | "" | 534/177 | WGS: CBYQ010002339.1:23390-23923 |
| S. habrochaites | "" | 534/177 | WGS: CBYS010000564.1:16354-16887 |
| S. pennellii | "" | 534/177 | WGS: CCXL01007674.1:26924-27457 |
| Сем. Solanaceae, род Solanum, секция Melongena | |||
| S. melongena | Баклажан или паслен темноплодный | 543/180 | *** TSA: GBGZ01070312.1:241-783 |
| S. incanum | Паслен седой | 543/180 | TSA: GAYS01032656.1:86-628 |
| Сем. Solanaceae, род Solanum, секция Torva | |||
| S. torvum | Паслен страшный | 531/176 | KC884746.1 |
| Сем. Solanaceae, род Solanum, секция Brevantherum | |||
| S. mauritianum | Паслен маврикийский | 540/179 | ***MK473854 |
| Сем. Solanaceae, род Capsicum | |||
| C. annuum | Перец овощной | 540/179 | TSA: JW066104.1:723-1262 |
| C. annuum var. glabriusculum | То же | 540/179 | WGS: ASJV01098869.1:123188-123727 |
| C. chinense | Перец китайский | 552/184 | WGS: MCIT02000012.1:225210900-225211933 |
| Сем. Solanaceae, род Petunia | |||
| P. integrifolia | Петуния | 519/172 | TSA: GBRV01030828.1:76-594 |
| P. axillaris | "" | 513/170 | TSA: GBRU01005649.1:478-990 |
| P. exserta | "" | 519/172 | TSA: GBRT01014436.1:465-983 |
| Сем. Solanaceae, род Physalis | |||
| P. alkekengi var. franchetii | Физалис | 552/184 | TSA: IABG01055463.1:137-688 |
| P. peruviana | "" | 576/191 | TSA: IABH01030898.1:22-597 |
| Сем. Solanaceae, род Withania | |||
| W. somnifera | Ашваганда или физалис солнечно-листный | 531/176 | TSA: GBHJ01019844.1:241-771 |
| Сем. Solanaceae, род Nicotiana, секция Nicotiana | |||
| N. tabacum | Табак | 519/172 | TSA: GFCB01042641.1:105-623 |
| Сем. Solanaceae, род Nicotiana, секция Petunioides | |||
| N. attenuata | Табак | 519/172 | TSA: GBGF01001400.1:110-628 |
| Сем. Solanaceae, род Nicotiana, секция Tomentosae | |||
| N. tomentosiformis | Табак | 519/172 | XM_009591851.2 |
| Сем. Solanaceae, род Nicotiana, секция Sylvestres | |||
| N. sylvestris | Табак | 519/172 | XM_009765416.1 |
| Сем. Asteraceae, род Artemisia | |||
| A. annua cv. Huhao1 | Полынь однолетняя | 582/193 | WGS: PKPP01002962.1:130612-131193 |
Примечание. * – последовательности, аннотированные по результатам полногеномного секвенирования (WGS – Whole genome shotgun sequence) и секвенирования транскриптома (TSA – Transcriptome shotgun assembly); NC – nucleotide collection – аннотированные нуклеотидные последовательности. ** – https://solgenomics.net/; *** – последовательности, клонированные в нашей лаборатории (не опубликовано); **** – в скобках приведены названия видов согласно последней реклассификации видов картофеля (Spooner et al., 2014, 2018).
Паслен был представлен тремя видами подрода Leptostemonum: S. torvum (секция Torva), S. melongena и S. incanum (секция Melongena) (табл. 1). Мы расширили анализируемую группу пасленов, идентифицировав ген-гомолог INH2 паслена маврикийского S. mauritianum (подрод Brevantherum, секция Brevantherum).
Структура и полиморфизм нуклеотидных последовательностей гомологов INH2. Ген INH2 (INH2a) ингибитора вакуолярной инвертазы содержит единственный экзон. Помимо мРНК INH2a транскрипты INH2 могут быть представлены также двумя альтернативно-сплайсированными формами (Brummell et al., 2011). Анализ первичной структуры нуклеотидных последовательностей выявленных гомологов INH2 Solanaceae показал, что все они – гомологи INH2a и состоят из одного экзона, а их длина (ген = кДНК) варьирует от 513 пар нуклеотидов (п. н.) Petunia axillaris до 576 п. н. Physalis peruviana (табл. 1). Длина референсной кДНК S. tuberosum составила 537 п. н. Вариабельность длин последовательностей объяснялась наличием инсерций и делеций. Ген INH2 Capsicum chinense и P. peruviana отличался от INH2 остальных Solanaceae инсерциями 15 п. н. (301–315) и 18 п. н. (190–208) соответственно. Кроме того, на 5'-конце анализируемых генов присутствовала область, гипервариабельная из-за наличия инделей повторяющегося триплета (в основном AAC/AAT, кодирующих аминокислоту Asn). Для пасленов, физалисов и перцев в данной области были характерны инсерции AGC/AGT (Ser) и AGG (Arg), а для ашваганды – ACC (Thr) (рис. 1). Взятая для сравнения длина последовательности вида A. annua составляла 582 п. н. (табл. 1) и значительно отличалась от INH2-генов Solanaceae.
Рис. 1.
Вариабельность аминокислотных последовательностей гомологов INH2 у исследуемых видов Solanaceae. Радикальные замещения и индели (серый фон) выделены рамкой. Номера сверху соответствуют порядковому номеру а. о. в референсном белке INH2 S.tuberosum cv. DM.

Оценка полиморфизма INH2-последовательностей Solanaceae по сравнению с геном INH2a S. tuberosum cv. DM 1-3 516 R44 (XM_006349784.2) выявила 321 вариабельный сайт (SNPs). Вариабельность кДНК INH2a составила 53.5% (у всех анализируемых видов Solanaceae), 13.09% у секции Petota (10 видов) и 6.55% у секции Lycopersicon (9 видов). Интересно, что экзонный полиморфизм INH2 среди видов томата (секция Lycopersicon) был выше, чем у кодирующих последовательностей генов вакуолярной инвертазы TAI (5.36%) – белка-мишени ингибитора INH2 (Slugina et al., 2017). Данный факт свидетельствует о повышенной скорости накопления мутаций, сопровождающей эволюцию гена ингибитора вакуолярной инвертазы, что подчеркивает важность гена INH2 для эволюции аппарата углеводного метаболизма.
Структура и вариабельность аминокислотных последовательностей гомологов INH2. Выявленные последовательности генов-гомологов INH2 были транслированы. Размер гомологов INH2 Solanaceae составил от 170 до 191 а. о. (табл. 1). Все белки принадлежали к сем. PF04043 (Pfam database, http://pfam.wustl.edu/), объединяющему ингибиторы инвертаз и пектинметилэстераз, известных как белки с почти идентичной структурой, но распознающие разные белковые мишени (Scognamiglio et al., 2003). Все гомологи INH2 содержали N-концевую область, богатую остатками аспарагина, и консервативный каталитический домен PMEI (~145 а. о.). Поиск консервативных последовательностей, специфичных для гомологов INH2, обнаружил семь мотивов, четыре из которых присутствуют как в вакуолярном, так и в апопластном ингибиторе инвертаз (рис. 2). Мотивы I, II и III оказались характерными исключительно для INH2 Solanaceae, причем мотив II присутствовал только у половины анализируемых видов, а мотив III не был обнаружен у видов петунии. В целом данные анализа указывают на возможность использования мотива I при поиске и аннотировании гомологичных INH2 последовательностей внутри сем. Solanaceae.
Рис. 2.
Филогенетические отношения между исследуемыми видами Solanaceae. Дендрограмма построена на основе анализа полноразмерных аминокислотных последовательностей соответствующих гомологов INH2 методом максимального правдоподобия с использованием модели замен. Белок INH1 Solanum lycopersicum (NM_001247862.2) использован в качестве внешней группы по последовательности ингибитора инвертазы, а INH2 Artemisia annua – внешняя группа по отношению к видам семейства Solanaceae. Цифры в узлах – значения бутстрэпа, масштабная линия под дендрограммой – генетическая дистанция между видами. Справа от дендрограммы – схема консервативных мотивов в белковых последовательностях INH2 по сравнению с INH1; под схемой представлены последовательности мотивов I, II и III, специфичных для гомологов INH2.
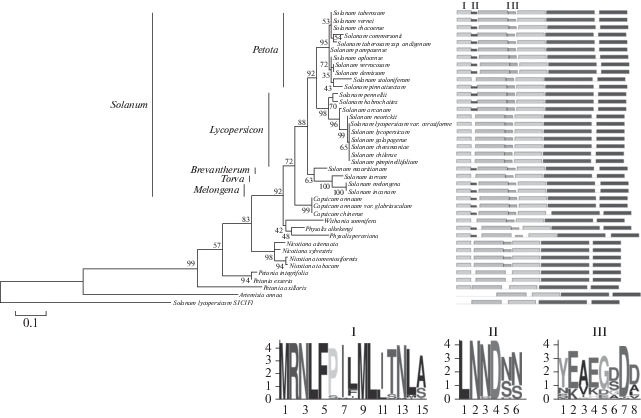
Всего в гомологах INH2 Solanaceae по сравнению с референсом было обнаружено 120 (60%) вариабельных сайтов, 13 из которых соответствовали 16 радикальным замещениям аминокислотных остатков, которые потенциально могут влиять на конформацию белка (рис. 1). Сравнение пространственной структуры выявленных в данной работе гомологов INH2 не обнаружило каких-либо существенных различий между ними. Все анализируемые белки формировали характерную для семейства PF04043 вторичную и третичную структуры и представляли собой N-концевую шпильку, состоящую из трех α-спиралей (где вторая и третья спирали были разделены аспарагин-богатой областью) и асимметричного четырехспирального пучка. При этом обе структуры были дополнительно стабилизированы двумя дисульфидными связями, образуемыми четырьмя остатками цистеина, консервативными для всех анализируемых гомологов INH2. Высокое структурное сходство гомологов INH2 предполагает сохранение ими основных свойств и функций ингибитора вакуолярной инвертазы.
Считается, что взаимодействие вакуолярной инвертазы и ее ингибитора происходит в вакуоле. Известно, что для посттрансляционной транспортировки в вакуоль молекула ингибитора INH2 должна иметь сигнальный пептид (Brummell et al., 2011).
На основе проведенного нами анализа аминокислотной последовательности INH2 было предсказано наличие сигнального пептида (длиной 15–24 а. о. в зависимости от образца), большая часть которого соответствовала мотиву I, у всех анализируемых ингибиторов Solanaceae. В последовательности INH2 вида Asteraceae A. annua (идентичность/перекрывание с INH2 S. tuberosum – 48%/77% соответственно) было предсказано наличие нетрансмембранного сигнального пептида длиной 45 а. о. Сходный длинный (40 а. о.) нетрансмембранный сигнальный пептид был предсказан в ходе дополнительного поиска у гомолога INH2 другого вида Asteraceae – Helianthus annuus. Полученные данные могут быть отражением эволюции гена ингибитора вакуолярной инвертазы после расхождения euasterids на клады campanulids (euasterids II), куда входят Asteraceae, и lamiids (euasterids I), включающие в себя Solanaceae. Мы предположили, что последовательность гомологов INH2 может быть использована для исследования филогении видов.
Филогения видов Solanaceae на основе аминокислотных последовательностей гомологов ингибитора вакуолярной инвертазы INH2. Ранее исследователи филогении INH2 предполагали, что гены ингибиторов инвертаз эволюционировали сравнительно недавно, одновременно с эволюцией сосудистых растений. Это привело к более специфичной регуляции активностей инвертаз (вакуолярной и апопластной) и оптимизации транспорта ассимилятов из источника в запасающие органы, и такая эволюция способствовала выживанию сосудистых растений (Wan et al., 2018).
Для оценки возможной эволюции гена INH2 внутри сем. Solanaceae аминокислотные последовательности идентифицированных нами гомологов INH2 (табл. 1) были использованы в построении филогенетической дендрограммы. Полученное дерево состояло из нескольких выраженных кластеров, соответствующих разделению видов Solanaceae на роды, подроды и секции (рис. 2). Топология дерева во многом совпадала с данными более ранней работы, где для определения филогении видов Solanaceae использовался суммарный набор белоккодирующих последовательностей геномов этих видов (Fukuoka et al., 2010), что подтверждает пригодность аминокислотной последовательности INH2 для филогенетического анализа растений.
Нами было показано, что баклажан S. melongena и его ближайший родственник S. incanum наиболее близки к S. torvum. Вместе с пасленом S. mauritianum другого подрода эти виды образуют отдельный кластер, объединяющий три секции – Melongena, Torva и Brevantherum. Данный кластер является сестринским по отношению к кластеру, образованному видами подрода Potatoe и состоящему из двух сестринских групп – видов секции Lycopersicon и видов секции Petota. По мере удаления от рода Solanum располагаются более древние виды: кластер перцев Capsicum; кластер, объединяющий физалис и ашваганду (физалис солнечно-листный); кластер Nicotiana; кластер Petunia (рис. 2).
Несмотря на столь хорошую кластеризацию вплоть до секций, разделение на уровне видов происходило не всегда. Так, перцы C. annuum никак не разошлись с C. chinense, что, однако, подтверждает предположение об одновременном происхождении данных видов от предшественника (Moscone et al., 2007). Из 13 известных видов томата (Peralta et al., 2008) мы анализировали 8 видов. Среди них на дендрограмме достоверно выделилась группа красноплодных видов, в которую вошел зеленоплодный вид S. chilense. При этом наиболее древние виды S. pennellii и S. habrochaites выделились в отдельный кластер в основании секции Lycopersicon (рис. 2). Ранее молекулярный анализ видов картофеля позволил сгруппировать их в 4 клады (Spooner et al., 2014, 2018). В настоящей работе исследуемые виды картофеля достоверно разошлись на две сестринские клады (рис. 2). Обе клады состояли в основном из видов “клады 4”. Однако одна из клад включала в себя и вид S. pinnatisectum, который относится к “кладе 1 + 2”, и вид S. stoloniferum, который может относиться как к “кладе 4”, так и к “кладе 1 + 2”. Одна из клад была представлена видами “клады 4” (согласно Spooner et al., 2014, 2018). Другая клада дополнительно поделилась на 2 подклады, включающие в себя помимо видов “клады 4” также виды “клады 1” (S. pinnatisectum) и “комплексной” (S. stoloniferum) (Spooner et al., 2014, 2018). Внутри подклад виды картофеля практически не разделились (значения бутстрэпа <50%) (рис. 2). Это может быть связано с высокой сложностью данной таксономической группы благодаря наличию межвидовой гибридизации, инрогрессии, аллополиплоидии, смеси полового и бесполого размножения и возможного недавнего расхождения видов (Spooner et al., 2008, 2014, 2018).
Мы полагаем, что точнее оценить филогению видов можно с использованием совместно последовательности INH2 и вакуолярной инвертазы VacInv, принимая во внимание взаимодействие данных белков in vivo и возможную коэволюцию кодирующих их генов.
Таким образом, в данном исследовании методами in silico и молекулярной биологии были идентифицированы гены-гомологи ингибитора вакуолярной инвертазы INH2 у различных видов семейства Solanaceae. С помощью структурно-филогенетического анализа был охарактеризован межвидовой полиморфизм данного гена и кодируемого им белка внутри семейства, отдельных родов и секций. Полученные результаты свидетельствуют о связи эволюции гена ингибитора вакуолярной инвертазы и возникновения разнообразия сосудистых растений, а также о возможности оценки филогении современных видов на основе полиморфизма INH2.
Авторы выражают благодарность С.Р. Майорову за предоставление растительного материала, а также Е.О. Шмельковой за значимую техническую помощь в проведении исследования.
Работа выполнена при финансовой поддержке РФФИ (грант 18–016–00108), Министерства науки и высшего образования Российской Федерации и ФНТП развития сельского хозяйства РФ на 2017–2025 гг. (подпрограмма “Развитие селекции и семеноводства картофеля в Российской Федерации”).
Список литературы
Bailey T.L., Elkan C. Fitting a mixture model by expectation maximization to discover motifs in biopolymers // Proc. Second Inter. Conf. Intell. Systems Mol. Biol. California: AAAI Press, Menlo Park, 1994. P. 28–36.
Bate N.J., Niu X., Wang Y., Reimann K.S., Helentjaris T.G. An invertase inhibitor from maize localizes to the embryo surrounding region during early kernel development // Plant Physiol. 2004. V. 134. № 1. P. 246–254.
Brummell D.A., Chen R.K., Harris J.C., Zhang H., Hamiaux C., Kralicek A.V., McKenzie M.J. Induction of vacuolar invertase inhibitor mRNA in potato tubers contributes to cold-induced sweetening resistance and includes spliced hybrid mRNA variants // J. Exp. Bot. 2011. V. 62. № 10. P. 3519–3534.
Choi Y., Sims G.E., Murphy S., Miller J.R., Chan A.P. Predicting the functional effect of amino acid substitutions and indels // PLoS One. 2012. V. 7. № 10:e46688
Di Matteo A., Giovane A., Raiola A., Camardella L., Bonivento D., De Lorenzo G., Cervone F., Bellincampi D., Tsernoglou D. Structural basis for the interaction between pectin methylesterase and a specific inhibitor protein // Plant Cell. 2005. V. 17. № 3. P. 849–858.
Fukuoka H., Yamaguchi H., Nunome T., Negoro S., Miyatake K., Ohyama A. Accumulation, functional annotation, and comparative analysis of expressed sequence tags in eggplant (Solanum melongena L.), the third pole of the genus Solanum species after tomato and potato // Gene. 2010. V. 450. № 1–2. P. 76–84.
Gebhardt C. The historical role of species from the Solanaceae plant family in genetic research // Theor. Appl. Genet. 2016. V. 129. № 12. P. 2281–2294.
Greiner S., Rausch T., Sonnewald U., Herbers K. Ectopic expression of a tobacco invertase inhibitor homolog prevents cold-induced sweetening of potato tubers // Nat. Biotechnol. 1999. V. 17. № 7. P. 708–711.
Hothorn M., Wolf S., Aloy P., Greiner S., Scheffzek K. Structural insights into the target specificity of plant invertase and pectin methylesterase inhibitory proteins // Plant Cell. 2004. V. 16. № 12. P. 3437–3447.
Kelley L.A., Mezulis S., Yates C.M., Wass M.N., Sternberg M.J. The Phyre2 web portal for protein modeling, prediction and analysis // Nat. Protoc. 2015. V. 10. № 6. P. 845–858.
Knapp S., Bohs L., Nee M., Spooner D.M. Solanaceae – a model for linking genomics with biodiversity // Comp. Funct. Genom. 2004. V. 5. P. 285–291.
Kumar S., Stecher G., Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets // Mol. Biol. Evol. 2016. V. 33. № 7. P. 1870–1874.
Link M., Rausch T., Greiner S. In Arabidopsis thaliana, the invertase inhibitors AtC/VIF1 and 2 exhibit distinct target enzyme specificities and expression profiles // FEBS Lett. 2004. V. 573. № 1–3. P. 105–110.
Machida-Hirano R. Diversity of potato genetic resources // Breed. Sci. 2015. V. 65. № 1. P. 26–40.
Moscone E.A., Scaldaferro M.A., Grabiele M., Cecchini N.M., García Y.S., Jarret R., Daviña J.R., Ducasse D.A., Barboza G.E., Ehrendorfer F. The evolution of chili peppers (Capsicum – Solanaceae): A cytogenetic perspective // Acta Hort. ISHS. 2007. V. 745. P. 137–170.
Peralta I.E., Spooner D.M., Knapp S. Taxonomy of wild tomatoes and their relatives (Solanum sect. Lycopersicoides, sect. Juglandifolia, sect. Lycopersicon; Solanaceae) // Syst. Bot. Monogr. 2008. V. 84. P. 1–186.
Pressey R. Invertase inhibitor from potatoes: purification, characterization, and reactivity with plant invertases // Plant Physiol. 1967. V. 42. № 12. P. 1780–1786.
Qin G., Zhu Z., Wang W., Cai J., Chen Y., Li L., Tian S. A tomato vacuolar invertase inhibitor mediates sucrose metabolism and influences fruit ripening // Plant Physiol. 2016. V. 172. № 3. P. 1596–1611.
Rausch T., Greiner S. Plant protein inhibitors of invertases // Biochim. Biophys. Acta. 2004. V. 16962:253e261.
Scognamiglio M.A., Ciardiello M.A., Tamburrini M., Carratore V., Rausch T., Camardella L. The plant invertase inhibitor shares structural properties and disulfide bridges arrangement with the pectin methylesterase inhibitor // J. Protein Chem. 2003. V. 22. P. 363–369.
Sherson S.M., Alford H.L., Forbes S.M., Wallace G., Smith S.M. Roles of cell-wall invertases and monosaccharide transporters in the growth and development of Arabidopsis // J. Exp. Bot. 2003. V. 54. P. 525–531.
Slugina M.A., Shchennikova A.V., Kochieva E.Z. TAI vacuolar invertase orthologs: the interspecific variability in tomato plants (Solanum section Lycopersicon) // Mol. Genet. Genomics. 2017. V. 292. № 5. P. 1123–1138.
Spooner D.M., Fajardo D., Salas A. Revision of the Solanum medians complex (Solanum section Petota) // Syst. Bot. 2008. V. 33. № 3. P. 579–588.
Spooner D.M., Ghislain M., Simon R., Jansky S.H., Gavrilenko T. Systematics, diversity, genetics, and evolution of wild and cultivated potatoes // Bot. Rev. 2014. V. 80. P. 283–383.
Spooner D.M., Ruess H., Arbizu C.I., Rodríguez F., Solís-Lemus C. Greatly reduced phylogenetic structure in the cultivated potato clade (Solanum section Petota pro parte) // Am. J. Bot. 2018. V. 105. № 1. P. 60–70.
Tymowska-Lalanne Z., Kreis M. Expression of the Arabidopsis thaliana invertase gene family // Planta. 1998. V. 207. № 2. P. 259–265.
Wan H., Wu L., Yang Y., Zhou G., Ruan Y.L. Evolution of sucrose metabolism: The dichotomy of invertases and beyond // Trends Plant Sci. 2018. V. 23. № 2. P. 163–177.
Дополнительные материалы отсутствуют.
Инструменты
Известия РАН. Серия биологическая


