Известия РАН. Серия биологическая, 2022, № 5, стр. 470-481
Разнообразие аллелей главного комплекса гистосовместимости у обыкновенного хомяка (Cricetus cricetus) в городской и сельской популяциях
Н. Ю. Феоктистова 1, *, И. Г. Мещерский 1, Т. Н. Карманова 1, А. В. Гуреева 1, А. В. Суров 1
1 Институт проблем экологии и эволюции им. А.Н. Северцова РАН
119071 Москва, Ленинский пр., 33, Россия
* E-mail: feoktistovanyu@gmail.com
Поступила в редакцию 03.04.2022
После доработки 11.04.2022
Принята к публикации 11.04.2022
- EDN: FLNDXK
- DOI: 10.31857/S1026347022050079
Аннотация
Известно, что городские популяции млекопитающих и птиц сталкиваются с большим разнообразием различных патогенов. На основе метода таргетного секвенирования на платформе Illumina определяли аллельное разнообразие экзона 2 гена DRB главного комплекса гистосовместимости в городской (г. Симферополь) и сельской популяциях обыкновенного хомяка. Для особей, обитающих в городе, отмечено большее количество аллелей, более высокие значения показателей гаплотипического и нуклеотидного разнообразия, меньшая доля особей с гомозиготным генотипом. Для обеих популяций характерно значительное превышение числа несинонимичных замен над синонимичными и почти каждому аллелю гена соответствует своя аминокислотная последовательность. Однако влияние положительного отбора на разнообразие вариантов антигенсвязывающих участков в аллелях гена DRB в условиях города выражено значительно сильнее. Полученные результаты позволяют предполагать, что устойчивость обыкновенных хомяков, обитающих в специфичных и разнообразных условиях городской среды к различного рода патогенной нагрузке выше, чем в сельских популяциях.
Стремительное распространение городских экосистем на планете можно рассматривать как качественно новый этап развития жизни на Земле. Предполагается, что к 2030 г. почти 10% суши будет урбанизировано (Schilthuizen, 2018). Увеличение площади территорий, занятых урбанизированным ландшафтом, неизбежно приводит к включению в городские экосистемы новых видов, ранее не проявлявших склонности к синантропизму. Хотя для многих видов городские условия являются абсолютно неприемлемыми, другие могут заселять отдельные городские биотопы, удовлетворяющие их биологическим потребностям. В то же время для отдельных видов городские условия оказываются благоприятными, и плотность населения таких видов в городах может быть даже выше, чем в естественных биотопах. В качестве примера можно привести виды, сокращающие численность в естественных биотопах, но успешно заселяющие города. Это, например, сокол сапсан (Falco peregrinus), освоивший за последние 30 лет города Северной Америки и Европы (Сорокин, 2002), или ястреб Купера (Accipiter cooperii), успешно заселяющий города США (Boggie, Mannan, 2014; Morinha et al., 2016). Так города фактически стали местом сохранения этих редких видов.
Изучение экологических особенностей городской среды, выявление положительных и отрицательных факторов, определяющих возможность существования здесь животных, растений, а в итоге и самого человека, является актуальной проблемой экологии. Одно из важнейших направлений в исследовании процессов синурбанизации – изучение механизмов адаптаций животных к новым условиям, а также определение параметров оценки их благополучия. Городская среда имеет множество специфических особенностей, включающих как абиотические (температурный режим, влажность, химическое, шумовое, световое загрязнение и т.д.), так и биотические факторы. Среди последних – обитание на одной территории ряда автохтонных и инвазивных видов, формирующих новые, ранее не существовавшие сообщества с повышенной плотностью населения, c нехарактерной кормовой базой и пр. (Luniak, 2004). Как следствие, в городе следует ожидать появление новых паразитарно-хозяинных отношений и более высокую паразитарную нагрузку. Известно, что в целом городские популяции млекопитающих и птиц чаще подвергаются заражению паразитами и встречаются с большим количеством патогенов, чем сельские (Gliwicz et al., 1980; Luniak, 2004). При этом вид сталкивается в городе с патогенами и паразитами, не характерными для естественных биотопов. Возникает вопрос, как иммунная система животных–синурбистов отвечает на эти городские “вызовы”. Оценка аллельного разнообразия генов главного комплекса гистосовместимости (major histocompatibility complex – МHC) может пролить свет на решение этой проблемы и служить показателем степени приспособленности вида к противодействию негативным факторам городской среды. Эти гены играют ключевую роль в иммунной защите организма, в том числе в обеспечении ответа на патогенную нагрузку (Klein, 1986; Hill et al., 1991; Potts, Wakeland 1993; Brown, Eklund 1994; Hedrick, 1994; Edwards, Potts 1996; Janeway et al., 2004; Acevedo-Whitehouse, Cunningham, 2006; Ujvari, Belov, 2011).
Исследование популяций белоногих хомячков (Peromyscus leucopus), обитающих в парках Нью-Йорка показало, что гены, ответственные за иммунные характеристики особей (так же как и ряд других, например, ответственных за переработку продуктов, богатых жирами), в городских условиях находятся под влиянием положительного отбора (Harris et al., 2013, 2016; Harris, Munshi-South, 2017). Исследования красных рысей (Lynx rufus) в г. Таузенд-Оукс (США), столкнувшихся с эпидемией чесотки, показали, что до и после эпидемии в популяции обнаружены разные наборы аллелей генов МНС и TLR (Tоll–Like Receptors – рецепторы, позволяющие распознавать консервативные структуры микроорганизмов и активирующие клеточный иммунный ответ). По всей видимости, эпидемию пережили только те животные, которые обладали подходящей комбинацией иммунных генов (Serieys et al., 2015).
Одним из видов млекопитающих, активно осваивающих урбанизированные территории, является обыкновенный хомяк (Cricetus cricetus). В естественных биотопах численность этого вида за последние полвека катастрофически снизилась, особенно в западной части ареала (Surov et al., 2016), вплоть до того, что в 2020 г. вид был включен в Красную книгу МСОП со статусом CR (виды, находящиеся на грани полного исчезновения) (Banaszek et al., 2020). В то же время поселения обыкновенного хомяка в городах, зачастую достигают высокой численности (Feoktistova et al., 2013; Суров и др., 2015; Surov et al., 2016; Feoktistova et al., 2017). К настоящему времени популяции этого вида обитают в Нальчике, Грозном, Кисловодске, Владимире, Омске, Туле, Рязани, Москве и некоторых других городах России (Феоктистова и др., 2019), известны из г. Нур-Султан в Казахстане (Феоктистова и др., 2020), а также из целого ряда городов Европы (Niethammer, 1982; Thorns, 1998; Endres, Weber, 1999; Kupfernagel, 2003; Losík et al., 2007; Schmelzer, Millesi, 2008; Banaszek, Ziomek, 2010; Čanády, 2013; Feoktistova et al., 2013; Matysek et al., 2013; Petrová et al., 2018; Buczek, 2019).
Объектом нашего исследования стала популяция обыкновенного хомяка, обитающая в г. Симферополь (Крым) – самая крупная из известных городских популяций этого вида (Товпинец и др., 2006; Феоктистова и др., 2016). Мы определяли аллельное разнообразие экзона 2 гена DRB, входящего в состав MHC класса II у млекопитающих. Полученные значения сравнивались с отмеченными для поселений хомяка, расположенных в сельском антропогенном ландшафте Крыма. Цель работы состояла в оценке влияния обитания в урбоценозе на особенности имунной системы данного вида.
Существенным затруднением при изучении аллельного разнообразия генов MHC является значительное число отличающих разные аллели нуклеотидных замен, а также, в ряде случаев, многокопийность. Для того, чтобы определить замены, отличающие последовательность каждого из присутствующих в генотипе животного аллелей, традиционно используют такие трудоемкие методы, как клонирование или анализ одноцепочечного конформационного полиморфизма (SSCP) с последующим секвенированием по Сэнгеру во многих повторностях. В нашей работе мы воспользовались технологией секвенирования нового поколения (NGS), в настоящее время успешно вытесняющей традиционные методы (Shiina et al., 2015). Возможность считывания до 250 п.н. при длине интересующего нас участка 246 п.н. позволила прямо определять каждый вариант последовательности без необходимости совмещения отдельных фрагментов молекулы.
МАТЕРИАЛЫ И МЕТОДЫ
Для проведения молекулярно-генетического анализа были использованы образцы тканей 20 особей обыкновенного хомяка, отловленных нами в городской черте Симферополя, и 20 особей, с окраин сельских поселений, расположенных далее 20 км от границ города. Образцы представляли собой зафиксированные в 96%-ном этаноле ткани уха или пальца (при отлове живых зверьков) или зафиксированная аналогичным образом мышечная ткань от мертвых животных, полученных от хозяев сельскохозяйственных участков.
Тотальную ДНК выделяли с использованием набора реагентов Diatom™ DNA Prep (OOO “Лаборатория Изоген”, Москва, Россия) по инструкции производителя.
Для амплификации экзона 2 гена DRB использовали праймеры, предложенные в работе Smulders et al., 2003:
Forward: 5-GAGTGTCATTTCTACAACGGGA-3;
Reverse: 5-CTCTCCGCGGCACAAAGGAA-3
Предварительно специфичность праймеров была проверена постановкой ПЦР с последующим секвенированием продукта методом Сэнгера. Полученные последовательности соответствовали представленным в работе (Smulders et al., 2003), но содержали значительное количество нераспознаваемых (двойных) позиций.
Определение нуклеотидных последовательностей отдельных аллелей было выполнено в ООО “Евроген Лаб.” на основе таргетного секвенирования ПЦР-продуктов, полученных с использованием указанных праймеров на платформе Illumina.
Подготовку индивидуальных библиотек проводили в соответствии с протоколом, описанным в руководстве “16S Metagenomic Sequencing Library Preparation” (Part # 15044223 Rev. B; Illumina) с увеличенным количеством циклов амплификации на первой стадии ПЦР (35 циклов). После получения ампликонов библиотеки были очищены и смешаны эквимолярно с помощью SequalPrep™ Normalization Plate Kit (“ThermoFisher”). Контроль качества полученных пулов библиотек был проведен с помощью системы Fragment Analyzer, количественный анализ – с помощью qPCR.
Пул библиотек был секвенирован на Illumina MiSeq с использованием реактивов MiSeq Reagent Kit v2 Nano: длина прочтений – 250 п.н. с двух сторон фрагментов, 500 циклов. Для контроля параметров секвенирования использовали библиотека фага PhiХ.
Файлы FASTQ были получены с помощью программного обеспечения bcl2fastq v2.17.1.14 Conversion Software (Illumina). При обработке файлов в формате FASTQ с парными чтениями для каждого образца на первом этапе анализа прямые и обратные чтения были слиты с помощьью инструмента Bbmerge из пакета BBTools v38.87 (Bushnell et al., 2017). Чтения, которые не сливались однозначно без замен, не учитывали. Слитые чтения были выровнены на референс одного из вариантов последовательности DRB exon 2 известных для обыкновенного хомяка (Crcr-DRB1*14 allele, GenBank AJ490324, Smulders et al., 2003) с помощью программного обеспечения Bowtie2 (Langmead, Salzberg, 2012) с опцией local. Из полученных выравниваний были извлечены чтения с длиной не менее 240 п.н. при длине референтной последовательности 246 п.н. (включая области посадки праймеров). Для дальнейшего анализа были использованы только образцы, для которых было получено не менее 50 последовательностей, отвечавших данному условию.
В наборе последовательностей, полученных для каждого образца, были определены уникальные варианты (аллели) и посчитана частота их встречаемости. Для дальнейшего анализа были отобраны варианты (а), представленные в каждом случае не менее, чем 10 повторностями; (б) составляющие не менее 5% от общего количества полученных для данной особи последовательностей и (в) представленные числом повторностей, составляющим не менее 25% от количества повторностей аллеля с более высокой частотой в данном образце.
Из отобранных на основании этих условий последовательностей были исключены участки, соответствующие областям посадки прямого (22 п.н.) и обратного (20 п.н.) праймеров, а также две начальные (консервативные) позиции – с тем, чтобы первая позиция последовательности соответствовала первой позиции триплета. Идентичность последовательностей аллелей, отмеченных у разных особей, определяли с использованием онлайн-сервиса FaBox 1.61 (Villesen, 2007).
Варианты последовательностей, отобранные для последующего анализа, а также соответствующие им последовательности аминокислот проверяли на соответствие ожидаемому участку генома с использованием функции BLAST на сайте NCBI (https://blast.ncbi.nlm.nih.gov).
Учитывая, что на различия в количестве повторностей разных аллелей, полученных для каждого образца, в определенной степени влияют случайные отклонения в ходе секвенирования, а также отсутствие данных о возможном количестве повторов данного локуса (многокопийность) у обыкновенного хомяка, описание индивидуальных генотипов по соотношению представленности разных аллелей в индивидуальных наборах не проводили. Исключение составляло определение гомозиготных генотипов, которые регистрировались для особей, у которых в полученным наборе последовательностей был отмечен лишь один вариант, отвечающий указанным выше условиям. Последующее сравнение двух анализируемых выборок проводили на основании набора аллелей, отмеченных в каждой выборке, с указанием на число животных, в генотипе которых данный аллель был отмечен.
Сравнение выборок было проведено в программе Arlequin v.3.11 (Excoffier et al., 2005) с использованием Fst и Φst критериев, а также с оценкой показателей гаплотипического (аллельного) и нуклеотидного разнообразия для каждой выборки.
Медианная сеть вариантов нуклеотидных последовательностей была построена методом Median Joining в программе Network 4.612 (Bandelt et al., 1999). Полученные варианты нуклеотидных последовательностей были транслированы, определено число вариантов аминокислотных последовательностей и их соответствие отмеченным аллелям гена для каждой выборки, а также для всей исследованной совокупности.
В аминокислотных последовательностях были определены позиции, потенциально ответственные за связывание антигена – на основе данных, приведенных в работе (Brown et al., 1993) и аналогично тому, как это было выполнено ранее для представителей рода Peromyscus (Richmond, Davey, 2003).
Характер нуклеотидных последовательностей в полученных выборках был проанализирован на количество и соотношение несинонимичных и синонимичных замен в программе MEGA X (Kumar et al., 2018). Также в этой программе был выполнен Z-test на оценку влияния положительного отбора на увеличение разнообразия аминокислотных последовательностей. Тест был проведен на основе модифицированного метода Nei-Gojobori с принимаемой вероятностью отношения частоты транзиций и трансверсий 2.0 и подтвержден бутстреп-тестом, включавшим 10 000 реплик. Тест был выполнен как для полного состава последовательностей (201 п.н., 67 кодонов), так и только для регионов, ответственных за связывание антигена (57 п.н., 19 кодонов).
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Результирующие файлы, содержавшие от 53 до 2996 последовательностей нужной длины, были получены для 17 особей, отловленных в городе, и 19 из сельских популяций. Для каждого образца было получено от 15 до 606 вариантов нуклеотидных последовательностей, от 1 до 4, из которых отвечали перечисленным выше условиям. После удаления участков посадки праймеров все признанные валидными последовательности имели длину 201 п.н., не содержали стоп-кодонов, а также делеций или вставок, сдвигающих рамку считывания. Всего в двух исследованных выборках было отмечено 25 вариантов (аллелей), последовательности которых депонированы в базе данных GenBank NCBI (табл. 1). При проверке с использованием функции BLAST для всех этих вариантов было установлено соответствие гену DRB1 MHC класса II у грызунов. Для 21 варианта максимальное сходство (91.1–99.4%) было с 6 последовательностями, известными для Cricetus cricetus (AJ490311, AJ490314, AJ490316-317, AJ490319, AJ490320, AJ490322-323), для 3 вариантов (Crcr-DRB*25, Crcr-DRB*26 и Crcr-DRB*29 из городской популяции) – с последовательностью, известной для Apodemus flavicollis (JQ858341, сходство 93.3–93.8%), для аллеля Crcr-DRB*27 (городская популяция) – с последовательностью Rattus norvegicus (AY626204, сходство 93.8%). Использование функции BLAST для соответствующих данным аллелям аминокислотных последовательностей также показало их соответствие бета цепи антигенов MHC класса II у грызунов (class II histocompatibility antigen, beta domain).
Таблица 1.
Аллели локуса DRB1, экзон 2, отмеченные в ходе исследования
| Аллель | Номер Генбанка | Количество особей в обобщенной выборке (n = 36) |
|---|---|---|
| Crcr-DRB*15 | ON081883 | 12 |
| Crcr-DRB*16 | ON081884 | 4 |
| Crcr-DRB*17 | ON081885 | 3 |
| Crcr-DRB*18 | ON081886 | 1 |
| Crcr-DRB*19 | ON081887 | 7 |
| Crcr-DRB*20 | ON081888 | 4 |
| Crcr-DRB*21 | ON081889 | 1 |
| Crcr-DRB*22 | ON081890 | 2 |
| Crcr-DRB*23 | ON081891 | 1 |
| Crcr-DRB*24 | ON081892 | 2 |
| Crcr-DRB*25 | ON081893 | 3 |
| Crcr-DRB*26 | ON081894 | 3 |
| Crcr-DRB*27 | ON081895 | 2 |
| Crcr-DRB*28 | ON081896 | 2 |
| Crcr-DRB*29 | ON081897 | 1 |
| Crcr-DRB*30 | ON081898 | 1 |
| Crcr-DRB*31 | ON081899 | 1 |
| Crcr-DRB*32 | ON081900 | 1 |
| Crcr-DRB*33 | ON081901 | 1 |
| Crcr-DRB*34 | ON081902 | 2 |
| Crcr-DRB*35 | ON081903 | 1 |
| Crcr-DRB*36 | ON081904 | 1 |
| Crcr-DRB*37 | ON081905 | 1 |
| Crcr-DRB*38 | ON081906 | 1 |
| Crcr-DRB*39 | ON081907 | 1 |
Между двумя исследованными выборками были отмечены достоверные различия как по частоте встречаемости разных аллелей (Fst = 0.0316, p = = 0.022), так и по степени различия в нуклеотидных последовательностях (Φst = 0.09027, p = 0.0001).
Количество аллелей, отмеченных в генотипе одного животного, варьировало от 1 (гомозиготность, отмечена у большинства особей) до 4. Характеристика разнообразия выборок представлена в табл. 2. Значения всех показателей разнообразия в городской популяции были заметно выше, чем в сельских.
Таблица 2.
Характеристика разнообразия исследованных выборок
| Городская популяция |
Сельская популяция | Обобщенная выборка | |
|---|---|---|---|
| Объем выборки (n) | 17 | 19 | 36 |
| Число отмеченных аллелей | 19 | 11 | 25 |
| Среднее число аллелей в генотипе особи | 1.71 | 1.42 | 1.56 |
| Доля особей с гомозиготным генотипом, %% | 47.15 | 79.0 | 61.1 |
| Индекс гаплотипического разнообразия | 0.961 | 0.866 | 0.932 |
| Индекс нуклеотидного разнообразия, %% | 10.15 | 8.14 | 9.69 |
Встречаемость разных аллелей в выборках показана на рис. 1. В загородной популяции частота одного из аллелей (Crcr-DRB*15, встречен у 47.4% особей) значительно превышала частоту остальных, встречавшихся не более, чем у 15.8% (аллели Crcr-DRB*16, Crcr-DRB*17 и Crcr-DRB*19, остальные с еще меньшей частотой) особей каждый. В городской популяции подобного доминирования какого-либо одного аллеля не наблюдалось. Максимальная частота (23.5% особей) здесь была отмечена у аллеля Crcr-DRB*19, и еще три аллеля (Crcr-DRB*15, Crcr-DRB*25 и Crcr-DRB*26) были отмечены у 17.6% особей каждый. В среднем каждый аллель был отмечен только у 7.5% особей в городской и у 5.7% особей в сельской популяции.
Рис. 1.
Доля особей (%%) в городской (справа, темная заливка) и сельской (слева, светлая заливка) популяциях, у которых был отмечен тот или иной аллель.
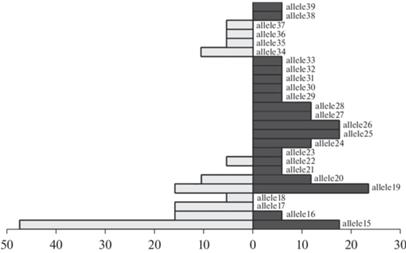
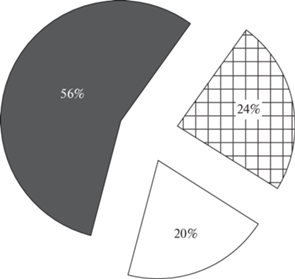
Большинство аллелей (73.7% в городской и 54.5% в сельской) были уникальными для каждой популяции. Из 25 отмеченных в нашем исследовании аллелей только 5 (20%) являлись общими для обеих выборок, остальные были встречены только в одной популяции каждый (рис. 1, 2). Два из них (Crcr-DRB*15 и Crcr-DRB*19) относились к числу относительно часто встречающихся в обеих популяциях и три были отмечены в обеих или в одной популяции только у 1–2 особей каждый.
Рис. 2.
Для суммарного числа аллелей – доля встреченных только в городской (серый цвет), только в сельской (штриховка) или в обеих (белый цвет) популяциях.
Медианная сеть последовательностей аллелей, отмеченных в городской и сельской популяциях, представлена на рис. 3.
Рис. 3.
Медианная сеть отмеченных вариантов последовательностей экзона 2 гена DRB. Темная заливка – городская, светлая заливка – сельская популяция, белые кружки – медианные векторы. (a) – диаметр кружков пропорционален встречаемости аллелей в обобщенной выборке, (б) – диаметр кружков одинаков. Числа соответствуют обозначениям аллелей, см. табл. 1.
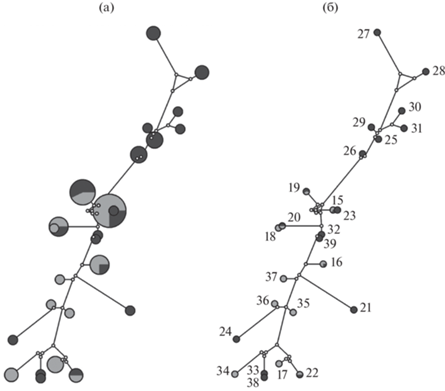
В центре сети расположены два из наиболее часто встречающихся общих для обеих выборок аллелей. Еще два общих аллеля также расположены ближе к центру. Одна из двух ветвей представлена аллелями, встречающимися либо в городской, либо в сельской популяции, другая – уникальными вариантами, встреченными только в городской популяции. Однако выявление конкретных филогенетических связей между отдельными вариантами в большинстве случаев невозможно из-за значительного количества неразрешаемых узлов.
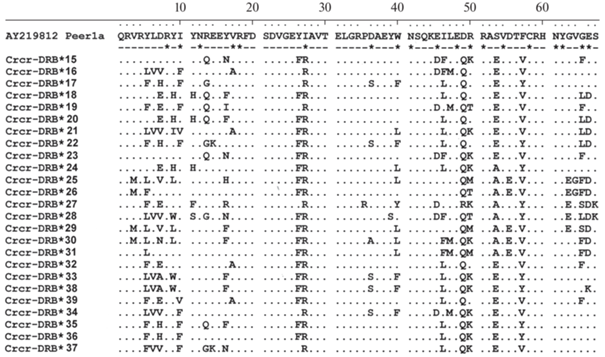
Большая часть вариантов нуклеотидных последовательностей (23 из 25) соответствовали оригинальным вариантам белка (рис. 4). Исключения составили два аллеля: присутствующий только в городской популяции Crcr-DRB*25, кодирует ту же аминокислоту, что и аллель Crcr-DRB*15, встречающийся в обеих популяциях. Аллель Crcr-DRB*18 уникальный для сельской популяции кодирует ту же аминокислоту, что и Crcr-DRB*22, также характерный для обеих популяций. Следует отметить, что аллели Crcr-DRB*25 и Crcr-DRB*18 были отмечены только у одного животного каждый.
Рис. 4.
Аминокислотные последовательности секвенированного участка экзона 2 гена DRB. В качестве референтной использована последовательность Peromyscus ereimicus (Richmond et al., 2003). Одинаковые аминокислотные остатки обозначены точками. Предположительно антигенсвязывающие позиции указаны звездочками.
Число несинонимичных замен в последовательностях аллелей существенно превышало число синонимичных как в каждой из исследованных выборок, так и в целом (табл. 3). При этом величина dN/dS в выборке из городской популяции была существенно выше, чем в сельской, как при сравнении полных последовательностей секвенированных участков, так и только антигенсвязывающих регионов.
Таблица 3.
Результаты оценки числа несинонимичных (dN) и синонимичных (dS) замен и результаты Z-теста на соответствие наблюдающихся разнообразия вариантов нуклеотидных последовательностей результату балансирующего отбора. Статистически достоверные значения выделены жирным шрифтом
| Полные последовательности секвенированных участков | Антигенсвязывающие регионы | |||||
|---|---|---|---|---|---|---|
| Городскаяпопуляция | Сельская популяция | Обобщенная выборка | Городскаяпопуляция | Сельская популяция | Обобщенная выборка | |
| dN (S.E.) |
15.71 (2.71) |
11.35 (2.60) |
14.32 (2.68) |
4.91 (1.52) |
4.86 (1.58) |
5.10 (1.57) |
| dS (S.E.) |
4.71 (1.14) |
4.69 (1.41) |
5.01 (1.26) |
1.04 (0.48) |
1.34 (0.75) |
1.18 (0.59) |
| dN/dS | 3.335 | 2.422 | 2.861 | 4.736 | 3.62 | 4.311 |
| Z | 1.123 | -0.094 | 0.5 | 2.109 | 0.961 | 1.791 |
| p(Z) | 0.132 | 1 | 0.309 | 0.019 | 0.169 | 0.038 |
В пределах кодируемых отсеквенированными участками экзона 2 гена DRB аминокислотных последовательностей 19 из 67 аминокислотных остатков предположительно являлись ответственными за связывание антигена (рис. 4).
Достоверное соответствие гипотезе воздействия балансирующего отбора было отмечено только для антигенсвязывающих регионов в обобщенной выборке и выборке из городской, но не из сельской популяции (табл. 3).
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Генетическое разнообразие является важнейшим фактором благополучного существования популяций. При этом его изучение должно сочетать оценку как по нейтральным генетическим маркерам митохондриальной и ядерной ДНК (в том числе, микросателлитным локусам), так и по генам, ответственным за адаптивные характеристики организма, в частности генам МНС (Pfrender et al., 2000; Reed, Frankham 2001; Hedrick, 2001; McKay, Latta, 2002; Luikart et a l., 2003; Palo et al., 2003; Gomez-Mestre, Tejedo, 2004; Ujvari et al., 2005; Ujvari, Belov, 2011; Shiina et al., 2015). Последние играют ключевую роль в обеспечении иммунных функций организма при инфекционной и паразитарной нагрузке, и наличие полиморфизма этих генов может свидетельствовать о благополучном состоянии популяций (Biedrzycka et al., 2011). Разнообразие генов MHC является важным фактором при формировании прогноза выживания для исчезающих популяций животных в дикой природе (Ujvari, Belov, 2011, Shiina et al., 2015).
Повышению полиморфизма аллелей MHC в индивидуальном генотипе способствует свойственная генам этого комплекса многокопийность – наличие более чем одного локуса определенного типа. Это явление широко распространено у млекопитающих: у лошадей (Fraser, Bailey, 1996), приматов (Bontrop et al., 1999; Khazand et al., 1999), крупного рогатого скота (Lewin et al., 1999), разных видов кошачьих (Kennedy et al., 2002), морских львов (Bowen et al., 2004). Отмечена многокопийность генов MHC и у грызунов, в частности, у песчанок (Gerbillurus paeba) (Harf, Sommer, 2005) и бобров (Castor fiber pohlei) (Babik et al., 2005). Вопрос о многокопийности гена DRB у обыкновенного хомяка до настоящего времени оставался открытым. При исследовании европейских популяций ни для одного из 70 образцов не было обнаружено наличие более двух аллелей (Smulders et al., 2003). Однако это исследование проводилось методом клонирования с амплификацией и секвенированием не более, чем 10 молекул (клонов) на образец. В нашем случае, при секвенировании многократно большего числа ампликонов, была показана возможность присутствия в генотипе обыкновенного хомяка до 4 разных последовательностей экзона 2 гена DRB, представленных со сходно высокой частотой.
Обитание в условиях города зачастую характеризуется обеднением генетического разнообразия, выявляемого по нейтральным маркерам (Kajdacsi et al., 2013; Chiappero et al., 2011; MunshiSouth et al., 2014; Феоктистова и др., 2016; 2019). Связано это как с ограниченным числом приспособившихся к городским условиям особей основателей, так и ограниченностью и изолированностью пригодных для обитания территорий, что способствует усилению инбридинга. Однако в условиях города животные становятся мишенью для большого числа нестандартных патогенов и паразитов из-за высокой плотности синантропных видов и разнообразных стрессовых факторов (Gliwicz et al., 1980; Luniak, 2004). Поэтому накопление большего высокого разнообразия генов, отвечающих за иммунные характеристики, является крайне актуальным для выживания видов-синурбистов.
Обыкновенный хомяк, как уже отмечалось, является видом, включенным в Красную книгу МСОП в связи с резким сокращением численности естественных популяций. Исследования генетического разнообразия по нейтральным маркерам (как по мтДНК, так и по микросаттелитным локусам), показали резкое сокращение генетического разнообразия его популяций в крайней западной части ареала, в то время как в популяциях из Восточной Европы разнообразие по этим маркерам остается достаточно высоким (Smulders et al., 2003; Neumann et al., 2004, 2005; Banaszek et al., 2011; Reiners et al., 2014).
Единственное исследование, оценивающие аллельное разнообразие генов MHC в популяциях обыкновенного хомяка было проведено также для естественных популяций западной части ареала вида (Smulders et al., 2003). Было выявлено резкое снижение разнообразия (вплоть до сохранения единственного аллеля экзона 2 гена DRB) в популяциях Франции и Голландии, тогда как анализ музейных образцов продемонстрировал, что еще в начале прошлого века в этих популяциях присутствовало, по крайней мере, еще 7 аллелей. С резким сокращением аллельного разнообразия DRB в крайней западной части ареала вида связывают снижение средней массы тела, ухудшение размножения и снижение устойчивости к паразитарной нагрузке. В то же время в современной популяции из Чехии (Восточная Европа) у 15 животных было отмечено 13 различных аллелей гена DRB, и большинство животных были гетерозиготными (Smulders et al., 2003). Учитывая, что обыкновенный хомяк в последние 50 лет начал активно заселять города, остро встал вопрос – насколько благополучными являются синурбические популяции этого вида? Ранее мы исследовали особенности распространения митохондриальных линий в популяциях обыкновенного хомяка в ряде городов Кавказа и Крыма (Феоктистова и др., 2016; 2019) и показали, что в городах разнообразие этих линий обеднено. Однако в городских популяциях обнаружены и уникальные варианты, не встречающиеся вне города. Так, на территории города Симферополя отмечено только три из семи вариантов объединенного участка гена цитохрома b и контрольного региона, представленных на Крымском полуострове. При этом два из них были уникальными для этого города.
Проведенный нами анализ аллельного состава гена DRB показал, что, напротив, именно городская популяция отличается повышенным разнообразием этого гена по всем показателям (число аллелей, отмеченных в популяции, среднее число аллелей в генотипе особи, рассчитанные для популяции индексы гаплотипического и нуклеотидного разнообразия). Число аллелей (11) в сельских популяциях обыкновенного хомяка на Крымском полуострове было несколько ниже, чем для благополучной негородской популяции Чехии (13), но для Симферополя это значение (19) было существенно большим. Хотя число несинонимичных замен превышало число синонимичных в обоих выборках с Крымского полуострова, соотношение dN/dS и значение показателя действия положительного отбора в городской популяции было заметно выше, чем сельской.
Полученные результаты позволяют предполагать, что представители городской популяции Симферополя хорошо “подготовлены” к противостоянию “городским вызовам”, обладая большей устойчивостью к инфекциям и паразитарной нагрузке. Скорее всего, это является следствием отбора к специфическим условиям городской среды.
Финансирование. Работа выполнена при финансовой поддержке грантов РФФИ (20-04-00102а) и (19-34-90059).
Список литературы
Сорокин А.Г. Современные проблемы сохранения и восстановления популяций редких птиц // Сохранение и восстановление биоразнообразия. Разд. V. М.: Изд-во Научного и учебно-методического центра, 2002. С. 131–147.
Суров А.В., Поплавская Н.С., Богомолов П.Л., Кропоткина М.В., Товпинец Н.Н., Кацман Е.А., Феоктистова Н.Ю. Синурбанизация обыкновенного хомяка (Cricetus cricetus L., 1758) // Росс. журн. биологических инвазий 2015. Т. 8. № 4. С. 105–116.
Товпинец Н.Н., Евстафьев И.Л., Карасева Е.В. Склонность к синантропии обыкновенного хомяка (Cricetus cricetus) по наблюдениям в Крыму. В сб.: Фауна в антропогенном ландшафте. Мат. териологической школы. 2006. № 8. 136–145.
Феоктистова Н.Ю., Мещерский И.Г., Суров А.В., Богомолов П.Л., Товпинец Н.Н., Поплавская Н.С. Генетическая структура городской популяции обыкновенного хомяка (Cricetus cricetus) // Генетика. 2016. Т. 52. № 2. С. 221–221.
Феоктистова Н.Ю., Мещерский И.Г., Богомолов П.Л., Мещерский С.И., Саян А.С., Ситникова Е.Ф., Власов А.А., Власова О.П., Суров А.В. К вопросу о границе между двумя филогруппами обыкновенного хомяка (Cricetus cricetus) (Cricetinae, Rodentia) // Поволжский экол. журн. 2019. № 4. С. 485–494.
Феоктистова Н.Ю., Мещерский И.Г., Богомолов П.Л., Мещерский С.И., Кацман Е.А., Пельгунова Л.А., Поташникова Е.П., Суров А.В. Непреднамеренно поставленный эксперимент–заселение вновь созданного городского парка видом-синурбистом обыкновенным хомяком Cricetus cricetus L., 1758 // Изв. РАН. Сер. биол. 2020. № 2. С. 224–232.
cevedo-Whitehouse K., Cunningham A.A. Is MHC enough for understanding wildlife immunogenetics? // Trends in Ecol. Evol. 2006. № 21(8). P. 433–438
Babik W., Durka W., Radwan J. (2005). Sequence diversity of the MHC DRB gene in the Eurasian beaver (Castor fiber) // Mol. Ecology. V. 14(14). P. 4249–4257.
Banaszek A., Ziomek J. The common hamster (Cricetus cricetus L) population in the city of Lublin // Ann. Univer. Mariae Currie Sklodowska Lublin Polonia. 2010. V. 65. № 1. P. 59–66.
Banaszek A., Jadwiszczak K.A., Ziomek J. Genetic variability and differentiation in the Polish common hamster (Cricetus cricetus L.): genetic consequences of agricultural habitat fragmentation // Mamm. Biol. 2011. V. 76(6). P. 665–671.
Banaszek A., Bogomolov P., Feoktistova N., La Haye M., Monecke S., Reiners T.E., Rusin M., Surov A., Weinhold U., Ziomek J. // Cricetus Cricetus. The IUCN Red List of Threatened Species. 2020.
https://doi.org/ -2. RLTS.T5529A111875852.en (accessed on 28 March 2020).https://doi.org/10.2305/IUCN.UK.2020
Bandelt H.-J., Forster P., Röhl A. Median-joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16. P. 37–48.
https://doi.org/10.1093/oxfordjournals.molbev.a026036
Biedrzycka A., Kloch A., Buczek M., Radwan J. Major histocompatibility complex DRB genes and blood parasite loads in fragmented populations of the spotted suslik Spermophilus suslicus // Mamm. Biology. 2011. V. 76(6). P. 672–677.
Boggie M.A., Mannan R.W. Examining seasonal patterns of space use to gauge how an accipiter responds to urbanization // Landscape and Urban Planning. 2014. V. 124 P. 34–42.
Bontrop R.E., Otting N., de Groot N.G., Doxiadis G.G. Major histocompatibility complex class II polymorphisms in primates // Immunological reviews. 1999. V. 167(1). P. 339–350.
Bowen L., Aldridge B.M., Gulland F., Van Bonn W., DeLong R., Melin S., Lowenstine L.J., Stott J.L., Johnson M.L. Class II multiformity generated by variable MHC-DRB region configurations in the California sea lion (Zalophus californianus) // Immunogenetics. 2004. V. 56(1). P. 12–27.
Brown J.H., Jardetzky T.S., Gorga J.C., Stern L.J., Urban R.G., Strominger J.L., Wiley D.C. Three-dimensional structure of the human class II histocompatibility antigen HLA-DR1 // Nature. 1993. V. 364(6432). P. 33–39.
Brown J.L., Eklund A. Kin recognition and the major histocompatibility complex: an integrative review // The American Naturalist. 1994. V. 143(3). P. 435–461.
Buczek T. Inwentaryzacja chomika europejskiego Cricetus cricetus na obszarze Górek Czechowskich w Lublinie. 2019
Bushnell B., Rood J., Singer E. BBMerge – Accurate paired shotgun read merging via overlap // PLoS ONE. 2017. V. 12(10). e0185056.
Chiappero M.B., PanzettaDutari G.M., Gomez D. et al. Contrasting genetic structure of urban and rural populations of the wild rodent Calomys musculinus (Cricetidae, Sigmodontinae) // Mammal. Biol. 2011. V. 76. P. 41–50.
https://doi.org/10.1016/j.mambio.2010.02.003
Čanády A. New site of the European hamster (Cricetus cricetus) in the urban environment of Košice city (Slovakia) // Zoology and Ecology. 2013. V. 23. P. 61–65.
Edwards S.V., Potts W.K., Polymorphism of genes in the major histocompatibility complex (MHC): Implications for conservation genetics of vertebrates. In: Molecular Genetic Approaches in Conservation (eds TB Smith and RK Wayne) Oxford University Press. N.Y., 1996. P. 214–237.
Endres J., Weber U. Möglichkeiten und Maßnahmen zur langfristigen Erhaltung des Feldhamsters (Cricetus cricetus L.) im Nordbereich der Universität Göttingen // Dokumentation zum PilotForschungsprojekt “Feldhamster” am Institut für Wildbiologie und Jagdkunde der Georg-August-Universität Göttingen, 1999. 263 p. (in German)
Excoffier L., Laval G., Schneider S. Arlequin ver. 3.0: An integrated software package for population genetics data analysis // Evol. Bioinform. Online. 2005. V. 1. P. 47–50. PMCID: PMC2658868
Feoktistova N.Y., Surov A.V., Tovpinetz N.N., Kropotkina M.V., Bogomolov P.L., Siutz C., Haberl W., Hoffmann I.E. The common hamster as a synurbist: a history of settlement in European cities // Zoologica Poloniae. 2013. V. 58(3–4). P. 116.
Feoktistova N.Y., Meschersky I.G., Bogomolov P.L., Sayan A.S., Poplavskaya N.S., Surov A.V. Phylogeographic structure of the Common hamster (Cricetus cricetus L.): Late Pleistocene connections between Caucasus and Western European populations // PLoS One. 2017. V. 12(11). e0187527.
Fraser D.G., Bailey E. Demonstration of three DRB locl in a domestic horse family // Immunogenetics. 1996. V. 44(6). P. 441–445.
Gomez-Mestre I., Tejedo M. Contrasting patterns of quantitative and neutral genetic variation in locally adapted populations of the natterjack toad, Bufo calamita. // Evolution. 2004. V. 58. P. 2343–2352.
Gliwicz J. Ecological aspects of synurbization of Striped Field Mouse Apodemus agrarius (Pall.) // Wiadomoœci Ekol. 1980. V. 26. P. 185–196.
Harf R., Sommer S. Association between major histocompatibility complex class II DRB alleles and parasite load in the hairy-footed gerbil, Gerbillurus paeba, in the southern Kalahari // Mol. Ecology. 2005. V. 14(1). P. 85–91.
Harris S.E., Munshi-South J., Obergfell C., O’Neill R. Signatures of rapid evolution in urban and rural transcriptomes of white-footed mice (Peromyscus leucopus) in the New York metropolitan area // PLoS One. 2013. V. 8(8). e74938.
Harris S.E., Munshi-South J. Scans for positive selection reveal candidate genes and local adaptation of Peromyscus leucopus populations to urbanization // bioRxiv. 2016. 038141.
Harris S.E., Munshi-South J. Signatures of positive selection and local adaptation to urbanization in white-footed mice (Peromyscus leucopus) // Mol. Ecology. 2017. V. 26(22). P. 6336–6350.
Hedrick P.W., Miller P.S. Rare alleles, MHC and captive breeding // In Conservation genetics. 1994. P. 187–204. Birkhäuser, Basel.
Hedrick P.W. Conservation genetics: Where are we now? Trends Ecol. Evol. 2001. V. 16. P. 629–636.
Hill A.V.S., Allsopp C.E.M., Kwiatowski D., Anstey N.M., Twumasi P., Rowe P.A., Bennett S., Brewster D., McMichael A.J., Greenwood B.M. Common West African HLA antigens are associated with protection from severe malaria // Nature. 1991. V. 352. P. 595–600.
Janeway C., Travers P., Walport M., Shlomchik M. Immunobiology. Garland Science. N.Y., 2004.
Kajdacsi B., Costa F., Hyseni C. et al. Urban population genetics of slum dwelling rats (Rattus norvegicus) in Salvador, Brazil // Mol. Ecol. 2013. V. 22. P. 5056–5070.
https://doi.org/10.1111/mec.1245517
Kennedy L., Ryvar R., Gaskell R., Addie D., Willoughby K., Carter S., Thomson W., Ollier W.E.R., Radford A. Sequence analysis of MHC DRB alleles in domestic cats from the United Kingdom // Immunogenetics. 2002. V. 54(5). P. 348–352.
Khazand M., Peiberg C., Nagy M., Sauermann U. Mhc-DQ-DRB haplotype analysis in the rhesus macaque: evidence for a number of different haplotypes displaying a low allelic polymorphism // Tissue antigen.1999. V. 54(6). P. 615–624.
Klein J., Figueroa F. Evolution of the major histocompatibility complex // Critical reviews in immunology. 1986. V. 6(4). P. 295–386.
Kumar S., Stecher G., Li M., Knyaz C., Tamura K. Mol. Biol. and Evol. 2018. V. 35. P. 1547–1549.
Kupfernagel C. Raumnutzung umgesiedelter Feldhamster Cricetus cricetus (Linnaeus, 1758) auf einer Ausgleichsfläche bei Braunschweig // Braunschweiger Naturkundliche Schriften. 2003. V. 6. № 4. P. 857–887.
Langmead B., Salzberg S. Fast gapped-read alignment with Bowtie 2. Nature Methods. 2012. V. 9. P. 357–359.
Lewin A.Y., Long C.P., Carroll T.N. The coevolution of new organizational forms // Organ. Science. 1999. V. 10(5). P. 535–550.
Losík J., Lisická L., Hříbková J., Tkadlec E. Demografická struktura a procesy v přírodní populaci křečk (Cricetus cricetus) na Olomoucku // Lynx (Praha), n. s. 2007. V. 38. P. 21–29.
Luikart G., England P.R., Tallmon D., Jordan S., Taberlet P. The power and promise of population genomics: from genotyping to genome typing // Nat. Rev. Genet. 2003. V. 4. P. 981–999.
Luniak M. Synurbization–adaptation of animal wildlife to urban development. In Proceedings 4th international urban wildlife symposium. 2004. (P. 50–55). Tucson: University of Arizona.
Matysek M., Hêdzak M., Kuc M., Osmolska A. The occurrence site of the common hamster Cricetus cricetus in Krakow // [In Polish with English summary] Chroñmy Przyr. Ojcz. 2013. V. 69(5). P. 430–436
McKay J.K., Latta R.G. Adaptive population divergence: markers, QTL and traits. Trends in ecology and evolution, 2002. V. 17(6). P. 285–291.
Morinha F., Ramos P.S., Gomes S., Mannan R.W., Guedes-Pinto H., Bastos E. Microsatellite markers suggest high genetic diversity in an urban population of Cooper’s hawks (Accipiter cooperii) // J. Genetics. 2016. V. 95(1). P. 19–24.
MunshiSouth J., Nagy C. Urban Park character istics, genetic variation, and historical demography of whitefooted mouse (Peromyscus leucopus) populations in New York City, Peer J., 2014. V. 2. P. 310–315.
https://doi.org/10.7717/peerj.310
Neumann K., Jansman H., Kayser A., Maak S., Gattermann R. Multiple bottlenecks in threatened western European populations of the common hamster Cricetus cricetus (L.) // Conservation Genetics. 2004. V. 5(2). P. 181–193.
Neumann K., Michaux J., Maak S., Jansman A.H., Kayser A., Mundt G., Gattermann R. Genetic spatial structure of European common hamsters (Cricetus cricetus) – a result of repeated range expansion and demographic bottlenecks // Mol. Ecol. 2005. V. 14. P. 1473–1483.
Niethammer J. Cricetus cricetus (Linnaeus, 1758) – Feldhamster // In: Handbuch der Säugetiere Europas / Eds. J. Niethammer, F. Krapp. Wiesbaden, 1982. Bd. 2/1, Rodentia II. P. 7–28.
Palo J.U., O’Hara R.B., Laugen A.T., Laurila A., Primmer C.R., Merila J. Latitudinal divergence of common frog (Rana temporaria) life history traits by natural selection: Evidence from a comparison of molecular and quantitative genetic data. // Mol. Ecol. 2003.V. 12. P. 1963–1978.
Petrová I., Petriláková M., Losík J., Gouveia A., Damugi I.E., Tkadlec E. Density-related pattern of variation in body growth, body size and annual productivity in the common hamster // Mamm. Biol. 2018. V. 91(1). P. 34–40.
Pfrender M.E., Spitze K., Hicks J., Morgan K., Latta L., Lynch M. Lack of concordance between genetic diversity estimates at the molecular and quantitative-trait levels // Conserv.Genet. 2000. V. 1. P. 263–269.
Potts W.K., Wakeland E.K. Evolution of MHC genetic diversity: a tale of incest, pestilence and sexual preference // Trends in Genetics. 1993. V. 9(12). P. 408–412.
Reed D.H., Frankham R. How closely correlated are molecular and quantitative measures of genetic variation? A meta-analysis // Evolution. 2001. V. 55. P. 1095–1110
Reiners T.E., Eidenschenk J., Neumann K., Nowak C. Preservation of genetic diversity in a wild and captive population of a rapidly declining mammal, the Common hamster of the French Alsace region // Mammal. Biol. 2014. V. 79(4). P. 240–246.
Richmond T.J., Davey C.A. The structure of DNA in the nucleosome core // Nature. 2003. V. 423(6936). P. 145–150.
Schilthuizen M. Darwin in de stad: evolutie in de urban jungle. Atlas Contact; 2018. May 23.
Schmelzer E., Millesi E. Surface activity patterns in a population of European hamsters (Cricetus cricetus) in an urban environment // In: The Common Hamster in Europe. Ecology, management, genetics, conservation, reintroduction. Proceedings Meeting of the International Hamster Workgroup / Ed. G. Nechay. 2008. P. 19–22.
Shiina T., Yamada Y., Aarnink A., Suzuki S., Masuya A., Ito S., Ido D., Yamanaka H., Iwatani C., Tsuchiya H., Ishigaki H., Itoh Y., Ogasawara K., Kulski J.K., Blancher A. Discovery of novel MHC-class I alleles and haplotypes in Filipino cynomolgus macaques (Macaca fascicularis) by pyrosequencing and Sanger sequencing // Immunogenetics, 2015. V. 67(10). P. 563–578.
Serieys L.E., Lea A., Pollinger J.P., Riley S.P., Wayne R.K. Disease and freeways drive genetic change in urban bobcat populations // Evol. Applic. 2015. V. 8(1). P. 75–92.
Smulders M.J.M., Snoek L.B., Booy G., Vosman B. Complete loss of MHC genetic diversity in the Common Hamster (Cricetus cricetus) population in the Netherlands. Consequences for conservation strategies // Cons. Genet. 2003. V. 4(4). P. 441–451.
Surov A., Banaszek A., Bogomolov P., Feoktistova N., Monecke S. Dramatic global decrease in the range and reproduction rate of the European hamster Cricetus cricetus // Endangered Species Res. 2016. V. 31. P. 119–145.
Thorns H.-J. Die Göttinger Feldhamster-Story // BUND Landesverband Niedersachsen e.V., BUNDmagazin. 1998 // 4. http://www.bundniedersachsen.de/service/bundmagazin/41998/die_goettinger_feldhamster_story/
Ujvari B., Olsson M., Madsen T. Discrepancy in mitochondrial and nuclear polymorphism in meadow vipers (Vipera ursinii) questions the unambiguous use of mtDNA in conservation studies // Amphibia-Reptilia. 2005. V. 26(3). P. 287–292.
Ujvari B., Belov K. Major histocompatibility complex (MHC) markers in conservation biology // Intern. J. Mol. Sci. 2011. V. 12(8). P. 5168–5186.
Villesen P. FaBox: an online toolbox for fasta sequences // Mol. Ecol. Notes. 2007. V. 7. P. 965–968.
Дополнительные материалы отсутствуют.
Инструменты
Известия РАН. Серия биологическая


