Кристаллография, 2021, T. 66, № 5, стр. 765-770
Структура мутантной формы белка-регулятора трансляции Hfq с удлиненной петлей L4
А. А. Алипов 1, Н. В. Леконцева 1, А. О. Михайлина 1, М. С. Фандо 1, С. В. Тищенко 1, А. Д. Никулин 1, *
1 Институт белка РАН
Пущино, Россия
* E-mail: nikulin@vega.protres.ru
Поступила в редакцию 17.09.2020
После доработки 03.10.2020
Принята к публикации 06.10.2020
Аннотация
Бактериальный белок Hfq принадлежит к обширному семейству Lsm белков, имеющих консервативную третичную структуру и формирующих стабильную четвертичную структуру в виде колец из нескольких мономеров. Бактериальные белки формируют гексамеры, а их архейные и эукариотические гомологи – гептамеры. Причина формирования высоко гомологичными Lsm белками с одинаковой третичной структурой мультимеров с различающимся числом мономеров не установлена и является предметом активной дискуссии. Экспериментально проверено влияние вариабельной по длине петли L4 на четвертичную структуру белка Hfq. Полученный белок сохраняет гексамерную четвертичную структуру, а удлиненная петля отходит от поверхности центральной части белка в разные стороны. Таким образом, не удалось изменить четвертичную структуру белка, что подтверждает ее высокую стабильность. При этом РНК-связывающие свойства мутантной формы белка практически не изменились.
ВВЕДЕНИЕ
РНК-связывающий белок Hfq является регулятором трансляции большого числа бактериальных генов [1–3]. Он способствует специфическому взаимодействию малых регуляторных РНК с мРНК, что приводит к активации или репрессии трансляции. Белок Hfqпринадлежит к обширному семейству Lsm белков, представители которого имеются в бактериях, археях и эукариотах. Их объединяет консервативная пятитяжевая β-структура с N-концевой α-спиралью общей протяженностью ~70 аминокислотных остатков (а.о.). На С-конце у Lsm белков наблюдается неупорядоченная и не консервативная последовательность от 3 до 30 а.о. [4]. Характерная особенность Lsm белков – формирование ими кольцевой четвертичной структуры из шести (бактериальные Hfq) или семи (эукариотические Sm/Lsm и архейные SmAP) мономеров [4]. Имеются единичные примеры формирования белками этого семейства пентамеров [5] и октамеров [6], но это исключение из общего правила. Начиная со времени объединения Lsm белков в один класс, перед исследователями стоял вопрос о причине формирования столь близкими по пространственной структуре белками четвертичной структуры с разным числом мономеров. Выдвигались гипотезы о влиянии на четвертичную структуру C-концевой части белка [7–10] и петли L4 между тяжами β3 и β4 [11, 12]. Первая гипотеза не получила подтверждения, поскольку все известные бактериальные белки Hfq формируют только гексамеры, несмотря на различную длину С-концов – от 3 до 25 а.о., а архейные и эукариотические белки формируют гептамеры, несмотря на значительное различие С-концевых последовательностей. Более того, экспериментально показано, что делеция С-концевой части белка Hfq не сказывается на его четвертичной структуре [13, 14]. Вторая гипотеза имеет больше оснований: все гексамерные белки имеют короткую последовательность петли L4 (включая Hfq-подобный архейный белок из Methanococcus jannaschii), а все гептамерные – удлиненную последовательность этой петли. Исключением является белок AF-SM2 (SmAP2 белок из археи Archeoglobus fulgidus), который может менять четвертичную структуру в зависимости от рН раствора или присутствия РНК [12]. Эта гипотеза неоднократно обсуждалась в литературе, но экспериментальной проверки влияния длины петли на четвертичную структуру Lsm белков не проводилось. В представленной работе получена генно-инженерная конструкция, несущая ген белка Hfq из P. aeruginosa со вставкой последовательности, кодирующей протяженную петлю L4 архейного SmAP белка. Белок был выделен, охарактеризованы его РНК-связывающие свойства, определена пространственная структура с помощью рентгеноструктурного анализа.
МАТЕРИАЛЫ И МЕТОДЫ
Клонирование и получение штамма-продуцента. Генетическую конструкцию, кодирующую ген белка PaeHfqL4 со вставкой, получали методом сайт-направленного мутагенеза, следуя протоколу QuikChange® Site-Directed Mutagenesis Kit (Stratagene, США). Нуклеотидная последовательность вставки соответствовала последовательности гена SmAP белка AF-SM1 из Archaeoglobus fulgidus (UNIPROT O29386), кодирующего длинную петлю L4 (рис. 1). Используемые праймеры (Синтол, Россия) содержали необходимую вставку:
Рис. 1.
Сравнение последовательностей белков AF-SM1 из Archaeoglobus fulgidus, Hfq из Pseudomonas aeruginosa дикого типа (PaeHfq) и с удлиненной петлей L4 (PaeHfqL4). Выделена последовательность удлиненной петли L4.

F-PaeHfqL4:
5'-TTCAGAACGGCGAAGTGGTGCGTAAAG TGGTCAGCCAGATGGTTTACA-3'
R-PaeHfqL4:
5'-CACTTCGCCGTTCTGAATTTCTTCCGCGGTGTTCTTCAGCAGGATGA-3'.
Для амплификации ДНК использовали KOD Hot Start ДНК-полимеразу. ДНК-матрицей в ПЦР служила плазмида рЕТ-22b(+)PaeHfq [15], несущая ген белка Hfq из P. aeruginosa дикого типа.
Выделение и очистку белков дикого типа PaeHfq и его мутантной формы PaeHfqL4 проводили по схеме, разработанной в [15]. Дополнительной стадией очистки стала гель-фильтрация. Сконцентрированный до 1 мл препарат белка наносили на колонку со смолой Superdex G75 (объем колонки 120 мл), уравновешенную буфером 200 мМ NaCl, 50 мМ Трис-HCl, pH 8.0.
Получение фрагментов РНК. Для анализа РНК-белковых взаимодействий использовали фрагменты двух разных РНК. Фрагмент малой некодирующей РНК DsrA длиной 42 нуклеотида (DsrA42; 5'-GGGUGUAACGAAUUUUUUAUUCGUUUGAAAUUCGUUACACCC-3') и фрагмент 5'-НТО rpoS мРНК длиной 40 нуклеотидов (rpoS40; 5'-GGCAUUUUGAAAUUCGUUACAAGGGGAAAUCCGUAAACCC-3') нарабатывали транскрипцией in vitro с рестрицированных по сайту EcoRI плазмид pUC18, содержащих гены фрагментов РНК под контролем промотора РНК-полимеразы фага Т7. Получение и очистка DsrA42 РНК описаны в [16]. Фрагмент гена мРНК rpoS40 клонировали в вектор pUC18 с использованием перекрывающихся праймеров (Синтол, Россия), причем прямой олигонуклеотид содержал последовательность Т7 промотора:
F-rpoS40:
5'-CTACTGCAAAGCTTAATACGACTCACTA TAGGGCAUUUUGAAAUUCGUUACAAGG-3'
R-rpoS40:
5'-ATCTAGCCCGGGTTTACGGATTTCCCC TTGTAACGAATTTC-3'.
Очистку наработанного транскрипцией in vitro фрагмента мРНК rpoS40 проводили по методике для очистки DsrA42 [16].
Кинетический анализ взаимодействия белков PaeHfq и PaeHfqL4 с фрагментами РНК проводили методом поверхностного плазмонного резонанса [17] на системе ProteOn XPR36 (Bio-Rad, США). Присоединение к фрагментам РНК биотинилированного олигонуклеотида проводили как описано в [16].
Биотинилированный фрагмент РНК наносили на сенсорный NLC-чип (Bio-Rad, США) c иммобилизованным авидином до достижения единиц ответа 500–1000. Поверхность чипа отмывали от несвязавшейся РНК 0.1%-ным раствором додецилсульфата натрия. Затем пропускали препарат белка с различной концентрацией (0.15–1.15 нМ) cо скоростью 30 мкл/мин. Серии разведений из пяти концентраций аналита (PaeHfq или PaeHfqL4) готовили с использованием буфера, содержащего 50 мМ Трис-HCl, pH25°С 7.5, 175 мМ NaCl, 2 мМ MgCl2, 0.05% Tween-20. Время ассоциации составляло 360 с, диссоциации – 2500 с. Все эксперименты по связыванию проводили при температуре 25°C. Серию из трех–пяти сенсограмм обсчитывали в программе BIAEvaluation с использованием модели Ленгмюра (1:1).
Кристаллизацию белка PaeHfqL4 проводили методом диффузии паров в висящей капле при 23°C. Препарат белка (1–2 мкл) с концентрацией 12 мг/мл в 200 мМ NaCl, 50 мМ Трис-HCl, pH 8.0, смешивали с равным объемом противораствора. Кристаллы белка получены в условиях № 3 набора JBScreen Nuc-Pro 1 (Jena Bioscience, Германия): 5% ПЭГ400, 50 мM PIPES 7.0, 30 мM MgCl2. Кристаллы появлялись в течение 3–5 дней и достигали размера 0.4 × 0.4 × 0.02 мм.Перед замораживанием кристаллы вымачивали в криорастворе с увеличенным содержанием ПЭГ 400.
Получение наборов кристаллографических данных. Дифракционный набор данных получен на детекторе DECTRIS PILATUS3S 6M линии ID23-1 синхротрона ESRF (Гренобль, Франция) с кристалла, охлажденного до температуры 100 K, на длине волны 0.97242 Å. Для сбора данных использовали метод качания–вращения с углом осцилляции 0.2°, суммарный угол вращения составил 200°. Первичные дифрактограммы были обработаны в программе XDS [18] и интегрированы в программе AIMLESS комплекса ССР4 [19]. Детали результата обработки и статистические характеристики набора данных представлены в табл. 1.
Таблица 1.
Статистические характеристики экспериментальных данных и уточнения структуры белка PaeHfqL4 (PDB ID 6XYJ)
| Длина волны, на которой получали дифракционные данные, Å | 0.9724 |
| Область разрешения, Å | 50–2.79 (2.89–2.79) |
| Пр. гр. ячейки кристалла | C21 |
| a, b, c, Å; α, β, γ, град | 76.8, 112.6, 78.0; 90.0, 119.4, 90.0 |
| Общее число отражений (рефлексов) | 52 850 (4964) |
| Из них уникальных | 14 417 (1396) |
| Избыточность | 3.7 (3.6) |
| Полнота, % | 98.7 (93.0) |
| Среднее значение I/σ(I) | 12.2 (0.8) |
| B-фактор Вильсона | 76.2 |
| R-merge | 0.079 (1.60) |
| R-meas | 0.093 (1.88) |
| R-pim | 0.047 (0.965) |
| CC1/2 | 1.0 (0.56) |
| CC* | 1.0 (0.85) |
| Число отражений, использованных при уточнении | 14 320 (1320) |
| Число отражений, использованных для расчета R-free | 710 (68) |
| Rwork | 0.218 (0.435) |
| Rfree | 0.279 (0.410) |
| CCwork | 0.954 (0.604) |
| CCfree | 0.929 (0.651) |
| Число неводородных атомов в модели | 3862 |
| СКО для длин связей, Å | 0.013 |
| СКО для валентных углов, град | 1.79 |
| Средний B-фактор для атомов | 105.8 |
| Число аминокислотных остатков на карте Рамачандрана | |
| В предпочтительных областях, % | 93 |
| В допустимых областях, % | 7 |
| В запрещенных областях, % | 0 |
Определение и уточнение структуры белка PaeHfqL4. Структуру белка определяли методом молекулярного замещения с использованием программы MorDA [20]. Исходная модель для решения была выбрана программой автоматически, на основе заданной аминокислотной последовательности белка PaeHfqL4. Найденное решение оценивали на соответствие экспериментальным данным по картам электронной плотности (ЭП) 2mFo-DFc и mFo-DFc. Уточнение структуры белка осуществляли в программе PHENIX [21]. На первом этапе проводили уточнение ориентации мономеров белка в гексамере, рассматривая каждый мономер в качестве независимого “твердого тела” с приписанным общим В-фактором для всех атомов, входящих в одну из шести субъединиц (rigid body refinement). На втором этапе уточнения использовали протокол “симуляции отжига” – молекулярной динамики с ограничениями по имеющимся экспериментальным дифракционным данным (simulated annealing). На этом этапе уточняли индивидуальные значения В-фактора для каждого атома.
После этих этапов в результирующей карте ЭП идентифицировали участок, который соответствовал добавленной к структуре белка Hfq петли L4 длиной 13 а.о., и в соответствующий участок ЭП вписывали недостающие а.о. с помощью программы COOT [22]. После завершения итеративного дополнения аминокислотных последовательностей мономеров белка до полных проводили несколько раундов уточнения структуры. На первых этапах использовали протокол “симуляции отжига” с уточнением индивидуальных значений В-фактора для каждого атома. На последних этапах уточнения, когда проводили тонкую подгонку значений длин связей, валентных и торсионных углов для подготовки финальной модели, использовали протокол без “симуляции отжига”, но с оптимизацией весовых параметров соотношения геометрии/свободной энергии модели.
Перед депонированием структуры белка в банк данных проводили тщательную проверку геометрии белка с помощью сервиса MolProbity, встроенного в программный комплекс PHENIX. Найденные ошибки исправляли с помощью программы молекулярной графики СООТ и подвергали повторному уточнению. Результирующая структура была уточнена в программе REFMAC комплекса ССР4 и депонирована в банк данных с присвоением PDBID 6XYJ. Статистические характеристики уточнения структуры приведены в табл. 1.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Структура белка Hfq из P. aeruginosa с удлиненной петлей L4. Для определения структуры белка Hfq из P. aeruginosa с удлиненной петлей L4 (PaeHfqL4) использовали метод молекулярного замещения. Поскольку ожидалось, что число мономеров в исследуемом белке будет отличаться от их количества в исходной структуре, для решения задачи молекулярного замещения использовали программу MorDA. Она позволяет при поиске решения задачи молекулярного замещения в автоматическом режиме тестировать ряд возможных вариантов исходных моделей, автоматически отбираемых из банка данных на основе гомологии первичных структур. Такой режим работы программы в данном случае позволяет значительно ускорить нахождение решения при неопределенности числа мономеров в асимметричной части ячейки кристалла. В результате работы программы наилучшей исходной моделью был выбран мономер белка Hfq из P. aeruginosa, причем асимметричная часть кристаллической ячейки содержала гексамер белка, а не гептамер, как ожидалось. Карта ЭП, рассчитанная по ориентированной модели гексамера белка, показала наличие для каждого из мономеров белка неописанного участка плотности в области между тяжами β3 и β4 (рис. 2). Этот участок был дополнен а.о., соответствующими последовательности вставки в петлю L4. Модель уточнена до хороших значений стереохимических параметров (с учетом разрешения кристаллографических данных 2.8 Å).
Рис. 2.
Этапы дополнения исходной структуры белка PaeHfq до полной структуры мутантной формы белка с удлиненной петлей L4 на примере одного из мономеров белка (стереопары). Карта электронной плотности 2Fo-DF показана в виде сетки со срезкой 1σ, разрешение 2.8 Å; а – наложение Сα-цепи исходной структуры PaeHfq на карту ЭП; б – наложение Сα-цепи модифицированной модели с добавленными а.о. удлиненной петли L4; в – наложение достроенной модели PaeHfqL4 на карту ЭП.
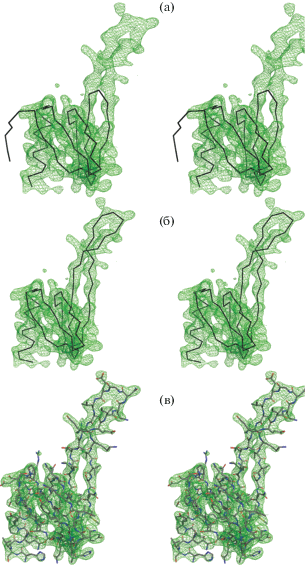
Полученная структура мутантной формы белка PaeHfq с удлиненной петлей L4 показала, что мономер белка сохраняет трехмерную структуру из N-концевой α-спирали и пяти β-тяжей. Вставка в петлю L4 из 13 а.о. в белке PaeHfqL4 приобрела конформацию β-шпильки (рис. 3). Однако в отличие от архейных гомологов, где петля L4 формирует плотно прилегающую к поверхности гептамера β-шпильку, в рассматриваемом случае удлиненная петля L4 отходит от основной части белка в разные стороны, а белок остается гексамером (рис. 4). В результате ожидаемого изменения четвертичной структуры белка не произошло. Возможными причинами этого могут быть компактность и высокая стабильность гексамера Hfq, не позволяющая сближать петли к его центру, а различия в последовательностях бактериального и архейных белков не способствуют формированию гидрофобных контактов и/или водородных связей между а.о. шпильки L4 и коровой частью белка, что способствовало бы “притягиванию” шпильки к ее поверхности. Таким образом, пока нельзя полностью отвергать гипотезу о существенном влиянии длины петли L4 на четвертичную структуру Lsm белков. Окончательный ответ можно получить только при создании мутантной формы Hfq с прилегающей к поверхности мономера длинной петли L4.
Рис. 3.
Структура мономеров исходного белка PaeHfq (а), мутантной формы белка PaeHfqL4 (б), мономера SmAP белка из Archaeoglobus fulgidus (AF-SM1) (в). Обозначены N- и С-концы мономеров, элементы β-слоя, положение петли L4. Цветом выделены удлиненные относительно PaeHfq петли L4 в мономерах PaeHfqL4 и AF-SM1, имеющие конформацию β-шпилек. Ориентация мономеров одинаковая относительно центральной части белка. Наложение главных цепей мономеров белков Hfq (светло-серый) и AF-SM1 (черный) (г). Обозначены N- и С-концы мономеров.
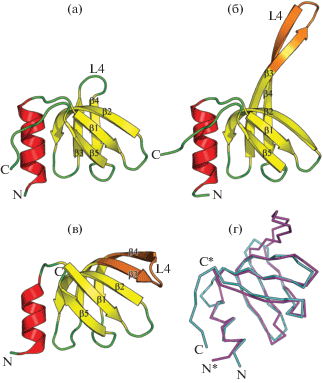
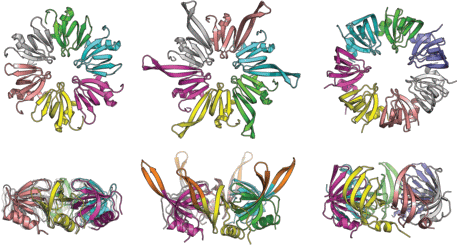
Рис. 4.
Сравнение четвертичной структуры белка PaeHfq дикого типа (слева), его мутантной формы PaeHfqL4 (в центре) и архейного гомолога AF-SM1 (справа). Вверху – вид со стороны дистальной (противоположной стороне с α-спиралями) поверхности белков; внизу – вид сбоку. Участки петель L4 выделены цветом.
Влияние удлинения петли L4 на РНК-связывающие свойства белка PaeHfq. Известно, что белок Hfq действует как РНК-шаперон, облегчая взаимодействие между малыми регуляторными РНК (мрРНК) и их мРНК-мишенями [2]. Так, после взаимодействия Hfq с лидерной последовательностью rpoS мРНК происходит связывание мРНК с DsrA мрРНК [23, 24]. Формирование тройного комплекса мРНК–Hfq–мрРНК приводит к образованию дуплекса rpoS–DsrA и последующей инициации трансляции мРНК. Участки связывания мРНК и мрРНК на белке различаются: мрРНК имеют, как правило, уридин-богатые участки и взаимодействуют с так называемым уридин-связывающим участком на белке, расположенным вблизи петли L4, а мРНК связывается с противоположной стороны гексамера [2]. Третий РНК-связывающий участок белка Hfq расположен на боковой поверхности гексамера белка и является “местом встречи” мРНК и мрРНК [25–28]. Для анализа влияния увеличенной петли L4 на взаимодействие белков со специфическими фрагментами мрРНК DrsA и мРНК rpoS были определены константы диссоциации РНК-белковых комплексов (табл. 2). Показано, что удлинение петли L4 практически не изменило сродство белка к фрагменту лидерной последовательности мРНК rpoS. Поскольку участок связывания мРНК удален от петли L4, это вполне ожидаемый результат. Сродство мутантной формы белка к DsrA мрРНК несколько увеличилось, что можно объяснить наличием дополнительных положительно заряженных а.о.в удлиненной петле. Тем не менее удлиненная петля L4 в целом не оказала значительного влияния на РНК-связывающие свойства белка Hfq, поскольку находится в отдалении от его РНК-связывающих участков.
Таблица 2.
Константы диссоциации (KD) РНК-белковых комплексов
| KD, нM | ||
|---|---|---|
| PaeHfq | PaeHfqL4 | |
| DsrA42 | 0.1 ± 0.01 | 0.04 ± 0.004 |
| RpoS40 | 0.03 ± 0.002 | 0.02 ± 0.007 |
Работа выполнена при поддержке Российского фонда фундаментальных исследований, проект № 18-04-00222.
Список литературы
Васильева Ю.М., Гарбер М.Б. // Молекулярная биология. 2002. Т. 36. № 6. С. 1.
Updegrove T.B., Zhang A., Storz G. // Curr. Opin. Microbiol. 2016. V. 30. P. 133. https://doi.org/10.1016/j.mib.2016.02.003
Sauer E. // RNA Biol. 2013. V. 10. № 4. P. 610.https://doi.org/10.4161/rna.24617
Мурина В.Н., Никулин А.Д. // Успехи биологической химии. 2011. Т. 51. С. 133.
Das D., Kozbial P., Axelrod H.L. et al. // Proteins. 2010. V. 75. № 2. P. 296. https://doi.org/10.1002/prot.22360
Naidoo N., Harrop S.J., Sobti M. et al. // J. Mol. Biol. 2008. V. 377. № 5. P. 1357. https://doi.org/10.1016/j.jmb.2008.01.007
Thore S., Mayer C., Sauter C. et al. // J. Biol. Chem. 2003. V. 278. № 2. P. 1239. https://doi.org/10.1074/jbc.M207685200
Fortas E., Piccirilli F., Malabirade A. et al. // Biosci. Rep. 2015. V. 35. № 2. P. 1. https://doi.org/10.1042/BSR20140128
Arluison V., Folichon M., Marco S. et al. // Eur. J. Biochem. 2004. V. 271. № 7. P. 1258. https://doi.org/10.1111/j.1432-1033.2004.04026.x
Beich-Frandsen M., Vecerek B., Konarev P.V. et al. // Nucl. Acids Res. 2011. V. 39. № 11. P. 4900. https://doi.org/10.1093/nar/gkq1346
Sauter C., Basquin J., Suck D. // Nucl. Acids Res. 2003. V. 31. № 14. P. 4091. https://doi.org/10.1093/nar/gkg480
Törö I., Basquin J., Teo-Dreher H., Suck D. // J. Mol. Biol. 2002. V. 320. № 1. P. 129. https://doi.org/10.1016/S0022-2836(02)00406-0
Beich-Frandsen M., Vecerek B., Sjoblom B. et al. // Acta Cryst. F. 2011. V. 67. № 5. P. 536. https://doi.org/10.1107/S174430911100786X
Olsen A.S., Møller-Jensen J., Brennan R.G., Valentin-Hansen P. // J. Mol. Biol. 2010. V. 404. № 2. P. 173. https://doi.org/10.1016/j.jmb.2010.09.038
Nikulin A., Stolboushkina E., Perederina A. et al. // Acta Cryst. D. 2005. V. 61. № 2. P. 141. https://doi.org/10.1107/S0907444904030008
Nikulin A., Mikhailina A., Lekontseva N. et al. // J. Biomol. Struct. Dyn. 2017. V. 35. № 8. P. 1615. https://doi.org/10.1080/07391102.2016.1189849
Katsamba P.S., Park S., Laird-Offringa I.A. // Methods. 2002. V. 26. № 2. P. 95. https://doi.org/10.1016/S1046-2023(02)00012-9
Kabsch W. // Acta Cryst. D. 2010. V. 66. № 2. P. 125. https://doi.org/10.1107/S0907444909047337
Winn M.D., Ballard C.C., Cowtan K.D. et al. // Acta Cryst. D. 2011. V. 67. № 4. P. 235. https://doi.org/10.1107/S0907444910045749
Vagin A., Lebedev A. // Acta Cryst. A. 2015. V. 71. № a1. P. s19. https://doi.org/10.1107/S2053273315099672
Afonine P.V., Grosse-Kunstleve R.W., Echols N. et al. // Acta Cryst. D. 2012. V. 68. № 4. P. 352. https://doi.org/10.1107/S0907444912001308
Emsley P., Cowtan K. // Acta Cryst. D. 2004. V. 60. № 12 P. 2126. https://doi.org/10.1107/S0907444904019158
Sledjeski D.D., Whitman C., Zhang A. // J. Bacteriol. 2005. V. 183. № 6. P. 1997. https://doi.org/10.1128/JB.183.6.1997-2005.2001
Vecerek B., Beich-Frandsen M., Resch A., Bläsi U. // Nucl. Acids Res. 2010. V. 38. № 4. P. 1284. https://doi.org/10.1093/nar/gkp1125
Chen J., Morita T., Gottesman S. // Front. Cell. Infect. Microbiol. 2019. V. 9. № 6. P. 201. https://doi.org/10.3389/fcimb.2019.00201
Sauer E., Schmidt S., Weichenrieder O. // Proc. Natl. Acad. Sci. 2012. V. 109. № 24. P. 9396. https://doi.org/10.1073/pnas.1202521109
Robinson K.E., Orans J., Kovach A.R. et al. // Nucl. Acids Res. 2013. V. 42. № 4. P. 2736. https://doi.org/10.1093/nar/gkt1171
Panja S., Schu D.J., Woodson S. A. // Nucl. Acids Res. 2013. V. 41. № 15. P. 7536. https://doi.org/10.1093/nar/gkt521
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография


