Кристаллография, 2023, T. 68, № 1, стр. 38-45
Объективные критерии определения стартовых параметров при моделировании структуры мицелл и липосом по данным малоуглового рентгеновского рассеяния
М. В. Петухов 1, 2, *, Э. В. Штыкова 1
1 Институт кристаллографии им. А.В. Шубникова ФНИЦ “Кристаллография и фотоника” РАН
Москва, Россия
2 Институт физической химии и электрохимии им. А.Н. Фрумкина РАН
Москва, Россия
* E-mail: pmxmvl@yandex.ru
Поступила в редакцию 20.09.2022
После доработки 22.09.2022
Принята к публикации 22.09.2022
- EDN: DQNXBJ
- DOI: 10.31857/S0023476123010204
Аннотация
Для изучения структуры гидрофобных мембранных белков используются матрицы, моделирующие клеточные мембраны и образованные молекулами соответствующих амфифильных веществ, например детергентами или липидами. Для изучения структуры белка, встроенного в искусственную мембрану, прежде всего необходимо определить структуру самой мембраны. Для решения этой задачи методом малоуглового рентгеновского рассеяния ранее были разработаны алгоритмы ELLLIP и ELLMIC, позволяющие построить модели эллипсоидальных везикул на основе атомной структуры мономера липида или детергента. Однако результат моделирования в значительной степени зависит от субъективной оценки стартовых значений структурных параметров матриц и может оказаться неверным в силу неоднозначности решения задач такого типа. Представлен независимый подход к определению стартовых размеров модельных мембран для дальнейшего их структурного моделирования, основанный на анализе функции распределения по расстояниям, полученной непосредственно из кривой малоуглового рассеяния.
ВВЕДЕНИЕ
Биологическая мембрана (БМ), состоящая из липидного бислоя как основы или матрицы и связанных с этой матрицей белков, является одним из основных компонентов живой клетки, обеспечивает ее целостность и выполняет ряд функций, необходимых для гомеостаза клеток и рецепции ими внешних и межклеточных сигналов. За большинство из этих функций ответственны интегральные и периферические мембранные белки. Периферические белки взаимодействуют с поверхностью мембраны и не пересекают ее. Их можно отмыть от мембраны с помощью солевых растворов без ее разрушения в отличие от интегральных белков, которые имеют обширные гидрофобные участки и в целом нерастворимы в воде. С липидами мембран интегральные белки связаны гидрофобными взаимодействиями. Эти белки частично погружены в толщу липидного бислоя или пронизывают его, оставляя на поверхности сравнительно небольшие гидрофильные участки. Для структурных исследований интегральных белков или их трансмембранных фрагментов необходимо создать соответствующую среду, имитирующую БМ. В обзоре [1] описаны известные варианты искусственной амфифильной среды на основе различных биоматериалов, обладающих такими типичными свойствами БМ, как сложная фибриллярная структура, вязкоупругие механические свойства, адгезивные последовательности, динамическая ферментативно-индуцированная природа, а также возможность контролируемого хранения и высвобождения таких биоактивных веществ, как, например, факторы роста. Эти биоматериалы изготавливаются из природных и синтетических полимерных систем, пористых материалов, гидрoгелей и других соединений. Однако для структурных исследований в растворе на сегодня наиболее успешными системами, имитирующими БМ, являются липидные двухслойные нанодиски [2–7] и липосомы, представляющие собой сферические или эллиптические липидные бислои [8–13]. Несомненным преимуществом липосом является то, что липидные бислои можно конструировать из разных композиций липидов, наилучшим образом подходящих для стабилизации в растворе соответствующего мембранного белка.
Еще одним важным миметиком, т.е. системой, имитирующей БМ, являются мицеллы. Наиболее активно их используют в спектроскопии ядерного магнитного резонанса [14]. В этом случае для стабилизации в растворе мембранного белка или его трансмембранного фрагмента природный бислой фосфолипидов заменяют на искусственную амфифильную среду, используя поверхностно-активные вещества – детергенты [15–17]. Наиболее часто для определения структуры трансмембранных пептидов используются мицеллы ДФХ (додецилфосфохолин) и ДСН (додецилсульфат натрия) [18–20].
Структурные исследования различных миметиков проводятся также с помощью малоуглового рентгеновского рассеяния (МУРР). Этот метод давно и успешно используется для определения наноструктур различных везикул и ориентированных мембран в растворе [21–28]. Структура всех БМ сходна: бислой состоит из двух поверхностных слоев полярных группировок с высокой электронной плотностью и находящейся между ними внутренней области с низкой электронной плотностью. Миметики на основе липосом имеют именно такое строение в отличие от мицелл, представляющих собой замкнутые сферические, эллипсоидальные или цилиндрические структуры с единственным гидрофильным слоем на поверхности и гидрофобной областью внутри мицеллы. Поэтому для исследования трансмембранных белков их встраивают в гидрофобные части липосом или мицелл. Направление по структурным исследованиям таких систем с помощью МУРР продолжает успешно развиваться. Недавно были опубликованы две взаимосвязанные работы, описывающие алгоритмы, позволяющие определять структуры липосом и мицелл по данным МУРР [29, 30]. В [29] описан новый подход, реализованный в компьютерной программе BILMIX, для восстановления профилей электронной плотности липидных бислоев в липосомах. В [30] представлены квазиатомистический подход и программа ELLLIP для моделирования формы и размера липосом. В совокупности оба подхода дают возможность по данным МУРР детально описать структуру миметиков, в том числе при их взаимодействии с белками [31].
Алгоритм ELLLIP [30], включенный в пакет программ ATSAS [32], позволяет строить модели эллипсоидальных везикул на основе атомной структуры мономера липида. Очевидно, что эта программа может быть адаптирована и для создания моделей однослойных мицелл с использованием при этом атомной структуры детергента. Такой подход реализован в программе ELLMIC. В квазиатомистическом подходе копии модели мономера липида (при моделировании структуры липосом) или детергента (для мицелл) совмещаются с квазиоднородной сеткой угловых направлений путем наложения длинной оси мономера на оси сетки. Для липосом моделируется бислой. Затем каждый мономер детергента или пара липидов для имитации бислоя сдвигаются от центра по соответствующей оси так, чтобы образовался эллипсоид с полуосями A, B, C. Отметим, что программы ELLLIP и ELLMIC позволяют зашумить положения отдельных пар липидов или мономеров детергента, так что они смещаются в соответствующем направлении для того, чтобы учесть флуктуации положения отдельных молекул в липосоме или мицелле. Это необходимо, поскольку отдельным молекулам липидов в бислое или детергентов в мицелле свойственна определенная подвижность. Для обеих программ входными параметрами являются значения полуосей моделируемого эллипсоида; эти параметры до сих пор определялись с помощью длительного скрининга в широком диапазоне значений. Такой подход не только очень затратен по времени, но и потенциально может привести к неверному решению вследствие неоднозначности обратной задачи МУРР. Поэтому в данном исследовании для усовершенствования квазиатомистического подхода была поставлена задача найти объективные критерии определения стартовых значений при моделировании структуры миметиков, т.е. разработать алгоритмы для оценки размеров (полуосей) модельных эллипсоидов непосредственно по кривой МУРР (без априорного 3D-моделирования), чтобы избежать возможной неверной интерпретации данных и исключить длительный скрининг искомых параметров.
МАТЕРИАЛЫ И МЕТОДЫ
Материалы. В работе использованы липосомы, приготовленные из смеси 90% диолеоилфосфатидилхолина (ДОФХ) и 10% диолеоилфосфатидилсерина (ДОФС) (Sigma, США) в концентрации 10 мг/мл. Оба липида приобретены у Avanti Polar Lipids, США. Получение липосом в растворе проведено по методике, описанной в [30, 33].
Мицеллы ДФХ готовили путем растворения в воде детергентов при концентрации выше критической концентрации мицеллообразования.
Квазиатомистическое моделирование структуры мицелл и липосом. Алгоритм ELLLIP для пакета программ ATSAS [32], позволяющий строить модели эллипсоидальных везикул на основе атомной структуры мономера липида, подробно описан в [30]. В настоящей работе этот подход адаптирован к моделированию структуры мицелл и реализован в программе ELLMIC. Разница по сравнению с ELLLIP заключается в том, что вместо двух слоев липидов генерируется только один (монослой).
Эксперимент и анализ данных МУРР. Экспериментальное исследование структуры липосом и мицелл с помощью малоуглового рассеяния синхротронного излучения проводили на синхротроне Petra III (DESY, EMBL, Гамбург) на линии P12 [34]. Станция P12 оснащена оборудованием для автоматической смены образцов и двумерным детектором Pilatus 2M (DECTRIS, Швейцария). Интенсивность рассеяния I(s) измерена в области значений волновых векторов 0.03 < s < 6.0 нм–1, где s = ($4\pi \sin \theta $)/λ, 2θ – угол рассеяния, λ = 0.124 нм – длина волны. Измерения проводили при температуре 20°C, за 1 с снимали 20 отдельных кривых рассеяния с целью отслеживания возможного радиационного повреждения. Первичную обработку данных проводили с помощью программы РRIMUS [35].
Функции распределения по расстояниям p(r), связанные с интенсивностью рассеяния I(s) преобразованием Фурье и необходимые для анализа структуры рассеивающих объектов, вычисляли в программе GNOM [36] в соответствии с уравнением [37]:
(1)
$p(r) = \frac{1}{{2{{{{\pi }}}^{2}}}}\mathop \smallint \limits_0^\infty \,srI(s){\text{sin}}(sr)ds.$Расчет интенсивностей МУРР от полученных с помощью программ ELLLIP и ELLMIC моделей липосом или мицелл с различными параметрами A, B, C проводили с помощью программы CRYSOL [38], рассчитывающей также расхождение между кривой рассеяния от модели Icalc и экспериментальной кривой Iexp, минимизируя невязку χ2:
(2)
${{\chi }}_{{}}^{{\text{2}}} = \frac{1}{{N - 1}}\sum\limits_j {{{{\left[ {\frac{{{{I}_{{{\text{exp}}}}}(s{}_{j}) - c{{I}_{{{\text{calc}}}}}({{s}_{j}})}}{{{{\sigma }}({{s}_{j}})}}} \right]}}^{2}},} $Кривые МУРР от геометрического тела (полого эллипсоида), построенного с помощью вспомогательной программы gen_dam_body, вычисляли программой CRYSOL [38] с последующим зашумлением с помощью программы ADDERRORS из пакета ATSAS. Эти кривые были необходимы для анализа и иллюстрации возможности использования функций распределения по расстояниям p(r) для определения стартовых геометрических параметров моделируемых липосом и мицелл.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Для разбавленных растворов частиц со случайной пространственной ориентацией рассеяние является изотропным и его интенсивность I(s) может быть описана интегралом, ограниченным максимальным размером рассеивающей частицы Dmax:
(3)
$I(s) = 4{{\pi }}\mathop \smallint \limits_0^{{{D}_{{{\text{max}}}}}} {\kern 1pt} p(r)\frac{{\sin (sr)}}{{sr}}dr.$В уравнении (3) функция распределения по расстояниям p(r) связана с интенсивностью рассеяния I(s) обратным преобразованием Фурье (также уравнение (1)).
По сути p(r) представляет собой гистограмму количеств отрезков между каждыми двумя точками в интервале между r и r + dr и распространяется на весь интервал [0, Dmax], где Dmax – максимальное расстояние между двумя точками внутри рассеивающей частицы. Таким образом, эта функция содержит информацию о форме и структуре частицы и позволяет оценивать ее максимальный размер Dmax из условия p(r) = 0 при r > > Dmax. Она используется для ab initio-восстановления формы рассеивающих частиц в однородном приближении с помощью метода конечных объемных элементов [39]. В этом методе для определения формы с низким разрешением используют минимизацию целевой функции с помощью имитации отжига в объеме поиска с диаметром Dmax для создания компактного взаимосвязанного ансамбля виртуальных атомов (шариков), рассеяние от которого соответствует экспериментальным данным. Такой подход реализован в программе DAMMIN [39].
Профиль интенсивности рассеяния и, следовательно, функция распределения по расстояниям p(r) зависят от контраста Δρ(r) между распределением плотности длины рассеяния частицы ρ(r) и плотностью растворителя ρs:
В случае, когда электронная плотность растворителя оказывается выше, чем электронная плотность какой-то части рассеивающего объекта, на профиле функции распределения по расстояниям p(r) появляются отрицательные значения. Такое рассеивающее тело нельзя рассматривать как однородное и использовать метод восстановления формы по экспериментальным данным МУРР с использованием программы DAMMIN. В том числе, это ограничение касается таких структур, как липосомы и мицеллы. Именно поэтому для определения их строения были разработаны программы BILMIX и ELLLIP/ELLMIC, основанные на принципиально других подходах и алгоритмах по сравнению с DAMMIN. Тем не менее соответствующая рассеянию от любого неоднородного тела функция распределения по расстояниям p(r) позволяет достаточно точно определить его максимальный размер Dmax, что значительно сокращает область возможных решений обратной задачи МУРР. Кроме того, поскольку функция распределения по расстояниям – это отображение рассеяния в прямом пространстве, с ее помощью легче анализировать особенности строения и характерные размеры образца, чем по профилю экспериментальной кривой МУРР. Это можно проиллюстрировать на примере рассеяния от полого эллипсоида, имитирующего липосому (рис. 1).
Рис. 1.
Анализ размеров рассеивающего тела по функции распределения по расстояниям p(r): а – шариковая модель полого эллипсоида вращения в разрезе, упрощенно имитирующего строение липосомы, с полуосями A = 15 нм, B = C = 10 нм и толщиной “бислоя” d = 5 нм; б – теоретическая кривая МУРР от данной модели эллипсоида; в – соответствие основных размеров эллипсоида функции распределения по расстояниям p(r), рассчитанной по теоретической кривой рассеяния, показанной на панели (б).

Для построения шариковой модели эллипсоида вращения были использованы параметры полуосей A = 15 нм, B = C = 10 нм и толщина “бислоя” d = 5 нм. Все указанные параметры близки к тем, которые обычно имеют место при получении липосом из молекул липида в растворе с помощью экструдера. Как видно из рис. 1, функция распределения по расстояниям p(r) (рис. 1в) точно отражает все заложенные в модель липосомы размеры, а также размеры внутренней полости и расстояние от центра “липосомы” до середины “бислоя”. Таким образом, анализируя профиль функции p(r), можно определить основные геометрические параметры рассеивающего объекта, которые затем можно использовать в моделировании структуры реального образца в том случае, если невозможно классическое ab initio-восстановление его формы по данным МУРР с использованием, например, программы DAMMIN.
В качестве примера возможности применения такого подхода к моделированию формы и размера реальных липосом в растворе была использована экспериментальная кривая МУРР от липосом, приготовленных из смеси 90% ДОФХ и 10% ДОФС, показанная на рис. 2.
Рис. 2.
Пример использования функции распределения по расстояниям для реальных липосом в растворе p(r): а – экспериментальная кривая МУРР от липосом, приготовленных из смеси 90% диолеоилфосфатидилхолина (ДОФХ) и 10% диолеоилфосфатидилсерина (ДОФС) (1), и теоретическая кривая рассеяния от функции p(r) (2), на вставке функция распределения по расстояниям для реальных липосом в растворе p(r); б – экспериментальная кривая МУРР от липосом (1) и теоретическая кривая рассеяния (2) от модели липосомы с размерами, полученными из анализа функции p(r).

Анализ экспериментальной кривой рассеяния от липосом проводили с помощью программы GNOM, в которой допускаются отрицательные значения p(r), поскольку в мицеллах и липосомах содержатся участки как с положительным (гидрофильные головы), так и с отрицательным (гидрофобные хвосты) контрастом. Эти участки на профиле функции p(r), показанной на вставке рис. 2а, соответствуют толщине липидного бислоя d = = 5.2 нм. Из функции p(r) также определяются полуоси эллипсоида, которым можно аппроксимировать липосому: А = 65 нм, B = 64 нм и C = = 59 нм. Реальные липосомы являлись двухкомпонентными и состояли из 90% ДОФХ и 10% ДОФС. Эти компоненты имеют близкое строение и одинаковый размер с молекулярной длиной 2.8 нм. То есть толщина бислоя должна быть ~5.6 нм или меньше при интеркаляции гидрофобных хвостов липидов. Поэтому для простоты моделирования использовали структуру атомного разрешения только ДОФХ. Полученная модель липосомы показана на рис. 2б. Зашумление поверхностного слоя составляло 0.8 нм. В результате кривая МУРР от этой модели визуально хорошо соответствует экспериментальным данным с χ2 = 8.2. Высокое значение χ2 в данном случае объясняется присутствием осцилляций на теоретической кривой рассеяния от модели липосомы, вызванные рассеянием от форм-фактора липосомы в целом. Отсутствие осцилляций на экспериментальной кривой объясняется усреднением интенсивностей от множества частиц с близкими, но не абсолютно одинаковыми структурными параметрами, т.е. присутствием полидисперсности. Таким образом, экспериментальная кривая МУРР от липосом состоит из двух важных составляющих: во-первых, это рассеяние от формы самой липосомы, во-вторых, рассеяние от форм-фактора липидного бислоя. Последнее в качестве огибающей определяет в целом профиль кривой МУРР от липосом или других похожих рассеивающих объектов. Проведенное моделирование и последующий анализ кривых рассеяния от полученных моделей показали, насколько чувствительным является изменение в ту или другую сторону стартового значения толщины бислоя d. Например, изменение величины d с учетом зашумления с 5.2 нм до 4.8 нм приводит к полному несоответствию экспериментальной и модельной кривых рассеяния. В то же время функция распределения по расстояниям p(r) дает возможность практически однозначно определить этот параметр, что значительно облегчает определение формы и размера липосом в растворе по данным МУРР. Также отметим, что изменение максимального размера при вычислении функции распределения по расстояниям p(r) не столь чувствительно для построения моделей липосом. Этот параметр следует выбирать минимальным из возможных значений, дающих хорошее соответствие с экспериментальными кривыми МУРР.
Для моделирования мицелл использовали эллипсоид с полуосями 4.0, 4.0 и 5.0 нм, построенный из мономеров ДФХ. Кривая рассеяния от такой мицеллы приведена на рис. 3, а соответствующая функция p(r) – на вставке к рис. 3. Из функции p(r) можно достаточно точно определить величины полуосей: А задается положением второго максимума функции (его половиной), а C получается из максимального размера Dmax (также половина). Полученные значения (A = 3.9 нм, C = = 5.2 нм) хорошо соответствуют заданным параметрам.
Рис. 3.
Теоретический пример использования функции распределения по расстояниям для мицелл в растворе: 1 – симулированные экспериментальные точки; 2 – теоретическая кривая рассеяния от функции p(r), на вставке функция распределения по расстояниям от симулированных мицелл в растворе p(r).

Далее предложенный подход применяли к экспериментальным данным от мицелл ДФХ. Экспериментальная кривая МУРР показана на рис. 4а, рассчитанная по ней p(r) дана на вставке к рис. 4а. Полученная функция p(r) соответствует следующим величинам полуосей эллипсоида: A = 2 нм, C = 3.6 нм. Эллипсоид с такими параметрами построен программой ELLMIC на основе мономера ДФХ (рис. 4б). Соответствующая кривая МУРР, рассчитанная программой CRYSOL, практически совпадает с экспериментальной кривой (рис. 4а) с χ2 = 1.2.
Рис. 4.
Пример использования функции распределения по расстояниям p(r) для реальных мицелл в растворе: а – экспериментальная кривая МУРР от мицелл ДФХ (1) и рассчитанная кривая рассеяния от модели мицеллы (2), на вставке функция распределения по расстояниям для реальных мицелл в растворе p(r); б – модель мицеллы с размерами, полученными из анализа функции p(r).
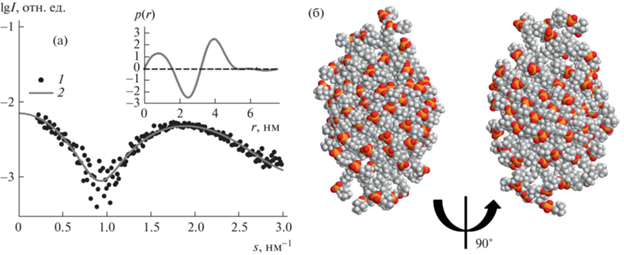
Таким образом, предложенный подход для анализа параметров мицеллы по функции распределения по расстояниям дает разумные значения полуосей, а квазиатомистичная модель мицеллы, построенная из мономеров, позволяет адекватно описать структуру мицеллы и обеспечить согласие с ее данными МУРР.
ЗАКЛЮЧЕНИЕ
Представлены подходы для определения структурных параметров эллиптических мицелл и липосом, используемых для изучения гидрофобных пептидов и белков методом рассеяния в растворе. Для получения величин полуосей эллипсоида используется функция распределения по расстояниям p(r), получаемая непосредственно из кривой рассеяния. Это является важным результатом, поскольку в общем случае данные МУРР при трехмерном моделировании являются потенциально неоднозначными и могут иметь несколько возможных решений. Отметим также универсальность предложенного подхода. Программы ELLLIP и ELLMIC можно использовать для моделирования структуры самых разных рассеивающих объектов, состоящих из повторяющихся элементов высокого разрешения.
Работа выполнена при поддержке Министерства науки и высшего образования в рамках Государственного задания ФНИЦ “Кристаллография и фотоника” РАН.
Список литературы
Jain P., Rauer S.B., Moller M. et al. // Biomacromolecules. 2022. V. 23. № 8. P. 3081. https://doi.org/10.1021/acs.biomac.2c00402
Bayburt T.H., Sligar S.G. // FEBS Lett. 2010. V. 584. № 9. P. 1721. https://doi.org/10.1016/j.febslet.2009.10.024
Knowles T.J., Finka R., Smith C. et al. // J. Am. Chem. Soc. 2009. V. 131. № 22. P. 7484. https://doi.org/10.1021/ja810046q
Lee S.C., Knowles T.J., Postis V.L. et al. // Nat. Protoc. 2016. V. 11. № 7. P. 1149. https://doi.org/10.1038/nprot.2016.070
Orwick M.C., Judge P.J., Procek J. et al. // Angew. Chem. Int. Ed. Engl. 2012. V. 51. № 19. P. 4653. https://doi.org/10.1002/anie.201201355
Smirnova I.A., Sjostrand D., Li F. et al. // Biochim. Biophys. Acta. 2016. V. 1858. № 12. P. 2984. https://doi.org/10.1016/j.bbamem.2016.09.004
Morrison K.A., Akram A., Mathews A. et al. // Biochem. J. 2016. V. 473. № 23. P. 4349. https://doi.org/10.1042/BCJ20160723
Pautot S., Frisken B.J., Weitz D.A. // Proc. Natl. Acad. Sci. USA. 2003. V. 100. № 19. P. 10718. https://doi.org/10.1073/pnas.1931005100
Hamada T., Miura Y., Komatsu Y. et al. // J. Phys. Chem. B. 2008. V. 112. № 47. P. 14678. https://doi.org/10.1021/jp807784j
Cheng H.T., Megha, London E. // J. Biol. Chem. 2009. V. 284. № 10. P. 6079. https://doi.org/10.1074/jbc.M806077200
Cheng H.T., London E. // Biophys. J. 2011. V. 100. № 11. P. 2671. https://doi.org/10.1016/j.bpj.2011.04.048
Chiantia S., London E. // Biophys. J. 2012. V. 103. № 11. P. 2311. https://doi.org/10.1016/j.bpj.2012.10.033
Elani Y., Purushothaman S., Booth P.J. et al. // Chem. Commun. (Camb.). 2015. V. 51. № 32. P. 6976. https://doi.org/10.1039/c5cc00712g
Mineev K.S., Nadezhdin K.D. // Nanotechnol. Rev. 2017. V. 6. № 1. P. 15. https://doi.org/10.1515/ntrev-2016-0074
Garavito R.M., Ferguson-Miller S. // J. Biol. Chem. 2001. V. 276. № 35. P. 32403. https://doi.org/10.1074/jbc.R100031200
Seddon A.M., Curnow P., Booth P.J. // Biochim. Biophys. Acta. 2004. V. 1666. № 1–2. P. 105. https://doi.org/10.1016/j.bbamem.2004.04.011
Tanford C., Reynolds J.A. // Biochim. Biophys. Acta. 1976. V. 457. № 2. P. 133. https://doi.org/10.1016/0304-4157(76)90009-5
MacKenzie K.R., Prestegard J.H., Engelman D.M. // Science. 1997. V. 276. № 5309. P. 131. https://doi.org/10.1126/science.276.5309.131
Pages G., Torres A.M., Ju P. et al. // Eur. Biophys. J. 2009. V. 39. № 1. P. 111. https://doi.org/10.1007/s00249-009-0433-1
Strandberg E., Ozdirekcan S., Rijkers D.T. et al. // Biophys. J. 2004. V. 86. № 6. P. 3709. https://doi.org/10.1529/biophysj.103.035402
Feigin L.A., Svergun D.I. Structure analysis by small-angle x-ray and neutron scattering. New York: Plenum Press, 1987. 335 p.
Shtykova E.V., Volkov V.V., Wang H.J. et al. // Langmuir. 2006. V. 22. № 19. P. 7994. https://doi.org/10.1021/la060879h
Jensen G.V., Lund R., Gummel J. et al. // J. Am. Chem. Soc. 2013. V. 135. № 19. P. 7214. https://doi.org/10.1021/ja312469n
Dwivedi D., Lepkova K. SAXS and SANS Techniques for Surfactant Characterization: Application in Corrosion Science. Application and Characterization of Surfactants. University of Tabriz: IntechOpen, 2017. 316 p.
Us’yarov O.G. // Colloid. J. 2016. V. 78. P. 698. https://doi.org/10.1134/S1061933X16050227
Razuvaeva E.V., Kulebyakina A.I., Streltsov D.R. et al. // Langmuir. 2018. V. 34. № 50. P. 15470. https://doi.org/10.1021/acs.langmuir.8b03379
Сыбачин А.В., Локова А.Ю., Спиридонов В.В. et al. // Высокомолекулярные соединения. Серия А. 2019. V. 61. № 3. P. 244. https://doi.org/10.1134/S2308112019030179
Zalygin A., Solovyeva D., Vaskan I. et al. // ChemistryOpen. 2020. V. 9. № 6. P. 641. https://doi.org/10.1002/open.201900276
Konarev P.V., Petoukhov M.V., Dadinova L.A. et al. // J. Appl. Cryst. 2020. V. 53. P. 236. https://doi.org/10.1107/S1600576719015656
Петухов М.В., Конарев П.В., Дадинова Л.А. et al. // Кристаллография. 2020. V. 65. № 2. P. 260. https://doi.org/10.31857/S0023476120020198
Kordyukova L.V., Konarev P.V., Fedorova N.V. et al. // Membranes (Basel). 2021. V. 11. № 10. P. 772. https://doi.org/10.3390/membranes11100772
Manalastas-Cantos K., Konarev P.V., Hajizadeh N.R. et al. // J. Appl. Cryst. 2021. V. 54. P. 343. https://doi.org/10.1107/S1600576720013412
Shilova L.A., Knyazev D.G., Fedorova N.V. et al. // Biochemistry (Moscow). Suppl. Ser. A. Membr. Cell Biol. 2017. V. 11. № 3. P. 225. https://doi.org/10.1134/S1990747817030072
Blanchet C.E., Spilotros A., Schwemmer F. et al. // J. Appl. Cryst. 2015. V. 48. № 2. P. 431. https://doi.org/10.1107/S160057671500254X
Konarev P.V., Volkov V.V., Sokolova A.V. et al. // J. Appl. Cryst. 2003. V. 36. P. 1277. https://doi.org/10.1107/S0021889803012779
Svergun D.I. // J. Appl. Cryst. 1992. V. 25. P. 495. https://doi.org/10.1107/S0021889892001663
Svergun D.I., Semenyuk A.V., Feigin L.A. // Acta Cryst. A. 1988. V. 44. P. 244. https://doi.org/10.1107/S0108767387011255
Svergun D.I., Barberato C., Koch M.H.J. // J. Appl. Cryst. 1995. V. 28. P. 768. https://doi.org/10.1107/S0021889895007047
Svergun D.I. // Biophys. J. 1999. V. 76. № 6. P. 2879. https://doi.org/10.1016/S0006-3495(99)77443-6
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография


