Лесоведение, 2022, № 5, стр. 530-539
Молекулярно-генетическая характеристика популяций сосны обыкновенной в Красноярском крае по панели 15 ядерных микросателлитных локусов
Д. Н. Шуваев a, *, А. А. Ибе b, Ю. Е. Щерба c, Т. В. Сухих b, Е. А. Шилкина b, М. А. Шеллер b, Е. А. Усова c, Е. В. Лисотова c, М. В. Репях c, О. М. Ступакова c
a Институт экологии растений и животных УрО РАН
620144 Екатеринбург, ул. 8 Марта, д. 202, Россия
b ФБУ “Рослесозащита” – “Центр защиты леса Красноярского края”
660036 Красноярск, Академгородок, д. 50 “а”, корп. 2, Россия
c Сибирский государственный университет науки и технологий им. академика М.Ф. Решетнева
660037 Красноярск, проспект им. газеты “Красноярский рабочий”, д. 31, Россия
* E-mail: denis.shuvaev@gmail.com
Поступила в редакцию 16.09.2021
После доработки 08.11.2021
Принята к публикации 02.02.2022
- EDN: NMPFCQ
- DOI: 10.31857/S002411482204009X
Аннотация
Для разработки метода ДНК-идентификации нелегально заготовленной древесины сосны обыкновенной (Pinus silvestris L.) отобрано 15 перспективных локусов ядерной микросателлитной ДНК. С данными локусами была исследована генетическая дифференциация в 10 выборках сосны обыкновенной, произрастающей на территории Красноярского края. Уровень межпопуляционной генетической дифференциации был низким (FST = 0.026). Корреляционная зависимость между генетическими и географическими дистанциями отсутствовала (r = 0.044, P > 0.05). Отсутствие кластеризации популяций свидетельствовало о недавних событиях резкого сокращения популяционной численности в прошлом, что могло быть вызвано рядом плейстоценовых оледенений. Отсутствие выраженной популяционно-генетической структуры не позволяет проводить точных оценок географического происхождения лесоматериалов сосны обыкновенной в исследуемой части ареала с помощью микросателлитных маркеров. Альтернативный подход к определению легальности заготовленной древесины подразумевает сравнение многолокусных профилей генотипов порубочных остатков и пней с сортиментом. На основе анализа частот генотипов 10 референтных выборок было установлено, что расчетная вероятность случайного совпадения профилей ДНК по 15 микросателлитным локусам составляет 4.2 × 10–11. Вероятность обнаружить идентичный генотип в выборке 1 млн деревьев составила менее 0.01%, что является приемлемой величиной в практике криминалистической экспертизы.
Незаконные рубки являются глобальной проблемой, которая ведет к серьезным экологическим последствиям, таким как обезлесение, снижение биоразнообразия и устойчивости экосистем. На незаконные рубки приходится от 10 до 35% всей лесозаготовки в РФ. В 2017 г. причиненный незаконными рубками ущерб превысил 11.5 млрд рублей, в 2018 г. данный показатель составил 11.6 млрд рублей. При этом за 2017 и 2018 годы общий объем незаконных рубок составил 2.8 млн куб. м (Косых, 2020). К хозяйственно ценным хвойным лесным породам, произрастающим на территории России, относятся сосна обыкновенная (Pinus sylvestris L.), виды лиственниц (Larix spp.), сосна сибирская (Pinus sibirica Du Tour), пихта сибирская (Abies sibirica L.) и ель сибирская (Picea obovata L.). На территории Красноярского края произрастает 14.2% от общероссийского запаса леса, значительную часть которого составляют насаждения сосны обыкновенной, играющей важную роль в экономике и экологической стабильности региона. Поэтому разработка мер противодействия незаконным рубкам является актуальной задачей.
Традиционный подход к экспертной оценке легальности древесины состоит в адаптации комплекса дендрохронологических методов применительно к целям криминалистического исследования. Избегая полемики относительно точности и границ применимости методов дендрохронологического анализа в рамках расследований фактов незаконных рубок (Синькевич, 2014; Майорова, Гончарук, 2015; Румянцев, 2017; Воронин и др., 2018), ‒ задача идентификации целого по частям представляется наименее дискуссионной и подкреплена хорошей методологической базой (Воронин и др., 2016). Тем не менее, точность экспертного заключения, основанного на дендрохронологическом подходе, в значительной степени зависит от места и условий произрастания дерева. В основе результирующего заключения дендрохронологической экспертизы лежат показатели коэффициентов корреляции, которые могут варьировать в достаточно широких пределах (0.83–0.98) (Пальчиков, Румянцев, 2009). Хотя высокий коэффициент корреляции (r = 0.98–0.99) свидетельствует о наличии сильной связи ‒ он отнюдь не устанавливает однозначного соответствия между частями дерева. Следовательно, дендрохронологический анализ позволяет построить лишь косвенную доказательную базу.
Методы генетического анализа в криминалистике уже длительное время с успехом применяют для идентификации личности подозреваемых в совершении преступлений. Аналогичным образом с помощью методов генетического анализа можно проводить высокоточную идентификацию целого по частям для сопоставления нелегально заготовленной древесины (хлысты, сортимент) и места рубки. Однако следует отметить, что идентификация географического происхождения древесины с помощью методов анализа ДНК имеет некоторые ограничения. Первое ограничение связано с получением достаточного количества интактной матрицы ДНК из древесины. Второе – наличие выраженной популяционно-генетической структуры у исследуемого вида. Например, если уровень межпопуляционной дифференциации вида достаточно высок (FST > 0.1), теоретически возможно провести анализ географического происхождения группы особей. В случае более низкого уровня межпопуляционной дифференциации проведение точной географической спецификации особей или их групп невозможно (Ogden, Linacre, 2015).
Популяционно-генетическая структура сосны обыкновенной была подробно исследована на большей части ареала с привлечением разнообразного и широкого спектра маркеров: морфологических, биохимических, аллозимных и генетических. Несмотря на выделение морфологами множества форм, рас и подвидов сосны обыкновенной, значимая межпопуляционная дифференциация была выявлена преимущественно в европейской части ареала (Санников и др., 2012), а также в Крыму и на Кавказе (Semerikov et al., 2020). В тоже время сибирская часть ареала сосны обыкновенной характеризуется крайне однородной структурой изменчивости и подразделяется на несколько больших региональных групп популяций: Западную Сибирь, Среднюю Сибирь, Восточную Сибирь и Приамурье. Однако между указанными популяционными группами уровень генетической дифференциации также остается низким (Санников и др., 2012). Возможной причиной сниженного генетического разнообразия и дифференциации популяций сосны из сибирской части ареала стали процессы послеледникового расселения вида из немногочисленных плейстоценовых рефугиумов (Semerikov et al., 2018). Таким образом, слабовыраженная генетическая структура популяций сосны обыкновенной в Сибири, по всей видимости, не может создать основу для идентификации географического происхождения лесоматериалов.
Альтернативным подходом к решению проблемы идентификации нелегальной древесины может стать метод анализа ДНК, основанный на сопоставлении многолокусных профилей генотипов. Суть метода заключается в попарном сравнительном анализе ДНК-профилей образцов изъятого круглого леса с образцами древесины (порубочные остатки, пни), изъятыми из незаконного места рубки. Вероятность случайного совпадения (PI) генотипических профилей образцов будет служить основой для экспертного заключения об их принадлежности одному дереву и может выступать основным доказательством в суде. Необходимость обладать информацией о предполагаемом незаконном месте рубки сильно ограничивает применимость подхода парных сравнений. Однако на сегодняшний день это единственный метод, способный предоставить высокую точность идентификации, которая недоступна другим методам, включая дендрохронологический анализ (Overview of current …, 2020).
Наиболее распространенными генетическими маркерами для индивидуального типирования являются маркеры, основанные на полиморфизме коротких повторов, – микросателлиты (SSR) (Tautz, 1993). Для сосны обыкновенной разработано и адаптировано большое количество локусов микросателлитной ДНК (Soranzo et al., 1998; Elsik et al., 2000; Liewlaksaneeyanawin et al., 2004; Sebastiani et al., 2012; Fang et al., 2014), что позволяет протестировать и отобрать хорошо воспроизводимые и изменчивые локусы для создания маркерной системы индивидуального типирования круглых лесоматериалов сосны обыкновенной.
Для надежной оценки мощности идентификационной панели микросателлитных маркеров необходимо создание представительной референтной базы генотипов по целевой древесной породе. Референтные популяции должны отражать данные о распределении аллельных частот генетических маркеров, поскольку от степени смещения частот аллелей зависит дискриминирующая сила метода идентификации. Поэтому референтную базу генотипов необходимо создавать с учетом популяционной структуры вида (Rowold, Herrera, 2005).
Ранее нами уже был проведен подбор начальной панели микросателлитных локусов (Шуваев и др., 2020а) с целью первичной оценки возможностей идентификационной мощности маркерной системы. Также в рамках отдельного исследования был найден наиболее эффективный протокол экстракции ДНК из древесины сосны обыкновенной (Шуваев и др., 2020б).
Целью настоящего исследования стала финальная корректировка идентификационной панели микросателлитных локусов на основе расширенной референтной базы выборок сосны обыкновенной.
ОБЪЕКТЫ И МЕТОДИКА
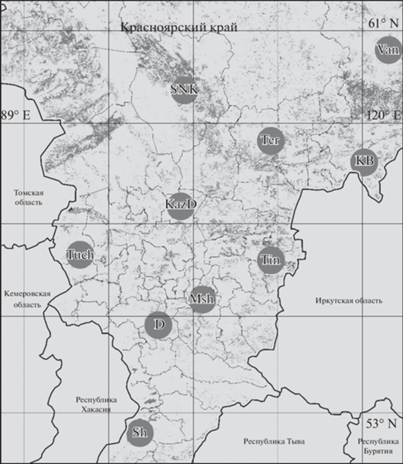
Объектом исследования послужили 10 выборок сосны обыкновенной, собранных в естественных популяциях Красноярского края. Объем каждой выборки составил 30 деревьев возрастом 60–80 лет. Всего было проанализировано 300 деревьев (рис. 1).
Суммарную ДНК экстрагировали из хвои по стандартному СТАВ-протоколу (Devey et al., 1996). С целью скрининга было дополнительно протестировано 18 пар праймеров для ядерных микросателлитных локусов сосны обыкновенной: lw_isotig20215, lw_isotig11166, lw_isotig12667, lw_isotig02138, lw_isotig02347, lw_isotig03088, lw_ isotig04931, lw_isotig06440, lw_isotig06215, lw_ isotig04204, lw_isotig04600, lw_isotig17679, lw_isotig00081, lw_isotig02842 (Fang et al., 2014), Lop3 (Liewlaksaneeyanawin et al., 2004), PtTx3016, PtTx2093, Ctg1376 (Ganea, Garcia Gil, 2011). Тестирование показало, что стабильная амплификация и достаточный уровень изменчивости были отмечены для двух новых локусов lw_isotig20215 и lw_isotig11166.
Таким образом, финальная идентификационная панель была доведена до 15 локусов ядерной микросателлитной ДНК: lw_isotig04195, lw_isotig04306, lw_isotig01420, lw_isotig07383, lw_isotig05123, lw_isotig20215, lw_isotig11166 (Fang et al., 2014), PtTx2146, PtTx3025, PtTx2123 (Elsik et al., 2000), PtTx4001, PtTx3107 (Ganea, Garcia Gil, 2011), Psyl17, Psyl42, Psyl57 (Sebastiani et al., 2012). Данный набор локусов был верифицирован на 10 популяционных выборках сосны обыкновенной для получения информации о распределении аллельных частот и выявления возможной популяционно-генетической структуры вида в сибирской части ареала.
Для постановки полимеразной цепной реакции (ПЦР) использовали коммерческий набор “GenPakPCRCore” (ООО “НПФ Генлаб”, Россия), согласно инструкции фирмы-производителя. ПЦР проводили на термоциклере T100 (Bio-Rad Laboratories, Inc., США). Протокол амплификации был общим для всех локусов: 95°С – 5 мин; (95°С – 30 с, 58°С – 90 с, 72°С – 45 с) × 32 цикла; 72°С – 7 мин; 4°С – ∞. Продукты амплификации локусов с 3-нуклеотидными повторами анализировали методом вертикального гель-электрофореза в 6%-м неденатурирующем полиакриламидном геле в TАE-буфере при 80 Вт в течение 3 ч в камерах для вертикального электрофореза VE-20 (Хеликон, Россия). Фрагменты динуклеотидных локусов: lw_isotig11166, lw_isotig20215, PtTx4001, Psyl17 и Psyl42 –разделяли в камерах для вертикального электрофореза MODEL S2 (Life Technologies, США) по методу, описанному в Elsik et al., 2000. В качестве стандартного маркера длин фрагментов использовали ДНК плазмиду pBR322, обработанную рестриктазой HPAII (СибЭнзим, Россия). Гели с 3-нуклеотидными повторами окрашивали бромистым этидием и визуализировали на трансиллюминаторе (Vilber Lourmat, Франция). Для гелей с динуклеотидными повторами использовали серебряное окрашивание. Генотипирование проводили при помощи программы Photo-Capt 12.4 (Vilber Lourmat, Франция). Для минимизации ошибок генотипирования электрофореграммы всех образцов были прогенотипированы независимо двумя специалистами, имеющими в качестве исходной информации о локусах только их аллельные размеры из первичных литературных источников. В случае несовпадения генотипических профилей образцов они были проанализированы заново, начиная с этапа экстракции ДНК.
Присутствие нуль-аллелей в локусах проверяли при помощи программы FreeNa (Chapuis, Estoup, 2007). Тесты на отклонение популяций от равновесия Харди-Вайнберга и неравновесие по сцеплению локусов были проведены в веб-интерфейсе программы Genepop, согласно настройкам по умолчанию (Raymond, Rousset, 1995; Rousset, 2008).
Оценки наблюдаемой и ожидаемой гетерозиготности (HO и HE), эффективного числа аллелей (Ne), межпопуляционной дифференциации (FST) по каждому локусу рассчитывали в программе GenAlEx 6.5 (Peakall, Smouse, 2006). Анализ кластеризации популяций методом главных координат (Kendall, Stuart, 1976) проводили на основе данных матрицы генетических дистанций DA (Nei et al., 1983). Степень зависимости между генетическими (DA) и географическими расстояниями оценивали при помощи коэффициента корреляции Пирсона. Значимость корреляции была определена тестом Мантела (Mantel, 1967) на основе 1000 пермутаций в программе GenAlEx 6.5. Иерархический анализ молекулярной изменчивости (AMOVA) был выполнен в программе Arlequin 3.5.2 (Excoffier, Lischer, 2010) при 10 000 пермутаций. С помощью байесовского подхода, реализованного в программе Structure 2.3.4 (Pritchard et al., 2000), была проведена кластеризация с последующей оценкой вклада кластерных генотипов в каждую исследуемую популяцию. Параметры симуляционного эксперимента включали модели Admixture и Locprior (Hubisz et al., 2009). Число исходов последовательных случайных событий MCMC (Monte Carlo Markov Chain) составило 300 тыс. при длине периода стабилизации (burn-in) 50 тыс. Всего было проведено 30 итераций для каждого запуска предполагаемого числа K кластеров от 1 до 10. Значение числа K кластеров оценивали путем вычисления апостериорной вероятности каждого кластера K (lnPr(X|K)) (Pritchard et al., 2000). Выравнивание вклада генотипов в каждый кластер по всем запускам с последующей визуализацией было проведено в веб-программе CLUMPACK (Kopelman et al., 2015). Мощность идентификационной панели оценивали с помощью показателей PI и Q, где PI ‒ вероятность случайного совпадения (Butler, 2005), Q – вероятность встречи генотипа, аналогичного ДНК-профилю исследуемого объекта среди N потенциальных индивидуумов (Перепечина, Гришечкин, 1996).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Присутствия нуль-аллелей в отобранных 15 локусах выявлено не было. Попарное тестирование локусов не выявило неравновесия по сцеплению (P > 0.05). Тест популяций на равновесие Харди-Вайнберга не обнаружил отклонений от равновесных частот генотипов ни в одной из исследуемых популяций (P > 0.05).
Суммарное число аллельных вариантов по 15 микросателлитным локусам составило 104. Примеры электрофореграмм нескольких локусов приведены на рис. 2. Наибольшее число аллелей было обнаружено у локуса lw_isotig20215 (NA = 14), а наименьшее – у локуса lw_isotig05123 (NA = 2) (табл. 1).
Рис. 2.
Электрофореграммы локусов lw_isotig20215, Psyl42, lw_isotig04306. M ‒ маркер молекулярного веса pBR322 после обработки рестриктазой HPAII.
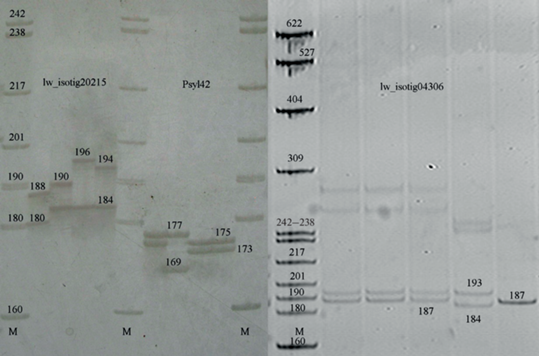
Таблица 1.
Усредненные показатели микросателлитных локусов по всем выборкам сосны обыкновенной
| № | Локус | HO ± S.E. | HE ± S.E. | NA | Ne± S.E. | FST | Диапазон аллелей |
|---|---|---|---|---|---|---|---|
| 1 | lw_isotig04195 | 0.313 ± 0.019 | 0.306 ± 0.015 | 4 | 1.448 ± 0.032 | 0.012 | 186–195 |
| 2 | lw_isotig04306 | 0.543 ± 0.009 | 0.570 ± 0.022 | 8 | 2.382 ± 0.123 | 0.017 | 172–193 |
| 3 | lw_isotig01420 | 0.630 ± 0.030 | 0.569 ± 0.018 | 5 | 2.357 ± 0.094 | 0.015 | 171–186 |
| 4 | lw_isotig07383 | 0.490 ± 0.041 | 0.543 ± 0.030 | 6 | 2.287 ± 0.168 | 0.049 | 188–203 |
| 5 | lw_isotig05123 | 0.383 ± 0.030 | 0.345 ± 0.018 | 2 | 1.535 ± 0.039 | 0.012 | 166–169 |
| 6 | lw_isotig20215 | 0.692 ± 0.049 | 0.754 ± 0.016 | 14 | 4.206 ± 0.261 | 0.022 | 178–204 |
| 7 | lw_isotig11166 | 0.869 ± 0.025 | 0.726 ± 0.012 | 8 | 3.705 ± 0.148 | 0.021 | 139–163 |
| 8 | PtTx3107 | 0.503 ± 0.027 | 0.659 ± 0.026 | 10 | 3.099 ± 0.253 | 0.032 | 153–183 |
| 9 | PtTx2146 | 0.733 ± 0.027 | 0.756 ± 0.008 | 10 | 4.130 ± 0.133 | 0.025 | 182–236 |
| 10 | PtTx3025 | 0.427 ± 0.026 | 0.408 ± 0.018 | 6 | 1.703 ± 0.052 | 0.017 | 272–314 |
| 11 | PtTx2123 | 0.287 ± 0.022 | 0.318 ± 0.018 | 3 | 1.477 ± 0.042 | 0.011 | 192–201 |
| 12 | PtTx4001 | 0.736 ± 0.033 | 0.647 ± 0.014 | 9 | 2.872 ± 0.111 | 0.053 | 201–225 |
| 13 | Psyl17 | 0.656 ± 0.042 | 0.661 ± 0.021 | 7 | 3.041 ± 0.169 | 0.049 | 221–249 |
| 14 | Psyl42 | 0.777 ± 0.032 | 0.713 ± 0.005 | 5 | 3.489 ± 0.061 | 0.033 | 169–177 |
| 15 | Psyl57 | 0.367 ± 0.040 | 0.378 ± 0.032 | 7 | 1.646 ± 0.082 | 0.025 | 187–205 |
Индивидуальный вклад локусов в межпопуляционные различия (FST) варьировал. Максимальное значение FST было обнаружено у локуса PtTx4001 (0.053), минимальное – у локуса PtTx2123 (0.011). Однако усредненное по всем локусам значение FST было низким и составило 0.026.
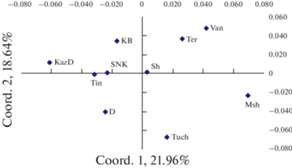
Анализ кластеризации выборок методом главных координат (рис. 3) и расчет коэффициента корреляционной зависимости между генетическими и географическими дистанциями продемонстрировали отсутствие взаимосвязи данных величин (r = 0.044, P > 0.05). Это свидетельствует об отсутствии генетико-географической спецификации популяций сосны обыкновенной на исследуемой территории.
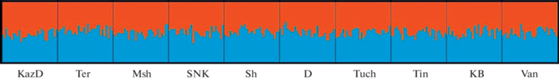
Кластеризация популяций по методу Structure также не выявила какой-либо структуры (рис. 4). Оценка числа K кластеров обнаружила лишь одну группу популяций, объединяющую все исследованные выборки сосны обыкновенной.
Иерархический анализ молекулярной изменчивости (AMOVA) показал, что большая часть изменчивости была сосредоточена внутри популяций (98.58%) и лишь небольшая доля приходилась на межпопуляционную (1.42%) компоненту изменчивости (табл. 2).
Таблица 2.
Результаты иерархического анализа молекулярной изменчивости (AMOVA) в 10 популяциях сосны обыкновенной
| Источник изменчивости | Степень свободы | Сумма квадратов | Компоненты дисперсии | Изменчивость, % | Индекс фиксации |
|---|---|---|---|---|---|
| Между 10 популяциями | 9 | 64.26 | 0.055 | 1.42 | FST = 0.014* |
| В пределах популяций | 590 | 2260.03 | 3.831 | 98.58 | |
| Сумма | 599 | 2324.29 | 3.886 | 100 | – |
Генетическая подразделенность популяций в пределах исследуемой части ареала оказалась низкой (FST = 2.6%). Исследуемая группа выборок сосны обыкновенной является, по всей видимости, частью большой популяции Средней Сибири. Высокая степень генетической однородности на достаточно обширном ареале является сильным свидетельством резкого сокращения популяционной численности в прошлом и последующего снижения генетического разнообразия (т.н. эффект бутылочного горлышка). Полученные результаты хорошо согласуются с исследованиями С.Н. Санникова с соавт. (Санников, Петрова, 2003) и В.Л. Семерикова с соавт. (Semerikov et al., 2018). Из работ которых следует, что основная волна расселения сосны обыкновенной двигалась в направлении востока Евразии, что сопровождалось резким снижением генетического разнообразия популяций переднего фронта волны расселения.
Таким образом, основываясь на полученных результатах кластеризации, следует сделать вывод, что возможность идентификации географического происхождения лесоматериалов сосны обыкновенной в Сибири крайне маловероятна при использовании генетического подхода.
Перспективным подходом к определению географического происхождения лесоматериалов могут стать методы, основанные на полногеномном анализе изменчивости SNP-локусов (Single Nucleotide Polymorphisms). Например, для сосны обыкновенной и ели европейской (Picea abies) разработано приблизительно 50 тыс. маркеров SNP с возможностью их анализа на микрочипе в автоматизированном режиме (Perry et al., 2020; Bernhardsson et al., 2021). Теоретически это позволяет построить геногеографическую карту с высокой степенью разрешения. Однако вопрос о дискриминирующей способности данного подхода в рамках точной идентификации географического происхождения лесоматериалов заслуживает отдельного исследования.
Альтернативным подходом к проблеме ДНК-идентификации легальности лесоматериалов является сравнительный анализ многолокусных генотипов. На основе полученных частот аллелей сосны обыкновенной был произведен расчет вероятности случайного совпадения (PI). Значение PI по 15 микросателлитным локусам составило 4.2 × 10–11, что дает вероятность обнаружения дерева с аналогичным генотипическим профилем приблизительно 1 на 25 млрд индивидуумов. Значение Q показывает вероятность встречи идентичного генотипа с поправкой на размер выборки (N) (Перепечина, Гришечкин, 1996). Размер выборки N = 106 деревьев соответствует значению Q в 0.0042%. Таким образом, вероятность обнаружения идентичного генотипа в выборке 1 млн деревьев составляет менее 0.01%. Данные показатели считаются достаточными для практики криминалистической экспертизы, что позволяет использовать разработанную панель микросателлитных локусов в расследовании фактов нелегального оборота древесины сосны обыкновенной.
ЗАКЛЮЧЕНИЕ
Популяционно-генетическая структура сосны обыкновенной не позволяет разработку надежных генетических методов идентификации географического происхождения деревьев или их групп в сибирской части ареала вида. Альтернативный подход к решению данной проблемы состоит в сравнении многолокусных профилей генотипов порубочных остатков и пней из незаконного места рубки с образцами сортимента сомнительного происхождения.
Итогом работ в данном направлении стала панель из 15 ядерных микросателлитных локусов для проведения сравнительного анализа генотипических профилей образцов сосны обыкновенной. Все локусы характеризовались высокой степенью воспроизводимости и достаточным уровнем изменчивости. На основе распределения аллельных частот локусов в референтных выборках были рассчитаны итоговые оценки мощности идентификационной панели, которые являлись достаточными для криминалистической экспертизы и давали вероятность ошибочного суждения менее 0.01%. Разработанную панель микросателлитных локусов можно рекомендовать для использования отделами мониторинга состояния лесных генетических ресурсов при ФБУ “Рослесозащита” в целях содействия органам МВД при расследовании фактов нелегальных рубок насаждений сосны обыкновенной. Кроме того, она может быть использована отделами ФБУ “Рослесозащита” в качестве стандартного набора локусов для целей паспортизации на объектах единого генетико-селекционного комплекса.
Список литературы
Воронин В.И., Грибунов О.П., Жаворонков Ю.М., Осколков В.А., Унжакова С.В. Судебно-ботаническая экспертиза с применением методов дендрохронологии при расследовании незаконной рубки лесных насаждений. Иркутск: Восточно-Сибирский институт МВД РФ, 2016. 200 с.
Воронин В.И., Унжакова С.В., Зырянов В.С. Судебная дендрохронология в криминалистике // Криминалистика: вчера, сегодня, завтра. 2018. Т. 4. № 8. С. 34‒40.
Косых В.А. Незаконный оборот леса и лесоматериалов в Российской Федерации: сущность, детерминация, проблемы противодействия: дис. … канд. юрид. наук: 12.00.08. Санкт-Петербург: ФГКОУ ВО СПбУ МВД России, 2020. 252 с.
Майорова Е.И., Гончарук Н.Ю. К вопросу использования дендрохронологического анализа в судебно-экспертной практике // Вестник МГУЛ ‒ Лесной вестник. 2015. Т. 19. № 5. С. 153‒158.
Пальчиков С.Б., Румянцев Д.Е. Контроль за законностью заготовки древесины на основе древесно-кольцевой информации // Устойчивое лесопользование. 2009. Т. 21. № 2. С. 12‒16.
Перепечина И.О., Гришечкин С.А. Вероятностные расчеты в ДНК-дактилоскопии. Методические рекомендации. М.: ЭКЦ МВД России, 1996. 16 с.
Румянцев Д.Е. Дендрохронологические экспертизы в лесном хозяйстве // Инноватика и экспертиза: научные труды. 2017. Т. 20. № 2. С. 111‒114.
Санников С.Н., Петрова И.В. Дифференциация популяций сосны обыкновенной. Екатеринбург: УрО РАН, 2003. 174 с.
Санников С.Н., Санникова Н.С., Петрова И.В. Очерки по теории лесной популяционной биологии. Екатеринбург: РИО УрО РАН, 2012. 248 с.
Синькевич С.М. Дендрохронология в судебной экспертизе: ограничения и перспективы // Вестник МГУЛ ‒ Лесной вестник. 2014. Т. 18. № 5. С. 166‒170.
Шуваев Д.Н., Ибе А.А., Щерба Ю.Е., Сухих Т.В., Шилкина Е.А., Усова Е.А., Лисотова Е.В., Репях М.В., Ступакова О.М. Разработка панели ядерных микросателлитных локусов для оценки легальности происхождения древесины сосны обыкновенной в Красноярском крае // Хвойные бореальной зоны. 2020. Т. 38. № 5–6. С. 298–305.
Шуваев Д.Н., Ибе А.А., Щерба Ю.Е., Сухих Т.В., Шилкина Е.А., Усова Е.А., Лисотова Е.В., Репях М.В., Ступакова О.М. Оценка эффективности протоколов экстракции ДНК из древесины сосны обыкновенной // Хвойные бореальной зоны. 2020. Т. 38. № 5–6. С. 291–297.
Bernhardsson C., Zan Y., Chen Z., Ingvarsson P.K., Wu H.X. Development of a highly efficient 50K single nucleotide polymorphism genotyping array for the large and complex genome of Norway spruce (Picea abies L. Karst) by whole genome resequencing and its transferability to other spruce species // Molecular Ecology Resources. 2021. № 21. P. 880‒896.
Butler J.M. Forensic DNA Typing: Biology, Technology and Genetics of STR Markers. 2nd Ed. Oxford: Elsevier, 2005. 647 p.
Chapuis M-P., Estoup A. Microsatellite Null Alleles and Estimation of Population Differentiation // Molecular Biology and Evolution. 2007. V. 24. № 3. P. 621‒631.
Devey M.E., Bell J.C., Smith D.N., Neale D.B., Moran G.F. A genetic linkage map for Pinus radiate based on RFLP, RAPD, and microsatellite markers // Theoretical and Applied Genetics. 1996. V. 92. № 6. P. 673–679.
Elsik C.G., Minihan V.T., Hall S.E., Scarpa A.M., Williams C.G. Low-copy microsatellite markers for Pinus taeda L. // Genome. 2000. V. 43. P. 550–555.
Excoffier L., Lischer H.E.L. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows // Molecular Ecology Resources. 2010. V. 10. № 3. P. 564–567.
Fang P., Niu S., Yuan H., Li Z., Zhang Y., Yuan L., Li W. Development and characterization of 25 EST-SSR markers in Pinus sylvestris var. mongolica (Pinaceae) // Applications in Plant Sciences. 2014. V. 2. № 1. P. 1–4.
Ganea S.L., Garcia Gil M.R. Multiplex Nuclear SSR Amplification in Scots Pine (Pinus sylvestris L.) // Bulletin UASVM Horticulture. 2011. V. 68. № 1. P. 47‒53.
Hubisz M.J., Falush D., Stephens M., Pritchard J.K. Inferring weak population structure with the assistance of sample group information // Molecular Ecology Resources. 2009. V. 9. № 5. P. 1322–1332.
Kendall M.G., Stuart A. The Advanced Theory of Statistics. Volume 3: Design and Analysis, and Time-Series. London: Charles Griffin & Co. Ltd., 1976. 585 p.
Kopelman N.M., Mayzel J., Jakobsson M., Rosenberg N.A., Mayrose I. Clumpak: a program for identifying clustering modes and packaging population structure inferences across K // Molecular Ecology Resources. 2015. V. 15. № 5. P. 1179–1191.
Liewlaksaneeyanawin C., Ritland C.E., El-Kassaby Y.A., Ritland K. Single-copy, species-transferable microsatellite markers developed from loblolly pine ESTs // Theoretical and Applied Genetics. 2004. V. 109. P. 361‒369.
Mantel N. The detection of disease clustering and a generalized regression approach // Cancer Research. 1967. V. 27. P. 209–220.
Nei M., Tajima F., Tateno Y. Accuracy of estimated phylogenetic trees from molecular data. II. Gene frequency data // J. Molecular Evolution. 1983. V. 19. № 94. P. 153–170.
Ogden R., Linacre A. Wildlife forensic science: A review of genetic geographic origin assignment // Forensic Science International: Genetics. 2015. V. 18. P. 152–159.
Overview of current practices in data analysis for wood identification: A guide for the different timber tracking methods / By ed. Schmitz N. GTTN, Global Timber Tracking Network: GTTN Secretariat: European Forest Institute and Thunen Institute, 2020. 143 p.
Peakall R., Smouse P.E. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research // Molecular Ecology Notes. 2006. V. 6. № 1. P. 288–295.
Perry A., Wachowiak W., Downing A., Talbot R., Cavers S. Development of a single nucleotide polymorphism array for population genomic studies in four European pine species // Molecular Ecology Resources. 2020. V. 20. № 6. P. 1697‒1705.
Pritchard J.K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data // Genetics. 2000. V. 155. № 2. P. 945–959.
Raymond M., Rousset F. GENEPOP (version 1.2): population genetics software for exact tests and ecumenicism // Journal of Heredity. 1995. V. 86. № 3. P. 248–249.
Rousset F. Genepop'007: a complete reimplementation of the Genepop software for Windows and Linux // Molecular Ecology Resources. 2008. V. 8. № 1. P. 103–106.
Rowold D.J., Herrera R.J. On human STR sub-population structure // Forensic Science International. 2005. V. 151. № 1. P. 59–69.
Sebastiani F., Pinzauti F., Kujala S.T., Gonzalez-Martinez S.C., Vendramin G.G. Novel polymorphic nuclear microsatellite markers for Pinus sylvestris L. // Conservation Genetics Resources. 2012. V. 4. № 2. P. 231–234.
Semerikov V.L., Semerikova S.A., Putintseva Y.A., Tarakanov V.V., Tikhonova I.V., Vidyakin A.I., Oreshkova N.V., Krutovsky K.V. Colonization history of Scots pine in Eastern Europe and North Asia based on mitochondrial DNA variation // Tree Genetics & Genomes. 2018. V. 14. № 8. P. 1–7.
Semerikov N.V., Petrova I.V., Sannikov S.N., Semerikova S.A., Tashev A.N., Lascoux M., Semerikov V.L. Cytoplasmic DNA variation does not support a recent contribution of Pinus sylvestris L. from the Caucasus to the main range // Tree Genetics & Genomes. 2020. V. 16. № 59. P. 1–11.
Soranzo N., Provan J., Powell W. Characterization of microsatellite loci in Pinus sylvestris L. // Molecular Ecology. 1998. V. 7. № 9. P. 1247–1263.
Tautz D. Notes on the definition and nomenclature of tandemly repetitive DNA sequences // DNA Fingerprinting: State of the Science. 1993. V. 34. № 2. P. 21–28.
Дополнительные материалы отсутствуют.