Микробиология, 2019, T. 88, № 4, стр. 438-449
Разнообразие bphA1-генов в микробном сообществе техногеннозагрязненной почвы и выделение новых бактерий рода Pseudomonas ‒ деструкторов бифенила/хлорбифенилов
А. О. Воронина a, Д. О. Егорова a, Е. С. Корсакова a, Е. Г. Плотникова a, *
a Институт экологии и генетики микроорганизмов Уральского отделения
Российской академии наук ‒ филиал ПФИЦ УрО РАН
614990 Пермь, Россия
* E-mail: peg@iegm.ru
Поступила в редакцию 13.11.2018
После доработки 19.12.2018
Принята к публикации 01.02.2019
Аннотация
Применение молекулярно-биологических и культуральных методов исследования микробного сообщества техногенной почвы, загрязненной трудноразлагаемыми токсичными (хлор)ароматическими соединениями, позволило оценить разнообразие ключевых генов (bphA1) разложения бифенила/полихлорированных бифенилов (ПХБ) и выделить новые бактерии-деструкторы бифенила/ПХБ. В результате клонирования ПЦР-продукта, полученного на матрице ДНК, выделенной из почвы, при использовании праймеров к генам α-субъединиц бифенил 2,3-диоксигеназ (bphA1) было выявлено два типа генов ароматических диоксигеназ (ДО), имеющих наибольшее сходство на уровне 97.8‒99.5% с генами, кодирующими кластер Риски α-субъединиц ДО, bphA1 некультивируемых бактерий. Из накопительной культуры, полученной при инкубировании образца почвы на бифениле, было выделено два деструктора бифенила рода Pseudomonas (штаммы VRP2-6 и VRP2-2), имеющих наибольшее сходство по генам 16S рРНК с типовыми штаммами видов P. taiwanensis (99%) и P. alcaligenes (100%) соответственно. Анализ нуклеотидных последовательностей bphA1 штаммов VRP2-6 и VRP2-2 показал сходство не выше 97.3% с bphA1-генами известных деструкторов бифенила/ПХБ рода Pseudomonas. Изолят VRP2-6 эффективно утилизировал орто- и пара-монохлорированные бифенилы и осуществлял деструкцию дихлорированного бифенила (2,4'-диХБ), окисляя как орто-, так и пара-хлорированные кольца молекулы бифенила. Новые штаммы псевдомонад могут представлять интерес при разработке биотехнологий, направленных на мониторинг и восстановление загрязненных бифенилом/ПХБ почв.
Проблема загрязнения окружающей среды трудноразлагаемыми токсичными органическими соединениями, поступающими в окружающую среду в результате работы промышленных предприятий, по-прежнему остается актуальной и привлекает все большее внимание исследователей, занимающихся проблемами экологии. К таким соединениям относятся бифенил и его хлорированные производные – полихлорированные бифенилы (ПХБ), которые Программой ООН по окружающей среде (ЮНЕП) отнесены к группе стойких органических загрязнителей (СОЗ) (http:// chm.pops.int). Бифенил и ПХБ негативно влияют на иммунную, нервную, репродуктивную и эндокринную системы человека, а продолжительное воздействие на организм человека этих токсикантов может вызвать серьезные заболевания, в том числе онкологические (Sharma et al., 2018). Бифенил, являясь компонентом нефти, каменного угля и природного газа, вызывает масштабное загрязнение окружающей среды, широко используется в химической промышленности для синтеза многих органических соединений (Nam et al., 2014). ПХБ, благодаря их исключительной устойчивости к физическим и химическим воздействиям, в XX веке широко использовали в различных отраслях промышленности. Наибольшее применение они получили в качестве компонентов, входящих в диэлектрические (в трансформаторах, конденсаторах), смазочно-охлаждающие жидкости (СОЖ), в лакокрасочные изделия, изоляционные материалы (Pieper, Seeger, 2008; Васильев, 2017). Несмотря на запрет промышленного выпуска и использования ПХБ (Стокгольмская конвенция, 2001 г.), до сих пор остается актуальной проблема их утилизации и восстановления почв, водоемов, загрязненных ПХБ (Васильева, Стрижакова, 2007; Sharma et al., 2018). Бифенил, ввиду меньшей токсичности и большей биодоступности, по сравнению с его хлорпроизводными, используется в качестве модельного соединения при изучении биологических процессов разложения ПХБ (Шумкова и соавт., 2015).
В последние годы все более интенсивно разрабатываются и внедряются новые технологии для детоксикации и восстановления загрязненных территорий с использованием биологического потенциала микроорганизмов (Sharma et al., 2018). Известны и хорошо охарактеризованы грамположительные и грамотрицательные бактерии, в том числе представители рода Pseudomonas, способные к утилизации или частичной трансформации бифенила и ПХБ (Master, Mohn, 2001; Furukawa et al., 2004; Adebusoye et al., 2007; Nam et al., 2014; Chakraborty, Das, 2016; Suenaga et al., 2017). Однако поиск и изучение активных бактерий-деструкторов, перспективных для использования в биотехнологиях, направленных на восстановлении загрязненных бифенилом/ПХБ территорий, остается актуальным.
Деструкция бифенила/ПХБ бактериями осуществляется до пентадиеновой и (хлор)бензойной кислот в четыре этапа. Первый этап – включение двух гидроксильных групп в ароматическое кольцо бифенила – происходит под действием фермента бифенил 2,3-диоксигеназы (БДО) с получением бифенил 2,3-дигидродиола, который на втором этапе окисляется бифенил 2,3-дигидродиол 2,3-дегидрогеназой (BphB) с образованием 2,3-дигидроксибифенила. На третьем этапе 2,3-дигидроксибифенил 1,2-диоксигеназа (BphC) осуществляет мета-расщепление ароматического кольца, в результате чего образуется 2-гидрокси-6-оксо-6-фенилгекса-2,4-диеновая кислота (ГОФДК). На последнем этапе конверсию ГОФДК до пентадиеновой и бензойной кислот катализирует 2-гидрокси-6-оксо-6-фенилгекса-2,4-диеноатгидролаза (BphD) (Pieper, Seeger, 2008). Ключевым ферментом разложения бифенила/ПХБ является бифенил 2,3-диоксигеназа (БДО) – мультикомпонентный фермент, включающий белки электрон-транспортной системы и терминальной оксигеназы, представляющей собой гексамер из трех α- и трех β-субъединиц (соответственно, BphA1 и BphA2), ферредоксина (BphA3) и ферредоксин редуктазы (BphA3). Считается, что именно α-субъединица играет ключевую роль в распознавании и связывании субстрата (Furukawa et al., 2004). Таким образом, ген bphA1 (кодирующий α‑субъединицу бифенил 2,3-диоксигеназы) является важным маркером при исследовании биодеградационного потенциала бактерий (Шумкова и соавт., 2015).
В рамках настоящей работы проводилось изучение микробного сообщества почвы с территории предприятия ОАО “Пермский завод смазок и СОЖ”, длительное время загрязняющейся (хлор)ароматическими соединениями (в том числе ПХБ). На предприятии более 60 лет производились смазочные материалы, смазочно-охлаждающие жидкости, пластичные смазки и технические жидкости (http://www.nge.ru/passport-description-161.htm).
Цель работы – определить наличие и оценить разнообразие ключевых генов деструкции бифенила (bphA-генов) в микробном сообществе почвы, отобранной на территории производства ОАО “Пермский завод смазок и СОЖ”, и выделить активные бактерии-деструкторы бифенила/ПХБ из исследуемой загрязненной почвы.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Образцы для исследований. В качестве материала для исследований с территории завода ОАО “Пермский завод смазок и СОЖ” (г. Пермь) в 2016 г. было отобрано 4 образца почвы. Образцы были взяты с глубины 5‒10 см из разных мест на территории завода. Для микробиологических и молекулярно-генетических исследований использовали суммарную почву, полученную при тщательном перемешивании отобранных образцов.
Выделение ДНК, ПЦР-амплификация и клонирование bphA1-генов. Выделение тотальной ДНК из образца почвы проводили с использованием коммерческого набора реактивов “MP Biomedicals” (США). Концентрацию ДНК определяли на приборе QubitTM Fluorometer, (“Invitrogen”, США) при применении реактивов производителя.
С матрицы тотальной ДНК проводили амплификацию bphA1-генов с праймерами BPHD-f3 и BPHD-r1 (табл. 1), специфичными к гену α-субъединицы бифенил-2,3-диоксигеназы, на приборе MyCycler (“Bio-Rad Laboratories”, США) согласно протоколу (Iwai et al., 2010). Продукты реакции разделяли методом электрофореза в 1%-ом агарозном геле при напряжении 10 В/см, окрашивали раствором бромистого этидия (5 мкг/мл) и фотографировали в УФ-свете с использованием системы гель-документирования Gel DocTM XR (“Bio-Rad Laboratories”, США).
Таблица 1.
Олигонуклеотидные праймеры
| Амплифицируемые фрагменты ДНК | Праймеры | Нуклеотидная последовательность, 5'-3' |
Ссылка |
|---|---|---|---|
| Ген 16S рРНК | Eub338 Eub518 |
ACTCCTACGGGAGGCAGCAG ATTACCGCGGCTGCTGG |
Fierer et al., 2005 |
| Ген 16S рРНК | 27F 1492R |
AGAGTTTGATC(A/C)TGGCTCAG ACGG(C/T)TACCTTGTTACGACTT |
Tiirola et al., 2002 |
| Гены, кодирующие α-субъединицу ГДО (Г+)* |
PAH-RHDαGPF PAH-RHDαGPR |
CGGCGCCGACAAYTTYGTNGG GGGGAACACGGTGCCRTGDATRAA |
Cébron et al., 2008 |
| Гены, кодирующие α-субъединицу ГДО (Г–)** |
PAH-RHDαGNF PAH-RHDαGNR |
GAGATGCATACCACGTKGGTTGGA AGCTGTTGTTCGGGAAGAYWGTGCMGTT |
Cébron et al., 2008 |
| Ген bphA1, кодирующий α-субъединицу БДО*** |
BPHD-f3 BPHD-r1 |
AACTGGAARTTYGCIGCVGA ACCCAGTTYTCICCRTCGTC |
Iwai et al., 2010 |
Полученные ПЦР-фрагменты bphA1-генов клонировали в клетках Е. сoli JM109 в составе вектора pTZ57R/T (“Thermo Scientific”, США). Последующий отбор рекомбинантных клонов осуществляли на основании “бело-голубого” теста, высевая трансформанты на среду Luria-Bertrani (LB) (Short Protocols in Molecular Biology, 1995), содержащую 50 мкг/мл ампициллина, 100 мМ IPTG, 20 мкг/мл X-gal (“Fermentas”, Литва). С ДНК-матрицы отобранных рекомбинантных клонов проводили амплификацию фрагментов bphA1-генов с праймерами BPHD-f3 и BPHD-r1 (Iwai et al., 2010). Анализ полиморфизма длин рестрикционных фрагментов bphA1-генов (ПДРФ-анализ), осуществляли с использованием эндонуклеазы рестрикции HhaI (“Fermentas”, Литва). Для подготовки к секвенированию клонированных в составе вектора pTZ57R/T фрагментов ДНК проводили амплификацию вставки с использованием стандартных праймеров M13 (М13F 5'-GTTTTCCCAGTCACGAC-3' и М13R 5'-CAGGAAACAGCTATGAC-3'), для которых имеются сайты связывания в векторе pTZ57R по обеим сторонам от полилинкера. Описание секвенирования и анализ клонированных последовательностей ДНК приведены ниже.
ПЦР-РВ анализ. Для характеристики микробного сообщества (МС) исследуемой почвы был использован метод полимеразной цепной реакции в режиме реального времени (ПЦР-РВ). Для оценки таксономического состава МС с матрицы тотальной ДНК почвы были амплифицированы гены 16S рРНК при использовании праймеров Eub338 (табл. 1) (Fierer et al., 2005). Для детекции генов, кодирующих α-субъединицу гидроксилирующих диоксигеназ (PAH-RHDα), участвующих в окислении различных полициклических ароматических углеводородов (ПАУ), бифенила/ПХБ, были использованы пары праймеров (табл. 1), разработанные для генов гидроксилирующих диоксигеназ грамположительных и грамотрицательных бактерий-деструкторов ПАУ, бифенила/ПХБ (Cébron et al., 2008). ПЦР-РВ выполняли в присутствии красителя Sybr Green I в наборах реактивов производства фирмы “Синтол” (Россия) и 2X Maxima SYBR Green/ROX qPCR Master Mix (“Thermo Scientific”, США) на приборе “CFX96 Touch™ Real-Time PCR Detection Systems” (“Bio-Rad Laboratories”, США) согласно стандартной методике (Jurelevicius et al., 2012).
Выделение штаммов-деструкторов бифенила/ПХБ осуществляли методом накопительного культивирования. В колбы Эрленмейера объемом 250 мл добавляли 50 мл минеральной среды К1 (Maltseva et al., 1999), образец почвы весом 0.5 г и бифенил (1 г/л) в качестве единственного источника углерода и энергии. Культивирование проводили в течение 1 мес. при температуре 28°С с аэрацией на термошейкере (120 об./мин). Полученную суспензию высевали на агаризованную среду К1, бифенил добавляли на крышку перевернутой чашки Петри и культивировали при температуре 28°С до появления колоний. Чистоту культур проверяли при их выращивании на богатой среде LB.
Морфо-физиологические признаки выделенных микроорганизмов изучали по общепринятым методикам (Методы общей бактериологии, 1983). Рост бактерий при изменении осмолярности среды изучали как на агаризованной минеральной среде Раймонда (Розанова, Назина, 1982) с добавлением бифенила в качестве единственного источника углерода и энергии, так и на богатой среде Раймонда (в состав среды входит дрожжевой экстракт ‒ 2.5 г/л, триптон ‒ 5 г/л), при концентрациях NaCl (г/л): 0, 30, 50, 70. Культивирование осуществляли в термостате при 28°С в течение 7 сут. Оценку роста бактериальных штаммов при различных температурных условиях проводили на агаризованных средах: минеральной К1 с бифенилом и богатой среде LB, при температурах: 4, 10, 28, 37 и 45°С. Рост колоний учитывали на 7 сут.
Способность бактерий разлагать ароматические соединения оценивали путем культивирования в жидкой среде K1 на термошейкере (120 об./мин) при 28°С и/или на агаризованной среде К1 на чашках Петри в термостате при 28°С. В качестве источника углерода и энергии использовали нафталин и фенантрен в виде мелкодисперсного порошка в концентрации 0.5 г/л, а также орто-фталевую, салициловую, пара-гидроксибензойную, бензойную, протокатеховую кислоты в виде водных растворов натриевых солей (в пересчете на кислоту ‒ 0.5 г/л). Толуол и фенол помещали на крышку перевернутой чашки Петри и культивировали бактерии в их парах. В ростовых экспериментах бифенил был использован в концентрации 0.5 и 1.0 г/л. Бифенил вносили в колбы в виде навески мелкодисперсного порошка с учетом объема среды культивирования. Рост штаммов на вышеперечисленных субстратах в жидкой среде оценивали путем измерения оптической плотности (ОП) культуры на спектрофотометре UV-Visible BioSpec-mini (“Shimadzu”, Япония) при λmax 600 нм. Удельную скорость роста штаммов рассчитывали по формуле: μ = (ln Nt ‒ ln N0)/t ‒ t0, где μ – удельная скорость роста, ч–1; ln Nt – натуральный логарифм значения ОП культуры при достижении стационарной фазы роста; lnN0 ‒ натуральный логарифм значения ОП культуры в начале логарифмической фазы роста; t – время культивирования при достижении стационарной фазы роста; t0 – время культивирования к моменту начала логарифмической фазы роста.
Генетическое типирование и идентификация бактерий-деструкторов. ДНК из чистых культур бактерий выделяли общепринятым методом (Short Protocols in Molecular Biology, 1995). Генетическое сходство/различие выделенных штаммов определяли методом ВОХ-ПЦР по стандартной методике (Versalovic et al., 1994).
Идентификацию бактерий осуществляли при амплификации гена 16S рРНК с использованием стандартных бактериальных праймеров 27F и 1492R (табл. 1), определении и анализе нуклеотидных последовательностей амплифицированных генов 16S рРНК (описание см. ниже). Поиск гомологичных последовательностей проводили в базе данных ezTaxon (http://www.ezbiocloud.net/ eztaxon).
Исследование ключевых генов деструкции бифенила/ПХБ (bphA1-генов) у выделенных штаммов осуществляли путем амплификации с праймерами BPHD-f3 и BPHD-r1 (табл. 1) (Iwai et al., 2010), с последующим секвенированием и анализом полученных нуклеотидных последовательностей bphA1-генов. Фрагменты bphA1-генов штаммов VRP2-2 и VRP2-6 зарегистрированы в базе данных GenBank под номерами KY978889.1 и KY978890.1 соответственно.
Секвенирование и анализ генов 16S рРНК и bphA1-генов. Определение нуклеотидных последовательностей проводили с применением набора реактивов Big Dye Terminator Cycle Sequencing Kit v. 3.1 (“Applied Biosystems”, США) на автоматическом секвенаторе Genetic Analyser 3500XL (“Applied Biosystems”, США) согласно рекомендациям производителя. Анализ нуклеотидных последовательностей осуществляли с использованием программ Sequence Scanner v. 2.0 и MEGA 6.0 (http://www.megasoftware.net). Поиск гомологичных последовательностей проводили в международной базе данных GenBank (http:/www.ncbi.nlm.nih.gov/ Blast.cgi). Для построения дерева сходства использовали алгоритм UPGMA (MEGA 6.0). Оценку статистической достоверности ветвления (“bootstrap-анализ”) проводили на основе 1000 альтернативных деревьев.
Плазмидная ДНК. Наличие плазмидной ДНК выявляли методом пульс-электрофореза с использованием прибора CHEF DR II (“Bio-Rad Laboratories”, США). Бактерии выращивали на минеральной среде K1 (10 мл) с добавлением бифенила (1 г/л) до ОП600 =1.0. Дальнейшую подготовку клеток, электрофорез и обработку полученных данных проводили, как описано ранее (Егорова и соавт., 2013).
Деструкция моно- и дихлорбифенилов. При исследовании деструкции 2-ХБ, 4‑ХБ и 2,4'-диХБ 1 мл отмытых дважды в среде К1 клеток (ОП600 = 2.0), выращенных на бифениле (1 г/л), переносили во флаконы объемом 5 мл с тефлоновыми крышками, добавляя субстраты до конечной концентрации, составляющей для моно-ХБ 250 мг/л, для 2,4'-диХБ – 44.6 мг/л, и инкубировали на шейкере (200 об./мин) при 28°С. Накопление хлорбензойных кислот в среде культивирования регистрировали методом ВЭЖХ при использовании хроматографа LC-20AD Prominance (“Shimadzu”, Япония) с колонкой (С-18 150 × 4.6 мм; “Sigma-Aldrich”, США) и УФ-детектором SPD-20A (при 205 нм) в системе ацетонитрил–0.1%-ная Н3РО4 (70 : 30). Идентификацию продуктов метаболизма проводили при сравнении времени удержания на колонке образовавшихся и стандартных соединений (2-ХБК, 4-ХБК) (Maltseva et al., 1999). Количество образовавшихся продуктов оценивали по величине площади и высоты пиков на хроматограмме относительно данных величин стандартных соединений. Образование других продуктов трансформации ХБ, 2-гидроксо-6-оксо-(хлорфенил)гекса-2,4-диеновых кислот (ГОФДК), определяли на спектрофотометре UV-Visible BioSpec-mini (‘Shimadzu”, Япония) при λmax от 390 до 440 нм (Maltseva et al., 1999). Для определения количества 2-ХБ, 4‑ХБ и 2,4'-ХБ весь реакционный объем экстрагировали смесью: конц. Н2SO4–12.5%-ный додецилсульфат натрия–гексан (1 : 10 : 25), в течение 60 мин при 30°С, скорость перемешивания 200 об./мин. Полученные экстракты обезвоживали Na2SO4 и анализировали методом ГХ‑МС на газовом хроматографе GC6890N (“Agilent Technology”, США) с масс-селективным детектором MSD5973N (“Agilent Technology”, США) согласно “РД 52.18.578-97. Методические указания…” (http://docs.cntd.ru/document/1200036915). Концентрацию хлорбифенилов рассчитывали согласно калибровочным графикам.
Статистика. Повторность опытов трехкратная. Полученные данные обрабатывали с использованием стандартных пакетов компьютерной программы Microsoft Excel. Все приведенные числовые данные проверены на достоверность при статистической обработке.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Молекулярно-генетическая характеристика микробного сообщества загрязненной почвы. С использованием метода количественного анализа (ПЦР-РВ) тотальной ДНК, выделенной из почвы, отобранной на территории завода ОАО “Пермский завод смазок и СОЖ”, было выявлено наличие бактериальных генов 16S рРНК в количестве 1.05 × 1011 (±1.59 × 108) копий гена на 1 г почвы. Методом ПЦР-РВ было обнаружено значительное количество копий PAH-RHDα-генов диоксигеназ (2.41 × 108 (±3.57 × 107) на 1 г почвы), участвующих в окислении ПАУ, бифенила грамотрицательными бактериями. В то же время, при использовании праймеров PAH-RHDαGPF и PAH-RHDαGPR (табл. 1) не было зафиксировано присутствие генов гидроксилирующих диоксигеназ грамположительных бактерий. Доля бактерий, способных к деградации ПАУ, бифенил/ПХБ (представленная соотношением количества копий гена PAH-RHDα к количеству копий гена 16S рРНК), в исследуемом микробном сообществе довольно высока и составляет около 10–3 в пересчете на 1 г почвы. Таким образом, в микробном сообществе исследуемой загрязненной почвы были обнаружены бактериальные гены, контролирующие начальные этапы деструкции полиароматических соединений (ПАУ, бифенила/ПХБ).
Для более точного определения наличия бактерий-деструкторов бифенила/ПХБ и исследования разнообразия генетических систем разложения этих соединений в исследуемом МС нами были использованы олигонуклеотидные праймеры, специфичные к гену α-субъединицы бифенил 2,3-диоксигеназы (табл. 1) (Iwai et al., 2010). С матрицы тотальной ДНК почвы был получен ПЦР-продукт искомого размера (около 500 п.н.), который был клонирован в клетках E. coli. В результате клонирования создана библиотека из 72 рекомбинантных клонов, содержащих bphA1-гены. Для выявления сходства и различий между клонированными фрагментами гена bphA1 был проведен ПДРФ-анализ с использованием эндонуклеазы рестрикции HhaI, результаты которого показали, что рестрикционный профиль клонированных участков ДНК отличался размером и числом рестрикционных фрагментов (данные не приводятся). Всего на основании ПДРФ-анализа было выявлено четыре геномогруппы клонированных bphA1-генов, у представителей которых были определены нуклеотидные последовательности и проведен сравнительный анализ с гомологичными последовательностями из базы данных GenBank (табл. 2).
Таблица 2.
Сравнение клонированных нуклеотидных последовательностей фрагментов генов гидроксилирующих диоксигеназ (bphA1-генов) с гомологичными последовательностями из базы данных GenBank
| Клон (геномогруппа)/ штамм, размер анализируемого фрагмента ДНК |
Гомологичные гены в GenBank | Номера сравниваемых нуклеотидных последовательностей в GenBank | Сходство, % |
Ссылка |
|---|---|---|---|---|
| Клон Pp64 (I), 451 п.н. |
Гены, кодирующие кластер Риски
α-субъединицы ДО; клоны LhIspA24/LhIspA20/LhIspA21, некультивируемые бактерии |
EF565833.1/ EF565845.1/ EF565830.1 |
97.8–99.5 | Aguirre de Cárcer et al., 2007 |
| Клон Pp116 (II), 427 п.н. |
||||
| Клон Pp123 (III), 448 п.н. |
||||
| Клон Pp105 (VI), 466 п.н. |
Ген фенилпропионат ДО (hca); клон AN-11, некультивируемая бактерия | DQ521968.1 | 99.3 | Нет данных |
| Ген бифенил ДО (bph); клон AN-70, некультивируемая бактерия |
DQ521951.1 | 99.0 | Нет данных | |
| bphA1; клон G7-12, некультивируемая бактерия | EF596918.1 | 98.4 | Vézina et al., 2008 | |
| bphА1; Pseudomonas putida, плазмида pKF715A | AP015030.1 | 90.7 | Suenaga et al., 2017 | |
| bph-оперон, Pseudomonas sp. Cam-1 | AY027651.1 | 90.5 | Master, Mohn, 2001 | |
| bphА1; Pseudomonas alcaliphila JAB1 | CP016162.1 | 90.7 | Ridl et al., 2018 | |
| bphА1; Pseudomonas furukawaii KF707 | AP014862.1 | 90.7 | Kimura et al., 2018 | |
| Штаммы VRP2-2 и VRP2-6, 453 п.н. | bphА1; Pseudomonas putida B6-2 | CP015202.1 | 97.3 | Li et al., 2009 |
| bphА1; Pseudomonas sp. B3B | AJ544517.1 | 97.1 | Kahl, Hofer, 2003 | |
| bphА1; Pseudomonas sp. B6K | AJ544520.2 | 93.0 | Kahl, Hofer, 2003 | |
| bphА1; Pseudomonas alcaligenes B-357 | EF596934.1 | 90.7 | Vézina et al., 2008 | |
| bph-оперон, Pseudomonas sp. Cam-1 | AY027651.1 | 90.0 | Master, Mohn, 2001 | |
| bphА1; Pseudomonas putida, плазмида pKF715A | AP015030.1 | 89.8 | Suenaga et al., 2017 | |
| bphА1; Pseudomonas alcaliphila JAB1 | CP016162.1 | 89.8 | Ridl et al., 2018 | |
| bphА1; Pseudomonas furukawaii KF707 | AP014862.1 | 89.8 | Kimura et al., 2018 | |
| bphА1; Pseudomonas sp. B4 | AJ544519.1 | 89.8 | Kahl, Hofer, 2003 |
Большинство клонированных фрагментов ДНК, отнесенных на основании ПДРФ-анализа к трем разным геномогруппам и составляющих 80% от общего количества полученных рекомбинантных клонов, имели высокий процент сходства (97.8‒99.5%) с генами, кодирующими кластер Риске α-субъединицы диоксигеназ, некультивируемых бактерий из почвы, длительное время загрязненной ПХБ (Aguirre de Cárcer et al., 2007). Секвенированный фрагмент ДНК клона Pp105 (VI геномогруппа) имел высокое сходство (98.4‒99.3%) с генами большой α-субъединицы бифенил-2,3-диоксигеназ (ген bphA1) некультивируемых бактерий (Vezina et al., 2008) и не более 90.5% сходства с подобными генами бактерий рода Pseudomonas (P. putida плазмида pKF715A, Pseudomonas sp. Cam-1, P. alcaliphila JAB1, P. pseudoalcaligenes KF707) (табл. 2). Полученные результаты указывают на присутствие гетерогенных генетических систем бактерий в микробном сообществе почвы, отобранной на территории предприятия ОАО “Пермский завод смазок и СОЖ”, которые принимают участие в разложении полиароматических соединений. Результаты исследований, проведенных с использованием молекулярно-биологических методов, в том числе методов высокопроизводительного секвенирования, свидетельствуют об огромном разнообразии ароматических диоксигеназ, в том числе бифенилдиоксигеназ (БДО), в МС почвы и других объектов окружающей среды. Показано, что гены бифенил диоксигеназ культивируемых бактерий составляют лишь небольшую часть по сравнению с многообразием генов БДО в метагеноме микробного сообщества почвы (Aguirre de Cárcer et al., 2007; Iwai et al., 2010; Standfuss-Gabisch et al., 2012; Jurelevicius et al., 2012).
Идентификация, морфо-физиологические и генетические характеристики выделенных бактерий. Методом накопительного культивирования из техногеннозагрязненной почвы было выделено 2 аэробных бактериальных штамма (обозначенных VRP2-2 и VRP2-6), способных использовать в качестве ростового субстрата незамещенный бифенил. Клетки штаммов представляли собой грамотрицательные подвижные палочки, не образующие спор. При росте на среде LB колонии изолята VRP2-2 были круглые, желтые, выпуклые, гладкие, блестящие, размером 1–3 мм. Колонии штамма VRP2-6 имели размер 2‒4 мм, кремово-желтый цвет, круглые, выпуклые, с волнистым краем, матовые. Клетки изолированных штаммов ‒ подвижные палочки правильной формы с закругленными концами, оксидазо- и каталазоположительные. Выделенные штаммы активно росли в диапазоне температур от 4 до 45°С на богатой среде (LB), и от 4 до 37°С на минеральной среде K1 с добавлением бифенила в качестве единственного источника углерода. Ростовые эксперименты при изменении осмолярности среды показали, что штаммы VRP2-2 и VRP2-6 способны к росту (как на среде LB, так и на среде К1 с бифенилом) при концентрации NaCl до 50 г/л.
Результаты генотипирования показали, что штаммы отличаются друг от друга на молекулярно-генетическом уровне (рис. 1). Отличались штаммы и наличием плазмид в клетках. Так, в клетках штамма VRP2-6, выращенного на среде К1 с бифенилом, методом пульс-электрофореза была обнаружена плазмида размером около 280 т.п.н. В другом штамме VRP2-2 плазмидной ДНК выявлено не было (рис. 2).
Рис. 1.
ВОХ-ПЦР-профили штаммов VRP2-2 (1) и VRP2-6 (2). М – маркер молекулярных масс O’GeneRuler™ 100 bp Plus DNA Ladder (“Fermentas”, Литва).
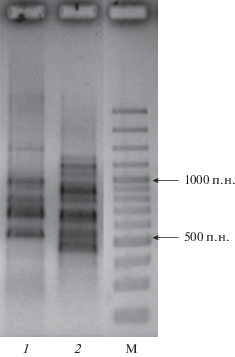
Рис. 2.
Электрофореграмма плазмидных ДНК штаммов рода Pseudomonas: 1 – маркер молекулярных масс “DNA Size Markers – Yeast Chromosoma” (“Bio-Rad Laboratories”, США), 2 – штамм VRP2-2, 3– штамм VRP2-6.

На основе анализа гена 16S рРНК штаммы VRP2-2 и VRP2-6 были отнесены к роду Pseudomonas и имели наибольшее сходство с типовым штаммом Pseudomonas alcaligenes NBRC 14159Т (100% сходства) и с типовым штаммом Pseudomonas taiwanensis BCRC 17751Т (99% сходства) соответственно. В литературе описан ряд штаммов псевдоманад ‒ активных деструкторов бифенила и ПХБ (Adebusoye et al., 2007; Hatamian-Zarmi et al., 2009; Li et al., 2009; Nam et al., 2014; Chakraborty, Das, 2016; Ridl et al., 2018), среди которых наиболее изученными являются штаммы (обозначенные KF), выделенные из техногенно загрязненной почвы с территории завода по производству бифенила в Японии (Furukawa et al., 1989). Установлено, что штаммы KF принадлежат к разным видам рода Pseudomonas, в том числе P. furukawaii (штамм KF707), P. putida (штаммы KF703, KF715), P. toyotomiensis (штамм KF710), P. aeruginosa (штамм KF702). Проведено полное секвенирование геномов этих штаммов, что позволило определить и проанализировать генетические структуры, лежащие в основе уникального метаболического потенциала этих бактерий, в том числе способности разлагать бифенил и ПХБ до безопасных соединений (Watanabe et al., 2015; Fujihara et al., 2015; Suenaga et al., 2015, 2017; Kimura et al., 2018).
Биодеградационные свойства изолированных бактерий. Установлено, что штаммы VRP2-2 и VRP2-6 используют в качестве единственного источника углерода и энергии не только бифенил, а также моноароматические углеводороды: орто-фталевую, бензойную кислоты. Штамм VRP2-6 активно рос на пара-гидроксибензойной, протокатеховой, салициловой кислотах, в то время как штамм VRP2-2 не утилизировал эти соединения (табл. 3). Штаммы не были способны к росту на нафталине, фенантрене, феноле, толуоле.
Таблица 3.
Рост штаммов VRP2-2 и VRP2-6 на ароматических соединениях
| Субстрат | Штаммы | |
|---|---|---|
| VRP2-2 | VRP2-6 | |
| Бифенил | ++ | +++ |
| Нафталин | – | – |
| Фенантрен | – | – |
| Фенол | – | – |
| Толуол | – | – |
| орто-Фталевая кислота | + | ++ |
| Салициловая кислота | – | +++ |
| пара-Гидроксибензойная кислота | – | +++ |
| Протокатеховая кислота | – (т.о.с.) |
+++ |
| Бензойная кислота | +++ | +++ |
На основании проведенных исследований можно предположить, что деградация бифенила штаммами VRP2-2 и VRP2-6 осуществляется по классическому пути через образование пентадиеновой и бензойной кислот, с последующим разложением бензойной кислоты (Pieper, Seeger, 2008). Установлено, что оба штамма активно растут на бензойной кислоте (табл. 3). Результаты ПЦР-анализа показали, что в геноме обоих штаммов присутствует ген benА (данные не приводятся), кодирующий малую субъединицу фермента бензоат 1,2-диоксигеназы – ключевого фермента разложения бензоата у бактерий, что указывает на присутствие метаболического пути разложения бензоата через образование ключевого интермедиата – пирокатехина (Ridi et al., 2018).
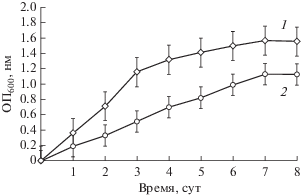
При выращивании в жидкой среде на бифениле штамм VRP2-6 демонстрировал более высокую скорость роста (0.098 ч–1), чем штамм VRP2-2 (0.042 ч–1) (рис. 3), и был проверен на способность осуществлять разложение хлорированных бифенилов. Показано, что штамм VRP2-6 эффективно утилизирует орто- и пара-моноХБ в высокой концентрации (250 мг/л), окисляя не хлорированное кольцо молекулы ХБ (табл. 4). При культивировании на 2-ХБ и 4-ХБ уже к 3 ч количество хлорбифенилов в среде уменьшалось, соответственно, в 4.9 и 4.3 раза. За 24 ч инкубации 2-ХБ был практически полностью утилизирован (97.1% от теоретически возможного), а разложение 4-ХБ осуществлено на 82.3%. В среде не были обнаружены промежуточные продукты метаболизма – 2-гидроксо-6-оксо-(хлорфенил)гекса-2,4-диеновые кислоты (ГОФДК), что может указывать на высокую активность ферментных систем деструкции моноХБ у штамма VRP2-6 (Maltseva et al., 1999). В исследуемые периоды времени в среде были зарегистрированы хлорбензойные кислоты в количестве 2.0‒3.5 мг/л (табл. 4). Наличие таких небольших количеств ХБК (не более 2% от теоретически возможного), может указывать на последующее разложение клетками штамма этих хлорсодержащих метаболитов. Известны немногочисленные штаммы рода Pseudomonas, способные полностью утилизировать образующиеся в процессе разложения монохлорбифенилов соответствующие ХБК (Chae et al., 2000; Kim, Picardal, 2001; Hatamian-Zarmi et al., 2009).
Таблица 4.
Деструкция хлорбифенилов штаммом Pseudomonas sp. VRP2-6
| Субстрат | Время инкубации, ч |
Содержание субстрата | Продукт деструкции | |||||
|---|---|---|---|---|---|---|---|---|
| мг/л | %* | ГОФДК | ХБК | мг/л | %* | |||
| λmax, нм | ОП, ед. | |||||||
| 0 | 250.00 ± 0.01 | 100.00 | Н.д | Н.д | 0.36 ± 0.02 | 0.17 | ||
| 2-ХБ | 3 | 51.45 ± 0.04 | 20.58 | Н.д | Н.д | 2-ХБК | 2.45 ± 0.05 | 1.18 |
| 24 | 7.43 ± 0.02 | 2.90 | Н.д | Н.д | 3.50 ± 0.03 | 1.68 | ||
| 0 | 250.00 ± 0.01 | 100.00 | Н.д | Н.д | 0.30 ± 0.03 | 0.16 | ||
| 4-ХБ | 3 | 57.80 ± 0.03 | 23.12 | Н.д | Н.д | 4-ХБК | 2.00 ± 0.04 | 0.96 |
| 24 | 44.30 ± 0.05 | 17.70 | Н.д | Н.д | 2.35 ± 0.03 | 1.13 | ||
| 0 | 44.60 ± 0.01 | 100.00 | Н.д. | Н.д. | Н.д. | Н.д. | ||
| 2,4′-ХБ | 3 | 40.80 ± 0.02 | 91.48 | 396 | 0.696 | 4-ХБК | 0.085 ± 0.002 | 0.27 |
| 24 | 35.70 ± 0.03 | 80.00 | 395 | 0.804 | 0.090 ± 0.005 | 0.28 | ||
Трансформация штаммом VRP2-6 дихлорированного бифенила (2,4'-диХБ) осуществлялась менее активно, чем монохлорбифенилов. Так, к 24 ч инкубации количество субстрата в среде культивирования оставалось на уровне 80% от начальной концентрации. Было зарегистрировано накопление продукта мета-расщепления дихлорбифенила ‒ 3,8-Cl ГОФДК с λmax = = 395/396 нм, что указывает на 2,3-диоксигенирование пара-хлорированного кольца 2,4'-диХБ (Maltseva et al., 1999). Кроме того, в среде культивирования происходило накопление небольшого количества 4-ХБК, что может свидетельствовать о трансформации 2,4'-диХБ по пути предпочтительного 2,3-диоксигенирования орто-хлорированного кольца (Maltseva et al., 1999). Таким образом, БДО штамма VRP2-6 может осуществлять окисление как орто-, так и пара-замещенного кольца молекулы 2,4'-диХБ. Ранее сообщалось, что штамм Pseudomonas sр. SA-6 использовал 2,4'-диХБ в качестве единственного источника углерода и энергии, окисляя орто-хлорированное кольцо молекулы бифенила, и, далее, осуществлял деструкцию образующейся 4-ХБК (Adebusoye et al., 2007). Для выявления особенностей метаболизма деструкции хлорбифенилов, различающихся количеством и положением хлора (орто-, пара-, мета-) в молекуле, а также продуктов их разложения – хлорбензоатов штаммом Pseudomonas sр. VRP2-6, предполагается проведение дальнейших исследований.
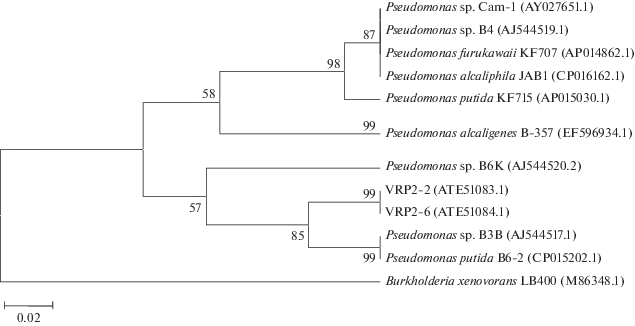
bphA1-Гены Pseudomonas sрp. VRP2-2 и VRP2-6. Методом ПЦР со специфичными праймерами (Iwai et al., 2010) установлено наличие у выделенных штаммов гена bphA1, кодирующего α-субъединицу бифенил 2,3-диоксигеназы – ключевого фермента деструкции бифенила. Данный факт подтверждает, что в разложении бифенила/ПХБ участвуют ферменты, кодируемые кластером bph-генов классического “верхнего” пути деструкции бифенила, осуществляющие конверсию бифенила до бензойной кислоты (Pieper, Seeger, 2008). Анализ нуклеотидных последовательностей секвенированных фрагментов генов bphA1 штаммов VRP2-2 и VRP2-6 показал их идентичность и наибольшее родство (сходство на уровне 89.8–97.3%) с генами α-субъединицы бифенил 2,3-диоксигеназы бактерий-деструкторов бифенила/ПХБ рода Pseudomonas (табл. 2). Филогенетический анализ транслированных аминокислотных последовательностей bphA1-генов штаммов VRP2-2 и VRP2-6 показал, что они формируют отдельную ветвь на дереве БДО (рис. 4). Эта ветвь наиболее близко располагается к ветви бифенил 2,3-диоксигеназ штаммов-деструкторов бифенила P. putida B6-2 (Li et al., 2009) и Pseudomonas sp. B3B (Kahl, Hofer, 2003). Уровни сходства bphA1-генов изолированных нами штаммов с таковыми вышеуказанных псевдомонад составляют 97.3 и 97.1% соответственно (табл. 2). Анализ генома деструктора ароматических соединений P. putida B6-2 показал, что в геноме содержится “классический” кластер bph-генов (bphABCKHJID) и гены (опероны), ответственные за разложение бензоата, катехола, пара-гидроксибензоата, салицилата (Li et al., 2009). Невысокий процент сходства (около 90%) bphA1-гены исследуемых штаммов имели с подобными генами хорошо охарактеризованных штаммов KF − P. furukawaii KF707 и P. putida KF715 (табл. 2, рис. 4), которые являются высокоактивными деструкторами хлорированных бифенилов, содержащих различное количества атомов хлора (в орто-, пара- и мета-положении) в молекуле бифенила, а также смесей ПХБ (Suenaga et al., 2017; Kimura et al., 2018). Ранее было установлено, что кластер bph-генов находится в одной из четырех плазмид штамма P. putida KF715, плазмиде pKF715A размером 483 т.п.н., тогда как у бесплазмидного штамма KF707 идентичные bph-гены локализованы в хромосоме (Kimura et al., 2018). Установлено, что мобильный элемент, содержащий bph-гены (плазмида pKF715A) может интегрироваться в хромосому (Suenaga et al., 2017). Исследование наших штаммов выявило наличие плазмиды размером около 280 т.п.н. у штамма VRP2-6 (рис. 2), что не исключает возможности нахождение bph-генов на плазмидной ДНК. Интересен факт наличия идентичных bphА1-генов у штамма VRP2-2 (сходство 100%), однако в клетках штамма не было обнаружено плазмид, что указывает на локализациию этих генетических структур в хромосоме. Исследование особенностей строения и функционирования генетических структур (в том числе мобильных элементов – плазмид, транспозонов), ответственных за разложение бифенила/ПХБ, у выделенных из одного биотопа штаммов-деструкторов будет продолжено.
Рис. 4.
Положение bphA1-генов штаммов Pseudomonas spp. VRP2-2 и VRP2-6 на филогенетическом дереве, построенном на основании сравнительного анализа транслированных аминокислотных последовательностей с использованием метода UPGMA. Цифрами показана достоверность ветвления, установленная с помощью “bootstrap”-анализа. В качестве аутгруппы использована аминокислотная последовательность большой субъединицы бифенил 2,3-диоксигeназы Burkholderia xenovorans LB400 (M86348.1).
Таким образом, результаты исследований показали, что в загрязненной почве (территория предприятия ОАО “Пермский завод смазок и СОЖ”) присутствует сообщество бактерий, способных осуществлять разложение ароматических соединений, в том числе бифенила и его токсичных хлорпроизводных (полихлорбифенилов). Методом накопительного культивирования из почвы выделены два активных деструктора бифенила рода Pseudomonas. Один из изолятов (штамм VRP2-6) эффективно утилизировал орто- и пара-монохлорированные бифенилы и осуществлял деструкцию дихлорбифенила (2,4'-диХБ). Филогенетический анализ генов (bphA1), кодирующих α‑субъединицу бифенил ДО, у выделенных псевдомонад (имеющих разную видовую принадлежность) показал их идентичность и высокое, но не превышающее 97.3% сходство с bphA1-генами известных деструкторов бифенила/ПХБ рода Pseudomonas. Такой тип bphA1-генов не был обнаружен при молекулярном исследовании общего пула ДНК, выделенной из загрязненной почвы, что можно объяснить присутствием небольшого количества культивируемых бактерий, содержащих аналогичные нуклеотидные последовательности (bphA1), в почве. В то же время, в исследуемой почвенной ДНК (с применением молекулярных методов ‒ амплификации, клонирования, секвенирования) были выявлены два других типа bphA1-подобных генов, наиболее филогенетически близких с генами ароматических диоксигеназ (в том числе, фенилпропионат ДО, бифенил ДО) некультивируемых бактерий. Полученные результаты не противоречат данным других исследователей, показавших, что бифенил диоксигеназы (гены БДО) культивируемых бактерий составляют лишь небольшую часть, по сравнению с многообразием БДО в метагеноме микробных сообществ почвы (Iwai et al., 2010; Standfuss-Gabisch et al., 2012; Jurelevicius et al., 2012). Предполагается дальнейшее всестороннее изучение новых штаммов Pseudomonas spp. VRP2-2 и VRP2-6 с целью возможного использования их генетического и метаболического потенциала при разработке биотехнологий, направленных на мониторинг и восстановление загрязненных бифенилом и ПХБ территорий.
ФИНАНСИРОВАНИЕ РАБОТЫ
Работа выполнена в рамках государственного задания (номер госрегистрации темы 01201353249).
Список литературы
Васильев А.В. Методологические подходы к классификации смазочно-охлаждающих жидкостей и оценка их токсикологического воздействия на человека и биосферу // Изв. Самарского научного центра РАН. 2017. Т. 19. № 5(2). С. 235‒241.
Васильева Г.К., Стрижакова Е.П. Биоремедиация почв и седиментов, загрязненных полихлорированными бифенилами // Микробиология. 2007. Т. 76. № 6. С. 725‒741.
Vasilyeva G.K., Strijakova E.R. Bioremediation of soils and sediments contaminated by polychlorinated biphenyls // Microbiology (Moscow). 2007. V. 76. P. 639–653.
Егорова Д.О., Корсакова Е.С., Демаков В.А., Плотникова Е.Г. Деструкция ароматических углеводородов штаммом Rhodococcus wratislavensis KT112-7, выделенным из отходов соледобывающего предприятия // Прикл. биохимия и микробиология. 2013. Т. 49. № 3. С. 267–278.
Egorova D.O., Korsakova E.S., Demakov V.A., Plotnikova E.G. Degradation of aromatic hydrocarbons by the Rhodococcus wratislaviensis KT112-7 isolated from waste products of a salt-mining plant // Appl. Biochem. Microbiol. 2013. V. 49. P. 244–255.
Методы общей бактериологии: Пер. с англ. / Под ред. Герхардта Ф. с соавторами. М.: Мир, 1983. Т. 1‒3.
Розанова Е.П., Назина Т.Н. Углеводородокисляющие бактерии и их активность в нефтяных пластах // Микробиология. 1982. Т. 51. С. 324‒348.
Rozanova E.P., Nazina T.N. Hydrocarbon-oxidizing bacteria and their activity in oil pools // Microbiology (Moscow). 1982. V. 51. P. 287–293.
Шумкова Е.С., Егорова Д.О., Боронникова С.В., Плотникова Е.Г. Полиморфизм генов bphA бактерий-деструкторов бифенила/полихлорированных бифенилов // Мол. биология. 2015. Т. 49. № 4. С. 638‒648.
Shumkova E.S., Egorova D.O., Boronnikova S.V., Plotnikova E.G. Polymorphism of the bphA genes in bacteria destructing biphenyl/chlorinated biphenils // Mol. Biol. (Moscow). 2015. V. 49. P. 569–580.
Adebusoye S.A., Picardal F.W., Ilori M.O., Amund O.O., Fuqua C., Grindle N. Growth on dichlorobiphenyls with chlorine substitution on each ring by bacteria isolated from contaminated African soils // Appl. Microbiol. Biotechnol. 2007. V. 74. P. 484–492.
Aguirre de Cárcer D., Martín M., Karlson U., Rivilla R. Changes in bacterial populations and in biphenyl dioxygenase gene diversity in a polychlorinated biphenyl-polluted soil after introduction of willow trees for rhizoremediation // Appl. Environ. Microbiol. 2007. V. 73. P. 6224–6232.
Cébron A., Norini M.P., Beguiristain T., Leyval C. Real-Time PCR quantification of PAH-ring hydroxylating dioxygenase (PAH-RHDα) genes from Gram positive and Gram negative bacteria in soil and sediment samples // J. Microbiol. Meth. 2008. V. 73. P. 148‒159.
Chae J.-C., Kim E., Park S.-H., Kim C.-K. Catabolic degradation of 4-chlorobiphenyl by Pseudomonas sp. DJ-12 via consecutive reaction of meta-cleavage and hydrolytic dechlorination // Biotechnol. Bioproc. Eng. 2000. V. 5. P. 449‒455.
Chakraborty J., Das S. Characterization of the metabolic pathway and catabolic gene expression in biphenyl degrading marine bacterium Pseudomonas aeruginosa JP-11 // Chemosphere. 2016. V. 144. P. 1706–1714.
Fierer N., Jackson J.A, Vilgalys R., Jackson R.B. Assessment of soil microbial community structure by use of taxon-specific quantitative PCR assays // Appl. Environ. Microbiol. 2005. V. 71. P. 4117–4120.
Fujihara H., Yamazoe A., Hosoyama A., Suenaga H., Kimura N., Hirose J., Watanabe T., Futagami T., Goto M., Furukawa K. Draft genome sequence of Pseudomonas aeruginosa KF702 (NBRC 110665), a polychlorinated biphenyl-degrading bacterium isolated from biphenyl-contaminated soil // Genome Announc. 2015. V. 3(3). e00517-15.
Furukawa K., Hayase N., Taira K., Tomizuka N. Molecular relationship of chromosomal genes encoding biphenyl/polychlorinated biphenyl catabolism: some soil bacteria possess a highly conserved bph operon // J. Bacteriol. 1989. V. 171. P. 5467‒5472.
Furukawa K., Suenaga H., Goto M. Biphenyl dioxygenases: functional versatilities and directed evolution // J. Bacteriol. 2004. V. 186. P. 5189–5196.
Hatamian-Zarmi A., Shojaosadati S.A., Vasheghani-Farahani E., Hosseinkhani S., Emamzadeh A. Extensive biodegradation of highly chlorinated biphenyl and Aroclor 1242 by Pseudomonas aeruginosa TMU56 isolated from contaminated soils // Int. Biodeterior. Biodegr. 2009. V. 63. P. 788‒794.
Iwai S., Chai B., Sul W.J., Cole J.R., Hashsham S.A., Tiedje J.M. Gene-targeted-metagenomics reveals extensive diversity of aromatic dioxygenase genes in the environment // ISME J. 2010. V. 4. P. 279–285.
Jurelevicius D., Alvarez V.M., Peixoto R., Rosado A.S., Seldin L. Bacterial polycyclic aromatic hydrocarbon ring-hydroxylating dioxygenases (PAH-RHD) encoding genes in different soils from King George Bay, Antarctic Peninsula // Appl. Soil Ecol. 2012. V. 55. P. 1‒9.
Kahl S., Hofer B. A genetic system for the rapid isolation of aromatic-ring-hydroxylating dioxygenase activities // Microbiology (SGM). 2003. V. 149. P. 1475–1481.
Kim S.F., Picardal W. Microbial growth on dichlorobiphenyls chlorinated on both rings as a sole carbon and energy source // Appl. Environ. Microbiol. 2001. V. 64. P. 1953–1955.
Kimura N., Watanabe T., Suenaga H., Fujihara H., Futagami T., Goto M., Hanada S., Hirose J. Pseudomonas furukawaii sp. nov., a polychlorinated biphenyl-degrading bacterium isolated from biphenyl-contaminated soil in Japan // Int. J. Syst. Evol. Microbiol. 2018. V. 68. P. 1429‒1435.
Li Q., Wang X., Yin G., Gai Z., Tang H., Ma C., Deng Z., Xu P. New metabolites in dibenzofuran cometabolic degradation by a biphenyl-cultivated Pseudomonas putida strain B6-2 // Environ. Sci. Technol. 2009. V. 43. P. 8635–8642.
Maltseva O.V., Tsoi T.V., Quensen III J.F., Fukuda M., Tiedje J.M. Degradation of anaerobic reductive dechlorination products of Aroclor 1242 by aerobic bacteria // Biodegradation. 1999. V. 10. P. 363‒371.
Master E.R., Mohn W.W. Induction of bphA, encoding biphenyl dioxygenase, in two polychlorinated biphenyl-de-grading bacteria, psychrotolerant Pseudomonas strain Cam-1 and mesophilic Burkholderia strain LB400 // Appl. Environ. Microbiol. 2001. V. 67. P. 2669‒2676.
Nam I.-H., Chon C.-M., Jung K.-Y., Kim J.-G. Biodegradation of biphenyl and 2-chlorobiphenyl by a Pseudomonas sp. KM-04 isolated from PCBs-contaminated coal mine soil // Bull. Environ. Contam. Toxicol. 2014. V. 93. P. 89–94.
Pieper D.H., Seeger M. Bacterial metabolism of polychlorinated biphenyls // J. Mol. Microbiol. Biotechnol. 2008. V. 15. P. 121–138.
Ridl J., Suman J., Fraraccio S., Hradilova M., Strejcek M., Cajthaml T., Zubrova A., Macek T., Strnad H., Uhlik O. Complete genome sequence of Pseudomonas alcaliphila JAB1 (=DSM 26533), a versatile degrader of organic pollutants // Stand. Genomic Sci. 2018. V. 13. P. 1‒10. https://doi.org/10.1186/s40793-017-0306-7
Sharma J.K., Gautam R.K., Nanekar S.V., Weber R., Singh B.K., Singh S.K., Juwarkar A.A. Advances and perspective in bioremediation of polychlorinated biphenyl-contaminated soils // Environ. Sci. Pollut. Res. 2018. V. 25. P. 16355‒16375.
Short Protocols in Molecular Biology. 3rd ed. / Eds. Ausbel F.M., Brent R., Kingston R.E., Moore D.D., Seidman J.G., Smith J.A., Struhl K. New York: John Wiley & Sons, 1995, 450 p.
Standfuss-Gabisch C., Al-Halbouni D., Hofer B. Characterization of biphenyl dioxygenase sequences and activities encoded by the metagenomes of highly polychlorobiphenyl contaminated soils // Appl. Environ. Microbiol. 2012. V. 78. P. 2706–2715.
Suenaga H., Fujihara H., Kimura N., Hirose J., Watanabe T., Futagami T., Goto M., Shimodaira J., Furukawa K. Insights into the genomic plasticity of Pseudomonas putida KF715, a strain with unique biphenyl-utilizing activity and genome instability properties // Environ. Microbiol. Rep. 2017. V. 9. P. 589‒598.
Suenaga H., Yamazoe A., Hosoyama A., Kimura N., Hirose J., Watanabe T., Fujihara H., Futagami T., Goto M., Furukawa K. Draft genome sequence of the polychlorinated biphenyl-degrading bacterium Pseudomonas putida KF703 (NBRC 110666) isolated from biphenyl-contaminated soil // Genome Announc. 2015. V. 3 (2). e00142-15.
Versalovic J., Schneider M., Frans J. de Bruijn., Lupski J.R. Genomic fingerprinting of bacteria using repetitive sequence-based polymerase chain reaction // Meth. Cell. Mol. Biol. 1994. V. 5. P. 25‒40.
Vezina J., Barriault D., Sylvestre M. Diversity of the C-terminal portion of the biphenyl dioxygenase large subunit // J. Mol. Microbiol. Biotechnol. 2008. V. 15. P. 139–151.
Watanabe T., Yamazoe A., Hosoyama A., Fujihara H., Suenaga H., Hirose J., Futagami T., Goto M., Kimura N., Furukawa K. Draft genome sequence of Pseudomonas toyotomiensis KF710, a polychlorinated biphenyl-degrading bacterium isolated from biphenyl-contaminated soil // Genome Announc. 2015. V. 3 (3). e00223-15.
Дополнительные материалы отсутствуют.