Микробиология, 2019, T. 88, № 4, стр. 426-437
Выявление хитинолитического потенциала у пресноводного планктомицета Planctomicrobium piriforme
И. С. Куличевская a, Д. Г. Наумов a, А. А. Иванова a, А. Л. Ракитин b, С. Н. Дедыш a, *
a Институт микробиологии им. С.Н. Виноградского, ФИЦ Биотехнологии РАН
119071 Москва, Россия
b Институт биоинженерии, ФИЦ Биотехнологии РАН
119071 Москва, Россия
* E-mail: dedysh@mail.ru
Поступила в редакцию 04.03.2019
После доработки 11.03.2019
Принята к публикации 29.03.2019
Аннотация
Представители филогенетической группы Planctomycetes населяют широкий спектр водных и наземных местообитаний, однако их функциональная роль в экосистемах изучена слабо. Одна из предполагаемых функций этих бактерий – это участие в деструкции ряда природных полимеров клеточных стенок растений и грибов. Первые экспериментальные доказательства способности планктомицетов к деструкции хитина были получены для Fimbriiglobus ruber SP5T, в геноме которого выявлен ген, кодирующий хитиназу семейства GH18, а также другие гены утилизации этого полисахарида. Филогенетический анализ хитиназы из Fimbriiglobus показал, что ее близкий гомолог имеется и у другого планктомицета, Planctomicrobium piriforme Р3Т. Экспериментальная проверка типового штамма Р3Т, а также нового изолята этого вида, штамма РХ70, выявила их способность к росту на аморфном хитине в качестве источника углерода и азота. Активности ферментов цепи деструкции хитина, определенные с использованием флуоресцентно меченых хитоолигосахаридов, были ассоциированы с клетками планктомицетов, но не с культуральной жидкостью. Скрининг базы данных аминокислотных последовательностей выявил наличие близких гомологов хитиназ F. ruber и P. piriforme у ряда других планктомицетов, представляющих различные таксономические группы. Более того, филогенетический анализ указал на планктомицеты как на вероятный источник множественных горизонтальных переносов гена хитиназы в бактерии других фил и даже в эукариоты. Выявление способности P. piriforme к деструкции и утилизации хитина позволяет внести коррективы в ранее опубликованное описание этого вида, а также расширить спектр планктомицетов с экспериментально подтвержденным хитинолитическим потенциалом.
Планктомицеты (Planctomycetes) – это филогенетическая группа домена Bacteria, представители которой населяют широкий круг природных и антропогенных местообитаний, характеризуются сложной организацией клеток, большими геномами и малоизученным функциональным потенциалом (Boedeker et al., 2017; Wiegand et al., 2018). Аннотирование геномов и исследования с использованием подходов метатранскриптомики указывают на возможную роль планктомицетов как компонентов гидролитических микробных сообществ (Ivanova et al., 2017, 2018; Dedysh, Ivanova, 2019), однако накопление экспериментальных доказательств этого положения методически сдерживается низкими скоростями роста этих бактерий, что затрудняет проведение рутинных лабораторных тестов. Ранее принятые представления о механизмах гидролиза полимеров бактериями, по всей видимости, также не вполне применимы для планктомицетов. Как показали исследования последнего времени, у ряда планктомицетов имеет место необычный механизм транспорта высокомолекулярных полимеров в клетку, в котором предположительно задействованы кратериформные структуры и фимбрии, являющиеся типичными поверхностными структурами клеток планктомицетов (Boedeker et al., 2017; Reintjes et al., 2017). Такой механизм позволяет аккумулировать макромолекулы полимерного субстрата в периплазматическом пространстве клеток и позволяет планктомицетам конкурировать за полимерные субстраты с быстрорастущими гидролитиками (Reintjes et al., 2017).
Одной из важных находок последних лет явилось доказательство способности планктомицетов к деструкции хитина, полученное для болотного представителя семейства Gemmataceae, Fimbriiglobus ruber SP5T (Ravin et al., 2018). Анализ генома этого планктомицета выявил наличие ряда генов, обусловливающих рост на хитине, в том числе кодирующих хитиназу из семейства GH18 гликозил-гидролаз, β-N-ацетилглюкозаминидазу из семейства GH20, а также полный набор ферментов, ответственных за дальнейшее использование N-ацетилглюкозамина как источника углерода и энергии. Все представители порядка Planctomycetales способны расти на N-ацетилглюкозамине (Staley et al., 1992), однако о наличии хитиназ у планктомицетов ранее известно не было. Ген, кодирующий гипотетическую хитиназу F. ruber SP5T, был клонирован и экспрессирован в E. coli с последующим успешным подтверждением хитинолитической активности рекомбинантного фермента (Ravin et al., 2018). Рост F. ruber SP5T на аморфном хитине был подтвержден экспериментально, однако он наблюдался только в том случае, когда этот биополимер использовался в качестве единственного источника азота, а не углерода. Такое целевое использование хитина в качестве источника азота обусловлено специфическими особенностями экосистемы, из которой выделен этот планктомицет – ультрапресных сфагновых болот, в которых имеется множество потенциальных источников углерода, но не источников минерального азота. Поскольку хитин и хитозан входят в состав клеточных стенок микромицетов и панцирей беспозвоночных, обильно представленных в кислых торфяниках, получение азота путем гидролиза хитина может являться одной из возможных стратегий выживания болотных планктомицетов.
Хитиназы или 1,4-β-поли-N-ацетилглюкозаминидазы (К.Ф. 3.2.1.14) – эндо-гликозил-гидролазы, катализирующие расщепление хитина и хитодекстринов (Nguyen et al., 2018; Oyeleye, Normi, 2018; Tamadoni Jahromi, Barzkar, 2018). Ферменты с этой активностью широко распространены в природе и отнесены в базе данных CAZy к четырем семействам: GH18, GH19, GH23 и GH48 (Lombard et al., 2014). Однако каждое из этих семейств помимо белков с хитинолитической активностью также содержит ферменты с иной субстратной специфичностью, что затрудняет предсказание энзиматической активности у экспериментально не исследованных представителей этих семейств. Обычно такие предсказания бывают обоснованными только в пределах уже охарактеризованных моноспецифичных подсемейств (Наумов, 2011). В 2017 году в качестве носителей самостоятельных энзиматических активностей в базе данных ExplorEnz (McDonald et al., 2009) были признаны два варианта экзо-хитиназы (К.Ф. 3.2.1.200 и К.Ф. 3.2.1.201) и эндо-хитодекстриназа (К.Ф. 3.2.1.202); белки с этими активностями отнесены в базе данных CAZy к семейству GH18, однако они там указаны как носители эндо-хитиназной активности (К.Ф. 3.2.1.14). Филогенетический анализ хитиназы семейства GH18 (клан GH-K) из Fimbriiglobus ruber SP5T с уже доказанной функцией (Ravin et al., 2018) показал, что один из ближайших гомологов этого фермента закодирован в геноме (GenBank, NZ_FOQD00000000.1) другого пресноводного планктомицета, P. piriforme DSM 26348Т. Этот планктомицет был выделен из образца торфа, отобранного с участка заболоченной литорали внутреннего Лещева озера на острове Валаам, и охарактеризован в качестве аэробного хемоорганотрофа с ограниченным гидролитическим потенциалом (Kulichevskaya et al., 2015). Оригинальное описание вида P. piriforme содержит указание на способность к росту на ламинарине, лихенане, ксилане, эскулине и желатине, но не на хитине и хитозане. Первичная проверка способности P. piriforme к росту на хитине, однако, проводилась с использованием кристаллического хитина из панцирей крабов (Kulichevskaya et al., 2015), тогда как в экспериментах с F. ruber SP5T была использована его аморфная форма (Ravin et al., 2018).
Целью настоящего исследования было экспериментально проверить наличие хитинолитического потенциала у типового штамма P. piriforme и нового изолята этого вида, а также провести детальный анализ филогении хитиназ, кодируемых в геномах этих бактерий.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Объекты исследования. В работе были использованы два штамма планктомицетов вида Planctomicrobium piriforme – типовой штамм Р3Т (=DSM 26348Т) и новый изолят этого вида, штамм РХ70. Последний был выделен из образца воды озера Мороцкое Вологодской области (58°43′28″ N, 37°39′07″ E) с помощью ранее описанной процедуры (Куличевская и соавт., 2006). Морфологические и фенотипические характеристики штамма РХ70 были идентичны таковым штамма Р3Т. Последовательность гена 16S рРНК штамма РХ70 (депонирована в GenBank под номером MK581019) имела 99.1% сходства с таковой у P. piriforme Р3Т, что позволило идентифицировать штамм РХ70 как представителя этого вида. Культуры планктомицетов поддерживали на модифицированной среде 621 (DSMZ), содержащей (г/л): пептон – 0.1; дрожжевой экстракт – 0.25; NH4NO3 – 0.1; 20 мл минерального раствора Хатнера (Staley et al., 1992). После стерилизации в среду добавляли 1 мл 5% раствора глюкозы и 1 мл/л раствора смеси витаминов (Staley et al., 1992); pH 6.5. В качестве желирующего агента использовали коммерческий препарат полисахарида микробного происхождения – фитагель (10 г/л; “Fluka”, США).
Наличие способности к росту на хитине проверяли с использованием жидкой среды, содержащей 20 мл/л минерального раствора Хатнера, 1 мл/л раствора смеси витаминов и 0.05 г/л дрожжевого экстракта. Аморфный хитин, приготовленный в соответствии с ранее описанной методикой (Sorokin et al., 2014), добавляли в среду в концентрации 0.1%. В первой серии экспериментов хитин служил единственным источником углерода, тогда как источником азота служил (NH4)2SO4 (0.01%). Во второй серии экспериментов источником углерода служила глюкоза (0.1 г/л), а хитин был добавлен как единственный источник азота, в контрольных вариантах эксперимента хитин был исключен. Опыты проводили в трехкратной повторности в 160 мл сывороточных флаконах, содержащих 30 мл среды при температуре инкубации 22°С. Начальная численность клеток, после внесения инокулята в среды, составляла 0.40 ± 0.11 × 108 мл–1 в случае штамма Р3Т и 0.50 ± 0.13 × 108 мл–1 в случае штамма РХ70. После 30-суточной инкубации аликвоты суспензий объемом 1 мл фиксировали 4% раствором свежеприготовленного раствора параформальдегида, как описано ранее (Dedysh et al., 2001). Подсчет числа клеток планктомицетов в культурах осуществляли путем гибридизации фиксированных проб суспензий с эквимолярной смесью 16S рРНК-специфичных олигонуклеотидных зондов PLA46 и PLA886, разработанных для детекции представителей филогенетической группы Planctomycetes (Neef et al., 1998). Олигонуклеотидные зонды были синтезированы фирмой “Синтол” (Москва, Россия). Гибридизацию проб проводили на покрытых 0.1% раствором желатина тефлоновых предметных стеклах с окошками. Препараты анализировали с использованием эпифлуоресцентного микроскопа Zeiss Axioplan 2 (Йена, Германия) со светофильтрами Zeiss 20 для Cy3-меченых зондов и Zeiss 02 для окраски ДАФИ. Численность клеток планктомицетов в пробах определяли путем учета количества гибридизованных с зондами клеток в 100 полях зрения микроскопа для каждого варианта опыта с последующим расчетом численности планктомицетов в 1 мл культуры планктомицета.
Определение активности ферментов, участвующих в разложении хитина. Хитинолитическую активность определяли в культурах клеток P. piriforme Р3Т, выращенных в жидкой среде с хитином в течении 30 сут, флуориметрическим методом по количеству высвобождаемого 4-метилумбеллиферона (4-МУ) с помощью набора реагентов для определения хитиназной активности Chitinase Assay Kit CS1030 (“Sigma”, США) в соответствии с рекомендациями производителя. Для определения типа хитинолитической активности ферментов, участвующих в гидролизе хитина и его производных, использовали следующие флуоресцентно меченые хитоолигосахариды: 4-МУ-β-D-N,N',N"-триацетил-хитотриозу (субстрат для определения активности эндохитиназы), 4-МУ-β-D-N,N'-диацетил-хитобиозид (для определения хитибиозидазной активности) и 4-МУ-N-ацетил-β-D-глюкозамин (для определения активности β-N-ацетилглюкозаминидазы). Осадок клеток получали путем центрифугирования 40 мл культуры при 8000 об./мин в течение 10 мин при 4°С на центрифуге Eppendorf 5810R (Германия). К осадку добавляли 0.4 мл 50 мМ фосфатного буфера (рН 6.0). Клетки разрушали с помощью ультразвукового гомогенизатора Bandelin Sonopuls HD 2200 (Германия) в течение 10 мин при 50% мощности прибора. Полученные лизаты клеток разделяли центрифугированием (12 000 об./мин в течение 20 мин) на фракции супернатанта и осадка; супернатант отбирали, а к осадку добавляли 100 мМ фосфатный буфер (рН 6) в объеме, равном объему супернатанта. Ферментативную активность измеряли в следующих фракциях исследуемых культур: (1) культуральной жидкости без клеток, (2) фракции неразрушенных клеток, (3) фракции разрушенных ультразвуком клеток и (4) и (5) фракциях супернатанта и осадка, полученных центрифугированием разрушенных клеток. Анализ ферментативной активности проводили в соответствии с рекомендациями производителя, в трех повторностях при 25°С в 100 мM фосфатном буфере (рН 6.0). Время инкубации исследуемого образца с флуоресцентно меченым субстратом составляло 30 мин. Интенсивность флуоресценции продуктов реакции измеряли на флуориметре Люмекс Флюорат 02 Панорама (Россия) при длине волны поглощения 360 нм и испускания 450 нм. За одну единицу хитинолитической активности принимали количество фермента, высвобождающего 1 нмоль 4-МУ из соответствующего субстрата за 1 мин в заданных условиях. Примеры использования этого метода для анализа хитинолитической активности различных микроорганизмов приведены в ранее опубликованных работах (Chernin et al., 1998; Huang et al., 2005).
Филогенетический анализ хитиназ. Полноразмерную аминокислотную последовательность хитиназы Fimbriiglobus ruber SP5T (GenPept, OWK46432.1) использовали для поиска близких гомологов в базе данных GenPept (раздел “non-redundant protein sequences”) с помощью программы blastp (http://www.nc-bi. nlm.nih.gov/). Для построения множественного выравнивания и дальнейшего филогенетического анализа были отобраны 133 ближайших гомолога, при этом близкородственные белки из штаммов одного вида и видов одного рода, как правило, не использовались. В качестве внешней группы использовали гипотетическую хитиназу Paludisphaera borealis (APW59709.1), а также несколько ее ближайших гомологов из планктомицетов (AGA26805.1, AMV39548.1, OJW15874.1 и OUT60416.1), других фил бактерий (AXA36678.1, EHO42689.1, OGD19389.1, OGD22781.1, OGD27033.1, OGU18841.1, PKO20101.1, PMP96649.1, RFT15254.1, RMH30546.1 и RPJ00663.1) и грибов (RKP14217.1).
Множественное выравнивание аминокислотных последовательностей проводили вручную в программе-редакторе BioEdit (http://www.mbio. nc-su.edu/BioEdit/bioedit.html), при этом учитывали результаты попарных выравниваний с помощью программы blastp. Результаты множественного выравнивания (после удаления наиболее вариабельных участков последовательностей) использовали для построения филогенетических деревьев с помощью программ PROTPARS (метод максимальной экономии, Protein Sequence Parsimony method, MP) и NEIGHBOR (метод ближайших соседей, Neighbor-Joining method, NJ) из пакета PHYLIP (http://evolution.gs.washington.edu/phylip.html). Статистическую надежность узлов оценивали с использованием бутстреп-анализа (по 1000 псевдореплик для каждого древа). Программу TreeView Win32 применяли для получения графических изображений деревьев. Для выявления в белках сигнальных последовательностей Sec/SPI-типа применяли программу SignalP 5.0 с настройками для грамотрицательных бактерий (http://www.cbs.dtu.dk/services/SignalP/).
Филогенетическое положение планктомицетов, у которых были выявлены кодирующие хитиназы гены, оценивали с помощью сравнительного анализа нуклеотидных последовательностей гена 16S рРНК и построения филогенетического дерева с использованием программного пакета ARB (http://www.arb-home.de).
РЕЗУЛЬТАТЫ
Экспериментальная проверка способности Planctomicrobium piriforme к росту на хитине. Микроскопический анализ культур двух штаммов P. piriforme, Р3Т и РХ70, развивающихся в течение 30 сут в среде с аморфным хитином, показал их способность к росту на этом полимерном субстрате. Клетки планктомицетов развивались преимущественно на частицах хитина, активно колонизуя их поверхность (рис. 1). Наиболее активный рост с увеличением численности клеток на два порядка был зарегистрирован при инкубации с хитином в качестве источника азота и глюкозой в качестве источника углерода (табл. 1). Во флаконах, содержащих хитин в качестве источника углерода и (NH4)2SO4 в качестве источника азота, численность клеток планктомицетов была в несколько раз ниже, хотя также превышала таковую в контроле более чем на порядок. В контрольных вариантах опыта, не содержащих хитин, наблюдали единичные, слабо флуоресцирующие малоактивные клетки. Таким образом, оба исследованных штамма P. piriforme были способны к использованию хитина в качестве источника азота и, менее активно, в качестве источника углерода.
Рис. 1.
Специфическая детекция клеток Planctomicrobium piriforme P3T (а, б) и РХ70 (в, г) на микрочастицах коллоидного хитина, использованного в инкубационных экспериментах в качестве источника азота. Фазово-контрастная микроскопия (а, в); флуоресцентные микрофотографии гибридизации клеток с Су3-мечеными зондами PLA46 и PLA886 (б, г). Стрелки указывают на микрочастицы хитина. Маркер – 10 мкм.
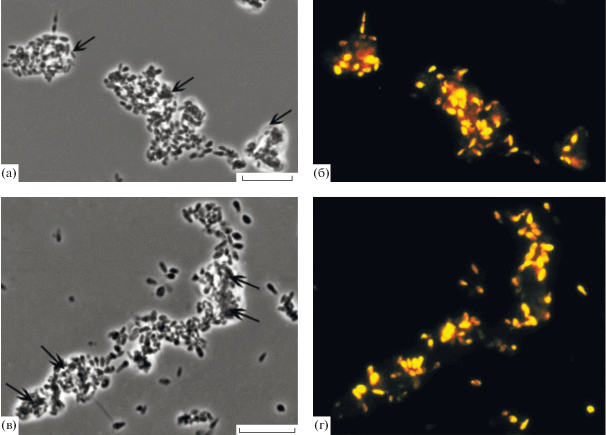
Таблица 1.
Численность клеток Planctomicrobium piriforme после 30 сут инкубации в среде с аморфным хитином в качестве источника углерода или азота
| Численность клеток, N × 108 мл–1 | |||||
|---|---|---|---|---|---|
| Штамм | xитин как источник С | xитин как источник N | |||
| опыт (+хитин, +${\text{NH}}_{4}^{ + }$) |
контроль (–хитин, +${\text{NH}}_{4}^{ + }$) |
опыт (+хитин, +глюкоза) |
контроль (–хитин, +глюкоза) |
||
| Р3T | I* | 38.41 ± 7.69 | 0.70 ± 0.28 | 137.47 ± 9.30 | 1.99 ± 0.79 |
| II | 35.24 ± 4.48 | 0.67 ± 0.24 | 140.54 ± 9.61 | 1.52 ± 0.41 | |
| III | 40.13 ± 8.07 | 0.75 ± 0.30 | 146.08 ± 8.86 | 1.33 ± 0.51 | |
| PX70 | I | 41.79 ± 7.60 | 1.39 ± 0.38 | 152.54 ± 7.96 | 1.71 ± 0.61 |
| II | 39.43 ± 5.04 | 1.08 ± 0.33 | 149.57 ± 6.05 | 1.57 ± 0.62 | |
| III | 42.65 ± 5.19 | 0.64 ± 0.23 | 157.08 ± 6.59 | 1.76 ± 0.56 | |
Активность ферментов цепи деструкции хитина. Измерение активности ферментов, задействованных в разложении хитина, – эндохитиназы, хитoбиозидазы и β-N-ацетилглюкозаминидазы – было проведено на примере штамма Р3Т (табл. 2). В культуральной жидкости всех исследуемых культур активностей этих ферментов выявлено не было. Активности, измеренные с использованием неразрушенных клеток, были эквивалентны таковым в лизате клеток без разделения на фракции. Сравнение активностей ферментов во фракциях супернатанта и осадка, полученных центрифугированием разрушенных клеток, показало, что большая часть активности ассоциирована с фракцией осадка. Таким образом, хитинолитические ферменты исследуемого планктомицета в основном локализованы на поверхности клеток, хотя некий пул ферментов, по всей видимости, может быть локализован и в периплазматическом пространстве. Активность эндохитиназы, первичного звена цепочки разложения хитина, была выше при росте на хитине как источнике азота. Активность β-N-ацетилглюкозаминидазы, напротив, была существенно выше в инкубациях с хитином в качестве источника углерода. Зарегистрированные величины активностей были сопоставимы с таковыми, полученными ранее для хитинолитической ацидобактерии Acidisarcina polymorpha (Belova et al., 2018).
Таблица 2.
Хитинолитическая активность в различных фракциях лизата клеток* Planctomicrobium piriforme Р3Т (ед. кл–1; N × 10–10), выращенных на среде с хитином как источнике азота или углерода. 1 – лизат клеток без разделения на фракции, 2 и 3 – фракции супернатанта и осадка, полученные центрифугированием разрушенных клеток
| Субстрат реакции(фермент) | Хитин как источник N | Хитин как источник С | ||||
|---|---|---|---|---|---|---|
| 1* | 2 | 3 | 1 | 2 | 3 | |
| 4-МУ-N-ацетил-β-D-глюкозамин (β-N-ацетилглюкозаминидаза) | 6.07 ± 0.43 | 0.25 ± 0.04 | 5.31 ± 0.12 | 146.18 ± 7.60 | 21.95 ± 1.11 | 121.56 ± 4.50 |
| 4-МУ-β-D-N,N'-диацетил-хитобиозид (хитобиозидаза) | 1.52 ± 0.10 | 0.61 ± 0.08 | 0.56 ± 0.07 | 20.83 ± 1.69 | 3.75 ± 0.49 | 16.88 ± 1.03 |
| 4-МУ-β-D-N,N',N''-триацетил-хитотриоза (эндохитиназа) | 4.55 ± 0.36 | 2.38 ± 0.15 | 1.97 ± 0.16 | 1.88 ± 0.14 | 0.38 ± 0.07 | 0.75 ± 0.08 |
Филогенетический анализ хитиназ, кодируемых в геномах планктомицетов. Согласно ранее опубликованному филогенетическому древу семейства GH18 гликозил-гидролаз (рис. S2A из работы Ravin et al., 2018), хитиназа Fimbriiglobus ruber (GenPept, OWK46432.1) имеет три близких гомолога из других планктомицетов: Phycisphaerae bacterium ST-NAGAB-D1 (AQT69353.1), Planctomicrobium piriforme (SFI04223.1) и Planctomyces sp. SH-PL14 (AMV17282.1). Поиск по базе данных NCBI показал, что на январь 2019 г. число доступных неидентичных аминокислотных последовательностей планктомицетов из данного подсемейства белков (≥30% идентичности; Наумов, Лившиц, 2001; Наумов, 2004) составляет 14, из которых четыре соответствуют метагеномам (PIE25743.1, REJ73149.1, REJ90827.1 и RLS36953.1). 13 из этих 14 белков (кроме AQT68755.1) имеют близкий размер (303–424 а.о.) и содержат только каталитический домен семейства GH18 гликозил-гидролаз. Все эти белки, а также их ближайшие гомологи из других организмов, были взяты для построения множественного выравнивания и дальнейшего филогенетического анализа. При этом был использован высоко консервативный фрагмент выравнивания, соответствующий участку аминокислотной последовательности хитиназы F. ruber между остатками 35 и 317. В качестве внешней группы служили гипотетические хитиназы планктомицетов семейства Isosphaeraceae, а также их близкие гомологи (представители другого подсемейства семейства GH18, см. раздел “Материалы и методы”).
Построение двумя разными алгоритмами – ближайших соседей (NJ) и максимальной экономии (MP) – филогенетического древа изучаемого пула из 133 белков выявило ряд интересных закономерностей. Большинство из этих белков (суммарно 83 белка) сформировали на MP-древе (рис. 2) восемь кластеров (III–X; на NJ-дереве образовался единый кластер IX/X), каждый из которых содержит представителей только из какой-то одной филы бактерий: Actinobacteria (кластер X), Bacteroidetes (VIII), Chloroflexi (III и IX), Firmicutes (IV) или Proteobacteria (V–VII). Белки этих восьми кластеров обладают тенденцией к образованию общего суперкластера на филогенетическом древе (47.7 и 62.2% бутстреп-поддержки при использовании MP- и NJ-алгоритмов соответственно). Белки кластеров IX (Chloroflexi) и X (Actinobacteria) образуют общий кластер с очень высокой достоверностью (99.6 и 99.8% на MP- и NJ-деревьях соответственно). На деревьях, построенных обоими алгоритмами, также прослеживается тенденция к совместной кластеризации шести кластеров V–X (51.2 и 84.3% на MP- и NJ-деревьях соответственно). Остальные 50 проанализированных белков имеют различное таксономическое происхождение и не формируют таксономически гомогенных кластеров: они распадаются на два крупных кластера I и II, а также отдельно лежащую последовательность – GenPept, RLT26932.1 – из пресноводного бактериального метагенома. Эти 50 белков наиболее представлены бактериальными филами Planctomycetes (14 белков), Bacteroidetes (12), Armatimonadetes (4) и Spirochaetes (4). Анализ N-концевой части аминокислотных последовательностей хитиназ F. ruber и P. piriforme, а также филогенетически близкого им белка из таксономически неохарактеризованного планктомицета SCGC AG-212-M04 (рис. 3а) показал наличие в них сигнальных последовательностей, ответственных за транспорт через цитоплазматическую мембрану.
Рис. 2.
Схема филогенетического древа исследуемого подсемейства семейства GH18 гликозил-гидролаз, построенного методом максимальной экономии. Статистическую надежность узлов древа оценивали с помощью бутстреп-анализа, около каждого узла указано число подтверждающих псевдореплик из 1000. Обозначены десять кластеров (I–X), для каждого указаны бутстреп-поддержка (внутри треугольника), число образующих кластер белков и таксономическая принадлежность организма-хозяина (фила бактерий). Кластеры I и II являются таксономически гетерогенными, их строение указано на рис. 3. В качестве внешней группы служили 17 белков другого подсемейства (бутстреп-поддержка 99.9% на MP- и NJ-древе), их список приведен в разделе “Материалы и методы”. Состав кластеров: кластер III (GBL19468.1, OQY79904.1, PID84970.1, RPI30819.1, RPJ19584.1), IV (ENZ14622.1, OUN05827.1, PWL58212.1, RGC53766.1, ROR23880.1, SFN86453.1, WP_018753820.1, WP_081848672.1, WP_097004517.1), V (ARU26837.1, KZN35416.1, KZN39632.1, KZN53538.1, PCK31609.1, PHI35543.1, PSU57434.1, PUA30549.1, RJG49451.1, RJG50427.1, WP_053062393.1, WP_087468929.1), VI (ABC29857.1, ABC32118.1), VII (AFE08340.1, AKQ63782.1, EPX60666.1, EPX65303.1, OJH39088.1, PTL77347.1, RKG89540.1, RKG98831.1, RKH17116.1, RKH25154.1, RKH31902.1, RKH84143.1, RKI00310.1, RKI09857.1, RKI39630.1, SES99311.1, WP_043323929.1, WP_083560334.1), VIII (ACU59678.1, AHJ96926.1, AOM79703.1, OQP54504.1, OQP66129.1, PJJ54913.1, RAV42254.1, RAV99470.1, RQO66045.1, SCC56941.1, SDO49040.1, SDP60453.1, SEW51767.1, SHE75132.1, SKC46620.1, WP_078014508.1, WP_079685366.1, WP_083252195.1), IX (WP_054532570.1, WP_110514307.1) и X (ADB32881.1, ALG15088.1, BAL91271.1, KOV81728.1, KOX02727.1, PIG50610.1, PKV27384.1, PRY19508.1, RLP93330.1, RQX10606.1, SDP32819.1, SEI95835.1, SES01854.1, SFC17705.1, WP_033431671.1, WP_052386612.1, WP_0867897890.1).
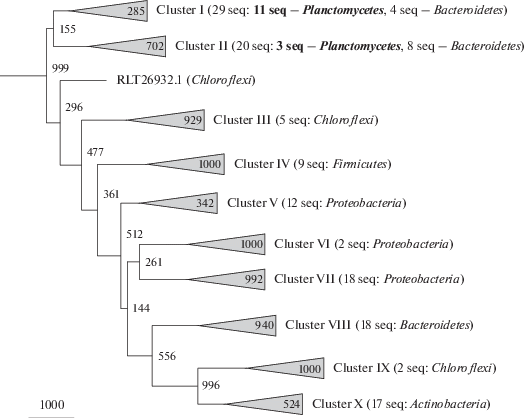
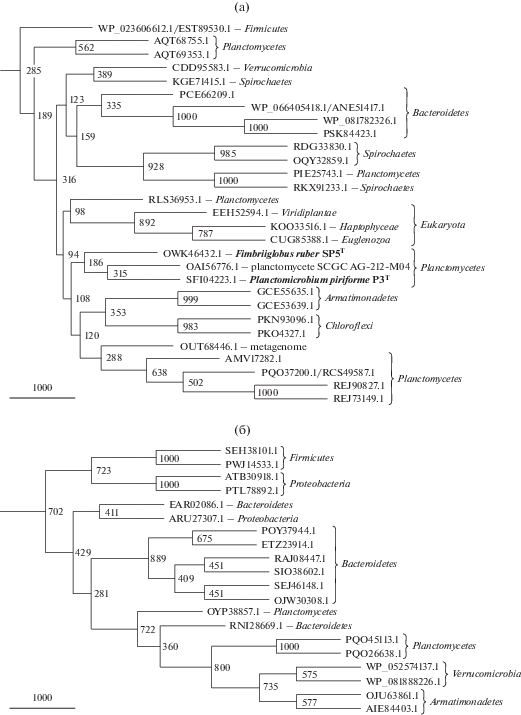
Рис. 3.
Строение кластеров I (а) и II (б) филогенетического древа, схема которого представлена на рис. 2. Условные обозначения те же. Жирным шрифтом указаны планктомицеты с экспериментально подтвержденной хитинолитической активностью.
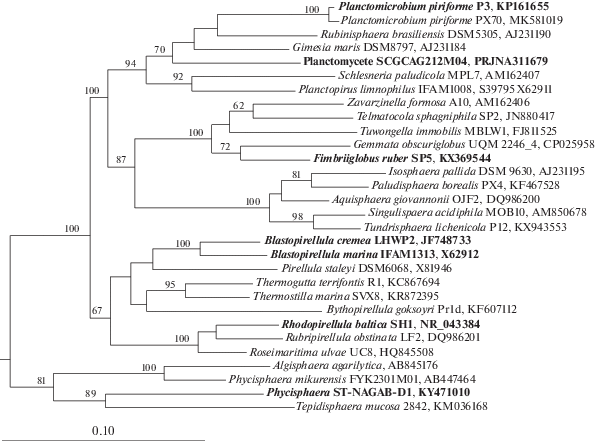
Видовая представленность планктомицетов с хитинолитическим потенциалом. Анализ филогенетического положения планктомицетов, у которых были обнаружены гены хитиназ, не выявил закономерностей в их таксономической принадлежности (рис. 4). Подавляющее большинство этих бактерий относятся к классу Planctomycetia, однако один потенциальный хитинолитик – неохарактеризованный планктомицет ST-NAGAB-D1 – имеется и в классе Phycisphaerae. В пределах Planctomycetia хитиназы выявлены у представителей родов Blastopirellula, Fimbriiglobus, Planctomicrobium, Rhodopirellula, а также неохарактеризованных планктомицетов группы родов Rubinisphaera-Gimesia.
Рис. 4.
Дендрограмма, построенная на основании сравнительного анализа последовательностей генов 16S рРНК планктомицетов (метод NJ). Жирным шрифтом выделены планктомицеты, в геномах которых закодированы хитиназы семейства GH18. Неохарактеризованный планктомицет, штамм SH-PL14, в геноме (GenBank, CP011270.1) которого также закодирована хитиназа, на дендрограмме отсутствует из-за низкого качества соответствующей последовательности гена 16S рРНК. Показаны величины бутстреп-поддержки >60. В качестве внешней группы использованы пять последовательности генов 16S рРНК анаммокс планктомицетов (AF375994, AF375995, AY254883, AY257181, AY254882). Маркер – 0.1 замена на нуклеотидную позицию.
ОБСУЖДЕНИЕ
Как показало настоящее исследование, способность к деструкции хитина среди планктомицетов присуща не только Fimbriiglobus ruber, но и представителям вида Planctomicrobium piriforme. В случае P. piriforme наличие хитинолитической активности удалось подтвердить экспериментально для двух независимо полученных изолятов – типового штамма Р3Т и штамма РХ70, выделенных из географически удаленных друг от друга экосистем. Эти планктомицеты с экспериментально подтвержденной способностью к росту на хитине, по всей видимости, далеко не исчерпывают существующего разнообразия планктомицетов-хитинолитиков. Как следует из филогенетического дерева (рис. 4), представители различных семейств этой филогенетической группы обладают хитинолитическим потенциалом, хотя последний имеется далеко не у всех планктомицетов.
Как в случае F. ruber, так и в случае P. piriforme способность к росту на хитине не удалось обнаружить экспериментально в ходе таксономического описания этих бактерий (Kulichevskaya et al., 2014, 2017). Отчасти это объясняется тем, что в соответствующих тестах была использована кристаллическая форма хитина из панцирей крабов (“Sigma”, США). В аморфной форме этот полимер, по всей видимости, из-за увеличения площади поверхности частиц, доступен более широкому кругу микроорганизмов, так как критическая фаза его разложения связана с адсорбцией клеток или внеклеточных хитиназ на поверхности частиц полимера. Еще одной причиной отрицательных результатов первоначальных тестов было включение хитина в среды в качестве источника углерода, тогда как планктомицеты, как показано в настоящей работе, предпочитают использовать его как источник азота. В случае F. ruber роста на хитине как источнике углерода не удалось зарегистрировать (Ravin et al., 2018). Таким образом, тесты, используемые в обычной практике для выявления хитинолитического потенциала микроорганизмов, мало подходят для планктомицетов, что сдерживает накопление знаний о существующем разнообразии планктомицетов-хитинолитиков. Механизмы деструкции хитина планктомицетами и локализация соответствующих ферментов в клетках этих бактерий требуют отдельных углубленных исследований. Как показало настоящее исследование, ферменты цепи деструкции хитина у планктомицетов ассоциированы с клетками и не экскретируются в среду культивирования.
Анализ топологии филогенетического дерева хитиназ (рис. 2 и 3) позволяет предположить, что в процессе эволюции имели место многочисленные горизонтальные переносы кодирующих их генов между бактериями разных фил. Расположение таксономически гомогенных филогенетических кластеров III–X указывает на их сравнительно позднее возникновение, т.е. они не могли появиться в результате расхождения таксонов. Группа из 50 белков, не принадлежащих к этим кластерам (кластеры I и II) и расположенных близко к корню древа, включает в себя представителей, по крайней мере, восьми фил бактерий, а также некоторых эукариот. Явными лидерами среди них оказались филы Planctomycetes и Bacteroidetes. Это может указывать на то, что гены бактерий одной из них и были предками всех генов белков анализируемого подсемейства. При этом представляется, что более вероятным из них кандидатом являются именно планктомицеты, поскольку они существенно менее представлены в списке полногеномных проектов (Наумов, Дедыш, 2018), которые в настоящее время являются основным источником биологических последовательностей.
Сравнительно широкое распространение генов хитиназ в геномах планктомицетов (рис. 3) и отсутствие закономерностей в их таксономической принадлежности (рис. 4) свидетельствуют, скорее, в пользу того, что они не наследовались вертикально от общего предка всех планктомицетов с потерями во многих филогенетических линиях, а распространялись посредством множественных горизонтальных переносов, которые сопровождались дупликациями и делециями генов. Как следствие, некоторые планктомицеты содержат более одного гена белков исследуемого подсемейства. Две хитиназы имеются у Phycisphaerae bacterium ST-NAGAB-D1 (AQT68755.1 и AQT69353.1), которые, вероятно, появились вследствие дупликации исходного гена. Две почти идентичных (99%) хитиназы из метагеномов (REJ73149.1 и REJ90827.1), вероятно, тоже принадлежали одному организму. Геном Blastopirellula marina кодирует сразу три потенциальных хитиназы: две из них (PQO26638.1 и PQO45113.1; рис. 3б) почти идентичны между собой (98%), явно соответствуя очень свежей дупликации, а третья (PQO37200.1; рис. 3а) – идентична белку из Blastopirellula cremea (RCS49587.1), который не имеет паралогов.
Обсуждая эволюционную историю хитиназ планктомицетов, следует отметить, что происхождение генов хитиназ бактерий разных фил путем горизонтальных переносов от планктомицетов касается только изучаемого подсемейства белков. Так в составе внешней группы на древе хитиназ есть пять белков из планктомицетов (список в разделе “Материалы и методы”). Четыре из них являются представителями семейства Isosphaeraceae и формируют на древе высокостабильный кластер (100% бутстреп-поддержки при использовании MP- и NJ-алгоритмов; рисунок не приводится), пятый белок принадлежит Rhodopirellula sp. (GenPept, OUT60416.1) и кластеризуется вместе с белком грибов (RKP14217.1). По всей видимости, планктомицетные белки из подсемейства, взятого в качестве внешней группы, появились в результате двух эволюционно относительно недавних горизонтальных переносов соответствующих генов в планктомицеты.
Исследованное в настоящей работе подсемейство хитиназ помимо планктомицетов представлено, главным образом, лишь у нескольких фил бактерий. Среди них вполне ожидаемая группа из четырех наиболее изученных таксонов: Actinobacteria, Bacteroidetes, Firmicutes и Proteobacteria. Менее прогнозируемым оказалось обнаружение многочисленных представителей филы Chloroflexi, которые наиболее равномерно распределены по филогенетическому древу (рис. 2 и 3). Топология древа позволяет утверждать, что произошло, по меньшей мере, четыре горизонтальных переноса генов исследуемого подсемейства белков в организмы этой филы. Наличие хитиназной активности у клеток некультивируемых Chloroflexi в активированном иле было продемонстрировано с использованием флуоресцентных субстратов (Kragelund et al., 2007), однако способность к гидролизу хитина у таксономически охарактеризованных представителей филы была отмечена пока лишь для рода Thermosporothrix (Yabe et al., 2010, 2016). По всей видимости, Chloroflexi – это еще одна группа бактерий, помимо планктомицетов, хитинолитический потенциал которых остается не раскрытым и заслуживает дальнейших исследований.
Выявление способности Planctomicrobium piriforme к деструкции хитина позволяет внести коррективы в ранее опубликованное описание этого вида:
Уточненное описание Planctomicrobium piriforme Kulichevskaya et al. 2015. Описание вида Planctomicrobium piriforme аналогично таковому в работе Куличевской и соавт. (Kulichevskaya et al., 2015), с добавлением следующих признаков: представители вида обладают хитиназной активностью и способны использовать хитин в качестве источника азота и углерода.
СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ
Настоящая статья не содержит результатов исследований, в которых в качестве объектов использовались люди или животные.
КОНФЛИКТ ИНТЕРЕСОВ
Авторы заявляют отсутствие конфликта интересов.
Вклад авторов: идея и схема исследования предложены С.Н. Дедыш. Эксперименты по культивированию и учету клеток планктомицетов проведены И.С. Куличевской. Активности ферментов цепи деструкции хитина определены А.Л. Ракитиным. Филогенетические анализы хитиназ и генов 16S рРНК планктомицетов проведены Д.Г. Наумовым и А.А. Ивановой соответственно. Текст статьи написан С.Н. Дедыш и Д.Г. Наумовым. Все авторы участвовали в обсуждении результатов.
Список литературы
Куличевская И.С., Панкратов Т.А., Дедыш С.Н. Выявление представителей Planctomycetes в сфагновых болотах с использованием молекулярных и культуральных подходов // Микробиология. 2006. Т. 75. № 3. С. 389–396.
Kulichevskaya I.S., Pankratov T.A., Dedysh S.N. Detection of representatives of the Planctomycetes in Sphagnum peat bogs by molecular and cultivation approaches // Microbiology (Mocow). 2006. V. 75. P. 329–335.
Наумов Д.Г. Иерархическая классификация гликозил-гидролаз // Биохимия. 2011. Т. 76. № 6. С. 764–780.
Naumoff D.G. Hierarchical classification of glycoside hydrolases // Biochemistry (Moscow). 2011. V. 76. P. 622–635.
Наумов Д.Г. Филогенетический анализ α-галактозидаз семейства GH27 // Мол. биология. 2004. Т. 38. № 3. С. 463–476.
Naumoff D.G. Phylogenetic analysis of α-galactosidases of the GH27 family // Mol. Biol. (Moscow). 2004. V. 38. P. 388–399.
Наумов Д.Г., Дедыш С.Н. Малоизученные группы бактерий – источник новых ферментов: β-галактозидазы из планктомицетов и веррукомикробий // Микробиология. 2018. Т. 87. № 6. С. 695–705.
Naumoff D.G., Dedysh S.N. Bacteria from poorly studied phyla as a potential source of new enzymes: β-galactosidases from Planctomycetes and Verrucomicrobia // Microbiology (Moscow). 2018. V. 87. P. 796–805.
Наумов Д.Г., Лившиц В.А. Молекулярная структура локуса утилизации сахарозы Lactobacillus plantarum: сравнение с Pediococcus pentosaceus // Мол. биология. 2001. Т. 35. № 1. С. 19–27.
Naumoff D.G., Livshits V.A. Molecular structure of the Lactobacillus plantarum sucrose utilization locus: comparison with Pediococcus pentosaceus // Mol. Biol. (Moscow). 2001. V. 35. P. 15–22.
Belova S.E., Ravin N.V., Pankratov T.A., Rakitin A.L., Ivanova A.A., Beletsky A.V., Mardanov A.V., Sinninghe Damsté J.S., Dedysh S.N. Hydrolytic capabilities as a key to environmental success: chitinolytic and cellulolytic Acidobacteria from acidic Sub-arctic soils and boreal peatlands // Front. Microbiol. 2018. V. 9. Article 2775.
Boedeker C., Schüler M., Reintjes G., Jeske O., van Teeseling M.C.F., Jogler M., Rast P., Borchert D., Devos D.P., Kucklick M., Schaffer M., Kolter R., van Niftrik L., Engelmann S., Amann R., Rohde M., Engelhardt H., Jogler C. Determining the bacterial cell biology of Planctomycetes // Nat. Commun. 2017. V. 8. Article 14853.
Chernin L.S., Winson M.K., Thompson J.M., Haran S., Bycroft B.W., Chet I., Williams P., Stewart A.B. Chitinolytic activity in Chromobacterium violaceum: substrate analysis and regulation by quorum sensing // J. Bacteriol. 1998. V. 180. P. 4435–4441.
Dedysh S.N., Derakshani M., Liesack W. Detection and enumeration of methanotrophs in acidic Sphagnum peat by 16S rRNA fluorescence in situ hybridization, including the use of newly developed oligonucleotide probes for Methylocella palustris // Appl. Environ. Microbiol. 2001. V. 67. P. 4850–4857.
Dedysh S.N., Ivanova A.A. Planctomycetes in boreal and subarctic wetlands: diversity patterns and potential ecological functions // FEMS Microbiol. Ecol. 2019. V. 95. Article fiy227. https://doi.org/10.1093/femsec/fiy227
Ivanova A.A., Naumoff D.G., Miroshnikov K.K., Liesack W., Dedysh S.N. Comparative genomics of four Isosphaeraceae planctomycetes: a common pool of plasmids and glycoside hydrolase genes shared by Paludisphaera borealis PX4T, Isosphaera pallida IS1BT, Singulisphaera acidiphila DSM 18658T, and strain SH-PL62 // Front. Microbiol. 2017. V. 8. Article 412.
Ivanova A.A., Wegner C.E., Kim Y., Lesack W., Dedysh S.N. Metatranscriptomics reveals the hydrolytic potential of peat-inhabiting Planctomycetes // Antonie van Leeuwenhoek. 2018. V. 111. P. 801–809.
Huang C.-J., Wang T.-K., Chung S.-C., Chen C.-Y. Identification of an antifungal chitinase from a potential biocontrol agent, Bacillus cereus 28-9 // J. Biochem. Mol. Biol. 2005. V. 38. P. 82–88.
Kragelund C., Levantesi C., Borger A., Thelen K., Eikelboom D., Tandoi V., Kong Y., van der Waarde J., Krooneman J., Rossetti S., Thomsen T.R., Nielsen P.H. Identity, abundance and ecophysiology of filamentous Chloroflexi species present in activated sludge treatment plants // FEMS Microbiol. Ecol. 2007. V. 59. P. 671–682.
Kulichevskaya I.S., Ivanova A.A., Detkova E.N., Rijpstra I.C., Sinninghe Damste J.S., Dedysh S.N. Planctomicrobium piriforme gen. nov., sp. nov., a stalked planctomycete from a littoral wetland of a boreal lake // Int. J. Syst. Evol. Microbiol. 2015. V. 69. P. 1659–1665.
Lombard V., Golaconda Ramulu H., Drula E., Coutinho P.M., Henrissat B. The carbohydrate-active enzymes database (CAZy) in 2013 // Nucl. Acids Res. 2014. V. 42 (Database issue). P. D490–D495.
McDonald A.G., Boyce S., Tipton K.F. ExplorEnz: the primary source of the IUBMB enzyme list // Nucl. Acids Res. 2009. V. 37 (Database issue). P. D593–D597.
Neef A., Amann R., Schlesner H., Schleifer K.H. Monitoring a widespread bacterial group: In situ detection of planctomycetes with 16S rRNA-targeted probes // Microbiology (SGM). 1998. V. 144. P. 3257–3266.
Nguyen S.T.C., Freund H.L., Kasanjian J., Berlemont R. Function, distribution, and annotation of characterized cellulases, xylanases, and chitinases from CAZy // Appl. Microbiol. Biotechnol. 2018. V. 102. P. 1629–1637.
Oyeleye A., Normi Y.M. Chitinase: diversity, limitations, and trends in engineering for suitable applications // Biosci. Rep. 2018. V. 38. Article BSR2018032300.
Ravin N.V., Rakitin A.L., Ivanova A.A., Beletsky A.V., Kulichevskaya I.S., Mardanov A.V., Dedysh S.N. Genome analysis of Fimbriiglobus ruber SP5T, a planctomycete with confirmed chitinolytic capability // Appl. Environ. Microbiol. 2018. V. 84. Article e02645-17.
Reintjes G., Arnosti C., Fuchs B.M., Amann R. An alternative polysaccharide uptake mechanism of marine bacteria // ISME J. 2017. V. 11. P. 1640–1650.
Sorokin D.Y., Gumerov V.M., Rakitin A.L., Beletsky A.V., Damsté J.S.S., Muyzer G., Mardanov A.V., Ravin N.V. Genome analysis of Chitinivibrio alkaliphilus gen. nov., sp. nov., a novel extremely haloalkaliphilic anaerobic chitinolytic bacterium from the candidate phylum Termite Group 3 // Environ. Microbiol. 2014. V. 16. P. 1549–1565.
Staley J.T., Fuerst J.A., Giovannoni S., Schlesner H. The Order Planctomycetales and the Genera Planctomyces, Pirellula, Gemmata, and Isosphaera // The Prokaryotes: A Handbook on the Biology of Bacteria: Ecophysiology, Isolation, Identification, Applications / Eds. Balows A., Trüper H.G., Dworkin M., Harder W., Schleifer K.-H. New York, New York, NY: Springer, 1992. P. 3710–3731.
Tamadoni Jahromi S., Barzkar N. Marine bacterial chitinase as sources of energy, eco-friendly agent, and industrial biocatalyst // Int. J. Biol. Macromol. 2018. V. 120. Pt. B. P. 2147–2154.
Wiegand S., Jogler M., Jogler C. On the maverick Planctomycetes // FEMS Microbiol. Rev. 2018. V. 42. P. 739–760.
Yabe S., Aiba Y., Sakai Y., Hazaka M., Yokota A. Thermosporothrix hazakensis gen. nov., sp. nov., isolated from compost, description of Thermosporothrichaceae fam. nov. within the class Ktedonobacteria Cavaletti et al. 2007 and emended description of the class Ktedonobacteria // Int. J. Syst. Evol. Microbiol. 2010. V. 60. P. 1794–1801.
Yabe S., Sakai Y., Yokota A. Thermosporothrix narukonensis sp. nov., belonging to the class Ktedonobacteria, isolated from leaves on geothermal soil, and emended description of the genus Thermosporothrix // Int. J. Syst. Evol. Microbiol. 2016. V. 66. P. 2152–2157.
Дополнительные материалы отсутствуют.


