Микробиология, 2019, T. 88, № 4, стр. 458-469
Характеристика бактериофага Pectobacterium carotovorum subsp. Carotovorum pp16, перспективного для биоконтроля мягкой гнили картофеля
М. В. Воронина a, Е. Н. Бугаева a, Д. М. Васильев a, А. П. Кабанова a, b, А. П. Баранник b, М. М. Шнейдер b, Е. Е. Куликов c, А. А. Корженков d, e, С. В. Тощаков c, d, А. Н. Игнатов a, К. А. Мирошников a, b, *
a Исследовательский центр “ФитоИнженерия”
141880 Московская обл., с. Рогачево, Россия
b Институт биоорганической химии им. М.М. Шемякина и Ю.А. Овчинникова РАН
117997 Москва, Россия
c Институт микробиологии им. С.Н. Виноградского, Федеральный исследовательский центр
“Фундаментальные основы биотехнологии” РАН
119071 Москва, Россия
d Балтийский федеральный университет им. И. Канта
236041 Калининград, Россия
e Национальный исследовательский центр “Курчатовский институт”
123182 Москва, Россия
* E-mail: kmi@ibch.ru
Поступила в редакцию 06.02.2019
После доработки 04.03.2019
Принята к публикации 29.03.2019
Аннотация
Бактериофаги пектобактерий, вызывающих заболевания картофеля (черную ножку и мягкую гниль), – сравнительно малоизученная группа фагов, которые имеют перспективы для технологических приложений в целях предотвращения потерь товарного и семенного картофеля. В настоящей статье представлена характеристика нового подовируса PP16, инфицирующего широкий спектр штаммов Pectobacterium carotovorum. По строению своего генома бактериофаг РР16 относится к отдельной филогенетической ветви рода Phimunavirus подсемейства Autographivirinae. Бактериофаг РР16 эффективно ингибирует развитие инфекции in vitro и in planta. Проведенный полевой эксперимент по обработке бактериофагом РР16 посадочного материала картофеля показал значительное повышение всхожести растений.
Грамотрицательные бактерии порядка Enterobacteriales, недавно выделенные в отдельное семейство Pectobacteriaceae (Adeolu et al., 2016), распространены в почвенной микробиоте и относятся к фитопатогенам, входящим в десятку наносящих наибольший урон в мировом сельском хозяйстве (Mansfield et al. 2012). Pectobacterium carotovorum subsp. carotovorum (Pcc) – наиболее распространенный в глобальном масштабе представитель этого семейства, обладающий наиболее широким спектром поражаемых видов растений (Pérombelon, 2002). На картофеле (Solanum tuberosum) Pcc вызывает черную ножку вегетирующих растений и мягкую (мокрую) гниль клубней во время хранения урожая и семенного материала. В отличие от похожих по вызываемым болезням фитопатогенов из родов Pectobacterium и Dickeya, обладающих низким внутривидовым разнообразием, бактерии Рсс высоко вариабельны (Pérombelon, 2002). По мере получения детальной информации о вирулентности, спектре растительных хозяев, температурных оптимумах патогенеза и, в особенности, деталях строения генома, некоторые группы штаммов, ранее относившихся к Рсс, были определены в новые виды и подвиды – P. carotovorum subsp. brasilense (Gardan et al., 2003), P. carotovorum gsp. maceratum (Shirshikov et al., 2018), Р. peruviense (Waleron et al., 2018), Р. polaris (Dees et al., 2017), P. punjabense (Sarfraz et al., 2018). В связи с ограничениями использования антибиотиков в сельском хозяйстве разработка альтернативных методов борьбы с бактериозами растений представляется актуальной. В частности, рассматриваются стратегии контроля фитопатогенных бактерий с помощью специфических бактериофагов (Balogh et al., 2010).
Целью работы было провести поиск высоко активных литических бактериофагов, инфекционных по отношению к патогенным штаммам Рсс, и дать исчерпывающее описание их свойств.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Объектом исследования был штамм Pectobacterium carotovorum F002 – хозяин изучаемого бактериофага.
Культуральные методы. Для культивирования штаммов бактерий родов Pectobacterium и Dickeya использовали стандартную среду LB и агаризованную среду LB (концентрация агара 1.5%). В качестве верхнего слоя при титровании фагов использовали ту же среду с 0.6% агара (Sambrook et al., 1989). Инкубацию планктонных и поверхностных культур проводили при 26–28°С.
Для наращивания фага в жидкой культуре 50 мл бульона LB инокулировали 1% (об.) ночной культуры Pcc штамма F002 и растили до середины логарифмической фазы (оптическая плотность при 600 нм от 0.5 до 0.6) при интенсивном перемешивании. Выросшую культуру инокулировали единичной бляшкой фага и инкубировали до проявления признаков лизиса. Затем добавляли 0.5% (об.) хлороформа, энергично встряхивали и через 1 ч лизаты центрифугировали при 10 000 g в течение 10 мин для удаления бактериальных фрагментов.
Очистка бактериофага РР16. После обработки хлороформом лизат фильтровали через мембранный фильтр с порами 0.22 мкм (“Millipore”, США), обрабатывали ДНКазой I (0.5 мг/мл, 60 мин). Фаг очищали ультрацентрифугированием (66 000 g, 120 мин, 4°C, ротор Beckman SW28) в ступенчатом градиенте плотности CsCl 0.5–1.7 г/мл. Суспензию фага РР16 диализовали против фагового буфера (10 мM Tris-HCl, pH 7.4, 10 мM MgSO4). Препарат очищенного фага хранили при 4°C.
Электронная микроскопия. Суспензию очищенного бактериофага наносили на сетки и контрастировали 1% водным раствором уранилацетата (Ackermann, 2009). Изображения получали с помощью электронного микроскопа JEOL JEM-CX100 (“JEOL”, Япония) с ускоряющим напряжением 100 кВ.
Определение инфекционного спектра бактериофага. Для определения способности фага РР16 инфицировать клетки бактерии-хозяина, по 5 мкл серийных разведений от 102 до 108 БОЕ/мл очищенного бактериофага наносили на чашку Петри с двуслойным агаром, инокулированным соответствующим штаммом. Затем чашки инкубировали при 37°С в течение ночи. Результаты регистрировали следующим образом: (–) – отсутствие роста фагов; (+) – зона ингибирования роста бактерий, разделяющаяся на отдельные зоны-бляшки при разведении препарата фага.
Определение параметров инфекции. Для оценки скорости адсорбции фаговых частиц штамм P. carotovorum F002 культивировали в жидкой среде LB до OD600 ~ 0.4, инфицировали фагом РР16 с множественностью инфекции (MOI) 0.1. Далее каждые 3 мин с начала инфицирования отбирали аликвоты 100 мкл и переносили в 800 мкл среды LB/50 мкл хлороформа. После лизиса бактерий смесь центрифугировали (10 000 g) и титровали для определения количества свободных частиц фага. Одностадийные кривые роста популяции фага определяли в соответствии с рекомендациями (Clokie, Kropinski, 2009). Стабильность фага оценивали путем инкубации суспензии фага с концентрацией 107 БОЕ/мл при различных температурах или в различных буферных растворах (20 мM Tris-HCl/20 мM цитрат Na/20 мM фосфат Na) доведенных до рН 4–9 с помощью NaOH).
Генотипирование фагочувствительных штаммов. Для определения генетического родства бактерий, инфицируемых фагом РР16, проводили фингерпринтинг геномов методом BOX-PCR (Martin et al., 1992). Геномную ДНК бактерий выделяли экстракцией фенолом (Sambrook et al., 1989), оценивали ее концентрацию по поглощению при 260 нм (NanoPhotometer NP60, “Implen”, Германия) и проводили ПЦР. Реакционная смесь объемом 25 мкл содержала: 67 мМ Трис-HCl (pH 8.8), 16.6 мМ (NH4)2SO4, 0.01% Твин-20, 2.5 мМ MgCl2, dNTP (“GE Healthcare”, США) (0.4 мM каждый), 0.6 мкМ праймер BOXA1R (5'-CTACGGCAAGGCGACGCTGACG-3'), 80 нг геномной ДНК, 1.0 U Taq-полимеразы (“Евроген”, Россия). Амплификацию осуществляли по схеме: начальная денатурация ДНК – 5 мин при 95°С, следующие 40 циклов: 95°С, 1 мин; 52°С, 1 мин; 72°С, 2 мин; завершающий цикл – 72°С, 5 мин. Амплифицированные фрагменты ДНК разделяли электрофорезом в 1.5% агарозном геле (80 В, буфер 1× TAE). В качестве маркера молекулярных масс был использован HyperLadder 1 kb (“Bioline”, Великобритания). Фингерпринты документировали и сравнивали с помощью системы Quantum-ST5 (“Vilber Lourmat”, Франция).
Выделение ДНК и секвенирование генома бактериофага РР16. Геномную ДНК бактериофага выделяли экстракцией фенолом (Clokie, Kropinski 2009) и фрагментировали ультразвуком. Фрагментные библиотеки конструировали с помощью набора Nebnext Ultra DNA library prep kit (“New England Biolabs”, США) и секвенировали на платформе Illumina MiSeq™ (США) с парными прочтениями длиной 150 п.н. Данные секвенирования фильтровали программой CLC Genomics Workbench 8.5 (“Qiagen”, Германия), перекрывающиеся прочтения соединяли программой SeqPrep tool (https://github.com/jstjohn/SeqPrep). Сборку генома проводили программой Spades 3.8.0 (Bankevich et al., 2012). Физические концы генома определяли секвенированием по Сэнгеру с помощью секвенатора ABI Prism 3130 (“Applied Biosystems”, США).
Аннотация генома и сравнительная геномика. Поиск открытых рамок считывания выполняли при помощи программы GeneMarkS (Besemer el al., 2001). Функции открытых рамок считывания (ORF) предсказывали путем выравнивания против последовательностей из базы данных NCBI nr программой PSI-BLAST (Altschul et al., 1997), поиск консервативных доменов выполняли программой HMMER (Finn el al., 2011). Поиск промоторных последовательностей в геноме осуществляли с помощью программы PHIRE (Lavigne et al., 2004). Филогенетическая классификация бактериофага была выполнена с помощью сервера VICTOR (Meier-Kolthoff, Göker, 2017). Значение средней нуклеотидной идентичности определяли с помощью скрипта ani.rb (https://github.com/lmrodriguezr/enveomics).
Депонирование нуклеотидных последовательностей в ГенБанк. Аннотированная полногеномная последовательность бактериофага PP16 (vM_PccP_PP16) депонирована в NCBI GenBank под номером KX278418.1.
Тестирование профилактического и лечебного воздействия бактериофага РР16 in planta. Эффект подавления фагом РР16 размягчения ткани картофеля, вызванного P. carotovorum subsp. carotovorum F002, выявляли опытами на срезах клубней (Czajkowski et al., 2015). Клубни картофеля сорта Айл оф Джура (“Cygnet Potato Breeders LTD”, Великобритания и ООО “Новый картофель”, Россия) поверхностно стерилизовали 70% этанолом в течение 3–5 мин, промывали водой и высушивали фильтровальной бумагой. Стерильным ножом клубни нарезали на диски толщиной 0.7 см. В каждом диске вырезали три углубления (5 × 5 × 5 мм) и вносили в них по 50 мкл суспензии, содержащей 106 КОЕ/мл Рсс F002 и 106 БОЕ/мл РР16 (MOI = 1). В качестве отрицательного контроля использовали 50 мкл воды, а в качестве положительного контроля 50 мкл суспензии, содержащей только Рсс F002 в той же концентрации. Защитный эффект фагов измеряли после инкубации в течение 72 ч при 26°C и 100% относительной влажности, оценивая диаметр мацерированной ткани в месте инокуляции. Опыт закладывали в трех повторностях на дисках из двух разных клубней.
Полевой эксперимент по обработке бактериофагом семенного материала. В исследовании использовали среднеустойчивый к бактериозам сорт картофеля Айл оф Джура, свободный от вирусной и грибной инфекций. Обработку семенных клубней картофеля проводили непосредственно перед посадкой путем распыления на клубни водного раствора, содержащего: фунгицид Эместо Квантум (“Bayer”, Германия) 40 мл/л; микроудобрение Изагри (Россия) 200 мл/л, лизат фага РР16, профильтрованного через 0.22 мкм фильтр, в конечной концентрации 108 БОЕ/мл. В контроле препарат бактериофага не добавляли. В предварительных экспериментах было установлено отсутствие негативного воздействия фунгицида и микроудобрения в рабочей концентрации на жизнеспособность и вирулентность бактериофага.
Эффективность действия препарата бактериофагов определяли по степени снижения развития бактериоза картофеля при оценке популяций патогенных бактерий на клубнях микробиологическими, молекулярными методами, а также по влиянию на всхожесть клубней и урожайность растений в фазе технической спелости. Место проведения эксперимента: с. Рогачево Дмитровского р-на Московской обл., Россия. Географические координаты – 56°26′00″ с.ш., 37°09′20″ в.д. Почва опытного участка дерново-подзолистая, легкосуглинистая, содержание гумуса 2.3%, подвижного фосфора 220 мг/кг, обменного калия 180 мг/кг; рН 5.7. Количество бактерий на 1 г почвы – 3.0 × 1010 КОЕ/г; параметры определяли согласно рекомендациям (Ягодин и соавт., 2002). В почве присутствует фоновое количество пектолитических бактерий (Humphris et al., 2015).
Посадка опыта произведена 27 мая, уборка – 15 сентября 2017 г. Делянки двухрядковые, количество высаженных клубней на делянке – 66 шт., повторность – двукратная. До посадки на поле внесены минеральные удобрения из расчета: азот : : фосфор : калий (90 : 100 : 200) кг действующего вещества/га. Ширина междурядий – 90 см, расстояние между клубнями в ряду – 27 см. В период вегетации проведено 7 обработок фунгицидами, микроудобрениями и инсектицидами. Количественную оценку содержания Рectobacterium sp. (без дифференциации на виды) проводили по стандартному протоколу ПЦР в реальном времени (RT-PCR) (Humphris et al., 2015).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
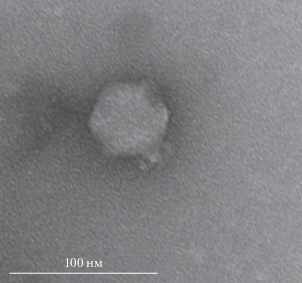
Свойства бактериофага РР16. Бактериофаг РР16 был выделен нами в 2012 г. из сточных вод в Московской области с использованием высоковирулентного штамма Рсс F002 (PB69), NCBI номер NZ_PDVY00000000.1 (Shirshikov et al., 2018) в качестве бактериального хозяина. Электронная микроскопия негативного контрастирования выявила морфологию вирусных частиц, характерную для семейства Podoviridae, с икосаэдрическим капсидом диаметром ~60 нм и коротким хвостом длиной ~15 нм (рис. 1). Таким образом, согласно универсальной номенклатуре вирусов бактерий (Lavigne et al., 2008), бактериофаг РР16 следует именовать vB_PccP_PP16. Обращает на себя внимание наличие выступов вокруг хвостового отростка, представляющих собой адсорбционный аппарат бактериофага (рис. 1). Такие выступы характерны для фагов родов KP34virus (Eriksson et al., 2015) и Phimunavirus (Buttimer et al., 2018) подсемейства Autographivirinae.
Рис. 1.
Электронно-микроскопическое изображение фага РР16, негативно контрастированного 1% уранилацетатом. Увеличение ×150 000. Шкала 100 нм.
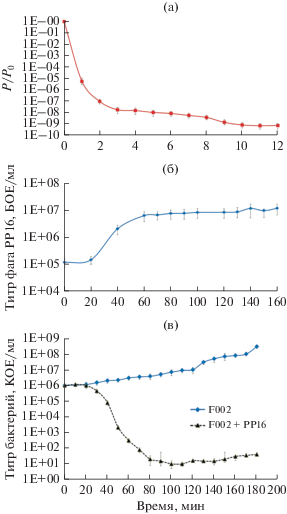
Бактериофаг РР16 эффективно инфицирует штамм Рсс F002, практически полностью адсорбируясь на клетках бактерий в течение 5 мин (рис. 2а) при 26°С. Фаг лизирует клетки полностью в течение 1 ч, при этом происходит полное просветление культуры клеток и формирование потомства фага (порядка 60 частиц на клетку) (рис. 2б). В течение 3 ч in vitro не происходит заметного роста фагоустойчивых мутантов клеток.
Рис. 2.
Биологические свойства фага РР16 при инфекции пектобактерий штамма Pcc F002. Эксперименты проведены в жидкой среде LB при 26°С. Множественность инфекции 0.1. а – адсорбция фага; б – одностадийная кривая роста; в – ингибирование популяции бактерий: 1 – контроль (без внесения фага); 2 – внесение фага.
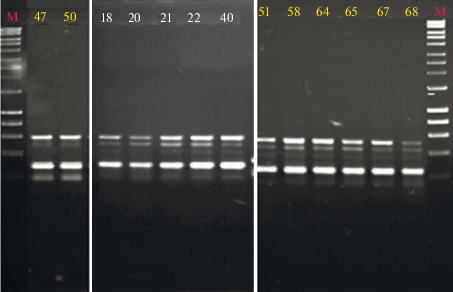
Инфекционный спектр бактериофага РР16. На газоне бактерий Рсс штамма F002 и других чувствительных бактерий фаг РР16 образует прозрачные негативные колонии размером 1–2 мм, окруженные мутным ореолом. Наличие подобного ореола обычно указывает на присутствие в частице бактериофага ферментов, осуществляющих деградацию липополисахаридов клеточной стенки бактерий. Бактериофаг проявляет инфекционность по отношению к 20 из 42 (47%) протестированных штаммов пектолитических бактерий (табл. 1). Инфекционный диапазон включает в себя бактерии P. carotovorum subsp. carotovorum (18 из 23, или 78%) и 2 из 3 штаммов P. carotovorum subsp. brasiliense (Pcb). Пектолитические патогены P. atrosepticum (Pat), P. parmentieri (Ppa), Dickeya solani (Dso) и D. dianthicola (Ddi) устойчивы к действию фага РР16. Генетическое разнообразие штаммов, чувствительных к действию бактериофага РР16, оценивали сравнением ДНК-фингерпринтов, полученных методом BOX-PCR (Martin et al., 1992). Этот метод амплификации фрагментов генома бактерий между консервативными межгенными регуляторными элементами BОХ‑-А, позволяет оценить филогенетическое родство штаммов разнообразных таксономических групп бактерий. Профили 18 чувствительных к фагу РР16 штаммов Pcc показывает принадлежность 16 из них к трем группам (рис. 3), что позволяет сделать вывод о распространенности в Центральной России ограниченного числа генотипов Рсс. В каждом профиле BOX-фингерпринта имеются мажорные полосы размером около 270 и 720 п.н., отличия между группами наблюдаются в количестве и интенсивности минорных полос. Только два штамма – F002 и близкородственный ему по геномной нуклеотидной последовательности F003 – имеют паттерны BOX-фингерпринтов, отличные от этих трех групп. Интересно, что все инфицируемые фагом РР16 представители таксона Рсс относятся к группе штаммов, характерных для Европейской части России и по сходству геномов отнесенных к предполагаемому новому геномовиду Pectobacterium carotovorum gsp. maceratum (Shirshikov et al., 2018). Коллекционные штаммы, считающиеся типовыми для Рсс, к бактериофагу РР16 устойчивы (табл. 1). Несмотря на довольно высокое генетическое родство штаммов, чувствительных к фагу РР16, его инфекционный спектр можно считать широким.
Таблица 1.
Инфекционный спектр бактериофага РР16
| № | Штамм | Другое обозначение | Год | Место | Вид | Определение таксона | BOX группа | РР16 |
|---|---|---|---|---|---|---|---|---|
| 1 | F002 | PB69 | 2012 | Московская обл. | Pcc* | NZ_PDVY00000000.1 | ND | + |
| 2 | F003 | PB70 | 2012 | Московская обл. | Pcc* | NZ_PDVZ00000000.1 | ND | + |
| 3 | F018 | 1947 | Московская обл. | Pcc* | NZ_PDVV00000000.1 | PccI | + | |
| 4 | F020 | 2003 | Московская обл. | Pcc* | 16S | PccI | + | |
| 5 | F021 | 2005 | Московская обл. | Pcc* | 16S | PccI | + | |
| 6 | F022 | 1993 | Московская обл. | Pcc* | 16S | PccI | + | |
| 7 | F047 | 1991 | Ленинградская обл. | Pcc* | 16S | PccII | + | |
| 8 | F050 | 1993 | Ленинградская обл | Pcc* | 16S | PccII | + | |
| 9 | F051 | 2005 | Московская обл. | Pcc* | 16S | PccIII | + | |
| 10 | F058 | 2003 | Московская обл. | Pcc* | 16S | PccIII | + | |
| 11 | F064 | 2005 | Московская обл. | Pcc* | 16S | PccIII | + | |
| 12 | F065 | 2003 | Московская обл. | Pcc* | 16S | PccIII | + | |
| 13 | F067 | 2004 | Московская обл. | Pcc* | 16S | PccIII | + | |
| 14 | F068 | 2005 | Московская обл. | Pcc* | 16S | PccIII | + | |
| 15 | F099 | 2005 | Московская обл. | Pcc* | 16S | PccIII | + | |
| 16 | F100 | 1993 | Тверская обл. | Pcc* | 16S, PCR | PccI | + | |
| 17 | F105 | 2005 | Московская обл. | Pcc* | 16S | PccIII | + | |
| 18 | F108 | 2003 | Московская обл. | Pcc* | 16S | PccI | + | |
| 19 | F118 | 2005 | Московская обл. | Pcc* | 16S, PCR | PccI | + | |
| 20 | F132 | 2005 | Московская обл. | Pcc* | 16S, PCR | PccI | + | |
| 21 | F135 | 1995 | Владимирская обл. | Pcc* | NZ_PDVX00000000.1 | PccI | + | |
| 22 | F008 | 1923 | ВКПМ | Pcc | 16S, PCR | ND | – | |
| 23 | F160 | NCPPB312 | ND | ВКПМ | Pcc | NZ_JQHJ00000000.1 | PccIV | – |
| 24 | F140 | 2012 | Московская обл. | Pcc | 16S, PCR | PccV | – | |
| 25 | F150 | 2014 | Московская обл. | Pcc | 16S, PCR | PccV | – | |
| 26 | F131 | 1998 | Калужская обл. | Pcc* | NZ_PDVW00000000.1 | PccVI | ||
| 27 | F126 | 2012 | Самарская обл. | Pcb | 16S, PCR | PcbI | + | |
| 28 | F128 | 2012 | Самарская обл. | Pcb | 16S, PCR | PcbI | + | |
| 29 | F152 | PB29 | 2014 | Московская обл. | Pcb | 16S, PCR | PcbII | – |
| 30 | F012 | 2010 | Воронежская обл. | Dso | NZ_PGOJ00000.1 | Dso | – | |
| 31 | F043 | 2010 | Московская обл. | Dso | 16S, PCR | Dso | – | |
| 32 | F082 | 1980 | Московская обл. | Dso | PCR | Dso | – | |
| 33 | F097 | 2011 | Калужская обл.. | Dso | PCR | Dso | – | |
| 34 | F119 | 2011 | Тверская обл. | Dso | PCR | Dso | – | |
| 35 | F156 | 2014 | Московская обл. | Dso | 16S, PCR | Dso | – | |
| 36 | F069 | 2011 | Московская обл. | Ddi | 16S, PCR | DdiI | – | |
| 37 | F077 | 2002 | Тюменская обл. | Ddi | 16S, PCR | DdiI | – | |
| 38 | F085 | 1979 | Московская обл. | Ddi | 16S, PCR | DdiII | – | |
| 39 | F090 | 1995 | Калужская обл. | Ddi | 16S, PCR | DdiII | – | |
| 40 | F162 | SCRI1043 | ND | Шотландия | Pat | NC_004547 | PatI | – |
| 41 | F163 | 21A | 1990 | Беларусь | Pat | NZ_CP009125 | PatII | – |
| 42 | F004 | PB72 | 2012 | Московская обл. | Pat | NZ_PDDK00000000.1 | PatII | – |
| 43 | F041 | 2011 | Московская обл. | Pat | 16S, PCR | PatI | – | |
| 44 | F048 | 2012 | Тверская обл. | Pat | 16S, PCR | PatI | – | |
| 45 | F148 | PB20 | 2013 | Московская обл. | Ppa | NZ_PDDJ00000000.1 | Ppa | – |
| 46 | F149 | 2013 | Тверская обл. | Ppa | 16S, PCR | Ppa | – |
Примечание. Pcc – P. carotovorum subsp. carotovorum, Pcc* – штаммы P. carotovorum subsp. carotovorum, относящиеся к перспективному геномовиду gsp maceratum (Shirshikov et al., 2018); Pcb – P. carotovorum subsp. brasiliense; Pat – P. atrosepticum; Ppa – P. parmentieri; Ddi – Dickeya dianthicola; Dso – D. solani. 16S – проведено секвенирование фрагмента гена 16S рРНК. PCR –определение таксономической принадлежности проведено стандартными диагностическими тестами (Humphris et al., 2015). ND – не определено/неизвестно.
Рис. 3.
BOX-фингерпринты геномов бактерий Pcc, чувствительных к фагу РР16. Электрофорез в 1.5% агарозном геле с окрашиванием бромидом этидия. Номера штаммов указаны на дорожках. М – маркер HyperLadder 1 kb.
Свойства генома бактериофага РР16. Собранный с 6179-кратным покрытием геном бактериофага РР16 представляет собой линейную двухцепочечную ДНК длиной 44268 п.н. и (Г + Ц) 51.9%. Из депонированных в GenBank геномов бактериофагов довольно высокое сходство последовательности РР16 имеется с геномами аннотированных, но детально не описанных фагов PPWS1 (LC063634.2) (Hirata et al., 2016), PPWS2 (LC375533.1) и BF25/12 (KT240186.1), инфицирующих P. carotovorum и Dickeya sp. соответственно. Сходство составляет от 77.9 до 87.70% и позволяет отнести РР16 к иному таксономическому виду, по сравнению с вышеупомянутыми вирусами.
Геном фага РР16 имеет организацию, характерную для фагов подсемейства Autographivirinae (рис. 4). Линейный геном имеет концевые повторы длиной 365 п.н., не кодирует собственных тРНК. Все 57 предсказанных открытых рамок считывания (ORF) расположены на одной цепи ДНК, функцию 31 (54%) из них можно предсказать анализом последовательности генов и закодированных ими белков. В геноме не было обнаружено генов, характерных для лизогенизации бактерии, то есть инфекционный цикл фага РР16 можно считать литическим. По функциям кодируемых ими белков ORF можно разделить на три группы.
Рис. 4.
Сравнительная схема структуры геномов фагов PP16, PPWS, PhiM1, KP34 и phiKMV. Серым цветом указано соответствие гомологичных участков геномов, насыщенность цвета отражает уровень идентичности последовательностей. Стрелками показаны белок-кодирующие гены.

Область ранних генов включает рамки, которые транскрибируются непосредственно после ввода фаговой ДНК в клетку-хозяин. Считается, что эти ORF вовлечены в перенаправление метаболизма клеточных белков на развитие инфекционного цикла фага за счет стимулирования или ингибирования их функций (Roucourt, Lavigne, 2009). Пять рамок, расположенных в области перед геном ДНК-праймазы (PP16_ORF14), высоко консервативны в геномах фагов PPWS1, BF25/12, фагов, отнесенных к роду phiM1-подобных (Buttimer et al., 2018), а также ряда неохарактеризованных подофагов, инфицирующих P. atrosepticum – Gaspode (MH807811.1), Lelidair (MH807814.1), Momine (MH807816.1), Nobby (MH807818.1) и Slant (MY817819.1). Другие гены ранней области кодируют белки, участвующие в репликации и репарации ДНК. Положение гена, кодирующего односубъединичную РНК-полимеразу (PP16_ORF37), в конце этой области характерно для представителей родов phiKMV, KP34 и phiM1-подобных бактериофагов, и отличается от “классических” Т7-подобных и SP6-подобных фагов подсемейства Autographivirinae.
Гены, кодирующие белки сборки и морфогенеза вириона РР16, также высоко консервативны по аминокислотным последовательностям и положению в геноме (рис. 4). Предсказанная с помощью программы HHPRED структура белка хвостового шипа (PP16_ORF57) характерна для этих компонентов адсорбционного аппарата фагов. В состав фаговых хвостовых шипов часто входят ферментативные домены, разрушающие или модифицирующие внешние полисахариды клеточной стенки бактерий в процессе адсорбции фага. Для PP16_ORF57 нами предсказана деацетилазная активность. Последовательность предсказанных элементов вторичной структуры PP16_ORF57 аналогична белку gp63.1 фага E. coli G7C, для которого определена трехмерная структура (Prokhorov et al., 2017). При этом наибольшее сходство аминокислотной последовательности PP16_ORF57 наблюдается не с белками хвостовых шипов близкородственных phiM1-подобных фагов, инфицирующих P. atrosepticum, а с белком gp40 филогенетически отдаленного фага РР2, инфицирующего P. carotovorum subsp. carotovorum (Lim et al., 2017). Также гены с последовательностью, гомологичной PP16_ORF57, обнаружены в областях профагов геномов различных штаммов P. carotovorum subsp. carotovorum и P. polaris. Можно предположить, что консервативная последовательность аминокислотных остатков обеспечивает наилучшую стерическую координацию белка хвостового шипа для связывания с цепочками О-полисахаридов именно P. carotovorum subsp. carotovorum, таким образом, определяя инфекционный диапазон бактериофага.
В кассете лизиса фага РР16 закодированы белки, которые осуществляют разрушение клетки хозяина в конце инфекционного цикла. Эндолизин (PP16_ORF56) имеет предсказанный N-концевой трансмембранный домен и может быть отнесен к группе signal–arrest–release (SAR) – эндолизинов. Совместно с белком пинхолином (PP16_ORF55) он обеспечивает проникновение через внутреннюю мембрану бактерии и деградацию пептидогликана клеточной стенки. Продукт первого гена в кассете (PP16_ORF54) имеет предсказанную N-концевую сигнальную последовательность и С-концевой трансмембранный домен, что относит его к классу u-спанинов или белков типа Rz1A (Young, 2014). Такое строение кассеты лизиса отмечено у phiM1-подобных фагов Pectobacterium (Buttimer et al., 2018) и KP34-подобных фагов Klebsiella (Eriksson et al., 2015). Однако следует отметить наличие дополнительного неперекрывающегося гена (PP16_ORF53), кодирующего белок с предсказанным трансмембранным доменом. Этот небольшой белок также может быть частью кассеты лизиса, выполняя роль цитоплазматического i-спанина (Rz-белка). В этой же позиции белки с заметным сходством аминокислотной последовательности закодированы в геномах фагов BF25/12 (BF2512_05), Momine (AXY81930.1), Nobby (AXY82036.1) и отнесенного к роду Phimunavirus фага Peat1 (YP_009224684.1). Участие этого белка в процессе лизиса хозяина требует экспериментального подтверждения, однако на основании биоинформационного анализа можно предположить, что наличие дополнительного гена в модуле лизиса характерно для phiM1-подобных фагов.
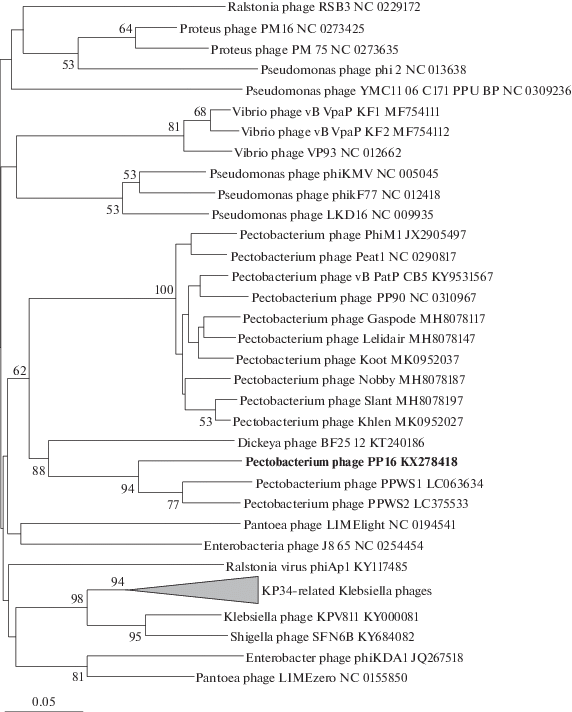
Сравнительная геномика. Для определения положения фага РР16 в подсемействе Autographivirinae был проведен филогенетический анализ полных геномных нуклеотидных последовательностей при помощи программы VICTOR с использованием формулы D4 (Meier-Kolthoff, Göker, 2017) (рис. 5). Полученная филограмма показывает, что фаги P. carotovorum subsp. carotovorum PP16, PPSW1, PPWS2, а также фаг Dickeya BF25/12 формируют отдельную группу на ветви, также включающей PhiM1-подобные фаги (Buttimer et al., 2018). Эта ветка располагается довольно близко от фагов рода KP34virus и группы, включающей фаги Vibrio VP93 и Pantoea LIMElight, которые были ранее описаны как эволюционно родственные группе KP34virus (рис. 5). Филогенетический анализ был также проведен с аминокислотными последовательностями генов, составляющих геномы фагов. Результат этого анализа представляет собой дерево, практически аналогичное по топологии предыдущему, но с меньшей достоверностью. Филогенетический анализ, основанный на сходстве белков, показывает, что фаг РР16 имеет достаточно близкое эволюционное родство с PhiM1-подобными фагами. Формирование отдельной ветви рода Phumunavirus, включающей PP16, PPSW1, PPSW2 и BF25/12, на основании средней идентичности нуклеотидных последовательностей геномов (от 77.9 до 87.7%), можно объяснить таксономическими различиями бактерий-хозяев. Все бактериофаги, отнесенные к роду Phimunavirus, инфицируют P. atrosepticum – родственный Pcc вид, но имеющий ряд отличий в спектре вирулентности, биохимических и генетических свойствах (Adeolu et al., 2016; Pérombelon, 2002). Приспособление к метаболизму имеющих общее эволюционное происхождение, но не одинаковых бактериальных хозяев привело к появлению различий в последовательности, но не общей архитектуре генома у фагов PP16, PPSW1, PPSW2 и BF25/12.
Рис. 5.
Филогенетическое древо вирусов семейства Autographivirinae, наиболее близких к вирусу PP16, установленное на основании полных нуклеотидных последовательностей при помощи сервиса VICTOR. Расстояния между геномами были получены при помощи полногеномного выравнивания, древо построено в программе FASTME методом минимальной эволюции, визуализировано в программе FigTree. Значения достоверности установлены при помощи 100 псевдо-репликаций и указаны для узлов, имеющих достоверность выше 50%. Ветвь фагов рода Kp34virus и родственных им свернута для улучшения восприятия древа. Шкала показывает рассчитанное расстояние между геномными последовательностями.
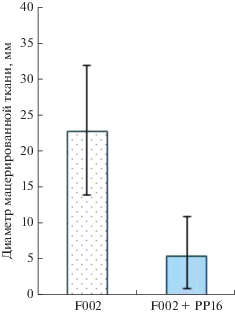
Защитное действие бактериофага при инфекции ткани клубня картофеля. Способность бактериофага РР16 влиять на развитие мягкой гнили при инокуляции дисков картофельных клубней штаммом Рсс F002 оценивали сравнением диаметра мацерированной ткани вокруг точки инокуляции через 72 ч в отсутствие и в присутствии бактериофага. При совместном нанесении патогенных бактерий и фага (MOI = 1) наблюдали достоверное 4-кратное уменьшение размера области поражения по сравнению с положительным контролем (рис. 6). Таким образом, бактериофаг РР16 способен эффективно замедлять развитие мягкой гнили ткани картофеля, вызванной штаммом Рсс F002.
Рис. 6.
Защитное действие бактериофага РР16 при совместной инокуляции ткани картофеля бактериофагом и штаммом P. carotovorum subsp. carotovorum F002 (справа) по сравнению с инокуляцией только бактерий штаммом F002 (слева). Эффект оценивали по диаметру мацерированной ткани в месте инокуляции после инкубации в течение 72 ч при 26°С.
Стимулирующее действие бактериофага в полевом эксперименте. Погодные условия вегетационного периода 2017 г. были благоприятными для развития бактериальных и грибных болезней картофеля. Май и июнь были холодными с большим количеством осадков. Период от посадки до появления всходов по этой причине составил 50 дней, что вдвое дольше обычного. Вплоть до третьей декады июля отмечалась избыточная влажность почвы.
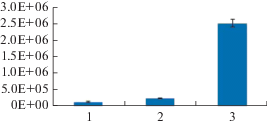
Первоначальный инфекционный фон Pcc в посадочном материале составлял в среднем 1.24 × × 103 КОЕ/клубень. В собранном урожае на здоровых клубнях контрольного варианта данный показатель составил 2.5 × 106 КОЕ/клубень, достигая концентрации, достаточной для проявления симптомов мягкой гнили (Pérombelon, 2002). В опыте с применением обработки бактериофагом РР16 концентрация бактерий Pcc в урожае была ниже в 10 раз и составила 2.32 × 105 КОЕ/клубень (рис. 7). Аналогично эксперименту на картофельных дисках, можно предположить, что ингибирование роста, но не полная элиминация популяции Рсс связаны с возникновением фагоустойчивых мутантов или наличием в микробном сообществе штаммов P. carotovorum, невосприимчивых к действию фага РР16 и продолжающих размножение в течение сезона вегетации. На фоне аномально низкой всхожести клубней в контроле, вызванной длительным периодом пребывания ростков в почве и развитием комплекса болезней до всходов, очень выраженным оказывается эффект стимулирования всхожести в случае обработки фагом – более, чем в 5 раз (85% по сравнению с 15% в зараженном контроле). Урожайность в контроле составила 0.7 кг/м2, в опытном варианте 4.65 кг/м2; соответственно, прибавка к урожайности контроля составила 567.5%. Столь высокая биологическая эффективность не может быть объяснена прямым действием фагового препарата на патогены, т.к. фаг РР16 не может быть инфекционным против всех представителей бактериального сообщества, ассоциированных с мягкой гнилью картофеля, и популяция пектобактерий на клубнях нового урожая также значительна.
Рис. 7.
Защитное действие бактериофага РР16 в полевом опыте. Численность популяции Pectobacterium spp.: 1 – клубни перед посадкой (до обработки), 2 – урожай клубней в варианте с обработкой бактериофагом, 3 – урожай клубней в контроле, без обработки бактериофагом.
В данный момент мы не можем предложить обоснованную гипотезу для объяснения влияния бактериофага на всхожесть растений и прибавку урожая картофеля. Однако очевидно, что стратегия обработки бактериофагами приводит к положительным результатам не только в случае защиты картофеля от мягкой гнили клубней при хранении собранного урожая, но и в полевых экспериментах. В дальнейшем исчерпывающе охарактеризованный бактериофаг P. carotovorum subsp. carotovorum PP16 может быть использован при конструировании перспективных препаратов (коктейлей) бактериофагов для комплексного биоконтроля заболеваний (мягкой гнили и черной ножки) картофеля.
ФИНАНСИРОВАНИЕ РАБОТЫ
Исследование, кроме исследования морфологии бактериофага, выполнено при поддержке гранта РНФ № 16-16-00073.
Список литературы
Ягодин Б.А., Жуков Ю.П., Кобзаренко В.И. Агрохимия / Под ред. Ягодина Б.А. М.: Колос, 2002. 584 с.
Ackermann H.-W. Basic Phage Electron Microscopy // Bacteriophages: Methods and Protocols. 2009. V. 1. P. 113–126.https://doi.org/10.1007/978-1-60327-164-6
Adeolu M., Alnajar S., Naushad S., Gupta R.S. Genome-based phylogeny and taxonomy of the ‘Enterobacteriales’: Proposal for Enterobacterales ord. nov. divided into the families Enterobacteriaceae, Erwiniaceae fam. nov., Pectobacteriaceae fam. nov., Yersiniaceae fam. nov., Hafniaceae fam. nov. // Int. J. Syst. Evol. Microbiol. 2016. V. 66. P. 5575–5599.https://doi.org/10.1099/ijsem.0.001485
Altschul S.F., Madden T.L., Schäffer A.A., Zhang J., Zhang Z., Miller W., Lipman D.J. Gapped BLAST and PSI-BLAST: A new generation of protein database search programs // Nucl. Acids Res. 1997. V. 25. P. 3389–3402.https://doi.org/10.1093/nar/25.17.3389
Balogh B., Jones J.B., Iriarte F.B., Momol M.T. Phage therapy for plant disease control // Curr. Pharm. Biotechnol. 2010. V. 11. P. 48–57.https://doi.org/10.2174/138920110790725302
Bankevich A., Nurk S., Antipov D., Gurevich A.A., Dvorkin M., Kulikov A.S., Lesin V.M., Nikolenko S.I., Pham S., Prjibelski A.D., Pyshkin A.V., Sirotkin A.V., Vyahhi N., Tesler G., Alekseyev M.A., Pevzner P.A. SPAdes: A new genome assembly algorithm and its applications to single-cell sequencing // J. Computat. Biol. 2012. V. 19. P. 455–477. https://doi.org/10.1089/cmb.2012.0021
Besemer J., Lomsadze A., Borodovsky M. GeneMarkS: A self-training method for prediction of gene starts in microbial genomes. Implications for finding sequence motifs in regulatory regions // Nucl. Acids Res. 2001. V. 29. P. 2607–2618. doi 10.1093/nar/29.12.2607
Buttimer C., Lucid A., Neve H., Franz C.M.A.P., O’Mahony J., Turner D., Lavigne R., Coffey A. Pectobacterium atrosepticum phage VB_PatP_CB5: a member of the proposed genus ‘Phimunavirus’// Viruses. 2018. V. 10. pii: E394.https://doi.org/10.3390/v10080394
Clokie M.R.J., Kropinski A.M. Bacteriophages: Methods and Protocols. V. 1: Isolation, Characterization, and Interactions // Methods in Molecular Biology. Humana press, 2009. https://doi.org/10.1007/978-1-60327-164-6
Czajkowski R., Ozymko Z., de Jager V., Siwinska J., Smolarska A., Ossowicki A., Narajczyk M., Lojkowska E. Genomic, proteomic and morphological characterization of two novel broad host lytic bacteriophages ΦPD10.3 and ΦPD23.1 infecting pectinolytic Pectobacterium spp. and Dickeya spp. // PLoS One. 2015. V. 10. Article e0119812.https://doi.org/10.1371/journal.pone.0119812
Dees M.W., Lysøe E., Rossmann S., Perminow J., Brurberg M.B. Pectobacterium Polaris sp. nov., isolated from potato (Solanum tuberosum) // Int. J. Syst. Evol. Microbiol. 2017. V. 67. P. 5222–5529.https://doi.org/10.1099/ijsem.0.002448
Eriksson H., Maciejewska B., Latka A., Majkowska-Skrobek G., Hellstrand M., Melefors Ö., Wang J.-T., Kropinski A.M., Drulis-Kawa Z., Nilsson A.S. A suggested new bacteriophage genus, ‘Kp34likevirus’, within the Autographivirinae subfamily of Podoviridae // Viruses. 2015. V. 7. P. 1804–1822.https://doi.org/10.3390/v7041804
Finn R.D., Clements J., Eddy S.R. HMMER Web Server: Interactive Sequence Similarity. Searching // Nucl. Acids Res. 2011. V. 39. Suppl. 2.https://doi.org/10.1093/nar/gkr367
Gardan L., Gouy C., Christen R., Samson R. Elevation of three subspecies of Pectobacterium carotovorum to species level: Pectobacterium atrosepticum sp. nov., Pectobacterium betavasculorum sp. nov. and Pectobacterium wasabiae sp. nov. // Int. J. Syst. Evol. Microbiol. 2003. V. 53 P. 381–391.https://doi.org/10.1099/ijs.0.02423-0
Hirata H., Kashihara M., Horiike T., Suzuki T., Dohra H., Netsu O., Tsuyumu S. Genome Sequence of Pectobacterium Carotovorum Phage PPWS1, Isolated from Japanese Horseradish [Eutrema Japonicum (Miq.) Koidz] Showing Soft-Rot Symptoms.// Genome Announcements. 2016. V. 4. № 2. pii: e01625-15. https://doi.org/10.1128/genomeA.01625-15
Humphris S.N., Cahill G., Elphinstone J.G., Kelly R., Parkinson N.M., Pritchard L., Toth I.K., Saddler G.S. Detection of the bacterial potato pathogens Pectobacterium and Dickeya spp. using conventional and real-time PCR // Methods in Molecular Biology. Clifton, N.J., 2015. P. 1–16.https://doi.org/10.1007/978-1-4939-2620-6_1
Lavigne R., Sun W.D., Volckaert V. PHIRE, a deterministic approach to reveal regulatory elements in bacteriophage genomes // Bioinformatics. 2004. V. 20. P. 629–635.https://doi.org/10.1093/bioinformatics/btg456
Lavigne R., Seto D., Mahadevan P., Ackermann H.-W., Kropinski A.M. Unifying classical and molecular taxonomic classification: Analysis of the Podoviridae using BLASTP-based tools // Rese. Microbiol. 2008. V. 159. P. 406–414. https://doi.org/10.1016/j.resmic.2008.03.005
Lim J.A., Heu S., Park J., Roh E. Genomic characterization of bacteriophage VB_PcaP_PP2 infecting Pectobacterium carotovorum subsp. carotovorum, a new member of a proposed genus in the subfamily Autographivirinae // Arch. Virol. 2017. V. 162. P. 2441–2444.https://doi.org/10.1007/s00705-017-3349-6
Mansfield J., Genin S., Magori S., Citovsky V., Sriariyanum M., Ronald P., Dow M., Verdier V., Beer S.V., Machado M.A., Toth I., Salmond G., Foster G.D. Top 10 plant pathogenic bacteria in molecular plant pathology // Mol. Plant Pathol. 2012. V. 13. P. 614–629.https://doi.org/10.1111/j.1364-3703.2012.00804.x
Martin B., Humbert O., Camara M., Guenzi E., Walker J., Mitchell T., Andrew P., Prudhomme M., Alloing G., Hakenbeck R., Morrison D.A., Boulnois G.J., Claverys J.-P. A highly conserved repeated DNA element located in the chromosome of Streptococcus pneumoniae // Nucl. Acids Res. 1992. V. 20. P. 3479–3483.https://doi.org/10.1093/nar/20.13.3479
Meier-Kolthoff J.P., Göker M. VICTOR: Genome-based phylogeny and classification of prokaryotic viruses // Bioinformatics. 2017. V. 33. P. 3396–3404.https://doi.org/10.1093/bioinformatics/btx440
Pérombelon M.C.M. Potato diseases caused by soft rot erwinias: an overview of pathogenesis // Plant Pathol. 2002. V. 51. P. 1–12.https://doi.org/10.1046/j.0032-0862.2001
Prokhorov N.S., Riccio C., Zdorovenko E.L., Shneider M.M., Browning C., Knirel Y.A., Leiman P.G., Letarov A.V. Function of bacteriophage G7C esterase tailspike in host cell adsorption // Mol. Microbiol. 2017. V. 105. P. 385–398. https://doi.org/10.1111/mmi.13710
Roucourt B., Lavigne R. The role of interactions between phage and bacterial proteins within the infected cell: a diverse and puzzling interactome // Environ. Microbiol. 2009. V. 11. P. 2789–2805.https://doi.org/10.1111/j.1462-2920.2009.02029.x
Sambrook J., Fritsch E.F., Maniatis T. Molecular Cloning: A Laboratory Manual. 2nd ed. Cold Spring Harbor Laboratory Press, 1989. 479 p.
Sarfraz S., Riaz K., Oulghazi S., Cigna J., Sahi S.T., Khan S.H., Faure D. Pectobacterium punjabense sp. nov., isolated from blackleg symptoms of potato plants in Pakistan // Int. J. Syst. Evol. Microbiol. 2018. V. 68. P. 3551–3556. https://doi.org/10.1099/ijsem.0.003029
Shirshikov F.V., Korzhenkov A.A., Miroshnikov K.K., Kabanova A.P., Barannik A.P., Ignatov A.N., Miroshnikov K.A. Draft genome sequences of new genomospecies “Candidatus Pectobacterium maceratum” strains, which cause soft rot in plants // Genome Announc. 2018. V. 6. Article e00260-18.https://doi.org/10.1128/genomeA.00260-18
Waleron M., Misztak A., Waleron M., Franczuk M., Wielgomas B., Waleron K. Transfer of Pectobacterium carotovorum subsp. carotovorum strains isolated from potatoes grown at high altitudes to Pectobacterium peruviense sp. nov. // Syst. Appl. Microbiol. 2018. V. 41. P. 85–93.https://doi.org/10.1016/J.SYAPM.2017.11.005
Young R. Phage lysis: Three steps, three choices, one outcome // J. Microbiol. 2014. V. 52. P. 243–258.https://doi.org/10.1007/s12275-014-4087-z
Дополнительные материалы отсутствуют.


