Микробиология, 2019, T. 88, № 4, стр. 490-496
Состав бактериальных сообществ рек острова новая Сибирь
О. С. Журлов a, *, Н. В. Немцева a, Д. А. Грудинин b, С. В. Левыкин b, И. Г. Яковлев b
a Институт клеточного и внутриклеточного симбиоза Уральского отделения Российской академии наук
460000 Оренбург, Россия
b Институт степи Уральского отделения Российской академии наук
460000 Оренбург, Россия
* E-mail: jurlov1968@mail.ru
Поступила в редакцию 24.08.2018
После доработки 12.03.2019
Принята к публикации 01.04.2019
Аннотация
В статье представлены результаты молекулярно-генетических исследований прокариотических сообществ речных экосистем центральной и юго-восточной части острова Новая Сибирь. В составе микробных сообществ доминирующее положение занимали представители 5 фил – Proteobacteria, Firmicutes, Actinobacteria, Bacteroidetes, Chloroflexi. При исследовании структуры прокариотических сообществ отмечено низкое содержание архей.
Актуальность исследований прокариотических сообществ островов арктических морей обусловлена не только активным освоением Арктики, но и слабой изученностью разнообразия прокариотической компоненты микробных сообществ этих местообитаний.
В настоящее время предпринимаются попытки оценки биологического разнообразия по федеральному и региональным законодательствам особо охраняемых природных территорий в береговой зоне арктических морей (Спиридонов и соавт., 2011). Однако сведений о структуре микробных сообществ речных экосистем, имеющих значение для функционирования арктических биоценозов, не достаточно.
Применение высокоэффективных молекулярно-генетических методов позволило значительно продвинуться в изучении микробного разнообразия различных биотопов криозоны, в частности, грунта в районах многолетней мерзлоты (Gilichinsky et al., 2003; Krivushin et al., 2010), а также содержимого кишечника ископаемых животных (Mardanov et al., 2012). На протяжении последнего десятилетия активно изучается микробное разнообразие вод Арктики и Антарктики (Baldwin et al., 2005; Vincent et al., 2009; Shtarkman et al., 2013). Однако в большей степени исследователей привлекают холодные океанические и морские экосистемы (Bowman et al., 2003; Li et al., 2015). Кроме того, в доступной нам литературе, мы не нашли информации о биоразнообразии микробных сообществ водоемов Новосибирских островов, имеются лишь отрывочные сведения о физико-географических особенностях (Гаккель, 1967), а также лимнологическая характеристика водоемов архипелага Новосибирских островов (Ушницкая и соавт., 2013).
Остров Новая Сибирь входит в состав архипелага Новосибирские острова и расположен в акватории Восточно-Сибирского моря. В 2011–2013 годах Русским географическим обществом были организованы экспедиции с участием сотрудников Института степи УрО РАН. В рамках экспедиции проводились комплексные исследования островов архипелага.
Для острова характерно преобладание тундровой растительности и отсутствие кустарниковых и древесных форм. Прибрежная акватория островов относится к водам, подвергшимся наименьшему антропогенному воздействию (Halpern et al., 2007). Особенностью гидрографической сети острова является меандрирование русла внутренних рек, что определяется рельефом местности, сформированным при участии криогенных (мерзлотных) процессов.
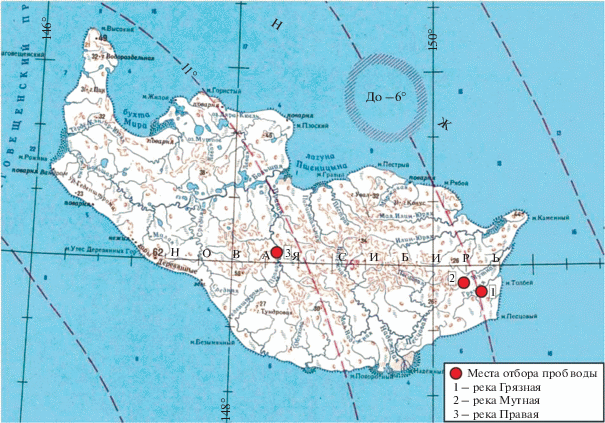
В летний период 2012 г. на острове Новая Сибирь из рек Грязная и Мутная, находящихся в восточной части острова, и реки Правая, протекающей в центральной части острова, были отобраны пробы воды для химического и микробиологического анализов.
Целью нашего исследования было провести анализ состава бактериальных сообществ речных экосистем острова Новая Сибирь при исследовании проб, отобранных из рек Грязная, Мутная и Правая.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Объектом исследования явились пробы воды рек Грязная, Мутная (юго-восточная часть) и Правая (центральная часть) острова Новая Сибирь. По типу питания водотоки Новосибирских островов можно отнести к рекам снежно-дождевого питания с некоторым преобладанием снежного, хотя осадки в течение года распределяются довольно равномерно. Незначительную долю воды реки получают при таянии льда лессово-ледовых формаций. Гидрологический режим рек соответствует источникам питания. Колебания уровня характеризуются тремя основными фазами: резко выраженным весенним половодьем, низкой летней меженью, изредка нарушаемой дождевыми паводками, и зимней меженью, когда у большинства рек сток полностью прекращается. Внутригодовое распределение стока рек отличается крайней неравномерностью. Максимум наблюдается во время весеннего половодья, когда проходит более 80% годового объема стока. Вода в реках мутная. Вследствие термического и динамического воздействия воды берега сильно разрушаются.
Отбор проб речной воды производили по маршруту следования экспедиции от побережья к центральной части острова Новая Сибирь (рис. 1). Температура воды в реках колебалась от 1.1 до 6.5°С. Пробы (общий объем 3 л) отбирали на мелководье с глубины 0.5 м примерно в 1 м от береговой линии с соблюдением нормативных требований ГОСТ 31942-2012 “Вода. Отбор проб для микробиологического анализа”. После этого каждую емкость укупоривали, удаляли загрязнение с ее внешней поверхности, наносили соответствующую маркировку и упаковывали в чистый пакет. При транспортировке пробы хранили в термоконтейнерах (4–5°С). В дальнейшем пробы концентрировали на стерильных поликарбонатных мембранных фильтрах с диаметром пор 0.22 мкм (“Millipore”, США) и до проведения молекулярно-генетического анализа хранили в стерильных емкостях в рефрижераторе (–70°С).
Выделение тотальной ДНК. Из фильтров тотальную ДНК выделяли комбинированным методом, включавшим механическую гомогенизацию в сочетании с методом ферментативного лизиса (Белькова, 2009). К образцам добавляли 400 мкл трис-солевого буфера (20 ммоль/л ЭДТА, 750 ммоль/л NaCl, 100 ммоль/л трис-HCl; pH 8.0) и гомогенизировали с помощью гомогенизатора TissueLyser LT (“QIAGEN”, Германия) с использованием лизирующего матрикса Е (“MP Biomedicals”, США) в течение 1 мин при частоте 50 Гц. После этого добавляли 50 мкл трис-солевого буфера с лизоцимом (50 мг/мл) и инкубировали 60 мин при 37°С. Затем в смесь вносили 10% раствор додецилсульфата натрия до конечной концентрации 1% и 2 мкл раствора протеиназы К (10 мг/мл), инкубировали 60 мин при 60°С. После экстракции смесью фенол–хлороформ–изоамиловый спирт (25 : 24 : 1) и последующей экстракции системой растворителей хлороформ–изоамиловый спирт (24 : 1). ДНК из водной фазы осаждали трехкратным объемом абсолютного этанола с добавлением 10 М ацетата аммония (1 : 10) при –20°С в течение 8 ч. После центрифугирования и двойной отмывки 80% этанолом ДНК высушивали и растворяли в 30 мкл деионизированной воды. Для исключения из результатов высокопроизводительного секвенирования возможной контаминации на этапе пробоподготовки использовали отрицательный контроль, для приготовления которого 100 мкл деионизированной автоклавированной воды обрабатывали с использованием описанной выше методики. Чистоту ДНК контролировали с помощью электрофореза в 1% агарозном геле и фотометрии на приборе NanoDrop 8000 (“Termo Fisher Scientific Inc.”, США). Концентрацию ДНК определяли на флуориметре Quantus (“Promega”, США) с применением набора Quanti Fluor dsDNA (“Promega”, США).
Высокопроизводительное секвенирование. ДНК-библиотеки для секвенирования были созданы по протоколу Illumina (http://support.illumina.com/documents/documentation/chemistrydocumentation/16s/16s-metagenomic-library-prep-guide-15044223-b.pdf), с праймерами к вариабельному участку V3–V4 гена 16S рРНК S-D-Bact-0341-b-S-17 и S-D-Bact-0785-a-A-21 (Klindworth et al., 2013). Секвенирование проводили на платформе MiSeq (“Illumina”, США) с использованием набора реактивов MiSeq Reagent Kit V3 PE6DD (“Illumina”, США) в Центре коллективного пользования научным оборудованием “Персистенция микроорганизмов” Института клеточного и внутриклеточного симбиоза УрО РАН.
Биоинформационная обработка. Оценку качества прочтений проводили с использованием программы FastQC vD.11.5. На следующем этапе объединяли парно-концевые прочтения, используя программу PEAR (Zhang et al., 2014). С помощью программы USEARCH v9.0.1623_win32(Edgar, 2010) осуществляли фильтрацию объединенных ридов по качеству и длине, используя команду fastq_filtr. Формирование ОТЕ проводили путем последовательной дерепликации и кластеризации на уровне 97%, что соответствует межвидовым различиям, используя алгоритм UPARSE (Edgar, 2013). Выбраковка химерных последовательностей была проведена согласно рекомендациям проекта Ribosomal Database Project с помощью сервиса Decipher chimera detection tool (http://decipher.cee.wisc.edu/FindChimeras.html). Таксономическую классификацию ОТЕ проводили с использованием базы данных RDP (http://rdp.cme.msu.edu).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Впервые проведены молекулярно-генетические исследования структуры бактериальных сообществ в образцах воды из трех рек (р. Грязная, р. Мутная и р. Правая) острова Новая Сибирь, отобранных в летний период 2012 г. В результате высокопроизводительного секвенирования участка гена 16S рРНК из образцов воды р. Грязная получено 127848 ридов (прочтений), длиной более 300 нуклеотидов. После применения биоинформатической обработки данных (фильтрации, дерепликации и кластеризации) получено 116 ОТЕ, из которых к домену Bacteria принадлежало 99.3%, а к домену Archaea – 0.4% из всех нуклеотидных последовательностей. Доля отдельных филогенетических групп от общего числа полученных для данного образца фрагментов гена 16S pPHK была различна в разных образцах.
В основном доминировали представители Proteobacteria – 69.7% (табл. 1). Доля других представителей микробного сообщества была значительно ниже: Chloroflexi – 7.7%, Firmicutes – 7.3%, Bacteroidetes – 5.1%, Actinobacteria – 3.8%. Для микробного сообщества р. Грязная было характерно низкое содержание представителей филы Verrucomicrobia (0.5%; 600 ридов), в сравнении с пробами из других рек – р. Мутная (5.3%; 5772 ридов) и р. Правая (1.8%; 3028 ридов). Доля фрагментов гена 16S рРНК, относящихся к филуму Crenarchaeota, составляла 0.4% (520 ридов).
Таблица 1.
Микробные сообщества речных экосистем о. Новая Сибирь
| Реки | Таксономический состав микробных сообществ* | |||||
|---|---|---|---|---|---|---|
| филы | % | семейства | % | роды | % | |
| Грязная | Proteobacteria | 69.7 | Rhodocyclaceae | 16.9 | Thauera | 15.6 |
| Chloroflexi | 7.7 | Alteromonadaceae | 12.5 | Bellilinea | 7.3 | |
| Firmicutes | 7.3 | Anaerolinaceae | 7.7 | Acinetobacter | 6.5 | |
| Bacteroidetes | 5.1 | Hyphomicrobiaceae | 6.9 | Cellvibrio | 6.5 | |
| Actinobacteria | 3.8 | Moraxellaceae | 6.6 | Hyphomicrobium | 6.1 | |
| Rhodospirillaceae | 3.4 | Pedobacter | 2.9 | |||
| Chromatiaceae | 3.0 | Rathayibacter | 2.3 | |||
| Мутная | Proteobacteria | 57.1 | Moraxellaceae | 17.2 | Acinetobacter | 17.1 |
| Firmicutes | 8.6 | Pedosphaeraceae | 4.4 | Pedosphaera | 4.4 | |
| Verrucomicrobia | 5.3 | Hyphomicrobiaceae | 4.3 | Stenoxybacter | 2.7 | |
| Bacteroidetes | 4.4 | Rhodocyclaceae | 4.1 | Rhodobium | 2.3 | |
| Actinobacteria | 3.2 | Neisseriaceae | 3.5 | Bellilinea | 1.9 | |
| Chloroflexi | 2.8 | Bacillaceae | 3.2 | Brachyspira | 1.8 | |
| Spirochaetes | 2.0 | Rhodospirillaceae | 3.2 | Methyloversatilis | 1.8 | |
| Правая | Proteobacteria | 53.1 | Xanthomonadaceae | 17.9 | Chlorobaculum | 6.6 |
| Actinobacteria | 8.0 | Rhodocyclaceae | 9.1 | Rhodoplanes | 5.9 | |
| Chlorobi | 6.6 | Chlorobiaceae | 6.6 | Saccharopolyspora | 5.7 | |
| Firmicutes | 5.5 | Hyphomicrobiaceae | 6.6 | Azoarcus | 5.0 | |
| Verrucomicrobia | 1.8 | Pseudonocardiaceae | 5.7 | Acinetobacter | 2.2 | |
| Bacteroidetes | 1.0 | Moraxellaceae | 2.2 | Coraliomargarita | 1.1 | |
| Bacillaceae | 1.4 | |||||
В пробах воды из реки Мутная определены 108 453 последовательности гена 16S рРНК, после обработки исходных данных получено 143 ОТЕ. Из них более 97% принадлежало домену Bacteria и 1.9% домену Archaea. Отличительной особенностью филогенетического состава микробного сообщества р. Мутная, по сравнению с сообществами р. Грязная и р. Правая, была значительная доля ридов, относящихся к представителям филы Spirochaetes (2.0%).
В пробах воды из реки Правая найдено 169 534 последовательностей фрагмента гена 16S рРНК длиной более 300 нуклеотидов, в результате кластеризации получено 151 ОТЕ, из них 99.9% принадлежало домену Bacteria и лишь 0.01% домену Archaea. Значительная доля фрагментов гена 16S pPHK представителей филы Chlorobi (6.6%; 11 123 рида) была обнаружена в пробах воды реки Правая.
Биоинформационный анализ выявил значительную часть неклассифицируемых на филогенетическом уровне последовательностей нуклеотидов. Относительная доля таких ридов в пробах воды из реки Грязная составляла 4.3% (5493 рида), р. Мутная – 9.8% (10 607 ридов) и р. Правая – 18% (30 423 рида).
Таким образом, в результате высокопроизводительного секвенирования гена 16S pPHK (по технологии Illumina) нами показано, что более 80% бактериальных сообществ речных экосистем острова Новая Сибирь составляют представители 5 фил: Proteobacteria, Actinobacteria, Firmicutes, Bacteroidetes и Chloroflexi. Доминирующее положение протеобактерий в микробных сообществах речных экосистем закономерно не только потому, что они широко распространены в пресных водоемах и грунтах в различных климатических зонах нашей планеты (Van Trappen et al., 2002), но и в связи с их основной ролью в метаболизме органических соединений.
Несмотря на то, что представители домена Archaea, в сравнении с бактериями, составляли меньшую часть от общего числа полученных нуклеотидных последовательностей, их относительная доля составляла от 0.01% (р. Правая) и 0.4% (р. Грязная) до 1.9% (р. Мутная). Большую часть архей в составе микробных сообществ рек острова Новая Сибирь составляли представители Crenarchaeota, по литературным данным они являются важной составной частью наземных биоценозов в экосистемах полярных морей (Gillan et al., 2007).
Дальнейший анализ на уровне класса, семейства и рода показал высокую гетерогенность таксономического состава микробных сообществ речных экосистем, несмотря на то, что анализировались варианты фрагмента гена 16S pPHK, содержащие более 1500 ридов (табл. 1).
В пробах воды из трех рек (р. Правая, р. Грязная, р. Мутная) среди представителей филы Proteobacteria доминировали микроорганизмы класса Gammaproteobacteria (26.8–28.6%). Бактерии, относящиеся к классам Betaproteobacteria (12.8–24%) и Alphaproteobacteria (10.9–15.2%), были представлены меньшим количеством фрагментов гена 16S pPHK. Другие классы содержали от 2 до 8 тысяч пар нуклеотидных последовательностей (ридов): Actinobacteria – от 2.6 до 7.5%, Bacilli – от 3.2 до 6.2%, Anaerolineae – от 2.7 до 7.7%. Кроме того, важной особенностью, по мнению авторов, является присутствие представителей домена Archaea и высокое содержание бактерий классов Gammaproteobacteria и Bacilli в составе прокариотических сообществ речных экосистем, свидетельствующее о поступлении в воду рек микроорганизмов с частицами грунта. Аналогичный феномен был отмечен нами ранее при исследовании воды из ручья, протекающего по дну Батагайского провала (Журлов и соавт., 2015), а также другими исследователями (Hauptmann et al., 2016). Необходимо отметить обнаружение значительной доли фрагмента гена 16S pPHK представителей класса Sphingobacteria (4.2%) в пробах воды из р. Грязная, микроорганизмов класса Pedosphaerae (4.4%) в р. Мутная и представителей класса Chlorobia (6.6%) и Clostridia (2.1%) в р. Правая.
На уровне семейств доминирующие позиции в составе микробных сообществ речных экосистем занимали представители Rhodocyclaceae (4.1–16.9%), Moraxellaceae (2.2–17.2%), Hyphomicrobiaceae (4.3–6.9%) и микроорганизмы семейства Bacillaceae (1.4–3.2%) в реках Мутная и Правая.
Спорообразующим бактериям (сем. Bacillaceae), обладающим мощным ферментативным аппаратом, отводится важная роль в минерализации органики. Однако, как было отмечено ранее, их видовое разнообразие в почвах северных широт значительно ниже, в сравнении с почвами умеренных широт (Гродницкая и соавт., 2013).
Качественный состав микробных сообществ речных экосистем на уровне рода отличался большей гетерогенностью. Доминирующее положение в составе микробного сообщества реки Грязная занимали микроорганизмы рода Thauera – 15.6% (класс Betaproteobacteria); представители рода Acinetobacter (класс Gammaproteobacteria) с высокой долей прочтений фрагмента гена 16S pPHK (2.2–17.1%) были обнаружены во всех трех реках. Микроорганизмы рода Bellilinea (класс Anaero-lineae) встречались в пробах из рек Грязная и Мутная, соответственно, 7.3 и 1.9% (табл. 1). Микроорганизмы рода Acinetobacter (класс Gammaproteobacteria) являются убиквитарными почвенными и водными сапрофитами (Bhargava et al., 2010).
Представителей рода Bellilinea (класс Anaerolineae) часто обнаруживают в составе иловых отложений. Они участвуют в процессах биодеградации поллютантов различного химического строения (Yamada et al., 2007; Ma et al., 2015). Для эффективного роста эти нитевидные, анаэробные, трудно культивируемые микроорганизмы нуждаются в ассоциации с другими бактериями (Yamada et al., 2006).
В пробе воды р. Мутная обнаружена относительно высокая доля фрагментов гена 16S pPHK представителей рода Brachyspira (1.8%; 1911 ридов). Они часто выделяются из кишечного тракта млекопитающих и птиц, их присутствие в пробах воды может быть связано с водоплавающими перелетными птицами (Mappley et al., 2014), гнездящимися на островах. Грамотрицательные бактерии рода Pedobacter (2.9%; 3669 ридов), обнаруженные в пробе воды р. Грязная, входят в состав микробных сообществ почв различных экстремальных сред (Viana et al., 2018).
Проведенные молекулярно-генетические исследования образцов воды рек острова Новая Сибирь являются первыми в изучении микробных сообществ речных экосистем Новосибирских островов.
Список литературы
Белькова Н.Л. Модифицированная методика выделения суммарной ДНК из водных проб и грунтовых вытяжек методом ферментативного лизиса // Молекулярно-генетические методы анализа микробных сообществ. Разнообразие микробных сообществ внутренних водоемов России: Учебно-методическое пособие. Ярославль: Изд-во ООО “Принтхаус”, 2009. С. 53–63.
Гаккель Я.Я. Новосибирские острова: физико-географическая характеристика архипелага. Л.: Гидрометеорологическое издательство, 1967. 211 с.
Гродницкая И.Д., Карпенко Л.В., Кнорре С.Н., Сырцов С.Н. Микробная активность торфяных почв заболоченных лиственничков и болота в криолитозоне центральной Эвенкии // Почвоведение. 2013. Т. 46. № 1. С. 67–79.
Grodnitskaya I.D., Karpenko L.V., Knorre A.A., Syrtsov S.N. Microbial activity of peat soils of boggy larch forests and bogs in the permafrost zone of central Evenkia // Euras. Soil Sci. 2013. V. 46. P. 61–73.
Журлов О.С., Грудинин Д.А., Яковлев И.Г. Филогенетический 16S метагеномный анализ и антибиотикорезистентность психротолерантных бактерий, выделенных из грунтов Батагайского Провала // Международный журнал прикладных и фундаментальных исследований. 2015. № 11-5. С. 648–651.
Спиридонов В.А., Гаврило М.В., Краснова Е.Д., Николаева Н.Г. Атлас биологического разнообразия морей и прибрежной Арктики. М.: WWF России, 2011. 64 с.
Ушницкая Л.А., Городничев Р.М., Спиридонова И.М., Пестрякова Л.А. Предварительная лимнологическая характеристика водоемов полуострова Фаддеевский (Новосибирские острова) // Международный журнал прикладных и фундаментальных исследований. 2013. № 8. С. 189–192.
Baldwin A.J., Moss J.A., Pakulski J.D. Microbial diversity in a Pacific Ocean transect from the Arctic to Antarctic circles // Aquat. Microb. Ecol. 2005. V. 102. P. 41–91.
Bhargava N., Sharma P., Capalash N. Quorum sensing in Acinetobacter: an emerging pathogen // Crit. Rev. Microbiol. 2010. V. 36. P. 349–360.
Bowman J.P.S., McCammon A., Gibson J.A.E. Prokaryotic metabolic activity and community structure in Antarctic continental shelf sediments // Appl. Environ. Microbiol. 2003. V. 69. P. 2448–2462.
Edgar R.C. Search and clustering orders of magnitude faster than BLAST // Bioinformatics. 2010. V. 26. P. 2460–2461.
Edgar R.C. UPARSE: highly accurate OTU sequences from microbial amplicon reads // Nat. Methods. 2013. V. 10. P. 996–998.
Gilichinsky D., Rivkina E., Shcherbakova V., Laurinavichuis K., Tiedje J. Supercooled water brines within permafrost – an unknown ecological niche for microorganisms: a model for astrobiology // Astrobiology. 2003. V. 3. P. 331–341.
Gillan D.C., Danis B. The archaebacterial communities in Antarctic bathypelagic sediments // Deep-Sea Research II. 2007. V. 54. P. 1682–1690.https://doi.org/10.1016/j.dsr2.2007.07.002
Halpern B.S., Selkoe K.A., Micheli F., Kappel C.V. Evaluating and ranking the vulnerability of global marine ecosystems to anthropogenic threats // Conserv. Biol. 2007. V. 21. P. 1301–1315.
Hauptmann A.L., Markussen T.N., Stibal M., Olsen N.S., Elberling B., Bælum J., Sicheritz-Pontén T., Jacobsen C.S. Upstream freshwater and terrestrial sources are differentially reflected in the bacterial community structure along a small Arctic river and its estuary // Front. Microbiol. 2016. V. 7. P. 1474.https://doi.org/10.3389/fmicb.2016.01474
Klindworth A., Pruesse E., Schweer T., Peplies J., Quast C., Horn M., Glöckner F.O. Evaluation of general 16S ribosomal RNA gene PCR primers for classical and next-generation sequencing-based diversity studies // Nucl. Acids Res. 2013. V. 41. P. 1–11.
Krivushin K.V., Shcherbakova V.A., Petrovskaya L.E., Rivkina E.M. Methanobacterium veterum sp. nov., from ancient Siberian permafrost // Int. J. Syst. Evol. Microbiol. 2010. V. 60. P. 455–459.
Li Y., Liu Q., Li C., Dong Y., Zhang W., Zhang W., Xiao T. Bacterial and archaeal community structures in the Arctic deep-sea sediment // Acta Oceanologica Sinica. 2015. V. 34. P. 93–113.https://doi.org/10.1007/s13131-015-0624-9
Ma W., Nie C., Su F., Cheng X., Yan Y., Chen B., Lun X. Migration and biotransformation of three selected endocrine disrupting chemicals in different river-based aquifers media recharge with reclaimed water // Int. Biodeteriorat. Biodegradat. 2015. V. 102. P. 298–307.https://doi.org/10.1016/j.ibiod.2015.03.022
Mappley L.J., La Ragione R.M., Woodward M.J. Brachyspira and its role in avian intestinal spirochaetosis // Vet. Microbiol. 2014. V. 168. P. 245–260. https://doi.org/10.1016/j.vetmic.2013.11.019
Mardanov A.V., Bulygina E.S., Nedoluzhko A.V., Kadnikov V.V., Beletskii A.V., Tsygankova S.V., Tikhonov A.N., Ravin N.V., Prokhorchuk E.B., Skryabin K.G. Molecular analisis of the intestinal microbiome composition of mammoth and woolly rhinoceros // Dokl. Biochem. Biophys. 2012. V. 445. P. 203–206.
Shtarkman Y.M., Koçer Z.A., Edgar R., Veerapaneni R.S., D’Elia T., Morris P.F., Rogers S.O. Subglacial Lake Vostok (Antarctica) accretion ice contains a diverse set of sequences from aquatic, marine and sediment-inhabiting Bacteria and Eukarya // PLoS One. 2013. V. 8. Article e67221. Doi.org/10.1371/journal.pone.0067221
Van Trappen S., Mergaert J., Van Eygen S., Dawyndt P., Cnockaert M.C., Swings J. Diversity of 746 heterotrophic bacteria isolated from microbial mats from ten Antarctic lakes // Syst. Appl. Microbiol. 2002. V. 25. P. 603–610.https://doi.org/10.1078/07232020260517742
Viana A T., Caetano T., Covas C., Santos T., Mendo S. Environmental superbugs: The case study of Pedobacter spp. // Environ. Pollut. 2018. V. 241. P. 1048–1055.https://doi.org/10.1016/j.envpol.2018.06.047
Vincent W.F., Whyte L.G., Lovejoy C., Greer C.W., Laurion I., Suttle C.A., Corbeil J., Mueller D.R. Arctic microbial ecosystems and impacts of extreme warming during the International Polar Year // Polar Sci. 2009. № 3. P. 171–180.
Yamada T., Sekiguchi Y., Hanada S., Imachi H., Ohashi A., Harada H., Kamataga Y. Anaerolinea thermolimosa sp. nov., Levilinea saccharolytica gen. nov., sp. nov. and Leptolinea tardivitalis gen. nov., sp. nov., novel filamentous anaerobes, and description of the new classes Anaerolineae classis nov. and Caldilineae classis nov. in the bacterial phylum Chloroflexi // Int. J. Syst. Evol. Microbiol. 2006. V. 56. P. 1331–1340.
Yamada T., Imachi H., Ohashi A., Harada H., Hanada S., Kamagata Y., Sekiguchi Y. Bellilinea caldifistulae gen. nov., sp. nov. and Longilinea arvoryzae gen. nov., sp. nov., strictly anaerobic, filamentous bacteria of the phylum Chloroflexi isolated from methanogenic propionate-degrading consortia // Int. J. Syst. Evol. Microbiol. 2007. V. 57. P. 2299–2306.https://doi.org/10.1099/ijs.0.65098-0
Zhang J., Kobert K., Stamatakis A. PEAR: a fast and accurate Illumina Paired-End read merger // Bioinformatics. 2014. V. 30. P. 614–620.
Дополнительные материалы отсутствуют.