Микробиология, 2019, T. 88, № 5, стр. 532-540
Анализ генных детерминант фототрофии у метанотрофных представителей Alphaproteobacteria
К. К. Мирошников a, С. Э. Белова a, С. Н. Дедыш a, *
a Институт микробиологии им. С.Н. Виноградского, Федеральный Исследовательский центр “Фундаментальные основы биотехнологии” РАН
119071 Москва, Россия
* E-mail: dedysh@mail.ru
Поступила в редакцию 25.03.2019
После доработки 23.04.2019
Принята к публикации 29.04.2019
Аннотация
Аэробные метанотрофные бактерии – экологически важная группа микроорганизмов, функционально специализированных на окислении парникового газа метана. Анализ накопленного за последние годы пула доступных геномных последовательностей метанотрофов позволил выявить ряд ранее скрытых метаболических возможностей этих бактерий. В их числе – выявление в геноме облигатного метанотрофа Methylocapsa palsarum NE2T кластера генов, кодирующего способность к аноксигенному фотосинтезу за счет функционирования фотосистемы II, характерной для пурпурных бактерий. Проведенный в настоящей работе расширенный поиск генетических детерминант фототрофии у метанотрофов подтвердил наличие таковых у ряда метанотрофных представителей Alphaproteobacteria, включая Methylocella silvestris BL2T и TVC, Methylocystis rosea SV97T и GW6, а также Methylocystis spp. штаммов SB2 и MitZ-2018. В геномах всех этих метанотрофов присутствует оперон pufABCLM, кодирующий белки светособирающего комплекса, наборы генов bch/chl, обеспечивающие биосинтез бактериохлорофилла, ген pucC, необходимый для транспорта хлорофиллов, а также гены crtFDC, crtL и crtB, ответственные за синтез каротиноидов. Последовательности генов в этих кластерах идентичны в пределах каждого из видов и близки у Methylocapsa и Methylocella. В геномах представителей Methylocystis наблюдается ряд перестановок, в том числе за счет инверсии группы генов, ответственных за биосинтез бактериохлорофилла и каротиноидов. Наличие генов pufLM выявлено и у нового изолята Methylocapsa palsarum, штамма NSB8, выделенного в настоящей работе из заболоченной тундры Европейского севера России. Наличие генов фототрофии в геномах всех штаммов вышеперечисленных видов свидетельствует об их функциональной значимости для этих бактерий и позволяет предположить реализацию способности к фототрофии в определенных условиях внешней среды.
Аэробные метанотрофные бактерии (метанотрофы) – экологически важная группа микроорганизмов, обеспечивающая биогенное окисление метана – второго по значимости парникового газа (Trotsenko, Murrell, 2008; Conrad, 2009). Подавляющее большинство известных метанотрофов используют исключительно С1 соединения в качестве источников углерода и энергии, однако известны и факультативные метанотрофы, способные использовать ряд органических кислот и спиртов (Dedysh et al., 2005; Dedysh, Dunfield, 2018), а также Н2 и СО2 (Mohammadi et al., 2017; Carere et al., 2017). Перечень ныне охарактеризованных метанотрофов включает представителей 23-х родов, принадлежащих к классам Alpha- и Gammaproteobacteria, также трех родов-кандидатов, относящихся к филе Verrucomicrobia (Dedysh, Knief, 2018). Введение геномики в практику микробиологических исследований позволило получить данные о геномном потенциале ряда метанотрофных бактерий и выявить ряд скрытых метаболических возможностей этих микроорганизмов. Примерами могут служить открытие изоформ ключевого фермента метанотрофов – метанмонооксигеназы (Baani, Liesack, 2008), альтернативных метанолдегидрогеназ (Keltjens et al., 2014) и гидрогеназ (Mohammadi et al., 2017; Carere et al., 2017) у метанотрофных бактерий.
Выполненный нами ранее анализ генома облигатного метанотрофа Methylocapsa palsarum NE2T, выделенного из тундровых почв Норвегии (Dedysh et al., 2015), выявил наличие генов, обеспечивающих способность к аноксигенному фото-синтезу за счет функционирования фотосистемы II, характерной для пурпурных бактерий (Miroshnikov et al., 2017). В их числе – гены, кодирующие светособирающий комплекс pufABCLM, реакционный центр puhA, а также гены, обеспечивающие синтез бактериохлорофиллов a и b и каротиноидов: спириллоксантина и гидроксисфероидена. Подобные кластеры генов были ранее выявлены у ряда факультативных метилотрофных бактерий рода Methylobacterium, населяющих филлосферу растений (Marx et al., 2012). Примечательно, что экспрессия этих генов и синтез бактериохлорофилла у представителей Methylobacterium были экспериментально подтверждены (Stiefel et al., 2013). Поиск аналогичных генов в других доступных на тот момент геномах метанотрофных бактерий показал их присутствие у Methylocella silvestris BL2T (Chen et al., 2010) и Methylocystis rosea SV97T (Wartiainen et al., 2006). Метанотрофы родов Methylocapsa, Methylocella и Methylocystis относятся к классу Alphaproteobacteria и являются типичными обитателями почв и болот бореальной и тундровой зон (Dedysh, 2009).
Так как способность метанотрофов к фототрофии до сих пор не была описана, данная работа ставила своей задачей поиск и филогенетический анализ генетических детерминант фототрофии у представителей метанотрофных бактерий и их сравнение с таковыми у филогенетически родственных фототрофов.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Объекты исследований. Объектами сравнительного анализа, проведенного в настоящей работе, были геномные последовательности следующих метанотрофных представителей класса Alphaproteobacteria: Methylocystis rosea штаммы SV97T (номер депонирования в NCBI GenBank GCA_000372845.1) и GW6 (GCA_003855495.1), Methylocella silvestris BL2T (GCA_000021745.1) и TVC (GCA_002891535.1) (Wang et al., 2018), Methylocapsa palsarum NE2T (GCA_900114285.1) (Miroshnikov et al., 2017), а также Methylocystis sp. SB2 (GCA_000499825.2) (Vorobev et al., 2014) и MitZ-2018 (GCA_003113245.1).
Поиск генов, детерминирующих способность к фототрофии. Поиск и сравнение последовательностей в базе данных NCBI GenBank осуществляли с использованием программы Blast (http:// blast.ncbi.nlm.nih.gov). Для поиска целевых генов в геномах метанотрофов в качестве референсных использовали последовательности соответствующих генов бактерий родов Rhodoblastus и Methylobacterium.
Анализ взаимного расположения детерминант фототрофии в геномах метанотрофных бактерий. Расположение целевых генов в геномах анализировали с помощью программного пакета UGENE (Okonechnikov et al., 2012). Сравнение организации кластеров генов проводили с помощью алгоритма BLAST, а визуализацию полученных результатов осуществляли с помощью программы Easyfig (Sullivan et al., 2011).
Анализ филогении генных детерминант фототрофии. Эволюционные связи генетических детерминант фототрофии у метанотрофов анализировали путем построения филогенетических деревьев на основании сравнительного анализа конкатенированных аминокислотных последовательностей белков реакционного центра фотосистемы II − Pu-fL и PufM. Массив последовательностей формировали на базе такового, опубликованного в работе Imhoff и соавторов (2018), путем дополнения его последовательностями из базы NCBI GenBank. Выравнивание последовательностей осуществляли с помощью алгоритма Clustal Omega (Sievers et al., 2011). Построение филогенетической дендрограммы проводили методом “максимального правдоподобия” с использованием программного пакета ARB (Ludwig et al., 2004).
Получение новых изолятов Methylocapsa palsarum с генными детерминантами фототрофии. Направленный поиск новых представителей этого вида метанотрофов проводили с использованием сфагнового торфа, отобранного из поверхностного слоя (глубина 4–8 см) болота субарктической тундры, Ямало-Ненецкий автономный округ, 65°35′00.1′′ N, 73°03′07′′ E. Посев торфяной суспензии проводили на агаризованную минеральную среду М2, с последующей инкубацией в эксикаторах с 20% метана в газовой фазе, как описано ранее (Belova et al., 2013). Идентификацию колоний метанотрофов проводили с помощью ПЦР-амплификации гена pmoA, кодирующего β‑субъединицу мембранной метанмонооксигеназы с использованием праймеров 189f и 682r (Holmes et al., 1995). Наличие генных детерминант фототрофии у полученных изолятов проверяли посредством ПЦР-амплификации фрагмента генов pufLM с использованием праймеров pu-fLf (Nagashima et al., 1997) и pufM750r (Achenbach et al., 2001). Определенные в настоящей работе последовательности генов pmoA и pufLM нового изолята Methylocapsa palsarum, штамма NSB8, депонированы в GenBank под номерами MK659581 и MK659582 соответственно.
Анализ распространения метанотрофов с фототрофным потенциалом. Распространение метанотрофов с фототрофным потенциалом в природных экосистемах анализировали путем поиска консервативных последовательностей РufM с уровнем сходства >86% с соответствующими последовательностями Methylocapsa palsarum NE2T в метагеномной базе данных IMG/M JGI (https:// img.jgi.doe.gov/). Для поиска использовали 50 пулов метагеномных последовательностей, полученных из наземных переувлажненных экосистем и пресноводных водоемов.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Гены, детерминирующие способность к фототрофии. Проведенный в настоящей работе поиск позволил существенно пополнить ранее опубликованный перечень метанотрофов с генными детерминантами фототрофии – Methylocapsa palsarum NE2T, Methylocella silvestris BL2T и Methylocystis rosea SV97T (Miroshnikov et al., 2017). К последнему был добавлен еще один представитель семейства Beijerinckiaceae – Methylocella silvestris TVC, а также три метанотрофа семейства Methylocystaceae – Methylocystis rosea GW6 и два штамма рода Methylocystis неустановленной видовой принадлежности – SB2 и MitZ-2018. Идентифицированные в геномах этих бактерий детерминанты фототрофии включали в себя оперон pufABCLM, кодирующий белки светособирающего комплекса, ген pucC, необходимый для транспорта хлорофиллов, наборы генов bch, обеспечивающие биосинтез бактериохлорофилла, а также гены crtFDC, crtL и crtB, ответственные за биосинтез каротиноидов (табл. 1). Интересной особенностью являлось наличие в геномах исследуемых метанотрофов двух паралогичных генов bchE и acsF (Pinta et al., 2002), позволяющих осуществлять синтез бактериохлорофиллов в аэробных и анаэробных условиях. В геномной последовательности Methylocystis sp. SB2 отсутствовали гены цепей светособирающих антенн, а оба штамма Methylocella silvestris утратили гены bchJ. Полный набор генов, аналогичный таковому у Methylocapsa palsarum NE2T и потенциально обеспечивающий функционирование фотосистемы II, был обнаружен в геномах штаммов вида Methylocystis rosea, а также Methylocystis sp. MitZ-2018.
Таблица 1.
Генные детерминанты фототрофии в геномах метанотрофных бактерий родов Methylocapsa, Methylocella и Methylocystis
| Вид | Methylocapsa palsarum |
Methylocella silvestris |
Methylocystis rosea |
Methylocystis sp. | ||||
|---|---|---|---|---|---|---|---|---|
| Штамм | NE2T | BL2T | TVC | SV97T | GW6 | SB2 | MitZ-2018 | |
| Биосинтез бактериохлорофилла | bchBNL | + | + | + | + | + | + | + |
| bchC | + | + | + | + | + | + | + | |
| bchE | + | – | – | + | + | + | + | |
| acsF | + | + | + | + | + | + | + | |
| bchF | + | – | – | + | + | + | + | |
| bchG | + | – | + | + | + | + | + | |
| bchIHOD | + | + | + | + | + | + | + | |
| bchJ | + | – | – | + | + | + | + | |
| bchM | + | + | + | + | + | + | + | |
| bchXYZ | + | + | + | + | + | + | + | |
| Синтез каротиноидов | crtB | + | + | + | + | + | + | + |
| crtC | + | + | + | + | + | + | + | |
| сrtD | + | + | + | + | + | + | + | |
| сrtF | + | + | + | + | + | + | + | |
| Гены фотосистемы II | pufAB | + | + | + | + | + | + | + |
| pufC | + | + | + | + | + | + | + | |
| pufLM | + | + | + | + | + | + | + | |
| puhA | + | + | + | + | + | + | + | |
| pucA | + | + | + | + | + | – | + | |
| pucB | + | + | + | + | + | – | + | |
Большинство генов, кодирующих способность к фототрофии, формировали единый кластер в геномах исследованных метанотрофов (рис. 1). Последовательность организации генов в этих кластерах была сходной в геномах Methylocapsa palsarum NE2T и штаммов Methylocella silvestris, тогда как в геномах представителей рода Methylocystis наблюдались некоторые перестановки за счет инверсии группы генов, ответственных за синтез бактериохлорофилла и каротиноидов. Подобные перестановки генов типичны для фототрофных бактерий, что свидетельствует об их достаточно высокой мобильности (Igarashi et al., 2001). В рамках исследуемого кластера наблюдались два участка с консервативным порядком генов. Первый из них включал гены pufABLMC, bchCXYZ и crtFDC, а второй – bchFNBHLM и pucC (рис. 1). Эта упорядоченность может быть связана с регуляцией транскрипции и трансляции этих генов.
Рис. 1.
Организация кластеров генов, кодирующих способность к аноксигенному фотосинтезу, у метанотрофных бактерий Methylocystis rosea GW6 (1), Methylocystis sp. Mitz-2018 (2), Methylocapsa palsarum NE2T (3), Methylocella silvestris TVC (4), а также филогенетически родственного фототрофа Rhodoblastus acidophilus DSM 137 (5). Цветовые обозначе-ния: А) гены, кодирующие фотосистему II, Б) гены биосинтеза каротиноидов, В) гены биосинтеза бактериохлорофилла.

В геномах метанотрофов класса Gammaproteobacteria и филы Verrucomicrobia гены, кодирующие способность к фототрофии, обнаружены не был-и.
Филогенетический анализ. Филогенетическое древо, отражающее результаты сравнительного анализа конкатенированных последовательностей белков PufLM исследуемых метанотрофов и гомологичных белков из неметанотрофных бактерий приведено на рис. 2. Для этого древа характерно наличие множества близкородственных кластеров, которые включают представителей различных семейств и даже порядков, что свидетельствует о высоком влиянии горизонтального переноса на эволюцию генов pufLM (Nagashima et al., 1997). Последовательности PufLM метанотрофов формировали на этом древе две группы: группу Methylocapsa–Methylocella и группу Methylocystis. Последовательности PufLM Methylocapsa palsarum NE2T и штаммов Methylocella silvestris обнаруживали высокое сходство (83–84% идентичности) с таковыми у Prosthecomicrobium hirschii, тогда как соответствующие белки представителей рода Methylocystis были наиболее близки (81–85% идентичности) таковым у метилотрофов рода Methylobacterium. Сообщение о наличии генных детерминант в геномах представителей Methylobacterium было опубликовано в 2012 году (Marx et al., 2012), тогда как их присутствие в геноме Prosthecomicrobium hirschii было обнаружено сравнительно недавно (Imhoff et al., 2018). Примечательно, что уровень сходства последовательностей PufLM Methylocapsa и Methylocella с таковыми у фототрофных представителей семейства Beijerinckiaceae, бактерий рода Rhodoblastus, был ниже и составлял лишь 74–76% (рис. 2). Таким образом, последовательности PufLM метанотрофов обнаруживали наибольшее филогенетическое родство с таковыми у аэробных фототрофных бактерий. Несмотря на высокое филогенетическое разнообразие последовательностей PufLM в пределах порядка Rhizobiales и их кластеризацию только на уровне рода (Imhoff et al., 2018), PufLM метанотрофных бактерий образуют на дереве всего два близко расположенных кластера. Это может говорить об общем происхождении и, возможно, общих принципах регуляции фотосинтетических генов у метанотрофных бактерий.
Рис. 2.
Филогенетическое древо, построенное на основании сравнительного анализа конкатенированных аминокислотных последовательностей РufLM метанотрофов родов Methylocystis, Methylocapsa, Methylocella (показаны жирным шрифтом) и других протеобактерий. Новый изолят Methylocapsa palsarum, выделенный в настоящей работе, отмечен звездочкой. Цифры в скобках обозначают число последовательностей в соответствующем кластере. Маркер, 0.1 замена на аминокислотную позицию.
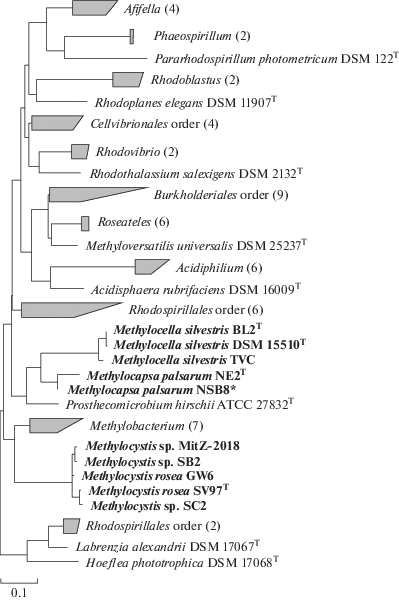
Получение нового изолята Methylocapsa palsarum с генными детерминантами фототрофии. Наличие генных детерминант фототрофии во всех ныне доступных геномах Methylocella silvestris и Methylocystis rosea позволяет предположить, что эта характеристика является видоспецифичной. Проверка справедливости такого предположения в отношении Methylocapsa palsarum была затруднена, так как этот вид был описан на основании только одного изолята, полученного из тундровой почвы Норвегии – штамма NE2T. В геномах других видов рода Methylocapsa – M. acidiphila и M. aurea – гены фотосистемы II отсутствуют. Для расширения спектра культивируемых представителей Methylocapsa palsarum в рамках настоящего исследования была проведена работа по получению новых изолятов этого вида метанотрофов из почвы заболоченной тундры Ханты-Мансийского АО. Полученный в результате этой работы изолят, штамм NSB8, обладал морфологическими и фенотипическими характеристиками, присущими метанотрофам вида Methylocapsa palsarum (Dedysh et al., 2015), обнаруживал высокое сходство (97.7% идентичности нуклеотидных последовательностей) фрагмента pmoA с таковым у штамма NE2T и был идентифицирован в качестве нового представителя этого вида. Фрагмент генов pufLM ожидаемой длины (~1500 нт) был успешно амплифицирован из ДНК штамма NSB8. Определение транслированной аминокислотной последовательности этого фрагмента показало ее высокое сходство (99% идентичности) с таковой у Methylocapsa palsarum NE2T (рис. 2). Наличие генов pufLM в геномах представителей этого вида, выделенных из географически отдаленных экосистем, позволяет предположить, что генные детерминанты фототрофии характерны для Methylocapsa palsarum и появились на уровне диверсификации вида.
Поиск фрагментов PufM, гомологичных таковым у Methylocapsa и Methylocella, в метагеномных базах данных. Для оценки широты распространения метанотрофов с генными детерминантами фототрофии нами был проведен поиск последовательностей PufM, гомологичных таковой у Methylocapsa palsarum NE2T, в метагеномах базы данных IMG. В результате этого поиска в метагеномах ряда переувлажненных экосистем Северной Америки было выявлено 54 последовательности, обнаруживающих более 86% идентичности с последовательностью PufM Methylocapsa palsarum NE2T. Этот уровень сходства характерен для кластера, содержащего последовательности PufML представителей родов Methylocapsa и Methylocella (рис. 2). Соответственно, найденные последовательности, вероятнее всего, принадлежат представителям этих двух родов метанотрофов. Из выявленных последовательностей, 16 обнаруживали более 90% идентичности с последовательностью PufM Methylocapsa palsarum NE2T (табл. 2) и с высокой степенью вероятности принадлежали представителям этого рода. Таким образом, результаты проведенного скрининга метагеномов свидетельствуют о широком распространении метанотрофов родов Methylocapsa и Methylocella с генными детерминантами фототрофии.
Таблица 2.
Фрагменты гена pufM, гомологичные таковому у Methylocapsa palsarum, в пулах метагеномных последовательностей базы IMG. В таблице представлены последовательности, обнаруживающие >90% идентичности с PufM Methylocapsa palsarum
| id гена | id генома | Место отбора проб | Уровень сходства | Длина |
|---|---|---|---|---|
| Ga0062381_100162653 | 3300004808 | Wetland sediment microbial communities from St. Louis River estuary, USA | 97% | 64 |
| Ga0209464_110620301 | 3300027778 | Wetland sediment microbial communities from St. Louis River estuary, USA | 96% | 72 |
| Ga0209397_121208741 | 3300027871 | Wetland microbial communities from Old Woman Creek Reserve in Ohio, USA | 92% | 99 |
| Ga0062381_129177731 | 3300004808 | Wetland sediment microbial communities from St. Louis River estuary, USA | 92% | 75 |
| Ga0209397_121208741 | 3300027871 | Wetland microbial communities from Old Woman Creek Reserve in Ohio | 92% | 99 |
| Ga0209293_125213841 | 3300027877 | Wetland microbial communities from Old Woman Creek Reserve in Ohio | 91% | 94 |
| Ga0062383_141038431 | 3300004778 | Wetland sediment microbial communities from St. Louis River estuary, USA | 91% | 66 |
| Ga0062383_137503791 | 3300004778 | Wetland sediment microbial communities from St. Louis River estuary, USA | 91% | 70 |
| Ga0062381_119755711 | 3300004808 | Wetland sediment microbial communities from St. Louis River estuary, USA | 91% | 87 |
| Ga0115028_120734551 | 3300009179 | Wetland microbial communities from Old Woman Creek Reserve in Ohio, USA | 91% | 168 |
| Ga0209464_105490842 | 3300027778 | Wetland sediment microbial communities from St. Louis River estuary, USA | 91% | 84 |
| Ga0209683_112251811 | 3300027840 | Wetland sediment microbial communities from St. Louis River estuary, USA, under dissolved organic matter induced mercury methylation – T4Bare2Fresh (SPAdes) | 91% | 112 |
| JGI20214J51650_111083381 | 3300003541 | Wetland sediment microbial communities from Twitchell Island in the Sacramento Delta | 90% | 183 |
| Ga0209682_106156472 | 3300027716 | Wetland sediment microbial communities from St. Louis River estuary, USA | 90% | 40 |
| Ga0062380_118035531 | 3300004779 | Wetland sediment microbial communities from St. Louis River estuary, USA | 90% | 64 |
| Ga0209496_130660761 | 3300027890 | Wetland microbial communities from Old Woman Creek Reserve in Ohio, USA | 90% | 96 |
Происхождение генов фототрофии в геномах ряда метанотрофов остается неясным. Примечательно, что в геномах метанотрофов видов Methylocapsa palsarum, Methylocella silvestris и Methylocystis rosea присутствует вся совокупность генов, необходимых для осуществления аноксигенной фототрофии, в то время как у других видов тех же родов она отсутствует. На сегодняшний день существует две гипотезы об эволюции генов аноксигенной фототрофии у бактерий. Первая гипотеза предполагает высокое влияние горизонтального переноса на распространение и эволюцию генов фототрофии (Nagashima et al., 1997; Igarashi et al., 2001). В соответствии с этой гипотезой метанотрофы могли получить способность к использованию энергии света от фототрофных представителей порядков Rhizobiales или Rhodospirillales (рис. 2). Аргументом в пользу этой гипотезы служат данные о высоком уровне кластеризации генов и частоте перестановок генов в рамках кластера. Вторая гипотеза предполагает раннее разделение групп фототрофных бактерий, с последующей независимой эволюцией соответствующих генов в пределах каждой из групп (Im-hoff et al., 2018). В рамках второго сценария, некоторые представители родов Methylocapsa, Methylocella и Methylocystis за счет специализации на метанотрофии могли утратить кластер генов фототрофии, присутствовавший у общего предка. Эта гипотеза подкрепляется высоким сходством организации кластеров генных детерминант фо-тотрофии в пределах отдельных видов метано-трофных представителей семейства Beijerinckiaceae и их отличием от таковых у Methylocystaceae.
Примечательно, что гены, кодирующие способность к фототрофии, были выявлены только у метанотрофов класса Alphaproteobacteria, многие из которых отличаются метаболической пластичностью и способны использовать не только С1 соединения, но и ряд других субстратов (Dedysh, Dunfield, 2018). Наиболее широким спектром используемых субстратов отличаются метанотрофы рода Methylocella (Dedysh et al., 2005; Dunfield, Dedysh, 2014). Некоторые представители родов Methylocystis и Methylocapsa в отсутствие метана способны к слабому росту на ацетате и этаноле (Dedysh, Dunfield, 2018). Недавно было показано, что метанотрофы рода Methylocapsa способны также к росту за счет использования метана в атмосферных концентрациях и ряда других атмосферных газов, таких как Н2, СО, N2 (Tveit et al., 2019). Таким образом, в качестве потенциальных доноров электронов для фотосинтеза у метанотрофов Alphaproteobacteria могут рассматриваться используемые ими органические соединения или водород. Возможно, способность к фототрофии является дополнительным источником энергии для метанотрофов в условиях отсутствия основного субстрата этих микроорганизмов – метана, или же позволяет взаимодействовать с растениями по аналогии с представителями рода Methylobacterium (Atamna-Ismaeel et al., 2012). Возможность реализации геномного потенциала к использованию энергии света, равно как и функциональная значимость фототрофии в арсе-нале экологических адаптаций метанотрофных бактерий заслуживают дальнейших исследований.
Список литературы
Achenbach L.A., Carey J., Madigan M.T. Photosynthetic and phylogenetic primers for detection of anoxygenic phototrophs in natural environments // Appl. Environ. Microbiol. 2001. V. 67. № 7. P. 2922–2926.
Atamna-Ismaeel N., Finkel O., Glaser F., von Mering C., Vorholt J.A., Koblížek M., Belkin S., Béjà O. Bacterial anoxygenic photosynthesis on plant leaf surfaces // Environ. Microbiol. Reports. 2012. V. 4. № 2. P. 209–216.
Baani M., Liesack W. Two isozymes of particulate methane monooxygenase with different methane oxidation kinetics are found in Methylocystis sp. strain SC2 // Proc. Natl. Acad. Sci. U. S. A. 2008. V. 105. № 29. P. 10203–10208.
Belova S.E., Kulichevskaya I.S., Bodelier P.L., Dedysh S.N. Methylocystis bryophila sp. nov., a facultatively methanotrophic bacterium from acidic Sphagnum peat, and emended description of the genus Methylocystis (ex Whittenbury et al., 1970) Bowman et al., 1993 // Int. J. Syst. Evol. Microbiol. 2013. V. 63. № 3. P. 1096–1104.
Carere C.R., Hards K., Houghton K.M., Power J.F., McDonald B., Collet C., Gapes D.J., Sparling R., Boyd E.S., Cook G.M., Greening C., Stott M.B. Mixotrophy drives niche expansion of verrucomicrobial methanotrophs // ISME J. 2017. V. 11. P. 2599–2610.
Chen Y., Crombie A., Rahman M. T., Dedysh S.N., Liesack W., Stott M.B., Alam M., Theisen A.R., Murrell J.C., Dunfield P.F. Complete genome sequence of the aerobic facultative methanotroph Methylocella silvestris BL2 // J. Bacteriol. 2010. V. 192. № 14. P. 3840–3841.
Conrad R. The global methane cycle: recent advances in understanding the microbial processes involved // Environ. Microbiol. Rep. 2009. V. 1. № 5. P. 285–292.
Dedysh S.N. Exploring methanotroph diversity in acidic northern wetlands: Molecular and cultivation-based studies // Microbiology (Moscow). 2009. V. 78. № 6. P. 655–669.
Dedysh S. N., Dunfield P.F. Facultative methane oxidizers // Taxonomy, Genomics and Ecophysiology of Hydrocarbon-Degrading Microbes. Handbook of Hydrocarbon and Lipid Microbiology / Ed. McGenity T.G. Springer, Cham., 2018. P. 1–20.
Dedysh S.N., Knief C., Dunfield P.F. Methylocella species are facultatively methanotrophic // J. Bacteriol. 2005. V. 187. P. 4665–4670.
Dedysh S.N., Didriksen A., Danilova O. V., Belova S.E., Liebner S., Svenning, M.M. Methylocapsa palsarum sp. nov., a methanotroph isolated from a subArctic discontinuous permafrost ecosystem // Int. J. Syst. Evol. Microbiol. 2015. V. 5. № 10. P. 3618–3624.
Dedysh S.N., Knief C. Diversity and phylogeny of described aerobic methanotrophs // Methane Biocatalysis: Paving the Way to Sustainability / Eds. Kalyuzhnaya M.G., Xing X.-H. Springer, Cham., 2018. P. 17–42.
Dunfield P.F., Dedysh S.N. Methylocella: a gourmand among methanotrophs // Trends Microbiol.. 2014. V. 22(7). P. 368–369.
Holmes A.J., Costello A., Lidstrom M.E., Murrell J.C. Evidence that participate methane monooxygenase and ammonia monooxygenase may be evolutionarily related // FEMS Microbiol. Lett . 1995. V. 132. № 3. P. 203–208.
Igarashi N., Harada J., Nagashima S., Matsuura K., Shimada K., Nagashima K.V. Horizontal transfer of the photosynthesis gene cluster and operon rearrangement in purple bacteria // J. Mol. Evol . 2001. V. 52. № 4. P. 333–341.
Imhoff J.F., Rahn T., Künzel S., Neulinger S.C. Photosynthesis is widely distributed among Proteobacteria as demonstrated by the phylogeny of PufLM reaction center proteins // Front. Microbiol. 2018. V. 8. Art. 2679. https://doi.org/10.3389/fmicb.2017.02679
Keltjens J.T., Pol A., Reimann J., den Camp H. J. O. PQQ-dependent methanol dehydrogenases: rare-earth elements make a difference // Appl. Microbiol. Biotechnol.. 2014. V. 98. № 14. P. 6163–6183.
Ludwig W., Strunk O., Westram R., Richter L., Meier H., Yadhukumar, Buchner A., Lai T, Steppi S., Jobb G., Förster W., Brettske I., Gerber S., Ginhart A.W., Gross O., Grumann S., Hermann S., Jost R., König A., Liss T., Lüßmann R., May M., Nonhoff B., Reichel B., Strehlow R., Stamatakis A., Stuckmann N., Vilbig A., Lenke M., Ludwig T., Bode A., Schleifer K-H. ARB: a software environment for sequence data // Nucleic Acids Res. 2004. V. 32. № 4. P. 1363–1371.
Marx C.J., Bringel F., Chistoserdova L., Moulin L., Haque M.F.U., Fleischman D.E., Gruffaz C., Jourand P., Knief C., Lee M.C., Muller E.E., Nadalig T., Peyraud R., Roselli S., Russ L., Goodwin L.A., Ivanova N., Kyrpides N., Lajus A., Land M.L., Mйdigue C., Mikhailova N., Nolan M., Woyke T., Stolyar S., Vorholt J.A., Vuilleumier S. Complete genome sequences of six strains of the genus Methylobacterium // J. Bacteriol. 2012. V. 194. P. 4746–4748.
Miroshnikov K.K., Didriksen A., Naumoff D.G., Huntemann M., Clum A., Pillay M., Palaniappan K., Varghese N., Mikhailova N., Mukherjee S., Reddy T.B.K., Daum C., Shapiro N., Ivanova N., Kyrpides N., Woyke T., Dedysh S.N., Svenning M.M. Draft genome sequence of Methylocapsa palsarum NE2T, an obligate methanotroph from subarctic soil // Genome Announc. 2017. V. 5. № 24. P. e00504–17.
Mohammadi S., Pol A., van Alen T.A., Jetten M.S., den Camp H.J.O. Methylacidiphilum fumariolicum SolV, a thermoacidophilic ‘Knallgas’ methanotroph with both an oxygen-sensitive and-insensitive hydrogenase // ISME J. 2017. V. 11. № 4. P. 945–958.
Nagashima K.V., Hiraishi A., Shimada K., Matsuura K. Horizontal transfer of genes coding for the photosynthetic reaction centers of purple bacteria // J. Mol. Evol. 1997. V. 45. № 2. P. 131–136.
Okonechnikov K., Golosova O., Fursov M., Ugene Team. Unipro UGENE: a unified bioinformatics toolkit // Bioinformatics. 2012. V. 28. №. 8. P. 1166–1167.
Pinta V., Picaud M., Reiss-Husson F., Astier C. Rubrivivax gelatinosus acsF (previously orf358) codes for a conserved, putative binuclear-iron-cluster-containing protein involved in aerobic oxidative cyclization of Mg-protoporphyrin IX monomethylester // J. Bacteriol. 2002. V. 184. № 3. P. 746–753.
Stiefel P., Zambelli T., Vorholt J.A. Isolation of optically targeted single bacteria by application of fluidic force microscopy to aerobic anoxygenic phototrophs from the phyllosphere //Appl. Environ. Microbiol. 2013. V. 79. № 16. P. 4895–4905.
Sievers F., Wilm A., Dineen D., Gibson T. J., Karplus K., Li W., Lopez R., McWilliam H., Remmert M., Söding J., Thompson J.D., Higgins D.G. Fast, scalable generation of high quality protein multiple sequence alignments using Clustal Omega // Mol. Syst. Biol. 2011. V. 7. Art. 539. https://doi.org/10.1038/msb.2011.75
Sullivan M.J., Petty N.K., Beatson S.A. Easyfig: a genome comparison visualizer // Bioinformatics. 2011. V. 27. № 7. P. 1009–1010.
Tveit A.T., Hestnes A.G., Robinson S., Schintlmeister A., Dedsyh S.N., Jehmlich N., von Bergen M., Herbold C., Wagner M., Richter A., Svenning M.M. Widespread soil bacterium that oxidizes atmospheric methane // Proc. Nat. Acad. Sci. USA. 2019. https://doi.org/10.1073/pnas.1817812116
Vorobev A. Jagadevan S., Jain S., Anantharaman K., Dick G.J., Vuilleumier S., Semrau J.D. Genomic and transcriptomic analyses of the facultative methanotroph Methylocystis sp. strain SB2 grown on methane or ethanol // Appl. Environ. Microbiol. 2014. V. 80. № 10. P. 3044–3052.
Wang J., Geng K., Haque M.F.U., Crombie A., Street L.E., Wookey P.A., Ma K., Murrell J.C., Pratscher J. Draft genome sequence of Methylocella silvestris TVC, a facultative methanotroph isolated from permafrost // Genome Announc. 2018. V. 6. № 8. P. e00040-1.
Wartiainen I., Hestnes A.G., McDonald I.R., Svenning M.M. Methylocystis rosea sp. nov., a novel methanotrophic bacterium from Arctic wetland soil, Svalbard, Norway (78 N) // J. Syst. Evol. Microbiol. 2006. V. 56. № 3. P. 541–547.
Дополнительные материалы отсутствуют.


