Микробиология, 2019, T. 88, № 6, стр. 730-734
Бактериофаги группы Т5 как потенциальные агенты фаготерапии
Е. Е. Куликов a, А. К. Голомидова a, *, В. В. Морозова c, Ю. Н. Козлова c, А. В. Летаров a, b
a Институт микробиологии им. С.Н. Виноградского, Федеральный исследовательский центр
“Фундаментальные основы биотехнологии” РАН
119071 Москва, Россия
b Московский государственный университет им. М.В. Ломоносова,
биологический факультет
119192 Москва, Россия
c Институт химической биологии и фундаментальной медицины
Сибирского отделения РАН
630090 Новосибирск, Россия
* E-mail: golomidova_alla@mail.ru
Поступила в редакцию 20.06.2019
После доработки 21.07.2019
Принята к публикации 29.07.2019
Аннотация
Бактериофаги группы Т5, инфицирующие большое количество видов и штаммов энтеробактерий, являются удобными объектами для получения терапевтических фаговых препаратов с широкой специфичностью. В данной работе рассматриваются особенности биологии фагов, родственных Т5, позволяющие им инфицировать широкий спектр штаммов бактерий, несмотря на весьма ограниченное количество известных белков – рецепторов фага. Способность бактериофагов специфически распознавать О-антиген бактерии значительно расширяет способность фага к инфекции и повышает ценность фагов группы Т5 для фаготерапии.
T5-подобные бактериофаги являются перспективными кандидатами в качестве агентов для фаговой терапии инфекций, вызываемых энтеробактериями. Их частицы устойчивы к различным условиям окружающей среды, а сами фаги не имеют генов потенциальных факторов патогенности, являются вирулентными и обладают широким хозяйским спектром (Sváb et al., 2018). В недавних исследованиях было предложено несколько T5-подобных бактериофагов в качестве потенциальных кандидатов для использования против различных патогенных видов сем. Enterobacteriaceae, включая Salmonella enterica serovar Typhimurium (Grover et al., 2015; Piya et al., 2015), штаммы Escherichia coli O157:H7 (Niu et al., 2012), с перспективными результатами применения T5‑подобного бактериофага CEV2 в комплексе с T4-подобным фагом CEV1 in vivo, полученными на овцах (Raya et al., 2011).
Установленными конечными рецепторами фага Т5 и родственных ему бактериофагов являются крупные трансмембранные белки-транспортеры внешней мембраны энтеробактерий, участвующие в переносе ионов d-металлов (железа, кобальта) в составе белковых и порфириновых комплексов – BtuB, FhuA и FepA (McCorquodale, Warner, 1988). За распознавание этих рецепторов и связывание с ними отвечает вирионный белок pb5 (Flayhan et al., 2012). Целью работы была экспериментальная оценка способности бактериофагов группы Т5 к инфекции широкого спектра различных штаммов и видов энтеробактерий, благодаря способности этих фагов распознавать различные серотипы бактериального О-антигена и различные белки внешней мембраны. В связи с ограниченным числом известных конечных клеточных рецепторов, узнаваемых фаговым белком pb5, для фагов группы Т5 особую актуальность приобретают исследования природного биоразнообразия Т5-подобных фагов с целью поиска новых типов pb5, узнающих другие рецепторы, и идентификация этих рецепторов.
Культивирование бактерий и бактериофагов, включая определение титров фага, проводили классическими методами, описанными в работе (Golomidova et al., 2016). Выделение и анализ липополисахаридов бактерий проводили методом ферментативного лизиса и окрашивания серебром в геле, описанном в работе (Kulikov et al., 2019). Для биоинформатического анализа были использованы программы пакета DNAStar Lasergene.
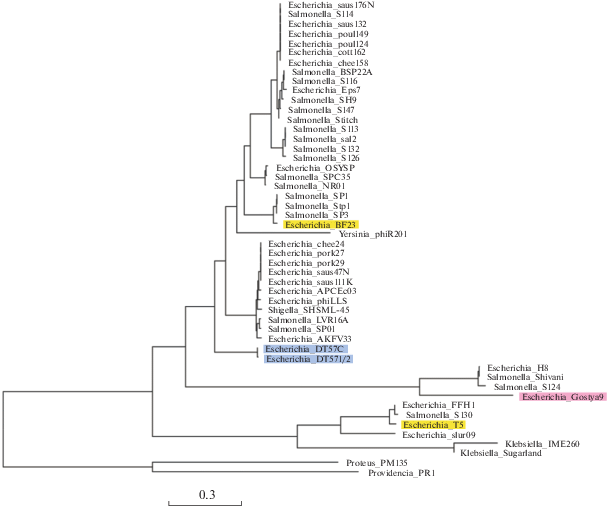
На основе анализа аминокислотных последовательностей рецептор-узнающего белка pb5 бактериофагов, родственных Т5, нами было построено филогенетическое дерево (рис. 1), состоящее из восьми основных кластеров, предположительно соответствующих бактериофагам с предположительно различными типами узнаваемого конечного рецептора. Это группа фагов Т5 (рецептор FhuA), BF23 (BtuB), H8 (FepA), группа, состоящая из фагов Proteus и Providencia и группа из фагов Klebsiella. В кластер фага H8 входит также и недавно описанный нами Т5-подобный бактериофаг Gostya9 (Golomidova et al., 2018). Рецептор этого фага пока не идентифицирован, но по имеющимся косвенным данным он может отличаться от белка FepA. Рецепторы последних двух групп также пока не известны. Несмотря на наблюдаемую кластеризацию, нет полной уверенности, что типов узнаваемых последовательностей действительно восемь, так как, например, достаточно удаленные друг от друга бактериофаги BF23 и DT57C/DT571/2 узнают один и тот же конечный рецептор – BtuB. При этом нужно отметить, что последовательности распознаваемых Т5-подобными фагами белков весьма консервативны у энтеробактерий (данные не приведены). Поэтому нескольких вариантов специфичности к конечному рецептору, вероятно, должно быть достаточно для успешной инфекции подавляющего большинства штаммов, особенно для такого вида, как E. coli.
Рис. 1.
Филогенетическое дерево аминокислотных последовательностей рецептор-узнающего белка pb5 различных бактериофагов группы Т5 (программа MegAlign, алгоритм neighbor-joining). Цветом выделены типовые представители отдельных кластеров.
Слой O-АГ, находящийся на поверхности бактериальной клетки, эффективно экранирует поверхность внешнего листка мембраны с конечными рецепторами от проникновения вируса (Broeker, Barbirz, 2017). Поэтому первичный и обратимый этап взаимодействия фага с клеткой обычно включает в себя транзитное взаимодействие белков адсорбционного аппарата фага с О-антигеном. Ранее мы показали, что выделенные нами T5-подобные бактериофаги DT57C и DT571/2 осуществляют такое взаимодействие с поверхностью клетки за счет разветвленных латеральных хвостовых фибрилл (lateral tail fibers, LTF), выполняющих функцию распознавания поверхностного О-антигена (ОПС) клетки бактерии-хозяина (Golomidova et al., 2016). Эти фибриллы состоят из двух белков – LtfA и LtfB. Мишенями для рецептор-узнающих доменов белков LtfA бактериофагов DT57C и DT571/2 являются О-полисахариды штаммов E. coli 4s – O22-подобные (для фага DT57C) или HS1/2 серотипа O87 (для фага DT571/2), а для белка LtfB – О-полисахарид серотипа О81 штамма HS3-104 (для обоих фагов).
Структура О-антигена может достаточно быстро изменяться благодаря различным мутациям и адаптациям белков, отвечающих за синтез О-антигена, что позволяет бактериям лучше приспосабливаться к существующему селективному давлению (DebRoy et al., 2018). По существующим на сегодня данным рецептор-узнающие домены LTF Т5-подобных фагов специфичны по отношению к одному серотипу О-АГ, либо по отношению к нескольким, очень близким в структурном отношении. Это ограничивает спектр хозяев каждого конкретного фага и затрудняет задачу разработки систем инженерии спектра хозяев на основе Т5‑подобных фагов. Мы исследовали шесть выделенных нами T5-подобных фагов на способность инфицировать различные штаммы энтеробактерий (64 штамма, выделенных из фекалий лошади и других природных объектов). Набор из шести бактериофагов (T5, DT57C, DT571/2, Gostya9, Shivani, CF17) оказался способен лизировать 30% исследованных бактериальных штаммов. В результате работы было показано, что T5-подобные бактериофаги способны к специфическому распознаванию различных серогрупп О-АГ энтеробактерий. Так, бактериофаг DT751/2 лизировал шесть штаммов E. coli (два из них серогрупп O87 и O81), DT57C – пять штаммов E. coli (два из них O22 и O81), CF17 – один штамм E. coli, два штамма Citrobacter spp., Shivani – один штамм Salmonella enterica, два штамма E. coli, T5 – два штамма E. coli, Gostya9 – 1 штамм E. coli (серогруппа О28).
Бактериофаг PM135 (GenBank NC_042090.1), также родственный Т5, был изолирован из сточных вод с использованием выделенного в клинике бактериального штамма Proteus mirabilis. Спектр штаммов-хозяев бактериофага был определен с использованием штаммов Proteus spp. (P. mirabilis, P. vulgaris, P. penneri). В результате было выявлено, что бактериофаг PM135 способен размножаться на 7 штаммах из 32 протестированных (данные не приведены). Из трех видов Proteus spp. фаг был не способен инфицировать только P. penneri, инфицировал же, преимущественно, штаммы P. mirabilis. Бактериофаг PM135 обладает неразветвленной латеральной хвостовой фибриллой (LTF; FDJ15_gp021) и рецептор-связывающим белком (pb5; FDJ15_gp003).
Судя по тому, что известных и предположительных вариантов фаговых конечных рецепторов оказывается весьма ограниченное количество, основным механизмом, лимитирующим фаговую инфекцию в случае Т5-подобных бактериофагов, оказывается экранирование поверхности клетки О-АГ. Ранее было показано, что бактериофаг Т5 специфически распознает полиманнозные О-АГ серогрупп О8 и О9, при этом в распознавании участвуют LTF (Heller, Braun, 1982). Для выделенных нами бактериофагов DT57C и DT571/2 была показана специфичность фагов с разветвленными LTF к трем различным вариантам О-АГ, при этом каждый компонент разветвленной фибриллы связывался с определенным вариантом О-АГ (Golomidova et al., 2016).
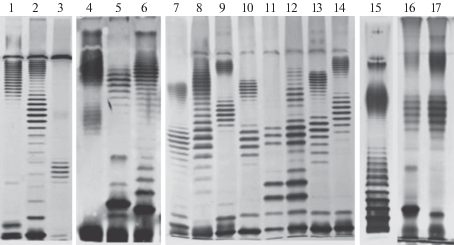
Для чувствительных к фагу штаммов нами был проведен анализ фракции липополисахаридов с помощью электрофореза в ДСН-ПААГ, результаты которого указывают на различия в паттернах подвижности ЛПС у бактерий, чувствительных к одному фагу (рис. 2). Таким образом, гипотеза о возможной полиспецифичности фаговых LTF получает экспериментальную поддержку. Обнаруженная нами способность бактериофагов группы Т5 специфически распознавать широкий спектр О-АГ бактерий кишечной группы и преодолевать барьер О-АГ указывает на то, что от фагов этой группы можно ожидать высокой эффективности при терапевтическом применении. Наличие в структуре вириона Т5-подобных фагов различных вариантов LTF, способных распознавать разнообразные варианты О-АГ, открывает возможности для направленного изменения специфичности бактериофагов группы Т5. Понимание принципов и характера взаимодействий, определяющих эффективность неспецифической защиты клеток бактерий от фагов, а также знание стратегий, которые фаги используют для преодоления этой защиты, позволит разработать требования к составу эффективных терапевтических коктейлей бактериофагов, а также выработать подходы к усилению действия фаговых препаратов.
Рис. 2.
Электрофореграммы липополисахаридов различных штаммов E. coli, поддерживающих рост бактериофагов группы Т5. Дорожки 1–3 – бактериофаг Shivani (штаммы SE147, A16, A13); 4–6 – бактериофаг CF17 (штаммы 1772, 1955, 2847); 7–8 – бактериофаг DT571/2 (штаммы HS1/2 (серогруппа O87), A11); 9–13 – бактериофаги DT571/2 и DT57C (A18, HS3/104, M21, M24, P1); 14 – бактериофаг DT57C (4S (O22-подобный серотип)); 15 – бактериофаг Gostya9 (штаммы F5 (серогруппа O28)); 16–17 – бактериофаг T5 (штаммы S1, S3). Видно, что паттерны ЛПС штаммов, инфицируемых одним и тем же фагом, способны значительно различаться.
Список литературы
Broeker N.K., Barbirz S. Not a barrier but a key: How bacteriophages exploit host’s O-antigen as an essential receptor to initiate infection // Mol. Microbiol. 2017. V. 105. P. 353–357.
DebRoy C., Fratamico P.M., Roberts E. Molecular serogrouping of Escherichia coli // Anim. Health Res. Rev. 2018. V. 19. P. 1–16. https://doi.org/10.1017/S1466252317000093
Flayhan A., Wien F., Paternostre M., Boulanger P., Breyton C. New insights into pb5, the receptor binding protein of bacteriophage T5, and its interaction with its Escherichia coli receptor FhuA // Biochimie. 2012. V. 94. P. 1982–1989.
Golomidova A.K., Kulikov E.E., Babenko V.V., Ivanov P.A., Prokhorov N.S., Letarov A.V. Escherichia coli bacteriophage Gostya9, representing a new species within the genus T5virus // Arch. Virol. 2019. V. 164. P. 879–884. https://doi.org/10.1007/s00705-018-4113-2
Golomidova A.K., Kulikov E.E., Prokhorov N.S., Guerrero-Ferreira R.C., Knirel Y.A., Kostryukova E.S., Tarasyan K.K., Letarov A.V. Branched lateral tail fiber organization in T5‑like bacteriophages DT57C and DT571/2 is revealed by genetic and functional analysis // Viruses. 2016. V. 8. pii: E26. https://doi.org/10.3390/v8010026
Grover J.M., Luna A.J., Wood T.L., Chamakura K.R., Kuty Everett G.F. Complete genome of Salmonella enterica serovar Typhimurium T5-like Siphophage Stitch // Genome Announc. 2015. V. 3. e01435-14. https://doi.org/10.1128/genomeA.01435-14
Heller K., Braun V. Polymannose O-antigens of Escherichia coli, the binding sites for the reversible adsorption of bacteriophage T5+ via the L-shaped tail fibers // J. Virol. 1982. V. 41. P. 222–227.
Kulikov E.E., Golomidova A.K., Prokhorov N.S., Ivanov P.A., Letarov A.V. High-throughput LPS profiling as a tool for revealing of bacteriophage infection strategies // Sci. Rep. 2019. V. 9. P. 2958.
McCorquodale D.J., Warner H.R. Bacteriophage T5 and related phages // The Bacteriophages / Ed. Calendar R. Boston, MA: Springer, 1988. V. 1. P. 439–475.
Niu Y.D., Stanford K., Kropinski A.M., Ackermann H.W., Johnson R.P., She Y.M., Ahmed R., Villegas A., McAllister T.A. Genomic, proteomic and physiological characterization of a T5-like bacteriophage for control of Shiga toxin-producing Escherichia coli O157:H7 // PLoS One. 2012. V. 7. P. e34585.
Piya D., Xie Y., Hernandez Morales A.C., Kuty Everett G.F. Complete genome sequence of Salmonella enterica serovar Typhimurium siphophage Shivani // Genome Announc. 2015. V. 3. https://doi.org/10.1128/genomeA.01443-14
Raya R.R., Oot R.A., Moore-Maley B., Wieland S., Callaway T.R., Kutter E.M., Brabban A.D. Naturally resident and exogenously applied T4-like and T5-like bacteriophages can reduce Escherichia coli O157:H7 levels in sheep guts // Bacteriophage. 2011. V. 1. P. 15–24.
Sváb D., Falgenhauer L., Rohde M., Szabó J., Chakraborty T., Tóth I. Identification and characterization of T5-like bacteriophages representing two novel subgroups from food products // Front Microbiol. 2018. V. 9. P. 202. https://doi.org/10.3389/fmicb.2018.00202
Дополнительные материалы отсутствуют.


