Микробиология, 2020, T. 89, № 1, стр. 99-110
Филогенетическое разнообразие микробных сообществ с поверхности полиэтилентерефталатных материалов при экспозиции в водных средах
Т. П. Турова a, *, Д. Ш. Соколова a, Т. Н. Назина a, Д. С. Груздев a, А. Б. Лаптев b
a Институт микробиологии им. С.Н. Виноградского, Федеральный исследовательский центр
“Фундаментальные основы биотехнологии” Российской академии наук
119071 Москва, Россия
b Федеральное государственное унитарное предприятие “Всероссийский научно-исследовательский институт авиационных материалов”
105005 Москва, Россия
* E-mail: tptour@rambler.ru
Поступила в редакцию 22.05.2019
После доработки 20.06.2019
Принята к публикации 29.09.2019
Аннотация
Целью настоящей работы было исследование состава сообществ микробных обрастаний образцов полиэтилентерефталатных (ПЭТФ) материалов, помещенных в водные среды различного происхождения (промышленные, морские, пресные) в разных климатических регионах. Методом высокопроизводительного секвенирования V3–V4 региона гена 16S рРНК показано доминирование представителей филумов Bacteroidetes, Gammaproteobacteria и Alphaproteobacteria во всех трех группах исследуемых образцов. Однако количественное соотношение как доминирующих, так и минорных филумов заметно различалось для каждой из групп полученных библиотек, а внутри групп менялось в зависимости от времени экспозиции в водных средах и проведения антимикробной обработки. На родовом уровне также заметно различались не только группы библиотек из образцов различного происхождения, но и отдельные сообщества внутри групп. С помощью программы iVikodak были предсказаны функциональные характеристики прокариот в полученных библиотеках гена 16S рРНК. Показано, что деградацию образцов ПЭТФ потенциально способны осуществлять микробные сообщества промышленных вод, в которых in silico обнаружены представители родов Pseudomonas и Acidovorax, возможные деструкторы ПЭТФ, обладающие ферментами катаболизма промежуточного метаболита этого процесса – терефталевой кислоты. Микрофотографии подтвердили локальное разрушение исходных образцов ПЭТФ в результате экспозиции в промышленных водах, в то время как разрушений образцов ПЭТФ в морской воде обнаружено не было.
На сегодняшний день создано большое количество полимерных материалов, и их преимущество очевидно: они почти не подвергаются биодеградации, не коррозируют под действием атмосферы, имеют высокие потребительские свойства, механические характеристики и низкую стоимость. Тем не менее, биостойкость пластика может вызывать серьезные экологические проблемы (Derraik, 2002; Thompson et al., 2004; Moore, 2008; Law et al., 2010).
С другой стороны, наиболее важным этапом при создании той или иной техники с использованием полимерных материалов является представлениеоб особенностях ее климатического исполнения. Поэтому предварительно определяется, при каком воздействии климатических, химических и биологических факторов может работать материал или изделие в течение назначенного периода эксплуатации (Каблов, 2012; Каблов, Старцев, 2015). Обоснованное планирование климатического исполнения изделий позволяет не только обеспечить их безопасную эксплуатацию в заданный период времени, но и значительно снизить затраты на ремонт и восстановление после поломок и разрушений элементов сложных технических систем при воздействии различных внешних факторов (Лаптев и соавт., 2018).
Изменение климата, увеличение эмиссии углекислого газа приводит к увеличению жизнеспособности и изменчивости микроорганизмов, что в последнее время инициирует более интенсивную деструкцию микроорганизмами высокомолекулярных синтетических материалов. На современном этапе развития техники и технологий одной из важных составляющих в их разработке и применении является проверка функциональных материалов на стойкость к микробиологическому воздействию.
Известно, что механизм микробного воздействия на полимерные материалы может включать в себя как непосредственное использование пластика в качестве источника углерода и энергии, так и влияние продуктов жизнедеятельности микроорганизмов (кислот, экзоферментов, биомассы и др.) на материал (Ghosh et al., 2013).
Одним из этапов в решении проблемы биоповреждений полимеров может являться изучение видового состава микробного сообщества, исследование экологических и физиолого-биохимических особенностей потенциальных деструкторов, определение возможных биохимических путей разрушения полимеров.
Полиэтилентерефталат (ПЭТФ) – полукристаллический полимер, относится к группе алифатически-ароматических полиэфиров, химически и термически стабилен. В России ПЭТФ используется для производства пищевых пленок и изготовления пластиковых бутылок для газированных напитков. Кроме того, полиэфирные материалы, к которым относится ПЭТФ, благодаря своим инновационным характеристикам, нашли широкое применение во многих отраслях. Наиболее часто они используются в строительстве и ремонте с использованием искусственного камня, в судостроении для создания биостойких корпусных деталей из композитных полимерных материалов, в качестве ремонтных компаундов в мото- и автомобилестроении. По химическому строению ПЭТФ представляет собой сложный термопластичный полиэфир терефталевой кислоты и этиленгликоля и является продуктом нефтехимического синтеза.
В немногочисленных публикациях отмечено разложение ПЭТФ бактериями родов Bacillus, Brevibacillus, Nocardia, Pseudomonas, Thermomonospora, Streptomyces и Ideonella (Kleeberg et al., 1998, 2005; Hadad et al., 2005; Ronkvist et al., 2009; Ribitsch et al., 2011; Sharon, Sharon, 2012; Acero et al., 2013; Miyakawa et al., 2015; Liu et al., 2019). Липазы (эстеразы), гидрогеназы и кутиназы, выделяемые микроорганизмами, являются ключевыми ферментами в процессе разрушения длинноцепочечных полиэфирных материалов (Sauvageau, 2004; Ronkvist et al., 2009). Однако пока только для бактерии Ideonella sakaiensis (Tanasupawat et al., 2016) наиболее достоверно показан путь разрушения ПЭТФ, включая данные о генетических детерминантах ключевых ферментов этого процесса, а также данные о структуре и активности этих ферментов (Yoshida et al., 2016; Liu et al., 2019). Согласно этим исследованиям, предполагаемый механизм разрушения ПЭТФ бактерией I. sakaiensis (а возможно, и другими бактериями-деструкторами ПЭТФ) приведен на рисунке S1 . Внеклеточный фермент ПЭТаза (фермент, гидролизующий ПЭТ, поли(этилен терефталат)гидролаза – EC 3.1.1.101) разлагает ПЭТФ до моно(2-гидроксиэтил)терефталевой кислоты (основной продукт) и терефталевой кислоты (минорный компонент). Моно(2-гидроксиэтил)терефталевая кислота гидролизуется МГЕТазой (моно(этилен терефталат)гидролазой – EC 3.1.1.102) до терефталевой кислоты и этиленгликоля. Терефталевая кислота доставляется в клетку с помощью специального белка-переносчика и последовательно катаболизируется двумя ферментами: 1,2-диоксигеназой терефталевой кислоты (EC 1.14.12.15) и 1,2-дигидрокси-3,5-циклогексадиен-1,4-дикарбоксилат дегидрогеназой (EC 1.3.1.53) до протокатеховой кислоты. Затем специальная 3,4-диоксигеназа (EC 1.11.13.) разрушает ароматическое кольцо протокатеховой кислоты (Yoshida et al., 2016).
Целью работы было определение филогенетического разнообразия бактерий методом высокопроизводительного секвенирования V3–V4 региона гена 16S рРНК в микробных обрастаниях, сформировавшихся на образцах полиэтилентерефталатных материалов, помещенных в водные среды (промышленные оборотные воды, морская вода и пресноводный водоем) в разных климатических регионах, а также in silico поиск ферментов биодеградации ПЭТФ у выявленных бактерий.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Подготовка образцов. Для определения биостойкости ПЭТФ, специально подготовленные образцы материала размером 100 мм × 10 мм × 2 мм помещали в водные среды на глубину 1 м в разных регионах России (г. Уфа, г. Геленджик (район Тонкого мыса), г. Можайск). Условия экспозиции образцов отличались температурой и минерализацией воды. В Уфе образцы помещали в водоблоки с пресной оборотной водой промышленного назначения завода “Уфаоргсинтез” (образцы U1P‒U4P – группа “industrial”). Для оборотной воды характерно постоянство температуры 24 ± 2°C, обусловленное технологическим процессом. В Геленджике образцы экспонировали в соленой морской воде Черного моря (образцы G1P‒G4P – группа “marine”) в период с 1 августа по 30 сентября 2018 г. Средняя, минимальная и максимальная тем-пература морской воды в Геленджике в августе 2018 г. составляла 26.8, 26.0 и 27.8°C соответственно, в сентябре – 25.1, 22.8 и 26.7°C соответственно (http://russia.pogoda360.ru/794677/). На глубине 1 м, в зоне экспонирования образцов ПЭТФ температура воды в этот период изменялась в более широком интервале от 34 до 18°C. В Подмосковье образец помещали в пресноводное Можайское водохранилище в районе д. Горетово (образец M1P – “fresh”) и инкубировали 10 сут (с 1 по 10 августа 2018 г.) при температуре воды от 17 до 23°C (http://weatherarchive.ru/Temperature/Mozhaysk/ August-2018). Образцы U1P, U2P, U3P и U4P инкубировали, соответственно, 10, 20, 40 и 60 сут, как и образцы G1P‒G4P.
На заводском водоблоке для корректировки роста микроорганизмов используется обработка химическими реагентами. При превышении численности микроорганизмов в воде свыше 105 кл./мл подается 10% щелочной раствор гипохлорита натрия, который обладает антисептическим и дезинфицирующим действием и широко используется в качестве бытового и промышленного дезинфектанта, средства очистки и обеззараживания воды. Образцы U2P‒U4P через 20 сут экспонирования, в соответствии с программой корректировки, одновременно подверглись такой обработке. Однако образец U2P после обработки сразу был извлечен, а образцы U3P и U4P еще на 20 и 40 сут оставлены в водной среде.
Химический состав водных сред измеряли однократно в начале эксперимента, согласно описанному в работе (Резников и соавт., 1970). Измерения проводили в трех повторностях.
Выделение ДНК. Биомассу с поверхности образцов ПЭФТ смывали этанолом (1 : 1, об./об.) и фильтровали через мембранный фильтр с диаметром пор 0.22 мкм (“Millipore”, США). Клеточная суспензия была высушена, обработана раствором, содержащим 0.15 M NaCl и 0.1 M Na2EDTA (pH 8.0), и использована для выделения ДНК с применением стандартного метода (Maniatis et al., 1984).
Высокопроизводительное секвенирование фрагментов гена 16S рРНК. C использованием ДНК, выделенной из обрастаний на образцах ПЭТФ, и специфичных праймеров Pro341F–Pro805R (Takahashi et al., 2014) был амплифицирован V3–V4 гипервариабельный регион гена 16S рРНК. Фрагменты генов 16S рРНК на матрице ДНК из каждого образца были амплифицированы в трех повторностях, которые затем были объединены и очищены с помощью электрофореза в 2% агарозном геле и набора реагентов Cleanup Standard gel extraction kit (“Евроген”, Россия). Высокопроизводительное секвенирование проводили на системе MiSeq (“Illumina”, США) c использованием набора реагентов MiSeq Reagent Kit v3 (600 cycles) (“Illumina”, США) в соответствии с рекомендациями производителя.
Биоинформатический анализ. Секвенированные фрагменты генов 16S рРНК были обрезаны с помощью программы trimmomatic 0.36 по алгоритму SLIDINGWINDOW: 4:15 (Bolger et al., 2014), правосторонние и левосторонние прочтения были объединены с помощью программы Se-qPrep (https://github.com/jstjohn/SeqPrep). Далее обработку последовательностей проводили при помощи пакета программ QIIME (Caporaso et al., 2010), что включало в себя демультиплексирование и фильтрацию химер. Библиотеки бактериального сообщества создавали с использованием QIIME и онлайн-ресурса SILVA (https:// www.arb-silva.de/ngs/). Для анализа библиотек с помощью QIIME последовательности предварительно объединяли в операционные таксономические единицы (OTE) c уровнем сходства 98%, которые затем идентифицировали с использованием онлайн-ресурса RDP (http://pyro.cme.msu.edu). Филогенетическое дерево, включающее 25 наиболее обильных ОТЕ, было построено с использованием пакета программ MEGA 7.0 (Kumar et al., 2016) на основании neighbor-joining алгоритма, при этом ближайшие референтные последовательности генов 16S рРНК были получены с использованием классификатора онлайн-ресурса SILVA.
Библиотеки фрагментов генов 16S рРНК бактерий из микробных обрастаний образцов ПЭТФ, полученные в результате секвенирования, депонированы в NCBI SRA под номером PRJNA545821.
Статистический анализ. Построение кривых разреженности осуществляли с использованием пакета программ QIIME. Индексы разнообразия считали, используя программу EstimateS (http:// purl.oclc.org/estimates). Для создания диаграмм Венна использовали онлайн-ресурс Venny (http:// bioinfogp.cnb.csic.es/tools/venny/). Онлайн-ресурс ClustVis (https://biit.cs.ut.ee/clustvis/) был использован для создания графика Principal Component Analysis (PCA) и тепловых карт (heatmaps) 50-ти наиболее обильных членов сообщества на родовом уровне. Используя данные о представленности таксонов в библиотеках и наиболее значимые различия условий экспозиции исследуемых образцов, были выявлены предполагаемые физиолого-биохимические характеристики бактериальных сообществ с использованием пакета программ iVikodak (Nagpal et al., 2019). Созданные по результатам этого анализа тепловые карты были построены с помощью онлайн-ресурса ClustVis.
Микроскопические методы. При исследовании бактериальных обрастаний и поверхности образцов ПЭТФ применяли сканирующий микроскоп TESCAN VEGA 3 XMU (Чехия) и систему энергодисперсионного микроанализа (ADVANCED AZTEC Energy, включая Inca Energy 350) на базе безазотного энергодисперсионного детектора X-MAX 50 STANDARD (“Oxford Instruments NanoAnalysis”, Великобритания).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Физико-химические условия водной среды. Химический состав, температура и pH водных сред, в которых экспонировались образцы ПЭТФ, приведены в табл. 1. Общая соленость, жесткость и минерализация вод промышленного происхождения (группа образцов “industrial”) заметно превышали аналогичные показатели чистой пресной воды Можайского водохранилища (образец “fresh”), хотя и не достигали значений морской воды (группа “marine”). Температура в процессе экспозиции существенно менялась (уменьшалась) только для морских образцов (группы “marine”); величина рН пресной воды составляла 7.4, а пробы промышленной воды были более щелочными и имели рН 8.3.
Таблица 1.
Физико-химические параметры водных сред, в которых экспонировали образцы ПЭТФ*
| Местообитание/сезон экспозиции | Общая соленость, г/л | Жесткость, мг/л | pH | Температура воды, °С | Содержание ионов, мг/л | |||
|---|---|---|---|---|---|---|---|---|
| Са2+ | Mg2+ | ${\text{SO}}_{4}^{{2 - }}$ | Cl– | |||||
| Чeрное море в районе г. Геленджик, август‒сентябрь (образцы G1P‒G4P) | 18.6 ± 0.1 | 65.6 ± 0.2 | 7.8 ± 0.1 | 26.8–25.1 | 250.9 | 660.9 | 1331.1 | 9818.5 |
| Пресная вода Можайского водохранилища, август (образец М1P) | 0.09–0.5 | 4.5 ± 0.2 | 7.4 ± 01 | 17–23 | 60.0 | 15.8 | 13.5 | 255.0 |
| Промышленная вода, г. Уфа, август‒сентябрь (образцы U1P‒U4P) | 1.3 ± 0.1 | 8.5 ± 0.2 | 8.3 ± 0.1 | 24 ± 2 | 160.4 | 56.2 | 358.0 | 183.0 |
Статистический анализ разнообразия сообществ бактерий на образцах ПЭТФ. В результате применения высокопроизводительного секвенирования V3–V4 региона гена 16S рРНК были получены библиотеки фрагментов гена 16S рРНК для каждой из 9 проб бактериальных обрастаний на подложке ПЭТФ. С использованием онлайн-ресурса SILVA были выявлены неидентифицированные последовательности (риды), которые были исключены из дальнейшего анализа. Кроме того, были удалены единичные риды (синглтоны), представленные только в одной из анализируемых библиотек. Оставшееся общее количество ридов для 9 библиотек составило 88583, из которых было сформировано 1028 ОТЕ с уровнем сходства последовательностей ≥98%.
Распределение ридов и ОТЕ по 9 библиотекам представлено в табл. 2. Кривые насыщения для всех библиотек были близки к максимальным значениям выхода на плато, что свидетельствовало о высоком уровне покрытия всех библиотек (рис. S2 ). Индексы разнообразия (табл. 2) оказались различными для библиотек из разных мест экспонирования образцов ПЭТФ. Для библиотек, полученных из образцов ПЭТФ, экспонировавшихся в воде Черного моря (группа “marine”) и в пресной воде Можайского водохранилища (“fresh”), индексы разнообразия Шеннона‒ У-ивера и Симпсона оказались высокими, а индексы доминирования Бергера‒Паркера, наоборот, низкими. Причем для библиотек группы “marine” в зависимости от времени экспонирования наблюдалось уменьшение индексов разнообразия и, соответственно, увеличение индексов доминирования. Это свидетельствует о постепенном отборе микрофлоры, наиболее приспособленной для роста в составе биопленки на поверхности ПЭТФ-подложки. Возможно, в этой группе образцов на отбор бактерий влияло также заметное понижение температуры морской воды. Для библиотек группы “industrial” индексы примерно такого же уровня наблюдались только для U1P, полученной из образца, экспонированного в промышленной воде только 10 сут и не подвергавшегося обработке антимикробным химическим реагентом. В библиотеке U2P, полученной из образца, экспонированного 20 сут и извлеченного из воды сразу после обработки, наблюдалось резкое падение индексов разнообразия и, наоборот, значительное увеличение индекса доминирования преобладающего филотипа до 70%, по-видимому, наиболее устойчивого к антимикробной обработке. Для библиотек образцов ПЭТФ U3P и U4P, оставленных в водной среде после обработки, наблюдалось постепенное увеличение индексов разнообразия и уменьшения индексов доминирования, однако значений, характерных для начала экспонирования, они так и не достигли. Таким образом, после химической обработки прежний уровень разнообразия микробного сообщества образцов ПЭТФ не восстановился, несмотря на длительный срок последующего экспонирования.
Таблица 2.
Индексы разнообразия библиотек фрагментов гена 16S рРНК из бактериальных обрастаний на поверхности подложек из ПЭТФ, экспонированных в водных средах
| Параметры | Библиотеки фрагментов гена 16S рРНК | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| Образец | U1P | U2P | U3P | U4P | G1P | G2P | G3P | G4P | M1P |
| Среда/местообитание | Промышленная вода (оборотная вода заводского водоблока, г. Уфа) | Морская вода (Черное море, Геленжик) | Пресная вода (Можайское водохранилище) | ||||||
| Время экспозиции, сут | 10 | 20 | 40 | 60 | 10 | 20 | 40 | 60 | 10 |
| Количество сиквенсов (ридов) | 12 421 | 16 647 | 13 690 | 10 001 | 5688 | 4338 | 5351 | 10 881 | 9568 |
| Количество ОТЕ | 365 | 256 | 226 | 281 | 266 | 284 | 192 | 256 | 469 |
| Индекс разнообразия Шеннона‒Уивера (H) | 4.7 | 1.8 | 3.3 | 4.1 | 4.8 | 4.8 | 4.4 | 4.3 | 5.0 |
| Обратный индекс разнообразия Симпсона (1/S) | 53.4 | 2.0 | 8.6 | 20.9 | 72.9 | 59.6 | 45.2 | 30.3 | 57.3 |
| Выровненность (H/Hmax) | 0.80 | 0.32 | 0.61 | 0.73 | 0.85 | 0.85 | 0.83 | 0.77 | 0.82 |
| Индекс доминирования Бергера‒Паркера (D) | 0.08 | 0.70 | 0.30 | 0.15 | 0.04 | 0.06 | 0.07 | 0.13 | 0.03 |
Для определения различий между исследуемыми библиотеками в отношении количества ОТЕ были построены диаграммы Венна. Соотношение между филотипами отдельных библиотек групп “industrial” и “marine” показано на рис. 1а и 1в. Среди библиотек группы “industrial” наибольшее разнообразие и количество ОТЕ наблюдалось для библиотеки U1P, она же содержала наибольшее количество уникальных филотипов (17.6%). Наименьшим было разнообразие в библиотеке U2P, содержащей, в основном, общие для всей группы филотипы и минимальное количество уникальных филотипов (2.3%). Всего общих филотипов для группы “industrial” оказалось 32.6%, а библиотеки U3P и U4P содержали только 4.1 и 4.5% уникальных филотипов соответственно. Таким образом, антимикробная обработка, проведенная после 20 сут экспозиции, вероятно, способствовала отбору бактерий, более приспособленных к условиям существования в промышленной оборотной воде.
Рис. 1.
Диаграммы Венна, показывающие различия количества ОТЕ между исследуемыми библиотеками бактерий, полученных с поверхности образцов ПЭТФ в трех различных местообитаниях. Соотношение между количеством ОТЕ в отдельных библиотеках групп “industrial” (a) и “marine” (б); соотношение количества ОТЕ для групп “industrial”, “marine” и образца “fresh” (в). Интенсивность цвета соответствует количеству ОТЕ в сегменте диаграммы.
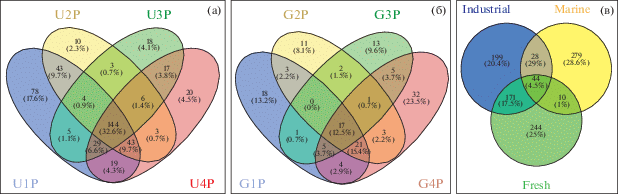
Библиотеки группы “marine” показали примерно одинаковое разнообразие в отношении количества ОТЕ, при этом количество уникальных для каждой библиотеки филотипов варьировало в пределах 2.2–8.3%, а количество общих филотипов составило 39.6%.
На диаграмме (рис. 1с) приведено количественное соотношение общих филотипов для групп “industrial”, “marine” и образца “fresh”. Ко-личество общих филотипов в библиотеках всех 3-х групп было незначительным (4.5%), при этом наибольшее количество уникальных филотипов по отношению к двум другим группам было представлено в библиотеках группы “marine”. В то же время, количество общих филотипов в библиотеках группы “industrial” и образца “fresh” было значительно выше (17.5%), чем их обоих с библиотеками группы “marine” (1.0–2.9%). Это свидетельствует о том, что количественный состав ОТЕ в библиотеках бактериальных сообществ образцов ПЭТФ, экспонированных в пресной (как чистой, так и оборотной) и в морской воде, заметно различается.
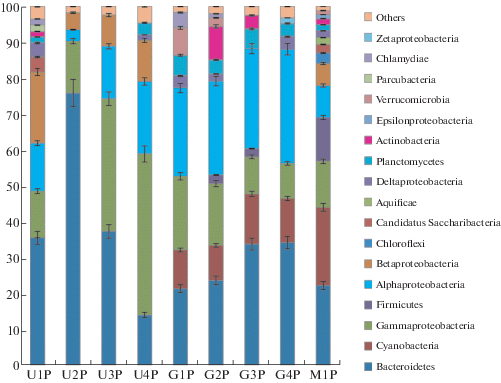
Филогенетическое разнообразие сообществ бактерий на образцах ПЭТФ. Во всех анализируемых библиотеках фрагментов генов 16S рРНК, полученных в результате высокопроизводительного секвенирования, были выявлены только бактерии, а архейный компонент не был обнаружен. Таксономическое распределение филотипов бактерий на уровне филумов, а для Proteobacteria – на уровне классов, показано на рис. 2. Представленными во всех анализируемых библиотеках и при этом доминирующими в большинстве из них оказались бактерии филумов Bacteroidetes (13.3–75.8%), Gammaproteobacteria (13.0–44.9%) и Alphaproteobacteria (3.2–31.8%). Однако в библиотеках групп “marine” и “industrial” наблюдались изменения долей этих доминирующих филумов в разные периоды экспозиции. В библиотеках группы “marine” при увеличении времени экспозиции возрастала доля Bacteroidetes (с 21.3 до 34.1%) и Alphaproteobacteria (с 24.6 до 31.8%), но уменьшалась доля Gammaproteobacteria (с 20.5 до 9.7%). В библиотеках группы “industrial” изменения соотношения доминирующих филумов были связаны как с продолжительностью экспозиции, так и с обработкой промышленной воды антимикробным реагентом. Это привело к увеличению последовательностей Bacteroidetes в библиотеке образца U2P, по сравнению с таковой U1P (с 35.5 до 75.8%), снижению доли последовательностей Alphaproteobacteria (c 13.3 до 3.2%), а доля Gammaproteobacteria практически не изменялась (13.0–14.4%). При дальнейшем экспонировании образцов U3P и U4P происходил обратный процесс: уменьшение доли Bacteroidetes (до 14.0%) и увеличение доли Alp-haproteobacteria (до 20.2%), а также значительное увеличение доли Gammaproteobacteria (до 44.9%) в библиотеках. Такая же закономерность отмечена и для последовательностей представителей класса Betaproteobacteria, которые в заметном количестве (4.8–19.8%) выявлены только в библиотеках образцов группы “industrial”. В библиотеке образца “fresh” их доля была незначительной (6.1%), а в библиотеках группы “marine” они встречались только как минорные компоненты сообщества (<1%). Исследуемые библиотеки также различались наличием последовательностей представителей филума Cyanobacteria, которые полностью отсутствовали в группе “industrial”, составляли заметную долю в группе “marine” (9.8–14.0%) и становились одним из доминантных компонентов в образце “fresh” (21.8%). В качестве минорных компонентов (<1%) в анализируемых сообществах встречались представители филумов Planctomycetes, Actinobacteria, Firmicutes, Deltaproteobacteria, Verrucomicrobia, Candidatus Saccharibacteria, Chlamydiae, Chloroflexi, Epsilonproteobacteria, Parcubacteria и Zetaproteobacteria.
Рис. 2.
Таксономическая принадлежность фрагментов генов 16S рРНК бактерий в библиотеках с поверхности образцов ПЭТФ на уровне филумов (для филума Proteobacteria на уровне классов), определенная с помощью RDP классификатора.
Группирование библиотек на родовом уровне приведено на графике PCA, построенном с помощью программы ClustVis (рис. S3 ) для 50 наиболее обильных родов. Результаты графика PCA свидетельствуют о том, что бактериальные сообщества исследуемых групп образцов достоверно различаются между собой на родовом уровне.
Таксономический состав 50 наиболее обильных родов в исследуемых сообществах представлен на тепловой карте, также сформированной в программе ClustVis (рис. 3), и в табл. S1 . Он значительно различался как между группами, так и в отдельных библиотеках внутри групп. В целом, наиболее разнообразными оказались представители филума Bacteroidetes, но состав их был различен для каждой из библиотек. В группе “industrial” наиболее разнообразной являлась библиотека U1P, где доминировали последовательности бактерий родов Chryseolinea (Bacteroidetes), Aquabacterium, Comamonas (Betaproteobacteria), Bdellovibrio (Deltaproteobacteria) и филума Candidatus Saccharibacteria. В библиотеке U2P сообщества образца, подвергнутого антимикробной обработке, отмечено резкое обеднение таксономического состава. Основным компонентом сообщества (около 70% от общего количества) стали некультивируемые и неидентифицированные бактерии филума Bacteroidetes, относящиеся к одному филотипу, вероятно, наиболее устойчивые к воздействию примененного антимикробного реагента. В образцах U3P и U4P при продолжении экспозиции произошло восстановление разнообразия таксономического состава бактериальных сообществ на родовом уровне, хотя этот состав изменялся в течение экспозиции. В библиотеке U3P преобладали последовательности бактерий родов Chryseobacterium (Bacteroidetes), Rheinheimera, Paraperlucidibaca (Gammaproteobacteria) и Acidovorax (Betaproteobacteria). В библиотеке U4P наблюдалось увеличение доли последовательностей Arenimonas и Pseudomonas (Gammaproteobacteria).
Рис. 3.
Тепловая карта (heatmap) распределения 50 доминантных родов по 9 библиотекам из образцов ПЭТФ. Сравнительное количество фрагментов последовательностей генов 16S рРНК бактерий определенных родов в библиотеках показано цветом. Представленность родов рассчитана как отношение количества ридов, принадлежащих представителям данного рода, к общему количеству ридов в каждой библиотеке.
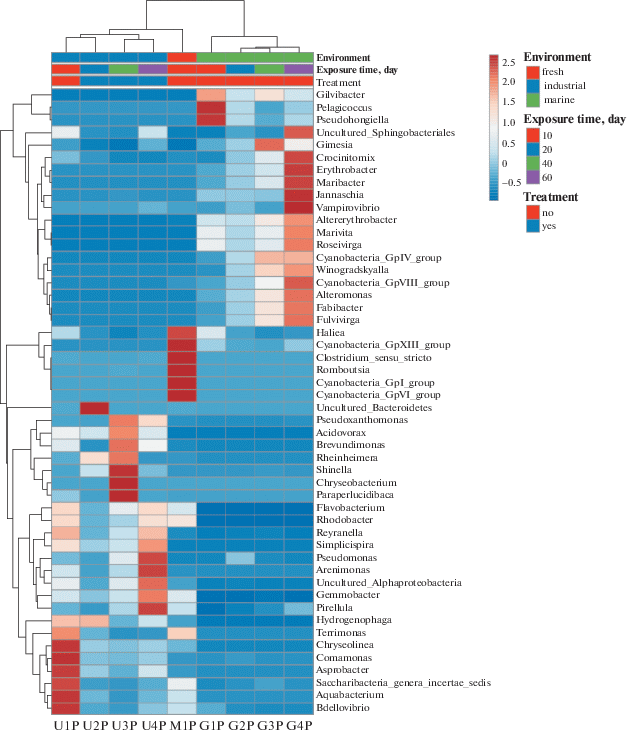
Таким образом, из списка родов бактерий, по данным литературы разрушающих ПЭТФ, в группе “industrial” выявлен только род Pseudomonas, представители которого встречаются во всех библиотеках группы, а доля их существенно увеличивалась к концу экспозиции, что, возможно, связано с участием их в процессе разрушения ПЭТФ. В группах образцов “marine” и “fresh” представители родов, содержащих предполагаемых бактерий-деструкторов ПЭТФ, не обнаружены. При этом в таксономическом составе группы “marine” на родовом уровне не отмечено заметного доминирования отдельных родов, как в группе “industrial”, кроме значительного увеличения к концу экспозиции бактерий рода Erythrobacter (Alphaproteobacteria) и неидентифицированных представителей филума Cyanobacteria. В бактериальном сообществе образца “fresh”, экспонировавшегося в воде Можайского водохранилища, преобладали фотосинтезирующие цианобактерии, а также представители родов Romboutsia и Clostridium (Firmicutes), которые в остальных сообществах практически отсутствовали.
Видовой состав изучаемых микробных сообществ был частично проанализирован в результате построения филогенетического дерева 25-ти наиболее обильных ОТЕ (рис. S4 ). В результате оказалось, что большинство представленных на дереве преобладающих ОТЕ, объединяющих более 100 ридов, являются компонентами библиотек группы “industrial”. Основными представленными на дереве филумами, как и в описанных выше анализах, оказались Bacteroidetes, Alpha- и Gammaproteobacteria, а также Betaproteobacteria и Cyanobacteria. Бактерия I. sakaiensis, у которой подробно изучен механизм деградации ПЭТФ, в составе исследуемых сообществ не обнаружена. Однако два филотипа в библиотеках сообществ группы “industrial” принадлежали бактерии близкого вида Acidovorax delafieldii, для которой показано наличие фермента, последовательности которого высоко гомологичны ПЭТазе I. sakaiensis (Joo et al., 2018). Возможно, что выявленные филотипы A. delafieldii принадлежат новым бактериям-деструкторам ПЭТФ.
Потенциальная способность изучаемых бактериальных сообществ к деградации ПЭТФ. Программа iVikodak позволяет на основании таксономического состава исследуемых бактериальных сообществ предсказывать их функциональные характеристики, используя геномные базы данных. На рис. S5 c использованием модуля программы “Global Mapper” представлен результат сравнительного анализа вклада исследуемых бактериальных сообществ в осуществление основных путей метаболизма согласно базе KEGG (Kanehisa, Goto, 2000). Поскольку карта пути деградации ПЭТФ в базе данных KEGG отсутствует, в анализ были включены, кроме основных путей метаболизма, большинство путей деградации различных ксенобиотиков, представленных в этой базе и выявленных программой в изучаемых сообществах. Согласно полученным результатам, наибольшим потенциальным функциональным разнообразием отличались сообщества образцов группы “industrial” U1P, U3P и U4P, что согласуется с большим таксономическим разнообразием этих библиотек и присутствием в их составе большинства наиболее обильных ОТЕ. Бактериальные сообщества образца U2P группы “industrial”, всех образцов группы “marine” и образца M1P “fresh” обладали меньшей потенциальной способностью в осуществлении анализируемых основных путей метаболизма. При анализе сообществ U1P, U3P и U4P группы “industrial” также была выявлена их потенциальная способность осуществлять большинство путей деградации ксенобиотиков, за исключением деградации гликозаминогликанов и атразина сообществом образца U2P, пути деградации бисфенола сообществами образцов группы “marine” и пути деградации нитротолуола сообществом M1P образца “fresh”.
Хотя путь деградации ПЭТФ в базе данных KEGG пока отсутствует, в составе пути “Деградация полициклических ароматических углеводородов” (“Polycyclic aromatic hydrocarbon degradation”) представлены реакции катаболизма терефталевой кислоты – основного промежуточного метаболита деградации ПЭТФ. Поэтому с помощью следующего модуля программы “Local Mapper” был проведен анализ ферментов пути “Деградация полициклических ароматических углеводородов”, возможно, образуемых исследуемыми природными сообществами. В результате этого анализа было предсказано образование двух ферментов, катаболизирующих терефталевую кислоту до протокатеховой кислоты сообществами группы “industrial”. Причем эти ферменты были наиболее обильно представлены в образце U1P и в меньшей степени обнаружены в образцах U3P и U4P (рис. S6 ). Эти результаты подтверждают потенциальную возможность деградации бактериями группы “industrial” промежуточного метаболита разрушения ПЭТФ – терефталевой кислоты.
Этот же модуль программы iVicodak позволяет оценить вклад бактерий различных таксонов в осуществление анализируемого пути метаболизма. Для пути “Деградация полициклических ароматических углеводородов” наиболее представленными были гаммапротеобактерии родов Pseudomonas и Aeromonas, а также бетапротеобактерии Acidovorax и Hydrogenophaga (рис. S7 а); в группе “marine” – альфапротеобактерии Erythrobacter, Marivita и Altererythrobacter (рис. S7 в); в образце “fresh” – бетапротеобактерии Aquabacterium (рис. S7 с). Поскольку, согласно литературным данным, именно для представителей рода Pseudomonas показана способность к деградации ПЭТФ, возможно, они и/или бактерии рода Acidovorax, близкие к I. sakaiensis, могут быть деструкторами ПЭТФ в исследуемых сообществах группы “industrial”.
Однако в образце U1P сообщества группы “industrial”, хотя и в качестве минорного компонента (до 2.1%), присутствовали также представители рода Comamonas; для некоторых штаммов этого рода описан процесс деградации 4-толуолкарбоксилата, в котором тоже образуется терефталевая кислота как промежуточный метаболит (Schlaveli et al., 1994; Sasoh et al., 2004). Этот путь не представлен в базе данных KEGG, однако в нем участвует тот же набор ферментов двойной функциональности (1.1.1.257, 1.2.1.62 и 1.14.1.28), осуществляющих путь деградации 4-метилбензойной кислоты, который в базе KEGG является частью пути “Деградация толуола” (“Toluene degradation”). Однако, согласно результатам использования модуля “Local Mapper” программы iVicodak, у представителей анализируемых сообществ этот набор ферментов отсутствует (рис. S8 ). Таким образом, наиболее вероятно, что терефталевая кислота, ферменты катаболизма которой обнаружены in silico у представителей исследуемых сообществ, действительно является промежуточным метаболитом деградации ПЭТФ.
Микроскопия микробных обрастаний на образцах ПЭТФ. Для визуализации микробных обрастаний на поверхности образцов ПЭТФ, а также для возможного обнаружения деструкции образцов после экспозиции в водной среде и последующей отмывки от микробных обрастаний был проведен микроскопический анализ. Микробные обрастания на образце U1P группы “industrial”, который экспонировался самое короткое время и не подвергался антимикробной обработке, представлены рыхлыми конгломератами бактериальных колоний, а структурированные микроэукариоты единичны (рис. 4а, 4б). При этом биопленка не покрывала всю поверхность образца, и видны участки исходного ПЭТФ. На микрофотографии этого же образца, отмытого от биопленки, видны заметные, но локальные разрушения ПЭТФ подложки (4в, 4г). По-видимому, эти разрушения связаны с воздействием тех участков биопленки, в которых были сосредоточены активные бактерии-деструкторы. Аналогичные закономерности наблюдались и для образцов U3P и U4P, на которых за длительный период экспозиции после антимикробной обработки восстановилась микрофлора, и биопленка почти полностью покрывала образец (рис. S9 ).
Рис. 4.
Микрофотографии биопленок на образце ПЭТФ U1P, инкубированном в промышленной воде (а, б), и отмытого от биопленок образца U1P (в, г), и микрофотографии биопленок образцов ПЭТФ, инкубированных в морской воде, G1P (д), G3P (е), G4P (ж), и отмытого от биопленки образца G4P (з). Сканирующий электронный микроскоп TESCAN VEGA 3 XMU.

Микробные обрастания на поверхности образцов из группы “marine” значительно отличались от описанных для группы “industrial”. На них обнаружена многослойная биопленка, покрывающая всю поверхность образца, даже на первом этапе экспозиции (рис. 4д‒4ж). При этом самую заметную часть биопленки составляли микроэукариоты, количество и разнообразие которых возрастало в течение экспозиции. Возможные конгломераты бактериальных колоний представлены в виде первого слоя на поверхности ПЭТФ-подложки либо на элементах микроэукариотных обрастаний. Однако на ПЭТФ-подложке образца G4P, экспонировавшегося самый длительный период времени, разрушения не обнаружены (рис. 4з). Это свидетельствует о том, что в морской воде при преобладании микроскопических эукариот деградация ПЭТФ, вероятно, не происходит, что соответствует результатам предсказанных процессов метаболизма для изучаемых сообществ.
Заключение. Проблема биостойкости полимерных материалов, включая ПЭТФ, рассматривается с противоположных точек зрения. С одной стороны, экологической, биостойкость препятствует природной деградации бытовых и промышленных отходов, изготовленных из этих материалов, и, тем самым, способствует их накоплению в наземных и водных местообитаниях. С другой стороны, технической, биостойкость требуется при конструировании промышленного оборудования, детали которого изготовлены из пластических материалов.
Материалы из ПЭТФ подвержены биологической деструкции в морской и пресной воде в разной степени, зависящей от климатической зоны и температуры воды, наличия или отсутствия бактерицидной обработки, но основным фактором является наличие специализированных штаммов, способных вырабатывать ферменты деградации ПЭТФ и промежуточного метаболита этого процесса – терефталевой кислоты до протокатеховой кислоты. Участие микроорганизмов в деструкции пластических материалов изучено еще очень слабо. В настоящее время Ideonella sakaiensis является единственной бактерией, для которой однозначно показана способность к деградации ПЭТФ и изучена структура ключевого фермента этого процесса (Yoshida et al., 2016). При выделении этой бактерии было исследовано 250 образцов из различных местообитаний, включая почву, осадки водоемов, промышленные стоки и активный ил со станции переработки пластиковых бутылок. Бактерия I. sakaiensis была выделена только при исследовании образца из последнего местообитания.
В наших исследованиях потенциальная способность к деградации ПЭТФ была обнаружена только для микробного сообщества промышленной оборотной воды завода “Уфаоргсинтез”, осуществляющего производство нефтехимической продукции, включая полимерные материалы. Биологический ил очистных сооружений, содержащий бактерии, грибы и беспозвоночные, за счет возвратных потоков попадает в системы охлаждающей воды промышленного предприятия, что приводит к образованию специализированной микробиоты в оборотной воде. Вследствие селективных условий наибольшее распространение получают штаммы, использующие попадающие в воду органические вещества, продукты нефтехимического синтеза и материалы, из которых изготовлено оборудование оборотного цикла охлаждающей воды. Можно предположить, что промышленные воды такого типа являются потенциальным источником для выделения новых бактерий-деструкторов полимерных материалов. В то же время, конструкторам оборудования, используемого в таких средах, следует учитывать возможность повреждения деталей, изготовленных из полимерных материалов.
В морской воде Черного моря и пресной чистой воде Можайского водохранилища микроорганизмы, потенциально способные к деструкции ПЭТФ не выявлены, хотя в биоинформатическом исследовании значительного количества микробиомов из различных местообитаний, и в частности, из морских, были выявлены гены потенциальных ПЭТФ-гидролаз (Danso et al., 2018). Однако при этом подчеркивалось, что ПЭТФ-гидролазы являются действительно редкими ферментами у микроорганизмов как наземных, так и морских местообитаний. Наиболее вероятно, что биодеградация ПЭТФ в природных местообитаниях в значительной степени зависит от состава обитающих в данных условиях микробных сообществ.
Таким образом, прогнозирование защиты конструкций от биодеструкции и, наоборот, разрушения полимерных материалов бактериями может быть оценено с помощью анализа видового состава и метаболического потенциала прокариот, формирующих биологические обрастания в среде, где эксплуатируется конструкция, или на поверхности утилизируемых материалов.
Список литературы
Лаптев А.Б., Николаев Е.В., Колпачков Е.Д. Термодинамические характеристики старения полимерных композиционных материалов в условиях реальной эксплуатации // Авиационные материалы и технологии. 2018. № 3(52). С. 80–88. https://doi.org/10.18577/2071-9140-2018-0-3-80-88
Каблов Е.Н. Стратегические направления развития материалов и технологий их переработки на период до 2030 г. // Авиационные материалы и технологии. 2012. № 8. С. 7–17.
Каблов Е.Н., Старцев О.В. Фундаментальные и прикладные исследования коррозии и старения материалов в климатических условиях (обзор) // Авиационные материалы и технологии. 2015. № 4. С. 38–52. https://doi.org/10.18577/2071-9140-2015-0-4-38-52
Резников А.А., Муликовская Е.П., Соколов И.Ю. Методы анализа природных вод. 3-е изд., переработ. и доп. М.: Недра, 1970. 488 с.
Acero E.H., Ribitsch D., Dellacher A., Zitzenbacher S., Marold A., Steinkellner G., Gruber K., Schwab H., Guebitz G.M. Surface engineering of a cutinase from Thermobifida cellulosilytica for improved polyester hydrolysis // Biotechnol. Bioeng. 2013. V. 110. P. 2581–2590.
Bolger A.M., Lohse M., Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data // Bioinformatics. 2014. V. 30. P. 2114–2120. https://doi.org/10.1093/bioinformatics/btu170
Caporaso J.G., Kuczynski J., Stombaugh J., Bittinger K., Bushman F.D., Costello E.K., Fierer N., Peña A.G., Goodrich J.K., Gordon J.I., Huttley G.A., Kelley S.T., Knights D., Koenig J.E., Ley R.E., Lozupone C.A., McDonald D., Muegge B.D., Pirrung M., Reeder J., Sevinsky J.R., Turnbaugh P.J., Walters W.A., Widmann J., Yatsunenko T., Zaneveld J., Knight R. QIIME allows analysis of high-throughput community sequencing data // Nat. Methods. 2010. V. 7. Iss. 5. P. 335–336.
Danso D., Schmeisser C., Chow J., Zimmermann W., Wei R., Leggewie C., Li X., Hazen T., Streit W.R. New insights into the function and global distribution of polyethylene terephthalate (PET) degrading bacteria and enzymes in marine and terrestrial metagenomes // Appl. Environ. Microbiol. 2018. V. 84. e02773-17. https://doi.org/10.1128/AEM.02773-17
Derraik J.G.B. The pollution of the marine environment by plastic debris: a review // Mar. Pollut. Bull. 2002. V. 44. P. 842–852.
Ghosh S., Pal S., Ray S. Study of microbes having potentiality for biodegradation of plastics // Environ. Sci. Pollut. Res. 2013. V. 20. P. 4339–4355. https://doi.org/10.1007/s11356-013-1706
Hadad D., Geresh S., Sivan A. Biodegradation of polyethylene by the thermophilic bacterium Brevibacillus borstelensis // J. Appl. Microbiol. 2005. V. 98. P. 1093–1100.
Joo S., Cho I.J., Seo H., Son H.F., Sagong H.-Y., Shin T.J., Choi S.Y., Lee S.Y., Kim K.-J. Structural insight into molecular mechanism of poly(ethylene terephthalate) degradation // Nat. Commun. 2018. V. 9. P. 382. https://doi.org/10.1038/s41467-018-02881-1
Kanehisa M., Goto S. KEGG: Kyoto encyclopedia of genes and genomes // Nucleic Acids Res. 2000. V. 28. P. 27–30.
Kleeberg I., Hetz C., Kroppenstedt R.M., Müller R.J., Deckwer W.D. Biodegradation of aliphatic-aromatic copolyesters by Thermomonospora fusca and other thermophilic compost isolates // Appl. Environ. Microbiol. 1998. V. 64. P. 1731–1735.
Kleeberg I., Welzel K., van den Heuvel J., Müller R.J., Deckwer W.D. Characterization of a new extracellular hydrolase from Thermobifida fusca degrading aliphatic-aromatic copolyesters // Biomacromolecules. 2005. V. 6. P. 262–270.
Kumar S., Stecher G., Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis Version 7.0 for Bigger Datasets // Mol. Biol. Evol. 2016. V. 33. P. 1870–1874. https://doi.org/10.1093/molbev/msw054
Law K.L., Morét-Ferguson S., Maximenko N.A., Proskurowski G., Peacock E.E., Hafner J., Reddy C.M. Plastic accumulation in the North Atlantic Subtropical Gyre // Science. 2010. V. 329. P. 1185–1188.
Liu C., Shi C., Zhu S., Wei R., Yin C.C. Structural and functional characterization of polyethylene terephthalate hydrolase from Ideonella sakaiensis // Biochem. Biophys. Res. Commun. 2019. V. 508. P. 289–294. https://doi.org/10.1016/j.bbrc.2018.11.148
Maniatis T., Frich E., Sambruk G. Methods in Genetic Engineering. Moscow: Molecular Cloning, 1984. P. 5–479.
Miyakawa T., Mizushima H., Ohtsuka J., Oda M., Kawai F., Tanokura M. Structural basis for the Ca2+-enhanced thermostability and activity of PET-degrading cutinase-like enzyme from Saccharomonospora viridis AHK190 // Appl. Microbiol. Biotechnol. 2015. V. 99. P. 4297–4307. https://doi.org/10.1007/s00253-014-6272-8
Moore C.J. Synthetic polymers in the marine environment: a rapidly increasing, long-term threat // Environ. Res. 2008. V. 108. P. 131–139.
Nagpal S., Haque M.M., Singh R., Mande S.S. iVikodak ‒ A platform and standard workflow for inferring, analyzing, comparing, and visualizing the functional potential of microbial communities // Front. Microbiol. 2019. V. 9. Article 3336. https://doi.org/10.3389/fmicb.2018.03336
Ribitsch D., Heumann S., Trotscha E., Herrero Acero E., Greimel K., Leber R., Birner-Gruenberger R., Deller S., Eiteljoerg I., Remler P., Weber T., Siegert P., Maurer K., Donelli I., Freddi G., Schwab H., Guebitz G.M. Hydrolysis of polyethylene terephthalate by para-nitrobenzylesterase from Bacillus subtilis // Biotechnol. Prog. 2011. V. 27. P. 951–960. https://doi.org/10.1002/btpr.610
Ronkvist A.M., Xie W., Lu W., Gross R.A. Cutinase-catalyzed hydrolysis of poly (ethylene terephthalate) // Macromolecules. 2009. V. 42. P. 5128–5138.
Sasoh M., Masai E., Ishibashi S., Hara H., Kamimura N., Miyauchi K., Fukuda M. Characterization of the terephthalate degradation genes of Comamonas sp. strain E6 // Appl. Environ. Microbiol. 2006. V. 72. P. 1825–1832. https://doi.org/10.1128/AEM.72.3.1825-1832.2006
Sauvageau D. Microbial esterase and the degradation of plasticizers // Dissertation. Department of Chemical Engineering McGill University Montreal, Quebec, Canada. 2004.
Schlaveli H.R., Weiss M.A., Leisinger T., Cook A.M. Terephtalate 1,2-dioxygenase system from Comamonas testosterone T-2: purification and some properties of the oxygenase component // J. Bacteriol. 1994. V. 176. P. 6644–6652.
Sharon M., Sharon C. Studies on biodegradation of polyethylene terephthalate: a synthetic polymer // J. Microbiol. Biotech. Res. 2012. V. 2. P. 248–257.
Takahashi S., Tomita J., Nishioka K., Hisada T., Nishijima M. Development of a prokaryotic universal primer for simultaneous analysis of Bacteria and Archaea using next-generation sequencing // PLoS One. 2014. V. 9. Article e105592. https://doi.org/10.1371/journal.pone.0105592
Tanasupawat S., Takehana T., Yoshida S., Hiraga K., Oda K. Ideonella sakaiensis sp. nov., isolated from a microbial consortium that degrades poly(ethylene terephthalate) // Int. J. Syst. Evol. Microbiol. 2016. V. 66. P. 2813–2818. https://doi.org/10.1099/ijsem.0.001058
Thompson R.C., Olsen Y., Mitchell R.P., Davis A., Rowland S.J., John A.W.G., McGonigle D., Russell A.E. Lost at sea: where is all the plastic? // Science. 2004. V. 304. P. 838.
Yoshida S., Hiraga K., Takehana T., Taniguchi I., Yamaji H., Maeda Y., Toyohara K., Miyamoto K., Kimura Y., Oda K. A bacterium that degrades and assimilates poly(ethylene terephthalate) // Science. 2016. V. 353. P. 759–759.
Дополнительные материалы
- скачать Turova.docx
- Таблица S1. Разнообразие доминируюших филотипов представителей домена Bacteria в библиотеках генов 16S rRNA* микроорганизмов, выросших на образцах ПЭТФ, экспонировавшихся в разных водных средах
- скачать Turova_1.tiff
- Рис. S1. Предполагаемые пути деградации ПЭТФ у бактерии I. sakaiensis (Yoshida et al., 2016).
- скачать Turova_2.tiff
- Рис. S2. Кривые насыщения, показывающие соотношение количества операционных таксономических единиц (ОТЕ) к общему количеству последовательностей фрагментов гена 16S рРНК в библиотеке, полученной на основе ДНК микроорганизмов образцов ПЭТФ. Последовательности сгруппированы в филотипы с уровнем сходства ≥ 98%.
- скачать Turova_3.tiff
- Рис. S3. График, показывающий группирование образцов ПЭТ в соответствии с составом их бактериальных сообществ, полученный методом главных компонент (principal components analysis, PCA).
- скачать Turova_4.tiff
- Рис. S4. Филогенетическое дерево, включающее 25 наиболее распространенных ОТЕ из библиотек генов 16S рРНК микроорганизмов образцов ПЭТ, инкубированных в различных средах. Операционные таксономические единицы бактерий I. sakaiensis, A. delafieldii и P. putida отмечены цветом. Масштаб показывает эволюционное расстояние, соответствующее 10 нуклеотидных замен на 100 нуклеотидов. Значения бутстреп поддержки, превышающие 80%, отмечены синими точками.
- скачать Turova_5.tiff
- Рис. S5. Тепловая карта, показывающая кластеризацию образцов ПЭТ на основе относительного процента функциональных профилей бактериального сообщества в программе iVikodak. Анализируемые ниже метаболические пути выделены черной рамкой.
- скачать Turova_6.tiff
- Рис. S6. Тепловая карта, показывающая кластеризацию образцов ПЭТ на основе процентного содержания ферментного профиля метаболического пути "Деградация полициклических ароматических углеводородов", полученного в программе iVikodak. Ферменты деградации терефталевой кислоты отмечены черным цветом.
- скачать Turova_7.tiff
- Рисунок S7a. Графики, показывающие роль ключевых микроорганизмов (и их таксономических линий) в осуществлении специфических функций в каждой группе образцов, экспонировавшихся в промышленной оборотной воде (А), морской воде (B) и чистой пресной воде (С).
- скачать Turova_8.tiff
- Рисунок S7b. Графики, показывающие роль ключевых микроорганизмов (и их таксономических линий) в осуществлении специфических функций в каждой группе образцов, экспонировавшихся в промышленной оборотной воде (А), морской воде (B) и чистой пресной воде (С).
- скачать Turova_9.tiff
- Рисунок S7c. Графики, показывающие роль ключевых микроорганизмов (и их таксономических линий) в осуществлении специфических функций в каждой группе образцов, экспонировавшихся в промышленной оборотной воде (А), морской воде (B) и чистой пресной воде (С).
- скачать Turova_10.tiff
- Рис. S8. Тепловая карта, показывающая кластеризацию образцов ПЭТ на основе процента iVicodak производного профиля метаболического пути "деградация толуола". Ферменты деградации 4-толуолкарбоксилата/4-толуолсульфоната отсутствуют.
- скачать Turova_11.tiff
- Рис. S9. Сканирующие электронные микрофотографии микробных биопленок на образцах ПЭТФ U3P (A) и U4P (C) и отмытых от биопленок образцов U3P (B) и U4P (D).