Микробиология, 2020, T. 89, № 3, стр. 334-343
Геномный анализ бактерии некультивируемого филума Riflebacteria выявил пути органотрофного метаболизма и диссимиляционного восстановления железа
В. В. Кадников a, А. В. Марданов a, А. В. Белецкий a, О. В. Карначук b, Н. В. Равин a, *
a Институт биоинженерии, ФИЦ Биотехнологии РАН
119071 Москва, Россия
b Лаборатория биохимии и молекулярной биологии, Томский государственный университет
634050 Томск, Россия
* E-mail: nravin@biengi.ac.ru
Поступила в редакцию 02.12.2019
После доработки 19.12.2019
Принята к публикации 20.12.2019
Аннотация
Кандидатный филум Riflebacteria был описан в результате анализа геномов, собранных из метагеномов различных анаэробных экосистем, но до настоящего времени ни один из представителей Riflebacteria не выделен в чистую культуру. Мы определили геном нового представителя Riflebacteria из метагенома микробного сообщества глубинного подземного резервуара термальных вод в Западной Сибири. Филогенетический анализ показал, что эта бактерия, названная Ch46, представляет новый род в филуме Riflebacteria, отличный от ранее описанного Candidatus Ozemobacter. Анализ генома бактерии Ch46 выявил предположительно секретируемые альфа-амилазы и различные транспортеры сахаров и пептидов. Реконструкция путей центрального метаболизма позволила предположить, что бактерия Ch46 является анаэробным органотрофом, способным сбраживать сахара и белковые субстраты. Известные пути автотрофной фиксации углерода, а также аэробная дыхательная цепь, у Ch46 отсутствуют. Генерация трансмембранного ионного градиента может обеспечиваться Rnf комлексом, а также натрий-транспортирующей NADH-хинон оксидоредуктазой. Наличие мультигемовых цитохромов с-типа указывает на возможность диссимиляционного восстановления Fe(III). Пути диссимиляционного восстановления сульфата, нитрата, нитрита, арсената и серы отсутствуют. В подземном водоносном горизонте Riflebacteria, вероятно, используют крахмал и аналогичные полисахариды, а также низкомолекулярные органические соединения. На основе результатов филогенетического и геномного анализа новую бактерию предлагается классифицировать как Candidatus Rifleibacterium amylolyticum.
Кандидатный филум Riflebacteria был впервые описан на основе филогенетического анализа генома, собранного из метагенома водоносного горизонта, залегающего на глубине 4–6 м около реки Колорадо, в районе г. Rifle, США (Anantharaman et al., 2016). В этой работе было получено 2540 геномов, представляющих 47 новых филогенетических линий уровня филума, один из которых, Candidatus Riflebacteria bacterium GWC2_50_8, был описан как представитель нового филума Riflebacteria (Anantharaman et al., 2016). Геномы двух других представителей Riflebacteria, HGW-Riflebacteria-1 и HGW-Riflebacteria-2, были получены из метагенома подземных вод, отобранных с глубины несколько сот метров в исследовательском центре Horonobe Underground Research Laboratory, Hokkaido, Япония (Hernsdorf et al., 2017). Еще один геном, uncultured bacterium CG2_30_54_10, был получен из метагенома воды источника Crystal Geyser (Utah, США) (Probst et al., 2017). Позднее были описаны геномы представителей Riflebacteria из подземных экосистем, – Candidatus Riflebacteria isolate UBA8953 (Parks et al., 2018) и Candidatus Ozemobacter sibiricus BY5 (Kadnikov et al., 2018), а также Rumen uncultured genome RUG334 из рубца коровы (Stewart et al., 2018).
О возможностях метаболизма Riflebacteria и их экологической роли известно мало. Отмечалось наличие в геномах Riflebacteria из подземных экосистем генов анаэробной сульфитредуктазы и гидрогеназы [FeFe] типа, на основании чего были высказаны предположения об участии этих бактерий в цикле серы (Hernsdorf et al., 2017; Anantharaman et al., 2018). Подробный анализ путей метаболизма был проведен только для Ca. Ozemobacter sibiricus BY5, геном которой был собран из метагенома подземного водоносного горизонта Западно-Сибирского региона (Kadnikov et al., 2018). Подземные термальные воды, залегающие в Западной Сибири на глубинах от 1 до 3 км в осадочных породах Мелового периода, доступны благодаря нефтепоисковым скважинам, через которые артезианская вода вытекает под естественным давлением (Frank et al., 2016; Kadnikov et al., 2018). Анализ этого генома показал, что Ca. Ozemobacter sibiricus BY5, по-видимому, является анаэробным органотрофом, способным сбраживать некоторые углеводы и белковые субстраты, а также осуществлять диссимиляционное восстановление Fe(III), при этом сульфитредуктаза в его геноме отсутствовала (Kadnikov et al., 2018).
Позднее мы проанализировали состав микробных сообществ (Кадников и соавт., 2017a, 2017b) и секвенировали метагеном подземных вод, вскрываемых другой артезианской скважиной, 5Р, расположенной в районе поселка Чажемто в Томской области (Kadnikov et al., 2017c). В этом сообществе около половины микроорганизмов составляют метаногенные археи, а вторую половину – бактерии различных некультивируемых линий (Кадников и соавт., 2017a). В частности, анализ состава сообщества по последовательностям генов 16S рРНК показал, что в нем присутствуют бактерии кандидатного филума Riflebacteria, филогенетически удаленные от Ca. Ozemobacter sibiricus BY5.
В этой работе, используя результаты метагеномного анализа, мы определили геном нового представителя Riflebacteria, охарактеризовали особенности его метаболизма, глобальное распространение и вероятную экологическую роль.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Характеристика скважины, отбор проб воды и выделение метагеномной ДНК. Объектом исследования были подземные воды, залегающие в осадочных породах мелового периода на глубине около 2 км, которые поступают на поверхность через нефтепоисковую скважину 5Р в районе пос. Чажемто Томской области. Образец воды отобрали в апреле 2016 года (Kadnikov et al., 2017с). Вода имела температуру около 20°С, нейтральный рН (7.43–7.6) и отрицательный окислительно-восстановительный потенциал (Eh от –304 до –338 mV).
Для сбора биомассы микроорганизмы из образца воды (объемом 20 литров) собирали на фильтре с диаметром пор 0.22 мкм. Фильтры гомогенизировали, растирая с жидким азотом, препарат метагеномной ДНК выделяли с помощью Power Soil DNA Isolation Kit (“MO BIO Laboratories Inc.”, Carlsbad, США). Всего было выделено около 1 мкг ДНК.
Секвенирование метагеномной ДНК и сборка геномов микроорганизмов – членов сообщества. Секвенирование метагеномной ДНК на Illumina HiSeq2500 (“Illumina Inc.”, США) описано нами ранее (Kadnikov et al., 2017с). В результате секвенирования (в формате парных чтений 2 × 250 нт) с последующей фильтрацией по качеству (Q > 33) было прочтено около 16.9 млрд нт. Сборку прочтений в контиги проводили с помощью программы metaSPAdes v. 3.7.1 (Nurk et al., 2017), как описано нами ранее (Kadnikov et al., 2017с). Для разбиения полученных контигов на кластеры, соответствующие геномам отдельных микроорганизмов (metagenome-assembled genome, MAG), использовали программу CONCOСT (Alneberg et al., 2014). Полноту собранных MAG и их “загрязнение” (т.е. наличие в MAG контигов, представляющих другие микроорганизмы), оценивали с помощью программы CheckM (Parks et al., 2015).
Таксономическую принадлежность собранных геномов определяли по базе GTDB с использованием программы GTDB-Tk v. 0.1.3 (Parks et al., 2018). Один из геномов, обозначенный как Ch46, был отнесен к кандидатному филуму Riflebacteria.
Дополнительно метагеномную ДНК секвенировали на MinION (“Oxford Nanopore”, Великобритания), используя Ligation Sequencing kit 1D согласно рекомендациям производителя. В результате секвенирования было получено 1 418 419 чтений суммарной длиной около 1.54 млрд нт. Эти длинные чтения использовали для объединения контигов, составляющих геном Ch46, в более длинные последовательности. Для этого c помощью программы BWA v0.7.15 (Li, Durbin, 2009) отбирали полученные на MinION прочтения, имеющие гомологию с последовательностями контигов Ch46. Контиги объединяли с использованием программы npScarf (Cao, 2017), при этом для заполнения пробелов между контигами использовали чтения Illumina из графа metaSPAdes.
Последовательность генома бактерии Ch46 депонирована в NCBI GenBank под номером JAACJG000000000.
Аннотация и анализ генома бактерии Ch46. Поиск генов и их аннотацию проводили с использованием сервера RAST, с последующей проверкой аннотации в результате сравнения последовательностей предсказанных белков с базами данных NCBI. Для предсказания N-концевых сигнальных пептидов использовали программы Signal P v. 5.0 (http://www.cbs.dtu.dk/services/SignalP/) и PRED-TAT (http://www.compgen.org/tools/PRED-TAT/), трансмембранные домены идентифицировали с помощью TMHMM Server v. 2.0 (http:// www.cbs.dtu.dk/services/TMHMM/). Программа iRep была использована для расчета индекса репликации геномной ДНК (Brown et al., 2016). Для классификации гидрогеназ использовали онлайн сервис HydDB (https://services.birc.au.dk/hyddb/) (Søndergaard et al., 2016).
Определение уровней сходства геномов и филогенетический анализ. Средний уровень идентичности аминокислотных последовательностей (average amino acid identity, AAI) между геномами определяли с помощью скрипта aai.rb из Enveomics Collection (Rodriguez-R, Konstantinidis, 2016). Значения ДНК–ДНК гибридизации in silico вычисляли с помощью сервиса GGDC2 (Meier-Kolthoff et al., 2013), доступного на сайте http://ggdc.dsmz.de/, используя рекомендованную формулу 2.
Для проведения филогенетического анализа на основе полногеномных данных была определена выборка, включающая геном Ch46, геномы 7 других представителей Riflebacteria (табл. 1) и геном представителя филогенетически близкого к Riflebacteria кандидатного филума Wallbacteria, UBP16_UBA6123 (GenBank GCA_002423385.1). Для этой выборки с помощью GTDB-Tk были найдены 120 консервативных однокопийных маркерных гена и построено множественное выравнивание конкатенированных последовательностей этих маркерных генов.
Таблица 1.
Основные характеристики геномов представителей Riflebacteria
| Организм/геном | GenBank | Размер генома, нт | Полнота генома, % | Число контигов | Г + Ц, % |
|---|---|---|---|---|---|
| Ca. Riflebacterium amylolyticus Ch46 | GCA_009917695.1 | 5 391 313 | 98 | 35 | 50.1 |
| GWC2_50_8 | GCA_001787855.1 | 4 386 086 | 76 | 881 | 49.6 |
| HGW-Riflebacteria-2 | GCA_002839435.1 | 5 226 436 | 89 | 199 | 50.1 |
| HGW-Riflebacteria-1 | GCA_002839445.1 | 5 678 741 | 98 | 50 | 49.9 |
| UBA8953 (sp002839445) | GCA_003446455.1 | 4 673 206 | 95 | 817 | 50 |
| RUG334 (sp900317935) | GCA_900317935.1 | 4 546 479 | 93 | 381 | 34.7 |
| Ca. Ozemobacter sibiricus BY5 | GCA_003327425.1 | 5 683 244 | 98 | 58 | 65.4 |
| CG2_30_54_10 | GCA_001872985.1 | 2 825 442 | 62 | 574 | 53.5 |
Полученное множественное выравнивание было использовано для построения филогенетического дерева методом Maximum Likelihood с помощью программы PhyML v. 3.3 (Guindon et al., 2010) с параметрами по умолчанию.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Сборка генома представителя кандидатного филума Riflebacteria. В результате секвенирования метагенома микробного сообщества подземных вод скважины 5Р по технологии Illumina было прочтено и собрано в контиги около 16.9 млрд нт (Kadnikov et al., 2017c). Кластеризацию контигов проводили с помощью программы CONCOCT. Один из полученных MAG, названный Ch46, был представлен 49 контигами общей длиной 5.4 млн нт, средняя кратность их прочтения составляла 23. Относительная доля этого генотипа в микробном сообществе, определяемая по доле MAG Ch46 во всем метагеноме, составляла 0.83%. Таксономическая классификация этого MAG с помощью Genome Taxonomy database показала, что он относится к кандидатному филуму Riflebacteria.
Для улучшения сборки генома бактерии Ch46 были использованы 1.4 млн длинных чтений (общим объемом около 1.5 млрд нт), полученных в результате нанопорового секвенирования. Это позволило объединить 49 контигов в 35, суммарная длина которых составила 5 391 313 нт (табл. 1). По оценке сервиса CheckM из 105 консервативных однокопийных генов в этом геноме присутствует 103 гена в одной копии и один – в двух копиях, что указывает на 98.31% полноту MAG Ch46 при возможном загрязнении посторонними последовательностями в 1.69%. По степени полноты и минимальному числу контигов собранный MAG является лучшим среди просеквенированных к настоящему времени геномов Riflebacteria (табл. 1).
В результате анализа генома Ch46 были обнаружены гены 16S и 23S рРНК, а также 45 генов транспортных РНК. По результатам аннотации генома предсказано 4623 потенциальных белок-кодирующих генов, функции лишь 1573 из которых могут быть предсказаны в результате сравнения с базами данных NCBI. Геном Ch46 содержит два CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) локуса, включающие 35 и 58 единиц повтор–спейсер, и набор генов, кодирующих CRISPR систему типа 1-С, расположенных в районе одного из локусов.
Анализ генома Ch46 выявил набор генов, кодирующих флагеллярный аппарат и аппарат хемотаксиса, а также набор белков пилей типа IV, обеспечивающих подвижность этой бактерии.
Для оценки скорости репликации ДНК бактерии Ch46 in situ, в подземных водах, мы использовали программу iRep (Brown et al., 2016). Значение индекса iRep для генома Ch46 составляло 1.33, что указывает на сравнительно медленный рост этих бактерий (около 33% клеток реплицировались во время отбора проб), но свидетельствует о том, что они представляют метаболически активную часть микробного сообщества.
Филогенетическое положение Ch46. Поиск в Genome Taxonomy database филогенетически родственных микроорганизмов на основе сходства геномов показал, что ближайшим родственником Ch46 является Candidatus Riflebacteria bacterium HGW-Riflebacteria-2 (Hernsdorf et al., 2017). Средняя идентичность аминокислотных последовательностей (AAI) этих двух геномов составляет 85.16%. Значения AAI между Ch46 и другими представителями Riflebacteria находятся в диапазоне от 51 до 78% (табл. 2). С учетом уровней пороговых значений AAI, предложенных для таксономической классификации некультивируемых микроорганизмов (65–95% для одного рода и 95–100% для одного вида; Konstantinidis et al., 2017), Ch46 и HGW-Riflebacteria-2 представляют разные виды одного рода. На принадлежность этих микроорганизмов к разным видам указывает и значение их ДНК–ДНК гибридизации in silico, оцениваемое в 24%.
Таблица 2.
Средняя идентичность аминокислотных последовательностей представителей Riflebacteria
| Организм/геном | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | |
|---|---|---|---|---|---|---|---|---|---|
| 1 | Ca. Riflebacterium amylolyticus Ch46 | 100 | 85 | 78 | 78 | 78 | 54 | 51 | 51 |
| 2 | HGW-Riflebacteria-2 | 85 | 100 | 79 | 78 | 78 | 54 | 51 | 51 |
| 3 | HGW-Riflebacteria-1 | 78 | 79 | 100 | 95 | 86 | 54 | 51 | 51 |
| 4 | UBA8953 (sp002839445) | 78 | 78 | 95 | 100 | 86 | 54 | 51 | 51 |
| 5 | GWC2_50_8 | 78 | 78 | 86 | 86 | 100 | 53 | 51 | 50 |
| 6 | RUG334 (sp900317935) | 54 | 54 | 54 | 54 | 53 | 100 | 48 | 48 |
| 7 | Ca. Ozemobacter sibiricus BY5 | 51 | 51 | 51 | 51 | 51 | 48 | 100 | 65 |
| 8 | CG2_30_54_10 | 51 | 51 | 51 | 51 | 50 | 48 | 65 | 100 |
Для анализа филогении Riflebacteria было построено филогенетическое дерево на основе конкатенированных последовательностей 43 консервативных маркерных генов, включающее Ch46 и другие доступные геномы Riflebacteria. Полученные результаты (рис. 1) подтверждают, что ближайшим родственником Ch46 является HGW-Riflebacteria-2. На дереве четко выделяются три кандидатных рода, предложенные в GTDB таксономии, – род UBA8953, включающий Ch46, UBA8953-sp002839445, HGW-Riflebacteria-1, HGW-Riflebacteria-2 и GWC2_50_ 8, род RUG334 с единственным видом sp900317935, а также ранее описанный род Candidatus Ozemobacter (Kadnikov et al., 2018), включающий Ca. Ozemobacter sibiricus BY5 и бактерию CG2_30_54_10 (в GTDB таксономии указан как род CG2-30-54-10).
Рис. 1.
Филогения кандидатного филума Riflebacteria, основанная на анализе конкатенированных последовательностей консервативных маркерных генов. В качестве внешней группы использован геном бактерии UBP16_UBA6123 из кандидатного филума Wallbacteria. Уровни поддержки ветвей определены с помощью Байесовского теста в PhyML.

Среди представителей кандидатного рода UBA8953 последовательность гена 16S рРНК известна только для HGW-Riflebacteria-1, уровень ее идентичности с 16S рРНК бактерии Ch46 составляет 98.1%. Последовательности генов 16S рРНК Ch46 и Ca. Ozemobacter sibiricus BY5 имеют 89.9% идентичность, что, как и уровень AAI, соответствует межродовым различиям.
Возможные субстраты и пути центрального метаболизма бактерии Ch46. Поиск генов гликозил-гидролаз, содержащих характерные для секретируемых белков N-концевые сигнальные последовательности, выявил только четыре альфа-амилазы (гены GQF41_2102, GQF41_2155, GQF41_3117, GQF41_3457) и пуллуланазу (GQF41_4095), которые могут обеспечивать внеклеточный гидролиз крахмала и сходных полисахаридов. Импорт мальтозы и мальтодекстрина в клетку может обеспечиваться транспортерами АВС-типа, а их последующая утилизация – соответствующими внутриклеточными гидролазами. Гены внеклеточных гидролитических ферментов, которые могли бы обеспечить гидролиз других полисахаридов, в геноме не обнаружены. Анализ генома показал наличие транспортеров и других сахаров, в том числе – глюкозы, маннозы, фруктозы и N-ацетил-β-глюкозамина. Метаболизм импортированных сахаров, по-видимому, связан с путями гликолиза. Так, N-ацетил-β-глюкозамин может фосфорилироваться с помощью N-ацетилглюкозаминкиназы NagC, а затем деацетилироваться N-ацетилглюкозамин-6-фосфат-деацетилазой NagA. На заключительном этапе глюкозамин-6-фосфат-деаминаза NagB превращают N-ацетил-D-глюкозамин-6-P во фруктозу-6-P, которая поступает в путь гликолиза.
При анализе генома выявлены гены всех ферментов пути гликолиза Эмбдена–Мейергофа, глюконеогенеза, а также пентозофосфатного пути, включая его окислительные и неокислительные стадии (рис. 2). Известные пути автотрофной фиксации углерода в геноме отсутствуют. В качестве запасного полисахарида Ch46 может использовать гликоген, о чем свидетельствует наличие генов ключевых ферментов, участвующих в его синтезе и деградации, глюкозо-1-фосфат-аденилтрансферазы, гликогенсинтазы, 1,4-альфа-глюкан соединяющего фермента, гликогенфосфорилазы и гликоген-расщепляющего фермента (glycogen debranching enzyme). Синтез другого запасного полисахарида и осмолита, трегалозы, обеспечивается трегалозосинтетазой, а также парой ферментов – мальтоолигозил трегалозосинтазой и мальтоолигозил трегалозогидролазой.
Рис. 2.
Основные пути метаболизма Ca. Riflebacterium amylolyticus Ch46. Обозначения ферментов: Amy, альфа амилаза; GH, гликозил-гидролаза; GK, глюкокиназа; PGI, глюкозо-6-фосфат изомераза; PFK, 6-фосфофруктокиназа; FBA, фруктозо-бисфосфат альдолаза; TIM, триозофосфатизомераза; GPDH, глицеральдегид 3-фосфат дегидрогеназа; PGK, фосфоглицераткиназа; PGM, фосфоглицератмутаза; PYK, пируваткиназа; G6PDH, глюкоза-6-фосфат 1-дегидрогеназа; PGL, 6-фосфоглюконолактоназа; PGD, 6-фосфоглюканат дегидрогеназа; RPE, рибулоза-фосфат 3-эпимераза; RPI, рибоза 5-фосфат изомераза; POR, пируват ферредоксин (флаводоксин) оксидоредуктаза; Atr, аминотрансферазы, OOR, ферредоксин-зависимые оксидоредуктазы 2-кетокислот; PPase, пирофосфатаза; Na-Nqr, Na+-транспортирующая NADH-хинон оксидоредуктаза; [FeFe] Hyd, гидрогеназа [FeFe] типа; FNOR, ферредоксин:NADP+ оксидоредуктаза; ASR, анаэробная сульфитредуктаза. Другие обозначения: GAP, GAP, глицеральдегид-3-фосфат; 3PG, 3-фосфоглицерат; 2PG, 2-фосфоглицерат; PEP, фосфоэнолпируват; Fd, ферредоксин; ox/red, окисленная и восстановленная формы; cyt, цитохром c; CoA, коэнзим А; PP, пирофосфат; Р, фосфат.
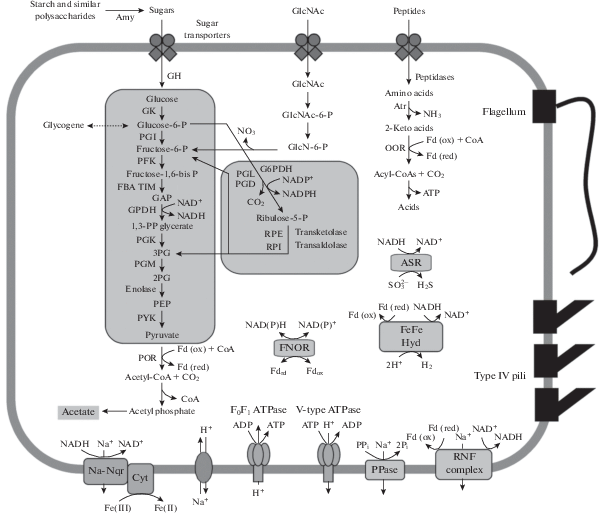
Пируват, образованный в результате гликолиза, может быть конвертирован в ацетил-КоА с помощью пируват-ферредоксин оксидоредуктазы. Окисление ацетил-КоА с образованием ацетата и генерацией АТФ может осуществляться в двустадийной реакции с помощью фосфат-ацетилтрансферазы и ацетаткиназы, в то время как ацетил-КоА-синтетазы не обнаружено. Наличие альдегид- и алкогольдегидрогеназ позволяет предположить, что, помимо ацетата, продуктами брожения могут быть и спирты. Ацетил-КоА, образующийся из пирувата, может поступать в цикл трикарбоновых кислот. Однако этот цикл является неполным (отсутствуют сукцинил-КоА синтетаза, сукцинатдегидрогеназа и малатдегидрогеназа) и, вероятно, используется только для биосинтетических целей.
Источником углерода и энергии для Ch46 могут быть также аминокислоты и короткие пептиды, импорт которых в клетку может обеспечиваться транспортерами АВС-типа. Импортированные или образовавшиеся в результате внутриклеточного гидролиза пептидов аминокислоты могут деаминироваться и конвертироваться в 2-кетокислоты (пируват, 2-оксоглутарат и др.). Затем 2-кетокислоты могут окисляться с помощью ферредоксин-зависимых оксидоредуктаз различной специфичности до ацил-КоА производных, которые затем расщепляются с образованием соответствующих кислот и продукцией АТФ (рис. 2).
Восстановленные продукты, образующиеся в путях ферментации, NADH и восстановленный ферредоксин, могут быть повторно окислены гидрогеназами [Fe-Fe] типа (Greening et al., 2016). Все гидрогеназы не содержат N-концевых сигнальных пептидов или трансмембранных доменов и, вероятно, локализованы в цитоплазме. В геноме имеется два кластера генов гидрогеназ. Первый из них (гены GQF41_0195-GQF41_0204) кодирует две трехсубъединичные гидрогеназы группы А3, которые могут осуществлять бифуркацию электронов между Н2, ферредоксином и NADH, при которой окисление ферредоксина сопрягается с окислением NADH и образованием молекулярного водорода (Schut, Adams, 2009; Greening et al., 2016). Второй кластер (GQF41_3791–GQF41_3797) помимо трехсубъединичной гидрогеназы группы А3 кодирует также [Fe-Fe] гидрогеназу группы В, которая может окислять восстановленный ферредоксин с образованием Н2 (Greening et al., 2016). В целом, активность этих гидрогеназ может обеспечивать повторное окисление NADH и восстановленного ферредоксина, образующихся в ходе ферментации.
Еще одним механизмом регенерации NAD+ может быть NADH-зависимое восстановление сульфита, осуществляемое цитоплазматической анаэробной сульфит редуктазой Asr. Этот фермент у бактерии Ch46 кодируется asrABC опероном (GQF41_2511–GQF41_2509), сходным с аналогичными оперонами у Clostridium и Salmonella (Hallenbeck et al., 1989). Гены анаэробной сульфитредуктазы присутствуют и в геномах трех других представителей кандидатного рода UBA8953, бактерий GWC2_50_8, HGW-Riflebacteria-1 и HGW-Riflebacteria-2, и было высказано предположение, что они могут осуществлять восстановление сульфита с использованием Asr комплекса (Anantharaman et al., 2018).
Генерация трансмембранного ионного градиента. Анализ генома бактерии Ch46 не выявил гены основных компонентов аэробной дыхательной цепи, а именно, NADH дегидрогеназы, мембранно-связанной сукцинатдегидрогеназы, цитохром-bc1-комплекса или альтернативного комплекса III и цитохромоксидаз. Известные пути диссимиляционного восстановления сульфата, нитрата, нитрита, арсената и серы также не обнаружены.
Однако бактерия Ch46 все же имеет несколько механизмов для генерации трансмембранного ионного градиента, который может быть использован мембранно-связанной АТФ синтазой F0F1 типа для синтеза АТФ. Во-первых, в геноме Ch46 имеется полный набор генов, кодирующих мембранно-связанный ион-транспортирующий Rnf комплекс (GQF41_3883–GQF41_3888). Rnf может функционировать либо анаболически, восстанавливая ферредоксин и окисляя NADH, либо катаболически, осуществляя окисление ферредоксина, восстановление NAD+ и перенос протонов или ионов натрия через цитоплазматическую мембрану (Biegel et al., 2011). Исходя из порядка генов, rnfCDGEAB, Rnf оперон бактерии Ch46 относится к типу 3, обычно обнаруживаемому в Firmicutes, где он действует катаболически, как запасающая энергию ферредоксин: NAD+ оксидоредуктаза. Два других фермента также могут способствовать генерации трансмембранного ионного градиента, – АТФаза V-типа, которая использует гидролиз АТФ, образующийся при ферментации, для транспорта протонов, и мембранно-связанная пирофосфатаза GQF41_0857, использующая гидролиз пирофосфата для транслокации протонов или ионов Na+. Отсутствие лизина в сигнатурной последовательности GNXX (K/A) (Malinen et al., 2007) указывает на то, что эта пирофосфатаза переносит ионы натрия, а не протоны. Баланс между концентрациями H+ и Na+ могут обеспечивать кодируемые в геноме H+/Na+ антипортеры.
Другим комплексом, сохраняющим энергию в форме трансмембранного ионного градиента, является Na+-транспортирующая NADH-хиноноксидоредуктаза, Na-NQR. Этот ферментативный комплекс является основным ионным насосом и точкой входа электронов в дыхательную цепь у различных патогенных и свободноживущих бактерий (Reyes-Prieto et al., 2014). Na-NQR представляет собой натрий-специфическую ионную помпу, имеющую общее с Rnf эволюционное происхождение. Na-NQR обычно включает шесть субъединиц (NqrA-F). В геноме Ch46 обнаружено два nqr оперона (GQF41_2620–GQF41_2616 и GQF41_4465–GQF41_4461), кодирующих субъединицы NqrB, C, D, E и F, а гены nqrA в обоих оперонах найдены не были. В отличие от других субъединиц, NqrA локализована в цитозоле, не содержит сайтов связывания кофакторов и ее функция неясна. Возможно, ген nqrA не был обнаружен, или эта субъединица не требуется для функционирования Na-NQR у Ch46.
Поскольку Na-NQR оксидоредуктаза передает электроны в пул хинонов цитоплазматической мембраны, у бактерии Ch46 должна существовать терминальная редуктаза, использующая эти электроны для восстановления терминального акцептора. Один из nrqBCDEF кластеров содержит также ген GQF41_2615 цитохрома с с 8 гем-связывающими мотивами (CxxCH). Также в геноме обнаружены два гена (GQF41_1560 и GQF41_1756), кодирующих цитохромы с с 10 гем-связывающими мотивами. Все три цитохрома содержат N-концевые сигнальные пептиды и трансмембранные домены, что указывает на их экспорт из клетки и локализацию на внешней стороне мембраны; это позволяет им контактировать с нерастворимым акцептором электронов. Такие мультигемовые цитохромы с-типа играют ключевую роль в переносе электронов из клетки к внеклеточному акцептору электронов у грамотрицательных бактерий Shewanella и Geobacter, осуществляющих диссимиляционное восстановление Fe(III) (Shi et al., 2007; Richter et al., 2012). Один из декагемовых цитохромов (GQF41_1560) содержат мотив (Ser/Thr)-Pro-(Ser/Thr), который может обеспечивать его связывание с гематитом, минералом оксида железа (Fe2O3), что было показано для Fe(III)-редуктазы Shewanella oneidensis (Lower et al., 2008). Адгезии к минералам Fe(III) также могут способствовать пили типа IV, набор генов которых обнаружен в геноме бактерии Ch46. Таким образом, можно предположить, что мультигемовые цитохромы могут принимать электроны из пула хинонов и обеспечивать внеклеточное восстановление Fe(III). Предсказанные пути метаболизма бактерии Ch46 приведены на рис. 2.
Общие характеристики метаболизма Riflebacteria. Имеется очень ограниченная информация о метаболических возможностях других представителей Riflebacteria. Хотя помимо Ch46 известно 7 геномов Riflebacteria, 4 из которых имеют полноту свыше 90%, анализ путей метаболизма на основе геномных данных был проведен только для бактерии Ca. Ozemobacter sibiricus BY5, представляющей другой род этого филума (Kadnikov et al., 2018). Как и Ch46, Ca. Ozemobacter sibiricus BY5 может использовать в качестве субстратов крахмал и его производные, а также простые сахара (включая N-ацетил-β-глюкозамин), аминокислоты и пептиды. Оба генома кодируют гликолитический путь Эмбдена–Мейергофа, полный пентозофосфатный путь и содержат неполный набор генов цикла трикарбоновых кислот, аэробная дыхательная цепь отсутствует в обеих геномах. Ca. Ozemobacter sibiricus BY5 также имеет гены, кодирующие цитоплазматические гидрогеназы [Fe-Fe] типа, Rnf комплекс и два кластера генов Na-NQR оксидоредуктаз с тем же набором субъединиц. Гены предполагаемых Fe(III) редуктаз у представителей родов Ca. Ozemobacter и UBA8953, однако, отличаются. Так, у Ch46, UBA8953-sp002839445, HGW-Riflebacteria-1, HGW-Riflebacteria-2 и GWC2_50_ 8 один из nrqBCDEF кластеров включает только ген восьмигемового цитохрома с, а у Ca. Ozemobacter sibiricus BY5 и CG2_30_54_10 вместо этого гена имеется 4 гена мультигемовых цитохромов с и ген ферритина. Другим отличием является отсутствие у Ca. Ozemobacter sibiricus BY5 и CG2_30_54_10 генов анаэробной сульфитредуктазы, имеющейся в геномах представителей UBA8953.
Глобальное распространение родственных Ch46 бактерий и их экологическая роль. Поиск в GenBank выявил 45 почти полных (>1400 нт) последовательностей генов 16S рРНК, имеющих идентичность выше 95% с 16S рРНК бактерии Ch46, в том числе 40 последовательностей с идентичностью выше 97%, т.е. представляющих тот же вид. Все они были детектированы в анаэробных местообитаниях в разных географических районах, – биореакторах для анаэробной переработки органических отходов и очистки сточных вод (31), подземных водах и источниках (10), осадках озер и рек (3 последовательности).
В микробном сообществе подземного водоносного горизонта, в котором была обнаружена бактерия Ch46, примерно половину прокариот составляют метаногенные археи, а вторую половину – бактерии филумов Firmicutes, Chloroflexi, Proteobacteria, Bacteroidetes и Ignavibacteriae (Кадников и соавт., 2017a). Среди бактерий этих филумов известны организмы, способные гидролизовать сложные полимеры. Так, представители филумов Ignavibacteriae и Chloroflexi (Anaerolineaceae), встречающиеся в подземных водах Западно-Сибирского региона, могут гидролизовать сложные полисахариды и белковые субстраты (Kadnikov et al., 2018). По-видимому, Riflebacteria в этих экосистемах могут специализироваться на гидролизе крахмала и аналогичных полисахаридов, сохранившихся в осадочных породах мелового периода с момента образования водоносных горизонтов или образованных автотрофами, а также использовать образованные гидролитиками низкомолекулярные органические соединения (простые сахара, пептиды, аминокислоты и т.п.), при этом образуя водород и ацетат, являющиеся субстратами для метаногенных архей. Вероятно, Riflebacteria развиваются в составе биопленок на поверхности минеральных пород, в богатых органикой условиях и контакте с нерастворимыми акцепторами электронов. Поскольку подземные водоносные горизонты Западно-Сибирского региона не являются полностью изолированными от поверхности и на протяжении миллионов лет пополнялись водами метеорного происхождения (Kadnikov et al., 2018), по-видимому, Riflebacteria попали сюда с поверхности и успешно колонизировали новую экологическую нишу.
Описание “Candidatus Rifleibacterium amylolyticum” Ch46. Геном бактерии Ch46 соответствует требованиям, предложенным для описания новых таксонов некультивируемых микроорганизмов (Konstantinidis et al., 2017), поэтому мы предлагаем описать новый вид как “Candidatus Rifleibacterium amylolyticus”. Название рода-кандидата соответствует ранее предложенному названию филума Riflebacteria (Anantharaman et al., 2016) с учетом правил наименования прокариот, а видовое название отражает предполагаемую способность бактерии гидролизовать крахмал.
“Candidatus Rifleibacterium” gen. nov. (Rif.le.i.bac.te'ri.um. N.L. neut. n. bacterium a rod; N.L. neut. n. Rifleibacterium a rod named after Rifle, Colorado).
“Candidatus Rifleibacterium amylolyticum” sp. nov. (a.my.lo.ly'ti,cum. Gr. neut. n. amylon, starch; N.L. masc. adj. lyticus (from Gr. masc. adj. lytikos), able to dissolve; N.L. neut. adj. amylolyticum, degrading starch). Не культивирован. Обнаружен в подземном водоносном горизонте в Западной Сибири. Предположительно является анаэробным органотрофом, получающим энергию за счет ферментации и анаэробного дыхания, может использовать в качестве субстратов крахмал, а также низкомолекулярные углеводы, аминокислоты и пептиды. Содержание Г + Ц в ДНК – 50.1 мол. %. Представлен геномом (GenBank JAACJG000000000), полученным из метагенома подземных термальных вод скважины 5Р (п. Чажемто, Томская область, Россия).
Список литературы
Кадников В.В., Франк Ю.А, Марданов А.В., Белецкий А.В., Ивасенко Д.А., Пименов Н.В., Карначук О.В., Равин Н.В. Некультивируемые бактерии и метаногенные археи доминируют в микробном сообществе подземных вод Западной Сибири // Микробиология. 2017a. Т. 86. С. 383–386.
Kadnikov V.V., Frank Y.A., Mardanov A.V., Beletskii A.V., Ivasenko D.A., Pimenov N.V., Karnachuk O.V., Ravin N.V. Uncultured bacteria and methanogenic archaea predominate in the microbial community of Western Siberian deep subsurface aquifer // Microbiology (Moscow). 2017a. V. 86. P. 412–415.
Кадников В.В., Франк Ю.А, Марданов А.В., Белецкий А.В., Ивасенко Д.А., Пименов Н.В., Карначук О.В., Равин Н.В. Вариабельность состава микробного сообщества резервуара подземных термальных вод в Западной Сибири // Микробиология. 2017b. Т. 86. С. 739–747.
Kadnikov V.V., Frank Y.A., Mardanov A.V., Beletsky A.V., Ivasenko D.A., Pimenov N.V., Karnachuk O.V., Ravin N.V. Variability of the composition of the microbial community of the deep subsurface thermal aquifer in Western Siberia // Microbiology (Moscow). 2017b. V. 86. P. 765–772.
Alneberg J., Bjarnason B.S., De Bruijn I., Schirmer M., Quick J., Ijaz U.Z., Lahti L., Loman N.J., Andersson A.F., Quince C. Binning metagenomic contigs by coverage and composition // Nat. Methods. 2014. V. 11. P. 1144–1146.
Anantharaman K., Brown C.T., Hug L.A., Sharon I., Castelle C.J., Probst A.J., Thomas B.C., Singh A., Wilkins M.J., Karaoz U., Brodie E.L., Williams K.H., Hubbard S.S., Banfield J.F. Thousands of microbial genomes shed light on interconnected biogeochemical processes in an aquifer system // Nat. Commun. 2016. V. 7. P. 13219.
Anantharaman K., Hausmann B., Jungbluth S.P., Kantor R.S., Lavy A., Warren L.A, Rappé M.S., Pester M., Loy A., Thomas B.C., Banfield J.F. Expanded diversity of microbial groups that shape the dissimilatory sulfur cycle // ISME J. 2018. V. 12. P. 1715–1728.
Biegel E., Schmidt S., Gonzalez J.M., Müller V. Biochemistry, evolution and physiological function of the Rnf complex, a novel ion-motive electron transport complex in prokaryotes // Cell. Mol. Life Sci. 2011. V. 68. P. 613–634.
Brown C.T., Olm M.R., Thomas B.C., Banfield J.F. Measurement of bacterial replication rates in microbial communities // Nat. Biotechnol. 2016. V. 34. P. 1256–1263.
Cao M.D., Nguyen S.H., Ganesamoorthy D., Elliott A.G., Cooper M.A., Coin L.J. Scaffolding and completing genome assemblies in real-time with nanopore sequencing // Nat. Commun. 2017. V. 8. P. 14515.
Frank Y.A., Kadnikov V.V., Gavrilov S.N., Banks D., Gerasimchuk A.L., Podosokorskaya O.A., Merkel A.Y., Chernyh N.A., Mardanov A.V., Ravin N.V., Karnachuk O.V., Bonch-Osmolovskaya E.A. Stable and variable parts of microbial community in siberian deep subsurface thermal aquifer system revealed in a long-term monitoring study // Front. Microbiol. 2016. V. 7. P. 2101.
Greening C., Biswas A., Carere C.R., Jackson C.J., Taylor M.C., Stott M.B., Cook G.M., Morales S.E. Genomic and metagenomic surveys of hydrogenase distribution indicate H2 is a widely utilised energy source for microbial growth and survival // ISME J. 2016. V. 10. P. 761–777.
Guindon S., Dufayard J.F., Lefort V., Anisimova M., Hordijk W., Gascuel O. New algorithms and methods to estimate maximum-likelihood phylogenies: assessing the performance of PhyML 3.0 // Syst. Biol. 2010. V. 59. P. 307–321.
Hallenbeck P.C., Clark M.A., Barrett E.L. Characterization of anaerobic sulfite reduction by Salmonella typhimurium and purification of the anaerobically induced sulfite reductase // J. Bacteriol. 1989. V. 171. P. 3008–3015.
Hernsdorf A.W., Amano Y., Miyakawa K., Ise K., Suzuki Y., Anantharaman K., Probst A., Burstein D., Thomas B.C., Banfield J.F. Potential for microbial H2 and metal transformations associated with novel bacteria and archaea in deep terrestrial subsurface sediments // ISME J. 2017. V. 11. P. 1915–1929.
Kadnikov V.V., Frank Y.A., Mardanov A.V., Beletsky A.V., Karnachuk O.V., Ravin N.V. Metagenome of the Siberian underground water reservoir // Genome Announc. 2017c. V. 5. P. e01317-17.
Kadnikov V.V., Mardanov A.V., Beletsky A.V., Banks D., Pimenov N.V., Frank Y.A., Karnachuk O.V., Ravin N.V. A metagenomic window into the 2-km-deep terrestrial subsurface aquifer revealed multiple pathways of organic matter decomposition // FEMS Microbiol. Ecol. 2018. V. 94. P. fiy152.
Konstantinidis K.T., Rosselló-Móra R., Amann R. Uncultivated microbes in need of their own taxonomy // ISME J. 2017. V. 11. P. 2399–2406.
Li H., Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform // Bioinformatics. 2009. V. 25. P. 1754–1760.
Lower B.H., Lins R.D., Oestreicher Z., Straatsma T.P., Hochella M.F.Jr., Shi L., Lower S.K. In vitro evolution of a peptide with a hematite binding motif that may constitute a natural metal-oxide binding archetype // Environ. Sci. Technol. 2008. V. 42. P. 3821–3827.
Malinen A.M., Belogurov G.A., Baykov A.A., Lahti R. Na+-pyrophosphatase: a novel primary sodium pump // Biochemistry. 2007. V. 46. P. 8872–8878.
Meier-Kolthoff J.P., Auch A.F., Klenk H.-P., Göker M. Genome sequence-based species delimitation with confidence intervals and improved distance functions // BMC Bioinform. 2013. V. 14. P. 60.
Nurk S., Meleshko D., Korobeynikov A., Pevzner P.A. metaSPAdes: a new versatile metagenomic assembler // Genome Res. 2017. V. 27. P. 824–834.
Parks D.H., Chuvochina M., Waite D.W., Rinke C., Skarshewski A., Chaumeil P.A., Hugenholtz P. A standardized bacterial taxonomy based on genome phylogeny substantially revises the tree of life // Nat. Biotechnol. 2018. V. 36. P. 996–1004.
Parks D.H., Imelfort M., Skennerton C.T., Hugenholtz P., Tyson G.W. CheckM: assessing the quality of microbial genomes recovered from isolates, single cells, and metagenomes // Genome Res. 2015. V. 25. P. 1043–1055.
Probst A.J., Castelle C.J., Singh A., Brown C.T., Anantharaman K., Sharon I., Hug L.A., Burstein D., Emerson J.B., Thomas B.C., Banfield J.F. Genomic resolution of a cold subsurface aquifer community provides metabolic insights for novel microbes adapted to high CO2 concentrations // Environ. Microbiol. 2017. V. 19. P. 459–474.
Reyes-Prieto A., Barquera B., Juarez O. Origin and evolution of the sodium-pumping NADH: ubiquinone oxidoreductase // PLoS One 2014. V. 9. e96696.
Richter K., Schicklberger M., Gescher J. Dissimilatory reduction of extracellular electron acceptors in anaerobic respiration // Appl. Environ. Microbiol. 2012. V. 78. P. 913–921.
Rodriguez-R L.M., Konstantinidis K.T. The enveomics collection: a toolbox for specialized analyses of microbial genomes and metagenomes // PeerJ Preprints. 2016. V. 4. P. e1900v1.
Schut G.J., Adams M.W. The iron-hydrogenase of Thermotoga maritima utilizes ferredoxin and NADH synergistically: a new perspective on anaerobic hydrogen production // J. Bacteriol. 2009. V. 191. P. 4451–4457.
Shi L., Squier T.C., Zachara J.M., Fredrickson J.K. Respiration of metal (hydr)oxides by Shewanella and Geobacter: a key role for multihaem c-type cytochromes // Mol. Microbiol. 2007. V. 65. P. 12–20.
Søndergaard D., Pedersen C.N., Greening C. HydDB: A web tool for hydrogenase classification and analysis // Sci Rep. 2016. V. 6. P. 34212.
Stewart R.D., Auffret M.D., Warr A., Wiser A.H., Press M.O., Langford K.W., Liachko I., Snelling T.J., Dewhurst R.J., Walker A.W., Roehe R., Watson M. Assembly of 913 microbial genomes from metagenomic sequencing of the cow rumen // Nat. Commun. 2018. V. 9. P. 870.
Дополнительные материалы отсутствуют.


